Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Polysomenreinigung aus Sojabohnen-Symbiose-Knötchen
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt ein Verfahren zur eukaryotischen Polysomenreinigung aus intakten Sojaknollen. Nach der Sequenzierung können Standardpipelines für die Genexpressionsanalyse verwendet werden, um differentiell exprimierte Gene auf Transkriptom- und Translatomebene zu identifizieren.
Zusammenfassung
Das Ziel dieses Protokolls ist es, eine Strategie zur Untersuchung des eukaryotischen Translatoms des Sojabohnen (Glycine max) symbiotischen Knötchens bereitzustellen. Dieser Artikel beschreibt Methoden, die optimiert wurden, um pflanzliche Polyribosomen und die zugehörigen mRNAs zu isolieren, die mittels RNA-Sequenzierung analysiert werden sollen. Zunächst werden zytoplasmatische Lysate durch Homogenisierung unter Polysomen- und RNA-erhaltenden Bedingungen aus ganzen, gefrorenen Sojabohnenknollen gewonnen. Dann werden Lysate durch Zentrifugation mit niedriger Geschwindigkeit entfernt, und 15% des Überstands werden für die Isolierung der Gesamt-RNA (TOTAL) verwendet. Das verbleibende gereinigte Lysat wird verwendet, um Polysomen durch Ultrazentrifugation durch ein zweilagiges Saccharosekissen (12% und 33,5%) zu isolieren. Polysom-assoziierte mRNA (PAR) wird nach der Resuspension aus polysomalen Pellets gereinigt. Sowohl TOTAL als auch PAR werden durch hochempfindliche Kapillarelektrophorese bewertet, um die Qualitätsstandards von Sequenzierungsbibliotheken für RNA-seq zu erfüllen. Als Beispiel für eine nachgeschaltete Anwendung können nach der Sequenzierung Standardpipelines für die Genexpressionsanalyse verwendet werden, um differentiell exprimierte Gene auf Transkriptom- und Translatomebene zu erhalten. Zusammenfassend lässt sich sagen, dass diese Methode in Kombination mit RNA-seq die Untersuchung der translationalen Regulation eukaryotischer mRNAs in einem komplexen Gewebe wie dem symbiotischen Knötchen ermöglicht.
Einleitung
Leguminosen wie Sojabohnen (Glycine max) können eine Symbiose mit bestimmten Bodenbakterien namens Rhizobien eingehen. Diese mutualistische Beziehung führt zur Bildung neuer Organe, der symbiotischen Knötchen, an den Pflanzenwurzeln. Die Knötchen sind die Pflanzenorgane, die die Bakterien beherbergen, und bestehen aus Wirtszellen, deren Zytoplasma mit einer spezialisierten Form von Rhizobien besiedelt ist, die Bakterioide genannt wird. Diese Bakterien katalysieren die Reduktion von Luftstickstoff (N2) zu Ammoniak, das im Gegenzug für Kohlenhydrate 1,2 auf die Pflanze übertragen wird.
Obwohl diese stickstofffixierende Symbiose eine der am besten untersuchten Pflanzen-Mikroben-Symbiosen ist, müssen noch viele Aspekte besser verstanden werden, wie zum Beispiel, wie Pflanzen, die verschiedenen abiotischen Stressbedingungen ausgesetzt sind, ihre Interaktion mit ihrem symbiotischen Partner modulieren und wie sich dies auf den Knötchenstoffwechsel auswirkt. Diese Prozesse könnten besser verstanden werden, indem das Knötchentranslatom (d.h. die Untergruppe der Boten-RNAs [mRNAs] aktiv translatiert) analysiert wird. Polyribosomen oder Polysomen sind Komplexe mehrerer Ribosomen, die mit mRNA assoziiert sind und häufig zur Untersuchung der Translationverwendet werden 3. Die Polysomen-Profiling-Methode besteht aus der Analyse der mRNAs, die mit Polysomen assoziiert sind, und wurde erfolgreich eingesetzt, um die posttranskriptionellen Mechanismen zu untersuchen, die die Genexpression steuern, die in verschiedenen biologischen Prozessen auftritt 4,5.
In der Vergangenheit konzentrierte sich die Genomexpressionsanalyse hauptsächlich auf die Bestimmung der mRNA-Häufigkeit 6,7,8,9. Es besteht jedoch ein Mangel an Korrelation zwischen Transkript- und Proteinspiegeln aufgrund der verschiedenen Stadien der posttranskriptionellen Regulation der Genexpression, insbesondere der Translation10,11,12. Darüber hinaus wurde keine Abhängigkeit zwischen den Veränderungen auf der Ebene des Transkriptoms und denen, die auf der Ebene des Translatoms13 auftreten, beobachtet. Die direkte Analyse des Satzes von mRNAs, die übersetzt werden, ermöglicht eine genauere und vollständigere Messung der Zellgenexpression (deren Endpunkt die Proteinhäufigkeit ist) als die, die erhalten wird, wenn nur mRNA-Spiegel analysiert werden14,15,16.
Dieses Protokoll beschreibt, wie pflanzliche Polysomen aus intakten Sojaknollen durch differentielle Zentrifugation durch ein zweischichtiges Saccharosekissen gereinigt werden (Abbildung 1). Da jedoch auch Bacteroid-abgeleitete Ribosomen in den Knötchen vorhanden sind, wird eine Mischung aus Ribosomen und RNA-Spezies gereinigt, obwohl die eukaryotischen die Hauptfraktion darstellen (90%-95%). Die anschließende RNA-Isolierung, Quantifizierung und Qualitätskontrolle wird ebenfalls beschrieben (Abbildung 1). Dieses Protokoll soll in Kombination mit RNA-seq experimentelle Ergebnisse zur translationalen Regulation eukaryotischer mRNAs in einem komplexen Gewebe wie dem symbiotischen Knötchen liefern.
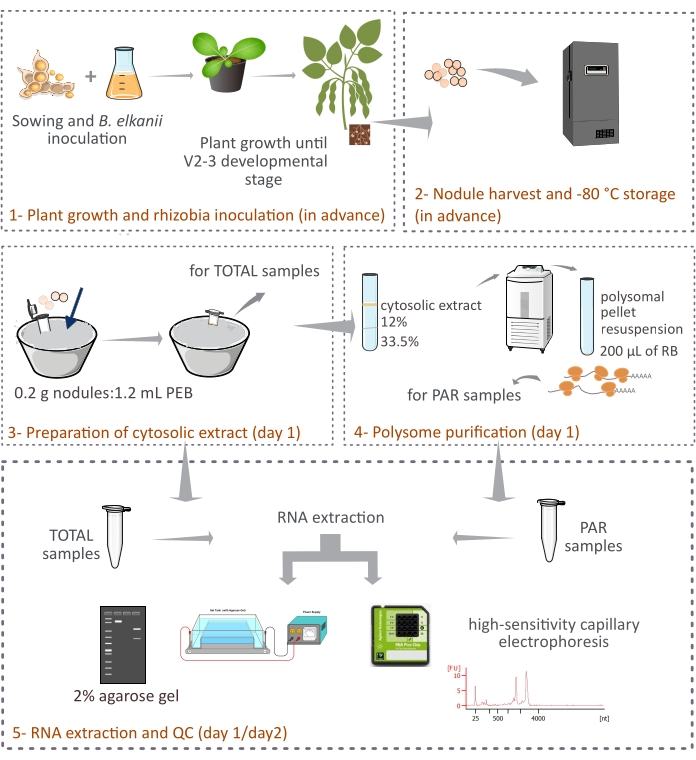
Abbildung 1: Schematische Übersicht über die vorgeschlagene Methodik zur eukaryotischen Polysomenreinigung aus symbiotischen Knötchen. Das Schema gibt einen Überblick über die Schritte, die im Protokoll von (1) Pflanzenwachstum und (2) Knötchenernte bis (3) Herstellung der zytosolischen Extrakte, (3) Gewinnung von GESAMTPROBEN und (4) PAR-Proben und (5) RNA-Extraktion und Qualitätskontrolle befolgt werden. Abkürzungen: PEB = Polysomenextraktionspuffer; RB = Resuspensionspuffer; TOTAL = Gesamt-RNA; PAR = Polysom-assoziierte mRNA. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Protokoll
1. Pflanzenwachstum und Rhizobienimpfung
- Um die benötigten Knötchen zu erzeugen, säen Sie die Sojabohnensamen Ihrer Wahl in das ausgewählte Substrat in einer Wachstumskammer unter kontrollierten Bedingungen.
HINWEIS: In diesem Protokoll wurden Samen in einer 0,5-l-Plastikflasche ausgesät, die mit einer Mischung aus Sand:Vermiculit (1:1) gefüllt war. Die Wachstumskammer wurde mit einer Tag/Nacht-Zyklustemperatur von 28 °C bzw. 20 °C bzw. einer Hell/Dunkel-Photoperiode von 16 h/8 h eingestellt. Die photosynthetisch aktive Strahlungsintensität betrug 620 μmol·m−2·s−1. - Bereiten Sie im Voraus flüssiges Hefe-Extrakt Mannitol (YEM)-medium vor (siehe Materialtabelle). Autoklavieren Sie das Medium.
- Am selben Tag der Saatgutaussaat wird ein Kolben mit 100 ml YEM mit dem Stamm Bradyrhizobium elkanii U1302 beimpft. Der Kolben wird bei 28 °C auf einem Orbitalschüttler bei 100 U/min inkubiert.
- Lassen Sie die Rhizobien 2 Tage wachsen und impfen Sie die Sämlinge mit 2 ml der Kultur.
2. Wassermangelbehandlung (optional)
HINWEIS: Dieses Protokoll beschreibt die Wassermangelbehandlung der Sojabohnenpflanzen. Dieser Teil kann je nach experimenteller Fragestellung geändert oder ganz weggelassen werden.
- Wachsen Sie die Sojabohnensämlinge für 19 Tage (V2-3-Entwicklungsstadium) ohne Wasserbeschränkung. Halten Sie das Substrat mit B&D-medium17 , ergänzt mit 0,5 mM KNO3, auf Feldkapazität.
- Am 20. Tag ziehen Sie die Bewässerung zurück.
HINWEIS: Hier wurde der Wassergehalt täglich durch Gravimetrie (gravimetrischer Wassergehalt) während der Wachstums- und Wasserdefizitperioden gemessen. - Messen Sie die Stomaleitfähigkeit in dreifacher Ausfertigung täglich auf der abaxialen Blattoberfläche mit einem Porometer, wie vom Hersteller angewiesen (siehe Materialtabelle), vom 20. Tag bis zum Ende der Wasserdefizitperiode.
ANMERKUNG: Das Ende der Wasserdefizitperiode wurde für jede Pflanze individuell bestimmt, wenn der Stomatalleitwert etwa 50% des am Tag 20 aus der Aussaat erhaltenen Wertes betrug.
3. Knötchenernte
- Sammeln Sie flüssigen Stickstoff in einem sauberen Behälter und kennzeichnen Sie 15 ml Röhrchen (eines für jede Pflanze).
VORSICHT: Behandeln Sie flüssigen Stickstoff mit Kryohandschuhen, Gesichtsschilden und Schutzbrillen. - Rufen Sie jede Pflanze einzeln ab. Schneiden Sie den oberirdischen Teil aus und werfen Sie ihn weg. Waschen Sie die Wurzel gründlich mit Wasser, um das restliche Substrat zu entfernen.
- Lösen Sie jeden Knoten von der Wurzel und sammeln Sie ihn in einem vorgekühlten 15-ml-Röhrchen. Die Röhrchen einfrieren und bei −80 °C lagern.
4. Herstellung von zytosolischen Extrakten
HINWEIS: Das Endziel dieses Protokolls ist es, qualitativ hochwertige Gesamt-RNA (TOTAL) und Polysom-assoziierte RNA (PAR) zu erhalten. Arbeiten Sie daher unter Bedingungen, die einen RNA-Abbau verhindern, halten Sie die Proben immer bei 4 °C und verwenden Sie RNase-freie Laborgeräte und -lösungen. Sofern nicht anders angegeben, werden alle Lösungen mit sterilem Reinstwasser hergestellt.
- Vorbereitung von Pufferlagerlösungen
- Bereiten Sie die in Tabelle 1 aufgeführten autoklavierbaren Stammlösungen vor, autoklavieren Sie sie 15 Minuten lang und bewahren Sie sie bei RT auf.
- Die in Tabelle 1 aufgeführten filtersterilisierbaren Stammlösungen werden vorbereitet, filtriert und bei −20 °C aufbewahrt.
- Bereiten Sie den Polysomenextraktionspuffer (PEB, siehe Tabelle 1) vor und halten Sie ihn auf Eis.
ANMERKUNG: Die angegebenen Volumina beziehen sich auf sechs Proben. - Kühlen Sie die Zentrifuge auf 4 °C ab und legen Sie 2 mL Mikrozentrifugenröhrchen auf Eis.
- Wiegen Sie ca. 0,2 g intakte Knötchen in einer Waagschale und geben Sie sie in ein vorgekühltes 2-ml-Röhrchen.
HINWEIS: Führen Sie diesen Schritt schnell durch, um das Auftauen der Proben zu vermeiden. Alternativ kann anstelle der 0,2 g ein geschätztes Knötchenvolumen von 0,4-0,5 ml verwendet werden. - Fügen Sie 1,2 ml PEB hinzu, lassen Sie die Probe 2 min auftauen und homogenisieren Sie mit einem Gewebeschleifer bis zur vollständigen Auflösung und Homogenisierung der Knötchen.
HINWEIS: Achten Sie darauf, die Knötchen nach dem Auftauen zu mahlen, da sie weich genug sind, um leicht mit Gewebeschleifern (siehe Materialtabelle) in Mikrozentrifugenröhrchen gestört zu werden. Gefrorene Knötchen sind schwer zu schleifen. Außerdem wird empfohlen, die Röhrchen in einem Tischkühler (0 °C) zu platzieren, um eine ordnungsgemäße Homogenisierung zu gewährleisten und die Proben kühl zu halten. - Inkubieren Sie die Proben auf Eis mit leichtem Rühren für 10 min oder bis alle Proben verarbeitet wurden.
- Bei 16.000 × g 15 min bei 4 °C zentrifugieren, um die Ablagerungen zu pelletieren. Gewinnen Sie den Überstand zurück und wiederholen Sie den Zentrifugenschritt.
- Der geklärte zytosolische Extrakt wird vorsichtig zurückgewonnen und ein Aliquot von 200 μL zur Isolierung in ein sauberes 1,5-ml-Mikrozentrifugenröhrchen überführt (TOTAL-Proben; siehe Abschnitt 7).
5. Herstellung von Saccharosekissen
HINWEIS: Dieses Protokoll verwendet ein zweilagiges Saccharosekissen (12% und 33,5%) in 13,2-ml-Ultrazentrifugenröhrchen (siehe Materialtabelle). Alle Lösungen werden mit sterilem Reinstwasser hergestellt.
- Bereiten Sie die Saccharose- und Salzbrühlösungen vor.
- Bereiten Sie 100 ml 2 M Saccharoselösung vor (68,5%, Tabelle 1).
- 10x Salzlösung vorbereiten (Tabelle 1).
- Bereiten Sie die beiden Schichten des Saccharosekissens wie in Tabelle 2 beschrieben vor. Siehe Tabelle 1 für die Zusammensetzung der antibiotischen (CHX und CHL) Stammlösungen.
HINWEIS: Die angegebenen Bände gelten für sechs Kissen. - Gießen Sie 4,5 ml der 33,5% igen Saccharoseschicht in die Zentrifugenröhrchen. Fügen Sie dann 4,5 ml der 12% igen Schicht vorsichtig und langsam mit einer P1000-Mikropipette hinzu. Die Röhrchen bis zur Zugabe des geklärten zytosolischen Extrakts auf Eis legen.
6. Polysomenreinigung
- Laden Sie 1 ml des geklärten zytosolischen Extrakts (Schritt 4.8) auf das Saccharosekissen, indem Sie vorsichtig auf die Seitenwand des Röhrchens pipettieren.
- Die Ultrazentrifugenröhrchen werden in vorgekühlte Eimer überführt und bei 217.874 × g (35.000 U/min im referenzierten Rotor [siehe Materialtabelle]) für 2 h bei 4 °C zentrifugiert.
- Der verbleibende zytosolische Extrakt und das Saccharosepolster sind zu verwerfen und das polysomale Pellet mit 200 μL vorgekühltem RB (zuvor gemäß Tabelle 1 hergestellt) zu resuspendieren.
- Inkubieren Sie für 30 min auf Eis und geben Sie dann die polysomale Resuspension in 1,5 ml vorgekühlte Röhrchen. Fahren Sie mit der RNA-Extraktion (PAR-Proben) fort.
7. RNA-Extraktion und Qualitätskontrolle
HINWEIS: Dieser Schritt wird für TOTAL (Schritt 4.8) und PAR-Proben (Schritt 6.4) ausgeführt.
- Bereiten Sie 75% EtOH mit sterilem Reinstwasser vor und kühlen Sie es auf −20 °C ab. Zusätzlich Chloroform und Isopropanol auf −20 °C abkühlen.
- Die Proben werden mit 750 μL des RNA-Isolierreagenz homogenisiert und 5 min bei RT inkubiert.
- Fügen Sie 200 μL kaltes Chloroform hinzu und schütteln Sie die Röhrchen kräftig für 15 s. Inkubieren bei RT für 10 min.
- Zentrifugieren bei 12.000 × g für 15 min bei 4 °C zur Phasentrennung. 500 μL der oberen wässrigen Phase in ein sauberes Röhrchen überführen, ohne die rosa organische Phase zu stören, und 375 μL kaltes Isopropanol und 0,5 μL RNase-freies Glykogen hinzufügen (siehe Materialtabelle). Gründlich mischen, indem Sie auf und ab pipettieren.
HINWEIS: Glykogen ist ein Träger, der als Co-Präzipitant verwendet wird, um die Nukleinsäureerholung aus Alkoholausfällung zu erhöhen. - Die Mischung 10 min bei 4 °C inkubieren und bei 12.000 × g 15 min zentrifugieren. Verwerfen Sie den Überstand.
- Waschen Sie den RNA-Niederschlag mit 1 ml kaltem 75% EtOH. Durch kurzes Wirbeln mischen.
HINWEIS: Zu diesem Zeitpunkt können die Proben über Nacht bei −20 °C gelagert werden. - Bei 7.500 × g 5 min bei 4 °C zentrifugieren, den Überstand vorsichtig mit einer Mikropipette entfernen und das RNA-Pellet an der Luft trocknen.
- Das Pellet wird in 50 μL RNasefreiem Wasser gelöst und bei 65 °C für 5 min inkubiert.
- Beurteilung der RNA-Konzentration und -Integrität mit hochempfindlicher Kapillarelektrophorese18 (siehe Materialtabelle) und/oder Elektrophorese an einem 2% RNase-freien Agarosegel19 (siehe Materialtabelle).
HINWEIS: Wenn die RNA-Proben nicht sofort verwendet oder zur Vorbereitung der RNA-seq-Bibliothek an die Sequenzierungsanlage geschickt werden, wird empfohlen, EtOH zu verwenden, um sie auszufällen.
8. RNA-Fällung
- Zentrifuge und EtOH auf 4 °C abkühlen.
- Bereiten Sie 3 M Natriumacetat vor.
- 70% EtOH mit sterilem Reinstwasser zubereiten und auf −20 °C abkühlen.
- Schätzen Sie das Probenvolumen. Fügen Sie 0,1 Volumen 3 M Natriumacetat, 3 Volumen kaltes EtOH und 0,5 μL RNase-freies Glykogen hinzu. Gründlich mischen.
- Bei −20 °C belassen, bis sie benötigt werden.
HINWEIS: RNA-gefällte Proben können bis zu 1 Jahr bei −80 °C gelagert werden.
9. Standard-Pipeline für die Genexpressionsanalyse
- Führen Sie durchschnittlich 40 Mio. Illumina-Paired-End-Lesevorgänge mit einer Leselänge >100 bp durch, um zuverlässige Transkriptom- und Translatomdatenanalysen durchzuführen.
HINWEIS: Die Standard-RNA-seq-Datenanalyse umfasst die folgenden Schritte 9.2-9.5. - Führen Sie eine Lesequalitätsprüfung mit FastQC (https://www.bioinformatics.babraham.ac.uk/projects/fastqc/) oder dem Multisample MultiQC20 durch.
- Entfernen Sie Adapter und minderwertige Sequenzen unter anderem mit Trimmomatic21, BBDuk (http://jgi.doe.gov/data-and-tools/bb-tools/), cutadapt22, Sickle23 oder Trim Galore (https://www.bioinformatics.babraham.ac.uk/projects/trim_galore/).
- Wenn ein Referenzgenom für die Art verfügbar ist, führen Sie eine Lesezuordnung gegen die Referenz durch, gefolgt von einer Quantifizierung der Abundanz mit Bowtie224, TopHat2 25 oder Salmon 26 und featureCounts27, neben anderen Open-Source-Tools.
- Führen Sie statistische Analysen zur Identifizierung differentiell exprimierter Gene durch, unter anderem mit edgeR28, DESeq229 oder limma30.
HINWEIS: Diese Open-Source-Software kann lokal über die Befehlszeile verwendet werden. Alternativ können sie über einen Webbrowser auf einem öffentlichen Server wie Galaxy31 oder GEOexplorer32 betrieben werden, die eine grafische Benutzeroberfläche bieten, so dass für ihre Nutzung keine Kommandozeilenkenntnisse erforderlich sind.
Ergebnisse
Die Quantitäts- und Qualitätsbewertung der mit dem oben genannten Verfahren gereinigten TOTAL- und PAR-Fraktionen ist entscheidend für den Erfolg, da für die meisten nachgelagerten Anwendungen, wie z. B. die RNA-Sequenzierung, qualitativ hochwertige Proben für die Bibliotheksvorbereitung und -sequenzierung von grundlegender Bedeutung sind. Darüber hinaus ermöglicht die Integrität der RNA-Moleküle die Erfassung einer Momentaufnahme des Genexpressionsprofils zum Zeitpunkt der Probenentnahme18
Diskussion
Die Untersuchung der Genexpressionsregulation auf translationaler Ebene ist entscheidend, um verschiedene biologische Prozesse besser zu verstehen, da der Endpunkt der Zellgenexpression die Proteinhäufigkeit13,14 ist. Dies kann beurteilt werden, indem das Translatom des interessierenden Gewebes oder Organismus analysiert wird, für das die polysomale Fraktion gereinigt und die zugehörigen mRNAs analysiert werden sollen 3,4,34,35,36....
Offenlegungen
Die Autoren haben keine Interessenkonflikte.
Danksagungen
Diese Forschung wurde durch CSIC I+D 2020 Grant Nr. 282, FVF 2017 Grant Nr. 210 und PEDECIBA (María Martha Sainz) finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Plant growth and rhizobia inoculation | |||
| Orbital shaker | Daihan Scientific | Model SHO-1D | |
| YEM-medium | Amresco | J850 (yeast extract) 0122 (mannitol) | |
| Water deficit treatment | |||
| KNO3 | Merck | 221295 | |
| Porometer | Decagon Device | Model SC-1 | |
| Scalpel | |||
| Preparation of cytosolic extracts | |||
| Brij L23 | Sigma-Aldrich | P1254 | |
| Centrifuge | Sigma | Model 2K15 | |
| Chloranphenicol | Sigma-Aldrich | C0378 | |
| Cycloheximide | Sigma-Aldrich | C7698 | |
| DOC | Sigma-Aldrich | 30970 | |
| DTT | Sigma-Aldrich | D9779 | |
| EGTA | Sigma-Aldrich | E3889 | |
| Igepal CA 360 | Sigma-Aldrich | I8896 | |
| KCl | Merck | 1.04936 | |
| MgCl2 | Sigma-Aldrich | M8266 | |
| Plastic tissue grinder | Fisher Scientific | 12649595 | |
| PMSF | Sigma-Aldrich | P7626 | |
| PTE | Sigma-Aldrich | P2393 | |
| Tris | Invitrogen | 15504-020 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween 20 | Sigma-Aldrich | P1379 | |
| Weighing dish | Deltalab | 1911103 | |
| Preparation of sucrose cushions | |||
| Sucrose | Invitrogen | 15503022 | |
| SW 40 Ti rotor | Beckman-Coulter | ||
| Ultracentrifuge | Beckman-Coulter | Optima L-100K | |
| Ultracentrifuge tubes | Beckman-Coulter | 344059 | 13.2 mL tubes |
| RNA extraction and quality control | |||
| Agarose | Thermo scientific | R0492 | |
| Bioanalyzer | Agilent | Model 2100. Eukaryote total RNA nano assay | |
| Chloroform | DI | 41191 | |
| Ethanol | Dorwil | UN1170 | |
| Isopropanol | Mallinckrodt | 3032-06 | |
| Glycogen | Sigma | 10814-010 | |
| TRIzol LS | Ambion | 102960028 | |
| Miscellaneous | |||
| Falcon tubes 15 mL | Biologix | 10-0152 | |
| Filter tips 10 µL | BioPointe Scientific | 321-4050 | |
| Filter tips 1000 µL | BioPointe Scientific | 361-1050 | |
| Filter tips 20 µL | BioPointe Scientific | 341-4050 | |
| Filter tips 200 µL | Tarsons | 528104 | |
| Microcentrifuge tubes 1.5 mL | Tarsons | 500010-N | |
| Microcentrifuge tubes 2.0 mL | Tarsons | 500020-N | |
| Sequencing company | Macrogen | ||
| Sterile 250 mL flask | Marienfeld | 4110207 |
Referenzen
- Limpens, E., et al. Cell- and tissue-specific transcriptome analyses of Medicago truncatula root nodules. PLoS ONE. 8 (5), 64377 (2013).
- Masson-Boivin, C., Giraud, E., Perret, X., Batut, J. Establishing nitrogen-fixing symbiosis with legumes: How many rhizobium recipes. Trends in Microbiology. 17 (10), 458-466 (2009).
- King, H. A., Gerber, A. P. Translatome profiling: Methods for genome-scale analysis of mRNA translation. Briefings in Functional Genomics. 15 (1), 22-31 (2014).
- Chassé, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), 15 (2017).
- Yángüez, E., Castro-Sanz, A. B., Fernández-Bautista, N., Oliveros, J. C., Castellano, M. M. Analysis of genome-wide changes in the translatome of Arabidopsis seedlings subjected to heat stress. PloS ONE. 8 (8), 71425 (2013).
- Brown, P. O., Botstein, D. Exploring the new world of the genome with DNA microarrays. Nature Genetics. 21, 33-37 (1999).
- Krishnamurthy, A., Ferl, R. J., Paul, A. -. L. Comparing RNA-Seq and microarray gene expression data in two zones of the Arabidopsis root apex relevant to spaceflight. Applications in Plant Sciences. 6 (11), 1197 (2018).
- Shulse, C. N., et al. High-throughput single-cell transcriptome profiling of plant cell types. Cell Reports. 27 (7), 2241-2247 (2019).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: A revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Kawaguchi, R., Girke, T., Bray, E. A., Bailey-Serres, J. Differential mRNA translation contributes to gene regulation under non-stress and dehydration stress conditions in Arabidopsis thaliana. The Plant Journal. 38 (5), 823-839 (2004).
- Larsson, O., Tian, B., Sonenberg, N. Toward a genome-wide landscape of translational control. Cold Spring Harbor Perspectives in Biology. 5 (1), 012302 (2013).
- Vogel, C., Marcotte, E. M. Insights into regulation of protein abundance from proteomics and transcriptomis analyses. Nature Reviews Genetics. 13 (4), 227-232 (2013).
- Tebaldi, T., et al. Widespread uncoupling between transcriptome and translatome variations after a stimulus in mammalian cells. BMC Genomics. 13 (220), 1-15 (2012).
- Wang, T., et al. Translating mRNAs strongly correlate to proteins in a multivariate manner and their translation ratios are phenotype specific. Nucleic Acids Research. 41 (9), 4743-4754 (2013).
- Ingolia, N. T. Ribosome profiling: New views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Lukoszek, R., Feist, P., Ignatova, Z. Insights into the adaptive response of Arabidopsis thaliana to prolonged thermal stress by ribosomal profiling and RNA-Seq. BMC Plant Biology. 16 (1), 1-13 (2016).
- Broughton, W. J., Dilworth, M. J. Control of leghaemoglobin synthesis in snake beans. Biochemical Journal. 125 (4), 1075-1080 (1971).
- Schroeder, A., et al. The RIN: An RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 1-14 (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing agarose gel electrophoresis of RNA nondenaturing agarose gel electrophoresis of RNA. Cold Spring Harbor Protocols. 2010 (6), 1-3 (2012).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: Summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Marcel, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- . Sickle: A sliding-window, adaptive, quality-based trimming tool for FastQ files (Version 1.33) Available from: https://github.com/najoshi/sickle (2011)
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Kim, D., et al. TopHat2: Accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biology. 14 (4), 1-13 (2013).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Liao, Y., Smyth, G. K., Shi, W. featureCounts: An efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30 (7), 923-930 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: A Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (550), 1-21 (2014).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2015).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Hunt, G. P., et al. GEOexplorer: A webserver for gene expression analysis and visualisation. Nucleic Acids Research. , (2022).
- Imbeaud, S., et al. Towards standardization of RNA quality assessment using user-independent classifiers of microcapillary electrophoresis traces. Nucleic Acids Research. 33 (6), 1-12 (2005).
- Di Paolo, A., et al. PDCD4 regulates axonal growth by translational repression of neurite growth-related genes and is modulated during nerve injury responses. RNA. 26 (11), 1637-1653 (2020).
- Smircich, P., et al. Ribosome profiling reveals translation control as a key mechanism generating differential gene expression in Trypanosoma cruzi. BMC Genomics. 16 (1), 443 (2015).
- Eastman, G., Sharlow, E. R., Lazo, J. S., Bloom, G. S., Sotelo-Silveira, J. R. Transcriptome and translatome regulation of pathogenesis in Alzheimer's disease model mice. Journal of Alzheimer's Disease. 86 (1), 365-386 (2022).
- Zanetti, M. E., Chang, I. -. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification of polyribosomal complexes of Arabidopsis for global analysis of gene expression. Plant physiology. 138 (2), 624-635 (2005).
- Mustroph, A., et al. Profiling translatomes of discrete cell populations resolves altered cellular priorities during hypoxia in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18843-11848 (2009).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature Reviews. Molecular Cell Biology. 16 (11), 651-664 (2015).
- Eastman, G., Smircich, P., Sotelo-Silveira, J. R. Following ribosome footprints to understand translation at a genome wide level. Computational and Structural Biotechnology Journal. 16, 167-176 (2018).
- Westermann, A. J., Gorski, S. A., Vogel, J. Dual RNA-seq of pathogen and host. Nature Reviews Microbiology. 10 (9), 618-630 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten