A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
טיהור פוליזום מגושים סימביוטיים של פולי סויה
In This Article
Summary
פרוטוקול זה מתאר שיטה לטיהור פוליזום אאוקריוטי מגושי סויה שלמים. לאחר הריצוף, ניתן להשתמש בצינורות סטנדרטיים לניתוח ביטוי גנים כדי לזהות גנים המתבטאים באופן דיפרנציאלי ברמות השעתוק והטרנסלטום.
Abstract
מטרת פרוטוקול זה היא לספק אסטרטגיה לחקר הטרנסלטום האאוקריוטי של הגולם הסימביוטי של פולי הסויה (גליצין מקס). מאמר זה מתאר שיטות הממוטבות לבידוד פוליריבוזומים שמקורם בצמחים ואת ה-mRNA הקשורים אליהם לניתוח באמצעות ריצוף RNA. ראשית, ליזאטים ציטופלסמיים מתקבלים באמצעות הומוגניזציה בתנאים משמרים פוליזום ורנ"א מגושים שלמים וקפואים של פולי סויה. לאחר מכן, הליזטים מנוקים על ידי צנטריפוגה במהירות נמוכה, ו-15% מהסופר-נאטנט משמש לבידוד RNA כולל (TOTAL). הליזאט המנוקה הנותר משמש לבידוד פוליזומים על ידי אולטרה-צנטריפוגציה באמצעות כרית סוכרוז דו-שכבתית (12% ו-33.5%). mRNA הקשור לפוליזום (PAR) מטוהר מכדורים פוליסומליים לאחר חידוש. הן TOTAL והן PAR מוערכים על ידי אלקטרופורזה נימית רגישה ביותר כדי לעמוד בתקני האיכות של ספריות ריצוף עבור RNA-seq. כדוגמה ליישום במורד הזרם, לאחר ריצוף, ניתן להשתמש בצינורות סטנדרטיים לניתוח ביטוי גנים כדי לקבל גנים המתבטאים באופן דיפרנציאלי ברמות השעתוק והטרנסלטום. לסיכום, שיטה זו, בשילוב עם RNA-seq, מאפשרת לחקור את הוויסות התרגומי של mRNA אאוקריוטי ברקמה מורכבת כגון הגולם הסימביוטי.
Introduction
צמחים קטניים, כגון סויה (גליצין מקס), יכולים לבסס סימביוזה עם חיידקי אדמה ספציפיים הנקראים ריזוביה. מערכת יחסים הדדית זו מעוררת את היווצרותם של איברים חדשים, הגושים הסימביוטיים, על שורשי הצמח. הגושים הם האיברים הצמחיים המארחים את החיידקים ומורכבים מתאים מארחים שהציטופלסמה שלהם מיושבת עם צורה מיוחדת של ריזוביה הנקראת חיידקים. חיידקים אלה מזרזים את הפחתת החנקן האטמוספרי (N 2) לאמוניה, אשר מועבר לצמח בתמורה לפחמימות 1,2.
אף על פי שסימביוזה זו של קיבוע חנקן היא אחת הסימביוזה בין צמחים למיקרובים הנחקרים ביותר, היבטים רבים עדיין צריכים להיות מובנים טוב יותר, כגון כיצד צמחים הנתונים לתנאי עקה אביוטיים שונים מווסתים את האינטראקציה שלהם עם השותף הסימביוטי שלהם וכיצד זה משפיע על חילוף החומרים של הגולם. ניתן היה להבין טוב יותר תהליכים אלה על ידי ניתוח טרנסלטום הגולם (כלומר, תת-הקבוצה של רנ"א שליח [mRNAs] המתורגמת באופן פעיל). פוליריבוזומים או פוליזומים הם קומפלקסים של ריבוזומים מרובים הקשורים ל-mRNA, המשמשים בדרך כלל לחקר תרגום3. שיטת פרופיל הפוליזום מורכבת מניתוח ה-mRNA הקשורים לפוליזומים ושימשה בהצלחה לחקר המנגנונים הפוסט-טרנספורמטיביים השולטים בביטוי גנים המתרחשים בתהליכים ביולוגיים מגוונים 4,5.
מבחינה היסטורית, ניתוח ביטוי הגנום התמקד בעיקר בקביעת שפע mRNA 6,7,8,9. עם זאת, קיים חוסר מתאם בין רמות התעתיק לרמות החלבון בשל השלבים השונים של ויסות פוסט-טרנספורמטיבי של ביטוי גנים, במיוחד תרגום10,11,12. יתר על כן, לא נצפתה תלות בין השינויים ברמת התעתיק לבין אלה המתרחשים ברמה של translatome13. הניתוח הישיר של קבוצת ה-mRNA המתורגמת מאפשר מדידה מדויקת ומלאה יותר של ביטוי הגנים בתאים (שנקודת הקצה שלהם היא שפע חלבונים) מזו המתקבלת כאשר מנותחות רק רמות mRNA14,15,16.
פרוטוקול זה מתאר כיצד פוליזומים ממקור צמחי מטוהרים מגושי סויה שלמים על-ידי צנטריפוגה דיפרנציאלית באמצעות כרית סוכרוז דו-שכבתית (איור 1). עם זאת, מכיוון שריבוזומים שמקורם בבקטרואיד נמצאים גם בגושים, תערובת של ריבוזומים ומיני RNA מטוהרים, למרות שהאאוקריוטים מייצגים את החלק העיקרי (90%-95%). כמו כן מתוארים הבידוד, הכימות ובקרת האיכות של הרנ"א הבאים (איור 1). פרוטוקול זה, בשילוב עם RNA-seq, אמור לספק תוצאות ניסיוניות על הוויסות התרגומי של mRNA אאוקריוטי ברקמה מורכבת כגון הגולם הסימביוטי.
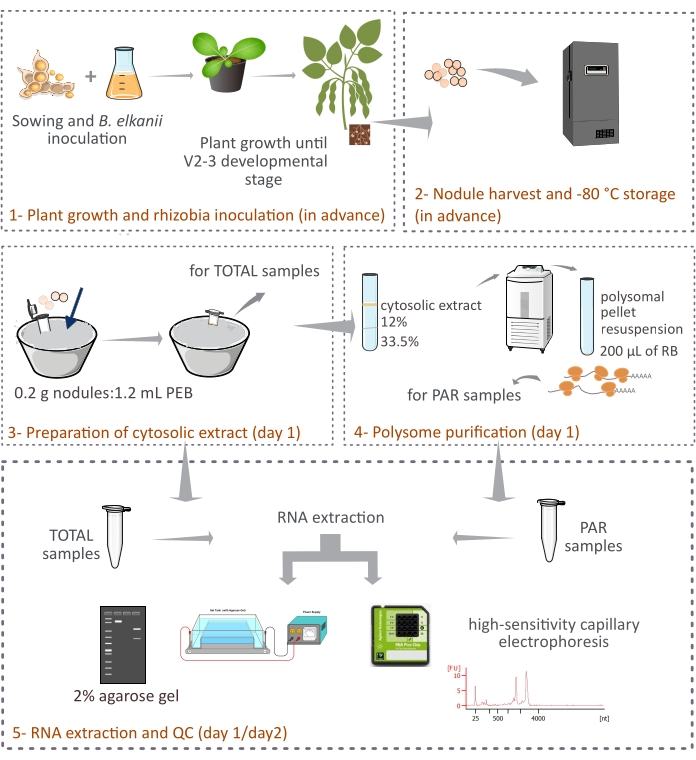
איור 1: סקירה סכמטית של המתודולוגיה המוצעת לטיהור פוליזום אאוקריוטי מגושים סימביוטיים. התוכנית נותנת סקירה של השלבים הבאים בפרוטוקול מ-(1) גידול צמחים ו-(2) קציר גולמים ועד (3) הכנת התמציות הציטוזוליות, (3) קבלת דגימות TOTAL ו-(4) דגימות PAR, ו-(5) מיצוי RNA ובקרת איכות. קיצורים: PEB = מאגר מיצוי פוליזום; RB = חיץ resuspension; סה"כ = סך רנ"א; PAR = mRNA הקשור לפוליזום. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
Protocol
1. גידול צמחים וחיסון ריזוביה
- כדי ליצור את הגושים הנדרשים, לזרוע את זרעי סויה של בחירה במצע שנבחר בתא הגידול בתנאים מבוקרים.
הערה: בפרוטוקול זה, זרעים נזרעו בבקבוק פלסטיק 0.5 ליטר מלא בתערובת של חול:ורמיקוליט (1:1). תא הגידול נקבע עם טמפרטורת מחזור יום / לילה של 28 °C (75 °F) / 20 ° C, בהתאמה, ו photoperiod אור / חושך של 16 h / 8 h, בהתאמה. עוצמת הקרינה הפעילה מבחינה פוטוסינתטית הייתה 620 μmol·m−2·s−1. - הכינו מראש תמצית שמרים נוזלית מניטול (YEM)-בינונית (ראו טבלת חומרים). אוטוקלאב את המדיום.
- באותו יום של זריעת זרעים, לחסן בקבוק אחד המכיל 100 מ"ל של YEM עם זן Bradyrhizobium elkanii U1302. דגירה של הבקבוקון ב-28 מעלות צלזיוס על שייקר מסלולי ב-100 סל"ד.
- תן ריזוביה לגדול במשך 2 ימים לחסן את השתילים עם 2 מ"ל של התרבות.
2. טיפול בגירעון מים (אופציונלי)
הערה: פרוטוקול זה מתאר את הטיפול בגירעון המים של צמחי הסויה. חלק זה ניתן לשינוי או להשמטה לחלוטין בהתאם לשאלה הניסויית העומדת על הפרק.
- לגדל את שתילי סויה במשך 19 ימים (V2-3 שלב התפתחותי) ללא הגבלת מים. שמור על המצע בקיבולת שדה עם B&D-בינוני17 בתוספת 0.5 mM KNO3.
- ביום 20, למשוך השקיה.
הערה: כאן, תכולת המים נמדדה מדי יום על ידי גרבימטריה (תכולת גרבימטרי במים) במהלך תקופות הגידול והגירעון במים. - יש למדוד את המוליכות הסטומטית במשולש מדי יום על פני העלה האבקסיאלי באמצעות פורומטר, בהתאם להוראות היצרן (ראו טבלת חומרים), מהיום ה-20 ועד תום תקופת גירעון המים.
הערה: סוף תקופת הגירעון במים נקבע בנפרד עבור כל צמח כאשר ערך ההולכה הסטומטית היה כ-50% מזה שהתקבל ביום 20 מהזריעה.
3. קציר גולם
- אסוף חנקן נוזלי במיכל נקי ותייג צינורות של 15 מ"ל (אחד לכל צמח).
התראה: טפל בחנקן נוזלי עם כפפות קריו, מגני פנים ומשקפי מגן. - יש לאחזר כל צמח בנפרד. חותכים ומשליכים את החלק האווירי. שטפו היטב את השורש במים כדי להסיר את כל המצע שנותר.
- לנתק כל גולם מן השורש ולאסוף אותם בצינור prechilled 15 מ"ל. הקפיא את הצינורות ואחסן אותם בטמפרטורה של −80 מעלות צלזיוס.
4. הכנת תמציות ציטוזוליות
הערה: המטרה הסופית של פרוטוקול זה היא להשיג RNA כולל באיכות גבוהה (TOTAL) ו- RNA הקשור לפוליזום (PAR). לכן, לעבוד בתנאים המונעים פירוק RNA, תמיד לשמור על דגימות ב 4 מעלות צלזיוס ולהשתמש בציוד מעבדה ללא RNase ופתרונות. אלא אם כן צוין, כל הפתרונות מוכנים עם מים סטריליים ultrapure.
- הכנת פתרונות מלאי בופר
- הכן את פתרונות המלאי הניתנים להחלפה אוטומטית המפורטים בטבלה 1, אוטוקלאב אותם למשך 15 דקות, ושמור ב- RT.
- הכן את פתרונות המלאי הניתנים לעיקור במסננים המפורטים בטבלה 1, סנן-עקר אותם ושמור אותם בטמפרטורה של −20 מעלות צלזיוס.
- הכינו את חיץ מיצוי הפוליסום (PEB, ראו טבלה 1) ושמרו אותו על קרח.
הערה: הכרכים שניתנו הם עבור שש דוגמאות. - מקררים את הצנטריפוגה ל-4 מעלות צלזיוס ומניחים 2 מ"ל צינורות מיקרוצנטריפוגה על קרח.
- שוקלים כ -0.2 גרם של גושים שלמים בכלי שקילה ומעבירים אותם לצינור 2 מ"ל מקורר מראש.
הערה: בצע שלב זה במהירות כדי למנוע הפשרה של הדגימות. לחלופין, ניתן להשתמש בנפח גולם מוערך של 0.4-0.5 מ"ל במקום 0.2 גרם. - מוסיפים 1.2 מ"ל של PEB, נותנים לדגימה להפשיר למשך 2 דקות, והומוגניים עם מטחנת רקמות עד לשיבוש מוחלט והומוגניות של הגושים.
הערה: הקפידו לטחון את הגושים לאחר ההפשרה מכיוון שהם יהיו רכים מספיק כדי להיות מופרעים בקלות עם מטחנות רקמות (ראו טבלת החומרים) בצינורות מיקרוצנטריפוגה. גושים קפואים קשה לטחון. כמו כן, מומלץ למקם את הצינורות בצידנית ספסל (0 מעלות צלזיוס) להומוגניזציה נכונה תוך שמירה על דגימות קרירות. - דגרו את הדגימות על הקרח בתסיסה עדינה למשך 10 דקות או עד שכל הדגימות עובדו.
- צנטריפוגה ב 16,000 × גרם במשך 15 דקות ב 4 מעלות צלזיוס כדי לזרוק את הפסולת. לשחזר את supernatant ולחזור על שלב צנטריפוגה.
- שחזר בזהירות את התמצית הציטוזולית המובהקת והעבר אליקוט של 200 μL לצינור מיקרוצנטריפוגה נקי של 1.5 מ"ל לבידוד כולל (דגימות TOTAL; ראה סעיף 7).
5. הכנת כריות סוכרוז
הערה: פרוטוקול זה משתמש בכרית סוכרוז דו-שכבתית (12% ו-33.5%) בצינורות אולטרה-צנטריפוגה בגודל 13.2 מ"ל (ראו טבלת חומרים). כל הפתרונות מוכנים עם מים סטריליים ultrapure.
- הכינו את תמיסות הסוכרוז והמלח.
- הכינו 100 מ"ל של תמיסת סוכרוז 2 M (68.5%, טבלה 1).
- הכינו 10x תמיסת מלח (טבלה 1).
- הכינו את שתי השכבות של כרית הסוכרוז כמתואר בטבלה 2. ראו טבלה 1 להרכב תמיסות המלאי האנטיביוטיות (CHX ו-CHL).
הערה: הכרכים שניתנו הם עבור שש כריות. - יוצקים 4.5 מ"ל של שכבת סוכרוז 33.5% לתוך צינורות צנטריפוגה. לאחר מכן, הוסיפו 4.5 מ"ל משכבת ה-12% בזהירות ובאיטיות עם מיקרופיפט P1000. שים את הצינורות על קרח עד תוספת של תמצית cytosolic הבהיר.
6. טיהור פוליזום
- טען 1 מ"ל של תמצית ציטוזולית מזוקקת (שלב 4.8) על גבי כרית סוכרוז על ידי ניפוח בזהירות על דופן הצינור.
- העבר את צינורות האולטרה-צנטריפוגה לדליים מוכנים מראש ולצנטריפוגה ב-217,874 × גרם (35,000 סל"ד ברוטור המוזכר [ראו טבלת החומרים]) למשך שעתיים ב-4 מעלות צלזיוס.
- יש להשליך את התמצית הציטוזולית הנותרת ואת כרית הסוכוז, ולהשהות את הגלולה הפוליסומלית עם 200 μL של RB מקורר מראש (שהוכן בעבר לפי טבלה 1).
- דגירה במשך 30 דקות על קרח ולאחר מכן להעביר את resuspension polysomal ל 1.5 מ"ל צינורות precooled. המשך עם מיצוי RNA (דגימות PAR).
7. מיצוי RNA ובקרת איכות
הערה: שלב זה מבוצע עבור דוגמאות TOTAL (שלב 4.8) ו- PAR (שלב 6.4).
- הכינו 75% EtOH עם מים סטריליים במיוחד וקיררו אותם ל-20°C-. בנוסף, יש לקרר כלורופורם ואיזופרופנול ל-20°C-.
- הומוגניזציה של הדגימות עם 750 μL של מגיב בידוד RNA ודגירה במשך 5 דקות ב- RT.
- יש להוסיף 200 μL של כלורופורם קר ולנער את הצינורות במרץ במשך 15 שניות. דגירה ב- RT במשך 10 דקות.
- צנטריפוגה בטמפרטורה של 12,000 × גרם למשך 15 דקות בטמפרטורה של 4 מעלות צלזיוס להפרדת פאזה. העבירו 500 μL מהפאזה המימית העליונה לצינור נקי מבלי להפריע לפאזה האורגנית הוורודה, והוסיפו 375 μL של איזופרופנול קר ו-0.5 μL של גליקוגן ללא RNase (ראו טבלת חומרים). מערבבים היטב על ידי צנרת למעלה ולמטה.
הערה: גליקוגן הוא נשא המשמש כמשקע משותף להגברת ההתאוששות מחומצת גרעין ממשקעי אלכוהול. - דגירה של התערובת למשך 10 דקות ב-4 מעלות צלזיוס וצנטריפוגה ב-12,000 × גרם למשך 15 דקות. השליכו את הסופר-נטנט.
- לשטוף את משקע RNA עם 1 מ"ל של קר 75% EtOH. מערבבים על ידי מערבולת קצרה.
הערה: בשלב זה, ניתן לאחסן את הדגימות בטמפרטורה של −20 מעלות צלזיוס למשך הלילה. - צנטריפוגה ב 7,500 × גרם במשך 5 דקות ב 4 מעלות צלזיוס, להסיר בזהירות את supernatant עם micropipette, ולייבש באוויר את כדור RNA.
- ממיסים את הכדור ב-50 μL של מים ללא RNase ודוגרים בטמפרטורה של 65°C למשך 5 דקות.
- הערך את ריכוז הרנ"א ותקינותו באמצעות אלקטרופורזה נימית רגישה ביותר18 (ראו טבלת החומרים) ו/או אלקטרופורזה על ג'ל אגרוז19 נטול RNase בשיעור של 2% (ראו טבלת חומרים).
הערה: אם דגימות ה- RNA אינן עומדות לשמש באופן מיידי או שהן עומדות להישלח למתקן הריצוף להכנת ספריית RNA-seq, מומלץ להשתמש ב- EtOH כדי לזרז אותן.
8. משקעי RNA
- מצננים את הצנטריפוגה ואת EtOH ל-4 מעלות צלזיוס.
- הכינו 3 M נתרן אצטט.
- הכינו 70% EtOH עם מים סטריליים במיוחד וקיררו אותם ל-20°C-.
- הערך את נפח המדגם. הוסף 0.1 כרכים של 3 M נתרן אצטט, 3 כרכים של EtOH קר, ו-0.5 μL של גליקוגן ללא RNase. מערבבים היטב.
- להשאיר בטמפרטורה של −20°C עד הצורך.
הערה: ניתן לאחסן דגימות מזרזות RNA למשך עד שנה אחת בטמפרטורה של −80 מעלות צלזיוס.
9. צינור סטנדרטי לניתוח ביטוי גנים
- בצע ממוצע של 40M קריאות קצה מזווגות של Illumina, עם אורך קריאה >100 bp, לניתוח בטוח של נתוני תמלול וטרנסלטום.
הערה: ניתוח נתוני RNA-seq סטנדרטי כולל את השלבים הבאים 9.2-9.5. - בצע בדיקת איכות קריאה באמצעות FastQC (https://www.bioinformatics.babraham.ac.uk/projects/fastqc/) או MultiQC20 מרובה הדגמות.
- הסר מתאם ורצפים באיכות נמוכה באמצעות Trimmomatic 21, BBDuk (http://jgi.doe.gov/data-and-tools/bb-tools/), cutadapt22, מגל23, או Trim Galore (https://www.bioinformatics.babraham.ac.uk/projects/trim_galore/), בין היתר.
- אם גנום ייחוס זמין עבור המין, בצע מיפוי קריאה כנגד ההתייחסות, ולאחר מכן כימות שפע, באמצעות Bowtie224, TopHat2 25 או סלמון 26, ו- featureCounts27, מלבד כלי קוד פתוח אחרים.
- בצע ניתוח סטטיסטי לזיהוי גנים מבוטא דיפרנציאלי באמצעות edgeR28, DESeq229, או limma30, בין היתר.
הערה: ניתן להשתמש בתוכנות קוד פתוח אלה באופן מקומי, דרך שורת הפקודה. לחלופין, ניתן להפעיל אותם באמצעות דפדפן אינטרנט בשרת ציבורי, כגון Galaxy31 או GEOexplorer32, המציעים ממשק משתמש גרפי, כך שאין צורך בידע בשורת הפקודה לצורך השימוש בהם.
תוצאות
הערכת הכמות והאיכות של שברים TOTAL ו- PAR המטוהרים באמצעות ההליך הנ"ל היא המפתח לקביעת הצלחתו, שכן עבור רוב היישומים במורד הזרם, כגון ריצוף RNA, דגימות באיכות גבוהה הן בסיסיות להכנת ספרייה וריצוף. יתר על כן, שלמות מולקולות הרנ"א מאפשרת לכידת תמונת מצב של פרופיל ביטוי הגנים ברגע איסוף הדגימה
Discussion
חקר ויסות ביטוי גנים ברמת התרגום הוא קריטי להבנה טובה יותר של תהליכים ביולוגיים שונים, שכן נקודת הקצה של ביטוי גנים בתאים היא שפע חלבונים13,14. ניתן להעריך זאת על ידי ניתוח הטרנסלטום של הרקמה או האורגניזם המעניין שעבורו יש לטהר את השבר הפוליסומלי ואת ה- mRNAs הקש...
Disclosures
למחברים אין ניגודי עניינים.
Acknowledgements
מחקר זה מומן על ידי מענק CSIC I+D 2020 מס ' 282, מענק FVF 2017 מס ' 210, ו- PEDECIBA (מריה מרתה סיינז).
Materials
| Name | Company | Catalog Number | Comments |
| Plant growth and rhizobia inoculation | |||
| Orbital shaker | Daihan Scientific | Model SHO-1D | |
| YEM-medium | Amresco | J850 (yeast extract) 0122 (mannitol) | |
| Water deficit treatment | |||
| KNO3 | Merck | 221295 | |
| Porometer | Decagon Device | Model SC-1 | |
| Scalpel | |||
| Preparation of cytosolic extracts | |||
| Brij L23 | Sigma-Aldrich | P1254 | |
| Centrifuge | Sigma | Model 2K15 | |
| Chloranphenicol | Sigma-Aldrich | C0378 | |
| Cycloheximide | Sigma-Aldrich | C7698 | |
| DOC | Sigma-Aldrich | 30970 | |
| DTT | Sigma-Aldrich | D9779 | |
| EGTA | Sigma-Aldrich | E3889 | |
| Igepal CA 360 | Sigma-Aldrich | I8896 | |
| KCl | Merck | 1.04936 | |
| MgCl2 | Sigma-Aldrich | M8266 | |
| Plastic tissue grinder | Fisher Scientific | 12649595 | |
| PMSF | Sigma-Aldrich | P7626 | |
| PTE | Sigma-Aldrich | P2393 | |
| Tris | Invitrogen | 15504-020 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween 20 | Sigma-Aldrich | P1379 | |
| Weighing dish | Deltalab | 1911103 | |
| Preparation of sucrose cushions | |||
| Sucrose | Invitrogen | 15503022 | |
| SW 40 Ti rotor | Beckman-Coulter | ||
| Ultracentrifuge | Beckman-Coulter | Optima L-100K | |
| Ultracentrifuge tubes | Beckman-Coulter | 344059 | 13.2 mL tubes |
| RNA extraction and quality control | |||
| Agarose | Thermo scientific | R0492 | |
| Bioanalyzer | Agilent | Model 2100. Eukaryote total RNA nano assay | |
| Chloroform | DI | 41191 | |
| Ethanol | Dorwil | UN1170 | |
| Isopropanol | Mallinckrodt | 3032-06 | |
| Glycogen | Sigma | 10814-010 | |
| TRIzol LS | Ambion | 102960028 | |
| Miscellaneous | |||
| Falcon tubes 15 mL | Biologix | 10-0152 | |
| Filter tips 10 µL | BioPointe Scientific | 321-4050 | |
| Filter tips 1000 µL | BioPointe Scientific | 361-1050 | |
| Filter tips 20 µL | BioPointe Scientific | 341-4050 | |
| Filter tips 200 µL | Tarsons | 528104 | |
| Microcentrifuge tubes 1.5 mL | Tarsons | 500010-N | |
| Microcentrifuge tubes 2.0 mL | Tarsons | 500020-N | |
| Sequencing company | Macrogen | ||
| Sterile 250 mL flask | Marienfeld | 4110207 |
References
- Limpens, E., et al. Cell- and tissue-specific transcriptome analyses of Medicago truncatula root nodules. PLoS ONE. 8 (5), 64377 (2013).
- Masson-Boivin, C., Giraud, E., Perret, X., Batut, J. Establishing nitrogen-fixing symbiosis with legumes: How many rhizobium recipes. Trends in Microbiology. 17 (10), 458-466 (2009).
- King, H. A., Gerber, A. P. Translatome profiling: Methods for genome-scale analysis of mRNA translation. Briefings in Functional Genomics. 15 (1), 22-31 (2014).
- Chassé, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), 15 (2017).
- Yángüez, E., Castro-Sanz, A. B., Fernández-Bautista, N., Oliveros, J. C., Castellano, M. M. Analysis of genome-wide changes in the translatome of Arabidopsis seedlings subjected to heat stress. PloS ONE. 8 (8), 71425 (2013).
- Brown, P. O., Botstein, D. Exploring the new world of the genome with DNA microarrays. Nature Genetics. 21, 33-37 (1999).
- Krishnamurthy, A., Ferl, R. J., Paul, A. -. L. Comparing RNA-Seq and microarray gene expression data in two zones of the Arabidopsis root apex relevant to spaceflight. Applications in Plant Sciences. 6 (11), 1197 (2018).
- Shulse, C. N., et al. High-throughput single-cell transcriptome profiling of plant cell types. Cell Reports. 27 (7), 2241-2247 (2019).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: A revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Kawaguchi, R., Girke, T., Bray, E. A., Bailey-Serres, J. Differential mRNA translation contributes to gene regulation under non-stress and dehydration stress conditions in Arabidopsis thaliana. The Plant Journal. 38 (5), 823-839 (2004).
- Larsson, O., Tian, B., Sonenberg, N. Toward a genome-wide landscape of translational control. Cold Spring Harbor Perspectives in Biology. 5 (1), 012302 (2013).
- Vogel, C., Marcotte, E. M. Insights into regulation of protein abundance from proteomics and transcriptomis analyses. Nature Reviews Genetics. 13 (4), 227-232 (2013).
- Tebaldi, T., et al. Widespread uncoupling between transcriptome and translatome variations after a stimulus in mammalian cells. BMC Genomics. 13 (220), 1-15 (2012).
- Wang, T., et al. Translating mRNAs strongly correlate to proteins in a multivariate manner and their translation ratios are phenotype specific. Nucleic Acids Research. 41 (9), 4743-4754 (2013).
- Ingolia, N. T. Ribosome profiling: New views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Lukoszek, R., Feist, P., Ignatova, Z. Insights into the adaptive response of Arabidopsis thaliana to prolonged thermal stress by ribosomal profiling and RNA-Seq. BMC Plant Biology. 16 (1), 1-13 (2016).
- Broughton, W. J., Dilworth, M. J. Control of leghaemoglobin synthesis in snake beans. Biochemical Journal. 125 (4), 1075-1080 (1971).
- Schroeder, A., et al. The RIN: An RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 1-14 (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing agarose gel electrophoresis of RNA nondenaturing agarose gel electrophoresis of RNA. Cold Spring Harbor Protocols. 2010 (6), 1-3 (2012).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: Summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Marcel, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- . Sickle: A sliding-window, adaptive, quality-based trimming tool for FastQ files (Version 1.33) Available from: https://github.com/najoshi/sickle (2011)
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Kim, D., et al. TopHat2: Accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biology. 14 (4), 1-13 (2013).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Liao, Y., Smyth, G. K., Shi, W. featureCounts: An efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30 (7), 923-930 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: A Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (550), 1-21 (2014).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2015).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Hunt, G. P., et al. GEOexplorer: A webserver for gene expression analysis and visualisation. Nucleic Acids Research. , (2022).
- Imbeaud, S., et al. Towards standardization of RNA quality assessment using user-independent classifiers of microcapillary electrophoresis traces. Nucleic Acids Research. 33 (6), 1-12 (2005).
- Di Paolo, A., et al. PDCD4 regulates axonal growth by translational repression of neurite growth-related genes and is modulated during nerve injury responses. RNA. 26 (11), 1637-1653 (2020).
- Smircich, P., et al. Ribosome profiling reveals translation control as a key mechanism generating differential gene expression in Trypanosoma cruzi. BMC Genomics. 16 (1), 443 (2015).
- Eastman, G., Sharlow, E. R., Lazo, J. S., Bloom, G. S., Sotelo-Silveira, J. R. Transcriptome and translatome regulation of pathogenesis in Alzheimer's disease model mice. Journal of Alzheimer's Disease. 86 (1), 365-386 (2022).
- Zanetti, M. E., Chang, I. -. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification of polyribosomal complexes of Arabidopsis for global analysis of gene expression. Plant physiology. 138 (2), 624-635 (2005).
- Mustroph, A., et al. Profiling translatomes of discrete cell populations resolves altered cellular priorities during hypoxia in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18843-11848 (2009).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature Reviews. Molecular Cell Biology. 16 (11), 651-664 (2015).
- Eastman, G., Smircich, P., Sotelo-Silveira, J. R. Following ribosome footprints to understand translation at a genome wide level. Computational and Structural Biotechnology Journal. 16, 167-176 (2018).
- Westermann, A. J., Gorski, S. A., Vogel, J. Dual RNA-seq of pathogen and host. Nature Reviews Microbiology. 10 (9), 618-630 (2012).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved