Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Purificación de polisomas a partir de nódulos simbióticos de soja
En este artículo
Resumen
Este protocolo describe un método para la purificación de polisomas eucariotas a partir de nódulos de soja intactos. Después de la secuenciación, se pueden utilizar tuberías estándar para el análisis de la expresión génica para identificar genes expresados diferencialmente a nivel de transcriptoma y translatomo.
Resumen
El objetivo de este protocolo es proporcionar una estrategia para estudiar el translatomo eucariota del nódulo simbiótico de soja (Glycine max). Este documento describe métodos optimizados para aislar polirribosomas derivados de plantas y sus ARNm asociados para ser analizados mediante secuenciación de ARN. En primer lugar, los lisados citoplasmáticos se obtienen mediante homogeneización en condiciones de preservación de polisomas y ARN a partir de nódulos de soja enteros congelados. Luego, los lisados se eliminan por centrifugación a baja velocidad, y el 15% del sobrenadante se usa para el aislamiento de ARN total (TOTAL). El lisado eliminado restante se utiliza para aislar polisomas por ultracentrifugación a través de un cojín de sacarosa de dos capas (12% y 33,5%). El ARNm asociado a polisomas (PAR) se purifica a partir de gránulos polisomales después de la resuspensión. Tanto TOTAL como PAR se evalúan mediante electroforesis capilar altamente sensible para cumplir con los estándares de calidad de las bibliotecas de secuenciación para RNA-seq. Como ejemplo de una aplicación posterior, después de la secuenciación, se pueden utilizar tuberías estándar para el análisis de la expresión génica para obtener genes expresados diferencialmente a nivel de transcriptoma y translatomo. En resumen, este método, en combinación con RNA-seq, permite estudiar la regulación traslacional de los ARNm eucariotas en un tejido complejo como es el nódulo simbiótico.
Introducción
Las plantas leguminosas, como la soja (Glycine max), pueden establecer simbiosis con bacterias específicas del suelo llamadas rizobios. Esta relación mutualista provoca la formación de nuevos órganos, los nódulos simbióticos, en las raíces de las plantas. Los nódulos son los órganos de la planta que albergan las bacterias y consisten en células huésped cuyo citoplasma está colonizado con una forma especializada de rizobios llamados bacteroides. Estos bacteroides catalizan la reducción del nitrógeno atmosférico (N2) en amoníaco, que se transfiere a la planta a cambio de carbohidratos 1,2.
Aunque esta simbiosis fijadora de nitrógeno es una de las simbiosis planta-microbio mejor estudiadas, quedan muchos aspectos por comprender mejor, como cómo las plantas sometidas a diferentes condiciones de estrés abiótico modulan su interacción con su pareja simbiótica y cómo esto afecta el metabolismo de los nódulos. Estos procesos podrían entenderse mejor analizando el translatomo de nódulos (es decir, el subconjunto de ARN mensajeros [ARNm] traducidos activamente). Los polirribosomas o polisomas son complejos de múltiples ribosomas asociados con el ARNm, comúnmente utilizados para estudiar la traducción3. El método de perfilado de polisomas consiste en el análisis de los ARNm asociados a polisomas y ha sido utilizado con éxito para estudiar los mecanismos posttranscripcionales que controlan la expresión génica que ocurre en diversos procesos biológicos 4,5.
Históricamente, el análisis de expresión del genoma se ha centrado principalmente en determinar la abundancia de ARNm 6,7,8,9. Sin embargo, existe una falta de correlación entre la transcripción y los niveles de proteínas debido a las diferentes etapas de la regulación posttranscripcional de la expresión génica, particularmente la traducción10,11,12. Además, no se ha observado dependencia entre los cambios a nivel del transcriptoma y los que ocurren a nivel del translatomo13. El análisis directo del conjunto de ARNm que se están traduciendo permite una medición más precisa y completa de la expresión génica celular (cuyo punto final es la abundancia de proteínas) que la obtenida cuando sólo se analizan los niveles de ARNm14,15,16.
Este protocolo describe cómo los polisomas derivados de plantas se purifican a partir de nódulos de soja intactos mediante centrifugación diferencial a través de un cojín de sacarosa de dos capas (Figura 1). Sin embargo, dado que los ribosomas derivados de bacteroides también están presentes en los nódulos, se purifica una mezcla de ribosomas y especies de ARN, aunque los eucariotas representan la fracción principal (90%-95%). También se describe el posterior aislamiento, cuantificación y control de calidad del ARN (Figura 1). Este protocolo, en combinación con RNA-seq, debería proporcionar resultados experimentales sobre la regulación traslacional de los ARNm eucariotas en un tejido complejo como el nódulo simbiótico.
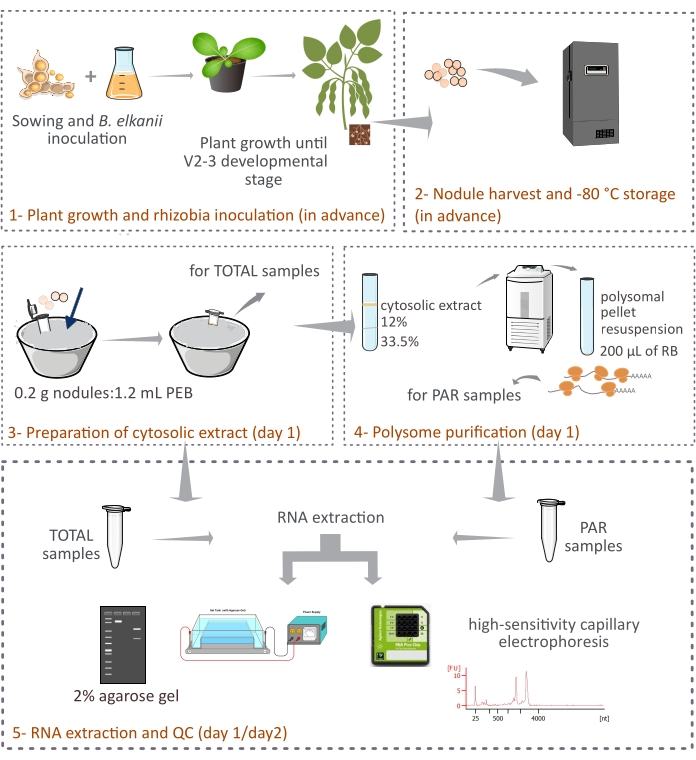
Figura 1: Resumen esquemático de la metodología propuesta para la purificación de polisomas eucariotas a partir de nódulos simbióticos. El esquema ofrece una visión general de los pasos seguidos en el protocolo desde (1) crecimiento de plantas y (2) recolección de nódulos hasta (3) preparación de los extractos citosólicos, (3) obtención de muestras TOTAL y (4) muestras PAR, y (5) extracción de ARN y control de calidad. Abreviaturas: PEB = tampón de extracción de polisomas; RB = tampón de resuspensión; TOTAL = ARN total; PAR = ARNm asociado a polisomas. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
1. Crecimiento de plantas e inoculación de rizobios
- Para generar los nódulos requeridos, siembre las semillas de soja de elección en el sustrato seleccionado en una cámara de crecimiento bajo condiciones controladas.
NOTA: En este protocolo, las semillas se sembraron en una botella de plástico de 0,5 L llena de una mezcla de arena: vermiculita (1:1). La cámara de crecimiento se estableció con una temperatura de ciclo día/noche de 28 °C / 20 °C, respectivamente, y un fotoperíodo de luz/oscuridad de 16 h/8 h, respectivamente. La intensidad de radiación fotosintéticamente activa fue de 620 μmol·m−2·s−1. - Prepare por adelantado el medio líquido de manitol de extracto de levadura (YEM) (consulte la Tabla de materiales). Autoclave el medio.
- El mismo día de la siembra de semillas, inocular un matraz que contenga 100 ml de YEM con la cepa Bradyrhizobium elkanii U1302. Incubar el matraz a 28 °C en un agitador orbital a 100 rpm.
- Deje que los rizobios crezcan durante 2 días e inocule las plántulas con 2 ml del cultivo.
2. Tratamiento del déficit hídrico (opcional)
NOTA: Este protocolo describe el tratamiento del déficit hídrico de las plantas de soja. Esta parte puede ser cambiada u omitida por completo dependiendo de la pregunta experimental en cuestión.
- Cultive las plántulas de soja durante 19 días (etapa de desarrollo V2-3) sin restricción de agua. Mantenga el sustrato a su capacidad de campo con B&D-medium17 suplementado con 0,5 mM KNO3.
- El día 20, retirar el riego.
NOTA: Aquí, el contenido de agua se midió diariamente por gravimetría (contenido gravimétrico de agua) durante los períodos de crecimiento y déficit de agua. - Mida la conductancia estomática por triplicado diariamente en la superficie abaxial de la hoja con un porómetro, según las instrucciones del fabricante (consulte la Tabla de materiales), desde el día 20 hasta el final del período de déficit hídrico.
NOTA: El final del período de déficit hídrico se determinó individualmente para cada planta cuando el valor de conductancia estomática fue aproximadamente del 50% del obtenido el día 20 de la siembra.
3. Recolección de nódulos
- Recoja el nitrógeno líquido en un recipiente limpio y etiquete los tubos de 15 ml (uno para cada planta).
PRECAUCIÓN: Manipule el nitrógeno líquido con guantes criogénicos, protectores faciales y gafas de seguridad. - Recupere cada planta individualmente. Cortar y desechar la parte aérea. Lave bien la raíz con agua para eliminar cualquier sustrato restante.
- Separe cada nódulo de la raíz y recójalos en un tubo preenfriado de 15 ml. Congele los tubos a presión y guárdelos a -80 °C.
4. Preparación de extractos citosólicos
NOTA: El objetivo final de este protocolo es obtener ARN total (TOTAL) y ARN asociado a polisomas (PAR) de alta calidad. Por lo tanto, trabaje en condiciones que eviten la degradación del ARN, manteniendo siempre las muestras a 4 °C y utilizando equipos y soluciones de laboratorio libres de RNasa. A menos que se especifique, todas las soluciones se preparan con agua ultrapura estéril.
- Preparación de soluciones de la reserva reguladora
- Preparar las soluciones madre esterilizables en autoclave enumeradas en la Tabla 1, esterilizarlas en autoclave durante 15 minutos y mantenerlas en RT.
- Preparar las soluciones madre esterilizables por filtración enumeradas en la Tabla 1, esterilizarlas por filtración y mantenerlas a −20 °C.
- Prepare el tampón de extracción de polisomas (PEB, ver Tabla 1) y manténgalo en hielo.
NOTA: Los volúmenes dados son para seis muestras. - Enfriar la centrífuga a 4 °C y colocar 2 ml de tubos de microcentrífuga sobre hielo.
- Pesar aproximadamente 0,2 g de nódulos intactos en una fuente de pesaje y transferirlos a un tubo preenfriado de 2 ml.
NOTA: Realice este paso rápidamente para evitar la descongelación de las muestras. Alternativamente, se puede utilizar un volumen de nódulo estimado de 0,4-0,5 ml en lugar de 0,2 g. - Agregue 1.2 ml de PEB, deje que la muestra se descongele durante 2 minutos y homogeneice con un molinillo de tejido hasta la interrupción y homogeneización completas de los nódulos.
NOTA: Asegúrese de moler los nódulos una vez descongelados, ya que serán lo suficientemente suaves como para ser fácilmente interrumpidos con molinos de tejido (consulte la Tabla de materiales) en tubos de microcentrífuga. Los nódulos congelados son difíciles de moler. Además, se recomienda colocar los tubos en un refrigerador de sobremesa (0 ° C) para una homogeneización adecuada mientras se mantienen las muestras frescas. - Incubar las muestras en hielo con una agitación suave durante 10 minutos o hasta que todas las muestras hayan sido procesadas.
- Centrifugar a 16.000 × g durante 15 min a 4 °C para granular los residuos. Recupere el sobrenadante y repita el paso de centrifugación.
- Recuperar cuidadosamente el extracto citosólico clarificado y transferir una alícuota de 200 μL a un tubo de microcentrífuga limpio de 1,5 ml para el aislamiento TOTAL (muestras TOTAL; ver sección 7).
5. Preparación de cojines de sacarosa
NOTA: Este protocolo utiliza un cojín de sacarosa de dos capas (12% y 33.5%) en tubos de ultracentrífuga de 13.2 ml (consulte la Tabla de materiales). Todas las soluciones se preparan con agua ultrapura estéril.
- Prepare las soluciones caldo de sacarosa y sal.
- Preparar 100 mL de solución de sacarosa 2 M (68,5%, Tabla 1).
- Prepare una solución salina 10x (Tabla 1).
- Prepare las dos capas del cojín de sacarosa como se describe en la Tabla 2. Ver Tabla 1 para la composición de las soluciones madre de antibióticos (CHX y CHL).
NOTA: Los volúmenes dados son para seis cojines. - Vierta 4,5 ml de la capa de sacarosa al 33,5% en los tubos de la centrífuga. Luego, agregue 4.5 ml de la capa al 12% con cuidado y lentamente con una micropipeta P1000. Poner los tubos en hielo hasta la adición del extracto citosólico clarificado.
6. Purificación de polisomas
- Cargar 1 ml del extracto citosólico clarificado (paso 4.8) sobre el cojín de sacarosa pipeteando cuidadosamente sobre la pared lateral del tubo.
- Transfiera los tubos de la ultracentrífuga a cucharones preenfriados y centrífuga a 217.874 × g (35.000 rpm en el rotor de referencia [consulte la tabla de materiales]) durante 2 h a 4 °C.
- Desechar el extracto citosólico restante y el cojín de sacarosa, y resuspender el pellet polisómico con 200 μL de RB preenfriado (previamente preparado según la Tabla 1).
- Incubar durante 30 minutos en hielo y luego transferir la resuspensión polisomal a tubos preenfriados de 1,5 ml. Proceder con la extracción de ARN (muestras PAR).
7. Extracción de ARN y control de calidad
NOTA: Este paso se realiza para las muestras TOTAL (paso 4.8) y PAR (paso 6.4).
- Preparar EtOH al 75% con agua ultrapura estéril y enfriar a -20 °C. Además, enfriar el cloroformo y el isopropanol a -20 °C.
- Homogeneizar las muestras con 750 μL del reactivo de aislamiento de ARN e incubar durante 5 min a RT.
- Añadir 200 μL de cloroformo frío y agitar los tubos vigorosamente durante 15 s. Incubar en RT durante 10 min.
- Centrifugar a 12.000 × g durante 15 min a 4 °C para la separación de fases. Transfiera 500 μL de la fase acuosa superior a un tubo limpio sin alterar la fase orgánica rosada, y agregue 375 μL de isopropanol frío y 0,5 μL de glucógeno libre de RNasa (consulte la Tabla de materiales). Mezclar bien pipeteando hacia arriba y hacia abajo.
NOTA: El glucógeno es un portador utilizado como coprecipitante para aumentar la recuperación de ácidos nucleicos de la precipitación de alcohol. - Incubar la mezcla durante 10 min a 4 °C y centrifugar a 12.000 × g durante 15 min. Deseche el sobrenadante.
- Lavar el precipitado de ARN con 1 mL de EtOH frío al 75%. Mezclar por vórtice breve.
NOTA: En este punto, las muestras se pueden almacenar a -20 °C durante la noche. - Centrifugar a 7.500 × g durante 5 min a 4 °C, retirar con cuidado el sobrenadante con una micropipeta y secar al aire el pellet de ARN.
- Disolver el pellet en 50 μL de agua libre de RNasa e incubar a 65 °C durante 5 min.
- Evaluar la concentración e integridad de ARN con electroforesis capilar altamente sensible18 (ver la Tabla de materiales) y/o electroforesis en un gel de agarosa libre de RNasa al 2%19 (ver la Tabla de materiales).
NOTA: Si las muestras de ARN no se van a utilizar inmediatamente o se van a enviar a la instalación de secuenciación para la preparación de la biblioteca de ARN-seq, se recomienda utilizar EtOH para precipitarlas.
8. Precipitación de ARN
- Enfriar la centrífuga y EtOH a 4 °C.
- Preparar acetato de sodio 3 M.
- Prepare EtOH al 70% con agua ultrapura estéril y enfríela a -20 °C.
- Estimar el volumen de la muestra. Agregue 0.1 volúmenes de acetato de sodio 3 M, 3 volúmenes de EtOH frío y 0.5 μL de glucógeno libre de RNasa. Homogeneizar.
- Dejar a -20 °C hasta que sea necesario.
NOTA: Las muestras precipitadas de ARN pueden almacenarse hasta 1 año a −80 °C.
9. Tubería estándar para el análisis de la expresión génica
- Realice un promedio de 40 millones de lecturas de extremo emparejado de Illumina, con una longitud de lectura >100 pb, para un análisis confiable de datos de transcriptoma y translatomo.
NOTA: El análisis de datos estándar de RNA-seq incluye los siguientes pasos 9.2-9.5. - Realice inspecciones de calidad de lectura utilizando FastQC (https://www.bioinformatics.babraham.ac.uk/projects/fastqc/) o MultiQC20 multimuestra.
- Retire el adaptador y las secuencias de baja calidad utilizando Trimmomatic21, BBDuk (http://jgi.doe.gov/data-and-tools/bb-tools/), cutadapt 22, hoz23 o Trim Galore (https://www.bioinformatics.babraham.ac.uk/projects/trim_galore/), entre otros.
- Si se dispone de un genoma de referencia para la especie, realice un mapeo de lectura contra la referencia, seguido de la cuantificación de la abundancia, utilizando Bowtie2 24, TopHat225 o Salmon26, y featureCounts27, además de otras herramientas de código abierto.
- Realizar análisis estadísticos para la identificación diferencial de genes expresados utilizando edgeR28, DESeq229 o limma30, entre otros.
NOTA: Este software de código abierto se puede utilizar localmente, a través de la línea de comandos. Alternativamente, pueden ser operados a través de un navegador web en un servidor público, como Galaxy31 o GEOexplorer32, que ofrecen una interfaz gráfica de usuario, por lo que no se requieren conocimientos de línea de comandos para su uso.
Resultados
La evaluación de la cantidad y calidad de las fracciones TOTAL y PAR purificadas con el procedimiento mencionado anteriormente es clave para determinar su éxito, ya que para la mayoría de las aplicaciones posteriores, como la secuenciación de ARN, las muestras de alta calidad son fundamentales para la preparación y secuenciación de bibliotecas. Además, la integridad de las moléculas de ARN permite la captura de una instantánea del perfil de expresión génica en el momento de la recolección de la muestra
Discusión
El estudio de la regulación de la expresión génica a nivel traslacional es fundamental para comprender mejor los diferentes procesos biológicos, ya que el punto final de la expresión génica celular es la abundancia de proteínas13,14. Esto se puede evaluar analizando el translatomo del tejido u organismo de interés para el cual se debe purificar la fracción polisomal y analizar sus ARNm asociados 3,4,34,35,36.
Divulgaciones
Los autores no tienen conflictos de intereses.
Agradecimientos
Esta investigación ha sido financiada por la subvención I+D 2020 del CSIC nº 282, la subvención FVF 2017 nº 210 y PEDECIBA (María Martha Sainz).
Materiales
| Name | Company | Catalog Number | Comments |
| Plant growth and rhizobia inoculation | |||
| Orbital shaker | Daihan Scientific | Model SHO-1D | |
| YEM-medium | Amresco | J850 (yeast extract) 0122 (mannitol) | |
| Water deficit treatment | |||
| KNO3 | Merck | 221295 | |
| Porometer | Decagon Device | Model SC-1 | |
| Scalpel | |||
| Preparation of cytosolic extracts | |||
| Brij L23 | Sigma-Aldrich | P1254 | |
| Centrifuge | Sigma | Model 2K15 | |
| Chloranphenicol | Sigma-Aldrich | C0378 | |
| Cycloheximide | Sigma-Aldrich | C7698 | |
| DOC | Sigma-Aldrich | 30970 | |
| DTT | Sigma-Aldrich | D9779 | |
| EGTA | Sigma-Aldrich | E3889 | |
| Igepal CA 360 | Sigma-Aldrich | I8896 | |
| KCl | Merck | 1.04936 | |
| MgCl2 | Sigma-Aldrich | M8266 | |
| Plastic tissue grinder | Fisher Scientific | 12649595 | |
| PMSF | Sigma-Aldrich | P7626 | |
| PTE | Sigma-Aldrich | P2393 | |
| Tris | Invitrogen | 15504-020 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween 20 | Sigma-Aldrich | P1379 | |
| Weighing dish | Deltalab | 1911103 | |
| Preparation of sucrose cushions | |||
| Sucrose | Invitrogen | 15503022 | |
| SW 40 Ti rotor | Beckman-Coulter | ||
| Ultracentrifuge | Beckman-Coulter | Optima L-100K | |
| Ultracentrifuge tubes | Beckman-Coulter | 344059 | 13.2 mL tubes |
| RNA extraction and quality control | |||
| Agarose | Thermo scientific | R0492 | |
| Bioanalyzer | Agilent | Model 2100. Eukaryote total RNA nano assay | |
| Chloroform | DI | 41191 | |
| Ethanol | Dorwil | UN1170 | |
| Isopropanol | Mallinckrodt | 3032-06 | |
| Glycogen | Sigma | 10814-010 | |
| TRIzol LS | Ambion | 102960028 | |
| Miscellaneous | |||
| Falcon tubes 15 mL | Biologix | 10-0152 | |
| Filter tips 10 µL | BioPointe Scientific | 321-4050 | |
| Filter tips 1000 µL | BioPointe Scientific | 361-1050 | |
| Filter tips 20 µL | BioPointe Scientific | 341-4050 | |
| Filter tips 200 µL | Tarsons | 528104 | |
| Microcentrifuge tubes 1.5 mL | Tarsons | 500010-N | |
| Microcentrifuge tubes 2.0 mL | Tarsons | 500020-N | |
| Sequencing company | Macrogen | ||
| Sterile 250 mL flask | Marienfeld | 4110207 |
Referencias
- Limpens, E., et al. Cell- and tissue-specific transcriptome analyses of Medicago truncatula root nodules. PLoS ONE. 8 (5), 64377 (2013).
- Masson-Boivin, C., Giraud, E., Perret, X., Batut, J. Establishing nitrogen-fixing symbiosis with legumes: How many rhizobium recipes. Trends in Microbiology. 17 (10), 458-466 (2009).
- King, H. A., Gerber, A. P. Translatome profiling: Methods for genome-scale analysis of mRNA translation. Briefings in Functional Genomics. 15 (1), 22-31 (2014).
- Chassé, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), 15 (2017).
- Yángüez, E., Castro-Sanz, A. B., Fernández-Bautista, N., Oliveros, J. C., Castellano, M. M. Analysis of genome-wide changes in the translatome of Arabidopsis seedlings subjected to heat stress. PloS ONE. 8 (8), 71425 (2013).
- Brown, P. O., Botstein, D. Exploring the new world of the genome with DNA microarrays. Nature Genetics. 21, 33-37 (1999).
- Krishnamurthy, A., Ferl, R. J., Paul, A. -. L. Comparing RNA-Seq and microarray gene expression data in two zones of the Arabidopsis root apex relevant to spaceflight. Applications in Plant Sciences. 6 (11), 1197 (2018).
- Shulse, C. N., et al. High-throughput single-cell transcriptome profiling of plant cell types. Cell Reports. 27 (7), 2241-2247 (2019).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: A revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Kawaguchi, R., Girke, T., Bray, E. A., Bailey-Serres, J. Differential mRNA translation contributes to gene regulation under non-stress and dehydration stress conditions in Arabidopsis thaliana. The Plant Journal. 38 (5), 823-839 (2004).
- Larsson, O., Tian, B., Sonenberg, N. Toward a genome-wide landscape of translational control. Cold Spring Harbor Perspectives in Biology. 5 (1), 012302 (2013).
- Vogel, C., Marcotte, E. M. Insights into regulation of protein abundance from proteomics and transcriptomis analyses. Nature Reviews Genetics. 13 (4), 227-232 (2013).
- Tebaldi, T., et al. Widespread uncoupling between transcriptome and translatome variations after a stimulus in mammalian cells. BMC Genomics. 13 (220), 1-15 (2012).
- Wang, T., et al. Translating mRNAs strongly correlate to proteins in a multivariate manner and their translation ratios are phenotype specific. Nucleic Acids Research. 41 (9), 4743-4754 (2013).
- Ingolia, N. T. Ribosome profiling: New views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Lukoszek, R., Feist, P., Ignatova, Z. Insights into the adaptive response of Arabidopsis thaliana to prolonged thermal stress by ribosomal profiling and RNA-Seq. BMC Plant Biology. 16 (1), 1-13 (2016).
- Broughton, W. J., Dilworth, M. J. Control of leghaemoglobin synthesis in snake beans. Biochemical Journal. 125 (4), 1075-1080 (1971).
- Schroeder, A., et al. The RIN: An RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 1-14 (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing agarose gel electrophoresis of RNA nondenaturing agarose gel electrophoresis of RNA. Cold Spring Harbor Protocols. 2010 (6), 1-3 (2012).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: Summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Marcel, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- . Sickle: A sliding-window, adaptive, quality-based trimming tool for FastQ files (Version 1.33) Available from: https://github.com/najoshi/sickle (2011)
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Kim, D., et al. TopHat2: Accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biology. 14 (4), 1-13 (2013).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Liao, Y., Smyth, G. K., Shi, W. featureCounts: An efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30 (7), 923-930 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: A Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (550), 1-21 (2014).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2015).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Hunt, G. P., et al. GEOexplorer: A webserver for gene expression analysis and visualisation. Nucleic Acids Research. , (2022).
- Imbeaud, S., et al. Towards standardization of RNA quality assessment using user-independent classifiers of microcapillary electrophoresis traces. Nucleic Acids Research. 33 (6), 1-12 (2005).
- Di Paolo, A., et al. PDCD4 regulates axonal growth by translational repression of neurite growth-related genes and is modulated during nerve injury responses. RNA. 26 (11), 1637-1653 (2020).
- Smircich, P., et al. Ribosome profiling reveals translation control as a key mechanism generating differential gene expression in Trypanosoma cruzi. BMC Genomics. 16 (1), 443 (2015).
- Eastman, G., Sharlow, E. R., Lazo, J. S., Bloom, G. S., Sotelo-Silveira, J. R. Transcriptome and translatome regulation of pathogenesis in Alzheimer's disease model mice. Journal of Alzheimer's Disease. 86 (1), 365-386 (2022).
- Zanetti, M. E., Chang, I. -. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification of polyribosomal complexes of Arabidopsis for global analysis of gene expression. Plant physiology. 138 (2), 624-635 (2005).
- Mustroph, A., et al. Profiling translatomes of discrete cell populations resolves altered cellular priorities during hypoxia in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18843-11848 (2009).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature Reviews. Molecular Cell Biology. 16 (11), 651-664 (2015).
- Eastman, G., Smircich, P., Sotelo-Silveira, J. R. Following ribosome footprints to understand translation at a genome wide level. Computational and Structural Biotechnology Journal. 16, 167-176 (2018).
- Westermann, A. J., Gorski, S. A., Vogel, J. Dual RNA-seq of pathogen and host. Nature Reviews Microbiology. 10 (9), 618-630 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados