Method Article
棘阿米巴属的定量运动性
摘要
该程序描述了如何可视化、跟踪和量化 棘阿米巴属。 运动性。
摘要
棘阿米巴 角膜炎是一种严重的眼部感染,会带来治疗挑战并可能导致失明。尽管它无处不在并且在接触水后可能会污染隐形眼镜,但这种病原体的自然行为仍然难以捉摸。了解 棘阿米巴的运动 模式可以告诉我们它如何定植隐形眼镜并污染患者的角膜。幸运的是, 棘阿米巴 spp. 通过明场显微镜从 4 倍放大开始可见。以前的技术已经开发出来来量化 棘阿米巴 运动与细胞病变效应或电场暴露不足。在这里,我们描述了一种跟踪和量化 棘阿米巴属的方法。 长期运动(数小时至数天),这是一种适用于多种变形虫菌株、表面和变形虫营养状况的方案。该程序与确定许多核心运动量化密切相关,例如距离、速度、限制和方向性,这些对于监测感染、增殖或行为改变的不同阶段是必要的。
引言
由于棘阿米巴角膜炎 (AK) 患病率的增加,棘阿米巴研究近年来急剧增加,棘阿米巴是棘阿米巴附着在角膜上的寄生虫感染1。虽然 AK 的爆发可归因于不适当的隐形眼镜护理或无效的隐形眼镜护理解决方案 2,3,4,5,但目前没有要求证明市场上任何产品的棘阿米巴消毒功效。然而,科学界和标准组织正在努力研究量化隐形眼镜护理产品消毒效果所需的方案 6,7。此外,由于其在细胞和功能方面与人类巨噬细胞相似,棘阿米巴除了阿米巴本身带来的致病性外,还被认为在宿主和传播其他人类病原体8 方面发挥着重要作用。
最近的技术已经被描述能够量化棘阿米巴运动 - 通常不高度受布朗运动的影响 9,10 - 关于细胞病变效应或暴露于电场不足 9,11,以及使用棘阿米巴作为可追踪病毒载体的巨型病毒研究中运动分析的进步12,13.在过去的 20 年里,使用新的软件程序(例如此处使用的成像软件)以及新算法和深度学习技术,细胞和粒子跟踪也取得了持续而显着的改进14。然而,这是一个相对较新且不断发展的科学领域,涉及台式研究、临床应用和行业标准,并且缺乏关于可视化和跟踪这种变形虫的方法的已发表数据,特别是为了量化依从隐形眼镜后或隐形眼镜消毒期间或之后的行为变化。扩展到长期视觉监控的其他领域也支持了这项工作 15,16,17。由于棘阿米巴本质上具有挑战性 - 包括对质粒的一般抗性(可以赋予荧光)、变形虫消耗和破坏标准细胞染料的能力,以及独特的外部蛋白质组成 - 使抗体标记变得困难 - 其他细胞可用的方法使它们在明场成像以外的环境中可见在该生物体中不可用。因此,量化这种变形虫的运动性已经证明了对该领域的显着补充。使用此处描述的方法,我们已经能够确定变形虫在没有营养物质的情况下保持运动至少12 小时 18,并且受到消毒过程挑战并在消毒过程中停止运动的变形虫可以在消毒后恢复其运动能力,如果它们没有完全裂解19。
该协议详细介绍了如何在显微镜下直观地跟踪和量化变形虫的运动。主要步骤是使用图像之间的适当焦点和时间在明场中记录变形虫,使用成像软件将图像转换为二进制,然后使用成像软件的跟踪插件设置实验参数并跟踪每个变形虫以确定所需的测量值,例如速度、距离和限制。在此之后,可以量化变形虫或变形虫种群的趋化性,以定义方向性。该方法的主要贡献是可视化和量化营养支持、表面粘附、消毒挑战或其他环境改变(例如与哺乳动物细胞培养物共存)的不同状态下的变形虫行为。
研究方案
1. 棘阿米巴 制剂
注:该实验方案已针对 ATCC 50655、30872、50702、30010、30461、50370、50703、30137、PRA-115 和 PRA-411 进行了验证。这是一种 BSL2 病原体,应在 BSL2 通风橱和实验室内进行研究。
- 用 30 mL AC6 培养基填充 T75 培养瓶,从样品瓶或准备好的塞子20 中制备预培养物,并在无菌中加入一个塞子或样品瓶的内容物。将培养瓶在 26-30 °C 下孵育 3-5 天,直到培养瓶汇合 50% 至 80%。
- 在需要细胞的前一天传代细胞,以产生均匀的滋养体群体。
- 根据菌株的需要,摇晃和/或轻快敲击主培养物 2 次以去除粘附的滋养体。
- 用 30 mL AC6 培养基填充 T75 培养瓶。向通道中加入 2 mL 主培养物。将培养瓶在 26-30 °C 下孵育 18-24 小时。
- 收获前,在显微镜下以 4 倍放大倍率目视检查滋养体种群。确保滋养体粘附且均匀。
- 轻快地敲击培养瓶 2 次以去除粘附的滋养体。将内容物倒入 50 mL 锥形管中。
- 在平衡离心机中以 500 x g 旋转试管 5 分钟以沉淀变形虫。倒出或移出上清液培养基并丢弃。在 2-10 mL 的 1/4 林格溶液中稀释沉淀。
- 涡旋样品。将 10 μL 样品添加到一次性血细胞计数器中,并计算 棘阿米巴的 CFU/mL。
- 根据血细胞计数器计数,在 1/4 林格溶液中将变形虫沉淀稀释至 7.5 x 103 个细胞/mL 的浓度。
- 将变形虫种子在表面上,可以是玻璃、塑料或非营养琼脂,以及各种孔形状,具体取决于实验需要,如下所述。
- 96 孔板:用 200 μL 棘阿米巴 悬浮液接种每个孔,并用盖子封闭板。
- 48 孔板:用 1 mL 棘阿米巴 悬浮液接种每个孔,并用盖子封闭板。
- 流通池:通过无菌铝制流通池的无菌端口缓慢加入 4 mL 棘阿米巴 悬浮液,避免腔室内出现气泡。添加悬架后,将端口夹紧关闭。
- 在成像之前,在开始显微镜记录之前,让变形虫至少粘附在表面 30 分钟。
2. 可视化和记录棘阿米巴
- 在明场中以 4 倍放大倍率观察变形虫。背景应为浅灰色,变形虫应为黑色。调整光线和焦点,使变形虫是实心黑眼圈而不是半透明的(因此,略微失焦, 图 1。
注意:跟踪的最佳放大倍数为 4 倍,原因如下:在跟踪过程中离开或进入视野的变形虫不应包含在数据分析中,因此较低的放大倍数最能支持一段时间内分析中包含的最大变形虫数量。变形虫应在明场中显示为黑色实心圆圈,以便将它们转换为二进制以进行跟踪分析。这在较低的放大倍率下最容易实现,因为较高的放大倍率很容易揭示变形虫的半透明性质和内部结构。一般来说,可以在较低的放大倍率下在单个视场中捕获更多的变形虫,从而支持更稳健的数据分析。 - 在成像软件中设置记录程序,以定期记录图像以跟踪变形虫。允许准确跟踪的图像之间的最长时间为 30 秒,文件大小和跟踪细节的最佳时间建议为 12 秒或 24 秒。
- 变形虫可以一次跟踪多天,但请注意这种记录会产生的文件大小以及处理超大文件的固有困难。为避免这种情况,请以一定的间隔记录一段时间的变形虫。例如,每 12 小时记录 1 小时,持续 5 天。
- 如果显微镜和程序允许,使用板孔的 XY 坐标或流通池的多个位置在单个会话中记录多个孔。如果需要,将一个孔或流通池的多个切片拼接在一起,以在 4 倍放大倍率下创建更大的视野。
注意:可以记录多少个位置的唯一限制是显微镜载物台在图像间隔时间内在不同位置之间移动的速度(例如,每 12 秒、24 秒等拍摄一次图像)。但是,这不是必需的,并且跟踪从单个位置制作的视频仍然可以具有足够数量的轨道,以进行稳健的统计分析。本研究使用了 Nikon Eclipse Ti-U 显微镜,并利用自动可移动载物台一次记录多个孔中的图像。但是,任何具有可编程记录功能的显微镜都可以工作。显微镜必须连接到计算机,并能够将图像记录到程序或硬盘驱动器中。
3. 变形虫大小分析
- 在成像软件中打开显微镜文件。将打开 Bio-formats 导入选项。在对话框中,确保满足以下条件:堆栈查看;查看方式:Selection-Hyperstack;内存管理:勾选 Use virtual stack;Color mode(颜色模式):默认。
- 确保未激活任何其他下拉菜单或复选框。单击 Ok(确定)。
- 打开 bio-formats 系列选项。选择所需的 Series。如果一次只记录一个位置,则此处可能只有一个选项。单击 Ok(确定)。
- 选择 Image > Duplicate(图像复制 ),通过仅选择一个 C 通道和一个 Z 通道来复制一次正在使用的单个井。
- 从这里开始,仅使用复制的图像进行操作和分析,而不是原始图像。选择 Image > Type > 8 位。选择 Process > Subtract Background (处理背景)。
- 将滚动球设置为 10.0。检查 Light Background (光背景)。检查 Sliding Paraboloid。
- 选择 Process > Enhance Contrast(增强对比度)。对于滋养体,将饱和像素设置为 .1%,对于一组细胞和聚集体,将饱和像素设置为 0.3%。
- 选择 Image > Adjust > Threshold (默认>黑白)”。Make Binary 进程将打开。选择 Default ,然后选中 Black background (of Binary Masks)。
- 积极选择阈值,以便大多数背景点不可见,但每个变形虫的某些部分可见。
- 如果成像软件反转了图像,背景是白色的,变形虫是黑色的,请转到 编辑>反转 将其固定为黑色背景和白色变形虫。
- 选择 Process> Binary > Close,以防由于细胞失焦或细胞运动而在外膜中具有较小的空间。
- 选择 Process > Binary > Fill holes (填充漏洞处理二进制文件)。使用形状绘制工具和 Edit > Fill 删除非变形虫伪影。如果此时背景为白色,变形虫为黑色,请选择 Edit > Invert。此时,图像应表示 图 2 中的二进制图片。
- 要记录每个变形虫的大小,请选择 Analyze > Analyze Particles。套装大小:10-Infinitiy,循环度:0-1,显示:无。选中 Only Display Results (仅显示结果) 和 Summarize (汇总)。
- 保存显示的 CSV 文件。选择 File > Save As > Tiff 并选择 Save As Tiff 并将名称编辑为所需的规范。
4. 准备用于跟踪的显微镜文件(运动分析)
- 在成像软件中打开显微镜文件。Bio-Formats Import Options 将打开。
- 在对话框中,确保满足以下条件:堆栈查看;查看方式:Selection-Hyperstack。对于 memory management(内存管理),选中 Use Virtual Stack(使用虚拟堆栈)。单击 Ok(确定)。
- 将打开 Bio-Formats 系列选项。选择所需的 Series。如果一次只记录一个位置,则此处可能只有一个选项。单击 Ok(确定)。
- 选择 Image > Duplicate。从这里开始,只处理视频的复制部分,而不是处理原始文件。
注意:复制当时正在跟踪的视频部分。为此,需要知道与实验所需的分钟数相对应的帧。例如,如果要跟踪第 1 小时并每 24 秒拍摄一次图像,则第 1 小时内应有 150 帧。因此,第 1 小时将是帧 1-150,第 2 小时将是 151-300,依此类推。 - 选择 Image > Type > 8-bit。选择 Process > Subtract Background (处理背景)。将 Rolling ball 设置为 10.0。
- 检查 Light Background (光背景)。检查 Sliding Paraboloid。选择 处理>增强对比度 并将其设置为 0.1%。
- 选中 All x# Slices (例如,所有 150 个切片)。取消选中 Normalize (规范化)。选择 Image > Adjust > Threshold(默认>黑白))。
- Make Binary 进程将打开。选择 Default (默认)。检查 Black Background (of Binary Masks)。
- 阈值,因此大多数背景点不可见,但变形虫的某些部分是可见的。在此步骤中,如果背景为白色且变形虫为黑色,请选择 Edit > Invert (编辑反转)。背景应该是黑色的,变形虫应该是白色的。
- 选择 Process > Binary > Close (处理二进制文件关闭)。选择 Process > Binary > Fill holes (填充漏洞处理二进制文件)。选择 File > Save As > Tiff 并选择 Save As Tiff 并将名称编辑为所需的规范。现在,该文件已准备好进行跟踪。
5. 通过跟踪分析运动性
- 从具有跟踪打开所需的 tiff 文件的成像软件中,转到 Trackmate > 跟踪的插件>。此时将打开 Trackmate 的版本。
- 选择 Next (下一步)。从检测器下拉菜单中选择 LoG 检测器 。设置估计的斑点直径:35.0 微米,阈值 1.0。取消选中 Use median filter 和 Do sub-pixel localization。
- 单击第一个、中间和结束切片处的 预览 。确保所有变形虫都由紫色圆圈捕获,并且不包括背景缺陷或伪影。
- 选择 Next (下一步)。它将开始处理,此步骤可能需要几分钟才能完成。屏幕顶部的检测栏将指示该过程已完成多少。
- 出现提示时选择 Next (下一步 )。在初始阈值中,再次选择 Next (下一步 ),而不选择任何内容。选择视图时选择 Hyperstack Displayer 。选择 Next (下一步)。
- 设置 filter on spot(筛选点),然后选择 Next(下一步 ),而不选择任何内容。选择跟踪链接;为此,请选择 LAP Tracker (而不是 Simple LAP Tracker)。选择 Next (下一步)。
- 在帧到帧链接时,将最大距离设置为 40 μm。On Track Segment Closing (轨道段间隙缩小) 选中 Allow Gap Closing (允许间隙缩小)。将最大距离设置为 100 μm,最大帧间隙设置为 4。请勿为“轨道段拆分”或“轨道段合并”选择任何内容。
- 选择 Next (下一步)。执行跟踪可能需要很长时间。完成后,窗口应显示:Tracking done in the x s 在底部。选择 Next (下一步)。
- 跟踪完成后,再次选择 Next (下一步 )。在轨道上设置过滤器:选择左下角的 + 号,为轨道中的点数添加过滤器。将滤镜设置为至少 93% 的帧的 Above(例如,150 帧至少需要 140 个点)。
注意:该软件的某些版本需要选择相反的等效值,例如低于至少 7%,这可以通过向左或向右拖动一条线来达到所需的数字来完成。 - 选择 Next (下一步)。将显示 Display Options。在保存 Trackmate XML 文件或 Tracks CSV 文件之前,请确保每一帧中的轨迹不会因变形虫的移动方式而变形或奇怪。
- 如果需要删除轨道,请单击 TrackScheme。TrackScheme 将显示所有帧中变形虫的所有轨迹和接触点。
注意:删除轨迹的可能原因是:在录制过程中,变形虫在视野中走动/离开了视野,与变形虫的实际路径相比,轨迹的形状不合适,或者由于某种原因,根据实验参数,轨迹或变形虫在数据中是异常值,等等。 - 单击变形虫以突出显示屏幕上的绿色斑点,在 Trackscheme 中,该斑点周围将有一个绿色方块。这将指示需要删除哪个轨道或点。
- 右键单击需要删除的轨道,然后选择 Whole Track。按 Delete 按钮。
- 查看所有跟踪后,单击跟踪软件弹出窗口左下角的 Save 保存跟踪文件。使用视频名称保存 XML 文件。点击 恢复 返回跟踪软件弹出窗口。
- 单击 Tracks 并选择左侧的 Tracks 。单击 Export to CSV 并保存 CSV 文件。
- 如果电子表格无法识别生成的 CSV,请执行以下步骤。
- 在记事本中打开保存的 CSV 文件。点击 Control + A 和 Control + C 复制所有内容。
- 打开电子表格,然后单击 Control + V 将其粘贴到电子表格中。所有数据都将位于一个单元格中。
- 在数据仍处于选中状态的单元格中,转到 Data > Text to Columns。选择 Delimited > Next (下一步)。选择 Comma > Next > Finish。将此文件另存为新的 CSV。
- 跟踪 CSV 将有许多可用于分析的参数(图 3)。将所需参数复制并粘贴到新的 CSV 文件(图 4)进行分析。此处使用的参数如下所述。
- 总行进距离:变形虫行进的总距离(以 μm 为单位)。
- Max distance traveled:变形虫路径上从起点开始的最大距离 - 这可能不一定是终点
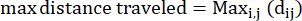
其中 dij 是到轨道上任何点 I 到任何点 j 的距离。 - 限制比:限制比表示变形虫离开起点的效率:它是从起点沿直线移动(接近 1 的值),还是相对靠近起点(接近 0 的值)。
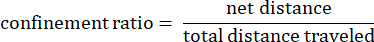
- 跟踪平均速度:变形虫在总行进距离上的平均速度,以微米/秒为单位。
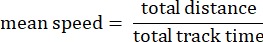
- Track displacement:变形虫起点与终点之间的距离(以微米为单位)。
注:所有成像软件分析参数及其定义均可在此处找到:https://imagej.net/plugins/trackmate/analyzers/。请记住,每个保存的文件都是当时分析的轨道的摘要。为了产生稳健的数据分析,需要根据数据情况适当地组合多次重复、小时等的结果(图 5 和 图 6)。
结果
要使用这种方法取得成功,需要考虑几个关键的整体部分。第一个是变形虫和显微镜的物理设置,第二个是正确利用成像软件,第三个是以有意义的方式导出和分析成像数据。
在显微镜记录开始之前,确保变形虫正确聚焦在显微镜载物台上至关重要(图 1)。与其将注意力集中在最佳 Z 平面上的变形虫上以获得单个细节,不如将其略微失焦,以便它们在细节观察时变成实心黑点,而不是半透明状态。以这种方式查看它们将允许成像软件和跟踪软件在文件转换为二进制时更容易地找到每个单独的变形虫(图 2)。了解成像软件中的目标是什么 - 也就是说,让每个变形虫都显示为一个明确的白色圆圈,这可能有助于读者在明场中拍摄原始图像时找到合适的焦点。然后,在明确定义每个圆圈后,跟踪软件将能够正确跟踪每个变形虫的运动并收集每个变形虫的个体运动指标。
在按照上面列出的成像软件和跟踪软件协议中的分步说明并参考相关视频以协助使用成像软件的关键第一步之后,是时候导出成像软件数据进行定量和数据分析了。跟踪软件的原始输出将类似于 图 3 中所示。在这里,重要的是要检查它导出的轨道中的点数是否与预期一致(例如,如果要求每个轨道中存在 93% 的帧,而视频中总共有 100 帧,则每个导出的轨道应大于或等于 93 个点)。如果点不符合要求,则可能需要返回跟踪软件步骤以确保正确设置所需的参数。
从原始导出开始,开始构建数据分析表(图 4)。请务必记住,每个轨道都表示单个复制中的单个变形虫。因此,如图 4、 图 5 和 图 6 所示,必须对帧集或时间点的所有轨迹进行平均(图 4),并且必须对整个系列中的每个帧集或时间点进行此过程(即,如果总共拍摄了 3 小时的视频,并且数据点按小时划分, 然后每个重复将有 3 帧集/小时进行分析, 图 5)。最后,从每个重复中每个帧集中的平均值组合中,必须将每个重复的平均数据合并到一个空间中,以便可以分析多个重复中的数据以确定每个时间点的真实平均值和标准偏差(图 6)。
每个仿行的平均值将用于数据分析,所有仿行的合并平均值和标准差或标准误差将用于图形表示(如图 7 中的示例所示)。如果像我们在这里所做的那样分析数据,即在多个时间点跟踪多个样本,则使用 2 向重复测量方差分析和事后 Tukey 检验来分析它们是最合适的。这使我们能够确定每个时间点样品之间的差异,并确定每个样品内随时间变化的差异。

图 1:变形虫在明场显微镜下显示为不透明的深色形式。 以 4 倍放大倍率放大变形虫的代表性图像,略微失焦,显示为纯色黑眼圈。比例尺 = 500 μm。 请点击此处查看此图的较大版本。
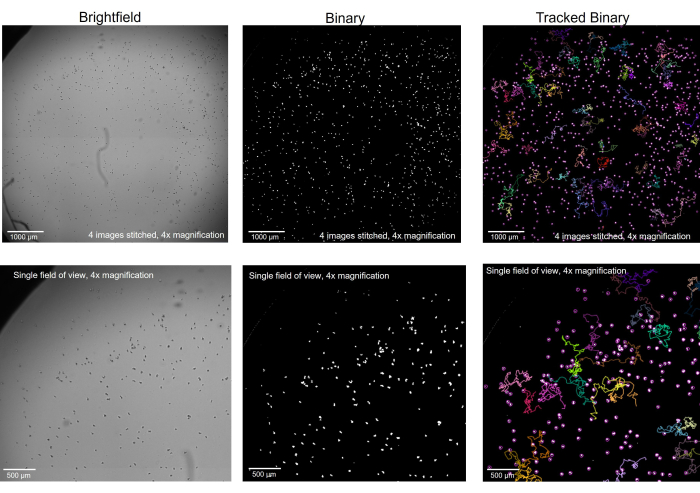
图 2:在明场显微镜下成像的变形虫转换为二进制并进行跟踪。 在明场(左)中,4 倍放大倍率的变形虫被转换为去除伪影的二进制变形虫(中),跟踪后显示选定的轨迹(右)。顶行:通过将 4 张图像拼接在一起成像的整个孔,比例尺 = 1000 μm。底行:拼接前单个图像的尺寸,比例尺 = 500 μm。 请单击此处查看此图的较大版本。
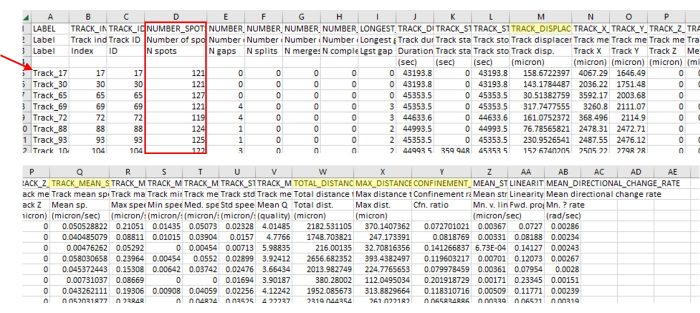
图 3:成像和跟踪软件的即时数据分析输出包含多种数据类型。 跟踪的代表性输出如图 2 所示,这是一个单视频。请注意轨道编号(红色箭头)跳过编号,表示轨道跟踪软件正确删除了与参数不对齐的轨道。在此示例中,识别的 5385 个轨道中只有 71 个与参数对齐。考虑到如此严格的指导方针,这个比例是正常的。这个轨迹的数量也与该协议中使用的变形虫数量一致(例如,如果每孔用 200 μL 的 7.5 x 103 CFU/mL 接种 96 孔板,那么应该预期孔中大约有 1,500 个变形虫,并且在 4 针视野中可见的变形虫数量大致相同。当变形虫在视野中进出时,足迹可能会比变形虫多得多,从而产生新的足迹)。这个轨道编号在 图 2 中再次很明显,与大量点相比,只有少数轨道可见。始终检查轨道中的点数(红色框),以确保仅包含正确的轨道长度。例如,在这里,我们包含了超过 93% 的帧,并且此部分有 127 帧,因此所有点数都应高于 118。以黄色突出显示的是实验方案中提到的测量结果。 请单击此处查看此图的较大版本。
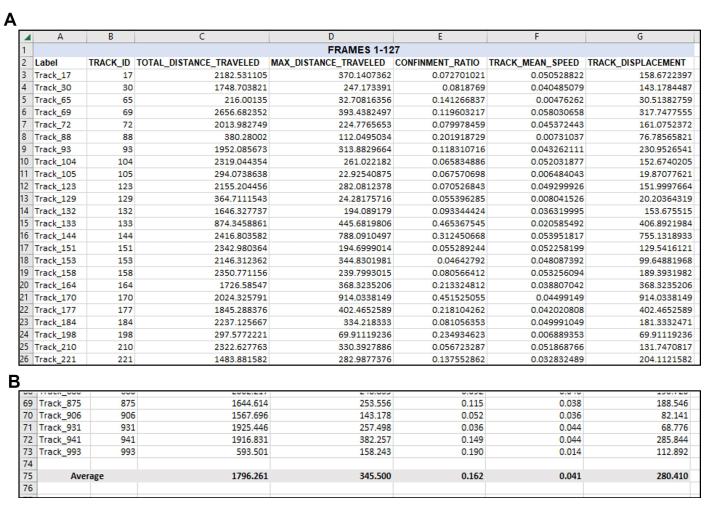
图 4:在初始数据输出后根据需要重新组织数据。 导出后所需数据的组织。(A) 选择用于量化的相关测量值是从跟踪软件导出中复制的(图 3)并粘贴到新的电子表格中。(B) 在新电子表格中组织所需的测量值后,应计算每个轨迹的平均值。因此,在这个具有代表性的示例中,此单个视频中每个可用轨道的总距离、最大距离、限制、平均速度和位移已在每次测量中平均为一个奇异数。请再次注意,共有 71 个轨道满足第 1 帧到第 127 帧的参数。从分析的每个视频或帧集中可能会有不同数量的合适轨道,如图 5 所示。 请单击此处查看此图的较大版本。
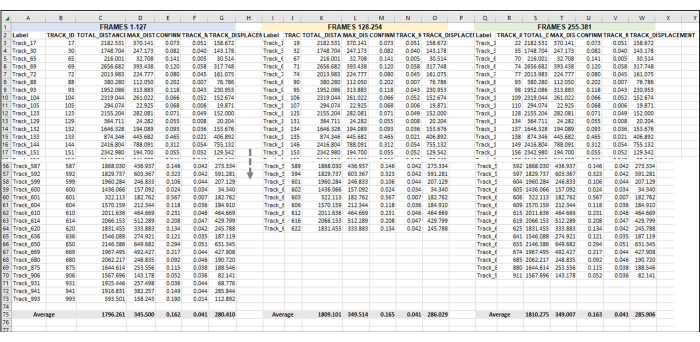
图 5:为分析整个图像系列或视频而设置的序列帧。 这是在数据分析中添加连续视频的代表性图像。假设第 1-127 帧构成视频的第 1 小时(这里,1 小时由每 28 秒拍摄一次图像组成)。第 128 帧到第 254 帧将是视频的第 2 小时,帧 255-381 将是视频的第 3 小时。将所有小时的导出数据(如图 3 中导出的)合并到一个工作表中,以便一次处理所有数据。如图 4 所示,每个帧集/小时的所有可用轨道的数据将被平均。 图 5 中的所有内容都是来自一个重复或一个样本的数据。如果有 3 个重复,那么将有 3 个布局相同的单独电子表格选项卡,这些选项卡中的数据将被输入到一个新选项卡中,如图 6 所示。 请单击此处查看此图的较大版本。
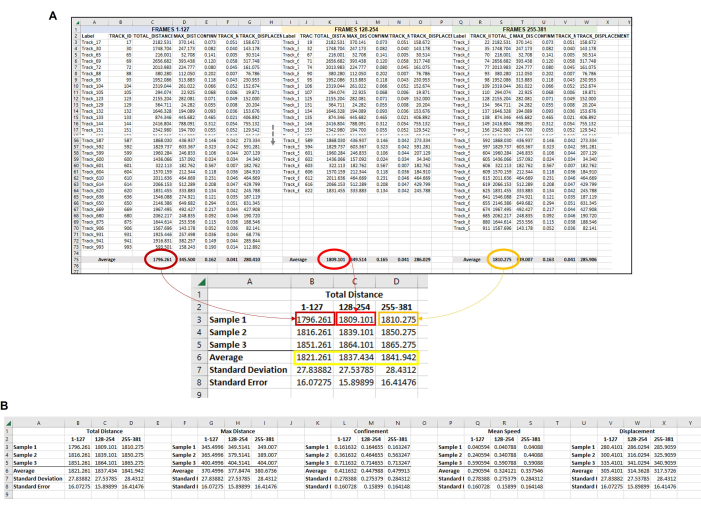
图 6:来自每个框架集中所有变形虫的复合数据,用于表示单个重复。 (A) 图 5 中每帧集的所有平均值都需要收集在一个中心位置,以便完成最终的数据分析。在此代表性图像中,显示了前三个帧集(或小时)的 Total Distance 平均值。必须对正在运行的每个样品/条件/重复重复此操作。然后,必须对每个帧集中每个样本的平均值进行平均(黄色框)。这是可以计算标准偏差和标准误差的最终数据点。每个样本的单独平均值(此处为总距离样本 1 的 B3、B4 和 B5 中的数字;C3、C4 和 C5 用于总距离采样 2 等)将用于运行统计分析。对于所有必需的测量(最大距离、限制、平均速度、位移等),将重复此过程。(B) 要制作量化的图形表示,请使用黄色框中的数字作为数据点,用于最终图形。 请单击此处查看此图的较大版本。
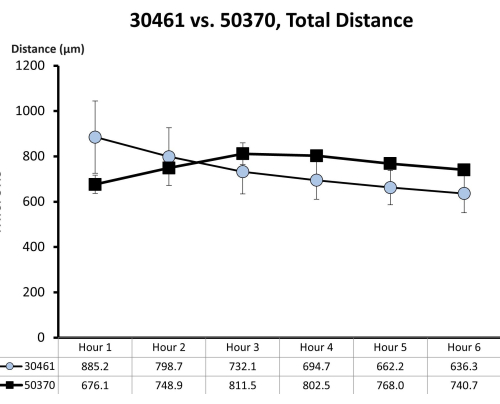
图 7:ATCC 30461 和 ATCC 50370 的原始运动数据。原始数据(此处表示为每小时的平均 ± SE)是先前收集的,未为先前的研究发表18,19。这种运动定量表明,这两种变形虫在玻璃表面上的 1/4 林格溶液中,它们的前 6 小时运动(以该小时内行进的总距离(以微米为单位)是相似的。每个时间点代表三个单独的重复,每个重复由 20-200 个 amoeba 轨迹组成。请单击此处查看此图的较大版本。
讨论
能够跟踪和量化变形虫的运动,例如棘阿米巴,它们是不受低速布朗运动影响的粘附微生物 9,10,揭示了有关变形虫行为的大量信息,可以大大增强对 AK 预防方法的启发。该协议已在最近的出版物中详细说明,并且数据已通过其他类似数据分析证实或与其他类似数据分析相结合18,19。我们在这里注意到,使用这种方法详细描述的变形虫的速度与其他小组公布的速度相似。该协议因其基本上能够用于对光透明的表面或变形虫处理而著称,但重要的是要注意,虽然该协议可能会被修改以与其他生物一起使用,但到目前为止,它仅针对棘阿米巴进行了优化。上述方案在实验室中一直运行良好,但提供了以下更改,用于更改方法以适应其他需求或解决困难情况。
确定跟踪参数:在上述协议中,列出了帧到帧链接、间隙闭合、最大帧间隙和轨道上的点数,这些参数最适用于最近的出版物。但是,其他参数可能更适合其他需求。此外,可能还有一些其他人想要使用但未在此处使用的参数(例如轨迹过滤器下轨迹的 Maximum Distance)。有关每个参数及其描述的更多信息,请访问网站(参见参考文献21)。在决定哪些参数和过滤器与其他实验最相关以及如何设置它们时,请考虑特定项目的统计合理性;例如,人们是否希望来自离开然后又回到视野中的变形虫的数据?或者,人们是否希望保留来自根本没有移动但仍然被标记为轨迹的变形虫的数据?以及录制图像时帧数和帧之间的时间长度有什么意义。
改变斑点大小:我们发现,根据标准实验室可用的当前技术和软件,任何软件或任何显微镜设置都非常困难,要准确跟踪聚集体中变形虫的数量,因为它会随时间变化。如果跟踪一个斑点的大小并尝试将该大小与其中的生物体数量相关联,请使用重复实验后生成的标准曲线来数学预测聚集体的大小。例如,通过在孔中接种一系列变形虫来产生聚集体,例如每孔 8、16、32、125、250、500、1,000 和 2,000 个细胞。生成每个孔不同时间点 24 h 的延时图像。对每个球体(具有已知数量的变形虫)进行二维面积分析,并生成标准曲线作为变形虫数量与球体大小随时间变化的函数。这个实验至少重复一式三份,以便为我们提供生成的任何曲线的适当标准差。
确定变形虫的方向性:虽然这对于特定研究可能不是必需的,但了解变形虫的方向性可能会有所帮助。这将提供有关治疗或实验的趋化效果的数据。此数据还可用于创建视觉 (图形) 和定量数据。这可以通过趋化性和迁移工具获得,该工具是成像软件的免费插件。网站上提供了该指南以及示例图像和视频(参见 材料表)。
详细的运动动力学:其他小组已经通过高度先进和发达的统计分析研究了扩散动力学和扩散轨迹,超出了这里讨论的范围 9,10,22。这些可以根据用户的需求进行考虑。
与任何成功的方法一样,本手稿中详述的方案已经经过多轮故障排除以实现一致性。虽然可以通过每秒拍摄一张图像来制作出色的高质量可跟踪视频(或者显微镜能够以最快的速度拍摄,可能不到一秒),但这会产生非常大的文件,这可能很难处理。这实际上仅适用于最多录制几分钟的非常短的视频。相反,通过每隔几秒钟拍摄一张图像,我们发现根据该物种的速度,变形虫的行为仍然非常可追踪,并且可以在数小时或数天内创建可行的视频。我们发现图像之间的最大时间为 30 秒,这是成像软件准确跟踪变形虫所需的时间。用户选择的时间间隔应使用显微镜记录图像的速度、每孔需要多少张图像以及每个间隔记录多少孔的已知约束来考虑。同样,本协议中提到的有关最大位移、允许的帧间隙、所需的最小帧数等参数已由本实验室通过反复试验确定,以创建轨道信息,其中包括最完整的变形虫轨道,并忽略由不完整的轨道产生的噪声、在视频中加入或离开舞台的变形虫,或由成像软件混淆产生的错误,例如当两个变形虫在轨道中相遇然后分离时。在创建不断相互交互的微生物的极长(数小时到数天)视频时,这类错误和不完整的轨道是可以理解的,这就是为什么必须拒绝很大一部分错误轨道的原因。应该注意的是,根据本实验室的经验,协议中的填充孔步骤对于减少成像软件跟踪变形虫的错误非常重要。通过确保每个变形虫都是一个实心圆圈,而不是有时是甜甜圈或 c 形,该软件更有可能成功跟踪每个变形虫。
此外,如前所述,用户可以使用许多参数来分析图像或视频。根据实验需求,我们始终从分析总距离、最大距离、速度和位移中受益最大。这些在以前的出版物中通过图形解释进行了深入讨论(包括使用不同的应变和表面)18,19。这四个参数允许用户推断变形虫行进线性距离的能力以及行进需要多长时间,这有助于了解它们与隐形眼镜污染相关的行为。检索和分析这些参数是一项艰巨的工作,如我们的图所示。在处理成像软件输出中的大量电子表格时,我们通过创建锁定的电子表格模板来自动计算所有必需的分析,从而减少了无意的错误。但是,对此方法的可能改进是编写一个可以处理此数据并对其进行排序和分析的脚本。
总之,这里描述了一种在许多不同条件下测量 棘阿米巴 大小和运动的可访问且准确的方法。我们已经证明这种方法可以应用于许多不同的变形虫菌株,并概述了虽然可能有简单的参数来获得运动信息,但这种实验设置可以高度适应任何特定需求。
披露声明
所有作者均为 Alcon Research, LLC 的员工。
致谢
这项工作由 Alcon Research, LLC 资助。
材料
| Name | Company | Catalog Number | Comments |
| ¼ Ringer’s solution | ThermoFisher Scientific | BR0052G | Oxoid Ringers Solution Tablets. Follow directions to make one-quarter strength instead of full strength Ringers. |
| 10 µL pipette | Eppendorf Research | 3123000039 | 2 µL-20 µL single channel |
| 10 µL pipette tips | Neptune Scientific, San Diego, CA, USA | BT10.N | 10 µL Universal Barrier Tip |
| 48 well plate | Millipore Sigma, | CLS3548 | Corning Costar TC-Treated Multiple Well Plate: polystyrene plate, flat bottom wells, sterile, with lid |
| 50 mL conical tubes (1 for each sample, 1 for each pass 2 sample) | Fisher Scientific | Falcon 352098 | Falcon 50 mL High Clarity Conical Centrifuge Tubes, polypropylene |
| 96-well plate | Millipore Sigma, Burlington, MA, USA | CLS3596 | Corning Costar TC-Treated Multiple Well Plate: polystyrene plate, flat bottom wells, sterile, with lid |
| Acanthamoeba | American Type Culture Collection, Manassas, VA, USA | ATCC 30461 | This protocol has been verified for ATCC 50655, 30872, 50702, 30010, 30461, 50370, 50703, 30137, PRA-115, and PRA-411 |
| Axenic culture media (AC6) | Made in house | n/a | Containing 20 g biosate peptone, 5 g glucose, 0.3 g KH2PO4, 10 µg vitamin B12, and 1 glass5 mg l-methionine per liter of distilled deionized water. Adjust pH to 6.6-6.95 with 1 M NaOH and autoclave at 121 °C for 20 min, store at room temperature for up to 3 months. |
| Centrifuge and appropriate rotor | Thermo Scientific, Waltham, MA, USA | Sorvall ST 40R | Any equivalent centrifuge and rotor are acceptable so long as it can spin 50 mL conical tubes at 500 x g for 5 min |
| Chemotaxis and Migration Tool | Free software based on ImageJ, available at https://ibidi.com/chemotaxis-analysis/171-chemotaxis-and-migration-tool.html | ||
| Disposable hemocytometer | Bulldog Bio, Portsmouth, NH, USA | DHC-N01 | Neubauer Improved 2-Chip Disposable Hemocytometer, Individually packaged, Nonpyrogenic |
| ImageJ software with Trackmate plugin (this protocol written with Trackmate version 6.0.2.) | Free software developed by the National Institutes of Health, available at imagej.net. Trackmate plugin available at https://imagej.net/plugins/trackmate/ | ||
| Microscope, preferably with automated moveable stage | Nikon, Tokyo, Japan | A Nikon Eclipse Ti-U Microscope was used in this study and the automated moveable stage was utilized to be able to record images in multiple wells at a time. | |
| NIS-Elements software | Nikon | ||
| Serological pipette | Fisher Scientific, Hampton, NH, USA | BrandTech 26331 | BrandTech accu-jet pro Pipet Controller |
| Serological pipette tips | VWR | 5 mL: 76201-710 10 mL: 170356 25 mL: 89130-900 50 mL: 75816-088 | VWR Serological Pipette, Non-Pyrogenic |
| Sterile aluminum transmission flow cell | Biosurface Technologies Corporation, Bozeman, MT, USA | FC 81-AL | Anodized aluminum single channel transmission flow cell with 96-well plate footprint for use with an inverted microscope |
| T75 Flasks | VWR, Radnor, PA, USA | 734-2316 | VWR Tissue Culture Flask, 182.5 cm, Surface treated, Plug seal cap, Sterile |
参考文献
- Randag, A. C., et al. The rising incidence of Acanthamoeba keratitis: A 7-year nationwide survey and clinical assessment of risk factors and functional outcomes. PLoS One. 14 (9), e0222092 (2019).
- Szentmary, N., et al. Acanthamoeba keratitis - Clinical signs, differential diagnosis and treatment. J Curr Ophthalmol. 31 (1), 16-23 (2019).
- Carnt, N., et al. Acanthamoeba keratitis: confirmation of the UK outbreak and a prospective case-control study identifying contributing risk factors. Br J Ophthalmol. 102 (12), 1621-1628 (2018).
- Verani, J. R., et al. National outbreak of Acanthamoeba keratitis associated with use of a contact lens solution, United States. Emerg Infect Dis. 15 (8), 1236-1242 (2009).
- Tu, E. Y., Joslin, C. E. Recent outbreaks of atypical contact lens-related keratitis: what have we learned. Am J Ophthalmol. 150 (5), 602-608 (2010).
- International Organization for Standardization. ISO 14729:2001/A1. Ophthalmic optics-Contact lens care products-Microbiological requirements and test methods for products and regimens for hygienic management of contact lenses. International Organization for Standardization. , (2010).
- . ASC Z80, Parent Committee Meeting Available from: https://www.thevisioncouncil.org/sites/default/files/ASCZ80_ParentCommitteeMinutes_February_27_2018_FINALMar19-2018.pdf (2023)
- Rayamajhee, B., et al. Acanthamoeba, an environmental phagocyte enhancing survival and transmission of human pathogens. Trends Parasitol. 38 (11), 975-990 (2022).
- Reverey, J. F., et al. Superdiffusion dominates intracellular particle motion in the supercrowded cytoplasm of pathogenic Acanthamoeba castellanii. Sci Rep. 5, 11690 (2015).
- Thapa, S., Lukat, N., Selhuber-Unkel, C., Cherstvy, A. G., Metzler, R. Transient superdiffusion of polydisperse vacuoles in highly motile amoeboid cells. J Chem Phys. 150 (14), 144901 (2019).
- Rudell, J. C., et al. Acanthamoeba migration in an electric field. Invest Ophthalmol Vis Sci. 54 (6), 4225-4233 (2013).
- Fukaya, S., Aoki, K., Kobayashi, M., Takemura, M. Kinetic analysis of the motility of giant virus-infected amoebae using phase-contrast microscopic images. Front Microbiol. 10, 3014 (2019).
- Fukaya, S., Takemura, M. Kinetic analysis of Acanthamoeba castellanii infected with giant viruses quantitatively revealed process of morphological and behavioral changes in host cells. Microbiol Spectr. 9 (1), 0036821 (2021).
- Cheng, H. J., Hsu, C. H., Hung, C. L., Lin, C. Y. A review for cell and particle tracking on microscopy images using algorithms and deep learning technologies. Biomed J. 45 (3), 465-471 (2022).
- Mathieu, E., et al. Time-lapse lens-free imaging of cell migration in diverse physical microenvironments. Lab Chip. 16 (17), 3304-3316 (2016).
- Svensson, C. M., Medyukhina, A., Belyaev, I., Al-Zaben, N., Figge, M. T. Untangling cell tracks: Quantifying cell migration by time lapse image data analysis. Cytometry A. 93 (3), 357-370 (2018).
- Piltti, K. M., et al. Live-cell time-lapse imaging and single-cell tracking of in vitro cultured neural stem cells - Tools for analyzing dynamics of cell cycle, migration, and lineage selection. Methods. 133, 81-90 (2018).
- Campolo, A., et al. Continuous real-time motility analysis of Acanthamoeba reveals sustained movement in absence of nutrients. Pathogens. 10 (8), 995 (2021).
- Campolo, A., Patterson, B., Lara, E., Shannon, P., Crary, M. Complete recovery of Acanthamoeba motility among surviving organisms after contact lens care disinfection. Microorganisms. 11 (2), 299 (2023).
- Crary, M. J., Walters, R., Shannon, P., Gabriel, M. M. Variables affecting the recovery of Acanthamoeba trophozoites. Pathogens. 10 (2), 221 (2021).
- . imagej Available from: https://imagej.net/plugins/trackmate/tutorials/getting-started (2024)
- Cherstvy, A. G., Nagel, O., Beta, C., Metzler, R. Non-Gaussianity, population heterogeneity, and transient superdiffusion in the spreading dynamics of amoeboid cells. Phys Chem Chem Phys. 20 (35), 23034-23054 (2018).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。