Method Article
Acanthamoeba spp.의 정량화 운동성
요약
이 절차에서는 Acanthamoeba spp 를 시각화, 추적 및 정량화하는 방법에 대해 설명합니다.운동성.
초록
아칸타메바 각막염은 치료에 어려움을 초래하고 실명으로 이어질 수 있는 심각한 안구 감염입니다. 어디에나 존재하며 물에 노출된 후 콘택트렌즈가 오염될 가능성에도 불구하고 이 병원체의 자연적인 행동은 여전히 파악하기 어렵습니다. 아칸타메바의 움직임 패턴을 이해하면 어떻게 콘택트렌즈에 서식하고 환자의 각막을 오염시키는지에 대한 정보를 얻을 수 있습니다. Acanthamoeba spp. 4x 배율에서 시작하는 명시야 현미경을 통해 볼 수 있습니다. 이전의 기술은 세포병증 효과 또는 전기장에 대한 노출 부족과 관련하여 Acanthamoeba 운동성을 정량화하기 위해 개발되었습니다. 여기에서는 Acanthamoeba spp 를 추적하고 정량화하는 방법을 설명합니다.Motility Long-term (Hours to Days)은 여러 아메바 균주, 표면 및 아메바의 영양 상태에 적용할 수 있는 프로토콜입니다. 이 절차는 감염, 증식 또는 행동 변화의 여러 단계를 모니터링하는 데 필요한 거리, 속도, 제한 및 방향성과 같은 많은 핵심 운동성 정량화를 결정하는 데 필수적입니다.
서문
아칸타메바가 각막1에 부착된 후 발생하는 기생충 감염인 아칸타메바 각막염(AK)의 유병률이 증가함에 따라 최근 몇 년 동안 아칸타메바 연구가 급격히 증가했습니다. AK의 발병은 부적절한 콘택트렌즈관리 또는 비효율적인 콘택트렌즈 관리 솔루션에 기인할 수 있지만2,3,4,5 현재 시장에 나와 있는 모든 제품에 대해 Acanthamoeba 소독 효능을 입증하기 위한 요구 사항은 없습니다. 그러나 과학계와 표준 기구 내에서는 콘택트렌즈 관리 제품의 소독 효능을 정량화하는 데 필요한 프로토콜을 검토하기 위한 지속적인 노력이 이루어지고 있다 6,7. 또한, 세포 및 기능적 측면이 인간 대식세포와 유사하기 때문에 아칸타메바는 아메바 자체가 가져오는 병원성 외에도 다른 인간병원체8를 숙주하고 전파하는 데 중요한 역할을 하는 것으로 알려져 있습니다.
세포병증 효과(cytopathic effect) 또는 전기장(9,11)에 대한 노출 부족과 관련하여 일반적으로 브라운 운동(Brownian movement)9,10에 크게 영향을 받지 않는 아칸타메바(Acanthamoeba) 운동성을 정량화할 수 있는 최근의 기술들이 설명되었으며, 아칸타메바를 추적 가능한 바이러스 벡터로 사용하는 거대 바이러스 연구의 운동성 분석의 발전도 이루어졌다(12). 13. 또한 지난 20년 동안 여기에 사용된 이미징 소프트웨어와 같은 새로운 소프트웨어 프로그램, 새로운 알고리즘 및 딥 러닝 기술을 사용하여 세포 및 입자 추적에서 지속적이고 상당한 개선이 이루어졌습니다14. 그러나 이 아메바는 벤치탑 연구, 임상 적용 및 산업 표준과 관련하여 비교적 새롭고 성장하는 과학 분야이며, 특히 콘택트렌즈 준수 후 또는 콘택트렌즈 소독 중 또는 후의 행동 변화를 정량화하기 위해 이 아메바를 시각화하고 추적하는 방법에 대해 발표된 데이터가 부족합니다. 장기 육안 모니터링으로 확장되는 다른 분야도 이러한 노력을 지원했습니다 15,16,17. 아칸타메바는 플라스미드(형광을 유발할 수 있음)에 대한 일반적인 내성, 표준 세포 염료를 섭취하고 파괴하는 아메바의 능력, 항체 태그 지정을 어렵게 만드는 독특한 외부 단백질 구성 등 본질적으로 까다로운 특성으로 인해 명시야 이미징 이외의 환경에서 다른 세포가 볼 수 있는 방법을 이 유기체에서 사용할 수 없었습니다. 따라서 이 아메바의 운동성을 정량화하는 것은 이 분야에 상당한 추가를 보여주었습니다. 본 명세서에 기술된 방법을 사용하여, 우리는 아메바가 영양분 없이 적어도 12시간 동안 운동성 상태를 유지한다는 것을 확인할 수 있었다18 그리고 소독 과정에 도전하여 소독 중에 운동성을 정지시킨 아메바는 완전히 용해되지 않은 경우 소독 후 운동성을 회복할 수 있다19.
이 프로토콜은 아메바의 운동성을 현미경으로 육안으로 추적하고 정량화하는 방법을 자세히 설명합니다. 주요 단계는 이미지 사이의 적절한 초점과 타이밍을 사용하여 명시야에서 아메바를 기록하고, 이미징 소프트웨어를 사용하여 이미지를 이진으로 변환한 다음, 이미징 소프트웨어의 추적 플러그인을 사용하여 실험 매개변수를 설정하고 각 아메바를 추적하여 속도, 거리 및 감금과 같은 필요한 측정값을 결정하는 것입니다. 그 후, 방향성을 정의하기 위해 아메바 또는 아메바 개체군의 화학주성을 정량화할 수 있습니다. 이 방법의 주요 기여는 영양 지원, 표면 부착, 소독 문제 또는 포유류 세포 배양과의 공존과 같은 기타 환경 변화의 다양한 상태 동안 아메바의 행동을 시각화하고 정량화하는 것입니다.
프로토콜
1. 아칸타메바 준비
참고: 이 프로토콜은 ATCC 50655, 30872, 50702, 30010, 30461, 50370, 50703, 30137, PRA-115 및 PRA-411에 대해 검증되었습니다. 이것은 BSL2 병원체이며 BSL2 후드 및 실험실에서 작업해야 합니다.
- T75 플라스크에 30mL의 AC6 배지를 충전하여 샘플 바이알 또는 준비된 플러그20 에서 마스터 배양액을 생성하고 하나의 플러그 또는 샘플 바이알의 내용물을 무균 방식으로 추가합니다. 플라스크가 50%에서 80%까지 합류할 때까지 26-30°C에서 3-5일 동안 플라스크를 배양합니다.
- 세포가 필요하기 전날 세포를 통과시켜 균질한 영양류 집단을 만듭니다.
- 균주의 필요에 따라 흔들어 주거나 마스터 배양액을 2번 세게 두드려 부착된 영양류를 제거합니다.
- T75 플라스크에 30mL의 AC6 배지를 채웁니다. 마스터 배양액 2mL를 통로에 추가합니다. 플라스크를 26-30 °C에서 18-24 시간 동안 배양합니다.
- 수확하기 전에 현미경으로 4배 확대하여 영양성 영양 생물 개체군을 육안으로 검사합니다. trophozoites가 부착되고 균일한지 확인하십시오.
- 플라스크를 세게 2번 두드려 부착된 영양류석을 제거합니다. 내용물을 50mL 원뿔형 튜브에 붓습니다.
- 균형 잡힌 원심분리기에서 500 x g 으로 튜브를 5분 동안 회전시켜 아메바를 펠릿화합니다. 상층액 매체를 붓거나 피펫팅하고 폐기합니다. 펠릿을 1/4 링거 용액 2-10mL에 희석합니다.
- 샘플을 소용돌이치게 합니다. 일회용 혈구계에 10μL의 샘플을 추가하고 Acanthamoeba의 CFU/mL를 계산합니다.
- 혈구계 수에 따라 아메바 펠릿을 1/4 링거 용액에 7.5 x 103 cells/mL의 농도로 희석합니다.
- 유리, 플라스틱 또는 비영양 한천일 수 있는 표면에 아메바를 종자하고 아래에 설명된 대로 실험적 요구 사항에 따라 다양한 우물 모양으로 만듭니다.
- 96웰 플레이트: 각 웰에 200μL의 Acanthamoeba 현탁액을 파종하고 플레이트를 뚜껑으로 둘러쌉니다.
- 48웰 플레이트: 각 웰에 1mL의 Acanthamoeba 현탁액을 파종하고 플레이트를 뚜껑으로 둘러쌉니다.
- 플로우 셀: 챔버 내 기포를 방지하여 멸균 알루미늄 플로우 셀의 멸균 포트를 통해 4mL의 Acanthamoeba 현탁액을 천천히 추가합니다. Clamp 서스펜션이 추가된 후 닫힌 포트.
- 이미징하기 전에 현미경 기록을 시작하기 전에 아메바가 표면에 최소 30분 동안 부착되도록 하십시오.
2. 아칸타메바 시각화 및 기록
- 명시야에서 4x 배율로 아메바를 시각화합니다. 배경은 밝은 회색으로 나타나야 하고 아메바는 검은색이어야 합니다. 아메바가 반투명이 아닌 단색의 다크서클이 되도록 빛과 초점을 조정합니다(따라서 초점이 약간 맞지 않음, 그림 1).
참고: 추적 중에 시야를 벗어나거나 시야에 들어오는 아메바는 데이터 분석에 포함되어서는 안 되므로 더 낮은 배율이 일정 기간 동안 분석에 포함된 최대 아메바 수를 가장 잘 지원합니다. 아메바는 추적 분석을 위해 이진법으로 변환하기 위해 명시야에서 단색 검은색 원으로 나타나야 합니다. 이것은 낮은 배율에서 가장 쉽게 얻을 수 있는데, 높은 배율은 아메바의 반투명한 특성과 내부 구조를 쉽게 드러내기 때문입니다. 일반적으로 더 낮은 배율의 단일 시야에서 더 많은 아메바를 캡처할 수 있어 보다 강력한 데이터 분석을 지원합니다. - 아메바를 추적하기 위해 일정한 간격으로 이미지를 녹화하도록 이미징 소프트웨어의 녹화 프로그램을 설정합니다. 정확한 추적이 가능한 이미지 사이의 가장 긴 시간은 30초이며, 파일 크기 및 추적 세부 사항에 대한 최적의 시간은 12초 또는 24초로 제안됩니다.
- 아메바는 한 번에 여러 일 동안 추적할 수 있지만 이러한 종류의 녹화로 인해 생성되는 파일 크기와 매우 큰 파일을 조작하는 데 내재된 어려움에 유의해야 합니다. 이를 피하려면 특정 간격으로 특정 시간 섹션에 대한 아메바를 기록하십시오. 예를 들어 5일 동안 12시간마다 1시간을 기록합니다.
- 현미경과 프로그램이 허용하는 경우, 플레이트 웰의 XY 좌표 또는 플로우 셀의 여러 위치를 사용하여 단일 세션에서 여러 웰을 기록할 수 있습니다. 필요한 경우 하나의 웰 또는 플로우 셀의 여러 섹션을 스티칭하여 4배 배율에서 더 큰 시야를 생성합니다.
참고: 기록할 수 있는 위치 수에 대한 유일한 제약 조건은 현미경 stage가 이미지 간격 시간 내에서 위치 사이를 얼마나 빨리 이동할 수 있는지입니다(예: 12초, 24초 등마다 이미지 촬영). 그러나 이는 필수는 아니며 단일 위치에서 만든 추적 비디오에는 강력한 통계 분석을 위한 충분한 수의 추적이 있을 수 있습니다. 이 연구에는 Nikon Eclipse Ti-U 현미경이 사용되었으며 자동화된 이동식 스테이지를 사용하여 한 번에 여러 웰의 이미지를 기록했습니다. 그러나 프로그래밍 가능한 기록 기능이 있는 모든 현미경이 작동합니다. 현미경은 컴퓨터에 연결되어야 하며 프로그램이나 하드 드라이브에 이미지를 기록할 수 있어야 합니다.
3. 아메바 크기 분석
- 이미징 소프트웨어에서 현미경 파일을 엽니다. Bio-formats 가져오기 옵션이 열립니다. 대화 상자 내에서 다음 사항이 true인지 확인하십시오. 스택 보기; 보기 대상: 선택-하이퍼스택; 메모리 관리: 가상 스택 사용을 선택합니다. 컬러 모드: 기본값.
- 다른 드롭다운 메뉴나 확인란이 활성화되어 있지 않은지 확인합니다. 딸깍 하는 소리 Ok.
- 바이오 형식 시리즈 옵션을 엽니다. 필요한 시리즈를 선택하십시오. 한 번에 하나의 위치만 기록되는 경우 여기에는 하나의 옵션만 있을 수 있습니다. 딸깍 하는 소리 Ok.
- [이미지] > [복제]를 선택하면 하나의 C 채널과 하나의 Z 채널만 선택하여 한 번에 작업 중인 단일 웰을 복제할 수 있습니다.
- 이제부터는 원본이 아닌 조작 및 분석을 위해 복제된 이미지로만 작업하십시오. 이미지 > 유형을 8비트> 선택합니다. [프로세스] > [배경 빼기]를 선택합니다.
- 롤링 볼을 10.0으로 설정합니다. 밝은 배경을 확인하십시오. 슬라이딩 포물면을 확인하십시오.
- [프로세스]> [대비를 향상시키다]를 선택합니다. 포화 픽셀을 trophozoites의 경우 .1%로 설정하고 셀 및 응집체 그룹의 경우 0.3%로 설정합니다.
- 이미지를 선택하고 임계값(기본값 > 흑백)> 조정합니다> Make Binary 프로세스가 열립니다. Default를 선택하고 Black background (of Binary Masks)를 선택합니다.
- 대부분의 배경 반점은 보이지 않지만 각 아메바의 일부는 볼 수 있도록 임계값을 적극적으로 선택합니다.
- 이미징 소프트웨어가 이미지를 반전시켰고 배경이 흰색이고 아메바가 검은색인 경우 편집 > 반전 으로 이동하여 검은색 배경과 흰색 아메바로 수정합니다.
- Process(프로세스> Binary > Close를 선택합니다. 세포의 초점이 맞지 않거나 세포의 운동성이 어긋나 외막에 약간의 공간이 있는 경우 이를 선택합니다.
- [Process > Binary >]를 선택하여 구멍을 채웁니다. 아메바가 아닌 아티팩트는 도형 그리기 도구와 Edit > Fill을 사용하여 제거합니다. 이 시점에서 배경이 흰색이고 아메바가 검은색이면 [편집] > [반전]을 선택합니다. 이 시점에서 이미지는 그림 2의 이진 그림을 나타내야 합니다.
- 각 아메바의 크기를 기록하려면 [분석] > [입자 분석]을 선택합니다. 세트 크기: 10-무한대, 원형도: 0-1, 표시: 없음. 결과만 표시하고 요약을 선택합니다.
- 표시되는 CSV 파일을 저장합니다. 파일 > Tiff> 다른 이름으로 저장 을 선택하고 이름을 원하는 사양으로 편집합니다.
4. 추적을 위한 현미경 파일 준비(운동성 분석)
- 이미징 소프트웨어에서 현미경 파일을 엽니다. Bio-Formats 가져오기 옵션이 열립니다.
- 대화 상자 내에서 다음 사항이 true인지 확인하십시오. 스택 보기; 보기 대상: 선택-하이퍼스택. 메모리 관리의 경우 Use Virtual Stack(가상 스택 사용)을 선택합니다. 딸깍 하는 소리 Ok.
- Bio-Formats 시리즈 옵션이 열립니다. 필요한 시리즈를 선택하십시오. 한 번에 하나의 위치만 기록되는 경우 여기에는 하나의 옵션만 있을 수 있습니다. 딸깍 하는 소리 Ok.
- [이미지]> [복제]를 선택합니다. 이제부터는 원본 파일에서 작업하는 대신 비디오의 중복된 부분에서만 작업하십시오.
참고: 해당 시간에 추적 중인 비디오 섹션을 복제합니다. 이렇게 하려면 실험에 필요한 시간(분)에 해당하는 프레임을 알아야 합니다. 예를 들어, 1st hour를 추적하고 24 초마다 이미지를 촬영하려면 1sthour에 150 프레임이 있어야합니다. 따라서 1번째 시간은 프레임 1-150, 2번째 시간은 151-300 등이 됩니다. - 8비트> 이미지 > 유형을 선택합니다. [프로세스] > [배경 빼기]를 선택합니다. 롤링 볼을 10.0으로 설정합니다.
- 밝은 배경을 확인하십시오. 슬라이딩 포물면을 확인하십시오. 프로세스 > Enhance Contrast를 선택하고 0.1%로 설정합니다.
- 모든 x# 조각(예: 150개 조각 모두)을 선택합니다. Normalize를 선택 취소합니다. 이미지를 선택하고 임계값을 조정> >(기본값 > 흑백)합니다.
- Make Binary 프로세스가 열립니다. 기본값을 선택합니다. (바이너리 마스크의) 검은색 배경을 확인합니다.
- 임계값을 공격적으로 설정하면 대부분의 배경 반점은 보이지 않지만 아메바의 일부는 볼 수 있습니다. 이 단계에서 배경이 흰색이고 아메바가 검은색이면 [편집] > [반전]을 선택합니다. 배경은 검은색이어야 하고 아메바는 흰색이어야 합니다.
- [Process] > [Binary >]를 선택합니다. [Process > Binary >]를 선택하여 구멍을 채웁니다. 파일 > Tiff> 다른 이름으로 저장을 선택하고 이름을 원하는 사양으로 편집합니다. 이제 파일을 추적할 준비가 되었습니다.
5. 트래킹을 통한 운동성 분석
- 트래킹에 필요한 tiff 파일이 있는 이미징 소프트웨어에서 플러그인 > 트래킹 > 트랙메이트로 이동합니다. Trackmate 버전이 열립니다.
- 다음(Next)을 선택합니다. 탐지기 드롭다운 메뉴에서 LoG 탐지기를 선택합니다. 예상 블롭 직경 설정: 35.0미크론, 임계값 1.0. Use median filter(중앙값 필터 사용) 및 Do sub-pixel localization(하위 픽셀 지역화 수행)을 선택 취소합니다.
- 첫 번째, 중간 및 끝 슬라이스에서 미리보기 를 클릭합니다. 모든 아메바가 보라색 원으로 캡처되고 배경 결함이나 아티팩트가 포함되지 않았는지 확인합니다.
- 다음(Next)을 선택합니다. 처리가 시작되고 이 단계를 수행하는 데 몇 분 정도 걸릴 수 있습니다. 화면 상단의 감지 표시줄은 프로세스가 얼마나 완료되었는지 나타냅니다.
- 메시지가 표시되면 다음을 선택합니다. 초기 임계값 설정에서 아무 것도 선택하지 않고 다음을 다시 선택합니다. 뷰를 선택할 때 Hyperstack Displayer 를 선택합니다. 다음(Next)을 선택합니다.
- 스폿에 필터를 설정하고 아무 것도 선택하지 않고 다음을 선택합니다. 트래커를 선택하십시오. 이렇게 하려면 Simple LAP Tracker(단순 LAP 트래커) 대신 LAP Tracker(LAP 트래 커)를 선택합니다. 다음(Next)을 선택합니다.
- 프레임 간 연결에서 최대 거리를 40μm로 설정합니다. 트랙에서 세그먼트 갭 닫기(Segment gap closing)를 선택합니다. 갭 닫기 허용(Allow Gap Closing)을 선택합니다. 최대 거리를 100μm로 설정하고 최대 프레임 간격을 4로 설정합니다. 트랙 세그먼트 분할 또는 트랙 세그먼트 병합에 대해 아무 것도 선택하지 마십시오.
- 다음(Next)을 선택합니다. 추적을 실행하는 데 상당한 시간이 걸릴 수 있습니다. 완료되면 창 하단에 추적 완료 x s가 표시됩니다. 다음(Next)을 선택합니다.
- 추적이 완료되면 다음을 다시 선택합니다. 트랙에 필터 설정: 왼쪽 아래에 있는 + 기호를 선택하여 트랙의 지점 수에 대한 필터를 추가합니다. 프레임의 93% 이상에 대해 필터를 위(Above)로 설정합니다(예: 150개의 프레임에는 최소 140개의 스폿이 필요함).
알림: 이 소프트웨어의 일부 버전에서는 원하는 숫자에 도달하기 위해 선을 왼쪽이나 오른쪽으로 드래그하여 수행할 수 있는 최소 7% 미만과 같은 반대 항목을 선택해야 합니다. - 다음(Next)을 선택합니다. 디스플레이 옵션이 나타납니다. Trackmate XML 파일 또는 Tracks CSV 파일을 저장하기 전에 각 프레임의 트랙이 아메바가 움직이는 방식이 변형되거나 이상하지 않은지 확인합니다.
- 트랙을 삭제해야 하는 경우 TrackScheme을 클릭합니다. TrackScheme은 모든 프레임에서 아메바의 모든 트랙과 접점을 표시합니다.
참고: 트랙을 삭제할 수 있는 이유는 아메바가 녹화 중에 시야를 켜거나 벗었거나, 트랙이 아메바의 실제 경로와 비교하여 부적절한 모양이거나, 어떤 이유로 트랙 또는 아메바가 실험 매개변수에 따른 데이터에서 이상치인 경우입니다. - 아메바를 클릭하여 화면에서 초록색 지점을 강조 표시하고 Trackscheme에서 해당 지점을 둘러싼 녹색 사각형이 있습니다. 이것은 어떤 트랙이나 지점을 제거해야 하는지 나타냅니다.
- 제거해야 하는 트랙을 마우스 오른쪽 버튼으로 클릭하고 Whole Track을 선택합니다. 삭제 버튼을 누릅니다.
- 모든 트랙을 검토한 후 추적 소프트웨어 팝업의 왼쪽 하단에 있는 저장 을 클릭하여 추적 파일을 저장합니다. 비디오 이름을 사용하여 XML 파일을 저장합니다. 재개 를 클릭하여 추적 소프트웨어 팝업으로 돌아갑니다.
- 트랙을 클릭하고 왼쪽에서 트랙을 선택합니다. CSV로 내보내기를 클릭하고 CSV 파일을 저장합니다.
- 스프레드시트가 결과 CSV를 인식하지 못하는 경우 다음 단계를 실행합니다.
- 메모장에서 저장된 CSV 파일을 엽니다. Ctrl + A 및 Control + C 를 클릭하여 모든 내용을 복사합니다.
- 스프레드시트를 열고 Control + V 를 클릭하여 스프레드시트에 붙여넣습니다. 모든 데이터가 하나의 셀에 있습니다.
- 데이터가 있는 셀을 선택한 상태에서 Data > Text to Columns로 이동합니다. [Delimited > Next]를 선택합니다. [다음> [마침]> 쉼표를 선택합니다. 이 파일을 새 CSV로 저장합니다.
- 추적 CSV에는 분석에 사용할 수 있는 많은 매개변수가 있습니다(그림 3). 원하는 매개변수를 복사하여 새 CSV 파일(그림 4)에 붙여넣어 분석합니다. 여기에 사용된 매개변수는 아래에 설명되어 있습니다.
- 총 이동 거리: 아메바가 이동한 총 거리(μm)입니다.
- 최대 이동 거리: 시작점에서 아메바의 경로상에 있는 최대 거리 - 반드시 끝점이 아닐 수도 있습니다.
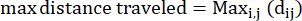
여기서 dij는 트랙의 모든 지점 j에서 모든 지점 i까지의 거리입니다. - 감금 비율: 감금 비율은 아메바가 시작 지점에서 직선으로 이동했는지(1에 가까운 값) 또는 시작 지점에 상대적으로 가깝게 머물렀는지(0에 가까운 값)를 나타냅니다.
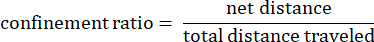
- 추적 평균 속도: 총 이동 거리 동안 아메바의 평균 속도(초당 미크론)입니다.
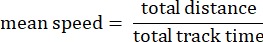
- 트랙 변위: 아메바의 시작점과 끝점 사이의 거리(미크론)입니다.
참고: 모든 이미징 소프트웨어 분석 매개변수와 그 정의는 여기에서 찾을 수 있습니다: https://imagej.net/plugins/trackmate/analyzers/. 저장된 각 파일은 해당 시점에 분석된 트랙의 요약임을 기억하십시오. 강력한 데이터 분석을 생성하려면 여러 반복실험, 시간 등의 결과를 데이터에 적합하게 적절하게 결합해야 합니다(그림 5 및 그림 6).
결과
이 방법을 성공적으로 사용하려면 고려해야 할 몇 가지 중요한 전체 요소가 있습니다. 첫 번째는 아메바와 현미경의 물리적 설정이고, 두 번째는 이미징 소프트웨어의 적절한 활용이며, 세 번째는 이미징 데이터를 의미 있는 방식으로 내보내고 분석하는 것입니다.
현미경 기록이 시작되기 전에 아메바가 현미경 스테이지에 적절하게 초점을 맞추는지 확인하는 것이 중요합니다(그림 1). 개별 세부 사항을 위해 가장 최적의 Z 평면에서 아메바에 초점을 맞추는 것보다는, 여기서는 아메바를 매우 약간 초점에서 벗어나 자세히 볼 때 반투명 상태와 대조적으로 실선 검은 점이 되도록 보는 것이 이상적입니다. 이러한 방식으로 아메바를 보면 이미징 소프트웨어와 추적 소프트웨어가 파일을 이진수로 변환할 때 각 개별 아메바를 훨씬 더 쉽게 찾을 수 있습니다(그림 2). 이미징 소프트웨어의 목표가 무엇인지, 즉 각 아메바가 잘 정의된 흰색 원으로 표시되는 것이 무엇인지 알면 독자가 명시야에서 원본 이미지를 촬영할 때 적절한 초점을 찾는 데 도움이 될 수 있습니다. 그런 다음 각 원이 명확하게 정의되면 추적 소프트웨어는 각 아메바의 운동성을 적절하게 추적하고 각 아메바에 대한 개별 운동성 메트릭을 수집할 수 있습니다.
이미징 소프트웨어 및 추적 소프트웨어에 대해 위에 나열된 프로토콜의 단계별 지침을 따르고 이미징 소프트웨어 사용의 중요한 첫 번째 단계를 지원하기 위해 관련 비디오를 참조한 후에는 정량화 및 데이터 분석을 위해 이미징 소프트웨어 데이터를 내보낼 차례입니다. 추적 소프트웨어의 원시 출력은 그림 3에서 볼 수 있는 것과 유사합니다. 여기서 내보낸 트랙의 스폿 수가 예상한 것에 맞는지 확인하는 것이 중요합니다(예: 모든 트랙에 프레임의 93%가 있도록 요청하고 비디오에 총 100개의 프레임이 있는 경우 내보낸 모든 트랙은 93개 이상의 스폿을 가져야 합니다). 스폿이 요구 사항과 일치하지 않으면 필요한 매개변수가 올바르게 설정되었는지 확인하기 위해 추적 소프트웨어 단계를 다시 수행해야 할 수 있습니다.
원시 내보내기에서 데이터 분석 시트 작성을 시작합니다(그림 4). 모든 트랙은 단일 복제 내에서 단일 아메바를 나타낸다는 것을 기억하는 것이 중요합니다. 따라서, 그림 4, 그림 5 및 그림 6에서 볼 수 있듯이, 프레임셋 또는 시점에 대한 모든 트랙은 평균화되어야 하며(그림 4), 이 프로세스는 전체 시리즈의 모든 프레임 세트 또는 시점에 대해 수행되어야 합니다(즉, 총 3시간의 비디오가 촬영되고 데이터 포인트가 시간으로 나뉘어진 경우, 그런 다음 반복당 분석해야 할 3개의 프레임 세트/시간이 있습니다( 그림 5). 마지막으로, 각 반복실험에 설정된 각 프레임의 평균 조합에서 각 반복실험의 평균 데이터를 하나의 공간으로 결합해야 여러 반복실험의 데이터를 분석하여 시점별 실제 평균 및 표준 편차를 결정할 수 있습니다(그림 6).
각 반복실험의 평균은 데이터 분석에 사용되며, 모든 반복실험의 결합된 평균과 표준 편차 또는 표준 오차는 그래픽 표현에 사용됩니다( 그림 7의 예에서 볼 수 있음). 여기에서 수행한 것처럼 여러 시점에 걸쳐 여러 샘플을 추적하는 데이터를 분석하는 경우 사후 Tukey의 테스트와 함께 2방향 반복 측정 ANOVA를 사용하여 데이터를 분석하는 것이 가장 적합합니다. 이를 통해 모든 시점에서 샘플 간의 차이를 결정할 수 있으며 시간 경과에 따른 각 샘플 내의 차이를 결정할 수도 있습니다.

그림 1: Amemiba는 brightfield microscopy에서 불투명하고 어두운 형태로 나타납니다. 아메바의 대표 이미지를 4배 확대하여 초점이 약간 맞지 않아 단색 다크서클로 보입니다. 스케일 바 = 500 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
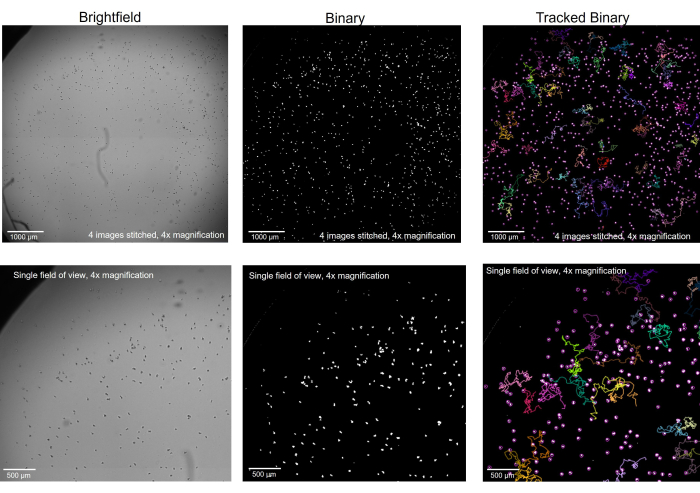
그림 2: brightfield microcopy에서 이미지화한 Amemiba를 binary로 변환하여 추적한 결과. 명시야(왼쪽)에서 4배 배율로 촬영한 아메바는 아티팩트가 제거된 이진법으로 변환되었으며(가운데), 추적 후 일부 트랙이 표시되었습니다(오른쪽). 맨 윗줄: 4개의 이미지를 함께 연결하여 이미지화한 전체 웰, 스케일 바 = 1000 μm. 맨 아래 줄: 스티칭 전 단일 이미지의 크기, 스케일 바 = 500μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
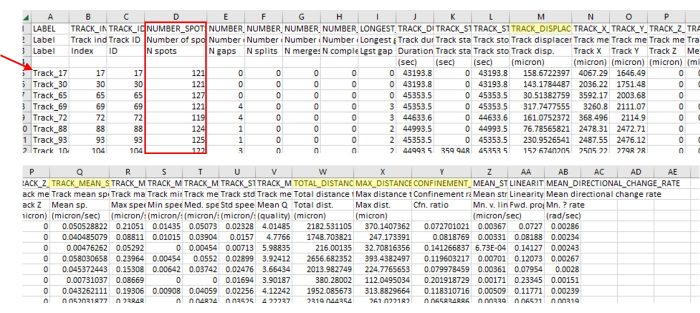
그림 3: 이미징 및 추적 소프트웨어의 즉각적인 데이터 분석 출력에는 다양한 데이터 유형이 포함되어 있습니다. 트래킹의 대표적인 출력은 단일 비디오인 그림 2에 나와 있습니다. 트랙 번호(빨간색 화살표)는 숫자를 건너뛰는데, 이는 추적 소프트웨어가 매개변수와 정렬되지 않은 트랙을 올바르게 제거했음을 나타냅니다. 이 예에서는 5385개 중 71개만 매개변수와 정렬된 트랙을 식별했습니다. 이 비율은 엄격한 지침을 감안할 때 정상입니다. 이 트랙의 수는 이 프로토콜에 사용된 아메바의 수와도 일치합니다(예를 들어, 96웰 플레이트에 웰당 7.5 x 103CFU /mL의 200μL가 파종된 경우 웰에 약 1,500개의 아메바가 있고 4-스티치 시야에서 대략 그만큼의 아메바를 볼 수 있습니다. 아메바보다 훨씬 더 많은 발자국이 있을 가능성이 높으며, 아메바는 시야 안팎을 걸으며 새로운 발자국을 생성합니다. 이 트랙 번호는 그림 2에서 다시 한 번 분명하게 나타나며, 많은 수의 스폿에 비해 몇 개의 트랙만 볼 수 있습니다. 항상 트랙의 지점 수(빨간색 상자)를 확인하여 올바른 트랙 길이만 포함되었는지 확인하십시오. 예를 들어, 여기서는 프레임의 93% 이상을 포함하고 이 섹션에는 127개의 프레임이 있으므로 모든 스폿 수는 118 이상이어야 합니다. 노란색으로 강조 표시된 것은 프로토콜에 언급된 측정값입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
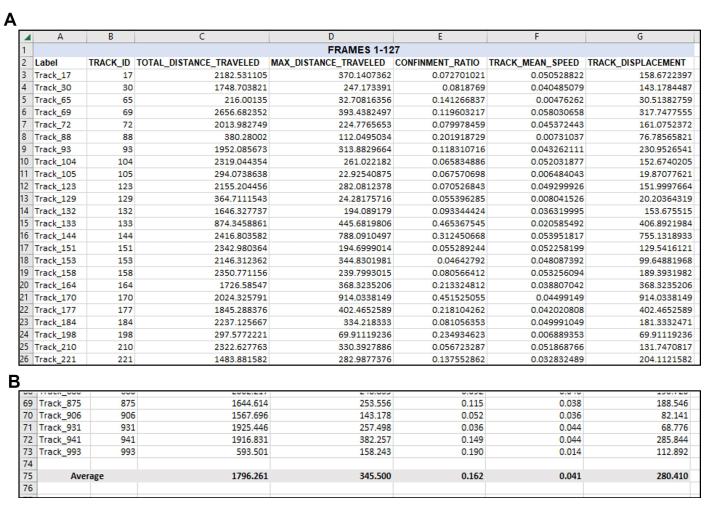
그림 4: 초기 데이터 출력 후 필요에 따라 재구성된 데이터. 내보내기 후 원하는 데이터의 구성. (A) 정량화하기 위해 선택한 관련 측정값은 추적 소프트웨어 내보내기(그림 3)에서 복사하여 새 스프레드시트에 붙여넣었습니다. (B) 원하는 측정값을 새 스프레드시트에 구성한 후 각 트랙의 평균을 계산해야 합니다. 따라서, 이 대표적인 예에서, 이 단일 비디오에서 사용 가능한 모든 트랙의 총 거리, 최대 거리, 구속, 평균 속도 및 변위는 각 측정에 대해 단일 숫자로 평균화되었습니다. 총 71개의 트랙이 프레임 1-127의 매개변수를 충족했다는 점에 다시 한 번 유의하십시오. 그림 5에서 볼 수 있듯이 분석된 각 비디오 또는 프레임 세트에서 적합한 트랙의 수가 다를 수 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
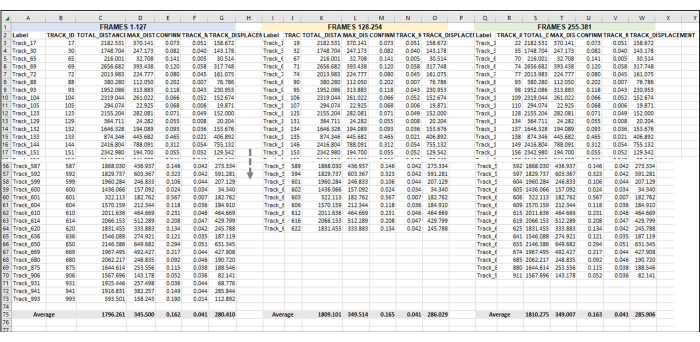
그림 5: 전체 이미지 시리즈 또는 비디오의 분석을 위해 설정된 순차 프레임. 데이터 분석에 순차적 비디오가 추가된 대표적인 이미지입니다. 프레임 1-127이 비디오의 1시간을 구성한다고 가정합니다(여기서 1시간은 28초마다 한 번씩 이미지를 촬영하는 것으로 구성됨). 프레임 128-254는 비디오의 2시간이고 프레임 255-381은 비디오의 3시간입니다. 모든 시간에서 내보낸 데이터( 그림 3 참조)를 하나의 시트로 결합하여 한 번에 모두 작업할 수 있습니다. 그림 4에서와 같이 각 프레임 세트/h에 대해 사용 가능한 모든 트랙의 데이터가 평균화됩니다. 그림 5 의 모든 것은 하나의 반복실험 또는 하나의 샘플에서 얻은 데이터입니다. 3개의 반복실험이 있는 경우 그림 6과 같이 3개의 동일하게 배치된 별도의 스프레드시트 탭이 있고 이러한 탭의 데이터는 새 탭에 공급됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
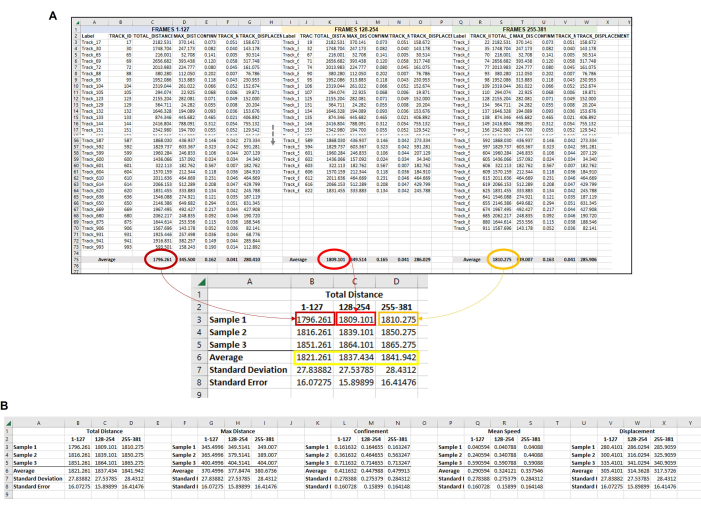
그림 6: 단일 복제를 나타내는 데 사용되는 각 프레임셋의 모든 아메바의 복합 데이터. (A) 그림 5 에 설정된 각 프레임의 모든 평균은 최종 데이터 분석을 완료할 수 있도록 중앙 위치에 수집되어야 합니다. 이 대표 이미지에는 처음 세 프레임 세트(또는 시간)의 총 거리 평균이 표시됩니다. 실행 중인 각 샘플/조건/반복실험에 대해 이 작업을 반복해야 합니다. 그런 다음 각 프레임 세트에서 각 샘플의 평균을 평균해야 합니다(노란색 상자). 이것은 표준 편차와 표준 오차를 계산할 수 있는 최종 데이터 포인트입니다. 각 샘플의 개별 평균(여기서는 총 거리 샘플 1에 대한 B3, B4 및 B5의 숫자; C3, C4 및 C5(총 거리 샘플 2 등) 통계 분석을 실행하는 데 사용됩니다. 이 프로세스는 필요한 모든 측정(최대 거리, 구속, 평균 속도, 변위 등)에 대해 반복됩니다. (B) 정량화를 그래픽으로 표현하려면 노란색 상자의 숫자를 최종 그래프에 사용할 데이터 포인트로 사용합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
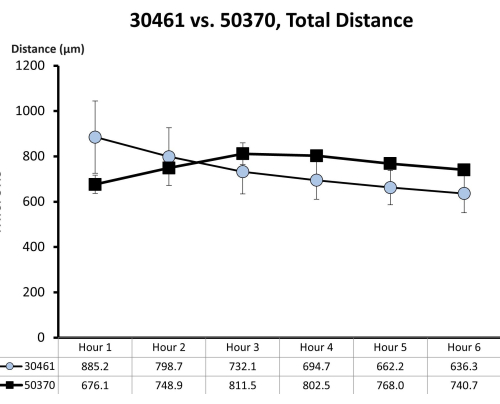
그림 7: ATCC 30461 및 ATCC 50370에 대한 원시 운동성 데이터. 원시 데이터(여기서는 각 시간에 대한 평균 ± SE로 표시)는 이전에 수집되었으며 선행 연구를 위해 게시되지 않았습니다18,19. 이 운동성 정량화는 이 두 아메바가 유리 표면의 1/4 링거 용액에서 처음 6시간의 운동성(해당 시간 동안 이동한 미크론 단위의 총 거리로 측정)이 유사하다는 것을 보여줍니다. 각 시점은 각각 20-200개의 아메바 트랙으로 구성된 세 개의 개별 복제를 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
저속 브라운 운동(low-velocity Brownian movement)9,10에 영향을 받지 않는 부착성 미생물인 아칸타메바(Acanthamoeba)와 같은 아메바의 운동성을 추적하고 정량화할 수 있게 되면 아메바의 행동에 대한 상당한 양의 정보를 얻을 수 있으며 AK 예방 방법에 대한 깨달음을 크게 높일 수 있습니다. 이 프로토콜은 최근 간행물에 상세히 기술되어 있으며, 데이터는 다른 유사한 데이터 분석에 의해 확인되거나 결합되었다(18,19). 여기서 우리는 이 방법을 사용하여 자세히 설명된 아메바의 속도가 다른 그룹에서 발표한 것과 유사하다는 점에 주목합니다. 이 프로토콜은 기본적으로 빛에 투명한 모든 표면 또는 아메바 처리에 사용할 수 있는 능력으로 유명하지만, 이 프로토콜이 잠재적으로 다른 유기체와 함께 작동하도록 수정될 수 있지만 지금까지는 Acanthamoeba에 대해서만 최적화되었다는 점에 유의하는 것이 중요합니다. 위의 프로토콜은 실험실에서 일관되게 잘 작동했지만 다른 요구 사항에 맞게 방법을 변경하거나 어려운 상황을 해결하기 위해 다음과 같은 변경 사항이 제공됩니다.
추적 매개변수 결정: 위의 프로토콜에서는 프레임 간 연결, 갭 닫기, 최대 프레임 갭 및 트랙의 스폿 수가 나열되었으며, 이는 최근 출판물에 가장 적용 가능한 것입니다. 그러나 다른 매개 변수가 다른 요구 사항에 더 적합할 수 있습니다. 또한 다른 사람들이 사용하고 싶지만 여기에 사용되지 않은 매개변수가 있을 수 있습니다(예: 트랙 필터 아래의 트랙의 최대 거리). 각 매개변수에 대한 자세한 내용과 매개변수가 설명하는 내용은 여기 웹사이트에서 액세스할 수 있습니다(참조21 참조). 어떤 매개변수와 필터가 다른 실험과 가장 관련성이 높은지, 그리고 어떻게 설정해야 하는지 결정할 때 특정 프로젝트에 대해 통계적으로 합리적인 것이 무엇인지 생각해 보십시오. 예를 들어, 아메바가 시야에서 걸어 나갔다가 다시 시야로 들어오는 데이터를 원할까요? 아니면 전혀 움직이지 않았지만 여전히 트랙으로 태그가 지정된 아메바의 데이터를 유지하고 싶습니까? 그리고 이미지가 기록된 프레임의 수와 프레임 사이의 시간 길이에 대해 의미가 있는 것은 무엇입니까?
블롭 크기 변경: 표준 실험실에서 사용할 수 있는 최신 기술 및 소프트웨어를 기반으로 하는 소프트웨어 또는 현미경 설정으로는 시간이 지남에 따라 변화하는 아메바의 수를 집계에서 정확하게 추적하는 것이 매우 어렵다는 것을 발견했습니다. 블롭의 크기를 추적하고 그 크기를 그 안에 있는 유기체의 수와 연관시키려는 경우, 반복적인 실험 후에 생성된 표준 곡선을 사용하여 응집체의 크기를 수학적으로 예측합니다. 예를 들어, 웰당 8개, 16개, 32개, 125개, 250개, 500개, 1,000개, 2,000개 세포와 같은 다양한 수의 아메바가 있는 웰에 응집체를 파종하여 만들었습니다. 24시간 동안 각 웰의 다양한 시점에서 타임랩스 이미지가 생성되었습니다. 각 스페로이드(알려진 수의 아메바)는 2차원 영역에 대해 분석되었으며, 시간 경과에 따른 아메바 수 대 스페로이드 크기의 함수로 표준 곡선이 생성되었습니다. 이 실험은 생성된 모든 곡선의 적절한 표준 편차를 제공하기 위해 최소 세 번 반복되었습니다.
아메바의 방향성 결정: 특정 연구에 필요하지 않을 수 있지만 아메바의 방향성을 이해하는 데 도움이 될 수 있습니다. 이것은 치료 또는 실험의 화학주성 효과에 대한 데이터를 제공할 것입니다. 이 데이터는 시각적(그래픽) 및 정량적 데이터를 생성하는 데에도 사용할 수 있습니다. 이는 이미징 소프트웨어용 무료 플러그인인 Chemotaxis and Migration Tool을 통해 사용할 수 있습니다. 웹 사이트에서 응용 프로그램 가이드, 샘플 이미지 및 동영상과 함께 사용할 수 있습니다( 자료 표 참조).
상세한 운동 역학 : 다른 그룹들은 여기에서 논의 된 것 이상의 고도로 발전된 통계 분석을 통해 확산 역학 및 확산 궤적을 조사했습니다 9,10,22. 이는 사용자의 요구에 따라 고려될 수 있습니다.
여느 성공적인 방법과 마찬가지로 이 원고에 자세히 설명된 프로토콜은 일관성을 달성하기 위해 여러 차례의 문제 해결을 거쳤습니다. 초당 이미지를 촬영하여(또는 현미경이 할 수 있는 한 1초 미만일 수 있음) 추적 가능한 우수한 고품질 비디오를 만들 수 있지만, 이렇게 하면 작업하기 어려울 수 있는 매우 큰 파일이 생성됩니다. 이것은 최대 몇 분 동안 녹화되는 매우 짧은 기간의 비디오에만 현실적으로 적합합니다. 반대로, 몇 초마다 이미지를 촬영함으로써 아메바의 행동은 이 종의 속도에 따라 여전히 매우 추적 가능하며 몇 시간 또는 며칠에 걸쳐 실행 가능한 비디오를 만들 수 있다는 것을 발견했습니다. 이미지 사이의 최대 시간은 30초이며, 이는 이미징 소프트웨어로 아메바를 정확하게 추적하는 데 필요한 시간입니다. 사용자가 선택하는 시간 간격은 현미경이 이미지를 얼마나 빨리 기록할 수 있는지, 웰당 얼마나 많은 이미지가 필요한지, 각 간격에서 얼마나 많은 웰이 기록되는지에 대한 알려진 제약 조건을 사용하여 고려해야 합니다. 마찬가지로, 이 프로토콜에서 언급한 최대 변위, 허용 프레임 갭, 필요한 최소 프레임 등에 대한 매개변수는 본 연구실에서 시행착오를 통해 결정되었으며, 가장 완전한 아메바 트랙을 포함하는 트랙 정보를 생성하고 불완전한 트랙, 비디오 중간에 스테이지에 합류하거나 퇴장하는 아메바, 또는 두 개의 아메바가 트랙 중간에 만나 분리되는 것과 같은 이미징 소프트웨어 혼동으로 인해 생성된 오류로 인해 생성된 노이즈를 무시합니다. 이러한 종류의 오류와 불완전한 트랙은 서로 끊임없이 상호 작용하는 미세한 생물학적 유기체에 대한 매우 긴(몇 시간에서 며칠) 비디오를 만들 때 당연히 높으며 이것이 잘못된 트랙의 매우 많은 부분을 거부해야 하는 이유입니다. 이 실험실의 경험에 따르면 프로토콜의 구멍 채우기 단계는 이미징 소프트웨어가 아메바를 추적하는 방법의 오류를 줄이는 데 중요하다는 점에 유의해야 합니다. 각 아메바가 때때로 도넛 또는 C 모양 대신 실선 원인지 확인함으로써 소프트웨어는 각 아메바를 성공적으로 추적 할 수 있습니다.
또한, 논의된 바와 같이, 이미지 또는 비디오를 분석하기 위해 사용자가 사용할 수 있는 많은 매개변수가 있습니다. 실험적 요구 사항을 기반으로 총 거리, 최대 거리, 속도 및 변위 분석을 통해 지속적으로 가장 많은 이점을 얻었습니다. 이것들은 이전 출판물18,19에서 그래픽 해석과 함께 심층적으로 논의됩니다(다양한 변형 및 표면 사용 포함). 이 네 가지 매개변수를 통해 사용자는 아메바가 선형 거리를 이동할 수 있는 능력과 그렇게 하는 데 걸리는 시간을 추정할 수 있으며, 이는 콘택트렌즈 오염과 관련된 아메바의 행동을 이해하는 데 도움이 됩니다. 이러한 매개 변수를 검색하고 분석하는 것은 그림에 자세히 설명되어 있듯이 큰 작업입니다. 이미징 소프트웨어 출력에서 크고 수많은 스프레드시트로 작업하는 동안 필요한 모든 분석을 자동으로 계산하는 잠긴 스프레드시트 템플릿을 만들어 의도하지 않은 오류를 제한했습니다. 그러나 이 방법을 개선할 수 있는 방법은 이 데이터를 처리하고 정렬 및 분석할 수 있는 스크립트를 작성하는 것입니다.
결론적으로, 다양한 조건에서 Acanthamoeba 크기와 운동성을 측정하는 접근 가능하고 정확한 방법이 여기에 설명되어 있습니다. 우리는 이 방법이 다양한 아메바 균주에 적용될 수 있음을 보여주었으며 운동성 정보를 얻기 위한 간단한 매개변수가 있을 수 있지만 이 실험 설정은 특정 요구 사항에 맞게 고도로 맞춤화될 수 있음을 설명했습니다.
공개
모든 저자는 Alcon Research, LLC의 직원입니다.
감사의 말
이 연구는 Alcon Research, LLC의 자금 지원을 받았습니다.
자료
| Name | Company | Catalog Number | Comments |
| ¼ Ringer’s solution | ThermoFisher Scientific | BR0052G | Oxoid Ringers Solution Tablets. Follow directions to make one-quarter strength instead of full strength Ringers. |
| 10 µL pipette | Eppendorf Research | 3123000039 | 2 µL-20 µL single channel |
| 10 µL pipette tips | Neptune Scientific, San Diego, CA, USA | BT10.N | 10 µL Universal Barrier Tip |
| 48 well plate | Millipore Sigma, | CLS3548 | Corning Costar TC-Treated Multiple Well Plate: polystyrene plate, flat bottom wells, sterile, with lid |
| 50 mL conical tubes (1 for each sample, 1 for each pass 2 sample) | Fisher Scientific | Falcon 352098 | Falcon 50 mL High Clarity Conical Centrifuge Tubes, polypropylene |
| 96-well plate | Millipore Sigma, Burlington, MA, USA | CLS3596 | Corning Costar TC-Treated Multiple Well Plate: polystyrene plate, flat bottom wells, sterile, with lid |
| Acanthamoeba | American Type Culture Collection, Manassas, VA, USA | ATCC 30461 | This protocol has been verified for ATCC 50655, 30872, 50702, 30010, 30461, 50370, 50703, 30137, PRA-115, and PRA-411 |
| Axenic culture media (AC6) | Made in house | n/a | Containing 20 g biosate peptone, 5 g glucose, 0.3 g KH2PO4, 10 µg vitamin B12, and 1 glass5 mg l-methionine per liter of distilled deionized water. Adjust pH to 6.6-6.95 with 1 M NaOH and autoclave at 121 °C for 20 min, store at room temperature for up to 3 months. |
| Centrifuge and appropriate rotor | Thermo Scientific, Waltham, MA, USA | Sorvall ST 40R | Any equivalent centrifuge and rotor are acceptable so long as it can spin 50 mL conical tubes at 500 x g for 5 min |
| Chemotaxis and Migration Tool | Free software based on ImageJ, available at https://ibidi.com/chemotaxis-analysis/171-chemotaxis-and-migration-tool.html | ||
| Disposable hemocytometer | Bulldog Bio, Portsmouth, NH, USA | DHC-N01 | Neubauer Improved 2-Chip Disposable Hemocytometer, Individually packaged, Nonpyrogenic |
| ImageJ software with Trackmate plugin (this protocol written with Trackmate version 6.0.2.) | Free software developed by the National Institutes of Health, available at imagej.net. Trackmate plugin available at https://imagej.net/plugins/trackmate/ | ||
| Microscope, preferably with automated moveable stage | Nikon, Tokyo, Japan | A Nikon Eclipse Ti-U Microscope was used in this study and the automated moveable stage was utilized to be able to record images in multiple wells at a time. | |
| NIS-Elements software | Nikon | ||
| Serological pipette | Fisher Scientific, Hampton, NH, USA | BrandTech 26331 | BrandTech accu-jet pro Pipet Controller |
| Serological pipette tips | VWR | 5 mL: 76201-710 10 mL: 170356 25 mL: 89130-900 50 mL: 75816-088 | VWR Serological Pipette, Non-Pyrogenic |
| Sterile aluminum transmission flow cell | Biosurface Technologies Corporation, Bozeman, MT, USA | FC 81-AL | Anodized aluminum single channel transmission flow cell with 96-well plate footprint for use with an inverted microscope |
| T75 Flasks | VWR, Radnor, PA, USA | 734-2316 | VWR Tissue Culture Flask, 182.5 cm, Surface treated, Plug seal cap, Sterile |
참고문헌
- Randag, A. C., et al. The rising incidence of Acanthamoeba keratitis: A 7-year nationwide survey and clinical assessment of risk factors and functional outcomes. PLoS One. 14 (9), e0222092 (2019).
- Szentmary, N., et al. Acanthamoeba keratitis - Clinical signs, differential diagnosis and treatment. J Curr Ophthalmol. 31 (1), 16-23 (2019).
- Carnt, N., et al. Acanthamoeba keratitis: confirmation of the UK outbreak and a prospective case-control study identifying contributing risk factors. Br J Ophthalmol. 102 (12), 1621-1628 (2018).
- Verani, J. R., et al. National outbreak of Acanthamoeba keratitis associated with use of a contact lens solution, United States. Emerg Infect Dis. 15 (8), 1236-1242 (2009).
- Tu, E. Y., Joslin, C. E. Recent outbreaks of atypical contact lens-related keratitis: what have we learned. Am J Ophthalmol. 150 (5), 602-608 (2010).
- International Organization for Standardization. ISO 14729:2001/A1. Ophthalmic optics-Contact lens care products-Microbiological requirements and test methods for products and regimens for hygienic management of contact lenses. International Organization for Standardization. , (2010).
- . ASC Z80, Parent Committee Meeting Available from: https://www.thevisioncouncil.org/sites/default/files/ASCZ80_ParentCommitteeMinutes_February_27_2018_FINALMar19-2018.pdf (2023)
- Rayamajhee, B., et al. Acanthamoeba, an environmental phagocyte enhancing survival and transmission of human pathogens. Trends Parasitol. 38 (11), 975-990 (2022).
- Reverey, J. F., et al. Superdiffusion dominates intracellular particle motion in the supercrowded cytoplasm of pathogenic Acanthamoeba castellanii. Sci Rep. 5, 11690 (2015).
- Thapa, S., Lukat, N., Selhuber-Unkel, C., Cherstvy, A. G., Metzler, R. Transient superdiffusion of polydisperse vacuoles in highly motile amoeboid cells. J Chem Phys. 150 (14), 144901 (2019).
- Rudell, J. C., et al. Acanthamoeba migration in an electric field. Invest Ophthalmol Vis Sci. 54 (6), 4225-4233 (2013).
- Fukaya, S., Aoki, K., Kobayashi, M., Takemura, M. Kinetic analysis of the motility of giant virus-infected amoebae using phase-contrast microscopic images. Front Microbiol. 10, 3014 (2019).
- Fukaya, S., Takemura, M. Kinetic analysis of Acanthamoeba castellanii infected with giant viruses quantitatively revealed process of morphological and behavioral changes in host cells. Microbiol Spectr. 9 (1), 0036821 (2021).
- Cheng, H. J., Hsu, C. H., Hung, C. L., Lin, C. Y. A review for cell and particle tracking on microscopy images using algorithms and deep learning technologies. Biomed J. 45 (3), 465-471 (2022).
- Mathieu, E., et al. Time-lapse lens-free imaging of cell migration in diverse physical microenvironments. Lab Chip. 16 (17), 3304-3316 (2016).
- Svensson, C. M., Medyukhina, A., Belyaev, I., Al-Zaben, N., Figge, M. T. Untangling cell tracks: Quantifying cell migration by time lapse image data analysis. Cytometry A. 93 (3), 357-370 (2018).
- Piltti, K. M., et al. Live-cell time-lapse imaging and single-cell tracking of in vitro cultured neural stem cells - Tools for analyzing dynamics of cell cycle, migration, and lineage selection. Methods. 133, 81-90 (2018).
- Campolo, A., et al. Continuous real-time motility analysis of Acanthamoeba reveals sustained movement in absence of nutrients. Pathogens. 10 (8), 995 (2021).
- Campolo, A., Patterson, B., Lara, E., Shannon, P., Crary, M. Complete recovery of Acanthamoeba motility among surviving organisms after contact lens care disinfection. Microorganisms. 11 (2), 299 (2023).
- Crary, M. J., Walters, R., Shannon, P., Gabriel, M. M. Variables affecting the recovery of Acanthamoeba trophozoites. Pathogens. 10 (2), 221 (2021).
- . imagej Available from: https://imagej.net/plugins/trackmate/tutorials/getting-started (2024)
- Cherstvy, A. G., Nagel, O., Beta, C., Metzler, R. Non-Gaussianity, population heterogeneity, and transient superdiffusion in the spreading dynamics of amoeboid cells. Phys Chem Chem Phys. 20 (35), 23034-23054 (2018).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유