Method Article
Quantification d’Acanthamoeba spp. Motilité
Dans cet article
Résumé
Cette procédure décrit comment visualiser, suivre et quantifier Acanthamoeba spp. motilité.
Résumé
La kératite à Acanthamoeba est une infection oculaire grave qui pose des problèmes de traitement et peut entraîner la cécité. Malgré son omniprésence et la contamination potentielle des lentilles de contact après une exposition à l’eau, le comportement naturel de cet agent pathogène reste insaisissable. Comprendre les mouvements de l’Acanthamoeba peut nous renseigner sur la façon dont il colonise les lentilles de contact et contamine la cornée du patient. Il est heureux qu’Acanthamoeba spp. sont visibles par microscopie à fond clair à partir d’un grossissement de 4x. Des techniques antérieures ont été développées pour quantifier la motilité d’Acanthamoeba en ce qui concerne les effets cytopathiques ou la sous-exposition au champ électrique. Nous décrivons ici une méthode pour suivre et quantifier Acanthamoeba spp. motilité à long terme (heures à jours), qui est un protocole applicable à plusieurs souches d’amibes, surfaces et état nutritionnel des amibes. Cette procédure est pertinente pour déterminer de nombreuses quantifications de motilité de base, telles que la distance, la vitesse, le confinement et la directionnalité, qui sont nécessaires pour surveiller les différents stades de l’infection, de la prolifération ou du changement de comportement.
Introduction
La recherche sur Acanthamoeba a considérablement augmenté ces dernières années en raison de la prévalence accrue de la kératite à Acanthamoeba (AK), qui est une infection parasitaire suite à la fixation d’Acanthamoeba à la cornée1. Bien que les éclosions de KA puissent être attribuées à un entretien inapproprié des lentilles de contact ou à des solutions inefficaces pour l’entretien des lentilles de contact 2,3,4,5, il n’existe actuellement aucune exigence pour démontrer l’efficacité de la désinfection d’Acanthamoeba pour tout produit sur le marché. Cependant, la communauté scientifique et les organismes de normalisation s’efforcent actuellement d’examiner les protocoles nécessaires pour quantifier l’efficacité de désinfection des produits d’entretien des lentilles de contact 6,7. De plus, en raison de sa ressemblance cellulaire et fonctionnelle avec les macrophages humains, Acanthamoeba a été noté comme ayant un rôle important dans l’hébergement et la dissémination d’autres agents pathogènes humains8, en plus de la pathogénicité que l’amibe elle-même apporte.
Des techniques récentes ont été décrites qui ont permis de quantifier la motilité d’Acanthamoeba - qui n’est généralement pas très sujette au mouvement brownien 9,10 - en ce qui concerne les effets cytopathiques ou la sous-exposition au champ électrique 9,11, ainsi que les progrès de l’analyse de la motilité dans la recherche sur les virus géants utilisant Acanthamoeba comme vecteur viral traçable 12, 13. Planche à billets Au cours des 20 dernières années, le suivi des cellules et des particules s’est également amélioré de manière constante et significative à l’aide de nouveaux logiciels tels que le logiciel d’imagerie utilisé ici, ainsi que de nouveaux algorithmes et de technologies d’apprentissage profond14. Cependant, il s’agit d’un domaine scientifique relativement nouveau et en pleine croissance en ce qui concerne la recherche sur paillasse, les applications cliniques et les normes industrielles, et il y a eu une pénurie de données publiées concernant les méthodes de visualisation et de suivi de cette amibe, en particulier afin de quantifier les changements de comportement après l’adhésion aux lentilles de contact ou pendant ou après la désinfection des lentilles de contact. D’autres domaines s’étendant à la surveillance visuelle à long terme ont soutenu cet effort 15,16,17. En raison de la nature intrinsèquement difficile d’Acanthamoeba - y compris une résistance générale aux plasmides (qui pourraient conférer la fluorescence), la capacité de l’amibe à consommer et à détruire les colorants cellulaires standard et une composition protéique externe unique - rendant difficile le marquage des anticorps - les méthodes disponibles pour d’autres cellules qui les rendent visibles dans des contextes autres que l’imagerie en fond clair ont été inutilisables dans cet organisme. Ainsi, la quantification de la motilité de cette amibe a démontré un ajout significatif au domaine. En utilisant la méthode décrite ici, nous avons pu constater que les amibes restent mobiles pendant au moins 12 h sans nutriments18 et que les amibes qui sont confrontées à un processus de désinfection et cessent leur motilité pendant la désinfection peuvent retrouver leur motilité après la désinfection si elles ne sont pas complètement lysées19.
Ce protocole détaille comment suivre visuellement et quantifier la motilité des amibes au microscope. Les premières étapes consistent à enregistrer les amibes en fond clair en utilisant la mise au point et le timing appropriés entre les images, à transformer les images en binaires à l’aide d’un logiciel d’imagerie, puis à utiliser le plug-in de suivi d’un logiciel d’imagerie pour définir les paramètres expérimentaux et suivre chaque amibe pour déterminer les mesures requises telles que la vitesse, la distance et le confinement. Suite à cela, il est possible de quantifier la chimiotaxie d’une amibe ou d’une population d’amibes afin de définir la directionnalité. La principale contribution de cette méthode est de visualiser et de quantifier le comportement des amibes lors de différents états de soutien nutritionnel, d’adhérence de surface, de défi de désinfection ou d’autres altérations environnementales telles que la cohabitation avec la culture de cellules de mammifères.
Protocole
1. Préparation de l’Acanthamoeba
REMARQUE :Ce protocole a été vérifié pour les ATCC 50655, 30872, 50702, 30010, 30461, 50370, 50703, 30137, PRA-115 et PRA-411. Il s’agit d’un agent pathogène BSL2 qui doit être travaillé dans une hotte et un laboratoire BSL2.
- Créez une culture mère à partir d’un flacon d’échantillon ou d’un bouchon20 préparé en remplissant un flacon T75 de 30 ml de milieu AC6 et en y ajoutant de manière aseptique un bouchon ou le contenu d’un flacon d’échantillon. Incuber le ballon pendant 3 à 5 jours à 26-30 °C jusqu’à ce qu’il soit confluent à 50 % à 80 %.
- Passez les cellules la veille de leur besoin pour créer une population homogène de trophozoïtes.
- Selon les besoins de la souche, secouez et/ou frappez vivement la culture maîtresse 2x pour déloger les trophozoïtes collés.
- Remplissez une fiole T75 avec 30 mL de milieu AC6. Ajouter 2 mL de la culture maîtresse dans le passage. Incuber le ballon pendant 18-24 h à 26-30 °C.
- Avant la récolte, inspectez visuellement la population de trophozoïtes sous un grossissement de 4x au microscope. Assurez-vous que les trophozoïtes sont adhérents et uniformes.
- Frappez vivement le ballon 2x pour déloger les trophozoïtes collés. Verser le contenu dans un tube conique de 50 ml.
- Faites tourner le tube à 500 x g pendant 5 min dans une centrifugeuse équilibrée pour granuler l’amibe. Versez ou pipetez le milieu surnageant et jetez-le. Diluer la pastille dans 2 à 10 mL de solution 1/4 de Ringer.
- Vortex l’échantillon. Ajouter 10 μL d’échantillon dans un hémocytomètre jetable et compter les UFC/mL de l’Acanthamoeba.
- D’après le nombre de globules de l’hémocytomètre, diluer la pastille d’amibe à une concentration de 7,5 x 103 cellules/mL dans une solution de Ringer 1/4.
- Grainez l’amibe sur une surface, qui peut être en verre, en plastique ou en gélose non nutritive, et une variété de formes de puits en fonction des besoins expérimentaux, comme décrit ci-dessous.
- Plaque à 96 puits : Ensemencez chaque puits avec 200 μL de suspension d’Acanthamoeba et entourez la plaque avec le couvercle.
- Plaque à 48 puits : Ensemencer chaque puits avec 1 mL de suspension d’Acanthamoeba et entourer la plaque avec le couvercle.
- Cellule d’écoulement : Ajouter lentement 4 ml de suspension d’Acanthamoeba à travers les orifices stériles d’une cellule d’écoulement en aluminium stérile, en évitant les bulles dans la chambre. Serrez les ports fermés après l’ajout de la suspension.
- Avant l’imagerie, laissez les amibes adhérer à la surface pendant au moins 30 minutes avant de commencer l’enregistrement microscopique.
2. Visualisation et enregistrement d’Acanthamoeba
- Visualisez l’amibe à un grossissement de 4x en fond clair. L’arrière-plan doit apparaître gris clair et l’amibe doit être noire. Ajustez la lumière et la mise au point de manière à ce que les amibes soient des cercles sombres solides au lieu d’être translucides (donc, légèrement floues, Figure 1.).
REMARQUE : Le grossissement optimal est de 4x pour le suivi pour plusieurs raisons : Les amibes qui quittent ou entrent dans le champ de vision pendant le suivi ne doivent pas être incluses dans l’analyse des données, donc le grossissement le plus faible soutient le mieux le nombre maximum d’amibes incluses dans l’analyse au cours d’une période donnée. Les amibes doivent apparaître sous forme de cercles noirs solides en fond clair afin de les convertir en binaire pour l’analyse de suivi. C’est plus facile à réaliser avec un grossissement plus faible, car des grossissements plus élevés révèlent facilement la nature translucide et les structures internes de l’amibe. En général, il est possible de capturer plus d’amibes dans un seul champ de vision avec un grossissement plus faible, ce qui permet une analyse plus robuste des données. - Réglez les programmes d’enregistrement dans le logiciel d’imagerie pour enregistrer une image à intervalles réguliers afin de suivre l’amibe. Le temps le plus long entre les images qui permettra un suivi précis est de 30 s, le temps optimal pour la taille du fichier et les détails du suivi est suggéré à 12 s ou 24 s.
- Les amibes peuvent être suivies pendant plusieurs jours à la fois, mais soyez conscient de la taille des fichiers que ce type d’enregistrement créera et de la difficulté inhérente à la manipulation de fichiers extrêmement volumineux. Pour éviter cela, enregistrez les amibes pendant des périodes de temps à certains intervalles. Par exemple, enregistrez pendant 1 h toutes les 12 h pendant 5 jours.
- Si le microscope et le programme le permettent, enregistrez plusieurs puits en une seule session en utilisant les coordonnées XY des puits de plaque ou plusieurs emplacements d’une cellule d’écoulement. Si nécessaire, assemblez plusieurs sections d’un puits ou d’une cellule d’écoulement pour créer un champ de vision plus grand avec un grossissement de 4x.
REMARQUE : La seule contrainte sur le nombre d’emplacements pouvant être enregistrés est la vitesse à laquelle la platine du microscope peut se déplacer entre les emplacements dans l’intervalle de temps (par exemple, prendre une image toutes les 12 s, 24 s, etc.). Cependant, ce n’est pas obligatoire, et les vidéos de suivi réalisées à partir d’un seul endroit peuvent toujours avoir un nombre suffisant de traces pour une analyse statistique robuste. Un microscope Nikon Eclipse Ti-U a été utilisé dans cette étude, et la platine mobile automatisée a été utilisée pour enregistrer des images dans plusieurs puits à la fois. Cependant, tout microscope doté d’une capacité d’enregistrement programmable fonctionnera. Le microscope doit se connecter à un ordinateur et être capable d’enregistrer des images sur un programme ou un disque dur.
3. Analyse de la taille des amibes
- Ouvrez le fichier du microscope dans le logiciel d’imagerie. Les options d’importation de bioformats s’ouvriront. Dans la boîte de dialogue, assurez-vous que les conditions suivantes sont vraies : Affichage de la pile ; Voir avec : Sélection-Hyperstack ; Gestion de la mémoire : cochez Utiliser la pile virtuelle ; Mode couleur : Par défaut.
- Assurez-vous qu’aucun des autres menus déroulants ou cases à cocher n’est activé. Cliquez sur OK.
- Ouvrez les options de série des bioformats. Choisissez la série dont vous avez besoin. Si un seul emplacement est enregistré à la fois, il n’y aura probablement qu’une seule option ici. Cliquez sur OK.
- Choisissez Image > Dupliquer pour dupliquer le puits unique en cours d’utilisation à la fois en choisissant un seul canal C et un seul canal Z.
- À partir de là, ne travaillez qu’avec l’image dupliquée à des fins de manipulation et d’analyse, et non avec l’original. Choisissez le type de > d’image > 8 bits. Choisissez Traiter > Soustraire l’arrière-plan.
- Réglez la balle roulante sur 10,0. Vérifiez Fond clair. Vérifiez Paraboloïde coulissant.
- Choisissez Process > Enhance Contrast. Définissez les pixels saturés sur 0,1 % pour les trophozoïtes ou 0,3 % pour un groupe de cellules et d’agrégats.
- Choisissez Image > Ajuster > seuil (par défaut > N&B). Le processus Make Binary s’ouvrira. Sélectionnez Par défaut et cochez Fond noir (des masques binaires).
- Sélectionnez le seuil de manière agressive de sorte que la plupart des taches d’arrière-plan ne soient pas visibles, mais qu’une partie de chaque amibe soit visible.
- Si le logiciel d’imagerie a inversé l’image et que l’arrière-plan est blanc et que l’amibe est noire, allez dans Modifier > Inverser pour la fixer sur un fond noir et une amibe blanche.
- Choisissez Processus> Binaire > Fermer, au cas où les cellules auraient un léger espace dans la membrane externe en raison d’une mise au point ou d’une motilité de la cellule.
- Choisissez Traiter > binaire > remplir les trous. Supprimez les artefacts non amibes à l’aide des outils de dessin de forme et de Modifier > remplissage. Si, à ce stade, l’arrière-plan est blanc et l’amibe est noire, choisissez Modifier > Inverser. À ce stade, l’image doit représenter les images binaires de la figure 2.
- Pour enregistrer la taille de chaque amibe, choisissez Analyser > Analyser les particules. Taille de l’ensemble : 10-infini, Circularité : 0-1, Spectacle : rien. Cochez uniquement Afficher les résultats et Résumer.
- Enregistrez les fichiers CSV qui s’affichent. Choisissez Fichier > Enregistrer sous > Tiff et modifiez le nom selon les spécifications souhaitées.
4. Préparation des fichiers microscopes pour le suivi (analyse de motilité)
- Ouvrez le fichier du microscope dans le logiciel d’imagerie. Les options d’importation de Bio-Formats s’ouvriront.
- Dans la boîte de dialogue, assurez-vous que les conditions suivantes sont vraies : Affichage de la pile ; Vue avec : Selection-Hyperstack. Pour la gestion de la mémoire, cochez Utiliser la pile virtuelle. Cliquez sur OK.
- Les options de la série Bio-Formats s’ouvriront. Choisissez la série dont vous avez besoin. Si un seul emplacement est enregistré à la fois, il n’y aura probablement qu’une seule option ici. Cliquez sur OK.
- Choisissez Image > Dupliquer. À partir de là, ne travaillez que sur la section dupliquée de la vidéo au lieu de travailler sur le fichier d’origine.
REMARQUE : Dupliquez la section de la vidéo en cours de suivi à ce moment-là. Pour ce faire, il faut connaître les images qui correspondent aux minutes nécessaires à l’expérience. Par exemple, si l’on veut suivre la 1èreheure et prendre des images toutes les 24 s, il devrait y avoir 150 images dans la 1èreheure. Par conséquent, la 1èreheure sera les images 1-150, la 2èmeheure sera 151-300, et ainsi de suite. - Choisissez le type de > d’image > 8 bits. Choisissez Traiter > Soustraire l’arrière-plan. Réglez la balle roulante sur 10,0.
- Vérifiez Fond clair. Vérifiez Paraboloïde coulissant. Choisissez Processus > Améliorer le contraste et réglez-le sur 0,1 %.
- Cochez Toutes les tranches x# (par exemple, toutes les 150 tranches). Décochez la case Normaliser. Choisissez Image > Ajuster > seuil (par défaut > N&B).
- Le processus Make Binary s’ouvrira. Sélectionnez Par défaut. Vérifiez le fond noir (des masques binaires).
- Seuil agressif, de sorte que la plupart des taches d’arrière-plan ne sont pas visibles, mais certaines parties de l’amibe sont visibles. À cette étape, si l’arrière-plan est blanc et que l’amibe est noire, choisissez Modifier > Inverser. L’arrière-plan doit être noir et l’amibe doit être blanche.
- Choisissez Traiter > binaire > Fermer. Choisissez Traiter > binaire > remplir les trous. Choisissez Fichier > Enregistrer sous > Tiff et modifiez le nom selon les spécifications souhaitées. Le fichier est maintenant prêt pour le suivi.
5. Analyser la motilité grâce au suivi
- À partir du logiciel d’imagerie avec le fichier tiff nécessaire pour suivre l’ouverture, allez dans Plugins > Tracking > Trackmate. La version de Trackmate s’ouvrira.
- Choisissez Suivant. Sélectionnez le détecteur LoG dans le menu déroulant du détecteur. Définir le diamètre estimé de la goutte : 35,0 microns, seuil 1,0. Décochez Utiliser le filtre médian et Effectuer la localisation des sous-pixels.
- Cliquez sur Aperçu au niveau de la première, du milieu et de la fin. Assurez-vous que toutes les amibes sont capturées par un cercle violet et que les défauts ou artefacts d’arrière-plan ne sont pas inclus.
- Choisissez Suivant. Le traitement commencera et cette étape peut prendre quelques minutes. Une barre de détection en haut de l’écran indiquera la partie du processus terminée.
- Choisissez Suivant lorsque vous y êtes invité. Dans le seuillage initial, choisissez à nouveau Suivant sans rien sélectionner. Sélectionnez Hyperstack Displayer lors de la sélection d’une vue. Choisissez Suivant.
- Définissez le filtre sur les spots et choisissez Suivant sans rien sélectionner. Sélectionnez un traceur ; pour ce faire, sélectionnez LAP Tracker (au lieu de Simple LAP Tracker). Choisissez Suivant.
- Lors de la liaison d’image à image, réglez la distance maximale à 40 μm. Sur la piste : Vérification de la fermeture de l’écart : Autoriser la fermeture de l’écart. Réglez la distance maximale pour cela à 100 μm et l’écart maximal du cadre à 4. Ne sélectionnez rien pour la division ou la fusion de segments de piste.
- Choisissez Suivant. L’exécution du suivi peut prendre beaucoup de temps. Lorsqu’elle est terminée, la fenêtre doit indiquer : Suivi effectué en x s en bas. Choisissez Suivant.
- Choisissez à nouveau Suivant une fois le suivi terminé. Pour définir des filtres sur les pistes : Sélectionnez le signe + en bas à gauche pour ajouter un filtre pour le nombre de places dans les pistes. Réglez le filtre sur Au-dessus pour au moins 93 % des images (par exemple, 150 images nécessiteraient un minimum de 140 spots).
REMARQUE : Certaines versions de ce logiciel nécessitent la sélection de l’équivalent opposé, tel que Inférieur à au moins 7%, ce qui peut être fait en faisant glisser une ligne vers la gauche ou la droite pour atteindre le nombre souhaité. - Choisissez Suivant. Les options d’affichage apparaîtront. Assurez-vous que les pistes de chaque image ne sont pas déformées ou étranges dans la façon dont l’amibe se déplace avant d’enregistrer le fichier XML Trackmate ou le fichier CSV Trackmate.
- Si une piste doit être supprimée, cliquez sur TrackScheme. TrackScheme affichera toutes les pistes et les points de contact de l’amibe sur tous les cadres.
REMARQUE : Les raisons possibles de supprimer une piste sont qu’une amibe a marché sur / hors du champ de vision pendant l’enregistrement, qu’une piste a une forme inappropriée par rapport à la trajectoire réelle de l’amibe, ou pour une raison quelconque, que la piste ou l’amibe est une valeur aberrante dans les données selon les paramètres expérimentaux, etc. - Cliquez sur l’amibe pour mettre en surbrillance la tache verte sur l’écran et dans Trackscheme, la tache sera entourée d’un carré vert. Cela indiquera quelle piste ou quel endroit doit être supprimé.
- Faites un clic droit sur la piste qui doit être supprimée et sélectionnez Piste entière. Appuyez sur le bouton Supprimer .
- Après avoir examiné toutes les pistes, enregistrez le fichier de suivi en cliquant sur Enregistrer en bas à gauche de la fenêtre contextuelle du logiciel de suivi. Enregistrez le fichier XML en utilisant le nom de la vidéo. Cliquez sur Reprendre pour revenir à la fenêtre contextuelle du logiciel de suivi.
- Cliquez sur Pistes et sélectionnez Pistes sur la gauche. Cliquez sur Exporter au format CSV et enregistrez le fichier CSV.
- Si la feuille de calcul ne reconnaît pas le fichier CSV résultant, exécutez les étapes suivantes.
- Ouvrez le fichier CSV enregistré dans le Bloc-notes. Cliquez sur Ctrl + A et Ctrl + C pour copier tout le contenu.
- Ouvrez la feuille de calcul et cliquez sur Ctrl + V pour la coller dans la feuille de calcul. Toutes les données seront dans une seule cellule.
- Avec les cellules avec des données toujours sélectionnées, accédez à Données > Texte aux colonnes. Choisissez Délimité > Suivant. Choisissez Virgule > > Terminer. Enregistrez ce fichier en tant que nouveau fichier CSV.
- Le CSV de suivi dispose de nombreux paramètres disponibles pour l’analyse (Figure 3). Copiez et collez les paramètres souhaités dans un nouveau fichier CSV (Figure 4) à analyser. Les paramètres utilisés ici sont décrits ci-dessous.
- Distance totale parcourue : distance totale parcourue par une amibe en μm.
- Distance maximale parcourue : la distance maximale sur le chemin d’une amibe à partir de son point de départ - ce n’est pas nécessairement le point d’arrivée
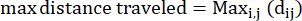
Où dij est la distance entre n’importe quel point I et n’importe quel point j sur la piste. - Rapport de confinement : Le rapport de confinement indique l’efficacité d’une amibe à s’éloigner de son point de départ : a-t-elle voyagé en ligne droite à partir de son point de départ (une valeur proche de 1), ou est-elle restée relativement proche de son point de départ (une valeur proche de 0).
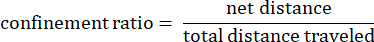
- Suivre la vitesse moyenne : vitesse moyenne de l’amibe sur la distance totale parcourue en microns par seconde.
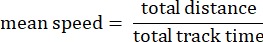
- Déplacement de la piste : la distance entre le point de départ d’une amibe et le point d’arrivée en microns.
REMARQUE : Tous les paramètres d’analyse du logiciel d’imagerie et leurs définitions se trouvent ici : https://imagej.net/plugins/trackmate/analyzers/. N’oubliez pas que chaque fichier sauvegardé est un résumé des pistes analysées à ce moment-là. Pour produire une analyse de données robuste, il faut combiner de manière appropriée les résultats de plusieurs répétitions, heures, etc., selon ce qui convient aux données (figures 5 et 6).
Résultats
Pour réussir en utilisant cette méthode, il y a plusieurs éléments globaux cruciaux à prendre en compte. Le premier est la configuration physique de l’amibe et du microscope, le deuxième est l’utilisation correcte du logiciel d’imagerie, et le troisième est l’exportation et l’analyse des données d’imagerie de manière significative.
Avant de commencer l’enregistrement microscopique, il est essentiel de s’assurer que l’amibe est correctement focalisée sur la platine du microscope (Figure 1). Plutôt que de se concentrer sur l’amibe au niveau Z le plus optimal pour les détails individuels, il est idéal de les voir très légèrement floues afin qu’elles deviennent des points noirs solides par opposition à leur état translucide lorsqu’elles sont observées en détail. En les visualisant de cette manière, le logiciel d’imagerie et le logiciel de suivi pourront trouver chaque amibe individuelle beaucoup plus facilement lorsque le fichier sera converti en binaire (Figure 2). Savoir quel est l’objectif dans le logiciel d’imagerie, c’est-à-dire que chaque amibe apparaisse comme un cercle blanc bien défini, peut aider le lecteur à trouver la bonne mise au point lors de la prise des images originales en fond clair. Ensuite, avec chaque cercle clairement défini, le logiciel de suivi sera en mesure de suivre correctement la motilité de chaque amibe et de recueillir les mesures de motilité individuelles pour chaque amibe.
Après avoir suivi les instructions étape par étape dans le protocole répertorié ci-dessus pour le logiciel d’imagerie et le logiciel de suivi et après avoir fait référence à la vidéo associée pour aider aux premières étapes cruciales de l’utilisation d’un logiciel d’imagerie, il est temps d’exporter les données du logiciel d’imagerie pour la quantification et l’analyse des données. Le résultat brut du logiciel de suivi sera similaire à ce que l’on voit dans la figure 3. Ici, il est important de vérifier que le nombre de spots dans les pistes qu’il a exportées est correct pour ce qui est attendu (par exemple, si l’on demande que 93% des images soient présentes dans chaque piste et que l’on a 100 images au total dans la vidéo, chaque piste exportée doit avoir plus ou moins de 93 spots). Si les spots ne correspondent pas aux exigences, il peut être nécessaire de revenir aux étapes du logiciel de suivi pour s’assurer que les paramètres requis ont été correctement définis.
À partir de l’exportation brute, commencez à construire la feuille d’analyse des données (Figure 4). Il est important de se rappeler que chaque morceau représente une amibe singulière au sein d’une réplique singulière. Par conséquent, comme le montrent les figures 4, 5 et 6, la moyenne de toutes les pistes d’un jeu d’images ou d’un point temporel (figure 4), et ce processus doit être effectué pour chaque ensemble d’images ou point temporel d’une série entière (c’est-à-dire, si un total de 3 h de vidéo sont pris et que les points de données sont divisés par l’heure, puis il y aura trois jeux d’images/heures à analyser par répétition, Figure 5). Enfin, à partir de cette combinaison de moyennes dans chaque ensemble de trames dans chaque répétition, les données moyennées de chaque réplication doivent être combinées dans un seul espace afin que les données de plusieurs répétitions puissent être analysées pour déterminer la moyenne réelle et l’écart-type par point temporel (Figure 6).
Les moyennes de chaque répétition seront utilisées pour l’analyse des données, et la moyenne combinée et l’écart-type ou l’erreur-type de toutes les répliques seront utilisés pour la représentation graphique (comme le montre un exemple de la figure 7). Si l’on analyse des données comme nous l’avons fait ici, c’est-à-dire que plusieurs échantillons sont suivis sur plusieurs points temporels, il est plus approprié de les analyser à l’aide d’une ANOVA à 2 voies avec un test de Tukey a posteriori. Cela nous permet de déterminer les différences entre les échantillons à chaque point temporel et également de déterminer les différences à l’intérieur de chaque échantillon au fil du temps.

Figure 1 : Les amibes apparaissent sous forme de formes sombres opaques en microscopie à fond clair. Zoom sur l’image représentative de l’amibe à un grossissement de 4x, légèrement floue pour apparaître comme des cernes solides. Barre d’échelle = 500 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
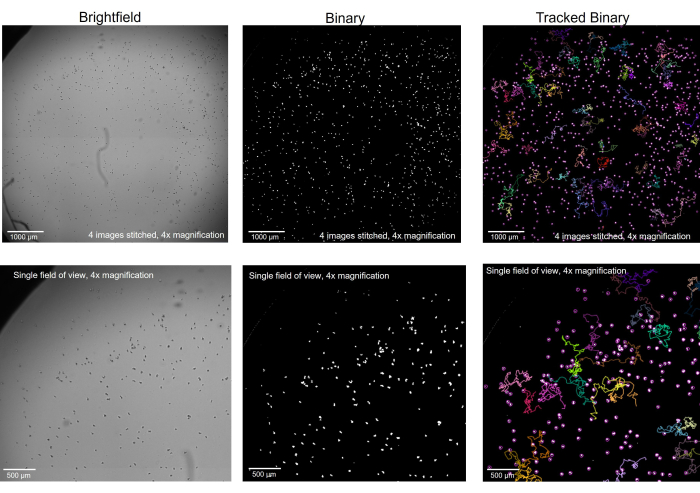
Figure 2 : Amibe imagée en microscopie à fond clair convertie en binaire et suivie. Amoeba à un grossissement de 4x, en fond clair (à gauche), a été converti en binaire avec les artefacts supprimés (au milieu) et, après le suivi, avec des pistes sélectionnées affichées (à droite). Rangée du haut : un tout bien imagé en assemblant 4 images ensemble, barre d’échelle = 1000 μm. Rangée du bas : la taille d’une seule image avant l’assemblage, barre d’échelle = 500 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
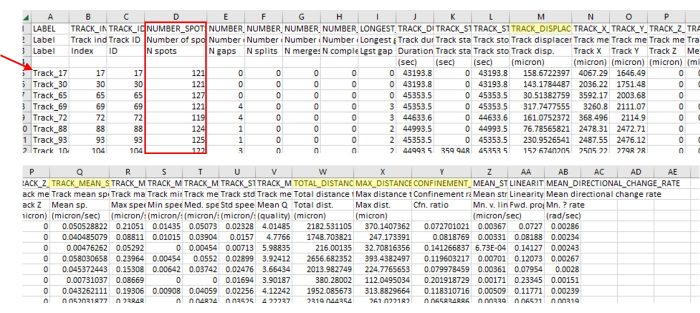
Figure 3 : Le résultat de l’analyse immédiate des données du logiciel d’imagerie et de suivi contient une grande variété de types de données. Le résultat représentatif du suivi est illustré à la figure 2, qui est une vidéo singulière. Notez les numéros de piste (flèche rouge) qui sautent les numéros, indiquant que le logiciel de suivi a correctement supprimé les pistes qui ne correspondaient pas aux paramètres. Dans cet exemple, seules 71 pistes sur 5385 identifiées étaient alignées avec les paramètres. Ce ratio est normal, compte tenu de ces directives strictes. Ce nombre de pistes correspond également au nombre d’amibes utilisé dans ce protocole (par exemple, si une plaque de 96 puits est ensemencée avec 200 μL de 7,5 x 103 UFC/ml par puits, alors on devrait s’attendre à environ 1 500 amibes dans le puits et à peu près autant visibles dans un champ de vision à 4 points. Il y aura probablement beaucoup plus de traces qu’il n’y a d’amibes, car les amibes se promènent dans et hors du champ de vision, générant de nouvelles traces. Ce numéro de trace est à nouveau apparent sur la figure 2, où seules quelques traces sont visibles par rapport à un grand nombre de taches. Vérifiez toujours le nombre de places dans le rail (case rouge) pour vous assurer que seules les longueurs de rail correctes sont incluses. Par exemple, ici, nous incluons plus de 93 % des images, et il y avait 127 images dans cette section, donc tous les nombres de taches doivent être supérieurs à 118. Les mesures mentionnées dans le protocole sont surlignées en jaune. Veuillez cliquer ici pour voir une version agrandie de cette figure.
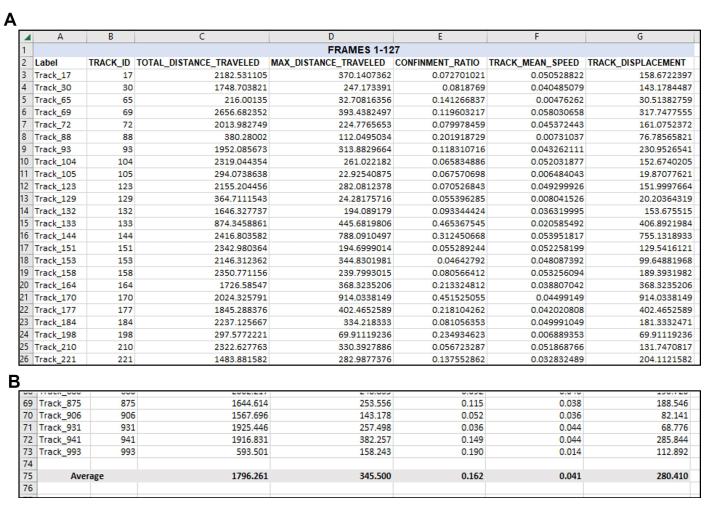
Figure 4 : Données réorganisées au besoin à la suite de la sortie initiale des données. Organisation des données souhaitées après exportation. (A) Les mesures pertinentes choisies pour la quantification ont été copiées à partir de l’exportation du logiciel de suivi (figure 3) et collées dans une nouvelle feuille de calcul. (B) Une fois que les mesures souhaitées ont été organisées dans la nouvelle feuille de calcul, la moyenne de chaque piste doit être calculée. Ainsi, dans cet exemple représentatif, la distance totale, la distance maximale, le confinement, la vitesse moyenne et le déplacement de chaque piste utilisable dans cette vidéo singulière ont été moyennés à un nombre unique pour chaque mesure. Notez à nouveau ici qu’un total de 71 pistes répondaient aux paramètres pour les images 1 à 127. Il y aura probablement un nombre différent de pistes appropriées pour chaque vidéo ou jeu d’images analysé, comme le montre la figure 5. Veuillez cliquer ici pour voir une version agrandie de cette figure.
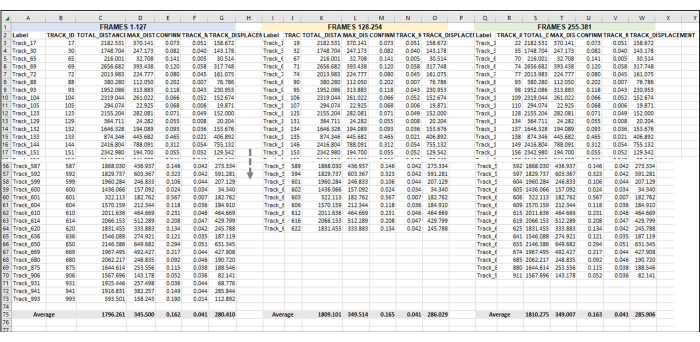
Figure 5 : Ensemble d’images séquentielles pour l’analyse d’une série entière d’images ou d’une vidéo. Il s’agit d’une image représentative de l’ajout de vidéos séquentielles à l’analyse des données. En supposant que les images 1 à 127 constituent l’heure 1 de la vidéo (ici, 1 h est constituée de la prise d’une image toutes les 28 s). Les images 128 à 254 seront l’heure 2 de la vidéo et les images 255 à 381 l’heure 3 de la vidéo. Combinez les données exportées (comme cela a été exporté dans la figure 3) de toutes les heures en une seule feuille pour travailler avec toutes les données en une seule fois. Comme nous l’avons fait à la figure 4, la moyenne des données de toutes les pistes utilisables pour chaque jeu de trames/h est calculée. Tout ce qui figure dans la figure 5 est les données d’une répétition ou d’un échantillon. S’il y a 3 répétitions, il y aura 3 onglets de feuille de calcul séparés disposés de la même manière, et les données de ceux-ci seront introduites dans un nouvel onglet, comme le montre la figure 6. Veuillez cliquer ici pour voir une version agrandie de cette figure.
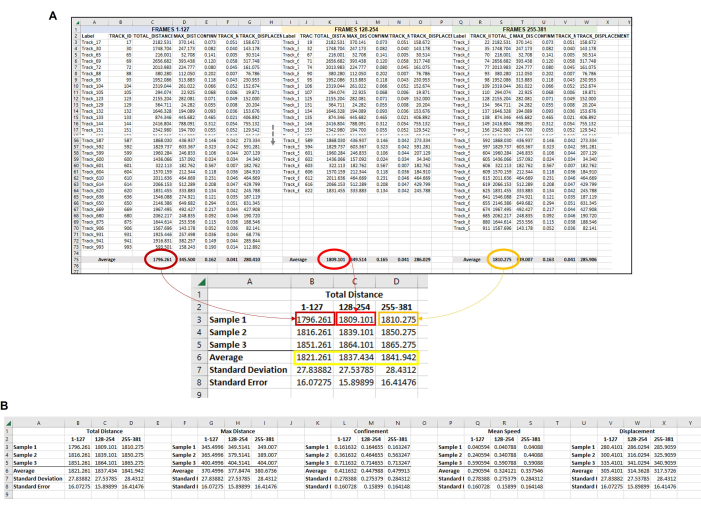
Figure 6 : Données composites de toutes les amibes de chaque jeu de cadres utilisées pour représenter une seule répétition. (A) Toutes les moyennes de chaque trame de la figure 5 doivent être rassemblées dans un emplacement central afin que l’analyse finale des données puisse être effectuée. Dans cette image représentative, les moyennes de distance totale des trois premiers jeux de cadres (ou heures) sont affichées. Cette opération doit être répétée pour chaque échantillon/condition/répétition exécuté. Ensuite, la moyenne des moyennes de chaque échantillon de chaque jeu de trames doit être calculée (boîte jaune). Il s’agit du dernier point de données à partir duquel l’écart-type et l’erreur-type peuvent être calculés. Les moyennes individuelles de chaque échantillon (ici, les chiffres en B3, B4 et B5 pour l’échantillon de distance totale 1 ; C3, C4 et C5 pour la distance totale de l’échantillon 2, etc.) sera utilisé pour exécuter l’analyse statistique. Ce processus sera répété pour toutes les mesures requises (distance maximale, confinement, vitesse moyenne, déplacement, etc.) (B) Pour faire une représentation graphique de la quantification, utilisez les chiffres de la boîte jaune comme points de données à utiliser pour le graphique final. Veuillez cliquer ici pour voir une version agrandie de cette figure.
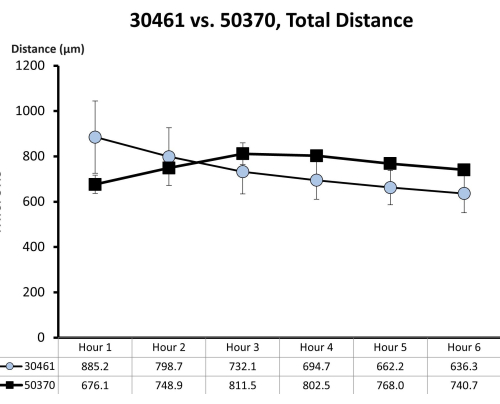
Figure 7 : Données brutes de motilité pour ATCC 30461 et ATCC 50370. Les données brutes (représentées ici sous forme de moyenne ± ET pour chaque heure) ont été collectées précédemment et n’ont pas été publiées pour les études antérieures18,19. Cette quantification de la motilité démontre que ces deux amibes sont similaires dans leurs 6 premières h de motilité (mesurée par la distance totale en microns parcourus au cours de cette heure) dans une solution de Ringer sur une surface en verre. Chaque point temporel représente trois répétitions distinctes, chacune composée de 20 à 200 traces d’amibes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Être capable de suivre et de quantifier la motilité des amibes telles que les Acanthamoeba, qui sont des organismes microscopiques adhérents non affectés par le mouvement brownien à basse vitesse 9,10, révèle une quantité substantielle d’informations sur le comportement des amibes et peut considérablement améliorer l’éclairage sur les méthodes de prévention de l’AK. Ce protocole a été détaillé dans des publications récentes, et les données ont été confirmées ou combinées à d’autres analyses de données similaires18,19. Notons ici que la vitesse des amibes détaillée à l’aide de cette méthode est similaire à ce qui a été publié par d’autres groupes. Ce protocole se distingue par sa capacité à être utilisé sur pratiquement n’importe quelle surface ou traitement d’amibe, qui est transparent à la lumière, mais il est important de noter que même si ce protocole pourrait potentiellement être modifié pour fonctionner avec d’autres organismes, il n’a été optimisé jusqu’à présent que pour Acanthamoeba. Le protocole ci-dessus a toujours bien fonctionné en laboratoire, mais les modifications suivantes sont apportées pour modifier la méthode afin de l’adapter à d’autres besoins ou de résoudre des situations difficiles.
Détermination des paramètres de suivi : Dans le protocole ci-dessus, la liaison d’image à image, la fermeture de l’espace, l’écart maximal entre les images et le nombre de points sur la piste ont été répertoriés, qui étaient les plus applicables pour les publications récentes. Cependant, il est possible que d’autres paramètres soient mieux adaptés à d’autres besoins. De plus, il peut y avoir des paramètres que d’autres aimeraient utiliser mais qui n’ont pas été utilisés ici (tels que la distance maximale d’une piste sous les filtres de piste). Plus d’informations sur chaque paramètre et ce qu’ils décrivent peuvent être consultées ici sur le site Web (voir référence21). Lorsque vous décidez quels paramètres et filtres sont les plus pertinents pour d’autres expériences et comment ils doivent être définis, réfléchissez à ce qui est statistiquement rationnel pour un projet spécifique ; Par exemple, voudrait-on des données d’une amibe qui s’éloigne puis revient dans le champ de vision ? Ou voudrait-on conserver les données d’une amibe qui n’a pas beaucoup bougé du tout mais qui a quand même été étiquetée comme une piste ? et ce qui a du sens pour le nombre d’images et la durée entre les images lorsque les images ont été enregistrées.
Modification de la taille des gouttes : Nous avons constaté qu’il est extrêmement difficile, avec un logiciel ou une configuration de microscope - sur la base des technologies actuelles et des logiciels disponibles pour les laboratoires standard - de suivre avec précision le nombre d’amibes dans un agrégat au fur et à mesure qu’il change au fil du temps. Si vous suivez la taille d’une goutte et essayez de corréler cette taille avec le nombre d’organismes qu’elle contient, utilisez une courbe standard générée après des expériences répétées pour prédire mathématiquement la taille d’un agrégat. Par exemple, les agrégats ont été créés en enfonçant des puits avec une gamme de nombres d’amibes, tels que 8, 16, 32, 125, 250, 500, 1 000 et 2 000 cellules par puits. Des images en accéléré à différents points temporels de chaque puits pendant 24 heures ont été générées. Chaque sphéroïde (qui était d’un nombre connu d’amibes) a été analysé pour une zone bidimensionnelle, et une courbe standard a été générée en fonction du nombre d’amibes par rapport à la taille du sphéroïde au fil du temps. Cette expérience a été répétée au moins en trois exemplaires pour nous donner un écart-type approprié de toute courbe générée.
Déterminer la directionnalité de l’amibe : Bien que cela ne soit peut-être pas nécessaire pour des études particulières, il peut être utile de comprendre la directionnalité de l’amibe. Cela fournirait des données sur les effets chimiotactiques du traitement ou de l’expérience. Ces données peuvent également être utilisées pour créer des données visuelles (graphiques) et quantitatives. Il est disponible via l’outil de chimiotaxie et de migration, qui est un plug-in gratuit pour les logiciels d’imagerie. Il est disponible sur le site Web, accompagné du guide d’application et d’exemples d’images et de films (voir la table des matières).
Dynamique détaillée du mouvement : d’autres groupes ont examiné la dynamique d’expansion et les trajectoires diffusives avec des analyses statistiques très avancées et développées au-delà de ce qui est discuté ici 9,10,22. Ceux-ci pourraient être envisagés en fonction des besoins de l’utilisateur.
Comme pour toute méthode réussie, le protocole détaillé dans ce manuscrit a fait l’objet de nombreuses séries de dépannage pour atteindre la cohérence. Bien qu’il soit possible de réaliser d’excellentes vidéos traçables de haute qualité en prenant une image toutes les secondes (ou, aussi vite que le microscope le peut, cela peut prendre moins d’une seconde), cela crée des fichiers extrêmement volumineux, qui peuvent être difficiles à utiliser. Cela ne convient de manière réaliste qu’aux vidéos à très court terme enregistrées pendant quelques minutes au maximum. À l’inverse, en prenant une image toutes les quelques secondes, nous avons constaté que le comportement des amibes est toujours très traçable en fonction de la vitesse de cette espèce, et il est possible de créer des vidéos exploitables pendant des heures ou des jours. Nous avons constaté que le temps maximum entre les images est de 30 s, ce qui est le temps nécessaire pour que les amibes soient suivies avec précision par un logiciel d’imagerie. L’intervalle de temps choisi par l’utilisateur doit être pris en compte à l’aide des contraintes connues de la vitesse à laquelle le microscope peut enregistrer les images, du nombre d’images par puits nécessaires et du nombre de puits enregistrés à chaque intervalle. De même, les paramètres mentionnés dans ce protocole concernant le déplacement maximal, les écarts d’images autorisés, les images minimales nécessaires, etc., ont été déterminés par essais et erreurs par ce laboratoire pour créer des informations sur les pistes qui incluent les pistes d’amibes les plus complètes et ignorent le bruit créé par les pistes incomplètes, les amibes qui rejoignent ou quittent la scène au milieu de la vidéo, ou les erreurs créées par la confusion du logiciel d’imagerie, comme lorsque deux amibes se rencontrent au milieu de la piste puis se séparent. Ce genre d’erreurs et de traces incomplètes est naturellement élevé lors de la création de vidéos extrêmement longues (de quelques heures à quelques jours) d’organismes biologiques microscopiques qui interagissent constamment les uns avec les autres et sont la raison pour laquelle une très grande partie des pistes erronées doivent être rejetées. Il convient de noter que l’étape de remplissage des trous dans le protocole est, d’après l’expérience de ce laboratoire, importante pour réduire les erreurs dans la façon dont le logiciel d’imagerie suit les amibes. En s’assurant que chaque amibe est un cercle solide au lieu d’un beignet ou d’une forme en C, le logiciel a beaucoup plus de chances de pouvoir suivre chaque amibe avec succès.
De plus, comme nous l’avons vu, il existe de nombreux paramètres à la disposition d’un utilisateur pour l’analyse d’une image ou d’une vidéo. Sur la base des besoins expérimentaux, nous avons toujours tiré le meilleur parti de l’analyse de la distance totale, de la distance maximale, de la vitesse et du déplacement. Celles-ci sont discutées en profondeur (y compris en utilisant différentes déformations et surfaces) avec des interprétations graphiques dans des publications précédentes18,19. Ces quatre paramètres permettent à un utilisateur d’extrapoler la capacité d’une amibe à parcourir des distances linéaires et le temps qu’il lui faut pour le faire, ce qui aide à comprendre son comportement en ce qui concerne la contamination des lentilles de contact. La récupération et l’analyse de ces paramètres est un travail important, comme le détaillent nos chiffres. En travaillant avec de grandes et nombreuses feuilles de calcul à partir de la sortie de logiciels d’imagerie, nous avons limité les erreurs involontaires en créant des modèles de feuilles de calcul verrouillées qui calculaient automatiquement toutes les analyses requises. Cependant, une amélioration possible de cette méthode serait d’écrire un script capable de gérer ces données, de les trier et de les analyser.
En conclusion, une méthode accessible et précise pour mesurer la taille et la motilité d’Acanthamoeba dans de nombreuses conditions différentes est décrite ici. Nous avons démontré que cette méthode peut être appliquée à de nombreuses souches différentes d’amibes et souligné que, bien qu’il puisse y avoir des paramètres simples pour obtenir des informations sur la motilité, cette configuration expérimentale peut être hautement adaptée à des besoins spécifiques.
Déclarations de divulgation
Tous les auteurs sont des employés d’Alcon Research, LLC.
Remerciements
Ce travail a été financé par Alcon Research, LLC.
matériels
| Name | Company | Catalog Number | Comments |
| ¼ Ringer’s solution | ThermoFisher Scientific | BR0052G | Oxoid Ringers Solution Tablets. Follow directions to make one-quarter strength instead of full strength Ringers. |
| 10 µL pipette | Eppendorf Research | 3123000039 | 2 µL-20 µL single channel |
| 10 µL pipette tips | Neptune Scientific, San Diego, CA, USA | BT10.N | 10 µL Universal Barrier Tip |
| 48 well plate | Millipore Sigma, | CLS3548 | Corning Costar TC-Treated Multiple Well Plate: polystyrene plate, flat bottom wells, sterile, with lid |
| 50 mL conical tubes (1 for each sample, 1 for each pass 2 sample) | Fisher Scientific | Falcon 352098 | Falcon 50 mL High Clarity Conical Centrifuge Tubes, polypropylene |
| 96-well plate | Millipore Sigma, Burlington, MA, USA | CLS3596 | Corning Costar TC-Treated Multiple Well Plate: polystyrene plate, flat bottom wells, sterile, with lid |
| Acanthamoeba | American Type Culture Collection, Manassas, VA, USA | ATCC 30461 | This protocol has been verified for ATCC 50655, 30872, 50702, 30010, 30461, 50370, 50703, 30137, PRA-115, and PRA-411 |
| Axenic culture media (AC6) | Made in house | n/a | Containing 20 g biosate peptone, 5 g glucose, 0.3 g KH2PO4, 10 µg vitamin B12, and 1 glass5 mg l-methionine per liter of distilled deionized water. Adjust pH to 6.6-6.95 with 1 M NaOH and autoclave at 121 °C for 20 min, store at room temperature for up to 3 months. |
| Centrifuge and appropriate rotor | Thermo Scientific, Waltham, MA, USA | Sorvall ST 40R | Any equivalent centrifuge and rotor are acceptable so long as it can spin 50 mL conical tubes at 500 x g for 5 min |
| Chemotaxis and Migration Tool | Free software based on ImageJ, available at https://ibidi.com/chemotaxis-analysis/171-chemotaxis-and-migration-tool.html | ||
| Disposable hemocytometer | Bulldog Bio, Portsmouth, NH, USA | DHC-N01 | Neubauer Improved 2-Chip Disposable Hemocytometer, Individually packaged, Nonpyrogenic |
| ImageJ software with Trackmate plugin (this protocol written with Trackmate version 6.0.2.) | Free software developed by the National Institutes of Health, available at imagej.net. Trackmate plugin available at https://imagej.net/plugins/trackmate/ | ||
| Microscope, preferably with automated moveable stage | Nikon, Tokyo, Japan | A Nikon Eclipse Ti-U Microscope was used in this study and the automated moveable stage was utilized to be able to record images in multiple wells at a time. | |
| NIS-Elements software | Nikon | ||
| Serological pipette | Fisher Scientific, Hampton, NH, USA | BrandTech 26331 | BrandTech accu-jet pro Pipet Controller |
| Serological pipette tips | VWR | 5 mL: 76201-710 10 mL: 170356 25 mL: 89130-900 50 mL: 75816-088 | VWR Serological Pipette, Non-Pyrogenic |
| Sterile aluminum transmission flow cell | Biosurface Technologies Corporation, Bozeman, MT, USA | FC 81-AL | Anodized aluminum single channel transmission flow cell with 96-well plate footprint for use with an inverted microscope |
| T75 Flasks | VWR, Radnor, PA, USA | 734-2316 | VWR Tissue Culture Flask, 182.5 cm, Surface treated, Plug seal cap, Sterile |
Références
- Randag, A. C., et al. The rising incidence of Acanthamoeba keratitis: A 7-year nationwide survey and clinical assessment of risk factors and functional outcomes. PLoS One. 14 (9), e0222092 (2019).
- Szentmary, N., et al. Acanthamoeba keratitis - Clinical signs, differential diagnosis and treatment. J Curr Ophthalmol. 31 (1), 16-23 (2019).
- Carnt, N., et al. Acanthamoeba keratitis: confirmation of the UK outbreak and a prospective case-control study identifying contributing risk factors. Br J Ophthalmol. 102 (12), 1621-1628 (2018).
- Verani, J. R., et al. National outbreak of Acanthamoeba keratitis associated with use of a contact lens solution, United States. Emerg Infect Dis. 15 (8), 1236-1242 (2009).
- Tu, E. Y., Joslin, C. E. Recent outbreaks of atypical contact lens-related keratitis: what have we learned. Am J Ophthalmol. 150 (5), 602-608 (2010).
- International Organization for Standardization. ISO 14729:2001/A1. Ophthalmic optics-Contact lens care products-Microbiological requirements and test methods for products and regimens for hygienic management of contact lenses. International Organization for Standardization. , (2010).
- . ASC Z80, Parent Committee Meeting Available from: https://www.thevisioncouncil.org/sites/default/files/ASCZ80_ParentCommitteeMinutes_February_27_2018_FINALMar19-2018.pdf (2023)
- Rayamajhee, B., et al. Acanthamoeba, an environmental phagocyte enhancing survival and transmission of human pathogens. Trends Parasitol. 38 (11), 975-990 (2022).
- Reverey, J. F., et al. Superdiffusion dominates intracellular particle motion in the supercrowded cytoplasm of pathogenic Acanthamoeba castellanii. Sci Rep. 5, 11690 (2015).
- Thapa, S., Lukat, N., Selhuber-Unkel, C., Cherstvy, A. G., Metzler, R. Transient superdiffusion of polydisperse vacuoles in highly motile amoeboid cells. J Chem Phys. 150 (14), 144901 (2019).
- Rudell, J. C., et al. Acanthamoeba migration in an electric field. Invest Ophthalmol Vis Sci. 54 (6), 4225-4233 (2013).
- Fukaya, S., Aoki, K., Kobayashi, M., Takemura, M. Kinetic analysis of the motility of giant virus-infected amoebae using phase-contrast microscopic images. Front Microbiol. 10, 3014 (2019).
- Fukaya, S., Takemura, M. Kinetic analysis of Acanthamoeba castellanii infected with giant viruses quantitatively revealed process of morphological and behavioral changes in host cells. Microbiol Spectr. 9 (1), 0036821 (2021).
- Cheng, H. J., Hsu, C. H., Hung, C. L., Lin, C. Y. A review for cell and particle tracking on microscopy images using algorithms and deep learning technologies. Biomed J. 45 (3), 465-471 (2022).
- Mathieu, E., et al. Time-lapse lens-free imaging of cell migration in diverse physical microenvironments. Lab Chip. 16 (17), 3304-3316 (2016).
- Svensson, C. M., Medyukhina, A., Belyaev, I., Al-Zaben, N., Figge, M. T. Untangling cell tracks: Quantifying cell migration by time lapse image data analysis. Cytometry A. 93 (3), 357-370 (2018).
- Piltti, K. M., et al. Live-cell time-lapse imaging and single-cell tracking of in vitro cultured neural stem cells - Tools for analyzing dynamics of cell cycle, migration, and lineage selection. Methods. 133, 81-90 (2018).
- Campolo, A., et al. Continuous real-time motility analysis of Acanthamoeba reveals sustained movement in absence of nutrients. Pathogens. 10 (8), 995 (2021).
- Campolo, A., Patterson, B., Lara, E., Shannon, P., Crary, M. Complete recovery of Acanthamoeba motility among surviving organisms after contact lens care disinfection. Microorganisms. 11 (2), 299 (2023).
- Crary, M. J., Walters, R., Shannon, P., Gabriel, M. M. Variables affecting the recovery of Acanthamoeba trophozoites. Pathogens. 10 (2), 221 (2021).
- . imagej Available from: https://imagej.net/plugins/trackmate/tutorials/getting-started (2024)
- Cherstvy, A. G., Nagel, O., Beta, C., Metzler, R. Non-Gaussianity, population heterogeneity, and transient superdiffusion in the spreading dynamics of amoeboid cells. Phys Chem Chem Phys. 20 (35), 23034-23054 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon