Method Article
כימות של Acanthamoeba spp. תנועתיות
In This Article
Summary
הליך זה מתאר כיצד לדמיין, לעקוב ולכמת Acanthamoeba spp. תנועתיות.
Abstract
Acanthamoeba keratitis הוא זיהום עיני חמור המהווה אתגרי טיפול ויכול להוביל לעיוורון. למרות היותו נפוץ בכל מקום וזיהום פוטנציאלי של עדשות מגע לאחר חשיפה למים, ההתנהגות הטבעית של פתוגן זה נותרה חמקמקה. הבנת דפוסי התנועה של Acanthamoeba יכולה ללמד אותנו על האופן שבו הוא מאכלס עדשות מגע ומזהם את הקרנית של המטופל. מזל כי Acanthamoeba spp. ניתן לראות באמצעות מיקרוסקופ ברייטפילד החל מהגדלה פי 4. טכניקות קודמות פותחו כדי לכמת את תנועתיות Acanthamoeba ביחס להשפעות ציטופתיות או תת-חשיפה לשדה החשמלי. כאן, אנו מתארים שיטה לעקוב ולכמת Acanthamoeba spp. תנועתיות לטווח ארוך (שעות עד ימים), שהוא פרוטוקול החל על זנים מרובים של אמבות, משטחים ומצב תזונתי של אמבה. הליך זה הוא חלק בלתי נפרד מקביעת כימות תנועתיות ליבה רבות, כגון מרחק, מהירות, כליאה וכיווניות, הנחוצות לניטור שלבים שונים של זיהום, התפשטות או שינוי התנהגותי.
Introduction
מחקר Acanthamoeba גדל באופן דרמטי בשנים האחרונות בשל שכיחות מוגברת של Acanthamoeba keratitis (AK), שהוא זיהום טפילי בעקבות ההתקשרות של Acanthamoeba לקרנית1. בעוד שניתן לייחס התפרצויות של AK לטיפול לא הולם בעדשות מגע או פתרונות לא יעילים לטיפול בעדשות מגע 2,3,4,5, אין כיום דרישות להוכחת יעילות חיטוי Acanthamoeba עבור מוצר כלשהו בשוק. עם זאת, קיים מאמץ מתמשך בקהילה המדעית ובתוך ארגוני התקנים לבחון את הפרוטוקולים הדרושים לכימות יעילות החיטוי של מוצרים לטיפול בעדשות מגע 6,7. יתר על כן, בשל דמיונו בהיבטים תאיים ותפקודיים למקרופאגים אנושיים, Acanthamoeba צוין כבעל תפקיד משמעותי באירוח והפצה של פתוגנים אנושיים אחרים8, בנוסף לפתוגנים שהאמבה עצמה מביאה.
לאחרונה תוארו טכניקות שהצליחו לכמת את תנועתיות האקנתמואבה - שבדרך כלל אינה כפופה מאוד לתנועה בראונית 9,10 - ביחס להשפעות ציטופתיות או תת-חשיפה לשדה החשמלי 9,11, כמו גם התקדמות בניתוח תנועתיות במחקר וירוסים ענק באמצעות Acanthamoeba כווקטור נגיפי12 הניתן למעקב, 13. כמו כן, חלו שיפורים תכופים ומשמעותיים במעקב אחר תאים וחלקיקים במהלך 20 השנים האחרונות באמצעות תוכנות חדשות כגון תוכנת ההדמיה המשמשת כאן, ואלגוריתמים חדשים וטכנולוגיות למידה עמוקה14. עם זאת, זהו תחום מדעי חדש יחסית וצומח בכל הנוגע למחקרי בנצ'טופ, יישום קליני ותקנים תעשייתיים, ופורסמו נתונים מועטים לגבי שיטות להדמיה ומעקב אחר אמבה זו, במיוחד על מנת לכמת שינויים התנהגותיים בעקבות היצמדות לעדשות מגע או במהלך או אחרי חיטוי עדשות מגע. תחומים אחרים שהתרחבו לניטור חזותי ארוך טווח תמכו במאמץ זה 15,16,17. בשל אופיו המאתגר מטבעו של Acanthamoeba - כולל עמידות כללית לפלסמידים (שעלולים להעניק פלואורסצנטיות), יכולתה של האמבה לצרוך ולהשמיד צבעי תאים סטנדרטיים, והרכב חלבוני חיצוני ייחודי - המקשה על תיוג נוגדנים - שיטות הזמינות לתאים אחרים שהופכות אותם לגלויים בסביבות שאינן הדמיה של שדה בהיר לא היו שמישות באורגניזם זה. לפיכך, כימות התנועתיות של אמבה זו הוכיח תוספת משמעותית לתחום. באמצעות השיטה המתוארת כאן, הצלחנו לוודא כי אמבה נשארת תנועתית לפחות 12 שעות ללא חומרים מזינים18 וכי אמבה המאותגרת בתהליך חיטוי ומפסיקה תנועתיות במהלך החיטוי יכולה לשחזר את תנועתיותה לאחר החיטוי אם היא לא עברה ליזה מלאה19.
פרוטוקול זה מפרט כיצד לעקוב ולכמת חזותית את תנועתיות האמבה באופן מיקרוסקופי. השלבים העיקריים הם להקליט אמבה בשדה בהיר באמצעות המיקוד והתזמון המתאימים בין תמונות, להפוך את התמונות לבינאריות באמצעות תוכנת הדמיה, ולאחר מכן להשתמש בתוסף מעקב של תוכנת הדמיה כדי להגדיר את הפרמטרים הניסיוניים ולעקוב אחר כל אמבה כדי לקבוע את המדידות הנדרשות כגון מהירות, מרחק וכליאה. לאחר מכן, ניתן לכמת את הכימוטקסיס של אוכלוסיית אמבה או אמבה על מנת להגדיר כיווניות. תרומתה העיקרית של שיטה זו היא לדמיין ולכמת התנהגות אמבה במהלך מצבים שונים של תמיכה תזונתית, היצמדות לפני השטח, אתגר חיטוי או שינויים סביבתיים אחרים כגון מגורים משותפים עם תרבית תאים של יונקים.
Protocol
1. הכנת Acanthamoeba
הערה: פרוטוקול זה אומת עבור ATCC 50655, 30872, 50702, 30010, 30461, 50370, 50703, 30137, PRA-115 ו- PRA-411. זהו פתוגן BSL2 ויש לעבוד עליו בתוך מכסה מנוע BSL2 ומעבדה.
- צור תרבות אב מבקבוקון לדוגמה או תקעמוכן 20 על ידי מילוי בקבוק T75 עם 30 מ"ל של מדיום AC6 ולהוסיף לו תקע אחד או את התוכן של בקבוקון לדוגמה. לדגור על הבקבוק במשך 3-5 ימים ב 26-30 מעלות צלזיוס עד שהבקבוק הוא 50% עד 80% במפגש.
- מעבר התאים יום לפני שהם נדרשים כדי ליצור אוכלוסייה הומוגנית של trophozoites.
- בהתאם לצרכי הזן, נערו ו/או הכו במהירות את תרבות האב פי 2 כדי לעקור טרופוזואיטים דבוקים.
- מלאו בקבוק T75 ב-30 מ"ל של AC6 בינוני. הוסף 2 מ"ל של תרבות המאסטר למעבר. לדגור את הבקבוק במשך 18-24 שעות ב 26-30 מעלות צלזיוס.
- לפני הקציר, בדקו ויזואלית את אוכלוסיית הטרופוזואיט תחת הגדלה פי 4 במיקרוסקופ. ודא trophozoites הם דבקים ואחידים.
- הכה במהירות את הבקבוק 2x כדי לעקור trophozoites דבק. יוצקים את התוכן לתוך צינור חרוטי 50 מ"ל.
- סובב את הצינור ב 500 x גרם במשך 5 דקות בצנטריפוגה מאוזנת כדי לגלול את האמבה. יוצקים או פיפטה את המדיה supernatant ולהשליך. לדלל את הגלולה ב 2-10 מ"ל של 1/4 פתרון רינגר.
- מערבלים את המדגם. הוסף 10 μL של דגימה להמוציטומטר חד פעמי וספור את CFU/mL של Acanthamoeba.
- בהתבסס על ספירת ההמוציטומטר, לדלל את גלולת האמבה לריכוז של 7.5 x 103 תאים/מ"ל בתמיסת רינגר 1/4.
- אמבה זרעית על משטח, שיכולה להיות זכוכית, פלסטיק או אגר שאינו מזין, ומגוון צורות באר בהתאם לצרכי הניסוי כמתואר להלן.
- צלחת 96 באר: זרעו כל באר עם 200 μL של תרחיף Acanthamoeba וסגרו את הצלחת עם המכסה.
- צלחת 48 באר: זרעו כל באר עם 1 מ"ל של השעיית Acanthamoeba וסגרו את הצלחת עם המכסה.
- תא זרימה: הוסף באיטיות 4 מ"ל של תרחיף Acanthamoeba דרך היציאות הסטריליות של תא זרימת אלומיניום סטרילי, תוך הימנעות מבועות בתא. הידוק היציאות נסגרו לאחר הוספת המתלים.
- לפני ההדמיה, יש לאפשר לאמבה לפחות 30 דקות להיצמד לפני השטח לפני תחילת ההקלטה המיקרוסקופית.
2. הדמיה והקלטה Acanthamoeba
- דמיינו את האמבה בהגדלה של פי 4 בשדה בהיר. הרקע צריך להיראות אפור בהיר, והאמבה צריכה להיות שחורה. התאימו את האור והמיקוד כך שהאמבה תהיה עיגולים כהים מוצקים במקום שקופים (כך, מעט מחוץ למיקוד, איור 1).
הערה: ההגדלה האופטימלית היא פי 4 למעקב מכמה סיבות: אמבות שעוזבות או נכנסות לשדה הראייה במהלך המעקב לא צריכות להיכלל בניתוח נתונים, ולכן ההגדלה התחתונה תומכת בצורה הטובה ביותר במספר המרבי של אמבות הכלולות בניתוח בפרק זמן. אמבה צריכה להופיע כעיגולים שחורים מוצקים בשדה בהיר כדי להמיר אותם לבינאריים לצורך ניתוח מעקב. זה הכי קל להשיג בהגדלה נמוכה יותר, מכיוון שהגדלות גבוהות יותר חושפות בקלות את הטבע השקוף ואת המבנים הפנימיים של האמבה. באופן כללי, ניתן ללכוד יותר אמבות בשדה ראייה יחיד בהגדלה נמוכה יותר, מה שתומך בניתוח נתונים חזק יותר. - הגדר את תוכניות ההקלטה בתוכנת ההדמיה להקליט תמונה במרווחי זמן קבועים כדי לעקוב אחר האמבה. הזמן הארוך ביותר בין תמונות שיאפשר מעקב מדויק הוא 30 שניות, הזמן האופטימלי לגודל הקובץ ופרטי המעקב מומלץ ב- 12 שניות או 24 שניות.
- ניתן לעקוב אחר אמבות במשך מספר ימים בכל פעם, אך היו מודעים לגודל הקובץ שהקלטה מסוג זה תיצור ולקושי המובנה במניפולציה של קבצים גדולים במיוחד. כדי להימנע מכך, רשום אמבות לקטעי זמן בפרקי זמן מסוימים. לדוגמה, הקלט במשך שעה אחת כל 12 שעות במשך 5 ימים.
- אם המיקרוסקופ והתוכנית מאפשרים, רשום בארות מרובות בהפעלה אחת באמצעות קואורדינטות XY של בארות הלוחות או מיקומים מרובים של תא זרימה. במידת הצורך, תפרו יחד מקטעים מרובים של באר אחת או תא זרימה אחד כדי ליצור שדה ראייה גדול יותר בהגדלה של פי 4.
הערה: המגבלה היחידה על מספר המיקומים שניתן להקליט היא כמה מהר שלב המיקרוסקופ יכול לנוע בין מיקומים בתוך מרווח הזמן של התמונה (למשל, צילום תמונה כל 12 שניות, 24 שניות וכו'). עם זאת, זה לא נדרש, ומעקב אחר קטעי וידאו שנעשו ממיקום יחיד עדיין יכול להיות מספר מספיק של מסלולים לניתוח סטטיסטי חזק. במחקר זה נעשה שימוש במיקרוסקופ Nikon Eclipse Ti-U, והשלב האוטומטי הניתן להזזה שימש להקלטת תמונות במספר בארות בו זמנית. עם זאת, כל מיקרוסקופ עם יכולת הקלטה ניתנת לתכנות יעבוד. המיקרוסקופ חייב להתחבר למחשב ולהיות מסוגל להקליט תמונות לתוכנית או לכונן קשיח.
3. ניתוח גודל אמבה
- פתח את קובץ המיקרוסקופ בתוכנת ההדמיה. אפשרויות ייבוא של פורמטים ביולוגיים ייפתחו. בתיבת הדו-שיח, ודא שמתקיימים התנאים הבאים: תצוגת מחסנית; תצוגה עם: בחירה-Hyperstack; ניהול זיכרון: סמן את השתמש במחסנית וירטואלית; מצב צבע: ברירת מחדל.
- ודא שאף אחד מהתפריטים הנפתחים או תיבות הסימון האחרים אינו מופעל. לחץ על אישור.
- פתח אפשרויות סדרה של תבניות ביולוגיות. בחר איזו סדרה נדרשת. אם רק מיקום אחד נרשם בכל פעם, סביר להניח שתהיה כאן רק אפשרות אחת. לחץ על אישור.
- בחרו 'תמונה' >'שכפל ' כדי לשכפל את הבאר היחידה שעובדים איתה בכל פעם, על-ידי בחירה בערוץ C אחד בלבד ובערוץ Z אחד בלבד.
- מכאן והלאה, לעבוד רק עם התמונה המשוכפלת לצורך מניפולציה וניתוח, לא עם המקור. בחר תמונה > הקלד > 8 סיביות. בחר תהליך > חיסור רקע.
- הגדר את הכדור המתגלגל ל- 10.0. בדוק את רקע האור. בדוק פרבולואיד הזזה.
- בחר תהליך > לשפר את הניגודיות. קבעו פיקסלים רוויים על 0.1% לטרופוזואיטים או 0.3% לקבוצת תאים וצברים.
- בחרו 'תמונה' > 'התאם את סף >' (> B&W המוגדר כברירת מחדל). Make Binary process ייפתח. בחר ברירת מחדל וסמן רקע שחור (של מסיכות בינאריות).
- בחר את הסף באגרסיביות כך שרוב כתמי הרקע לא יהיו גלויים, אך חלק כלשהו מכל אמבה יהיה גלוי.
- אם תוכנת ההדמיה הפכה את התמונה והרקע לבן, והאמבה שחורה, עבור אל ערוך > הפוך כדי לתקן אותה לרקע שחור ולאמבה לבנה.
- בחר תהליך> בינארי > סגור, במקרה שלתאים יש רווח קל בקרום החיצוני עקב היותם מחוץ למיקוד או תנועתיות של התא.
- בחר תהליך > > מילוי חורים בינאריים. הסר לכלוכים שאינם אמבה באמצעות כלי ציור הצורות וערוך מילוי >. אם בשלב זה הרקע לבן והאמבה שחורה, בחרו 'עריכה' >'היפוך'. בשלב זה, התמונה אמורה לייצג את התמונות הבינאריות באיור 2.
- כדי לתעד את הגודל של כל אמבה, בחר נתח > נתח חלקיקים. גודל הסט: 10-infinitiy, מעגליות: 0-1, הצגה: כלום. סמן רק את הצג תוצאות וסכם.
- שמור את קבצי ה- CSV שמופיעים. בחרו 'קובץ' >'שמור בשם' > Tiff וערכו את השם למפרט הרצוי.
4. הכנת קבצי מיקרוסקופ למעקב (ניתוח תנועתיות)
- פתח את קובץ המיקרוסקופ בתוכנת ההדמיה. אפשרויות ייבוא של פורמטים ביולוגיים ייפתחו.
- בתיבת הדו-שיח, ודא שמתקיימים התנאים הבאים: תצוגת מחסנית; הצג באמצעות: בחירה-Hyperstack. לניהול זיכרון, סמן את השתמש במחסנית וירטואלית. לחץ על אישור.
- אפשרויות סדרת הפורמטים הביולוגיים ייפתחו. בחר איזו סדרה נדרשת. אם רק מיקום אחד נרשם בכל פעם, סביר להניח שתהיה כאן רק אפשרות אחת. לחץ על אישור.
- בחרו 'תמונה' >'שכפל'. מכאן והלאה, עבוד רק על החלק המשוכפל של הסרטון במקום לעבוד על הקובץ המקורי.
הערה: שכפל את המקטע של הסרטון שנמצא במעקב באותו זמן. לשם כך יש לדעת את המסגרות התואמות את הדקות הנדרשות לניסוי. לדוגמה, אם מישהו רוצה לעקוב אחר השעההראשונה ולצלם תמונות כל 24 שניות, צריך להיות 150 פריימים בשעה1. לכן, 1שעה רחוב יהיה מסגרות 1-150, 2nd שעה יהיה 151-300, וכן הלאה. - בחר תמונה > הקלד > 8 סיביות. בחר תהליך > חיסור רקע. הגדר את כדור הגלגול ל- 10.0.
- בדוק את רקע האור. בדוק פרבולואיד הזזה. בחר תהליך > לשפר את הניגודיות והגדר אותה על 0.1%.
- סמן את כל פרוסות x# (לדוגמה, כל 150 הפרוסות). בטל את הסימון של Normalize. בחרו 'תמונה' > 'התאם את סף >' (> B&W המוגדר כברירת מחדל).
- Make Binary process ייפתח. בחר ברירת מחדל. בדוק את הרקע השחור (של מסכות בינאריות).
- סף אגרסיבי, כך שרוב נקודות הרקע אינן גלויות, אך חלקים מסוימים של האמבה גלויים. בשלב זה, אם הרקע לבן והאמבה שחורה, בחר Edit > Invert. הרקע צריך להיות שחור, והאמבה צריכה להיות לבנה.
- בחר תהליך > בינארי > סגור. בחר תהליך > > מילוי חורים בינאריים. בחר File > Save As >- Tiff וערוך את השם למפרט הרצוי. הקובץ מוכן כעת למעקב.
5. ניתוח תנועתיות באמצעות מעקב
- מתוכנת ההדמיה עם קובץ tiff הדרוש כדי לעקוב פתוח, עבור אל תוספים > מעקב > Trackmate. הגרסה של Trackmate תיפתח.
- בחר הבא. בחר את גלאי LoG מהתפריט הנפתח של הגלאי. הגדר קוטר כתם משוער: 35.0 מיקרון, סף 1.0. בטל את הסימון של האפשרות השתמש במסנן חציון ובצע לוקליזציה של תת-פיקסלים.
- לחצו על 'תצוגה מקדימה' בפרוסה הראשונה, האמצעית והסופית. ודא שכל האמבות נלכדות על ידי עיגול סגול ושפגמי רקע או ממצאים אינם כלולים.
- בחר הבא. זה יתחיל עיבוד שלב זה עשוי לקחת כמה דקות לעשות. פס זיהוי בחלק העליון של המסך יציין כמה מהתהליך הושלם.
- בחר הבא כשתתבקש. בסף התחלתי, בחר הבא שוב מבלי לבחור דבר. בחר Hyperstack Displayer בעת בחירת תצוגה. בחר הבא.
- הגדר מסנן בנקודות ובחר הבא מבלי לבחור דבר. בחר כלי מעקב; לשם כך, בחר LAP Tracker (במקום Simple LAP Tracker). בחר הבא.
- בקישור מסגרת למסגרת, הגדר את המרחק המרבי ל- 40 מיקרומטר. בסגירת פער במקטע מסלול, בדוק אפשר סגירת פער. הגדר את המרחק המרבי לכך ל- 100 מיקרומטר ואת מרווח המסגרת המרבי ל- 4. אל תבחר דבר עבור פיצול מקטע מסלול או מיזוג מקטע מסלול.
- בחר הבא. ביצוע המעקב עשוי להימשך זמן רב. כאשר הוא הושלם, החלון צריך לומר: מעקב נעשה x s בתחתית. בחר הבא.
- בחר הבא שוב לאחר השלמת המעקב. הגדרת מסננים ברצועות: בחר/י בסימן + בפינה השמאלית התחתונה כדי להוסיף מסנן עבור מספר המקומות ברצועות. הגדר את המסנן ל'מעל' עבור 93% מהמסגרות לפחות (לדוגמה, 150 מסגרות יזדקקו למינימום של 140 ספוטים).
הערה: גרסאות מסוימות של תוכנה זו ידרשו בחירת המקבילה ההפוכה, כגון מתחת לפחות 7%, אשר ניתן לעשות זאת על ידי גרירת קו שמאלה או ימינה כדי להגיע למספר הרצוי. - בחר הבא. אפשרויות תצוגה יופיעו. ודא שהרצועות בכל מסגרת אינן מעוותות או מוזרות באופן התנועה של האמבה לפני שמירת קובץ ה- XML של Trackmate או קובץ ה- CSV של Trackmate.
- אם יש צורך למחוק רצועה, לחץ על TrackScheme. TrackScheme יציג את כל הרצועות ונקודות המגע של האמבה בכל המסגרות.
הערה: סיבות אפשריות למחיקת רצועה הן אמבה שהלכה על/ירדה משדה הראייה במהלך ההקלטה, מסלול מעוצב בצורה לא הולמת בהשוואה לנתיב האמיתי של האמבה, או מסיבה כלשהי, המסלול או האמבה הם חריגים בנתונים על פי הפרמטרים הניסיוניים וכו '. - לחץ על האמבה כדי להדגיש את הכתם ירוק על המסך וב- Trackscheme לכתם יהיה ריבוע ירוק המקיף אותו. פעולה זו תציין איזה מסלול או נקודה יש להסיר.
- לחץ לחיצה ימנית על הרצועה שיש להסיר ובחר מסלול שלם. לחץ על הלחצן מחק .
- לאחר סקירת כל הרצועות, שמור את קובץ המעקב על ידי לחיצה להציל בפינה השמאלית התחתונה של חלון הקופץ של תוכנת המעקב. שמור את קובץ ה- XML באמצעות שם הווידאו. לחץ על המשך כדי לחזור לחלון המוקפץ של תוכנת המעקב.
- לחץ על Tracks ובחר Tracks מימין. לחץ על ייצוא ל- CSV ושמור את קובץ ה- CSV.
- אם הגיליון האלקטרוני אינו מזהה את ה- CSV המתקבל, בצע את השלבים הבאים.
- פתח את קובץ ה- CSV שנשמר בפנקס הרשימות. לחץ על Control + A ועל Control + C כדי להעתיק את כל התוכן.
- פתח את הגיליון האלקטרוני ולחץ על Control + V כדי להדביק אותו בגיליון האלקטרוני. כל הנתונים יהיו בתא אחד.
- כאשר התאים עם הנתונים עדיין נבחרים, עבור אל נתונים > טקסט לעמודות. בחר מופרד > הבא. בחר פסיק > הבא > סיום. שמור קובץ זה כקובץ CSV החדש.
- ל-CSV המעקב יהיו פרמטרים רבים זמינים לניתוח (איור 3). העתק והדבק את הפרמטרים הרצויים לקובץ CSV חדש (איור 4) כדי לנתח. הפרמטר המשמש כאן מתואר להלן.
- סה"כ מרחק נסעה: המרחק הכולל שעברה אמבה במיקרומטר.
- המרחק המרבי שעברה: המרחק המרבי בנתיב האמבה מנקודת ההתחלה שלה - זו לא בהכרח נקודת הסיום
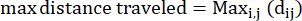
כאשר dij הוא המרחק לכל נקודה I לכל נקודה j על המסלול. - יחס כליאה: יחס הכליאה מציין עד כמה האמבה הייתה יעילה בהתרחקות מנקודת ההתחלה שלה: האם היא נעה בקו ישר הרחק מנקודת ההתחלה שלה (ערך קרוב ל-1), או שהיא נשארה קרוב יחסית לנקודת ההתחלה שלה (ערך קרוב ל-0).
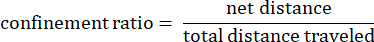
- מהירות ממוצעת: מהירות ממוצעת של האמבה על פני המרחק הכולל שעברה במיקרונים לשנייה.
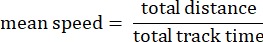
- עקבת מסלול: המרחק בין נקודת ההתחלה של אמבה לנקודת הסיום במיקרונים.
הערה: ניתן למצוא את כל הפרמטרים של ניתוח תוכנות הדמיה וההגדרות שלהם כאן: https://imagej.net/plugins/trackmate/analyzers/. זכור כי כל קובץ שנשמר הוא סיכום של הרצועות שניתחו באותו זמן. כדי לייצר ניתוח נתונים חזק, יהיה צורך לשלב כראוי את התוצאות ממספר שכפולים, שעות וכו', כפי שמתאים לנתונים (איור 5 ואיור 6).
תוצאות
כדי להשיג הצלחה באמצעות שיטה זו, ישנם מספר חלקים כלליים מכריעים שיש לקחת בחשבון. הראשון הוא ההתקנה הפיזית של האמבה והמיקרוסקופ, השני הוא ניצול נכון של תוכנת ההדמיה, והשלישי הוא ייצוא וניתוח נתוני ההדמיה בצורה משמעותית.
לפני תחילת ההקלטה המיקרוסקופית, חיוני לוודא שהאמבה ממוקדת כראוי בשלב המיקרוסקופ (איור 1). במקום להתמקד באמבה במישור Z האופטימלי ביותר עבור פרטים בודדים, כאן אידיאלי לראות אותן מעט מאוד מחוץ לפוקוס כך שהן יהפכו לנקודות שחורות מוצקות בניגוד למצבן השקוף כאשר מסתכלים עליהן בפירוט. צפייה בהם בדרך זו תאפשר לתוכנת ההדמיה ולתוכנת המעקב למצוא כל אמבה בודדת בקלות רבה יותר כאשר הקובץ מומר לבינארי (איור 2). הידיעה מה המטרה בתוכנת ההדמיה - כלומר, שכל אמבה תופיע כעיגול לבן מוגדר היטב, עשויה לעזור לקורא למצוא את המיקוד הנכון בעת צילום התמונות המקוריות בשדה בהיר. לאחר מכן, כאשר כל עיגול מוגדר בבירור, תוכנת המעקב תוכל לעקוב כראוי אחר התנועתיות של כל אמבה ולאסוף את מדדי התנועתיות האינדיבידואליים עבור כל אמבה.
לאחר ביצוע ההוראות שלב אחר שלב בפרוטוקול המפורט לעיל עבור תוכנת ההדמיה ותוכנת המעקב והפניה לווידאו המשויך כדי לסייע בשלבים הראשונים המכריעים של השימוש בתוכנת ההדמיה, הגיע הזמן לייצא את נתוני תוכנת ההדמיה לצורך כימות וניתוח נתונים. התפוקה הגולמית מתוכנת המעקב תהיה דומה למה שנראה באיור 3. כאן, חשוב לבדוק שמספר המקומות במסלולים שהוא ייצא נכון למצופה (למשל, אם מישהו ביקש ש-93% מהפריימים יהיו נוכחים בכל רצועה והיו לו 100 פריימים בסך הכל בסרטון, כל רצועה מיוצאת צריכה להיות גדולה או שווה ל-93 נקודות). אם הכתמים אינם תואמים את הדרישות, ייתכן שיהיה צורך לחזור על שלבי תוכנת המעקב כדי להבטיח שהפרמטרים הנדרשים הוגדרו כראוי.
מהייצוא הגולמי, התחל לבנות את גיליון ניתוח הנתונים (איור 4). חשוב לזכור שכל רצועה מייצגת אמבה סינגולרית בתוך העתק יחיד. לכן, כפי שניתן לראות באיור 4, איור 5 ואיור 6, כל הרצועות עבור ערכת פריימים או נקודת זמן חייבות להיות ממוצעות (איור 4), ותהליך זה חייב להיעשות עבור כל קבוצת פריימים או נקודת זמן בסדרה שלמה (כלומר, אם מצלמים סך של 3 שעות וידאו ונקודות הנתונים מחולקות לפי שעה, לאחר מכן יהיו שלוש קבוצות פריימים / שעות לנתח לכל שכפול, איור 5). לבסוף, משילוב זה של ממוצעים בכל מסגרת בכל שכפול, יש לשלב את הנתונים הממוצעים מכל שכפול למרחב אחד, כך שניתן יהיה לנתח את הנתונים על פני מספר עותקים משוכפלים כדי לקבוע את הממוצע האמיתי וסטיית התקן לכל נקודת זמן (איור 6).
הממוצעים מכל משוכפל ישמשו לניתוח נתונים, והממוצע המשולב וסטיית התקן או שגיאת התקן מכל המשוכפלים ישמשו לייצוג גרפי (כפי שניתן לראות בדוגמה באיור 7). אם מנתחים נתונים כפי שעשינו כאן, כלומר דגימות מרובות שנמצאות במעקב על פני נקודות זמן מרובות, הכי מתאים לנתח אותם באמצעות מדידה חוזרת דו-כיוונית ANOVA עם מבחן טוקי פוסט הוק. זה מאפשר לנו לקבוע את ההבדלים בין הדגימות בכל נקודת זמן וגם לקבוע את ההבדלים בתוך כל מדגם לאורך זמן.

איור 1: אמבה מופיעה כצורות כהות אטומות במיקרוסקופ שדה בהיר. מוגדל בתמונה מייצגת של אמבה בהגדלה פי 4, מעט מחוץ למיקוד כדי להופיע כעיגולים כהים מוצקים. סרגל קנה מידה = 500 מיקרומטר. לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
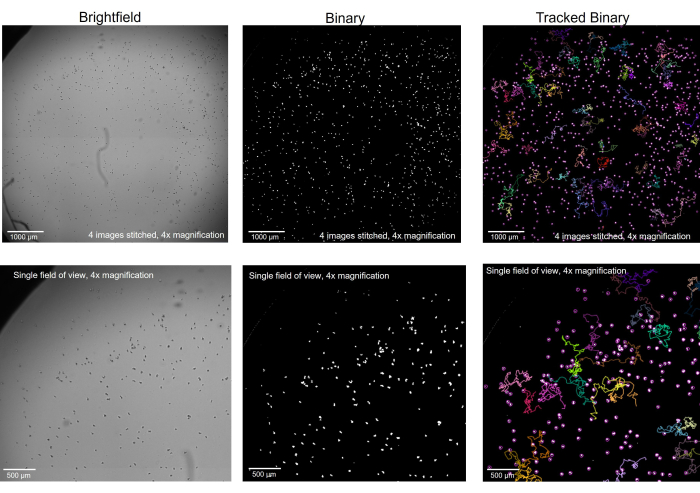
איור 2: אמבה שצולמה במיקרוסקופ שדה בהיר שהומרה לבינארית ונמצאת במעקב. אמבה בהגדלה פי 4, בשדה בהיר (משמאל), הומרה לבינארית עם חפצים שהוסרו (באמצע), ולאחר מעקב, עם רצועות נבחרות המוצגות (מימין). שורה עליונה: תמונה שלמה היטב על ידי תפירת 4 תמונות יחד, סרגל קנה מידה = 1000 מיקרומטר. שורה תחתונה: גודל תמונה בודדת לפני התפירה, סרגל קנה מידה = 500 מיקרומטר. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
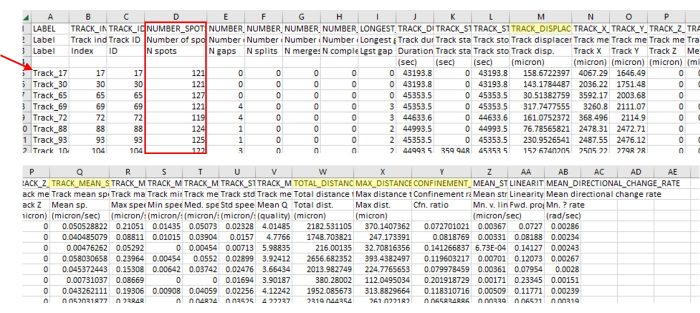
איור 3: פלט ניתוח נתונים מיידי מתוכנת ההדמיה והמעקב מכיל מגוון רחב של סוגי נתונים. הפלט המייצג מהמעקב מוצג באיור 2, שהוא סרטון יחיד. שים לב למספרי הדילוג על מספרי המסלולים (חץ אדום), המציינים שתוכנת המעקב הסירה כראוי רצועות שלא התיישרו עם הפרמטרים. בדוגמה זו, רק 71 מתוך 5385 מסלולים מזוהים תואמים לפרמטרים. יחס זה הוא נורמלי, בהתחשב בהנחיות קפדניות כאלה. מספר רצועות זה תואם גם את מספר האמבות המשמשות בפרוטוקול זה (לדוגמה, אם לוחית בת 96 בארות נזרעת עם 200 μL של 7.5 x 103 CFU/mL לכל באר, אז יש לצפות לכ-1,500 אמבות בבאר ובערך למספר זה הנראה בשדה ראייה של 4 תפרים. סביר להניח שיהיו הרבה יותר מסלולים מאשר אמבה כאשר אמבה הולכת על שדה הראייה ומחוצה לו, ומייצרת מסלולים חדשים). מספר מסלול זה ניכר שוב באיור 2, שם ניתן לראות רק מסלולים מעטים בהשוואה למספר רב של נקודות. בדוק תמיד את מספר המקומות במסלול (תיבה אדומה) כדי לוודא שרק אורכי המסלול הנכונים כלולים. לדוגמה, כאן, אנו כוללים יותר מ 93% של מסגרות, והיו 127 מסגרות בסעיף זה, כך שכל מספרי כתמים צריך להיות מעל 118. מודגשות בצהוב המדידות שהוזכרו בפרוטוקול. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
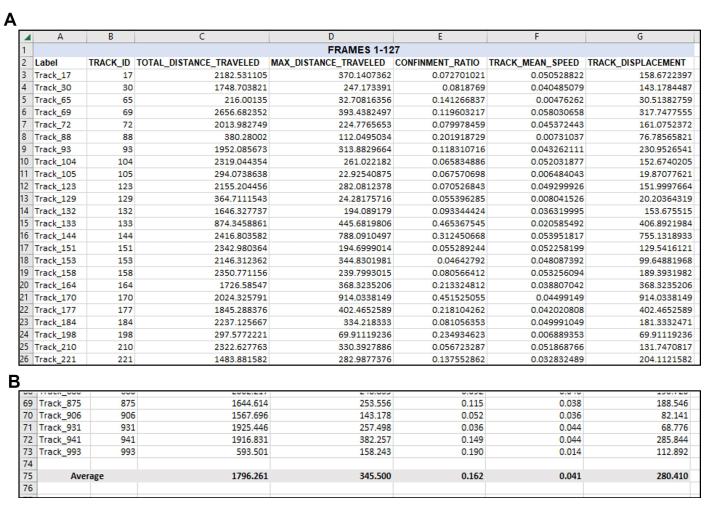
איור 4: הנתונים מאורגנים מחדש לפי הצורך לאחר פלט הנתונים הראשוני. ארגון הנתונים המבוקשים לאחר הייצוא. (A) המדידות הרלוונטיות שנבחרו לכימות הועתקו מייצוא תוכנת המעקב (איור 3) והודבקו בגיליון אלקטרוני חדש. (ב) לאחר ארגון המדידות הרצויות בגיליון האלקטרוני החדש, יש לחשב את הממוצע של כל רצועה. כך, בדוגמה מייצגת זו, המרחק הכולל, המרחק המרבי, הכליאה, המהירות הממוצעת והתזוזה של כל רצועה שמישה בסרטון יחיד זה הוערכו למספר יחיד עבור כל מדידה. שימו לב שוב כאן שבסך הכל 71 רצועות עמדו בפרמטרים של מסגרות 1-127. סביר להניח שיהיה מספר שונה של רצועות מתאימות מכל סרטון או ערכת פריימים שנותחו, כפי שניתן לראות באיור 5. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
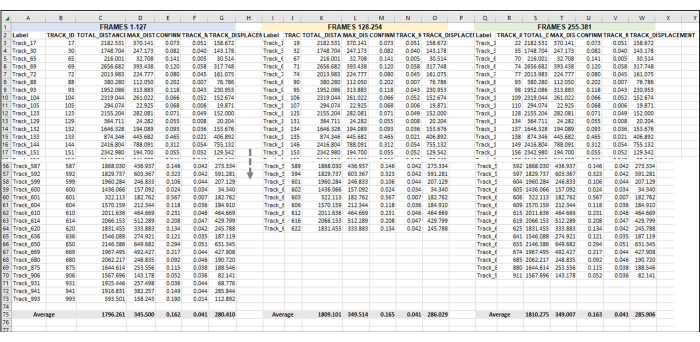
איור 5: מסגרות רציפות שהוגדרו לניתוח של סדרת תמונות או סרטון שלמים. זוהי תמונה מייצגת של הוספת סרטונים עוקבים לניתוח נתונים. בהנחה שפריימים 1-127 מהווים את שעה 1 של הסרטון (כאן, שעה אחת מורכבת מצילום תמונה אחת ל-28 שניות). מסגרות 128-254 יהיו שעה 2 של וידאו, ומסגרות 255-381 יהיו שעה 3 של וידאו. שלב את הנתונים המיוצאים (כפי שיוצאו באיור 3) מכל השעות לגיליון אחד כדי לעבוד איתם בבת אחת. כפי שנעשה באיור 4, הנתונים מכל המסלולים השמישים עבור כל ערכת פריימים יהיו ממוצעים. כל דבר באיור 5 הוא נתונים מהעתק אחד או מדגם אחד. אם יש 3 עותקים משוכפלים, אז יהיו 3 כרטיסיות גיליון אלקטרוני נפרדות זהות, והנתונים מהן יוזנו לכרטיסייה חדשה, כפי שמודגם באיור 6. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
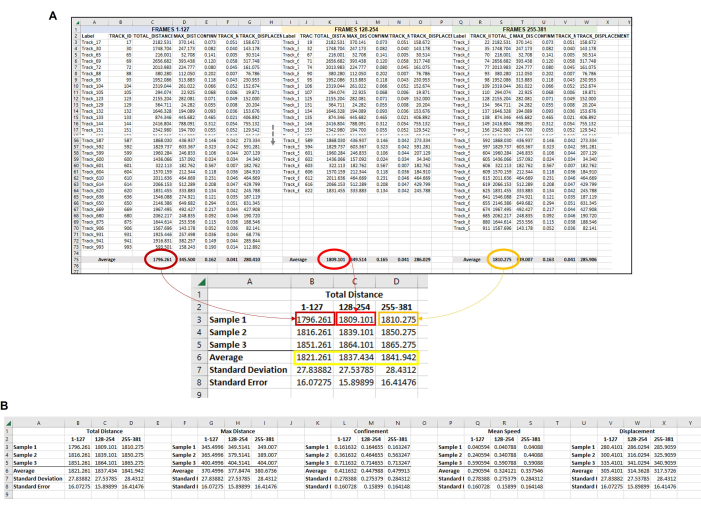
איור 6: נתונים מורכבים מכל האמבות בכל ערכת מסגרות המשמשות לייצוג שכפול יחיד. (A) יש לאסוף את כל הממוצעים מכל מסגרת שנקבעה באיור 5 במיקום מרכזי כדי שניתן יהיה להשלים את ניתוח הנתונים הסופי. בתמונה מייצגת זו מוצגים ממוצעי המרחק הכולל משלוש ערכות המסגרות (או השעות) הראשונות. יש לחזור על פעולה זו עבור כל דגימה/תנאי/שכפול שמופעל. לאחר מכן, יש לחשב את הממוצעים מכל מדגם מכל ערכת מסגרות (תיבה צהובה). זוהי נקודת הנתונים הסופית שממנה ניתן לחשב סטיית תקן ושגיאת תקן. הממוצעים הנפרדים מכל מדגם (כאן, המספרים ב- B3, B4 ו- B5 עבור מדגם המרחק הכולל 1; C3, C4 ו- C5 עבור מדגם מרחק כולל 2 וכו') ישמש להפעלת הניתוח הסטטיסטי. תהליך זה יחזור על עצמו בכל המדידות הנדרשות (מרחק מקסימלי, כליאה, מהירות ממוצעת, תזוזה וכו ') (B) כדי ליצור ייצוג גרפי של הכימות, השתמש במספרים בתיבה הצהובה כנקודות נתונים לשימוש עבור הגרף הסופי. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
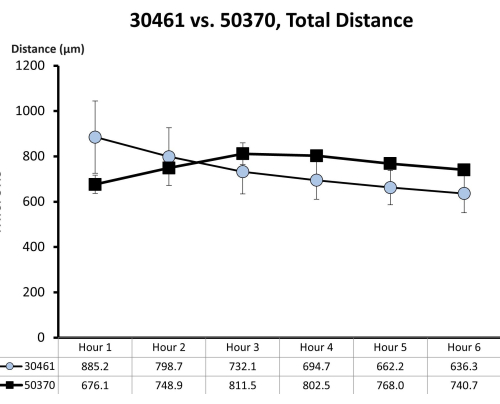
איור 7: נתוני תנועתיות גולמיים עבור ATCC 30461 ו-ATCC 50370. נתונים גולמיים (המיוצגים כאן כממוצע ± SE עבור כל שעה) נאספו בעבר ולא פורסמו עבור מחקרים קודמים 18,19. כימות תנועתיות זה מדגים כי שתי אמבות אלה דומות ב-6 השעות הראשונות של תנועתיותן (כפי שנמדדו על ידי המרחק הכולל במיקרונים שנסעו באותה שעה) בתמיסת רינגר של 1/4 על משטח זכוכית. כל נקודת זמן מייצגת שלושה העתקים נפרדים, שכל אחד מהם מורכב מ-20-200 רצועות אמבה. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
Discussion
היכולת לעקוב ולכמת את תנועתיות האמבה כגון Acanthamoeba, שהם אורגניזמים מיקרוסקופיים דבקים שאינם מושפעים מתנועה בראונית במהירות נמוכה 9,10, חושפת כמות משמעותית של מידע על התנהגות אמבה ויכולה לשפר מאוד את ההארה לגבי שיטות למניעת AK. פרוטוקול זה פורט בפרסומים האחרונים, והנתונים אושרו על ידי או בשילוב עם ניתוחי נתונים דומים אחרים18,19. נציין כאן כי מהירות האמבה המפורטת בשיטה זו דומה למה שפורסם על ידי קבוצות אחרות. פרוטוקול זה ראוי לציון ביכולתו לשמש למעשה על כל טיפול פני שטח או אמבה, שהוא שקוף לאור, אך חשוב לציין כי בעוד פרוטוקול זה יכול להיות שונה באופן פוטנציאלי לעבוד עם אורגניזמים אחרים, הוא עבר אופטימיזציה רק עד כה עבור Acanthamoeba. הפרוטוקול הנ"ל עבד היטב באופן עקבי במעבדה, אך השינויים הבאים לשינוי השיטה כך שתתאים לצרכים אחרים או לפתרון בעיות במצבים קשים מסופקים.
קביעת פרמטרי מעקב: בפרוטוקול לעיל פורטו קישור מסגרת למסגרת, סגירת פערים, פער מסגרת מקסימלי ומספר המקומות על המסלול, שהיו הישימים ביותר עבור פרסומים אחרונים. עם זאת, ייתכן כי פרמטרים אחרים יהיו מתאימים יותר לצרכים אחרים. יתר על כן, ייתכנו פרמטרים שאחרים ירצו להשתמש בהם אך לא נעשה בהם שימוש כאן (כגון המרחק המרבי של מסלול תחת מסנני מסלול). מידע נוסף על כל פרמטר ומה הוא מתאר ניתן למצוא כאן באתר (ראה הפניה21). כאשר מחליטים אילו פרמטרים ומסננים הם הרלוונטיים ביותר לניסויים אחרים וכיצד יש להגדיר אותם, חשבו מה רציונלי סטטיסטית לפרויקט מסוים; לדוגמה, האם מישהו ירצה נתונים מאמבה שיוצאת משדה הראייה ואז חוזרת אליו? או שמא נרצה לשמור את הנתונים מאמבה שלא זזה הרבה אך עדיין תויגה כרצועה? ומה הגיוני למספר הפריימים ומשך הזמן בין המסגרות שבהן התמונות הוקלטו.
שינוי גודל הכתמים: מצאנו כי קשה מאוד עם כל תוכנה או כל מערך מיקרוסקופ - בהתבסס על טכנולוגיות ותוכנות עדכניות הזמינות למעבדות סטנדרטיות - לעקוב במדויק אחר מספר האמבות במצטבר כפי שהוא משתנה לאורך זמן. אם אתם עוקבים אחר גודלו של כתם ומנסים להתאים אותו למספר האורגניזמים שבו, השתמשו בעקומה סטנדרטית שנוצרה לאחר ניסויים חוזרים ונשנים כדי לחזות מתמטית את גודלו של צבר. לדוגמה, אגרגטים נוצרו על ידי זריעת בארות עם מגוון מספרים של אמבה, כגון 8, 16, 32, 125, 250, 500, 1,000 ו -2,000 תאים לכל באר. נוצרו תמונות Timelapse בנקודות זמן שונות של כל באר במשך 24 שעות. כל ספרואיד (שהיה במספר ידוע של אמבות) נותח עבור שטח דו-ממדי, ועקומה סטנדרטית נוצרה כפונקציה של ספירת אמבות לעומת גודל ספרואיד לאורך זמן. ניסוי זה חזר על עצמו לפחות פי שלושה כדי לתת לנו סטיית תקן מתאימה של כל עקומה שנוצרה.
קביעת הכיווניות של האמבה: בעוד שזה לא יכול להיות הכרחי עבור מחקרים מסוימים, זה יכול להיות מועיל להבין את הכיווניות של האמבה. זה יספק נתונים על ההשפעות הכימוטקטיות של הטיפול או הניסוי. נתונים אלה יכולים לשמש גם ליצירת נתונים חזותיים (גרפיים) וכמותיים. זה זמין באמצעות Chemotaxis and Migration Tool, שהוא תוסף חינם עבור תוכנת הדמיה. הוא זמין באתר יחד עם מדריך היישום ותמונות וסרטים לדוגמה (ראה טבלת חומרים).
דינמיקת תנועה מפורטת: קבוצות אחרות בחנו את דינמיקת ההתפשטות ואת המסלולים הדיפוזיים באמצעות ניתוחים סטטיסטיים מתקדמים ומפותחים ביותר מעבר למה שנדון כאן 9,10,22. אלה יכולים להיחשב על בסיס הצרכים של המשתמש.
כמו בכל שיטה מוצלחת, הפרוטוקול המפורט בכתב יד זה עבר סבבים רבים של פתרון בעיות כדי להשיג עקביות. בעוד שניתן ליצור סרטונים מצוינים באיכות גבוהה הניתנים למעקב על ידי צילום תמונה בכל שנייה (או מהר ככל שהמיקרוסקופ מסוגל, זה יכול להיות פחות משנייה), זה יוצר קבצים גדולים במיוחד, שיכולים להיות קשים לעבודה איתם. זה מתאים באופן מציאותי רק לסרטונים לטווח קצר מאוד שהוקלטו במשך מקסימום של כמה דקות. לעומת זאת, על ידי צילום תמונה כל כמה שניות, מצאנו כי התנהגות האמבה עדיין ניתנת למעקב מאוד בהתבסס על המהירות של מין זה, וניתן ליצור סרטונים מעשיים במשך שעות או ימים. מצאנו כי משך הזמן המרבי בין תמונות הוא 30 שניות, שהוא הזמן הדרוש למעקב מדויק אחר אמבה על ידי תוכנת הדמיה. יש לקחת בחשבון את מרווח הזמן שהמשתמש בוחר באמצעות האילוצים הידועים של כמה מהר המיקרוסקופ יכול להקליט תמונות, כמה תמונות לכל באר דרושות, וכמה בארות נרשמות בכל מרווח. באופן דומה, הפרמטרים המוזכרים בפרוטוקול זה לגבי תזוזה מקסימלית, מרווחי פריימים מותרים, מינימום פריימים דרושים וכן הלאה, נקבעו באמצעות ניסוי וטעייה על ידי מעבדה זו כדי ליצור מידע מסלול הכולל את רצועות האמבה השלמות ביותר ולהתעלם מרעש שנוצר על ידי רצועות לא שלמות, אמבה המצטרפת או עוזבת את הבמה באמצע וידאו, או שגיאות הנוצרות על ידי בלבול תוכנת הדמיה כגון כאשר שתי אמבות נפגשות באמצע המסילה ואז נפרדות. טעויות מסוג זה ומסלולים חלקיים הם גבוהים באופן מובן בעת יצירת סרטונים ארוכים במיוחד (שעות עד ימים) של אורגניזמים ביולוגיים מיקרוסקופיים הנמצאים באינטראקציה מתמדת זה עם זה, והם הסיבה לכך שיש לדחות חלק גדול מאוד מהמסלולים השגויים. יש לציין כי שלב מילוי החורים בפרוטוקול הוא, מניסיונה של מעבדה זו, חשוב להפחתת טעויות באופן שבו תוכנת ההדמיה עוקבת אחר אמבה. על ידי וידוא שכל אמבה היא עיגול מוצק במקום, לפעמים, סופגנייה או צורת c, סביר הרבה יותר שהתוכנה תוכל לעקוב אחר כל אמבה בהצלחה.
יתר על כן, כפי שנדון, ישנם פרמטרים רבים הזמינים למשתמש לניתוח תמונה או וידאו. בהתבסס על צרכי הניסוי, הפקנו באופן עקבי את המרב ביותר מניתוח המרחק הכולל, המרחק המרבי, המהירות והתזוזה. אלה נידונים לעומק (כולל שימוש בזנים ומשטחים שונים) עם פרשנויות גרפיות בפרסומים קודמים18,19. ארבעת הפרמטרים הללו מאפשרים למשתמש להסיק את יכולתה של אמבה לנוע מרחקים ליניאריים וכמה זמן לוקח לה לעשות זאת, מה שמסייע בהבנת התנהגותה בכל הקשור לזיהום עדשות מגע. אחזור וניתוח פרמטרים אלה היא עבודה גדולה, כפי שמפורט בנתונים שלנו. במהלך העבודה עם גיליונות אלקטרוניים גדולים ורבים מפלט תוכנת ההדמיה, הגבלנו שגיאות לא מכוונות על-ידי יצירת תבניות גיליון אלקטרוני נעולות שחישבו באופן אוטומטי את כל הניתוחים הנדרשים. עם זאת, שיפור אפשרי לשיטה זו יהיה לכתוב סקריפט שיכול לטפל בנתונים אלה ולמיין ולנתח אותם.
לסיכום, מתוארת כאן שיטה נגישה ומדויקת למדידת גודל ותנועתיות אקנתמואבה בתנאים רבים ושונים. הוכחנו כי ניתן ליישם שיטה זו על זנים רבים ושונים של אמבה, ותיארנו כי בעוד שעשויים להיות פרמטרים פשוטים להשגת מידע תנועתיות, מערך ניסויי זה יכול להיות מותאם מאוד לכל צורך ספציפי.
Disclosures
כל הכותבים הם עובדי Alcon Research, LLC.
Acknowledgements
עבודה זו מומנה על ידי Alcon Research, LLC.
Materials
| Name | Company | Catalog Number | Comments |
| ¼ Ringer’s solution | ThermoFisher Scientific | BR0052G | Oxoid Ringers Solution Tablets. Follow directions to make one-quarter strength instead of full strength Ringers. |
| 10 µL pipette | Eppendorf Research | 3123000039 | 2 µL-20 µL single channel |
| 10 µL pipette tips | Neptune Scientific, San Diego, CA, USA | BT10.N | 10 µL Universal Barrier Tip |
| 48 well plate | Millipore Sigma, | CLS3548 | Corning Costar TC-Treated Multiple Well Plate: polystyrene plate, flat bottom wells, sterile, with lid |
| 50 mL conical tubes (1 for each sample, 1 for each pass 2 sample) | Fisher Scientific | Falcon 352098 | Falcon 50 mL High Clarity Conical Centrifuge Tubes, polypropylene |
| 96-well plate | Millipore Sigma, Burlington, MA, USA | CLS3596 | Corning Costar TC-Treated Multiple Well Plate: polystyrene plate, flat bottom wells, sterile, with lid |
| Acanthamoeba | American Type Culture Collection, Manassas, VA, USA | ATCC 30461 | This protocol has been verified for ATCC 50655, 30872, 50702, 30010, 30461, 50370, 50703, 30137, PRA-115, and PRA-411 |
| Axenic culture media (AC6) | Made in house | n/a | Containing 20 g biosate peptone, 5 g glucose, 0.3 g KH2PO4, 10 µg vitamin B12, and 1 glass5 mg l-methionine per liter of distilled deionized water. Adjust pH to 6.6-6.95 with 1 M NaOH and autoclave at 121 °C for 20 min, store at room temperature for up to 3 months. |
| Centrifuge and appropriate rotor | Thermo Scientific, Waltham, MA, USA | Sorvall ST 40R | Any equivalent centrifuge and rotor are acceptable so long as it can spin 50 mL conical tubes at 500 x g for 5 min |
| Chemotaxis and Migration Tool | Free software based on ImageJ, available at https://ibidi.com/chemotaxis-analysis/171-chemotaxis-and-migration-tool.html | ||
| Disposable hemocytometer | Bulldog Bio, Portsmouth, NH, USA | DHC-N01 | Neubauer Improved 2-Chip Disposable Hemocytometer, Individually packaged, Nonpyrogenic |
| ImageJ software with Trackmate plugin (this protocol written with Trackmate version 6.0.2.) | Free software developed by the National Institutes of Health, available at imagej.net. Trackmate plugin available at https://imagej.net/plugins/trackmate/ | ||
| Microscope, preferably with automated moveable stage | Nikon, Tokyo, Japan | A Nikon Eclipse Ti-U Microscope was used in this study and the automated moveable stage was utilized to be able to record images in multiple wells at a time. | |
| NIS-Elements software | Nikon | ||
| Serological pipette | Fisher Scientific, Hampton, NH, USA | BrandTech 26331 | BrandTech accu-jet pro Pipet Controller |
| Serological pipette tips | VWR | 5 mL: 76201-710 10 mL: 170356 25 mL: 89130-900 50 mL: 75816-088 | VWR Serological Pipette, Non-Pyrogenic |
| Sterile aluminum transmission flow cell | Biosurface Technologies Corporation, Bozeman, MT, USA | FC 81-AL | Anodized aluminum single channel transmission flow cell with 96-well plate footprint for use with an inverted microscope |
| T75 Flasks | VWR, Radnor, PA, USA | 734-2316 | VWR Tissue Culture Flask, 182.5 cm, Surface treated, Plug seal cap, Sterile |
References
- Randag, A. C., et al. The rising incidence of Acanthamoeba keratitis: A 7-year nationwide survey and clinical assessment of risk factors and functional outcomes. PLoS One. 14 (9), e0222092 (2019).
- Szentmary, N., et al. Acanthamoeba keratitis - Clinical signs, differential diagnosis and treatment. J Curr Ophthalmol. 31 (1), 16-23 (2019).
- Carnt, N., et al. Acanthamoeba keratitis: confirmation of the UK outbreak and a prospective case-control study identifying contributing risk factors. Br J Ophthalmol. 102 (12), 1621-1628 (2018).
- Verani, J. R., et al. National outbreak of Acanthamoeba keratitis associated with use of a contact lens solution, United States. Emerg Infect Dis. 15 (8), 1236-1242 (2009).
- Tu, E. Y., Joslin, C. E. Recent outbreaks of atypical contact lens-related keratitis: what have we learned. Am J Ophthalmol. 150 (5), 602-608 (2010).
- International Organization for Standardization. ISO 14729:2001/A1. Ophthalmic optics-Contact lens care products-Microbiological requirements and test methods for products and regimens for hygienic management of contact lenses. International Organization for Standardization. , (2010).
- . ASC Z80, Parent Committee Meeting Available from: https://www.thevisioncouncil.org/sites/default/files/ASCZ80_ParentCommitteeMinutes_February_27_2018_FINALMar19-2018.pdf (2023)
- Rayamajhee, B., et al. Acanthamoeba, an environmental phagocyte enhancing survival and transmission of human pathogens. Trends Parasitol. 38 (11), 975-990 (2022).
- Reverey, J. F., et al. Superdiffusion dominates intracellular particle motion in the supercrowded cytoplasm of pathogenic Acanthamoeba castellanii. Sci Rep. 5, 11690 (2015).
- Thapa, S., Lukat, N., Selhuber-Unkel, C., Cherstvy, A. G., Metzler, R. Transient superdiffusion of polydisperse vacuoles in highly motile amoeboid cells. J Chem Phys. 150 (14), 144901 (2019).
- Rudell, J. C., et al. Acanthamoeba migration in an electric field. Invest Ophthalmol Vis Sci. 54 (6), 4225-4233 (2013).
- Fukaya, S., Aoki, K., Kobayashi, M., Takemura, M. Kinetic analysis of the motility of giant virus-infected amoebae using phase-contrast microscopic images. Front Microbiol. 10, 3014 (2019).
- Fukaya, S., Takemura, M. Kinetic analysis of Acanthamoeba castellanii infected with giant viruses quantitatively revealed process of morphological and behavioral changes in host cells. Microbiol Spectr. 9 (1), 0036821 (2021).
- Cheng, H. J., Hsu, C. H., Hung, C. L., Lin, C. Y. A review for cell and particle tracking on microscopy images using algorithms and deep learning technologies. Biomed J. 45 (3), 465-471 (2022).
- Mathieu, E., et al. Time-lapse lens-free imaging of cell migration in diverse physical microenvironments. Lab Chip. 16 (17), 3304-3316 (2016).
- Svensson, C. M., Medyukhina, A., Belyaev, I., Al-Zaben, N., Figge, M. T. Untangling cell tracks: Quantifying cell migration by time lapse image data analysis. Cytometry A. 93 (3), 357-370 (2018).
- Piltti, K. M., et al. Live-cell time-lapse imaging and single-cell tracking of in vitro cultured neural stem cells - Tools for analyzing dynamics of cell cycle, migration, and lineage selection. Methods. 133, 81-90 (2018).
- Campolo, A., et al. Continuous real-time motility analysis of Acanthamoeba reveals sustained movement in absence of nutrients. Pathogens. 10 (8), 995 (2021).
- Campolo, A., Patterson, B., Lara, E., Shannon, P., Crary, M. Complete recovery of Acanthamoeba motility among surviving organisms after contact lens care disinfection. Microorganisms. 11 (2), 299 (2023).
- Crary, M. J., Walters, R., Shannon, P., Gabriel, M. M. Variables affecting the recovery of Acanthamoeba trophozoites. Pathogens. 10 (2), 221 (2021).
- . imagej Available from: https://imagej.net/plugins/trackmate/tutorials/getting-started (2024)
- Cherstvy, A. G., Nagel, O., Beta, C., Metzler, R. Non-Gaussianity, population heterogeneity, and transient superdiffusion in the spreading dynamics of amoeboid cells. Phys Chem Chem Phys. 20 (35), 23034-23054 (2018).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved