Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Reinigung und Visualisierung von Influenza A Virus Ribonukleoproteinkomplexe
In diesem Artikel
Zusammenfassung
Das Genom der Influenza A-Virus besteht aus acht separaten Komplexen von RNA und Proteinen, genannt virale Ribonukleoproteinkomplexe (vRNPs). Dieses Papier beschreibt die Glycerin Gradientenreinigung und Transmissionselektronenmikroskopie Visualisierung von Influenza-A-vRNPs.
Zusammenfassung
Das Influenza-A-Virus-Genom besteht aus acht negativen Sinne, einsträngige RNA-Moleküle, die individuell mit mehreren Kopien des Influenza-A-Nukleoprotein (NP) in virale ribonulceoprotein Partikel (vRNPs) verpackt. Die Influenza vRNPs sind innerhalb der Virushülle umgeben. Während Eindringen in die Zelle, jedoch sind diese vRNP Komplexe in das Zytoplasma freigesetzt, wo sie Zugriff auf den Host Kerntransport Maschinen zu gewinnen. Um die nukleare Import von Influenza vRNPs und die Replikation des Influenza-Genom-Studie, ist es sinnvoll, mit isolierten vRNPs Arbeit, so dass andere Komponenten des Virus nicht mit diesen Prozessen stören. Hier beschreiben wir ein Verfahren, um diese vRNPs von der Influenza A-Virus zu reinigen. Das Verfahren beginnt mit der Unterbrechung der Influenza-A-Virion mit Reinigungsmitteln, um die vRNP Komplexe aus den umhüllten Virion freizugeben. Die vRNPs werden dann von den anderen Komponenten des Influenza-A-Virion auf einem 33-70% diskontinuierliche Glycerin-Gradienten durch Geschwindigkeit Sedimentation abgeschieden. Die Fraktionen aus der Glycerin-Gradienten gewonnen werden dann über SDS-PAGE nach Färbung mit Coomassie-Blau analysiert. Die Peak-Fraktionen, die NP werden dann gepoolt und konzentriert durch Zentrifugation. Nach Einengen wird die Integrität der vRNPs durch Visualisierung der vRNPs durch Transmissionselektronenmikroskopie nach negativen Färbung überprüft. Das Glycerin Gradientenreinigung ist eine Abwandlung davon aus Kemler
Protokoll
Teil 1: Störung des Influenza-A-Virion
- Add 750 ul der MNT-Puffer (20 mM MES, 150 mM NaCl, 30 mM Tris, pH 7,5) in einem Beckman Polycarbonat-Zentrifugenröhrchen (11 mm x 34 mm) entwickelt, um in eine TLA-120,2 Rotor für den Einsatz in einem Beckman Optima fit Max-E Ultrazentrifuge.
- Add 500 ul des Influenza A Virus (H3N2 X-31 A/AICHI/68 Stamm; 2 mg / ml) in die Röhre. Mix das Virus mit der MNT-Puffer durch Auf-und Abpipettieren mehrmals.
- Zentrifuge für 10 Minuten bei 109.000 xg, 4 ° C in einem Beckman Optima Max-E-Zentrifuge mit einem Rotor TLA-120,2.
- Entfernen Sie den Überstand und das Pellet in 500 ul Störung Puffer (100 mM KCl, 5 mM MgCl 2, 5% (w / v) Glycerin, 50 mM Octylglucosid, 10 mg / ml Lysolecithin, 1,5 mM Dithiothreitol, 100 mM MES, pH 5,5).
- Vortex kräftig, und dann am 31. schütteln ° C für 20 Minuten in einem Eppendorf Thermomixer.
Teil 2: Glycerol Gradient Sediment Velocity Zentrifugation
- Bereiten Sie eine Glycerin-Gradienten, indem Sie die folgenden Mengen an Glycerin in einem Beckman UltraClear Zentrifugenröhrchen (13 mm x 51 mm): 1 ml 70% (v / v) Glycerin, 0,75 ml 50% Glycerin, 0,375 ml 40% Glycerin und 1,8 ml 33% Glycerin. Glycerin-Lösungen werden durch Mischen reinen Glycerins mit NM-Puffer (150 mM NaCl, 50 mM MES, pH 5,5) hergestellt. Ferner einen Ausgleich Röhre, die sowohl Glycerin und Puffer.
- Vortex die gestörte virale Muster wieder, und laden Sie es auf die Glycerin-Gradienten.
- Centrifuge des Gradienten für 3,75 Stunden bei 217.000 xg, 4 ° C in einem Beckman MLS-50 Ausschwingrotor Rotor.
- Nach der Zentrifugation abgeschlossen ist, manuell zu sammeln 250 ul Aliquots des Gradienten ab dem oberen Ende der Röhre. Keep Fraktionen auf Eis oder bei 4 ° C.
Teil 3: Analyse der Glycerol Gradientenfraktionen durch SDS-Polyacrylamidgelelektrophorese
- Nehmen Sie 20 ul von jeder Fraktion in ein frisches Röhrchen.
- Add 5 ul 5x SDS-PAGE-Probenpuffer.
- Hitze bis 95 ° C für 5 Minuten.
- Spin down die Proben kurz und laden Sie sie auf ein 10% Polyacrylamidgel und umfassen Molekulargewichtsmarker in einer der Vertiefungen.
- Führen Sie die Proben auf dem Gel, bis der Bromphenolblau loading Farbstoff erreicht in der Nähe der Unterseite des Gels.
- Stain das Gel mit Coomassie-Blau.
Teil 4: Die Konzentration der vRNP Fraktionen
- Wählen Sie die Glycerin-haltigen Fraktionen hauptsächlich NP, kombinieren Sie sie und verteilen sie in zwei Beckman Polycarbonat-Zentrifugenröhrchen (11 mm x 34 mm).
- Füllen Sie jedes Röhrchen mit Diethylether (DEPC)-Behandlung Reinstwasser und Pipette nach oben und unten mehrmals zu mischen.
- Zentrifuge für 4,5 Stunden 157.000 xg, 4 ° C in einem Beckman TLA-120,2 Rotor.
- Entfernen Sie den Überstand und das Pellet in 50 ul DEPC-behandeltem Wasser.
- Falls gewünscht, können die A 280 der konzentrierten vRNPs gemessen werden. Als NP ist der Hauptbestandteil in der gepoolten Fraktionen, kann eine Schätzung der Molarität der NP anhand ihrer Extinktionskoeffizienten von 55.350 M -1 cm -1 (als via ProtParam 3 ermittelt) werden.
- Aliquot des gereinigten vRNPs, und speichern Sie sie bei -80 ° C für eine spätere Verwendung.
Teil 5: Negative Färbung von vRNPs
- Verdünnen Sie 1 ul des gereinigten vRNP in 9 ul frisch gemacht und gefiltert, zweimal MNT-Puffer (20 mM MES, 150 mM NaCl, 30 mM Tris, pH 7,5). Andere Puffer sind auch OK, um die vRNPs verdünnen und auf die Probe Raster (Schritt 5,5) waschen, aber nicht verwenden, wenn PBS Uranylacetat für negative Färbung verwendet wird.
- Glimmentladung eine Probe (Kupfer TEM) Gitter zuvor beschichtet mit einer parlodion und Kohlenstoff-Film für 30 Sekunden.
- Halten Sie den frisch glow-entladen Probe Raster mit einer Pinzette, und wenden Sie einen 5-ul Tropfen verdünnt vRNP auf die Probe Netz.
- Lassen Sie die Tropfen der Probe in der Startaufstellung für 8 Minuten.
- Während der Wartezeit, platzieren Sie einen kleinen Streifen Parafilm auf der Bank, und verzichten 2 Tropfen mit 10 ul jeder MNT-Puffer (mit dem Netz zu waschen), und ein 80-ul Tropfen des frisch zubereiteten Färbelösung (1% Ammoniummolybdat oder 1 % Uranylacetat) auf die Parafilm.
- Nach der 8-Minuten Adsorption, Docht off Teil der Probelösung aus der Probe Gitter mit einem Stück Filterpapier (Schnitt in Dreiecke). Lassen Sie sich nicht die Probe Gitter trocknen lassen.
- Wash Probe Gitter in 2 Tropfen mit 10 ul jeder MNT-Puffer für eine Gesamtzeit von 1 Minute. Dies geschieht durch vorsichtiges Absenken der Probe Raster auf dem Tropfen, und dann wicking off Teil der Probelösung aus der Probe Gitter mit einem Stück Filterpapier (ohne dass die Probe Raster trocken) getan.
- Unmittelbar nach dem Feuchtigkeitstransport aus den letzten Tropfen Puffer von Probe Raster, komplettly tauchen die Probe Raster innerhalb der Fleck Tropfen und 1 Minute warten.
- Völlig Docht Sie den Fleck Lösung von der Probe Gitter mit einem Stück Filterpapier.
- Lassen Sie die Probe Gitter an der Luft trocknen für mehrere Minuten vor der Beobachtung unter dem Transmissions-Elektronenmikroskop.
Teil 6: Repräsentative Ergebnisse:

Abbildung 1
Wie Influenza-A-NP (~ 56 kDa) ist das wichtigste Protein in Viren Ribonukleoproteinkomplexe gefunden, die NP-Band in der Regel ist das stärkste Band in den Fraktionen mit den vRNPs (Abbildung 1). Zusätzlich zu NP, jeder Influenza vRNP enthält auch eine Kopie eines trimeren RNA-Polymerase (Molekulargewichte von 82, 86 und 86,5 kDa)-Komplex. Diese kann, muss aber nicht sichtbar sein, durch Coomassie Blau-Färbung, weil ihr Reichtum ist gering, dass der NP verglichen. Die Polymerasen können jedoch erkannt werden, wenn statt eines Commassie Bläue, ein silberner des Gels erfolgt gefärbt werden. Die Influenza-Matrixprotein M1 (~ 28 kDa) sollten minimal in den Fraktionen, wo der NP-Protein-Peak-Fraktionen vorhanden sind.
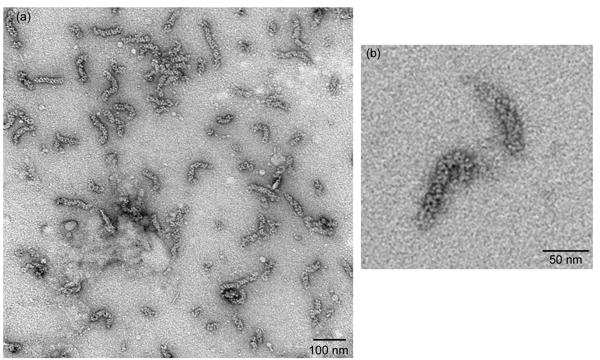
Abbildung 2
Negativ-gefärbten vRNPs als visualisierte unter einem Transmissions-Elektronenmikroskopie sollte Ausbeute vRNPs, dass stabförmige Teilchen mit variabler Länge, die etwa 30 nm bis 120 nm in der Länge (Abbildung 2a) sind ähnlich. Die oligomeren NP als eine Kette von NP-Moleküle, die zu einem Doppel-Helix-Struktur wiederholen wird gefaltet organisiert ist, so Schlaufen an beiden Enden dieser stäbchenförmige Partikel können manchmal zu sehen (Abb. 2b).
Diskussion
Die Reinigung von vRNPs wird über das Verfahren durch Kemler et al. (1994) beschrieben wird. 1 Wir und andere haben auch dieses Protokoll verwendet, um vRNPs zu isolieren, um ihre nuklearen Import-Studie. 2,4,5
Wir empfehlen die Verwendung von RNase-freien Spitzen und Rohre beim Manipulieren vRNPs weil das virale Genom von RNA besteht, und damit verschlechtert sich leicht in die Gegenwart von RNA. Darüber hinaus sollten alle Puffer in Wasser, RNase frei gemac...
Danksagungen
Diese Arbeit wurde durch Zuschüsse der Canada Foundation for Innovation (CFI), die Canadian Institute of Health Research (CIHR) und dem Natural Sciences and Engineering Research Council of Canada (NSERC) unterstützt.
Materialien
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| H3N2 X-31 A/AICHI/68 influenza A | Charles River Laboratories | 490715 | ||
| Tris | Sigma | T1503 | ||
| MES | Sigma | M-8250 | ||
| Glycerol | Fisher | G33-1 | ||
| Octylglucoside | Sigma | O-8001 | ||
| Lysolecithin | Sigma | L-4129 | ||
| Dithiothreitol | Sigma | D-9779 | ||
| Coomassie brilliant blue G-250 | Kodak | 1367796 | ||
| diethyl pyrocarbonate (DEPC)-treated water | Invitrogen | 750024 | ||
| Uranyl Acetate | Ted Pella | 19481 | ||
| Ammonium Molybdate | Fisher | A-674 | ||
| Optima MAX-E Ultracentrifuge | Beckman Coulter | 434491 | ||
| MLS-50 Rotor Package, Swinging Bucket | Beckman Coulter | 367280 | ||
| TLA-120.2 Rotor Assembly, Fixed-Angle, Titanium | Beckman Coulter | 362046 | ||
| Eppendorf Thermomixer | Brinkman | 022670000 |
Referenzen
- Kemler, I., Whittaker, G., Helenius, A. Nuclear import of microinjected influenza virus ribonucleoproteins. Virology. 202, 1028-1033 (1994).
- Wu, W. W. H., Weaver, L. L., Panté, N. Ultrastructural analysis of the nuclear localization sequences on influenza a ribonucleoprotein complexes. J Mol Biol. 374, 910-916 (2007).
- Gasteiger, E., Walker, J. M. . The Proteomics Protocols Handbook. , 571-607 (2005).
- Wu, W. W. H., Sun, Y. H. B., Panté, . Nuclear import of influenza A viral ribonucleoprotein complexes is mediated by two nuclear localization sequences on viral nucleoprotein. Virol J. 4, 49-49 (2007).
- Babcock, H. P., Chen, C., Zhuang, X. Using single-particle tracking to study nuclear trafficking of viral genes. Biophys J. 87, 2749-2758 (2004).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten