Method Article
Visualizing Einzel-Molekül-DNA-Replikation mit Fluoreszenz-Mikroskopie
In diesem Artikel
Zusammenfassung
Dieses Protokoll zeigt eine einfache Einzelmolekül-Fluoreszenz-Mikroskopie-Technik zur Visualisierung der DNA-Replikation von einzelnen replisomes in Echtzeit.
Zusammenfassung
Wir beschreiben eine einfache Fluoreszenzmikroskopie-basierte Echtzeit-Methode zur Beobachtung der DNA-Replikation in der Einzel-Molekül-Ebene. Eine kreisförmige, Gabel DNA Vorlage wird auf einer funktionalisierten Deckglas befestigt und umfassend nach Einführung der Replikation Proteinen und Nukleotiden (Abbildung 1) repliziert. Die wachsende Produkt doppelsträngige DNA (dsDNA) ist mit Laminar-Flow erweitert und visualisiert mit Hilfe eines interkalierende Farbstoff. Die Messung der Position des wachsenden DNA-Ende in Echtzeit ermöglicht eine präzise Bestimmung der Replikation Rate (Abbildung 2). Darüber hinaus gibt die Länge der abgeschlossenen DNA-Produkte auf dem Prozessivität der Replikation. Dieses Experiment kann sehr einfach und schnell durchgeführt werden und erfordert nur ein Fluoreszenzmikroskop mit einer relativ empfindlichen Kamera.
Protokoll
Vor der Durchführung einer Einzel-Molekül-Replikation Experiment, müssen ein paar Materialien im Voraus vorbereitet werden.
1. DNA Replication Vorlage
Das Substrat für die Replikation Reaktion ist ein biotinylierter, tailed M13 Rolling-Circle unter Verwendung von Standardverfahren molekularbiologischen Techniken 1,2.
Werkstoffe: M13mp18 Einzelstrang-DNA, Biotinylated Schwanz Oligonukleotid-Primer (5'-Biotin-T 36 AATTCGTAAT CATGGTCATAGCTGTTTCCT-3 '), T7-DNA-Polymerase, Heat Block, Phenol / Isoamylalkohol / Chloroform
- Glühen des biotinylierten Oligo Schwanz der M13 durch Zugabe einer 10-fachen Überschuss von Oligo in TBS-Puffer (330 nM Schwanz Oligo, 33 nM M13).
- Erhitzen Sie die Mischung auf 65 ° C in der Hitze zu blockieren. Einmal erhitzt, schalten Sie die Hitze blockieren und ermöglichen ein langsames Abkühlen auf richtig glühen die Grundierung.
- Fügen Sie die grundierte M13 zu einer Mischung von 64 nM T7 DNA-Polymerase und T7-Replikation Puffer mit dNTPs und MgCl 2.
- Inkubieren Sie die Reaktion bei 37 ° C für 12 Minuten und löschen mit 100 mM EDTA.
- Purify das ausgefüllte Produkt DNA mit Phenol / Chloroform / Isoamylalkohol-Extraktion und Dialyse in TE-Puffer oder einem anderen geeigneten Pufferspeicher und bestimmen DNA-Konzentration (Back-Extrakt jedes organische Phase, um restliche grundiert M13 wiederherzustellen).
- Eine Iteration der Vorlage Vorbereitung kann leicht mehrere Milliliter nanomolaren Konzentrationsbereich Vorlage, genug für Hunderte bis Tausende von Einzel-Molekül-Experimente.
2. Funktionalisierte Deckgläser
Zur Befestigung der DNA, die Deckglas wird das Glas zunächst mit einem Aminosilan, die dann an biotinylierte PEG-Moleküle gekoppelt ist funktionalisiert. Diese Beschichtung hilft, die unspezifische Wechselwirkungen der DNA-Replikation und Proteine mit der Oberfläche 1,3 zu reduzieren.
Materialien: Glas-Deckgläschen, Küvetten, 3-Aminopropyltriethoxysilan, Methoxy-PEG5k-NHS-Ester, Biotin-PEG5k-NHSester, Aceton, 1M KOH, Ethanol, Backofen, Ultraschallbad
- Reinigen Sie die Deckgläschen, indem man sie in Küvetten und Befüllen der Gläser mit Ethanol. Ultraschallbad für 30 Minuten und spülen Sie die Gläser füllen und mit 1 M KOH. Ultraschallbad für 30 Minuten sowohl wäscht wiederholen.
- Zum ersten Funktionalisierung Reaktion, müssen alle Spuren von Wasser entfernt werden. Füllt die Krüge mit Aceton und beschallen für 10 min. Spülen Sie die Gläser nochmals mit Aceton.
- Bereiten Sie eine 2% v / v Lösung des Silans Reagenz in Aceton. Hinzufügen, um die Küvetten und agitieren für 2 Minuten. Diese Reaktion koppelt die Alkoxygruppe des Aminosilan auf das Glas, so dass eine reaktive Amin für die nächsten Kupplungsschritt. Die Reaktion wird mit einem großen Überschuss an Wasser.
- Trocknen Sie die Deckgläschen durch Backen sie bei 110 ° C in den Ofen für 30 Minuten.
- Bereiten Sie eine 50:1 methoxy: Biotin PEG-Gemisch in 100 mM NaHCO 3 pH 8,2. Ziel für rund 0,2% w / v biotinylierten PEG.
- Je 100 ul der PEG-Gemisch auf ein trockenes silanisiert Deckglas und legen Sie eine weitere Deckglas an der Spitze. Inklusive einem Deckglas spacer erlauben Trennung der Deckgläser.
- Inkubieren Sie die Deckgläser in der PEG-Lösung für 3 Stunden, dann trennen Sie jedes Paar von Deckgläsern und waschen gründlich mit Wasser. Seien Sie vorsichtig, damit die Deckgläser funktionalisierten Seitenketten als nur einmal an Seite mit PEG beschichtet werden.
- Trocknen Sie die Deckgläser und speichern unter Vakuum. Die Oberflächen bleiben für mindestens einen Monat stabil, so Dutzende von Deckgläsern können in einer Batch gemacht werden und nach Bedarf verwendet.
Diese Materialien sollten vor der Zeit gemacht werden, und dann für jedes Experiment verwendet. Zu Beginn jedes Einzel-Molekül-Experiment, von der Montage der Flusskammer beginnen.
3. Flusskammer Vorbereitung
Das Experiment wird mit Hilfe einer einfachen Flusskammer mit einem funktionalisierten Deckglas, doppelseitiges Klebeband, ein Quarz-Objektträger und Schläuche hergestellt. Eine Flusskammer wird für jede Einzel-Molekül-Experiment 1,4 zubereitet.
Material: Doppelseitiges Klebeband, Rasierklinge, Quartz slide mit Bohrungen für Schläuche, Quick-dry Epoxy, funktionalisierten Deckglas (siehe oben), Streptavidin-Lösung (25 ul 1 mg / mL in PBS), Tubing, Blocking-Puffer (20 mM Tris pH 7,5, 2 mM EDTA, 50 mM NaCl, 0,2 mg / ml BSA, 0,005% Tween-20)
- Beginnen Sie, indem 20-25 ml Blocking-Puffer in einem Exsikkator, um mögliche Luftblasen für spätere Schritte zu entfernen.
- Werfen Sie einen PEG-funktionalisierten Deckglas und breitete 125 von PBS-verdünnten Streptavidin-Lösung über die Oberfläche. Lassen Sie diese für 20 Minuten inkubieren während der Vorbereitung in anderen Teilen der Kammer, so dass Streptavidin an die Oberfläche Biotine binden.
- Schneiden Sie ein Stück zweiseitigd-Band zu passen die Größe des Quarz-Objektträger. Markieren Sie die Position des Schlauches Löcher auf dem Band mit einem Bleistift, so dass ein Überblick über die Flusskammer auf dem Band (1 mg / mL) gezogen werden können.
- Machen Sie einen 2 mm breiten Rechteck um die Löcher. Dies wird als der Strömungskanal dienen, so achten Sie darauf, geraden Seiten zu machen und lassen Raum um das Rohr Einlass und Auslass. Falls gewünscht, können mehrere Kanäle oder verschiedene Größen verwendet werden.
- Schneiden Sie entlang der gezeichneten Kontur, so dass gerade, saubere Schnitte zu machen, dass kein Klebstoff ragt in den Kanal.
- Reinigen Sie den Quarz gründlich mit Aceton oder Ethanol, um Klebstoff von der vorherigen Durchflusszelle Bau zu entfernen.
- Finden Sie die beste Ausrichtung des Kanals skizzieren, entfernen Sie die eine Seite der Rückseite und setzt vorsichtig das Band auf den Quarz-Objektträger. Seien Sie vorsichtig, um das Band richtig auszurichten, da die Ein-und Auslass Löcher zu bleiben entsperrt brauchen.
- Cut Rohrlängen für die Einlass-und Auslass der Durchflusszelle. Es hilft, die Ende der Röhre bei etwa einem 30 ° Winkel geschnitten, so dass das Rohr nicht drücken wird gegen die Kammer unten flach. Legen Sie die Rohre auf irgendeine Art von Unterstützung sie für die einfache Befestigung an der Messzelle in die nächsten Schritte zu suspendieren.
- Spülen Sie die Streptavidin-beschichteten Deckglas gründlich mit Wasser ab und trocknen mit Druckluft. Denken Sie daran, dass nur eine Seite funktionalisiert wird, darauf achten, nicht das Deckglas umdrehen. Entfernen Sie die andere Seite der Band sichern und Ort der Quarz-Objektträger auf das Deckglas.
- Leicht auf dem Deckglas drücken, um Push-out keine Luft in den Klebstoff der Falle. So vermeiden Sie Luftblasen immer in den Strömungskanal.
- Verschließen Sie die Seiten der Kammer mit Epoxy. Legen Sie das ausgeschnittene Schlauch in die Löcher der Quarz und Dichtung in Stelle mit Epoxidharz. Dies muss für ein paar Minuten trocknen lassen.
- Nach dem Trocknen beginnen Blockierung der Oberfläche, indem einige der entgasten Blockierungspuffer durch eine der Röhren. Ein 21-Gauge-Nadel passt perfekt in die 0,76 mm Schlauch. Flush ein paar Mal, um Luftblasen zu entfernen und damit die Kammer für mindestens eine halbe Stunde zu inkubieren.
4. Single-Molecule Replication Experiment
Werkstoffe: Vorbereitet Flusskammer, SYTOX Orange, TIRF-Mikroskop, 532 nm Laser, CCD-Kamera, Computer mit der Bildaufnahme-Software, Rolling-circle DNA-Substrat, Replication Proteine
- Nach der Durchflusszelle blockiert wurde, ist alles bereit für die Einzel-Molekül-Experiment zu beginnen.
- Nehmen Sie die Durchflusszelle und legen Sie es auf dem Mikroskoptisch. Halten Sie die Kammer in Platz mit Bühne Clips und sicher sein, dass der Strömungskanal geradeaus entlang der y-Achse positioniert ist.
- Schließen Sie die Durchflusszelle Auslaufrohr der Spritzenpumpe mit einem größeren Durchmesser Anschluss Schlauch oder einer Nadel. Setzen Sie den Einlass in Blocking-Puffer und ziehen wieder auf die Spritze, um die Luft in den Schlauch zu entfernen. Eine sanfte Bewegung aus dem Auslaufrohr wird klar, keine Luftblasen in der Messzelle gefangen.
- Von der Stammlösung DNA-Template zu 25 pM in 1 ml Blocking-Puffer. Durchfluss in die Kammer zu moderaten Durchfluss gute Flächendeckung von DNA ermöglichen. 0,025 ml / min für 30 Minuten gut funktioniert. Dies kann je nachdem, wie viele DNA-Moleküle auf der Oberfläche für jede Charge von Deckgläsern oder wie viele sind für das Experiment gewünscht variiert werden.
- Sobald DNA ist in ausspülen, überflüssige DNA mit Blockierungspuffer. Wash für mindestens 200 Flow-Zelle Bände loswerden alle freien DNA.
- Begin Entgasung der Replikation Puffer. Für die E. coli-Replikation Versuche, dies zu tun bei 37 ° C, um das Risiko von Blasen in der beheizten Flusskammer zu reduzieren.
- Schalten Sie den Laser und Kamera. Die gekühlte CCD muss Temperatur zu erreichen, bevor es verwendet werden kann.
- Wenn der Durchführung einer 37 ° C Experiment, schalten die Heizung auf. Seien Sie sicher, dass das Ziel ist in Kontakt mit der Durchflusszelle oder es wird ein Kühlkörper später.
- Nach dem Waschen und der Entgasung kann die Reaktion hergestellt werden. Mix Nukleotide, DTT und SYTOX in die Replikation Puffer. Dann fügen Sie Proteine und vorsichtig mischen.
- Durchfluss des Reaktionsgemisches in die Messzelle. Diese Strömungsgeschwindigkeit muss ausreichend sein, um dsDNA strecken. Bei einem 2-mm breit, 100-um hohe Strömungskanal 0,04 ml / min gut funktioniert.
- Nehmen Sie sich Zeit für das Gemisch in die Kammer eintreten und beginnen Bildgebung. Ein guter Tipp ist es, das Blickfeld auf die Seite des Kanals zu bewegen und sich auf den Klebstoff. Dadurch wird sichergestellt, Sie sind in der Nähe der Oberfläche zu fokussieren.
- Passen Laserleistung, Mikroskop zu konzentrieren, und TIRF Winkel. Halten Sie die Leistung gering, um irgendeine Photospaltung der gefärbten DNA zu vermeiden. Erwerben Sie bei 1-5 Frames / Sekunde für einige Minuten in Abhängigkeit von der Rate der DNA-Replikation. Wenn gewünscht, wiederholen, um mehr Replikation Bahnen zu bekommen oder zu verschieben, um ein neues Feld mehr Moleküle sehen.
- Nachdem das Reaktionsgemisch fast leer ist, ist es eine gute Idee, in mehr Puffer mit S flowYTOX um zu sehen, anderen Bereichen der Blick. Unter mehreren Bildern verschiedener repliziert Moleküle bietet Statistiken für Prozessivität Entschlossenheit.
5. Repräsentative Ergebnisse
Aktiv replizierenden Moleküle werden leicht als wachsenden Linien der gefärbten DNA gesehen. In unseren Experimenten, fließende 25 pM der M13 Substrat gibt 100-1000 Moleküle in einem 60x, 125 um x 125 um Sichtfeld (Abbildung 1). Performing Replikation Experimente mit dem E. coli replisome Proteine bei 37 ° C (siehe Materialien für weitere Details) gibt 5-50 replizierenden Molekülen pro Gesichtsfeld. Bei vergleichbaren Konzentrationen der T7 replisome, die auf dem Substrat initiiert wesentlich effizienter, beobachten wir> 70% der Moleküle in einem Feld zu replizieren. Diese Bedingungen ergeben ein Produkt Dichte so hoch, dass einzelne Moleküle schwer zu lösen und zu analysieren, so dass die T7 Experimente sind bei niedrigeren Proteinkonzentrationen durchgeführt.
Datenanalyse: Um Tarifdaten, einfach Grundstück der Endpunkt der DNA gegen die Zeit und berechnen Sie die Steigung (Abbildung 2). Für Prozessivität, bestimmen die Gesamtlänge der DNA. Beide Zahlen werden sich an Basenpaare umgewandelt werden. Vor der Durchführung des Experiments, sollten Sie wissen, die Pixelgröße der Kamera an der richtigen Vergrößerung. Für Basenpaar Umwandlung ist eine gute Schätzung einfach konvertieren basierend auf den kristallographischen Länge der DNA, 2,9 bp / nm. Allerdings Laminar Flow wird nicht vollständig strecken die DNA, so dass ein DNA Längenkalibrierung, indem eine bekannte Länge DNA, zB 48.502 bp λ DNA, Befestigung an der Durchflusszelle mit einem biotinylierten Oligo und die Messung ihrer SYTOX gebeizt Länge an durchgeführt werden soll der Durchfluss für die Replikation Experiment verwendet. Ermittlung der Anzahl der Basenpaare / Pixel ermöglicht eine genaue Berechnung der Replikation Preise und processivities. Unter zahlreichen Bildern und Filmen liefert eine große Anzahl von Produkt-Molekül, so dass das Plotten von Geschwindigkeit und Prozessivität Distributionen (Abbildung 2) 1.
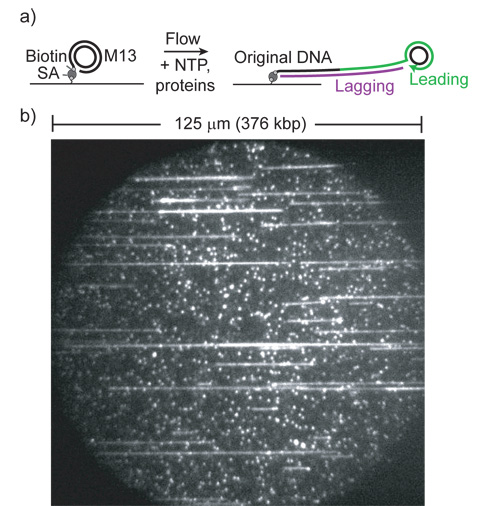
Abbildung 1: (aus Lit. 1). A) Cartoon des Rolling-Circle-Replikation Assay. (SA, Streptavidin). Leading-Strang-Synthese erweitert den Schwanz Verknüpfung des Kreises und der Oberfläche. Der Schwanz ist an dsDNA durch die nacheilenden Strang Polymerase umgewandelt und streckte von Laminar-Flow.
b) Beispiel-Sichtfeld mit SYTOX Orange und 532 nm Anregung. Kleine fluoreszierende Flecken angebunden werden, nicht repliziert Substraten. Beachten Sie die extreme Länge der Produkte und die große Anzahl der Produkte pro Feld (jeder Messzelle enthält> 5000 solcher Felder).

Abbildung 2: (nach Lit. 1). A, b) Kymographs typischer replizierenden Molekülen aus T7 (a) und E. coli (b) Experimente. c) Endpoint Flugbahnen von Molekülen aus a) und b) aufgetragen gegen die Zeit. Trajektorien sind mit lineare Regression anzupassen, um die Replikation Preise zu erhalten. Gezeigten Beispiele sind 99,4 bp s -1 und 467,1 bp s -1. d) Länge Verteilungen der Replikation Produkte, mit Einzel-exponentiellen Anpassung an Prozessivität erhalten: 25,3 ± 1,7 kbp (T7), 85,3 ± 6,1 kbp (E. coli). e) Rate Verteilungen der einzelnen Moleküls Bahnen, mit Einzel-Gauß-Fit. Mittel: 75,9 ± 4,8 bp s -1 (T7), 535,5 ± 39 bp s -1 (E. coli).
Diskussion
Eine kritische Kontrollpunkte prüft die Wirkung der Beize, SYTOX Orange, auf die Replikation Proteine von Interesse. Ein einfacher Weg, dies zu tun ist, um die Replikation Experiment in der Messzelle, wie beschrieben, aber mit dem Wegfall der SYTOX durchzuführen. Nachdem die Reaktionsmischung durch die Kammer geflossen ist, fügen Puffer mit SYTOX auf DNA-Färbung und untersuchen die Länge Verteilung der replizierten Moleküle. Alternativ können Standard-Bulk-Reaktionen verwendet werden, um eine Wirkung von SYTOX auf die Replikation Geschwindigkeit und Effizienz zu überprüfen.
Das hier beschriebene Experiment verwendet nur einen einzigen Strömungskanal. Dies kann leicht durch die Schaffung von Multi-Channel-Flow Kammern oder mit PDMS oder ähnliche mikrofluidischen Systemen geändert werden. Eine Erhöhung der Anzahl der Kanäle erleichtert Screening von Protein-Konzentrationen, Mutanten oder Inhibitor-Moleküle und erhöht die Geschwindigkeit der Replikation der Datenerhebung.
Wie bereits erwähnt, führen wir die E. coli-Replikation Experimente bei 37 ° C mit Hilfe eines selbst gebauten Aluminium-flow-Heizgerät, ein Widerstandsheizelement (Heizpatrone) und variable Spannungsversorgung. Das gibt gute Temperaturstabilität und vermeidet den Kauf eines objektiven Heizung. Zur Kalibrierung der Heizung haben wir einfach ein Loch in der Mitte eines Quarz-Durchflusszelle oben gebohrt, eingefügt ein Thermoelement in den Strömungskanal und floss Puffer als normal. Die Messung der Durchflusszelle Puffer Temperatur bei zunehmender Spannungen ermöglicht eine präzise Heizung.
Danksagungen
Samir Hamdan unterstützt bei der Entwicklung dieser Technik. E. coli-Proteine sind aus dem Labor von Prof. Nick Dixon, sind University of Wollongong und T7 Proteine aus Prof. Charles Richardson, Harvard Medical School. Die Arbeit wird durch die National Institutes of Health (GM077248 zu AMvO) und die Jane Coffin Childs Foundation (JJL) unterstützt.
Materialien
T7-Replikation: 40 mM Tris pH 7,5, 50 mM Kalium-Glutamat, 10 mM Magnesiumchlorid, 100 ug / mL BSA, mit 5 mM Dithiothreitol, 600 uM dNTPs, 300 nM ATP, 300 uM CTP und 15 nM SYTOX orange aufgenommen unmittelbar vor zu verwenden. Proteine hinzugefügt: 5 nM gp4 (Hexamer), 40 nM-Polymerase (1:1 gp5: Thioredoxin), 360 nm gp2.5 1,5.
E. coli-Replikation: 50 mM HEPES pH 7,9, 12 mM Magnesium-Acetat, 80 mM Kaliumchlorid, 100 ug / mL BSA mit 10 mM Dithiothreitol, 40 uM dNTPs, 200 nM rNTPs und 15 nM SYTOX orange aufgenommen unmittelbar vor dem Gebrauch. Proteine hinzugefügt: 30 nM DnaB (Hexamer), 180 nM DNAC (Monomer), 30 nM αεθ, 15 nM τ 2 γ 1 δδ'χψ oder τ 3 δδ'χψ, 30 nM β (Dimer), 300 nM DNAG, 250 nM SSB (Tetramer), 20 nM Pria, 40 nM Prib, 320 nM Pric, 480 nM dnaT 1,6
Referenzen
- Tanner, N. A., et al. Real-time single-molecule observation of rolling-circle DNA replication. Nucleic Acids Res. 37, e27(2009).
- Tabor, S., Huber, H. E., Richardson, C. C. Escherichia coli thioredoxin confers processivity on the DNA polymerase activity of the gene 5 protein of bacteriophage t7. J Biol Chem. 262, 16212-16223 (1987).
- Sofia, S. J., Premnath, V. V., Merrill, E. W. Poly(ethylene oxide) grafted to silicon surfaces: Grafting density and protein adsorption. Macromolecules. 31, 5059-5070 (1998).
- Tanner, N. A., van Oijen, A. M. Single-molecule observation of prokaryotic DNA replication. DNA replication: Methods and protocols. Vengrova, S., Dalgaard, J. Z. 521, Humana Press. New York. 397-410 (2009).
- Hamdan, S. M., Loparo, J. J., Takahashi, M., Richardson, C. C., van Oijen, A. M. Dynamics of DNA replication loops reveal temporal control of lagging-strand synthesis. Nature. 457, 336-339 (2009).
- Heller, R. C., Marians, K. J. Replication fork reactivation downstream of a blocked nascent leading strand. Nature. 439, 557-562 (2006).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten