Method Article
Visualización de una sola molécula de ADN de replicación con microscopía de fluorescencia
En este artículo
Resumen
Este protocolo demuestra un sencillo de una sola molécula de la técnica de microscopía de fluorescencia para la visualización de la replicación del ADN por replisomes individuales en tiempo real.
Resumen
Se describe un simple microscopía de fluorescencia basada en tiempo real el método para la observación de la replicación del ADN en el ámbito de una sola molécula. Una plantilla circular, ADN horquilla se une a un cubreobjetos funcionalizados y reproducido ampliamente después de la introducción de las proteínas de replicación y los nucleótidos (Figura 1). Los productos de mayor crecimiento de doble cadena de ADN (dsDNA) se amplía con flujo laminar y se visualizan mediante el uso de un colorante intercalante. La medición de la posición del extremo del ADN que crecen en tiempo real permite la determinación precisa de la tasa de replicación (Figura 2). Además, la duración de los productos de ADN completó los informes sobre la procesividad de la replicación. Este experimento se puede realizar con mucha facilidad y rapidez, y sólo requiere un microscopio de fluorescencia con una cámara bastante sensible.
Protocolo
Antes de realizar un experimento de replicación de una sola molécula, unos pocos tienen que estar preparados de antemano.
1. Patrón de ADN de replicación
El sustrato para la reacción de replicación es un biotina, cola M13 círculo rodante prepararse con técnicas de biología molecular 1,2.
Materiales: M13mp18 ADN de cadena sencilla, primer oligonucleótido biotinilado cola (5'-biotina-T 36 AATTCGTAAT CATGGTCATAGCTGTTTCCT-3 '), ADN polimerasa T7, bloque de calor, el alcohol fenol / isoamílico / cloroformo
- Recocer la cola oligo biotinilado a la M13 por la adición de un exceso de 10 veces de oligo en tampón TBS (330 nM cola oligo, 33 nM M13).
- Calentar la mezcla a 65 ° C en el bloque de calor. Una vez caliente, a su vez el bloque de calor y deje un enfriamiento lento para templar correctamente la cartilla.
- Agregue el M13 preparado para una mezcla de 64 nm de la polimerasa T7 ADN y la replicación T7 tampón que contiene dNTPs y MgCl 2.
- Incubar la reacción a 37 ° C durante 12 minutos y apagar con 100 mM EDTA.
- Purificar el lleno-en el ADN de productos con la extracción de alcohol de fenol / cloroformo / isoamílico y diálisis en buffer TE o tampón otro dispositivo de almacenamiento adecuado y determinar la concentración de ADN (Back-extracto de cada fase orgánica para recuperar los residuos preparados M13).
- Una repetición de la preparación de la plantilla puede hacer fácilmente varios mililitros de la plantilla de concentración nanomolar, suficiente para cientos de miles de experimentos de una sola molécula.
2. Cubreobjetos funcionalizados
Para unir el ADN a la cubreobjetos de vidrio, el vidrio es el primer funcionalizados con un aminosilano, que se acopla a las moléculas de PEG con biotina. Esta capa ayuda a reducir las interacciones no específicas de ADN y las proteínas de replicación con la superficie de 1,3.
Materiales: cubreobjetos de vidrio, cubetas, 3-aminopropiltrietoxisilano, éster de metoxi-PEG5k-NHS, biotina-PEG5k NHSester, acetona, KOH 1M, etanol, horno, baño de ultrasonido
- Limpiar a fondo el cubreobjetos de vidrio, colocándolas en la tinción de los frascos y llenar los frascos con etanol. Sonicar durante 30 minutos y enjuague los frascos y llenar con 1 M KOH. Sonicar durante 30 minutos Repita ambos lavados.
- Para la reacción de funcionalización en primer lugar, todos los rastros de agua necesitan ser removidos. Llenad las tinajas de acetona y ultrasonidos durante 10 minutos. Enjuague los frascos una vez más con acetona.
- Preparar un 2% v / v solución del reactivo de silano en acetona. Añadir a la cubetas y agitar durante 2 minutos. Esta reacción se acopla el grupo alcoxi de la aminosilano al vidrio, dejando una amina reactiva para la próxima etapa de acoplamiento. Detener la reacción con un gran exceso de agua.
- Seque el cubreobjetos de hornearlas a 110 ° C en el horno durante 30 minutos.
- Prepare una metoxi 50:1: biotina mezcla de PEG en 100 mM NaHCO 3 pH 8,2. Trate de alrededor del 0,2% w / v de PEG con biotina.
- Pipetear 100 L de la mezcla de PEG en un portaobjetos seco silanizada y otro lugar cubreobjetos en la parte superior. Incluyendo un separador cubreobjetos permitirá la separación de las laminillas.
- Incubar los cubreobjetos en la solución de PEG durante 3 horas, luego se separan cada par de cubreobjetos y lavar intensamente con agua. Tenga cuidado de mantener los cubreobjetos funcionalizada hacia arriba, ya que sólo una vez que lado va a ser recubierto con PEG.
- Seque el cubreobjetos y guardar al vacío. Las superficies se mantienen estables durante al menos un mes, por lo que decenas de cubreobjetos se puede hacer en un lote y se utiliza según sea necesario.
Estos materiales deben ser hechas antes de tiempo, y luego se usa para cada experimento. Para iniciar cada experimento de una sola molécula, comience el montaje de la cámara de flujo.
3. Preparación de la Cámara de flujo
El experimento se realiza con una cámara de flujo sencillo construido con un cubreobjetos funcionalizados, cinta de doble cara, una diapositiva de cuarzo y tubos. Una cámara de flujo se prepara para el 1,4 experimento cada molécula.
Materiales: cinta de doble cara, una hoja de afeitar, diapositivas de cuarzo con agujeros para la tubería, de secado rápido epoxi, cubreobjetos funcionalizados (ver arriba), la solución de estreptavidina (25 l de 1 mg / ml en PBS), tubos, tampón de bloqueo (20 mM Tris pH 7,5, 2 mM EDTA, 50 mM NaCl, 0,2 mg / ml de BSA, 0,005% de Tween-20)
- Comience colocando 20-25 ml de tampón de bloqueo en un desecador para eliminar cualquier burbuja de aire para los pasos posteriores.
- Tome un cubreobjetos PEG-funcionalizados y la propagación de la solución de estreptavidina 125 PBS diluido a lo largo de la superficie. Dejar esta tarea a incubar durante 20 minutos mientras se preparan otras partes de la cámara, permitiendo estreptavidina para unirse Biotins superficie.
- Corte un pedazo de doble carad cinta que coincida con el tamaño de la diapositiva de cuarzo. Marque la posición de los orificios de la tubería en la cinta con un lápiz de modo que un esquema de la cámara de flujo se pueden extraer de la cinta (1 mg / ml).
- Hacer una de 2 mm de ancho rectángulo alrededor de los agujeros. Esto servirá como canal de flujo, así que asegúrese de que los lados rectos y dejar espacio alrededor de la entrada y la salida de la tubería. Si lo desea, los canales múltiples o diferentes tamaños se pueden utilizar.
- Corte a lo largo de la línea trazada, por lo que los cortes rectos y limpio para asegurarse de que no sobresale adhesivo en el canal.
- Limpiar el cuarzo a fondo con acetona o etanol para quitar el adhesivo de la construcción de flujo de la celda anterior.
- Encontrar la mejor alineación de las líneas de canal, eliminar una parte de la superficie adhesiva y colocar con cuidado la cinta en el portaobjetos de cuarzo. Tenga cuidado de alinear correctamente la cinta, ya que los orificios de entrada y salida deben permanecer bloqueados.
- Cortar trozos de tubos para la entrada y salida de la celda de flujo. Que ayuda a cortar el extremo del tubo de aproximadamente un ángulo de 30 ° para que el tubo no se prensa plana contra el fondo de la cámara. Coloque los tubos en algún tipo de apoyo para suspender para la conexión fácil a la celda de flujo en los próximos pasos.
- Enjuague el portaobjetos recubiertas de estreptavidina a fondo con agua y secar con aire comprimido. Recuerde que sólo un lado es funcionalizados, tenga cuidado de no convertir el cubreobjetos encima. Retire el otro lado del soporte de la cinta y colocar la placa de cuarzo en el cubreobjetos.
- Presione suavemente el cubreobjetos para expulsar el aire atrapado en el adhesivo. Esto ayudará a evitar las burbujas de aire en el canal de flujo.
- Sello de los lados de la cámara con epoxy. Inserte la tubería cortada en los agujeros del cuarzo y el sello en su lugar con epoxy. Esto tiene que secar por unos minutos.
- Una vez seca, comenzará a bloquear la superficie tirando algunos de los buffer desgasificar el bloqueo a través de uno de los tubos. Una aguja de calibre 21 encaja perfectamente dentro de la tubería de 0,76 mm. Ras un par de veces para eliminar las burbujas de aire, y permitir que la cámara de incubación por lo menos durante media hora.
4. Una sola molécula de replicación Experimento
Materiales: cámara de flujo preparado, SYTOX Orange, microscopio TIRF, 532 nm láser, CCD de la cámara, ordenador con software de adquisición de imágenes, círculo rodante sustrato de ADN, las proteínas de replicación
- Después de la celda de flujo ha sido bloqueado, todo está listo para comenzar el experimento de una sola molécula.
- Tome la celda de flujo y colocarlo en la platina del microscopio. Mantenga la cámara en su lugar con clips de la etapa y estar seguro de que el canal de flujo esté recto a lo largo del eje.
- Conecte el tubo de salida de la celda de flujo de la bomba de jeringa con un tubo de diámetro de conector más grande o una aguja. Coloque la entrada en tampón de bloqueo y tire hacia atrás de la jeringa para eliminar el aire en el tubo. Un movimiento suave del tubo de salida va a ayudar a eliminar las burbujas de aire atrapadas en la celda de flujo.
- Diluir la plantilla de ADN de valores a 25 pM en 1 ml de tampón de bloqueo. Flujo en la cámara a una velocidad moderada para permitir el flujo de cobertura de la superficie buena de ADN. 0.025 ml / min durante 30 minutos funciona bien. Esto puede variar según el número de moléculas de ADN están en la superficie de cada lote de cubreobjetos o cuántos se desean para el experimento.
- Una vez que el ADN se encuentra en, lavar el exceso de ADN con el tampón de bloqueo. Lávese las manos por lo menos 200 células de flujo de los volúmenes de deshacerse de todo el ADN libre.
- Comience desgasificación del búfer de duplicación. Para el E. experimentos coli la replicación, haga lo siguiente a 37 ° C para reducir el riesgo de burbujas en la cámara de flujo caliente.
- Encienda el láser y la cámara. El CCD enfriado las necesidades para alcanzar la temperatura antes de que pueda ser utilizado.
- Si se realiza a 37 ° C experimento, a su vez la calefacción. Asegúrese de que el objetivo está en contacto con la celda de flujo o será un disipador de calor más tarde.
- Después de lavar y la desgasificación, la reacción puede ser preparado. Nucleótidos mezcla, la TDT, y SYTOX en el buffer de la replicación. A continuación, añadir proteínas y mezclar suavemente.
- El flujo de la mezcla de reacción en la celda de flujo. Esta velocidad de flujo debe ser suficiente para estirar dsDNA. Para un niño de 2 mm de ancho, de 100 m de altura canal de flujo de 0,04 ml / min funciona bien.
- Dé tiempo para que la mezcla entre en la cámara y comenzará a marcar. Un buen consejo es mover el campo de visión hacia el lado del canal y se centran en el material adhesivo. Esto asegurará que se encuentran cerca de la superficie para enfocar.
- Ajustar la potencia del láser, el enfoque microscopio, y el ángulo TIRF. Mantener el bajo consumo de energía para evitar cualquier photocleavage del ADN de colores. Adquirir en 5.1 fotogramas / segundo durante varios minutos dependiendo de la tasa de replicación del ADN. Si lo desea, repita a ser más largas trayectorias de replicación o trasladarse a un nuevo campo para ver más moléculas.
- Después de la mezcla de reacción está casi vacío, es una buena idea para el flujo de más de amortiguación con SYTOX para ver otros campos de la vista. Tomar varias imágenes de diferentes moléculas replicadas proporciona estadísticas para la determinación de procesividad.
5. Resultados representante
Moléculas de replicación activa son fácilmente visibles como líneas de crecimiento de ADN manchado. En nuestros experimentos, que fluye 25 horas del sustrato M13 ofrece 100-1000 moléculas en un 60x, 125 m x 125 m de campo de vista (Figura 1). La realización de experimentos de replicación utilizando el E. proteínas coli replisoma a 37 ° C (ver Materiales para más detalles) le da réplica moléculas 50-50 por campo de visión. En concentraciones equivalentes de la replisoma T7, que inicia en el sustrato mucho más eficiente, se observa> 70% de las moléculas en un campo de replicar. Estas condiciones producen una densidad del producto tan alto que las moléculas individuales son difíciles de resolver y analizar, de modo que los experimentos T7 se realizan en las concentraciones de proteína más bajos.
Análisis de datos: Para obtener la velocidad de datos, simplemente trazar el punto final de la ADN en función del tiempo y calcular la pendiente (Figura 2). Para procesividad, determinar la longitud total del ADN. Ambos de estos números tendrán que convertirse en pares de bases. Antes de realizar el experimento, usted debe saber el tamaño en píxeles de la cámara en un aumento adecuado. Para la conversión de pares de bases, una buena estimación es simplemente la conversión sobre la base de la longitud de cristalografía de ADN, 2,9 pb / nm. Sin embargo, el flujo laminar completamente no se estira el ADN, por lo que la calibración de la longitud del ADN se debe realizar mediante la adopción de un ADN de longitud conocida, por ejemplo, 48.502 pares de bases del ADN λ, adjuntando a la celda de flujo con un oligo biotinilado y medir su SYTOX manchada de longitud en el caudal utilizado para el experimento de replicación. Determinar el número de pares de bases / pixel permite un cálculo exacto de las tasas de repetición y processivities. Tomar numerosas imágenes y películas se ofrecen un gran número de moléculas de producto, lo que permite el trazado de las distribuciones de frecuencia y procesividad (Figura 2) 1.
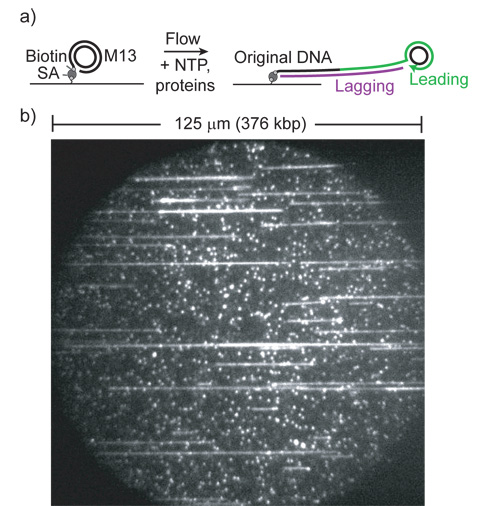
Figura 1: (De Ref 1.) Una historieta) de la prueba de la replicación de círculo rodante. (SA, estreptavidina). Principal capítulo de síntesis se extiende la cola que une el círculo y la superficie. La cola se convierte en ADN de doble cadena por la polimerasa de cadena retrasada y se extendía por el flujo laminar.
b) Ejemplo de campo de visión con SYTOX Orange y 532 nm de excitación. Pequeñas manchas fluorescentes están atados, no replicado sustratos. Tenga en cuenta la eslora máxima de los productos y el gran número de productos por campo (cada celda de flujo contiene> 5,000 campos de este tipo).

Figura 2: (adaptado de la Ref 1). A, b) Kymographs típica de las moléculas de la replicación del T7 (a) y E. coli (b) los experimentos. c) las trayectorias de extremos de las moléculas de a) y b) trazado en función del tiempo. Trayectorias están equipados con la regresión lineal para obtener las tasas de replicación. Ejemplos que se muestran son 99,4 pb s -1 y 467,1 pb s -1. d) Duración de la distribución de los productos de replicación, se ajustan a la decadencia exponencial simple para obtener procesividad: 25,3 ± 1,7 kpb (T7), 85,3 ± 6,1 kpb (E. coli). e) Tasa de las distribuciones de las trayectorias de una sola molécula, se ajustan a gaussianas sola. Medios: 75,9 ± 4,8 pb s -1 (T7), 535,5 ± 39 pb s -1 (E. coli).
Discusión
Un crítico de control es examinar el efecto de la mancha naranja, SYTOX, en la replicación de proteínas de interés. Una manera simple de hacer esto es llevar a cabo el experimento de replicación en la celda de flujo como se describe, pero con la omisión de SYTOX. Después de la mezcla de reacción ha fluido a través de la cámara, añadir tampón con SYTOX para teñir el ADN y examinar la distribución de la longitud de las moléculas de reproducirse. Por otra parte, las reacciones estándar a granel se pueden utilizar para comprobar ningún efecto sobre la tasa de SYTOX la replicación y la eficiencia.
El experimento que se describe aquí sólo utiliza un canal de flujo único. Esto se puede cambiar fácilmente mediante la creación de canales múltiples cámaras de flujo o el uso de PDMS o similares dispositivos de microfluídica. Aumentar el número de canales facilita en gran medida de detección de las concentraciones de proteínas, mutantes, o moléculas inhibidoras y aumenta la rapidez de la replicación de recolección de datos.
Como se ha mencionado, se realiza la E. experimentos coli la replicación a 37 ° C con una casa construida de aluminio calentador de flujo de células, un elemento de calefacción resistivo (calentador de cartucho) y fuente de alimentación variable. Esto le da estabilidad a la temperatura buena y evita la compra de un calentador de objetivo. Para calibrar la calefacción, simplemente un agujero en el centro de un importante flujo de celda de cuarzo, insertó un termopar en el canal de flujo y fluido de amortiguación de forma normal. La medición de la temperatura de la celda de flujo de amortiguación a tensiones cada vez permite un calentamiento preciso.
Agradecimientos
Samir Hamdan ayudó en el desarrollo de esta técnica. E. proteínas coli son del laboratorio del profesor Nick Diaz, las proteínas de la Universidad de Wollongong, y T7 son del Prof. Charles Richardson, Harvard Medical School. El trabajo es financiado por los Institutos Nacionales de Salud (GM077248 a amvo) y la Fundación Jane Coffin Niños (JJL).
Materiales
Replicación T7: 40 mM Tris pH 7,5, 50 mM de glutamato de potasio, cloruro de magnesio 10 mM, 100 mg / ml BSA, con 5 mM de ditiotreitol, 600 mM dNTPs, 300 mM ATP, 300 M CTP y 15 nM SYTOX naranja añadido inmediatamente antes de uso. Proteínas añadidas como: 5 nM gp4 (hexamer), 40 nM de la polimerasa (1:1 GP5: tiorredoxina), 360 nm gp2.5 1,5.
E. coli replicación: HEPES 50 mM pH 7,9, 12 mM de acetato de magnesio, cloruro de potasio 80 mM, 100 ug / ml de BSA con mM ditiotreitol 10, 40 mM dNTPs, 200 rNTPs M, y 15 nM SYTOX naranja añadido inmediatamente antes del uso. Proteínas añadidas como: 30 nM DnaB (hexamer), 180 nM DnaC (monómero), 30 nM αεθ, 15 nM τ γ 2 1 δδ'χψ o τ 3 δδ'χψ, 30 β nM (dímero), 300 nM dnaG, 250 nM SSB (tetrámero), 20 nM Pría, 40 nM PriB, 320 nM Pric, 480 nM DNAT 1,6
Referencias
- Tanner, N. A., et al. Real-time single-molecule observation of rolling-circle DNA replication. Nucleic Acids Res. 37, e27(2009).
- Tabor, S., Huber, H. E., Richardson, C. C. Escherichia coli thioredoxin confers processivity on the DNA polymerase activity of the gene 5 protein of bacteriophage t7. J Biol Chem. 262, 16212-16223 (1987).
- Sofia, S. J., Premnath, V. V., Merrill, E. W. Poly(ethylene oxide) grafted to silicon surfaces: Grafting density and protein adsorption. Macromolecules. 31, 5059-5070 (1998).
- Tanner, N. A., van Oijen, A. M. Single-molecule observation of prokaryotic DNA replication. DNA replication: Methods and protocols. Vengrova, S., Dalgaard, J. Z. 521, Humana Press. New York. 397-410 (2009).
- Hamdan, S. M., Loparo, J. J., Takahashi, M., Richardson, C. C., van Oijen, A. M. Dynamics of DNA replication loops reveal temporal control of lagging-strand synthesis. Nature. 457, 336-339 (2009).
- Heller, R. C., Marians, K. J. Replication fork reactivation downstream of a blocked nascent leading strand. Nature. 439, 557-562 (2006).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados