Method Article
Visualisation de la réplication d'ADN molécule unique par microscopie à fluorescence
Dans cet article
Résumé
Ce protocole illustre une simple molécule unique technique de microscopie par fluorescence pour la visualisation de la réplication de l'ADN par replisomes individuelles en temps réel.
Résumé
Nous décrivons un simple microscope à fluorescence-temps réel basé sur la méthode pour l'observation de réplication de l'ADN à la molécule seul niveau. Une circulaire, matrice d'ADN fourchue est attaché à une lamelle de verre fonctionnalisée et reproduit abondamment après l'introduction de protéines de réplication et de nucléotides (figure 1). Le produit de plus en plus l'ADN double-brin (ADNdb) est étendu avec un écoulement laminaire et visualisées en utilisant un colorant intercalant. Mesure de la position de l'extrémité d'ADN en croissance en temps réel permet la détermination précise du taux de réplication (figure 2). Par ailleurs, la longueur des produits d'ADN rempli des rapports sur la processivité de la réplication. Cette expérience peut être effectuée très facilement et rapidement et ne nécessite qu'un microscope à fluorescence avec un appareil assez sensible.
Protocole
Avant d'effectuer la réplication d'une expérience unique molécule, un peu de matériaux doivent être préparés à l'avance.
1. Modèle de réplication d'ADN
Le substrat pour la réaction de réplication est un biotinylé, queue M13 cercle roulant préparés en utilisant standards de biologie moléculaire 1,2 techniques.
Matériaux: M13mp18 ADN simple brin, biotinylé oligonucléotide amorce de queue (5'-biotine-T 36 AATTCGTAAT CATGGTCATAGCTGTTTCCT-3 '), l'ADN polymérase T7, un bloc de chaleur, de phénol / alcool isoamylique / Chloroforme
- Recuire les oligo biotinylé la queue à l'M13 en ajoutant un excès de 10 fois d'oligo dans du tampon TBS (330 nM queue oligo, 33 nM M13).
- Chauffer le mélange à 65 ° C dans le bloc thermique. Une fois chauffée, tournez le bloc de chauffage hors tension et permettre un refroidissement lent à bien recuit l'amorce.
- Ajouter la M13 amorcée à un mélange de 64 nM polymérase T7 ADN et le tampon de la réplication T7 contenant dNTPs et MgCl 2.
- Incuber la réaction à 37 ° C pendant 12 minutes et étancher avec 100 mM d'EDTA.
- Purifier l'remplie dans l'ADN des produits avec extraction phénol / chloroforme / isoamyle et dialyser dans un tampon TE ou d'autres tampons de stockage approprié et de déterminer la concentration d'ADN (Back-extraire chaque phase organique à récupérer tout résidu amorcée M13).
- Une itération de préparation modèle peut facilement faire plusieurs millilitres de modèle de concentration nanomolaire, assez pour des centaines de milliers de molécule unique d'expériences.
2. Lamelles fonctionnalisés
Pour la fixation à l'ADN de la lamelle de verre, le verre est d'abord fonctionnalisés avec un aminosilane, qui est ensuite couplé à des molécules de PEG biotinylé. Ce revêtement contribue à réduire les interactions non spécifiques de l'ADN et les protéines de réplication avec la surface 1,3.
Matériaux: lamelles de verre, des pots de coloration, 3-aminopropyltriéthoxysilane, méthoxy-PEG5k-NHS ester biotine-PEG5k-NHSester, l'acétone, 1M KOH, l'éthanol, Four Bain sonicateur,
- Nettoyez soigneusement les lamelles de verre en les plaçant dans des bocaux coloration et le remplissage des bocaux avec de l'éthanol. Soniquer pendant 30 minutes et rincer les bocaux et les remplir de 1 M KOH. Soniquer pendant 30 minutes Répéter deux lavages.
- Pour la réaction de fonctionnalisation d'abord, toute trace d'eau doivent être enlevés. Remplissez les bocaux avec de l'acétone et soniquer pendant 10 min. Rincez à nouveau les bocaux avec de l'acétone.
- Préparer un 2% v / v solution du réactif de silane dans l'acétone. Ajouter à la coloration des bocaux et agiter pendant 2 minutes. Cette réaction des couples du groupe alcoxy de l'aminosilane à la vitre, laissant une amine réactive pour la prochaine étape de couplage. Arrêter la réaction avec un large excès d'eau.
- Sécher les lamelles en les cuisant à 110 ° C dans le four pendant 30 minutes.
- Préparer un méthoxy 50:1: biotine PEG mélange dans 100 mM NaHCO 3 à pH 8,2. But pour environ 0,2% p / v PEG biotinylé.
- Pipeter 100 uL du mélange PEG sur une lamelle sèche silanisé et placer une autre lamelle sur le dessus. Y compris une entretoise lamelle de verre permettra la séparation des lamelles.
- Incuber les lamelles dans la solution de PEG pendant 3 heures, puis de séparer chaque paire de lamelles et laver abondamment avec de l'eau. Veillez à garder les lamelles latérales fonctionnalisées en tant que côté une fois sera recouvert avec du PEG.
- Sécher les lamelles et stocker sous vide. Les surfaces restent stables pendant au moins un mois, donc des dizaines de lamelles peuvent être faites dans un lot et utilisés au besoin.
Ces matériaux doivent être faites d'avance, puis utilisées pour chaque expérience. Pour commencer chaque expérience unique molécule, commencer par rassembler la chambre d'écoulement.
3. Préparation Chambre de circulation
L'expérience est réalisée en utilisant une chambre de flux simple, construit avec une lamelle fonctionnalisée, scotch double-face, une lame de quartz et les tubes. Une chambre d'écoulement est préparé pour le 1,4 expérimentation chaque molécule unique.
Matériaux: ruban adhésif double face, une lame de rasoir, glisser à quartz avec des trous pour tuyaux, à séchage rapide époxy, lamelle fonctionnalisés (voir ci-dessus), une solution de streptavidine (25 uL de 1 mg / ml dans le PBS), tubes de tampon de blocage (20 mM Tris pH 7,5, EDTA 2 mM, NaCl 50 mM, 0,2 mg / ml BSA, 0,005% de Tween-20)
- Commencez par placer 20-25 ml de tampon de blocage dans un dessiccateur pour éliminer les bulles d'air pour les étapes ultérieures.
- Prenez une lamelle de PEG-fonctionnalisés et la propagation de la solution de streptavidine 125 PBS-dilué sur la surface. Laissez ce à incuber pendant 20 minutes tout en préparant d'autres parties de la chambre, ce qui permet de lier streptavidine biotines surface.
- Coupez un morceau de double facebandes d pour correspondre à la taille de la diapositive à quartz. Marquer la position des trous de tuyau sur la bande avec un crayon afin que les grandes lignes de la chambre d'écoulement peuvent être tirées sur la bande (1 mg / ml).
- Faire un 2 mm de large rectangle autour des trous. Ce sera le canal d'écoulement, donc soyez sûr de faire les côtés droit et laisser de la place autour de l'entrée et la sortie de tube. Si désiré, des canaux multiples ou de tailles différentes peuvent être utilisées.
- Coupez le long du contour dessiné, rendant coupes droites et propres pour s'assurer que personne ne dépasse colle dans le canal.
- Nettoyez soigneusement l'acétone de quartz à l'aide ou l'éthanol pour enlever l'adhésif de la construction de cellules d'écoulement précédentes.
- Trouver le meilleur alignement du contour de canal, retirer un côté de l'adhésif et placer soigneusement le ruban sur la lame de quartz. Soyez prudent afin d'aligner la bande correctement, comme les trous d'entrée et de sortie doivent rester débloqué.
- Couper des longueurs de tuyaux pour l'entrée et la sortie de la cellule d'écoulement. Il aide à couper l'extrémité du tube à environ un angle de 30 ° pour que le tube ne sera pas de presse à plat contre le fond de chambre. Placer les tubes sur une sorte de soutien pour les suspendre pour une fixation facile à la cellule de flux dans les prochaines étapes.
- Rincer la lamelle recouvertes de streptavidine abondamment à l'eau et sécher à l'air comprimé. Rappelez-vous que seul un côté est fonctionnalisé; attention à ne pas tourner la lamelle sur. Retirer de l'autre côté du support cassette et placer la lame de quartz sur la lamelle.
- Appuyez légèrement sur la lamelle pour faire sortir tout l'air emprisonné dans la colle. Cela permettra d'éviter les bulles d'air d'entrer dans le canal d'écoulement.
- Scellez les côtés de la chambre à l'époxy. Insérer le tuyau coupé dans les trous du quartz et sceller en place à l'époxy. Cela doit sécher pendant quelques minutes.
- Une fois sèche, commencer à bloquer la surface en tirant partie de la mémoire tampon dégazée par un blocage de tubes. Une aiguille de calibre 21 correspond parfaitement à l'intérieur du tube de 0,76 mm. Rincez plusieurs fois pour enlever les bulles d'air, et permettre à la chambre à incuber pendant au moins une demi-heure.
4. Expérience de la réplication de molécules simples
Matériaux: chambre de flux Préparé, SYTOX d'Orange, un microscope TIRF, 532 nm laser, caméra CCD, de l'informatique avec le logiciel d'acquisition d'image, substrat d'ADN de cercle roulant, les protéines de réplication
- Après la cellule de débit a été bloqué, tout est prêt pour commencer l'expérience unique molécule.
- Prenez la cellule d'écoulement et le placer sur la platine du microscope. Tenir la chambre en place avec des clips scène et être sûr que le canal d'écoulement est positionné directement le long de l'axe y.
- Connectez le tube de sortie de flux de cellules à l'aide d'une pompe seringue tube diamètre de connecteur ou une aiguille. Placer l'entrée dans le tampon de blocage et tirez sur la seringue pour enlever tout l'air dans le tube. Un film de douceur du tube de sortie aidera à éclaircir les bulles d'air piégées dans la cellule d'écoulement.
- Diluer la matrice d'ADN d'actions à 25 pM dans 1 ml de tampon de blocage. Débit dans la chambre à débit modéré pour permettre une couverture de surface bien de l'ADN. 0,025 ml / min pendant 30 minutes fonctionne bien. Cela peut être varié en fonction du nombre de molécules d'ADN sont sur la surface de chaque lot de lamelles ou combien sont souhaitées pour l'expérience.
- Une fois que l'ADN est dans, laver tout excédant d'ADN à l'aide du tampon de blocage. Laver pendant au moins 200 flux de cellules volumes pour se débarrasser de tout l'ADN libre.
- Commencez dégazage le tampon de la réplication. Pour les E. expériences de réplication coli, cela à 37 ° C pour réduire le risque de bulles dans la chambre d'écoulement chauffé.
- Allumez le laser et la caméra. Le CCD refroidie doit atteindre la température avant de pouvoir être utilisé.
- Si vous effectuez une expérience de 37 ° C, tournez le chauffage. Soyez sûr l'objectif est en contact avec la cellule de flux ou il sera un dissipateur thermique plus tard.
- Après le lavage et le dégazage, la réaction peut être préparé. Nucléotides Mix, la TNT, et SYTOX dans le tampon de la réplication. Puis ajouter les protéines et les mélanger délicatement.
- Débit du mélange réactionnel dans la cellule de flux. Cette vitesse d'écoulement doit être suffisante pour étirer ADNdb. Pour une 2-mm de large, 100-um canal d'écoulement hauteur 0,04 ml / min fonctionne bien.
- Prévoyez du temps pour le mélange à pénétrer dans la chambre et commencer l'imagerie. Un bon conseil est de déplacer le champ de vision sur le côté du canal et de se concentrer sur le matériau adhésif. Cela permettra d'assurer vous êtes près de la surface de focalisation.
- Régler la puissance du laser, mise au point microscope, et l'angle de la FRBR. Gardez la faible puissance pour éviter toute photoclivage de l'ADN coloré. Acquérir à 1-5 images / seconde pendant plusieurs minutes, selon le taux de réplication de l'ADN. Si vous le souhaitez, répétez pour obtenir des trajectoires plus de réplication ou de passer à un nouveau champ pour voir plus de molécules.
- Après le mélange réactionnel est presque vide, c'est une bonne idée de circuler dans plusieurs tampons avec SYTOX pour voir d'autres champs de vue. Prendre des images multiples de différentes molécules reproduit fournit des statistiques pour la détermination de processivité.
5. Les résultats représentatifs
Molécules répliquant activement sont facilement considérées comme des lignes de plus en plus de l'ADN coloré. Dans nos expériences, coulant 25 pM du substrat M13 donne 100-1000 molécules dans un 60x, 125 um um champ x 125 de vue (figure 1). Réaliser des expériences de réplication en utilisant les E. protéines coli réplisome à 37 ° C (voir Matériaux pour plus de détails) donne des molécules de 50 à 50 répliquant par champ de vue. A des concentrations équivalentes de l'réplisome T7, qui initie sur le substrat beaucoup plus efficacement, nous observons> 70% des molécules dans un champ de répliquer. Ces conditions donnent une densité du produit si élevé que des molécules individuelles sont difficiles à résoudre et d'analyser, de sorte que les expériences sont réalisées à T7 concentrations de protéines plus faible.
Analyse des données: Pour obtenir des données de taux, il suffit de tracer l'extrémité de l'ADN en fonction du temps et de calculer la pente (figure 2). Pour processivité, déterminer la longueur totale de l'ADN. Ces deux numéros devront être convertis en paires de bases. Avant d'effectuer l'expérience, vous devriez connaître la taille de pixel de la caméra à l'agrandissement approprié. Pour la conversion de paires de bases, une bonne estimation est tout simplement la conversion basé sur la longueur cristallographique de l'ADN, 2,9 bp / nm. Cependant, un écoulement laminaire ne sera pas complètement étirer l'ADN, donc un calibrage de longueur d'ADN devraient être effectuées en prenant un ADN de longueur connue, par exemple 48 502 pb λ ADN, de le fixer à la cellule de flux avec un oligo biotinylé et en mesurant sa SYTOX teinté longueur à le débit utilisé pour l'expérience de la réplication. Déterminer le nombre de paires de base / pixel permet un calcul précis des taux de réplication et processivities. Prendre de nombreuses images et des films offrira un grand nombre de molécules de produits, permettant de traçage de distributions de taux et de processivité (figure 2) 1.
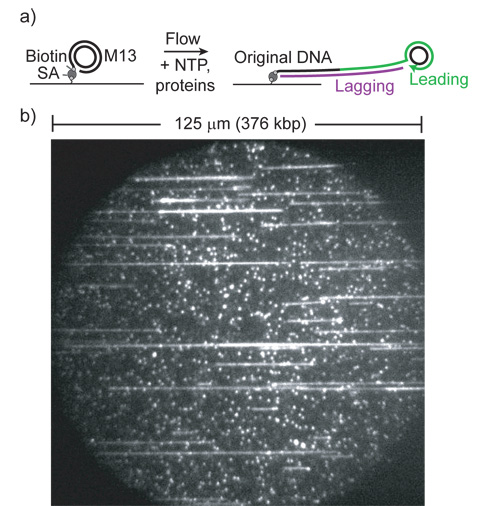
Figure 1: (A partir Ref 1.) Un dessin animé) de l'essai de réplication de cercle roulant. (SA, streptavidine). Diriger brin synthèse étend la queue reliant le cercle et la surface. La queue est converti en ADN double brin par la polymérase brin retardé et étirée par flux laminaire.
Exemple de terrain b) de vue avec SYTOX Orange et d'excitation 532 nm. De petites taches fluorescentes sont attachés, non répliquée substrats. Notez la longueur extrême des produits et le grand nombre de produits par le terrain (chaque cellule contient des flux> 5000 domaines tels).

Figure 2: (adapté de référence 1.) A, b) Kymographs des molécules typiques répliquant de T7 (a) et E. coli (b) des expériences. c) les trajectoires des molécules à partir de Endpoint a) et b) tracée en fonction du temps. Les trajectoires sont équipés de régression linéaire pour obtenir des taux de réplication. Les exemples montrés sont 99,4 pb s -1 et 467,1 pb s -1. d) Longueur distributions de produits de réplication, l'ajustement avec décroissance exponentielle simple à obtenir processivité: 25,3 ± 1,7 kpb (T7), 85,3 ± 6,1 kpb (E. coli). e) Taux de distribution des trajectoires seule molécule, l'ajustement des gaussiennes unique. Moyens: 75,9 ± 4,8 s -1 pb (T7), 535,5 ± 39 pb s -1 (E. coli).
Discussion
Une critique de contrôle n'est examinant l'effet de la tache orange, SYTOX, sur les protéines de réplication d'intérêt. Une façon simple de faire cela est de réaliser l'expérience de réplication dans la cellule de flux comme décrit mais avec l'omission de SYTOX. Après le mélange réactionnel a coulé dans la chambre, ajoutez tampon avec SYTOX pour colorer l'ADN et d'examiner la distribution des longueurs de molécules répliquées. Alternativement, les réactions en vrac standard peut être utilisé pour vérifier tout effet de SYTOX sur le taux de réplication et de l'efficacité.
L'expérience décrite ici utilise seulement un canal d'écoulement unique. Ceci peut être changé facilement en créant multi-canal chambres d'écoulement ou de l'utilisation de PDMS ou similaire dispositifs microfluidiques. Augmenter le nombre de canaux facilite grandement le dépistage des concentrations de protéines, des mutants, ou des molécules d'inhibiteur et augmente la rapidité de la réplication de données de collecte.
Comme mentionné précédemment, nous effectuons les E. expériences de réplication coli à 37 ° C à l'aide d'un chauffe-maison construite en aluminium flux de cellules, un élément résistif chauffant (cartouche chauffante) et une alimentation variable. Cela donne une bonne stabilité en température et évite l'achat d'un chauffe-objectif. Pour calibrer l'appareil de chauffage, nous avons simplement percé un trou dans le centre d'une cellule à écoulement supérieure à quartz, a inséré un thermocouple dans le canal d'écoulement et coulé tampon comme normale. Mesure de la température du tampon de flux de cellules à des tensions croissante permet un chauffage précis.
Remerciements
Samir Hamdan aidé dans le développement de cette technique. E. coli sont des protéines du laboratoire du professeur Nick Dixon, protéines de l'Université de Wollongong, et T7 sont du professeur Charles Richardson, Harvard Medical School. Le travail est soutenu par le National Institutes of Health (GM077248 d'AMvO) et la Fondation Jane Coffin Childs (JJL).
matériels
Réplication T7: 40 mM Tris pH 7,5, 50 mM de glutamate de potassium, chlorure de magnésium 10 mM, 100 ug / ml de BSA, avec 5 mM de dithiothréitol, 600 uM dNTP, 300 uM ATP, CTP 300 uM, et 15 nM SYTOX orange ajouté immédiatement avant utilisation. Protéines ajouté que: 5 nM GP4 (hexamère), 40 nM polymérase (1:1 gp5: thiorédoxine), 360 nm gp2.5 1,5.
La réplication de E. coli: 50 mM HEPES pH 7,9, 12 mM d'acétate de magnésium, chlorure de potassium 80 mM, 100 ug / ml de BSA avec 10 mM de dithiothréitol, 40 uM dNTP, 200 rNTP uM, et 15 nM SYTOX orange ajouté immédiatement avant utilisation. Protéines ajouté que: 30 nM DnaB (hexamère), 180 Nm DNAC (monomère), 30 nM αεθ, 15 nM τ γ 2 1 δδ'χψ ou τ 3 δδ'χψ, 30 nM β (dimère), 300 nM DnaG, 250 nM SSB (tétramère), 20 nM Pria, 40 nM Prib, 320 nM pric, 480 nM DNAT 1,6
Références
- Tanner, N. A., et al. Real-time single-molecule observation of rolling-circle DNA replication. Nucleic Acids Res. 37, e27(2009).
- Tabor, S., Huber, H. E., Richardson, C. C. Escherichia coli thioredoxin confers processivity on the DNA polymerase activity of the gene 5 protein of bacteriophage t7. J Biol Chem. 262, 16212-16223 (1987).
- Sofia, S. J., Premnath, V. V., Merrill, E. W. Poly(ethylene oxide) grafted to silicon surfaces: Grafting density and protein adsorption. Macromolecules. 31, 5059-5070 (1998).
- Tanner, N. A., van Oijen, A. M. Single-molecule observation of prokaryotic DNA replication. DNA replication: Methods and protocols. Vengrova, S., Dalgaard, J. Z. 521, Humana Press. New York. 397-410 (2009).
- Hamdan, S. M., Loparo, J. J., Takahashi, M., Richardson, C. C., van Oijen, A. M. Dynamics of DNA replication loops reveal temporal control of lagging-strand synthesis. Nature. 457, 336-339 (2009).
- Heller, R. C., Marians, K. J. Replication fork reactivation downstream of a blocked nascent leading strand. Nature. 439, 557-562 (2006).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon