Method Article
Replicação de DNA visualizar Único molécula com microscopia de fluorescência
Neste Artigo
Resumo
Este protocolo demonstra uma simples molécula única técnica de microscopia de fluorescência para a visualização de replicação do DNA por replisomes individuais em tempo real.
Resumo
Nós descrevemos um simples microscopia de fluorescência baseada em método em tempo real para observar a replicação do DNA ao nível de moléculas individuais. A circular, modelo de DNA bifurcada é anexado a uma lamela de vidro funcionalizados e replicados extensivamente após a introdução das proteínas de replicação e nucleotídeos (Figura 1). O crescimento do produto DNA dupla fita (dsDNA) é estendido com fluxo laminar e visualizados usando um corante intercalando. Medir a posição do fim de DNA crescente em tempo real permite a determinação precisa da taxa de replicação (Figura 2). Além disso, o comprimento dos produtos de DNA concluído relatórios sobre a processabilidade de replicação. Este experimento pode ser realizado muito facilmente e rapidamente e requer apenas um microscópio de fluorescência com uma câmera razoavelmente sensíveis.
Protocolo
Antes de realizar um experimento de replicação única molécula, alguns materiais precisam ser preparadas com antecedência.
1. Modelo de replicação do DNA
O substrato para a reação de replicação é um biotinilado, M13 círculo cauda rolando preparado usando o padrão de biologia molecular 1,2 técnicas.
Materiais: M13mp18 DNA de fita simples, primer oligonucleotídeo biotinilado cauda (5'-Biotina-T 36 AATTCGTAAT CATGGTCATAGCTGTTTCCT-3 '), T7 DNA polimerase, bloco de calor, fenol / álcool isoamílico / Clorofórmio
- Recozer a oligo cauda biotinilado para o M13, adicionando um excesso de 10 vezes de oligo em TBS tampão (330 nM cauda oligo, 33 nM M13).
- Aquecer a mistura a 65 ° C no bloco de calor. Uma vez aquecida, por sua vez o bloco de calor e deixe resfriamento lento corretamente anneal o primer.
- Adicione o M13 preparado para uma mistura de 64 nM DNA polimerase T7 e buffer de replicação T7 contendo dNTPs e MgCl 2.
- Incubar a reação a 37 ° C por 12 minutos e saciar com 100 mM EDTA.
- Purificar o DNA preenchido produto com fenol / clorofórmio / isoamílico extração de álcool e diálise em tampão TE ou buffer de armazenamento adequado e determinar a concentração de DNA (Back-extrair cada fase orgânica para recuperar qualquer residual preparado M13).
- Uma iteração do modelo de preparação pode facilmente fazer alguns ml de modelo de concentração nanomolar, o suficiente para centenas de milhares de single-molécula de experimentos.
2. Lamelas funcionalizados
Para prender a DNA para a lamela de vidro, o vidro é o primeiro funcionalizados com um aminosilane, que é acoplado a moléculas de PEG biotinilado. Este revestimento ajuda a reduzir as interações não específicas de DNA e proteínas de replicação com a superfície 1,3.
Materiais: lamínulas de vidro, potes de coloração, 3-aminopropyltriethoxysilane, Methoxy-PEG5k NHS-éster, Biotina-PEG5k-NHSester, acetona, 1M KOH, Etanol, Forno sonicador Bath,
- Limpam-se as lamínulas de vidro, colocando-os na coloração frascos e encher os frascos com etanol. Sonicado por 30 minutos e enxaguar os frascos e encher com 1 M KOH. Sonicado por 30 minutos Repita os dois lavagens.
- Para a reação de funcionalização em primeiro lugar, todos os vestígios de água precisam ser removidos. Enchei as talhas de acetona e sonicate por 10 min. Enxaguar os frascos novamente com acetona.
- Prepare uma solução 2% v / v do reagente silano em acetona. Adicionar ao tinas de coloração e agitar por 2 minutos. Esta reação casais do grupo de alcóxi de aminosilane ao vidro, deixando uma amina reativos para a etapa seguinte de acoplamento. Saciar a reação com um grande excesso de água.
- Seca as lamelas cozendo-los a 110 ° C no forno por 30 minutos.
- Prepare um metoxi 50:1: biotina mistura PEG em 100 mM NaHCO 3 pH 8.2. Apontar para cerca de 0,2% PEG w / v biotinilado.
- Pipetar 100 mL da mistura PEG em uma lamela seca silanizadas e colocar outra lamínula por cima. Incluindo um espaçador lamela de vidro vai permitir a separação das lamelas.
- Incubar as lamelas na solução de PEG por 3 horas, em seguida, separe cada par de lamelas e lavar abundantemente com água. Tenha cuidado para manter as lamelas funcionalizados lateral-se como apenas o lado uma vez serão revestidas com PEG.
- Seca as lamelas e armazenar sob vácuo. As superfícies de permanecer estável por pelo menos um mês, assim que dezenas de lamelas podem ser feitas em um lote e usado quando necessário.
Estes materiais devem ser feitas antes do tempo, e então usado para cada experimento. Para iniciar cada experimento molécula única, comece a montagem da câmara de fluxo.
3. Preparação câmara de fluxo
O experimento é realizado com uma câmara de fluxo simples construída com uma lamela funcionalizados, fita dupla face, uma lâmina de quartzo e tubo. Uma câmara de fluxo está preparada para 1,4 cada molécula única experiência.
Materiais: fita dupla face, lâmina de barbear, lâmina de quartzo com furos para tubos de epóxi, Quick-seca, lamela funcionalizados (veja acima), Estreptavidina solução (25 mL de 1 mg / mL em PBS), tubulação, tampão de bloqueio (20 mM Tris pH 7,5, 2 mM EDTA, 50 mM NaCl, 0,2 mg / mL BSA, 0,005% Tween-20)
- Comece colocando 20-25 mL de tampão de bloqueio em um dessecador para remover quaisquer bolhas de ar para etapas posteriores.
- Dê uma lamela PEG-funcionalizados e espalhar 125 de PBS-diluída solução de estreptavidina sobre a superfície. Deixar isso para incubar por 20 minutos enquanto prepara outras partes da câmara, permitindo estreptavidina para ligar a Biotins superfície.
- Corte um pedaço de dupla faced fita para combinar com o tamanho da lâmina de quartzo. Marcar a posição dos furos de tubo na fita com um lápis, para que um esboço da câmara de fluxo podem ser tiradas na fita (1 mg / mL).
- Faça um retângulo de 2 mm de largura em torno dos furos. Isso servirá como um canal de fluxo, por isso não deixe de fazer os lados em linha reta e deixar espaço ao redor da tubulação de entrada e saída. Se desejar, múltiplos canais ou tamanhos diferentes podem ser usados.
- Corte ao longo do contorno desenhado, tornando reto, cortes limpos para garantir que nenhum sobressaia adesivo para o canal.
- Limpe o quartzo, cuidadosamente, com acetona ou etanol para remover adesivo da construção anterior de fluxo de células.
- Encontrar o melhor alinhamento do contorno do canal, remove um dos lados da camada adesiva e coloque cuidadosamente a fita sobre a lâmina de quartzo. Tenha o cuidado de alinhar a fita corretamente, como os buracos de entrada e saída necessidade de permanecer desbloqueados.
- Cortar comprimentos de tubulação para a entrada e saída da célula de fluxo. Isso ajuda a cortar a extremidade do tubo em cerca de um ângulo de 30 ° para que o tubo não vai pressionar plana contra o fundo de câmara. Colocar os tubos em algum tipo de apoio a suspendê-las para fixação fácil a célula de fluxo nas próximas etapas.
- Lavar a lamela estreptavidina revestida abundantemente com água e seque com ar comprimido. Lembre-se que apenas um lado é funcionalizado; ter cuidado para não transformar a lamela mais. Remover o outro lado do suporte de fita e colocar a lâmina de quartzo para a lamela.
- Pressione levemente na lamela a empurrar para fora todo o ar aprisionado no adesivo. Isso ajudará a evitar as bolhas de ar entrando no canal de fluxo.
- Vedação dos lados da câmara com epóxi. Insira o tubo de corte nos orifícios do quartzo e foca no lugar com epóxi. Isso precisa secar por alguns minutos.
- Depois de seco, comece a bloquear a superfície, puxando alguns dos desgaseificados tampão de bloqueio através de um dos tubos. A agulha calibre 21 se encaixa perfeitamente dentro do tubo de 0,76 milímetros. Lavar algumas vezes para remover bolhas de ar, e permitir que a câmara para incubar por pelo menos meia hora.
4. Única molécula-Experimento de replicação
Materiais: câmara de fluxo preparados, SYTOX Orange, microscópio TIRF, 532 nm do laser, CCD da câmera, computador com software de aquisição de imagem, Rolling círculo substrato DNA, proteínas de replicação
- Após a célula de fluxo foi bloqueado, tudo está pronto para começar a experiência única molécula.
- Pegue a célula de fluxo e colocá-lo no palco microscópio. Segure a câmara no lugar com clipes de palco e ter certeza de que o canal de fluxo é posicionado em linha reta ao longo do eixo-y.
- Conecte o tubo de saída de fluxo de células para a bomba de seringa, usando um tubo conector maior diâmetro ou uma agulha. Coloque a entrada em tampão de bloqueio e puxe a seringa para remover qualquer ar no tubo. Um filme suave do tubo de saída vai ajudar a limpar as bolhas de ar aprisionado na célula de fluxo.
- Diluir o modelo de DNA de ações a 25 pM em 1 mL de tampão de bloqueio. Fluxo para a câmara de fluxo moderado para permitir a cobertura boa superfície de DNA. 0,025 mL / min por 30 minutos funciona bem. Isto pode ser variado com base em quantas moléculas de DNA estão na superfície para cada lote de lamelas ou quantos forem desejados para o experimento.
- Uma vez que o DNA se encontra, lavar DNA em excesso com tampão de bloqueio. Lavagem para pelo menos 200 o fluxo de células-volumes para se livrar de todo o DNA livre.
- Começar a desgaseificação do buffer de replicação. Para a E. experimentos de replicação coli, fazer isso a 37 ° C para reduzir o risco de bolhas na câmara de fluxo aquecido.
- Ligue o laser e câmera. O CCD refrigerado precisa atingir a temperatura antes que ele possa ser usado.
- Se realizar a 37 ° C experimento, por sua vez o aquecedor ligado. Certifique-se o objetivo está em contato com a célula de fluxo ou ele vai ser um dissipador de calor mais tarde.
- Após a lavagem e desgaseificação, a reação pode ser preparado. Nucleotídeos Mix, DTT, e SYTOX no buffer de replicação. Em seguida, adicione proteínas e misture delicadamente.
- Fluxo da mistura de reação dentro da célula de fluxo. Esta velocidade do fluxo deve ser suficiente para esticar dsDNA. Para a 2 mm de largura, canal de fluxo 100 mícrons de altura 0,04 mL / min funciona bem.
- Dar tempo para a mistura de entrar na câmara e começar a imagem. Uma boa dica é mover o campo de visão para o lado do canal e foco no material adesivo. Isso irá assegurar que você está perto da superfície para focagem.
- Ajustar a potência do laser, o foco do microscópio, e ângulo TIRF. Manter o baixo consumo de energia para evitar qualquer photocleavage do DNA manchado. Adquira com 1-5 frames / segundo por vários minutos, dependendo da taxa de replicação do DNA. Se desejar, repita para obter trajetórias mais de replicação ou mover para um novo campo para ver mais moléculas.
- Após a mistura de reação é quase vazio, ele é uma boa idéia a fluir com mais buffer com SYTOX para ver os campos de vista do outro. Tomando várias imagens de diferentes moléculas replicado fornece estatísticas para a determinação de processabilidade.
5. Resultados representante
Moléculas replicantes ativamente são facilmente visíveis como linhas crescente de DNA manchado. Em nossos experimentos, fluindo 25 pM do substrato M13 dá 100-1000 moléculas em um 60x 125 M x, campo 125 mM de vista (Figura 1). Realização de experimentos de replicação usando o E. proteínas coli replissoma a 37 ° C (veja Materiais para detalhes) dá moléculas replicantes 50-50 por campo de visão. Em concentrações equivalentes dos replissoma T7, que inicia no substrato com muito mais eficiência, observa-se> 70% das moléculas em um campo de replicar. Estas condições produzem uma densidade do produto tão alto que moléculas individuais são difíceis de resolver e analisar, de modo que o T7 experimentos são realizados em concentrações mais baixas de proteína.
Análise de dados: Para obter dados de taxa, basta traçar o ponto final do DNA em função do tempo e calcular a inclinação (Figura 2). Para processabilidade, determinar o comprimento total do DNA. Ambos estes números terão de ser convertido em pares de bases. Antes de executar o experimento, você deve saber o tamanho do pixel da câmera para a ampliação adequada. Para a conversão de pares de bases, uma boa estimativa é simplesmente a conversão com base no comprimento de cristalografia de DNA, 2,9 pb / nm. No entanto, o fluxo laminar não será completamente esticar o DNA, para uma calibragem de comprimento DNA deve ser realizado tomando um DNA comprimento conhecido, por exemplo, 48.502 bp λ DNA, anexá-lo à célula de fluxo com um oligo biotinilado e medir seu comprimento SYTOX manchada no a vazão utilizada para o experimento de replicação. Determinar o número de pares de bases / pixel permitirá o cálculo preciso das taxas de replicação e processivities. Levando inúmeras imagens e filmes irá fornecer um grande número de moléculas do produto, permitindo plotagem de taxa e processabilidade distribuições (Figura 2) 1.
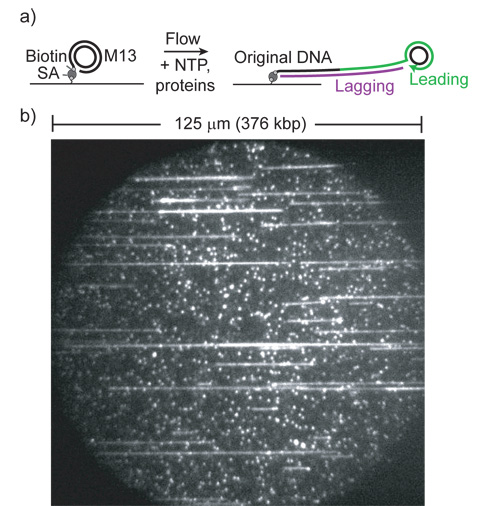
Figura 1: (De Ref 1.) Um dos desenhos animados) do ensaio de replicação rolling-círculo. (SA, estreptavidina). Levando-vertente de síntese se estende a cauda ligando o círculo e de superfície. A cauda é convertido para dsDNA pela polimerase vertente em atraso e se estendia pelo fluxo laminar.
b) Exemplo de campo de visão com SYTOX Laranja e 532 nm de excitação. Pequenas manchas fluorescentes são amarrados, não replicado substratos. Observe o comprimento do extremo dos produtos e ao grande número de produtos por campo (cada célula de fluxo contém> 5000 tais campos).

Figura 2: (adaptado da Ref. 1.) A, b) Kymographs típico de moléculas replicadoras de T7 (a) e E. coli (b) experimentos. c) as trajetórias Endpoint de moléculas de a) e b) plotados em função do tempo. Trajetórias estão equipados com a regressão linear para obter taxas de replicação. Exemplos mostrados são 99,4 bp s -1 e 467,1 bp s -1. d) Comprimento distribuições de produtos de replicação, se encaixam com decaimento exponencial simples para obter processabilidade: 25,3 ± 1,7 kbp (T7), 85,3 ± 6,1 kbp (E. coli). e) Taxa de distribuições de trajetórias única molécula, se encaixam com gaussianas único. Significa: 75,9 ± 4,8 s -1 bp (T7), 535,5 ± 39 bp s -1 (E. coli).
Discussão
Um crítico de controlo está a analisar o efeito da mancha, SYTOX Orange, sobre as proteínas de replicação de interesse. Uma maneira simples de fazer isso é realizar o experimento de replicação na célula de fluxo, como descrito, mas com a omissão de SYTOX. Após a mistura de reação fluiu através da câmara, adicionar tampão com SYTOX para manchar DNA e examinar a distribuição do comprimento das moléculas replicado. Alternativamente, reações granel padrão pode ser utilizado para verificar qualquer efeito sobre a taxa de SYTOX replicação e eficiência.
O experimento aqui descrito utiliza apenas um canal de fluxo único. Isto pode ser alterado facilmente através da criação de multi-canal de câmaras de fluxo ou usando PDMS ou similar dispositivos microfluídicos. Aumentar o número de canais facilita muito triagem das concentrações de proteínas, os mutantes, ou moléculas de inibidor e aumenta a rapidez da coleta de dados de replicação.
Como mencionado, realizamos a E. experimentos de replicação coli a 37 ° C utilizando uma casa construída em alumínio aquecedor de célula de fluxo, um elemento de aquecimento resistivo (aquecedor de cartucho) e fonte de alimentação variável. Isto dá boa estabilidade de temperatura e evita a compra de um aquecedor objetivo. Para calibrar o aquecedor, nós simplesmente um furo no centro de um alto fluxo de quartzo celular, inserido um termopar no canal de fluxo e fluiu buffer como normal. Medir o fluxo de temperatura tampão celular em tensões crescentes permite aquecimento precisas.
Agradecimentos
Samir Hamdan auxiliado no desenvolvimento desta técnica. E. coli são proteínas do laboratório do Prof Nick Dixon, proteínas Universidade de Wollongong, e T7 são do Prof Charles Richardson, Harvard Medical School. Trabalho é apoiado pelos Institutos Nacionais de Saúde (GM077248 para AMvO) e Jane Coffin Childs Foundation (JJL).
Materiais
T7 Replicação: 40 mM Tris pH 7,5, 50 mM glutamato de potássio, 10 mM de cloreto de magnésio, 100 mg / mL BSA, com 5 mM ditiotreitol, 600 mM dNTPs, 300 mM ATP, 300 mM CTP, e 15 nM SYTOX Laranja adicionado imediatamente antes uso. Proteínas adicionadas como: 5 nM gp4 (hexâmero), 40 nM polimerase (1:1 GP5: thioredoxin), 360 nm gp2.5 1,5.
E. coli replicação: 50 mM HEPES pH 7,9, 12 mM de acetato de magnésio, 80 mM de cloreto de potássio, 100 mg / mL BSA com 10 mM ditiotreitol, 40 mM dNTPs, 200 rNTPs mM, e 15 nM SYTOX Laranja adicionado imediatamente antes do uso. Proteínas adicionadas como: 30 nM DnaB (hexâmero), 180 nM DNAC (monômero), 30 nM αεθ, 15 nM 2 τ γ uma δδ'χψ ou τ 3 δδ'χψ, 30 β nM (dimer), 300 nM DnaG, 250 nM SSB (tetrâmero), 20 nM Pria, 40 nM PRIB, 320 nM Pric, 480 nM DNAT 1,6
Referências
- Tanner, N. A., et al. Real-time single-molecule observation of rolling-circle DNA replication. Nucleic Acids Res. 37, e27(2009).
- Tabor, S., Huber, H. E., Richardson, C. C. Escherichia coli thioredoxin confers processivity on the DNA polymerase activity of the gene 5 protein of bacteriophage t7. J Biol Chem. 262, 16212-16223 (1987).
- Sofia, S. J., Premnath, V. V., Merrill, E. W. Poly(ethylene oxide) grafted to silicon surfaces: Grafting density and protein adsorption. Macromolecules. 31, 5059-5070 (1998).
- Tanner, N. A., van Oijen, A. M. Single-molecule observation of prokaryotic DNA replication. DNA replication: Methods and protocols. Vengrova, S., Dalgaard, J. Z. 521, Humana Press. New York. 397-410 (2009).
- Hamdan, S. M., Loparo, J. J., Takahashi, M., Richardson, C. C., van Oijen, A. M. Dynamics of DNA replication loops reveal temporal control of lagging-strand synthesis. Nature. 457, 336-339 (2009).
- Heller, R. C., Marians, K. J. Replication fork reactivation downstream of a blocked nascent leading strand. Nature. 439, 557-562 (2006).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados