Method Article
Oligo Messe Profiling (OLIMP) der extrazellulären Polysacchariden
In diesem Artikel
Zusammenfassung
Eine schnelle Art und Weise beschrieben, Einblicke in die Struktur der Polysaccharide in einer extrazellulären Matrix zu gewinnen. Die Methode nutzt die Spezifität von Glykosylhydrolasen und die Empfindlichkeit der Massenspektrometrie ermöglicht winzige Mengen von Materialien analysiert werden. Diese Technik ist anpassungsfähig, um direkt auf das Gewebe selbst verwendet werden.
Zusammenfassung
Der direkte Kontakt der Zellen für die Umwelt ist in vielen Organismen durch eine extrazelluläre Matrix vermittelt. Ein gemeinsamer Aspekt der extrazellulären Matrix ist, dass sie komplexe Zuckerreste in Form von Glykoproteinen, Proteoglykanen und / oder Polysaccharide enthalten. Beispiele hierfür sind die extrazelluläre Matrix des Menschen und tierischen Zellen, die hauptsächlich aus fibrillären Proteinen und Proteoglykanen oder Polysaccharid Zellwände von Pflanzen und Pilzen, und die Proteoglykan / Glycolipid Basis Zellwände von Bakterien. Alle diese Glykostrukturen spielen entscheidende Rollen in Zell-Zell-und Zell-zu-Umfeld Kommunikation und Signalisierung.
Eine außerordentliche komplexeres Beispiel für eine extrazelluläre Matrix ist in die Wände der höheren Pflanzenzellen. Ihre Wand ist fast ausschließlich aus Zucker gemacht, bis zu 75% Trockengewicht, und besteht aus den am häufigsten vorkommende Biopolymere präsentieren auf diesem Planeten. Daher ist die Forschung durchgeführt, wie diese Materialien am besten zu nutzen als eine Kohlenstoff-neutrale erneuerbare Ressource zu petrochemischen Produkten aus fossilen Brennstoffen gewonnen zu ersetzen. Die größte Herausforderung für Brennstoff Umwandlung bleibt die Widerspenstigkeit der Wände enzymatische oder chemische Abbau aufgrund der einzigartigen Glykostrukturen in dieser einzigartigen Biokomposit.
Hier stellen wir ein Verfahren zur schnellen und empfindlichen Analyse der pflanzlichen Zellwand Glykostrukturen. Diese Methode Oligo Messe Profiling (OLIMP) ist die enzymatische Freisetzung von Oligosacchariden aus Wandmaterialien Erleichterung spezifische Glykosylhydrolasen und anschließende Analyse der gelösten Oligosaccharid Mischungen unter Verwendung von Matrix-Assisted Laser Desorption / Ionisation Time-of-flight Massenspektrometrie (MALDI-TOF/MS Basis ) 1 (Abbildung 1). OLIMP erfordert Wände von nur 5000 Zellen für eine vollständige Analyse können auf das Gewebe selbst 2 durchgeführt werden, und ist zugänglich für High-Throughput-Analysen 3. Während die absolute Menge des gelösten Oligosaccharide können nicht durch OLIMP bestimmt die relative Häufigkeit der verschiedenen Oligosaccharid-Ionen aus den Massenspektren geben Einblicke über die Substitution-Muster der nativen Polysaccharid in der Wand abgegrenzt werden.
OLIMP kann verwendet werden, um eine Vielzahl von Wand-Polymeren, die nur durch die Verfügbarkeit von bestimmten Enzymen 4 begrenzt zu analysieren. Zum Beispiel für die Analyse von Polymeren, die in der pflanzlichen Zellwand Enzyme sind für die Hemicellulosen Xyloglucan mit einem Xyloglucanase 5, 11, 12, 13, Xylan mit einem Endo-β-(1-4)-Xylanase 6,7 analysieren oder für Pektinstoffe Polysacchariden mit einer Kombination aus einer Polygalacturonase und Methylesterase 8. Darüber hinaus kann nach den gleichen Grundsätzen von OLIMP glycosylhydrolase und sogar Glycosyltransferase Aktivitäten überwacht und bestimmt werden 9.
Protokoll
Die OLIMP Methode beruht auf der großen Hemizellulose in den Zellwänden von zweikeimblättrigen Pflanzen, Xyloglucan Beispiel mit Hilfe eines Endoglucanase als glycosylhydrolase. Die Methode ist mit ganzen Arabidopsis-Keimlinge als Pflanzengewebe Quelle demonstriert. Enzyme und extrazelluläre Matrix-Material kann ersetzt je nach gewünschter Analyse mit dem gleichen Verfahren werden.
1. Zellwand isoliert
- Ernte fünf 5-Tage alte gesamten Arabidopsis-Keimlinge oder den entsprechenden Betrag von Ihrem gewünschten Material und übertragen Sie sie in ein 1,5 ml Reaktionsgefäß. Stellen Sie sicher, dass das Material in den Boden des Röhrchens befindet.
- Fügen Sie zwei 3mm Metallkugeln in das Reaktionsrohr auf der Probe und Snap-in flüssigem Stickstoff einfrieren.
- Grind die gefrorene Probe mit einer Kugelmühle (2:30 min, 25 Hz).
- 1ml 70% wässrigem Ethanol zur Probe. Entfernen Sie die Metallkugeln mit einem Magneten. Vortex gründlich.
- Centrifuge Probe bei 14.000 rpm für 10 Minuten, um Pellets der Alkohol unlösliche Rückstand enthält die Zellwand Material.
- Entfernen Sie vorsichtig den Überstand durch Absaugen. Achten Sie darauf, das Pellet stören.
- 1ml von Chloroform / Methanol (1:1 v / v) Lösung. Vortex gründlich, um das Pellet zu resuspendieren.
- Centrifuge Probe bei 14.000 rpm für 10 min und entfernen Sie vorsichtig den Überstand durch Absaugen. Achten Sie darauf, das Pellet stören.
- Trocknen Sie die Probe mit einem Konzentrator.
2. Solubilisierung von Oligosacchariden
- Für die Solubilisierung der jeweiligen Polysaccharid, das in Form seines Oligosaccharide resuspendieren die trockene Pellet in 25 &mgr; l eines geeigneten Puffers. Für die Endoglucanase 50mM Ammoniumformiat, pH4.5, verwendet wird. Add 0.2U der Endoglucanase. Vortex die Aussetzung und Spin Flüssigkeit nach unten.
- Inkubieren Sie die Probe für 16h bei 37 ° C in einem Inkubator, Schütteln bei 300rpm.
- Entfernen des Lösungsmittels der Verdauung (Wasser) mit einem Konzentrator.
3. MALDI-TOF-Analyse des freigesetzten Oligosaccharide
- Biorex MSZ 501 Kationenaustauscherharz Perlen werden verwendet, um Salze und Enzym aus der verdauten Probe zu entfernen. Zustand der Perlen, indem ein Aliquot in eine leere Spalte und Waschen der Beads mit reichlich Wasser.
- Add 5-10 Biorex Perlen, die verdaut und getrocknete Probe. Dann fügen Sie 10 &mgr; l Wasser, für kleinere Mengen an Material 5μl von Wasser verwendet werden können. Tauchen Sie die Perlen in der Lösung durch kurzes Drehen der Röhre. Inkubieren für mindestens 10 Minuten bei Raumtemperatur.
- Spot 2μl Matrix (2,5-Dihydroxybenzoesäure (DHB), 10mg/ml in Wasser) auf ein MALDI Zieltafel. Man dampft die Matrix Lösungsmittel unter Vakuum führt zu homogenen Matrix chemische Kristalle.
- Spot 2μl der entsalzt Probelösung auf der Matrix-Kristalle auf die Zielscheibe.
Wenn Sie eine große Anzahl von Proben vor Ort wollen nur an der Erkennung von maximal 3min zu halten. Dieser Zeitrahmen ist sichergestellt, dass der erste entdeckte Probe nicht trocken ist, noch. Warten Sie eine zusätzliche 2min zu einer Durchmischung wieder aufgelöst Matrix und Probe Oligosaccharidmoleküle in den letzten spotted Probe zu gewährleisten. Dann trocknen die Zielscheibe in einem Vakuum.
Die Probe / Matrix-Mischung sollte in weniger als 1 Minute, um eine homogene Kristallisation ermöglichen kristallisieren. - Legen Sie Zieltafel in ein MALDI-TOF-Massenspektrometer. Die Maschine sollte auf positive Reflektron-Modus mit einer Beschleunigungsspannung von 20000V und einer Verzögerung von 350 nm eingestellt werden; die ausgewählten Massenbereich sollte 500 bis 3000 Da werden (kann sich ändern, abhängig von der erwarteten Oligomere). Starten Auslösen des Laserstrahls und sammeln 100-200 Spektren pro Probe, die kompiliert werden, um einen repräsentativen Durchschnitt Spektrum (Abbildung 2A) zu erzeugen. Ionen repräsentieren spezifische Oligosaccharide können durch ihre Masse über Ladung (m / z)-Verhältnis ermittelt werden. Die Ionenintensitäten (Peakhöhe) aller Ionen von Interesse sollten bis zu 100%, was die relative Häufigkeit der einzelnen Oligosaccharid in der Probe (Abbildung 2B) hinzugefügt werden.
4. In situ OLIMP Analyse
OLIMP können auch direkt auf das Gewebe Weglassen jeder Wand Vorbereitungsschritte eingesetzt werden. Als Beispiel etiolierten Hypokotyledonen Arabidopsis als Pflanzengewebe Quelle verwendet werden. Auch hier ist eine Endoglucanase verwendet werden, um die Struktur der Hemicellulosen Xyloglucan zu bestimmen. Enzyme und Gewebe Material ersetzt je nach gewünschter Analyse mit dem gleichen Verfahren werden.
- Ernten im etiolierten Keimling und legen Sie es auf eine leere MALDI Zieltafel und lassen Sie die Sämlinge trocken.
- Add 0.5μl Endoglucanase (0.4U/μl, in 50 mM Ammoniumformiat aufgelöst, pH4.5) auf die getrocknete Keimling an der gewünschten Position. Stellen Sie sicher, dass das Enzym drop teilweise berührt die Zieltafel.
- Legen Sie die MALDI Zieltafel in einem geschlossenen Behälter mit sättigenden Feuchtigkeit zu vermeiden, dass das Enzym drop austrocknet. Die Platte in die Kammer für 16h bei 37 ° C.
- Dry das MALDI-Target-Platte mit dem Pflanzengewebe und das Enzym drop unter Vakuum.
- Add 0.5μl Matrix (DHB; 10mg / l) auf der jeweils getrocknet Enzym vor Ort. Lassen Sie es für 2min stehen.
- Trocknen Sie die MALDI Zieltafel unter Vakuum.
- Analysieren Sie die Proben mit der MALDI-TOF. Legen Sie Zieltafel mit dem Gewebe und das Enzym / Matrix-Spot (s) in ein MALDI-TOF-Massenspektrometer. Die Maschine sollte auf positive Reflektron-Modus mit einer Beschleunigungsspannung von 20000V und einer Verzögerung von 350 nm eingestellt werden; die ausgewählten Massenbereich sollte 500 bis 3000 Da werden (kann sich ändern, abhängig von der erwarteten Oligomere).
- Starten Auslösen des Laserstrahls auf die Matrix / Probe-Kristalle NEXT, um das Gewebe. Stellen Sie sicher, daß Sie nicht treffen das Gewebe selbst, wie es in einem solchen Fall die Zeit des Fluges der Moleküle wird ausgeschaltet. Sammeln Sie ca. 20-50 Spektren pro Spot, der kompiliert werden, um einen repräsentativen Durchschnitt Spektrum (Abbildung 3) zu erzeugen. Ionen repräsentieren spezifische Oligosaccharide können durch ihre Masse über Ladung (m / z)-Verhältnis ermittelt werden. Die Ionenintensitäten (ion Höhe) gemeldet und alle Ionen von Interesse sollten bis zu 100%, was die relative Häufigkeit der einzelnen Oligosaccharide in der Probe zugegeben werden.
5. Repräsentative Ergebnisse
Ein Beispiel für eine OLIMP Analyse der Hemicellulosen Xyloglucan in Arabidopsis-Keimlinge ist in Abbildung 2 dargestellt. Aufgrund der Masse Unterschiede der Ionen und der bekannten gut charakterisierte Enzym (Endoglucanase) Spezifität der Ionen kann auf bestimmte Oligosaccharid-Strukturen (Abbildung 2A) zugeordnet werden. Offensichtlich kann Strukturisomeren nicht unterschieden werden. Die Grundannahme für die Bestimmung der relativen Häufigkeit der verschiedenen Oligosaccharide (Abbildung 2B) ist, dass ihre Massenspektrometrie Responsefaktor sehr ähnlich für die Oligosaccharide ist. Wie hier gezeigt, ist die OLIMP Quantifizierung hoch reproduzierbar. Beachten Sie jedoch, dass Robustheit des Verfahrens stark abhängig von der Signal-Rausch-Verhältnis der verschiedenen Oligosaccharid-Ionen ist. Zum Beispiel Verunreinigungen mit Salzen oder geringere Mengen von Oligosacchariden abnehmen kann dieses Verhältnis.
Die OLIMP Analyse auf das Gewebe selbst (in-situ-Analyse) ermöglicht die Untersuchung von sehr kleinen und definierten Bereichen und beinhaltet sehr wenig Probenvorbereitung. In dem hier vorgestellten Beispiel (Abbildung 3) keine qualitativen (gleiche Ionen), aber quantitative Unterschiede (Variation in der Ionenintensitäten) wurden zwischen dem Spross und Wurzel-Gewebe des Arabidopsis Sämling beobachtet. Permutationen der OLIMP-Methode könnte somit das Gewebe "imaging" führen.
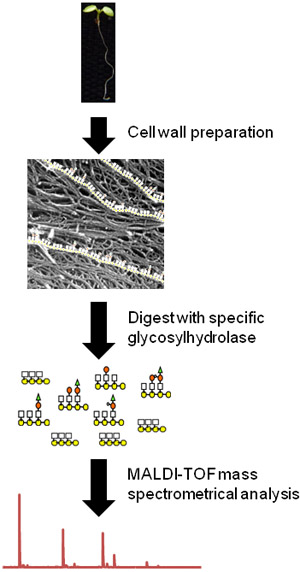
Abbildung 1. Flussdiagramm der OLIMP Verfahren mit ganzen Arabidopsis-Keimlinge als einer pflanzlichen Quelle. Zuerst wird das Gewebe mazeriert und Zellwand Material hergestellt (Bild aus Fujino et al modifiziert. 10). Dann Oligosaccharide einer bestimmten Wand Polysaccharid freigesetzt werden unter Verwendung eines spezifischen Hydrolase. Schließlich werden die relativen Häufigkeiten der löslichen Oligosacchariden bestimmt mittels MALDI-TOF-Massenspektrometrie.
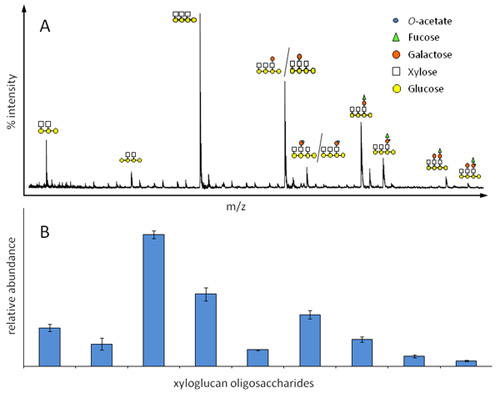
Abbildung 2. Relative Häufigkeit von Xyloglucan Oligosaccharide in etiolierten Keimlingen von Arabidopsis als von OLIMP bestimmt. A) Repräsentative Xyloglucan Oligosaccharid Massenspektrum; jedes Ion stellt eine spezifische Oligosaccharid-Struktur kann Strukturisomeren nicht unterschieden werden. B) Entsprechende Balkendiagramm für die Bestimmung der relativen Oligosaccharid Hülle und Fülle.
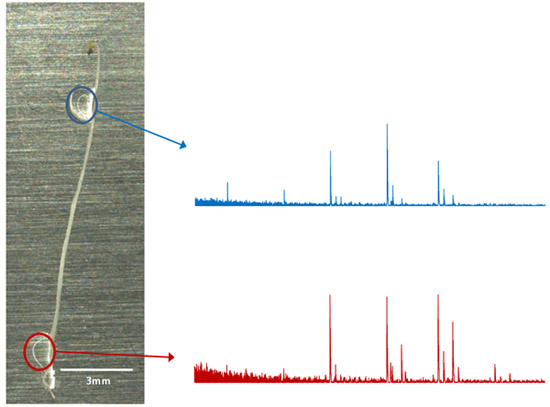
Abbildung 3. In situ OLIMP Analyse mit etiolierten Keimlingen von Arabidopsis als Beispiel. Jedes Enzym / Verdauung Tröpfchen (farbige Kreise) können unabhängig voneinander analysiert werden und eine entsprechende Massenspektren erhalten und analysiert werden.
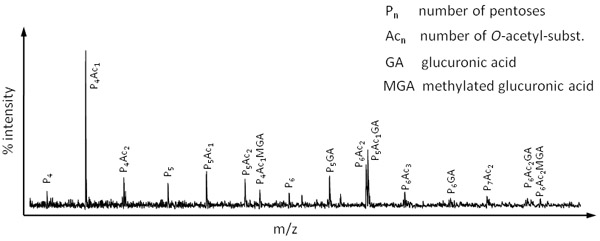
Abbildung 4. OLIMP Spektrum der Hemizellulose Xylan durch Verdauen von Miscanthus Blattmaterial mit einer Xylanase (Megazyme) erhalten.
Diskussion
Die OLIMP hier vorgestellte Methode ermöglicht eine sehr sensible und schnelle Analyse von Polymeren, die in der extrazellulären Matrix. OLIMP verbindet die enzymatische Freisetzung von Oligomeren mit anschließender MALDI-TOF-Analyse. Die Erzeugung eines MALDI-TOF-Spektrum dauert weniger als eine Minute, damit OLIMP ist geeignet für eine breite Palette von Anwendungen, einschließlich High-Throughput-Studien wie Mutantenscreens. OLIMP ist nicht auf pflanzliche Polysaccharide beschränkt, sondern kann potenziell ein breites Spektrum von Polymeren, die nur durch die Verfügbarkeit von bestimmten hydrolytische Enzyme beschränkt angewendet werden. Allerdings ist eine Beschränkung von OLIMP, dass eine absolute Fülle des Polymers nicht erreicht werden kann.
Wie bereits erwähnt OLIMP kann verwendet werden, um die Struktur einer Vielzahl von Polysacchariden in der extrazellulären Matrix von einer Vielfalt der Arten zu studieren. Als Beispiel stellt Abbildung 4 eine OLIMP Spektrum der wichtigsten Hemizellulose in Grasarten, Xylan. Hier wurde Zellwand Material aus den gemäßigten Gras Miscanthus abgeleitet verdaut mit einer Xylanase.
Offenlegungen
Danksagungen
Diese Arbeit wurde von der Energy Biosciences Institute gewähren OO0G01 finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| 2,5-dihydroxybenzoic acid | Sigma-Aldrich | 37550 | 10mg/mL in water |
| BioRex MSZ 501(D) Resin | Bio-Rad | 142-7425 | |
| Endoglucanase | Megazyme | E-CELTR | |
| Xylanase M6 | Megazyme | E-XYRU6 | |
| 3mm metal balls | Retsch | 22.455.0011 | |
| Beat mill | Retsch | Mixer Mill MM400 | |
| MALDI-TOF | Shimadzu Corporation | Axima Performance | |
| MALDI target plate | Kratos Analytical | DE4555TA | |
| SpeedVac | Eppendorf | Vacufuge 5301 | |
| Vacuum manifold | EMD Millipore | MSVMHTS00 | |
| Vacuum pump | Welch Allyn | DryFast Ultra 2032 |
Referenzen
- Lerouxel, O. Rapid structural phenotyping of plant cell wall mutants by enzymatic oligosaccharide fingerprinting. Plant Physiology. 130 (4), 1754-1754 (2002).
- Obel, N. Microanalysis of plant cell wall polysaccharides. Molecular Plant. 2 (5), 922-922 (2009).
- Mouille, G. Quantitative trait loci analysis of primary cell wall composition in Arabidopsis. Plant Physiology. 141 (3), 1035-1035 (2006).
- Bauer, S. Development and application of a suite of polysaccharide-degrading enzymes for analyzing plant cell walls. PNAS. 103 (30), 11417-11417 (2006).
- Pauly, M. A xyloglucan-specific endo-beta-1,4-glucanase from Aspergillus aculeatus: expression cloning in yeast, purification and characterization of the recombinant enzyme. Glycobiology. 9 (1), 93-93 (1999).
- Aboughe-Angone, S. Cell wall carbohydrates from fruit pulp of Argania spinosa: structural analysis of pectin and xyloglucan polysaccharides. Carbohydr Res. 343 (1), 67-67 (2008).
- Brown, D. M. Comparison of five xylan synthesis mutants reveals new insight into the mechanisms of xylan synthesis. Plant Journal. 52 (6), 1154-1154 (2007).
- Lee, C. The PARVUS gene is expressed in cells undergoing secondary wall thickening and is essential for glucuronoxylan biosynthesis. Plant Cell Physiol. 48 (12), 1659-1659 (2007).
- Obel, N. Pectin may hinder the unfolding of xyloglucan chains during cell deformation: implications of the mechanical performance of Arabidopsis hypocotyls with pectin alterations. Molecular Plant . , (2009).
- Cavalier, D. M., Keegstra, K. Two xyloglucan xylosyltransferases catalyze the addition of multiple xylosyl residues to cellohexaose. Journal of Biological Chemistry. 281 (45), 34197-34197 (2006).
- Leonard, R. Identification of an Arabidopsis gene encoding a GH95 alpha1,2-fucosidase active on xyloglucan oligo- and polysaccharides. Phytochemistry. 69 (10), 1983-1983 (2008).
- Cavalier, D. M., Keegstra, K. Two xyloglucan xylosyltransferases catalyze the addition of multiple xylosyl residues to cellohexaose. Journal of Biological Chemistry. 281 (45), 34197-34197 (2006).
- Lee, C. H. The irregular xylem9 mutant is deficient in xylan xylosyltransferase activity. Plant and Cell Physiology. 48 (11), 1624-1624 (2007).
- Iglesias, N. Apoplastic glycosidases active against xyloglucan oligosaccharides of Arabidopsis thaliana. Plant and Cell Physiology. 47 (1), 55-55 (2006).
- Fujino, T., Sone, Y., Mitsuishi, Y., Itoh, T. Characterization of cross-links between cellulose microfibrils, and their occurrence during elongation growth in pea epicotyl. Plant Cell Physiol. 41 (4), 486-486 (2000).
- Vanzin, G. F. The mur2 mutant of Arabidopsis thaliana lacks fucosylated xyloglucan because of a lesion in fucosyltransferase AtFUT1. PNAS. 99 (5), 3340-3340 (2002).
- Cavalier, D. M. Disrupting two Arabidopsis thaliana xylosyltransferase genes results in plants deficient in xyloglucan, a major primary cell wall component. Plant Cell. 20 (6), 1519-1519 (2008).
- Hilz, H. A comparison of liquid chromatography, capillary electrophoresis, and mass spectrometry methods to determine xyloglucan structures in black currants. Journal of Chromatography A. 1133 (1-2), 275-275 (2006).
- Pauly, M. Effects of the mur1 mutation on xyloglucans produced by suspension-cultured Arabidopsis thaliana cells. Planta. 214 (1), 67-67 (2001).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten