Method Article
Visualisierung von rekombinanten DNA-und Protein-Komplexe mit Atomic Force Microscopy
In diesem Artikel
Zusammenfassung
A-Tapping-Modus Rasterkraftmikroskop (AFM)-Methode zur Visualisierung von Plasmid-DNA, cytoplasmatische Proteine und DNA-Protein-Komplexen beschrieben. Das Verfahren umfasst das alternative Ansätze zur Vorbereitung der Proben für AFM folgenden biochemischen Manipulation. DNA mit spezifischen Protein interagiert Regionen sind in der Nähe-physiologischen Puffer Bedingungen beobachtet.
Zusammenfassung
Atomic Force Microscopy (AFM) ermöglicht die Visualisierung der einzelnen Proteine, DNA-Moleküle, Protein-Protein-Komplexe, und DNA-Protein-Komplexen. Am Ende des Mikroskops Freischwinger ist ein Nano-Sonde, die Traversen Bildbereiche von Nanometern bis Mikrometern, die Messung der Höhe von Makromolekülen ruht auf der Substratoberfläche zu einem bestimmten Zeitpunkt. Elektrostatische Kräfte verursachen Proteine, Lipide und Nukleinsäuren zu locker an das Substrat in zufälligen Orientierungen legen und erlauben Bildgebung. Die generierten Daten ähneln einer topographischen Karte, wo die Makromoleküle als dreidimensionale Teilchen diskrete Größen (Abbildung 1) 1,2 lösen. Tapping-Modus AFM beinhaltet die wiederholte Schwingung des Cantilevers, die Bildgebung des relativ weichen Biomaterialien wie DNA und Proteine ermöglicht. Einer der bemerkenswertesten Vorteile von AFM gegenüber anderen nanoskaligen Mikroskopie-Techniken ist die relative Anpassbarkeit an individuelle Proteine und makromolekulare Komplexe in wässrigen Puffern, darunter nahezu physiologischen gepufferten Bedingungen, in Echtzeit und ohne Flecken oder Beschichtung der Probe, die abgebildet werden zu visualisieren.
Die hier vorgestellte Methode beschreibt die Darstellung von DNA und einer immunoadsorbed Transkriptionsfaktor (dh den Glucocorticoid-Rezeptor, GR) in gepufferter Lösung (Abbildung 2). Immunoadsorbed Proteinen und Protein-Komplexe können aus den immunoadsorbing Antikörper-Bead-Pellet durch den Wettbewerb mit dem Antikörper-Epitop und dann abgebildet (Abbildung 2A) getrennt werden. Dies ermöglicht für die biochemische Manipulation von Biomolekülen von Interesse vor Bildgebung. Nach der Reinigung kann die DNA und Proteinen gemischt und die resultierende Interaktion Komplex kann auch abgebildet werden. Die Bindung von DNA auf Glimmer erfordert ein zweiwertiges Kation 3, wie Ni 2 + oder Mg 2 +, die Probenpuffer hinzugefügt werden noch werden halten Protein-Aktivität. Mit einem ähnlichen Ansatz hat AFM verwendet worden, um einzelne Enzyme, darunter RNA-Polymerase 4 und ein Reparatur-Enzym 5, gebunden, einzelne DNA-Stränge zu visualisieren. Diese Experimente liefern wichtige Einblicke in die Protein-Protein-und DNA-Protein-Interaktionen biophysikalischen statt auf der molekularen Ebene. Imaging einzelnen makromolekularen Teilchen mit AFM kann nützlich sein für die Bestimmung Teilchen Homogenität und zur Ermittlung der physikalischen Anordnung der Bestandteile der abgebildeten Partikel. Während die vorliegende Methode zur Visualisierung von GR-Chaperon-Protein-Komplexe 1,2 und DNA-Stränge, auf die die GR binden können entwickelt wurde, kann es im Großen und Ganzen zu bildgebenden DNA-und Protein-Proben angewandt werden aus einer Vielzahl von Quellen.
Protokoll
1. Vorbereitung DNA-und Protein-Proben, die abgebildet frei von Verunreinigungen sein
- Isolieren von Biomolekülen zu sein bebildert und in einem geeigneten wässrigen Puffer. Proteine, die abgebildet werden gereinigt kann die Verwendung von Flüssig-Chromatographie 6, gefolgt Immunadsorption durch die Entfernung der Antikörper-und Pellet 1,2 oder Profinity eXact Reinigung und Entfernung tag 7 (Abbildung 2A). Isolieren Sie DNA-Moleküle durch Miniprep Reinigung (Abbildung 2B) abgebildet werden.
- Bestätigen Zusammensetzung und Reinheit von Protein-Proben, die abgebildet mit SDS-PAGE und Western Blot werden. Für die DNA, bestätigen Sequenz und Reinheit durch Ausfüllen eines Restriktionsverdau und Gelelektrophorese.
- Inkubieren Proben in einem AFM Adsorption Puffer, um die Einhaltung der Probe auf Glimmer zu fördern. Für Protein-Proben können 10 mM HEPES-Puffer verwendet werden. Zusätzliche Low-Ionenstärke Salzen oder anderen Cofaktoren können ebenfalls hinzugefügt werden. Für die DNA-Proben umfassen ein zweiwertiges Kation (z. B. Mg 2 + oder Ni 2 +), um die Adsorption der Glimmer-Substrat zu fördern. Ein geeignetes Mg 2 +-haltigem Puffer für DNA ist 10 mM Tris, pH 7,5, 10 mM NaCl, 2 mM MgCl 2 3. Eine Alternative Ni 2 +-Puffer 10 mM HEPES, pH 6,8, 10 mM NiCl 2.
- Verdünnen Sie die Probe auf eine Konzentration von 5 ug / ml in der Adsorption Puffer. Vorsichtig mischen. Auf Eis, während das Mikroskop vorbereitet wird.
2. Montage AFM-Sonde
- Legen Sie die Flüssigkeit Zelle Sondenhalter auf den entsprechenden freitragenden Einbau-Dockingstation.
- Suchen Sie den langen, dicken Cantilevers (bezeichnet als 'B' Freischwinger) der scharfen Nitrid Hebel (SNL) Sonde für die Bildgebung verwendet werden. Sorgfältig übertragen, um die Flüssigkeit Zelle Sondenhalter, sicherzustellen, dass die Spitze aufrecht bleibt. Technischer Hinweis: Besondere Vorsicht bezahlt werden muss, während die Sonde auf der Durchreise sind, und lassen Sie sie von jeder beliebigen Höhe kann zu Schäden oder Zerstörung der Freischwinger oder Spitze. Ein microlever SNL (MSNL) Sonde kann als Alternative zu den SNL-Sonde verwendet werden.
- Entfernen Sie die Sonde Halter aus der Docking-Station und untersuchen die Cantilever unter einem Lichtmikroskop zu gewährleisten, ist es intakt und ordnungsgemäß in die Flüssigkeit Zelle Sondenhalter sitzt.
- Entfernen Sie vorsichtig die AFM Kopf aus dem Instrument Schwalbenschwanz Montage durch Anziehen der Rändelschraube Klemmschraube. Technischer Hinweis: Besondere Vorsicht bezahlt werden muss, während die Manipulation der AFM Kopf, und lassen Sie sie von selbst eine kurze Strecke (z. B. wenn sie nicht richtig in den Schwalbenschwanz Montage sitzend) kann zu erheblichen Schäden.
- Kehren Sie die AFM Kopf und fest drücken Sie die Flüssigkeit Zelle Sondenhalter auf die vier Stifte an der Unterseite des AFM Kopf. Vorsichtig wieder die AFM Kopf in das Gerät Schwalbenschwanz Montage. Lassen Sie die Rändelschraube Klemmschraube an die AFM Kopf in das Gerät zu befestigen.
3. Locating Cantileverspitze, Ausrichten Laser und Einstellen Photodetektor
- Mit dem AFM Instrument Software, suchen Sie die Cantilever-Spitze, indem Sie die Knöpfe der Onboard-Lichtmikroskop. Passen Sie die Beleuchtung des Mikroskops, wenn nötig bis zur Spitze Identifikation zu verbessern. Bringen Sie das Ende des Auslegers Spitze in der Nähe des Fadenkreuzes auf der Geräte-Software angezeigt.
- Bringen Sie die Cantilever-Spitze in den Fokus, indem die oben und unten Pfeile der Optik-Controller an die Software. Verwenden Sie langsam (S) oder medium (M) Geschwindigkeit, während die Anpassung der Optik.
- Richten Sie den Laser auf die Cantilever-Spitze mit dem Laser Einstellknöpfe. Bewegen Sie die Einstellknöpfe, bis der rote Laser in das weiße Ausleuchtung vor Ort. Trace entlang der Ausleger mit einem Zick-Zack-Muster, bis der Laser auf der Cantilever-Spitze positioniert ist. Wenn sie richtig ausgerichtet ist, wird eine starke Reflexion vor Ort in die Frontscheibe des AFM Kopf erscheinen.
- Zentrum der Photodetektor durch Anpassung der Photodetektor Noppen auf der AFM Kopf. Dies wird der Laserpunkt in das Fadenkreuz der Photodetektor Anzeige der Geräte-Software ausrichten. Das beobachtete Signal Summe sollte etwa 4-6 werden.
4. Positioning AFM Kopf-und Tuning-Cantilever
- Legen Sie eine frisch gespaltene Blatt Glimmer an einem Metall-AFM Probe-Disc auf die magnetisierten Probenhalter Spitze des AFM Halteplatte. Dieses Setup wird verwendet, um die AFM Kopf vor der Abbildung der DNA-oder Protein-Probe in Schritt 1 vorbereitet Position sein.
- Technischer Hinweis: Die magnetisierten Probenhalter und flüssige Zelle Sondenhalter sind dicker als andere Probe Befestigungsmechanismen und Sondenhalterungen. Wenn es derzeit keine ausreichenden Abstand zwischen den Glimmer-Probenhalter Montage-und AFM Kopf, bewegen Sie die AFM Kopf hoch, indem Sie den Rücktritt Symbol auf der Geräte-Software mehrere Male.
- Drehen Sie das AFM Halteplatte so die Probe Disc für das erste bildgebende positioniert ist. Sicherstellen, dass die Glimmer-Substrat unter dem AFM Kopf zentriert ist.
- Bewegen Sie die AFM Kopf nach unten in Richtung derOberfläche der Glimmer-Probenteller mit dem Schwerpunkt Oberfläche Steuerung der Geräte-Software. Wählen Sie das Medium (M) Geschwindigkeit auf der Z-Motor, und weiter in Richtung der Oberfläche, bis die AFM Spitze beträgt ca. 2 mm über dem Glimmer. Zu diesem Zeitpunkt, um mit dem langsamen (S) Z Motordrehzahl, um abrupte Kontakt zwischen der Sonde und der Oberfläche (im Folgenden als Absturz der Spitze) zu vermeiden wechseln.
- Fahren Sie mit der Sonde näher an den Glimmersubstrat Oberfläche zu bewegen, bis deutliche Merkmale auf der Glimmer-Oberfläche oder die Spitze Reflexion im Mittelpunkt stehen. Umschalten zwischen Oberfläche und Spitze Reflexion Brennebenen mit dem "Fokus auf:" Symbol aus der Gerätesoftware.
- Wählen Sie die Melodie Symbol aus der Geräte-Software und stellen Sie den Ausleger. Die Resonanzfrequenz der empfohlenen SNL und MSNL Cantileversonden ist 20-60 kHz. Wählen Sie einen 5% peak ausgeglichen. Technischer Hinweis: Ausreichende Cantilever-Tuning ist für eine ausreichende Probe Bildgebung unverzichtbar.
5. Imaging Glimmer Probenoberfläche
- Engage der Sonde auf die Glimmer-Oberfläche. Set ersten Scan Größe bis 10 um und Abtastrate bis 1 Hz. Integral gewinnen und Proportionalverstärkung kann zunächst auf 0,2 und 0,4 eingestellt werden, bzw.. Passen Sie Gewinne als erforderlich, um Spuren haben und verfolgen Scans etwa überlappen. Eine Verringerung der Amplitude Sollwert erhöht sich die Wechselwirkung zwischen der Sonde und der Glimmer und sicherzustellen Engagement.
- Erfassen kompletter Scan Bilder des 10 pm Feld. Abnahme Scanformat bis 5, 1 und 0,5 mu m und Capture abzuschließen Bilder der einzelnen Felder. Passen Gewinne und Abtastrate wie nötig. Die Aufnahme von Bildern der Glimmer-Oberfläche bietet eine Grundlage für den Vergleich der DNA-und Protein-haltigen Proben.
- Lösen Sie die Sonde mit einem einfachen Klick auf das Lösen Symbol auf der Geräte-Software.
6. Imaging von Biomolekülen von Interesse
- Vorsichtig mischen die DNA-oder Protein-Probe in Schritt 1 vorbereitet und bereiten Sie es auf der abgebildeten Glimmer-Oberfläche zu laden. Mix 5 ul der vorbereiteten Probe mit 45 ml frisches Adsorption Puffer (Endprobe Konzentration = 0,5 ug / ml).
- Vorsichtig 50 ul der 0,5 ug / ml Biomolekül-haltigen Lösung direkt auf die Glimmer-Oberfläche. Tun Sie dies, ohne die Sonde oder AFM Kopf und sorgen für die Probelösung (aber nicht Pipettenspitze) in Kontakt mit der Sonde.
- Pause 5 Minuten, damit die Biomoleküle in der Probe auf die Glimmer-Oberfläche haften.
- Am Ende der 5 Minuten, pipet eine zusätzliche 50-100 &mgr; l der Adsorption Puffer in die Flüssigkeit Zelle Sondenhalter darauf achten nicht zu berühren die Sonde, AFM Kopf, oder Glimmer mit der Pipettenspitze. Dies bildet einen Meniskus und damit für AFM-Tapping-Modus Bildgebung in der Flüssigkeit. Abbildung 3 zeigt die endgültige Anordnung der Flüssigkeit Zelle, Freischwinger, Probe, Probenhalterung und Meniskus.
- Danach ist der Photodetektor und richten Sie den Laser auf die Cantilever wie nötig. Technischer Hinweis: Zum Betrachten der Sondenspitze und Laser-Ausrichtung wird durch das Beugungsmuster, indem Probenpuffer in den Sondenhalter verursacht kompliziert. Manuell neu einstellen zu dem Ausleger, wenn nötig.
- Re-greifen die Sonde mit der Gerätesoftware. Einstellen der vertikalen Auslenkung, integrale Verstärkung und Proportionalverstärkung wie nötig. Dies wird den Prozess der Re-Imaging Abschnitt von Glimmer gescannt unmittelbar vor Zugabe Probe und Erfassen von Bildern.
- Erhöhen Scangröße (500 nm-10 um) in Regionen von Interesse zu identifizieren. Zoom-in und reduzieren Abtastrate bis 0,5 Hz bis Bildauflösung zu verbessern
- Sobald Imaging abgeschlossen ist, lösen Sie die Sonde durch die Auswahl zurückzutreten Symbol auf der Geräte-Software mehrere Male. Ausreichend Freiraum zwischen der AFM Spitze und Probe-Disc vor der Demontage des flüssigen Zell-oder Probe-Disc.
- Spülen Sie die Flüssigkeit Zelle Sondenhalter und Probenteller mit destilliertem Wasser, Salz die Bildung von Kristallen als Rest-Adsorption Puffer verdunstet zu verhindern. Dry mit Druckluft.
7. Repräsentative Ergebnisse:
Beispiele für AFM-Aufnahmen sind in Abbildung 4 dargestellt. Mica-Substrat (A) bietet eine molekular flachen Oberfläche, auf die DNA-und Protein adsorbieren kann. Imaging Glimmer vor Biomolekül Proben eine negative Kontrolle und Beurteilung der Bildgebung Lärm. Es bietet auch ein Maß an Gewissheit, dass der Ausleger richtig eingestellt ist und die anschließende Probe Bildgebung erfolgreich sein wird. Doppelsträngige Plasmid-DNA (B, D), mutmaßlich supercoiled, ist leicht durch seine asymmetrische Erscheinungsbild und einheitliche Ablage auf dem zuvor unauffällig Glimmersubstrat identifiziert. Protein-Komplexe von diskreten Partikelgrößen (C, E) sind ebenfalls eindeutig unterscheidbar von den Glimmer-Substrat. Partikelgröße Unterschiede zeigen Probe Heterogenität und kann nützlich sein, auf eine Angleichung Protein-Komplex Stöchiometrie oder biochemische Aktivität. Die allgemeine diagonal Form und die konsequente Ausrichtung der Protein Partikel in Panel C beobachtet ist ein bildgebendes Artefakt, wie Proteine erwartungsgemäß wäre auf dem Glimmer-Substrat orientiert nach dem Zufallsprinzip. Mögliche Ursachen der beobachteten Artefakt ist ein physischer Spitze Anomalie oder AFM in zu schnell von einer Scan-Rate. Während Spitze Faltung (siehe Abbildung 1B) verhindert absolute Längenmessung Berechnungen in der x-und y-Achsen, Höhenmessung (z-Achse) und die relative x-und y-Messungen kann dennoch nützlich für die Abschätzung biophysikalischen Eigenschaften der abgebildeten Biomolekülen.
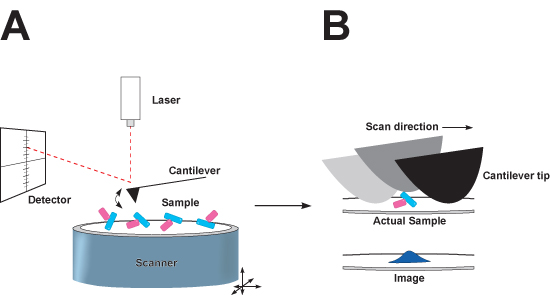
Abbildung 1: Schematische Darstellung der Tapping-Modus Rasterkraftmikroskopie (AFM). A, das Mikroskop. Am Ende des Auslegers ist eine scharfe Spitze, die bis schwingt auf und ab, während sie über die Oberfläche eines Glimmer-Substrat-Scans, um die biomolekularen Komplexen adsorbieren. Da der Scanner bewegt sich in x-und y-Richtung, wird ein Laserstrahl von der Rückseite des Cantilevers auf eine Position Sensitive Photodioden-Detektor reflektiert wird, um die vertikale (z) Abstand zur Spitze bewegt, wie es über die biomolekularen Komplexen auf den sitzenden Pässe Karte Glimmer. B. Verzerrung des Bildes in x-und y-Richtung durch die Spitze Faltung verursacht. Die nominelle Radius eines herkömmlichen Siliciumnitrid-Spitze ist größer als die Partikel, die abgebildet werden, und der Rand der Spitze in Kontakt mit der Probe beim Durchqueren der Oberfläche. Tipp Faltungsergebnisse in der abgebildeten Partikel erscheinen größer in der x-und y-Richtung, nicht aber die z-Richtung. Dieser Effekt kann durch die Verwendung von Spitzen mit einem kleineren nominal Spitzenradius minimiert werden.

Abbildung 2: Illustration der immunoadsorbed Protein-und DNA Probenvorbereitung. A, Veröffentlichung immunoadsorbed GR • Hsp70-Protein-Komplex aus dem monoklonalen Antikörper (mAb)-Protein A-Sepharose (PAS) Pellets. Inkubation des immunopellet mit einem Peptid, das die mAb Epitop erleichtert die Freigabe der GR • Hsp70-Protein-Komplex aus dem Pellet, so dass der Komplex aus dem Überstand und visualisiert durch AFM gesammelt werden. B, erfordert Vorbereitung der DNA für die AFM die Verwendung eines zweiwertigen Kations Adsorption Puffer. Die zweiwertigen Kations erhöht die Affinität der DNA für die Glimmer-Substrat.

Abbildung 3: Final Anordnung der AFM Spitze, flüssige Zelle Sondenhalter, Probe, Probenhalter und Puffer Meniskus. A, darauf zu achten, bei der Hinterlegung Probe und zusätzlichen Puffer auf die Glimmer-Substrat, um körperlichen Kontakt zwischen der Pipettenspitze und AFM Kopf, Flüssigkeit nennen, und Glimmer-Substrat zu vermeiden. B, Vergrößerung von A. Der Puffer Meniskus reicht von der Objektplatte, um die Flüssigkeit Zelle Sondenhalter.
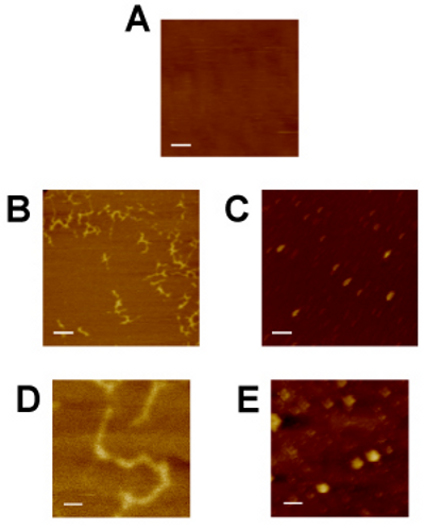
Abbildung 4: Atomic Force Aufnahmen von Glimmer-Substrat (A), 2xGal4-2xGRE-luciferease Plasmid-DNA 8 (B, D) und GR • Hsp70-Protein-Komplexe (C, E) in einer gepufferten Lösung. Mica dient als Kontrolle Bild mit der Visualisierung von DNA und Protein zu vergleichen. GR • Hsp70-Protein-Komplexe wurden durch Immunadsorption von GR mit hsp70 und dann aus dem Antikörper-Bead-Pellet freigesetzt mit einem mAb konkurrierenden Antikörper (dargestellt in Abbildung 2A) grundiert vorbereitet. DNA wurde durch konventionelle Plasmid Miniprep vorbereitet. Panels A, B und C ein Äquivalent Vergrößerung (Maßstab = 200 nm) sind, wie auch Panels D und E (Maßstab = 40 nm).
Diskussion
AFM bietet eine einzigartige mikroskopischen Technik in der Lage Bildgebung einzelnen unbeschichteten Biomolekülen in wässrigen und nahezu physiologischen gepufferten Lösungen in Echtzeit. Dies ermöglicht die Visualisierung der einzelnen Proteine und DNA-Moleküle, sowie Multiproteinkomplexen-und Protein-DNA-Komplexe. Imaging makromolekularen Teilchen mit AFM kann nützlich sein für die Beurteilung der Probe Homogenität und zur Ermittlung der physikalischen Anordnung der Bestandteile der beobachteten Teilchen. Dieser Ansatz der Beobachtung einzelner makromolekulare Partikel können eine sinnvolle Ergänzung sein zu herkömmlichen biochemischen Techniken, wie Immunpräzipitationsassays, Polyacrylamidgelelektrophorese und Size-Exclusion-Säulenchromatographie, die erhebliche summative Daten, die den 10 3 -10 11 einzelnen biomolekularen Komplexen von Interesse anwesend bieten in einer Probe.
Die Erforschung Multiproteinkomplex Stöchiometrie biomolekularen Wechselwirkungen und Cofaktor Anforderungen können alle untersuchten mittels AFM werden. Die hier vorgestellte Methode lässt sich an spezifischen biophysikalischen Fragestellungen von Interesse aufnehmen werden. Zum Beispiel mit einer Flüssigkeit Zelle Sondenhalter der Lage, den Austausch von Puffer, wird es möglich sein, Assay-Cofaktor für Protein-Komplexbildung. DNA-Protein-Baugruppen gebildet und getrennt werden, während sie abgebildet in near-real-time und sogar. DNA kann mit spezifischen Sequenzen von Interesse (zB ein mutmaßliches Response-Elementen oder neuartige Protein-Bindung Sequenz), mit der Hypothese bindendes Protein gemischt erzeugt werden, und abgebildet, einen direkten Nachweis von intermolekularen Wechselwirkungen liefern.
Tapping Mode-AFM-Bildgebung ist wesentlich weniger kompliziert, wenn trockene Proben statt Proben in wässrigen Lösungen, und lineare DNA steht und geschlossenen Kreis DNA-Plasmide haben beide auf diese Weise 3,9 abgebildet worden sind verwendet. Die hier vorgestellte Methode nutzt Proben in gepufferten Lösungen, um eine physiologische Bildgebung Umfeld zu schaffen. Andere bemerkenswerte Methoden der Bildgebung Proteine sollten ebenfalls berücksichtigt werden, einschließlich Nahfeld Infrarot-Mikroskopie 10. Die komplementäre Nutzung der Immunadsorption in Gemahlin mit AFM bietet die Möglichkeit, eine Vielzahl von Protein-Komplexe, mit gut etablierten biochemischen Techniken, und dann visualisieren folgende Entlassung aus dem immunoadsorbing Antikörper-Bead-Pellet. Zum Beispiel hat immunoadsorbed GR für seine Assoziation mit der molekularen Chaperon-Protein hsp70 1 sowie die Dynein Motorprotein untersucht 2 mit diesem Ansatz, um komplexe Größe und Stöchiometrien zu schätzen. Partikelgröße und biophysikalischen Eigenschaften (zB Festigkeit) haben sich als nützlich erwiesen bei der Ermittlung der Identität der Biomoleküle in AFM abgebildet Proben 11,12 beobachtet. Es ist auch möglich, Biomolekül Identität durch die Zugabe von einem interagierenden Biomolekül (zB ein Ligand oder monoklonale Antikörper), die zu seinem Ziel und verursachen eine Teilchengröße erhöhen binden würde, wenn das Ziel-Biomolekül vorhanden ist zu bestätigen.
Offenlegungen
Danksagungen
Diese Arbeit wurde vom National Institutes of Health Grants GM086822 finanziert. Die Autoren bedanken sich bei Dr. danken. Alec Pakhomov & Paul Wallace (Univ. of Washington Nanotechnologie User Facility (NTUF)) und Andrea Slade (Bruker AXS) für ihre technische Unterstützung durch Experten. DNA AFM wurde an der Univ durchgeführt. Washington NTUF, ein Mitglied der National Nanotechnology Infrastructure Network. Die 2xGal4-2xGRE-luciferease Plasmid wurde freundlicherweise von der Arbeitsgruppe von Dr. Keith Yamamoto (Univ. of California, San Francisco) zur Verfügung gestellt.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | |
|---|---|---|---|
| Dimension 3100 | Bruker | Dimension 3100 | |
| Dimension Fluidzelle | Bruker | DTFML-DD-HE | |
| Sharp-Nitrid-Hebel (SNL) Siliziumnitrid AFM-Sonde | Bruker | SNL-10 | |
| Microlever scharfe Nitrid Hebel (MSNL) Siliziumnitrid AFM-Sonde | Bruker | MSNL-10 | |
| NanoScope AFM Gerätesoftware | Bruker | 004-132-000 | |
| Metall AFM Objektplatten | Ted Pella | 16208 | |
| Grade V1 Mica Discs, 12 mm | Ted Pella | 50-12 |
Referenzen
- Murphy, P. J. M. Visualization and mechanism of assembly of a glucocorticoid receptorhsp70 complex that is primed for subsequent Hsp90-dependent opening of the steroid binding cleft. J Biol Chem. 278, 34764-34773 (2003).
- Harrell, J. M. Evidence for glucocorticoid receptor transport on microtubules by dynein. J Biol Chem. 279, 54647-54654 (2004).
- Pastre, D. Adsorption of DNA to mica mediated by divalent counterions: a theoretical and experimental study. Biophys J. 85, 2507-2518 (2003).
- Guthold, M. Direct observation of one-dimensional diffusion and transcription by Escherichia coli RNA polymerase. Biophys J. 77, 2284-2294 (1999).
- Petrucco, S., Volpi, G., Bolchi, A., Rivetti, C., Ottonello, S. A nick-sensing DNA 3'-repair enzyme from Arabidopsis. J Biol Chem. 277, 23675-23683 (2002).
- Murphy, P. J. M., Morishima, Y., Kovacs, J. J., Yao, T. P., Pratt, W. B. Regulation of the dynamics of hsp90 action on the glucocorticoid receptor by acetylation/deacetylation of the chaperone. J Biol Chem. 280, 33792-33799 (2005).
- Ruan, B., Fisher, K. E., Alexander, P. A., Doroshko, V., Bryan, P. N. Engineering subtilisin into a fluoride-triggered processing protease useful for one-step protein purification. Biochemistry. 43, 14539-14546 (2004).
- Meijsing, S. H., Elbi, C., Luecke, H. F., Hager, G. L., Yamamoto, K. R. The ligand binding domain controls glucocorticoid receptor dynamics independent of ligand release. Mol Cell Biol. 27, 2442-2451 (2007).
- Shen, X. C. A simple and effective sample preparation method for atomic force microscopy visualization of individual DNA molecules in situ. Mol Biol Rep. 38, 965-969 (2010).
- Paulite, M., Fakhraai, Z., Akhremitchev, B. B., Mueller, K., Walker, G. C. Assembly, tuning and use of an apertureless near field infrared microscope for protein imaging. J Vis Exp. , (2009).
- Cretu, A., Castagnino, P., Assoian, R. Studying the effects of matrix stiffness on cellular function using acrylamide-based hydrogels. J Vis Exp. , (2010).
- Brunger, A. T., Weninger, K., Bowen, M., Chu, S. Single-molecule studies of the neuronal SNARE fusion machinery. Annu Rev Biochem. 78, 903-928 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten