Method Article
Visualização do DNA recombinante e Complexos de Proteínas Usando Microscopia de Força Atômica
Neste Artigo
Resumo
Um modo de tocar microscópio de força atômica método (AFM) para a visualização de DNA plasmídeo, proteínas citoplasmáticas e proteína-DNA complexos é descrito. O método inclui abordagens alternativas para a preparação de amostras para AFM imagem após a manipulação bioquímica. DNA contendo regiões específicas interagindo proteínas são observados em condições quase fisiológica buffer.
Resumo
Microscopia de força atômica (AFM) permite a visualização de proteínas individuais, as moléculas de DNA, complexos proteína-proteína e DNA-proteína complexos. No final do cantilever do microscópio é uma sonda escala nanométrica, que atravessa áreas de imagem que variam de nanômetros a micrômetros, medir a elevação de macromoléculas descansando na superfície do substrato, em qualquer ponto. Forças eletrostáticas causa proteínas, lipídios e ácidos nucléicos para frouxamente anexar ao substrato em orientações aleatórias e permitir imagem. Os dados gerados se assemelham a um mapa topográfico, onde as macromoléculas como resolver tridimensional partículas de tamanhos distintos (Figura 1) 1,2. Tocando modo AFM envolve a oscilação repetida do cantilever, que permite imagens de biomateriais relativamente macio, tais como DNA e proteínas. Um dos benefícios notáveis da AFM em relação a outras técnicas de microscopia em nanoescala é a sua adaptabilidade em relação ao visualizar proteínas individuais e complexos macromoleculares em buffers aquosa, incluindo o quase fisiológica tamponada condições, em tempo real, e sem coloração ou revestimento da amostra a ser trabalhada.
O método aqui apresentado descreve a imagem de DNA e um fator de transcrição imunoadsorvido (isto é o receptor glicocorticóide, GR) em solução tampão (Figura 2). Imunoadsorvido proteínas e complexos de proteína pode ser separada da immunoadsorbing anticorpos talão de pelotas pela competição com o anticorpo epítopo e depois fotografada (Figura 2A). Isso permite a manipulação bioquímica do biomoléculas de interesse antes da imagem. Uma vez purificado, DNA e proteínas podem ser misturados e do complexo interagindo resultante pode ser trabalhada também. Ligação do DNA a mica requer um cátion divalente 3, tais como Ni 2 + ou Mg 2 +, que pode ser adicionado à amostra buffers ainda manter a atividade da proteína. Utilizando uma abordagem similar, AFM tem sido utilizado para visualizar diferentes enzimas, incluindo RNA polimerase 4 e uma enzima de reparo 5, ligado a cadeias de DNA individual. Esses experimentos fornecem uma visão significativa para a proteína-proteína e interações DNA-proteína biofísicos que ocorrem no nível molecular. Partículas macromolecular imagens individuais com AFM pode ser útil para determinar a homogeneidade de partículas e para a identificação do arranjo físico dos componentes constituintes das partículas com imagens. Enquanto o método foi desenvolvido para visualização dos complexos GR-chaperone da proteína 1,2 e DNA vertentes a que o GR pode ligar, ele pode ser aplicado de forma ampla para DNA de imagens e amostras de proteínas a partir de uma variedade de fontes.
Protocolo
1. Preparação de amostras de DNA e proteínas a ser trabalhada livre de contaminantes
- Biomoléculas isolar a ser trabalhada e coloque em um tampão adequado aquosa. Proteínas a ser trabalhada pode ser purificado por cromatografia líquida de 6 de imunoadsorção seguida da remoção de anticorpos e pellet 1,2, ou purificação Profinity eXact e remoção tag 7 (Figura 2A). Isolar moléculas de DNA a ser trabalhada por miniprep purificação (Figura 2B).
- Confirmar composição e pureza das amostras de proteína para ser fotografada usando SDS-PAGE e Western blotting. Para DNA, confirme seqüência e pureza, completando uma restrição digestão e eletroforese em gel de agarose.
- Incubar as amostras em um tampão de adsorção AFM para promover a adesão da amostra a mica. Para amostras de proteínas, 10 mM tampão HEPES pode ser usado. Adicionais de sais de baixa força iônica ou co-fatores podem ser adicionados outros também. Para amostras de DNA, incluem um cátion bivalente (por exemplo, Mg 2 + ou Ni 2 +) para promover a adsorção ao substrato mica. A Mg 2 + adequada, contendo tampão de DNA é de 10 mM Tris, pH 7,5, 10 mM NaCl, 2 mM MgCl 2 3. Ni uma alternativa buffer de 2 + é de 10 mM HEPES, pH 6.8, 10 mM NiCl 2.
- Diluir a amostra a uma concentração de 5 mg / ml em tampão de adsorção. Misturar suavemente. Loja no gelo, enquanto o microscópio está sendo preparada.
2. Montagem AFM sonda
- Coloque o suporte da sonda líquido da célula para a correspondente estação de acoplamento cantilever instalação.
- Localize o cantilever, longos e grossos (referido como o cantilever 'B') da sonda de nitreto de alavanca afiada (SNL) a ser utilizado para geração de imagens. Cuidadosamente transferi-lo para o titular líquido celular sonda, assegurando que a ponta permanece em pé. Nota técnica: Extremo cuidado deve ser pago, enquanto a sonda está em trânsito, como deixá-la cair de qualquer altura pode danificar ou destruir o cantilever ou ponta. A microlever SNL sonda (MSNL) pode ser usado como uma alternativa à sonda SNL.
- Remova o suporte da sonda da estação de ancoragem e examinar o cantilever sob um microscópio de luz para assegurar que ele está intacto e devidamente sentado no porta-líquido celular sonda.
- Remova cuidadosamente a cabeça AFM do conjunto se encaixam instrumento, apertando o parafuso de fixação serrilhado cabeça. Nota técnica: Extremo cuidado deve ser paga enquanto manipulando a cabeça AFM, como deixá-la cair até mesmo a uma curta distância (por exemplo, se não estão encaixados corretamente na montagem se encaixam) pode causar danos substanciais.
- Inverter a cabeça AFM e empurre o suporte da sonda líquido da célula para os quatro pinos na base da cabeça AFM. Voltar cuidadosamente a cabeça AFM no conjunto se encaixam instrumento. Solte o parafuso de fixação da cabeça serrilhada para prender a cabeça AFM no instrumento.
3. Localizando ponta cantilever, alinhando laser, e ajustando fotodetector
- Usando o software do aparelho AFM, localize a ponta cantilever movendo os botões do microscópio de luz a bordo. Ajustar a iluminação do microscópio, se necessário para melhorar a identificação ponta. Trazer o fim da ponta do cantilever em estreita proximidade da mira exibido no software do aparelho.
- Traga a ponta cantilever em foco, ajustando as setas para cima e para baixo do controlador de ótica sobre o software. Use lento (S) ou velocidade média (M) enquanto ajusta o sistema ótico.
- Alinhar o laser na ponta cantilever usando os botões de ajuste laser. Mova os botões de ajuste até o ponto de laser vermelho está dentro do local de iluminação branca. Traço ao longo do braço cantilever usando um padrão zig-zag até que o laser é posicionado na ponta cantilever. Quando devidamente alinhados, um ponto forte reflexo aparecerá na janela da frente da cabeça AFM.
- Centro do fotodetector, ajustando os botões fotodetector na cabeça AFM. Isso irá alinhar o ponto de laser na mira do visor fotodetector do software do aparelho. A soma do sinal observado deve ser de aproximadamente 4-6.
4. Posicionamento da cabeça e AFM cantilever ajuste
- Coloque uma folha recém-clivada de mica ligado a um disco de metal espécime AFM no porta-amostras magnetizadas em cima do suporte da placa AFM. Esta configuração será usada para posicionar a cabeça antes de AFM imagem da amostra de DNA ou proteína preparada na Etapa 1.
- Nota técnica: O porta-amostras magnetizadas e suporte da sonda líquidos celulares são mais espessas do que outra amostra de fixação de mecanismos e os detentores da sonda. Se existe actualmente depuração insuficiente entre o conjunto do suporte de amostra-mica e cabeça AFM, mover a cabeça até AFM, selecionando o ícone retirar no software do aparelho várias vezes.
- Gire o suporte da placa AFM para o disco de amostra é posicionada para a imagem inicial. Garantir o substrato de mica está centrada sob a cabeça AFM.
- Mova a cabeça para baixo em direção a AFMsuperfície do disco mica espécimes usando o controle de superfície foco do software do aparelho. Selecione a velocidade média (M) no motor Z, e continuar em direção à superfície até que a cabeça AFM é de aproximadamente 2 mm acima da mica. Neste momento, passar a usar o lento (S) Z velocidade do motor, a fim de evitar o contato brusco entre a sonda ea superfície (referido como bater a ponta).
- Proceder para mover a sonda mais próxima à superfície do substrato de mica até características distintas na superfície mica ou o reflexo ponta estão em foco. Alternar entre a superfície e aviões ponta reflexão focal usando a "concentrar-se:" ícone do software do aparelho.
- Selecionar o ícone de sintonia do software do aparelho e ajustar o cantilever. A frequência de ressonância do SNL recomendado e sondas cantilever MSNL é kHz 20-60. Selecione um pico de 5% offset. Nota técnica: tuning cantilever adequada é essencial para a imagem de amostra adequado.
5. Imagens da superfície da amostra mica
- Envolver a sonda sobre a superfície mica. Defina o tamanho da verificação inicial a 10 mm e taxa de varredura de 1 Hz. Ganho integral e ganho proporcional pode ser inicialmente definido como 0,2 e 0,4, respectivamente. Ajustar os ganhos, se necessário, a fim de ter traço e scans refazer cerca de sobreposição. Diminuindo a amplitude setpoint irá aumentar a interação entre a sonda ea mica e garantir engajamento.
- Capturar imagens varredura completa do campo M 10. Diminuir o tamanho de digitalização a 5, 1 e 0,5 micron e capturar imagens completas de cada campo. Ajustar os ganhos e velocidade de varredura, se necessário. Captura de imagens da superfície de mica fornece uma base para comparação com o DNA e proteínas contendo amostras.
- Desengatar a sonda com um único clique no ícone desengatar no software do aparelho.
6. Biomoléculas de interesse de imagem
- Delicadamente misturar o DNA ou proteínas da amostra preparada na Etapa 1 e se preparar para carregá-lo sobre a superfície de mica trabalhada. Mistura de 5 mL de amostra preparada com 45 mL de tampão de adsorção fresco (concentração da amostra final = 0,5 mg / ml).
- Cuidadosamente adicione 50 ul do 0,5 mg / solução biomolécula contendo ml diretamente na superfície mica. Fazer isso sem mover a cabeça da sonda ou AFM, e assegurar a solução da amostra (mas não ponta da pipeta) faz contato com a sonda.
- Pausa de 5 minutos para permitir que as biomoléculas na amostra para aderir à superfície mica.
- Na conclusão de 5 minutos, uma pipeta mL 50-100 adicionais de tampão de adsorção no suporte líquido celular sonda tendo o cuidado de não tocar na sonda, AFM cabeça, ou mica com a ponta da pipeta. Isto irá formar um menisco e permitir a AFM tocando imagem em modo de fluido. Figura 3 mostra o arranjo final da célula de fluidos, cantilever, amostra, titular da amostra, e menisco.
- Reajustar a célula fotoeléctrica e realinhar a laser para o cantilever se necessário. Nota técnica: ver a ponta da sonda e alinhamento a laser é complicado devido ao padrão de difração causada pela adição de tampão de amostra para o suporte da sonda. Manualmente sintonizar o cantilever, se necessário.
- Voltar a envolver a sonda usando o software do aparelho. Ajuste de deflexão vertical, ganho integral e ganho proporcional, conforme necessário. Isto irá iniciar o processo de re-imaging a seção de mica digitalizados imediatamente antes da adição da amostra e capturar imagens.
- Aumentar o tamanho do scan (500 nm-10 mm) para identificar regiões de interesse. Zoom-in e reduzir a taxa de varredura de 0,5 Hz para melhorar a resolução da imagem
- Uma vez que imagens é completa, desengatar a sonda selecionando retirar ícone no software do aparelho várias vezes. Garantir espaço suficiente entre a cabeça eo disco AFM espécime antes da desmontagem da célula de líquido ou disco espécime.
- Enxaguar o titular sonda líquido celular e disco de amostra com água destilada para evitar a formação de cristais de sal se evapora como tampão residual de adsorção. Seco com ar comprimido.
7. Resultados representativos:
Exemplos de imagens AFM são apresentados na Figura 4. Mica substrato (A) fornece uma superfície plana sobre a qual molecularmente DNA e proteínas podem absorver. Mica de imagem antes de amostras biomolécula fornece um controle negativo e avaliação do ruído de imagem. Ele também oferece um nível de garantia de que o cantilever está devidamente afinada e de imagem de amostra subseqüente será bem sucedido. Double-stranded DNA plasmídeo (B, D), presuntivamente superenrolado, é facilmente identificado por sua aparência assimétrica e uniforme depositar sobre o substrato de mica anteriormente normal. Complexos de proteínas de tamanhos de partículas discretas (C, E) também são distinguíveis exclusivamente a partir do substrato mica. Diferenças de tamanho de partículas indicam heterogeneidade da amostra, e pode ser útil para aproximar estequiometria proteínas complexas ou atividade bioquímica. A forma geral diagonal e orientação consistente do propartículas de proteína observado no painel C é um artefato de imagem, como as proteínas se expectedly ser orientado sobre o substrato de mica de uma forma aleatória. Possíveis causas do artefato observado é uma anormalidade ou física ponta AFM imagens em rápida demais de uma taxa de varredura. Enquanto convolução ponta (ilustrada na Figura 1B) impede que os cálculos de medição absoluta comprimento em eixos x e y, medida da altura (eixo z) e x relativa e medições y pode ser, no entanto útil para estimar propriedades biofísicas das biomoléculas com imagens.
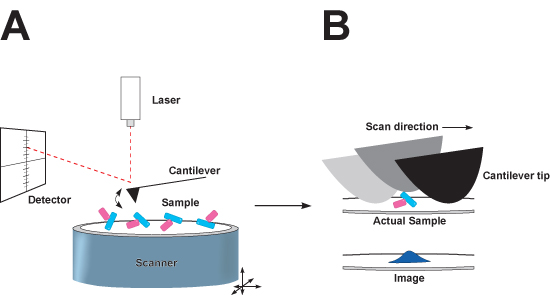
Figura 1: Apresentação esquemática do modo de tocar microscopia de força atômica (AFM). A, o microscópio. No final do cantilever é uma ponta afiada que oscila para cima e para baixo, uma vez que faz a varredura sobre a superfície de um substrato de mica a que adsorvem complexos biomolecular. Como o scanner se move nas direções x e y, um feixe de laser é refletida na parte de trás do cantilever em uma posição sensível detector fotodiodo para mapear a distância vertical (z) a ponta se move quando ele passa sobre os complexos biomolecular sentado no mica. B. Distorção da imagem nas direções x e y causada pela convolução da ponta. O raio nominal de uma ponta convencional nitreto de silício é maior do que as partículas a ser trabalhada, ea borda dos contatos ponta da amostra, uma vez que atravessa a superfície. Dica resultados convolução nas partículas com imagens que aparecem maiores na direções x e y, mas não a direção z. Este efeito pode ser minimizado através da utilização de pontas com um raio menor ponta nominal.

Figura 2: Ilustração de proteína imunoadsorvido e preparação de amostras de DNA. A Release, de GR imunoadsorvido • complexo de proteínas hsp70 do anticorpo monoclonal (mAb)-proteína A-Sepharose (PAS) da pelota. Incubando a immunopellet com um peptídeo contendo o epítopo mAb vai facilitar a liberação do GR • complexo de proteínas hsp70 do pellet, permitindo que o complexo para ser coletado do sobrenadante e visualizados por AFM. B, Preparação de DNA para AFM imagens requer o uso de um cátion divalente adsorção buffer. O cátion divalente aumenta a afinidade do DNA para o substrato de mica.

Figura 3: Disposição Final da cabeça AFM e suporte para sonda líquido celular, amostra, porta-espécime, e menisco buffer. A Care, devem ser tomados no momento do depósito de amostra e tampão adicionais sobre o substrato de mica para evitar o contato físico entre a ponta da pipeta e cabeça AFM, ligue para líquido, e substrato mica. B, Ampliação de A. O menisco tampão se estende a partir do disco de amostra para o titular líquido celular sonda.
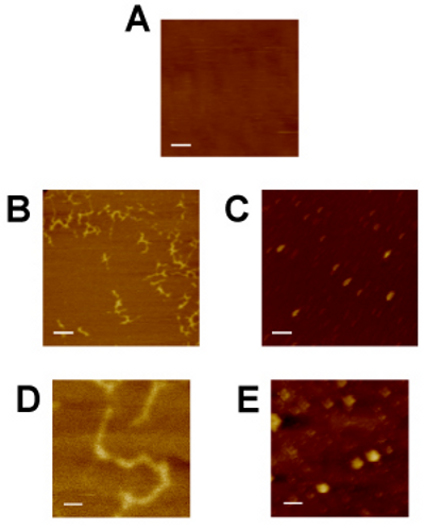
Figura 4: Micrografias de força atômica do substrato de mica (A), 2xGal4-2xGRE-luciferease DNA plasmídeo 8 (B, D) e GR • complexos de proteína hsp70 (C, E) em solução tampão. Mica serve como um controle de imagem para comparar com as visualizações de DNA e proteínas. GR • complexos de proteína hsp70 foram preparados por imunoadsorção de GR preparado com hsp70 e em seguida liberado a partir do pellet-anticorpo talão usando um anticorpo monoclonal concorrentes (ilustrada na Figura 2A). DNA foi preparado pela miniprep plasmídeo convencional. Painéis A, B e C são de uma ampliação equivalente (barra de escala = 200 nm), assim como os painéis D e E (barra de escala = 40 nm).
Discussão
AFM fornece uma técnica única microscópicas capazes de imagem individuais revestidos biomoléculas em solução aquosa e quase fisiológica soluções tamponadas em tempo real. Isto permite a visualização de proteínas e moléculas de DNA, bem como complexos multiprotein e proteína DNA-complexos. Partículas macromolecular de imagem com AFM pode ser útil para avaliar a homogeneidade da amostra e identificar o arranjo físico dos componentes constituintes das partículas observadas. Esta abordagem de observar partículas macromolecular indivíduo pode ser um complemento útil ao convencional técnicas bioquímicas, tais como ensaios de imunoprecipitação, eletroforese em gel de poliacrilamida e cromatografia tamanho da coluna de exclusão, que fornecem dados significativos sumativa representando o 10 3 -10 11 complexos biomolecular individuais de interesse presentes em uma amostra.
Investigação em estequiometria multiprotein complexos, interações biomoleculares e requisitos cofactor podem ser investigadas utilizando AFM. O método aqui apresentado pode ser adaptado para acomodar questões específicas de interesse biofísico. Por exemplo, utilizando um suporte da sonda líquido celular capazes de trocar de buffer, é possível aos requisitos do ensaio cofator para a formação de proteínas complexas. DNA-proteína assembléias podem ser formadas e dissociado quase em tempo real e até mesmo ao ser fotografada. DNA podem ser gerados com seqüências específicas de interesse (por exemplo, elementos de resposta presuntivo ou seqüência romance binding protein), misturado com a proteína a hipótese de ligação, e fotografada para fornecer evidências diretas de interações intermoleculares.
Tocando modo AFM é substancialmente menos complicado se as amostras secas são utilizadas em vez de amostras em soluções aquosas, e está DNA linear e plasmídeos fechado círculo de DNA têm sido fotografados tanto desta forma 3,9. O método aqui apresentado utiliza amostras em soluções tamponadas, a fim de proporcionar um ambiente de imagem mais fisiológica. Outros métodos de imagem notável de proteínas também devem ser considerados, incluindo perto de microscopia de campo infravermelho 10. A utilização complementar de imunoadsorção em consórcio com AFM fornece uma oportunidade para preparar uma miríade de complexos de proteínas, utilizando técnicas bem estabelecidas bioquímicos e, em seguida, visualizá-los após a libertação do immunoadsorbing anticorpos talão pellet. Por exemplo, GR imunoadsorvido foi ensaiada por sua associação com a proteína chaperone molecular hsp70 1, bem como a proteína dineína motor 2 usando essa abordagem, a fim de estimar o tamanho complexo e estequiometrias. Tamanho das partículas e características biofísicas (rigidez, por exemplo) têm sido úteis em determinar a identidade do biomoléculas observada em amostras de AFM imaged 11,12. Também é possível confirmar a identidade biomolécula pela adição de uma biomolécula interação (por exemplo, um ligante ou um anticorpo monoclonal), que se ligam ao seu alvo e causar um aumento de tamanho de partícula, se a biomolécula alvo está presente.
Divulgações
Agradecimentos
Este trabalho foi financiado pelo National Institutes of Health Grant GM086822. Os autores gostariam de agradecer as Dras. Alec Pakhomov & Paul Wallace (Univ. de Washington Facility Usuário Nanotecnologia (NTUF)) e Andrea Slade (Bruker AXS) para a sua assistência técnica especializada. DNA AFM foi realizado na Univ. de Washington NTUF, membro da Rede Nacional de Nanotecnologia Infra-estrutura. O plasmídeo 2xGal4-2xGRE-luciferease foi gentilmente cedido pelo laboratório do Dr. Keith Yamamoto (Univ. of California, San Francisco).
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | |
|---|---|---|---|
| Dimension 3100 | Bruker | Dimension 3100 | |
| Celular Fluid dimensão | Bruker | DTFML-DD-HE | |
| Alavanca de nitreto de Sharp (SNL) de nitreto de silício AFM sonda | Bruker | SNL-10 | |
| Microlever afiada nitreto de alavanca (MSNL) de nitreto de silício AFM sonda | Bruker | MSNL-10 | |
| Nanoscope software instrumento AFM | Bruker | 004-132-000 | |
| Espécime de metal discos AFM | Ted Pella | 16208 | |
| Grade V1 Discos Mica, 12 mm | Ted Pella | 50-12 |
Referências
- Murphy, P. J. M. Visualization and mechanism of assembly of a glucocorticoid receptorhsp70 complex that is primed for subsequent Hsp90-dependent opening of the steroid binding cleft. J Biol Chem. 278, 34764-34773 (2003).
- Harrell, J. M. Evidence for glucocorticoid receptor transport on microtubules by dynein. J Biol Chem. 279, 54647-54654 (2004).
- Pastre, D. Adsorption of DNA to mica mediated by divalent counterions: a theoretical and experimental study. Biophys J. 85, 2507-2518 (2003).
- Guthold, M. Direct observation of one-dimensional diffusion and transcription by Escherichia coli RNA polymerase. Biophys J. 77, 2284-2294 (1999).
- Petrucco, S., Volpi, G., Bolchi, A., Rivetti, C., Ottonello, S. A nick-sensing DNA 3'-repair enzyme from Arabidopsis. J Biol Chem. 277, 23675-23683 (2002).
- Murphy, P. J. M., Morishima, Y., Kovacs, J. J., Yao, T. P., Pratt, W. B. Regulation of the dynamics of hsp90 action on the glucocorticoid receptor by acetylation/deacetylation of the chaperone. J Biol Chem. 280, 33792-33799 (2005).
- Ruan, B., Fisher, K. E., Alexander, P. A., Doroshko, V., Bryan, P. N. Engineering subtilisin into a fluoride-triggered processing protease useful for one-step protein purification. Biochemistry. 43, 14539-14546 (2004).
- Meijsing, S. H., Elbi, C., Luecke, H. F., Hager, G. L., Yamamoto, K. R. The ligand binding domain controls glucocorticoid receptor dynamics independent of ligand release. Mol Cell Biol. 27, 2442-2451 (2007).
- Shen, X. C. A simple and effective sample preparation method for atomic force microscopy visualization of individual DNA molecules in situ. Mol Biol Rep. 38, 965-969 (2010).
- Paulite, M., Fakhraai, Z., Akhremitchev, B. B., Mueller, K., Walker, G. C. Assembly, tuning and use of an apertureless near field infrared microscope for protein imaging. J Vis Exp. , (2009).
- Cretu, A., Castagnino, P., Assoian, R. Studying the effects of matrix stiffness on cellular function using acrylamide-based hydrogels. J Vis Exp. , (2010).
- Brunger, A. T., Weninger, K., Bowen, M., Chu, S. Single-molecule studies of the neuronal SNARE fusion machinery. Annu Rev Biochem. 78, 903-928 (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados