Method Article
Visualizzazione del DNA ricombinante e Complessi proteina usando la microscopia a forza atomica
In questo articolo
Riepilogo
Un tapping mode microscopio a forza atomica (AFM), metodo per la visualizzazione di DNA plasmidico, proteine citoplasmatiche, e DNA-proteina complessi è descritta. Il metodo include approcci alternativi per la preparazione di campioni per l'imaging AFM seguenti manipolazione biochimica. Specifiche regioni di DNA che contiene le proteine che interagiscono sono osservati in quasi fisiologico condizioni di buffer.
Abstract
Microscopia a forza atomica (AFM) permette la visualizzazione delle singole proteine, molecole di DNA, proteina-proteina complessi, proteine e DNA-complessi. Sulla fine del cantilever del microscopio è una nano-scala della sonda, che attraversa aree dell'immagine che vanno da nanometri a micrometri, misurando l'elevazione delle macromolecole appoggiata sulla superficie del substrato in un qualsiasi punto. Forze elettrostatiche causa proteine, lipidi e acidi nucleici per fissare liberamente al substrato in orientamenti casuali e permesso di imaging. I dati generati assomigliare ad una mappa topografica, in cui le macromolecole come risolvere tridimensionale particelle di dimensioni discrete (Figura 1) 1,2. Toccando modalità AFM prevede l'oscillazione ripetuta del cantilever, che permette l'imaging di biomateriali relativamente morbido come il DNA e le proteine. Uno dei vantaggi notevoli di AFM rispetto ad altre tecniche di microscopia su scala nanometrica è la sua adattabilità rispetto di visualizzare singole proteine e complessi macromolecolari nei buffer acquosi, comprese le condizioni tamponata quasi fisiologico, in tempo reale, e senza macchie o rivestimento del campione da acquisire.
Il metodo presentato qui descrive l'immagine del DNA e un fattore di trascrizione immunoadsorbed (cioè il recettore dei glucocorticoidi, GR) in soluzione tamponata (Figura 2). Immunoadsorbed proteine e complessi proteici possono essere separati dalla immunoadsorbing anticorpo-tallone pellet dalla concorrenza con l'epitopo anticorpale e poi ripreso (Figura 2A). Questo permette la manipolazione biochimica del biomolecole di interesse prima di imaging. Una volta purificate, il DNA e le proteine possono essere mescolati e il complesso interagire risultante può essere immaginato come bene. Legame del DNA per mica richiede un catione bivalente 3, come Ni 2 + o Mg 2 +, che può essere aggiunto al buffer campione ancora mantenere l'attività della proteina. Utilizzando un approccio simile, AFM è stato utilizzato per visualizzare i singoli enzimi, tra cui la RNA polimerasi 4 e un enzima riparatore del 5, destinato a singoli filamenti di DNA. Questi esperimenti forniscono una visione significativa nella proteina-proteina e DNA-proteina interazioni biofisiche che si svolgono a livello molecolare. Particelle macromolecolari Imaging individuale con AFM può essere utile per determinare omogeneità delle particelle e per identificare la disposizione fisica dei componenti costituenti delle particelle immagine. Mentre l'attuale metodo è stato sviluppato per la visualizzazione di GR-chaperone complessi proteici e 1,2 DNA filoni a cui la GR può legare, può essere attuata in modo ampio al DNA di imaging e campioni di proteine da una varietà di fonti.
Protocollo
1. Preparazione di campioni di DNA e proteine per essere ripreso privo di contaminanti
- Isolare biomolecole di essere ripreso e posto in un buffer adatto acquosa. Le proteine per essere ripreso possono essere purificati mediante cromatografia liquida 6, immunoadsorbimento seguita da rimozione di anticorpi e pellet 1,2, o purificazione Profinity eXact e la rimozione tag 7 (Figura 2A). Isolare molecole di DNA ad essere ripreso dalla purificazione miniprep (Figura 2B).
- Confermare la composizione e la purezza dei campioni di proteine da acquisire mediante SDS-PAGE e Western blotting. Per il DNA, conferma la sequenza e la purezza completando una restrizione digerire ed elettroforesi su gel di agarosio.
- Incubare i campioni in un buffer di adsorbimento AFM per promuovere l'adesione del campione di mica. Per i campioni di proteine, 10 mM tampone HEPES possono essere utilizzati. Ulteriori bassa forza ionica sali o altri cofattori possono essere aggiunti pure. Per i campioni di DNA, includere un catione bivalente (es. Mg 2 + o Ni 2 +) per promuovere l'adsorbimento al substrato di mica. Un Mg 2 + adatto contenente tampone per il DNA è di 10 mM Tris, pH 7,5, 10 mM NaCl, 2 mM MgCl 2 3. Un'alternativa Ni 2 + tampone è di 10 HEPES mM, pH 6,8, 10 mM NiCl 2.
- Diluire il campione ad una concentrazione di 5 mg / ml in tampone di adsorbimento. Mescolare delicatamente. Memorizzare sul ghiaccio mentre il microscopio è in preparazione.
2. Montaggio della sonda AFM
- Mettere il liquido titolare sonda cella sul corrispondente docking station per l'installazione a sbalzo.
- Individuare il lungo, sbalzo di spessore (di seguito cantilever la 'B') della forte leva di nitruro (BN) sonda da utilizzare per l'imaging. Con attenzione trasferirlo al titolare liquido sonda cella, assicurando che la punta rimane in posizione eretta. Nota tecnica: la cura estrema deve essere pagato mentre la sonda è in transito, come cadere da qualsiasi altezza può danneggiare o distruggere la cantilever o la punta. Un microlever SNL (MSNL) sonda può essere utilizzata come alternativa alla sonda SNL.
- Rimuovere il supporto della sonda dalla docking station ed esaminare le sbalzo sotto un microscopio ottico per assicurarsi che sia integro e correttamente nel supporto liquido sonda cella.
- Rimuovere con attenzione la testa AFM dal gruppo di coda di rondine strumento stringendo la vite di fissaggio testa zigrinata. Nota tecnica: la cura estrema deve essere pagato mentre manipolando la testa AFM, farlo cadere anche da una breve distanza (ad esempio se non inserita correttamente nel montaggio a coda di rondine) possa arrecare danni sostanziali.
- Invertire la testa AFM e premere con forza il liquido titolare sonda cella sui quattro perni alla base della testa AFM. Cura restituire la testa AFM nel gruppo strumento a coda di rondine. Rilasciare la vite di serraggio testa zigrinata per fissare la testa AFM nello strumento.
3. Individuazione punta a sbalzo, allineamento laser, e regolando fotorilevatore
- Utilizzando il software dello strumento AFM, individuare la punta sbalzo muovendo le manopole del microscopio ottico a bordo. Regolare l'illuminazione del microscopio, se necessario, per migliorare l'identificazione punta. Portare la fine della punta a sbalzo in prossimità del mirino visualizzato sul software dello strumento.
- Tenga la punta del cantilever a fuoco regolando le frecce su e giù del regolatore ottica sul software. Usa lento (S) o media (M) velocità durante la regolazione delle ottiche.
- Allineare il laser per la punta a sbalzo utilizzando le manopole di regolazione laser. Spostare le manopole di regolazione fino a quando il punto laser rosso si trova all'interno della macchia illuminazione bianca. Traccia lungo il braccio a sbalzo con un percorso a zig-zag fino a quando il laser è posizionato sulla punta cantilever. Se correttamente allineata, un punto forte riflessione apparirà nella finestra anteriore della testa AFM.
- Centrare il fotorilevatore regolando le manopole fotorilevatore sulla testa AFM. Questo allineare il punto laser nel mirino del display fotodetettore del software dello strumento. La somma segnale osservato dovrebbe essere di circa 4-6.
4. Posizionamento testa AFM e messa a punto a sbalzo
- Posizionare un foglio appena spaccati di mica attaccato ad un disco metallico campione AFM sul titolare del campione magnetizzato in cima al portatarga AFM. Questa configurazione verrà utilizzato per posizionare la testina di imaging AFM prima il campione di DNA o proteine preparata nella fase 1.
- Nota tecnica: il titolare del campione magnetizzato e liquido titolare sonda cella sono più spessi di altro campione di fissaggio meccanismi e titolari sonda. Se non vi è attualmente insufficiente spazio tra il mica-campione di assemblaggio titolare e la testa AFM, muovere la testa AFM fino selezionando l'icona ritirare il software dello strumento più volte.
- Ruotare la piastra di supporto AFM in modo che il disco di preparato è posizionato per l'immagine iniziale. Assicurarsi che il substrato mica è centrata sotto la testa AFM.
- Spostare la testina di AFM verso ilsuperficie del campione mica disco utilizzando la superficie di controllo focus del software dello strumento. Selezionare il (M), velocità media del motore Z, e proseguire verso la superficie fino a quando la testa AFM è di circa 2 mm sopra la mica. A questo punto, passare ad usare il lento (S) Z velocità del motore al fine di evitare il contatto improvviso tra la sonda e la superficie (di seguito crash punta).
- Procedere per spostare la sonda più vicino alla superficie del substrato mica fino caratteristiche distinte sulla superficie mica o il riflesso punta sono a fuoco. Alterna tra la superficie e aerei riflessione punta focale utilizzando il "Focus on:" icona dal software dello strumento.
- Selezionare l'icona melodia dal software dello strumento e sintonizzare il cantilever. La frequenza di risonanza della BN raccomandata e le sonde a sbalzo MSNL è 20-60 kHz. Selezionare un picco del 5% di offset. Nota tecnica: messa a punto a sbalzo adeguata è essenziale per l'imaging campione adeguato.
5. Imaging mica superficie del campione
- Inserire la sonda sulla superficie di mica. Imposta la dimensione iniziale di scansione a 10 micron e velocità di scansione a 1 Hz. Guadagno integrale e guadagno proporzionale può essere inizialmente impostata a 0,2 e 0,4, rispettivamente. Regolare guadagni come necessario per avere scansioni di rintracciare e ripercorrere circa sovrappongono. Diminuire il setpoint ampiezza aumenterà l'interazione tra la sonda e la mica e garantire l'impegno.
- Catturare immagini scansione completa del campo 10 micron. Diminuire dimensioni di scansione a 5, 1 e 0,5 micron e catturare immagini complete di ogni campo. Regolare gli utili e le velocità di scansione, se necessario. Catturare le immagini della superficie mica fornisce una base di riferimento per il confronto con il DNA e proteine contenenti campioni.
- Sganciare la sonda con un semplice clic sul disimpegno icona sul software dello strumento.
6. Biomolecole Imaging di interesse
- Mescolare delicatamente il campione di DNA o delle proteine preparata nella fase 1 e si preparano a caricarlo sulla superficie mica immagine. Mescolare 5 ml di campione preparato con 45 ml di tampone fresco adsorbimento (concentrazione del campione finale = 0,5 mg / ml).
- Aggiungere con cautela 50 microlitri della mg 0,5 / ml di biomolecole contenenti soluzione direttamente sulla superficie di mica. Fate questo senza muovere la testa o la sonda AFM, e garantire la soluzione del campione (ma non punta della pipetta) entra in contatto con la sonda.
- Pausa di 5 minuti per consentire le biomolecole nel campione di aderire alla superficie mica.
- A conclusione di 5 minuti, pipetta un ulteriore 5-10 ml di buffer di adsorbimento nel supporto liquido sonda cella facendo attenzione a non toccare la sonda, testa AFM, mica o con la punta della pipetta. Questo formerà un menisco e consentire l'imaging AFM toccando in modo fluido. Figura 3 mostra la disposizione finale della cella fluido, a sbalzo, campione, portacampioni, e menisco.
- Regolare la cellula fotoelettrica e riallineare il laser per il cantilever, se necessario. Nota tecnica: la visualizzazione della punta della sonda e di allineamento laser è complicato a causa del modello di diffrazione causati con l'aggiunta di buffer di campione nel supporto sonda. Risintonizzare manualmente il cantilever, se necessario.
- Coinvolgere nuovamente la sonda utilizzando il software dello strumento. Regolare deflessione verticale, guadagno integrale, proporzionale e guadagno, se necessario. Questo inizierà il processo di re-imaging sezione di mica acquisiti immediatamente prima di assaggiare l'aggiunta e la cattura delle immagini.
- Aumentare la dimensione di scansione (500 nm-10 micron) per identificare le regioni di interesse. Zoom-in e ridurre i tassi di scansione a 0,5 Hz per migliorare la risoluzione delle immagini
- Una volta che l'imaging è completo, liberare la sonda selezionando ritirare icona sul software dello strumento più volte. Garantire uno spazio sufficiente tra la testa AFM e disco di preparato prima di smontare la cella di liquidi o disco di preparato.
- Sciacquare il liquido titolare sonda cella e disco di preparato con acqua distillata per evitare la formazione di cristalli di sale come residuo evapora buffer di adsorbimento. Asciugare con aria compressa.
7. Rappresentante dei risultati:
Esempi di immagini AFM sono presentati nella Figura 4. Mica substrato (A) fornisce una superficie piatta sulla quale molecolare del DNA e le proteine possono assorbire. Imaging mica prima di campioni biomolecole fornisce un controllo negativo e valutazione del rumore di imaging. Fornisce anche un livello di garanzia che il cantilever è correttamente sintonizzato e immagini campione successive avrà successo. DNA a doppia elica plasmide (B, D), presuntivamente superavvolto, è facilmente identificato dal suo aspetto asimmetrico e uniforme deposito sul supporto precedentemente mica irrilevante. Complessi proteici di dimensioni delle particelle discrete (C, E) sono univocamente distinguibili dal substrato di mica. Differenze di dimensione delle particelle indicano l'eterogeneità del campione, e può essere utile, per ravvicinare stechiometria complesso proteico o attività biochimica. La forma generale diagonale e l'orientamento costante della proparticelle di proteina osservata nel Panel C è un artefatto di imaging, come sarebbe da aspettarsi che le proteine essere orientato sul supporto mica in modo casuale. Possibili cause del manufatto osservato è una punta di anormalità fisica o AFM imaging troppo rapida di una velocità di scansione. Mentre convoluzione punta (illustrato in Figura 1B) impedisce in assoluto calcoli misurazione della lunghezza del assi X e Y, la misurazione dell'altezza (asse Z) e x y relativo e le misurazioni possono essere comunque utili per stimare le proprietà biofisiche delle biomolecole immagine.
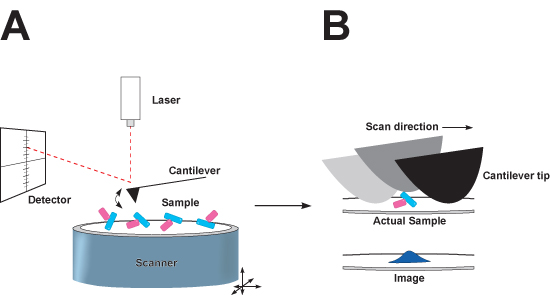
Figura 1: Rappresentazione schematica della toccando microscopia a forza atomica in modalità (AFM). A, il microscopio. Alla fine del cantilever è una punta acuminata che oscilla su e giù come la scansione sulla superficie di un substrato di mica a cui biomolecolari adsorbire complessi. Come lo scanner si muove nelle direzioni x e y, un raggio laser viene riflesso dal retro del cantilever su un rivelatore sensibile alla posizione fotodiodo di mappare la verticale (z) la distanza della punta si muove al passaggio sopra i complessi biomolecolari seduto sul mica. Distorsione B. dell'immagine nelle direzioni x ed y causati da convoluzione punta. Il raggio nominale di una punta convenzionale di nitruro di silicio è più grande delle particelle di essere ripreso, e il bordo dei contatti punta il campione mentre viaggia in superficie. Convoluzione risultati punta nelle particelle immaginato che appaiono più grandi della direzioni X e Y, ma non la direzione z.. Questo effetto può essere minimizzato con l'uso di punte con un raggio minore punta nominale.

Figura 2: Illustrazione di proteine immunoadsorbed e preparazione del campione di DNA. A, rilascio di GR immunoadsorbed • complesso proteico hsp70 dal anticorpi monoclonali (mAb)-proteina A-Sepharose (PAS) pellet. Incubando la immunopellet con un peptide contenente l'epitopo mAb faciliterà il rilascio del GR complesso proteico • hsp70 dal pellet, che consente il complesso da ritirare presso il surnatante e visualizzati da AFM. B, Preparazione del DNA per l'imaging AFM richiede l'uso di un buffer di adsorbimento di cationi bivalenti. Il catione bivalente aumenta l'affinità del DNA per il substrato di mica.

Figura 3: sistemazione finale della testa AFM, liquido titolare sonda cella, campione, portacampioni, e menisco del buffer. A, deve prestare attenzione al momento del deposito del campione e del buffer aggiuntivo sul substrato di mica per evitare il contatto fisico tra la punta della pipetta e la testa AFM, chiamata liquido, e il substrato di mica. B, ingrandimento di A. Il menisco tampone spazia dal disco di preparato al titolare liquido sonda cella.
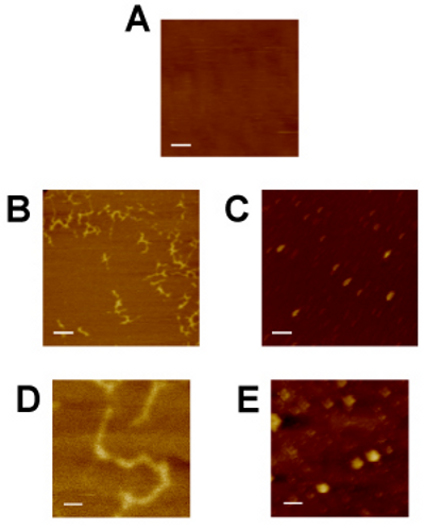
Figura 4: microscopio a forza atomica di substrato di mica (A), 2xGal4-2xGRE-luciferease plasmide DNA 8 (B, D), e GR • complessi proteici hsp70 (C, E) in soluzione tamponata. Mica serve come immagine di controllo da confrontare con le visualizzazioni di DNA e proteine. GR complessi proteici • hsp70 sono stati preparati da immunoadsorbimento di GR vaccinati con hsp70 e poi rilasciato dalla anticorpi tallone pellet utilizzando un anticorpo monoclonale concorrenti (illustrato in Figura 2A). Il DNA è stato preparato da convenzionale miniprep plasmide. Pannelli A, B e C sono di un ingrandimento equivalente (bar scala = 200 nm), così come i pannelli D ed E (barre di scala = 40 nm).
Discussione
AFM fornisce una tecnica microscopica unico in grado di immagini singole biomolecole rivestito in soluzioni tampone acquose e quasi fisiologico in tempo reale. Questo permette la visualizzazione delle singole proteine e molecole di DNA, così come complessi multiproteici e proteina-DNA complessi. Macromolecolari particelle di imaging con AFM può essere utile per valutare l'omogeneità del campione e per identificare la disposizione fisica dei componenti costituenti delle particelle osservate. Questo approccio di osservare particelle macromolecolari individuo può essere un utile complemento alle tradizionali tecniche biochimiche, come ad esempio test immunoprecipitazione, elettroforesi su gel di poliacrilamide, ed esclusione cromatografia dimensioni delle colonne, che forniscono dati significativi sommativa che rappresenta il 10 3 -10 11 complessi biomolecolari individuali di interesse attuale in un campione.
La ricerca sulla stechiometria multiproteici complesse, interazioni biomolecolari, e le esigenze cofattore possono essere indagati con AFM. Il metodo presentato qui possono essere adattate a specifiche questioni di interesse biofisico. Ad esempio, utilizzando un liquido titolare sonda cella in grado di scambiare tampone, è possibile cofattore requisiti test per la formazione delle proteine complesse. DNA-proteine gruppi possono essere formati e dissociato in near-real-time e anche mentre sta creando l'immagine. DNA può essere generato con sequenze specifiche di interesse (ad esempio una risposta di elementi presuntivi o di proteine sequenza romanzo vincolante), mescolato con la proteina ipotizzato vincolante, e ripreso a fornire una prova diretta di interazioni intermolecolari.
Toccando la modalità di imaging AFM è sostanzialmente meno complicato se i campioni a secco sono utilizzati piuttosto che campioni in soluzioni acquose, e stand DNA lineare e chiuso cerchio plasmidi di DNA sono stati entrambi ripreso in questo modo 3,9. Il metodo qui presentato utilizza campioni in soluzioni tampone al fine di fornire un ambiente di imaging più fisiologico. Altri metodi degni di nota di proteine di imaging dovrebbero essere considerati, tra cui microscopia a campo vicino infrarosso 10. L'uso complementare di immunoadsorbimento in consorte con AFM offre l'opportunità di preparare una miriade di complessi proteici, utilizzando consolidate tecniche biochimiche, e la loro successiva visualizzazione dopo la liberazione dal immunoadsorbing anticorpi tallone pellet. Per esempio, GR immunoadsorbed è stato testato per la sua associazione con la proteina chaperone molecolare hsp70 1 così come la proteina dineina motore 2 utilizzando questo approccio per valutare dimensioni complesse e stechiometrie. Dimensione delle particelle e le caratteristiche biofisiche (rigidità ad esempio) sono stati utili per accertare l'identità delle biomolecole osservati in campioni di AFM fotografato 11,12. E 'anche possibile confermare l'identità biomolecola con l'aggiunta di una biomolecola interagenti (ad esempio, un ligando o anticorpi monoclonali), che si legano al suo obiettivo e provocare un aumento delle dimensioni delle particelle se la biomolecola bersaglio è presente.
Divulgazioni
Riconoscimenti
Questo lavoro è stato finanziato dal National Institutes of Health di Grant GM086822. Gli autori desiderano ringraziare Drs. Alec Pakhomov & Paul Wallace (Univ. di Funzione utente Nanotechnology di Washington (NTUF)) e Andrea Slade (Bruker AXS) per la loro assistenza tecnico esperto. DNA imaging AFM è stato condotto presso l'Univ. NTUF di Washington, membro della Rete Nazionale Infrastrutture Nanotechnology. Il 2xGal4-2xGRE-luciferease plasmide è stato gentilmente fornito dal laboratorio del Dr. Keith Yamamoto (Univ. of California, San Francisco).
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | |
|---|---|---|---|
| Dimension 3100 | Bruker | Dimension 3100 | |
| Fluido dimensione cella | Bruker | DTFML-DD-HE | |
| Nitruro di leva Sharp (BN) in nitruro di silicio sonda AFM | Bruker | BN-10 | |
| Microlever forte nitruro di leva (MSNL) nitruro di silicio sonda AFM | Bruker | MSNL-10 | |
| Nanoscope AFM strumento software | Bruker | 004-132-000 | |
| Metallo AFM campione dischi | Ted Pella | 16208 | |
| Grado V1 Dischi Mica, 12 mm | Ted Pella | 50-12 |
Riferimenti
- Murphy, P. J. M. Visualization and mechanism of assembly of a glucocorticoid receptor•hsp70 complex that is primed for subsequent Hsp90-dependent opening of the steroid binding cleft. J Biol Chem. 278, 34764-34773 (2003).
- Harrell, J. M. Evidence for glucocorticoid receptor transport on microtubules by dynein. J Biol Chem. 279, 54647-54654 (2004).
- Pastre, D. Adsorption of DNA to mica mediated by divalent counterions: a theoretical and experimental study. Biophys J. 85, 2507-2518 (2003).
- Guthold, M. Direct observation of one-dimensional diffusion and transcription by Escherichia coli RNA polymerase. Biophys J. 77, 2284-2294 (1999).
- Petrucco, S., Volpi, G., Bolchi, A., Rivetti, C., Ottonello, S. A nick-sensing DNA 3'-repair enzyme from Arabidopsis. J Biol Chem. 277, 23675-23683 (2002).
- Murphy, P. J. M., Morishima, Y., Kovacs, J. J., Yao, T. P., Pratt, W. B. Regulation of the dynamics of hsp90 action on the glucocorticoid receptor by acetylation/deacetylation of the chaperone. J Biol Chem. 280, 33792-33799 (2005).
- Ruan, B., Fisher, K. E., Alexander, P. A., Doroshko, V., Bryan, P. N. Engineering subtilisin into a fluoride-triggered processing protease useful for one-step protein purification. Biochemistry. 43, 14539-14546 (2004).
- Meijsing, S. H., Elbi, C., Luecke, H. F., Hager, G. L., Yamamoto, K. R. The ligand binding domain controls glucocorticoid receptor dynamics independent of ligand release. Mol Cell Biol. 27, 2442-2451 (2007).
- Shen, X. C. A simple and effective sample preparation method for atomic force microscopy visualization of individual DNA molecules in situ. Mol Biol Rep. 38, 965-969 (2010).
- Paulite, M., Fakhraai, Z., Akhremitchev, B. B., Mueller, K., Walker, G. C. Assembly, tuning and use of an apertureless near field infrared microscope for protein imaging. J Vis Exp. , (2009).
- Cretu, A., Castagnino, P., Assoian, R. Studying the effects of matrix stiffness on cellular function using acrylamide-based hydrogels. J Vis Exp. , (2010).
- Brunger, A. T., Weninger, K., Bowen, M., Chu, S. Single-molecule studies of the neuronal SNARE fusion machinery. Annu Rev Biochem. 78, 903-928 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon