Method Article
Aseptische Labortechnik: Überzug Methoden
In diesem Artikel
Zusammenfassung
Beim Arbeiten mit und Reagenzien zur Kultur von Mikroorganismen, muss aseptischen Technik praktiziert, um sicherzustellen, Kontamination zu minimieren werden. Eine Vielzahl von Beschichtungsverfahren werden routinemäßig verwendet, um zu isolieren, verbreiten oder aufzuzählen Bakterien und Phagen, die alle Verfahren, die die Sterilität der experimentellen Materialien zu erhalten übernehmen.
Zusammenfassung
Mikroorganismen sind auf allen unbelebten Oberflächen schaffen allgegenwärtigen Quellen möglicher Kontamination im Labor. Experimentelle Erfolg beruht auf der Fähigkeit eines Wissenschaftlers auf Arbeitsflächen und Geräte zu sterilisieren sowie deren Kontakt mit sterilen Instrumenten und-Lösungen mit unsterilen Oberflächen. Hier präsentieren wir Ihnen die Schritte für mehrere Plating Verfahren routinemäßig im Labor verwendet werden, um zu isolieren, zu vermehren, oder aufzählen Mikroorganismen wie Bakterien und Phagen. Alle fünf Methoden integrieren aseptischen Technik oder Verfahren, die die Sterilität der experimentellen Materialien zu erhalten. Beschriebenen Verfahren umfassen (1) streifenfreie Beschichtung Bakterienkulturen, um einzelne Kolonien zu isolieren, (2) gießen-Plating und (3) Spread-Plating, lebensfähige Bakterien-Kolonien aufzählen, (4) Soft-Agar-Overlays zu Phagen-Plaques und enumerate isolieren, und ( 5) Replica-Plattierung, um Zellen aus einer Platte zur anderen in einer identischen räumlichen Muster zu übertragen. Diese Verfahren können bei t durchgeführt werdener Labortisch, vorausgesetzt, sie beinhalten nicht-pathogene Stämme von Mikroorganismen (Biosafety Level 1, BSL-1). Wenn die Arbeit mit BSL-2 Organismen, dann werden diese Manipulationen muss in einer biologischen Sicherheitswerkbank nehmen. Wenden Sie die aktuellste Ausgabe der Biosicherheit in mikrobiologischen und biomedizinischen Laboren (BMBL) sowie Material Safety Data Sheets (MSDS) für infektiöse Substanzen, um die Biohazard Klassifikation sowie die Sicherheitshinweise und Rückhalteeinrichtungen für den Mikroorganismus in Frage erforderlich zu bestimmen. Bakterienstämme und Phagen-Aktien können von der Forschung Forscher, Unternehmen und Sammlungen von bestimmten Organisationen wie der American Type Culture Collection (ATCC) bezogen werden beibehalten. Es wird empfohlen, nicht-pathogenen Stämme verwendet werden, wenn das Erlernen der verschiedenen Beschichtungsverfahren werden. Durch Befolgen der Verfahren in diesem Protokoll beschrieben, sollten die Studierenden in der Lage:
- Führen Plating Verfahren ohne eingeschleppte Verunreinigungenng Medien.
- Isolieren Sie einzelne Bakterienkolonien von der Streak-Plating-Verfahren.
- Verwendung Pour-Plattierung und Excel Plattierungsverfahren, um die Konzentration der Bakterien zu bestimmen.
- Führen Weichagar Overlays bei der Arbeit mit Phagen.
- Übertragen von Bakterienzellen aus einer Platte zur anderen unter Verwendung der Replika-abgeschieden werden.
- Da eine experimentelle Aufgabe, wählen Sie den entsprechenden Beschichtungsverfahren.
Protokoll
1. Vorbereiten einer sicheren und steriler Arbeitsraum
- Machen Sie sich mit allen Labor-Regeln und Sicherheitsvorkehrungen beim Arbeiten mit Mikroorganismen werden. Unabhängig von Biohazard Klassifizierung, werden alle Materialien, die in Kontakt mit Mikroorganismen kommen als infektiöse Abfälle und müssen vor der Entsorgung dekontaminiert werden. Folgen Sie Sicherheitsrichtlinien im Einklang mit den von institutionellen Environmental Health and Safety-Abteilungen zur Verfügung gestellt, die Einrichtung von entsprechenden Abfallbehälter für die sofortige und sachgerechte Entsorgung von möglicherweise kontaminiertem Material (biologische Gefahrstoffe).
- Sterilisieren Sie alle Instrumente, Lösungen und Medien vor, sie für Plating Verfahren.
- Räumen Sie alle Materialien, überladen Sie Ihren Arbeitsbereich auf dem Labortisch.
- Sauberen Arbeitsfläche mit einem Desinfektionsmittel, um mögliche Verunreinigungen zu minimieren.
- Richten Sie einen Bunsenbrenner und arbeiten langsam, vorsichtig und bewusst in den sterilen Bereich durch die Gegend updra erstelltft der Flamme.
- Wenn die Arbeit mit BSL-2 Organismen, richten Sie Ihren Arbeitsplatz in einer biologischen Sicherheitswerkbank. Ein Bunsenbrenner nicht im Schrank verwendet werden, da die Wärme aus der Flamme stört Luftstrom wesentlich, um seine Funktionalität.
- Ordnen Sie die Vorräte für das Verfahren auf dem Labortisch in der Nähe des sterilen Feldes benötigt. Achten Sie darauf, alle Materialien richtig beschriftet sind. Organisieren Sie den Arbeitsbereich, um Arbeit zu maximieren und unnötige Bewegungen zu minimieren wird die Belichtungszeit der experimentellen Materialien gegenüber Luftschadstoffen.
- Setzen Sie den Bunsenbrenner auf der rechten Seite auf der Bank.
- Ort Agarplatten oder Petrischalen auf der linken Seite.
- Anordnen Zellkulturen, Rohre, Flaschen und Kolben in der Mitte der Bank.
- Lösen Sie die Kappen von Rohren, Flaschen und Flaschen, so dass sie leicht mit einer Hand kann bei späteren Manipulationen eröffnet.
- Waschen Sie Ihre Hände gründlich mit Seife und warmem antiseptischenWasser vor dem Umgang mit Mikroorganismen.
2. Streak Platte Vorgehensweise: Isolierung von Bakterien-Kolonien mit dem Quadrant-Methode
Der Streifen-Platte Verfahren wurde entwickelt, um reine Kulturen von Bakterien oder Kolonien, aus gemischten Populationen zu isolieren durch einfache mechanische Trennung. Einzelne Kolonien werden von Millionen von Zellen, die in einem Cluster auf oder in einer Agar-Platte (1) besteht. Eine Kolonie, im Gegensatz zu einer einzelnen Zelle ist, die mit bloßem Auge. Theoretisch werden alle Zellen in einer Kolonie von einer Bakterie anfänglich auf der Platte abgeleitet und sind somit als Klon, oder Cluster von genetisch identischen Zellen bezeichnet.
- Bakterien vorhanden in einer Vielzahl von Formen und Größen. Zum Beispiel werden einzelne Zellen von Escherichia coli stabförmigen mit einer durchschnittlichen Länge von 2 um und Breite von 0,5 um, während Streptococcus Zellen kugelförmig mit einem mittleren Durchmesser von 1 um ist. Manche Bakterien (such wie E. coli) als einzelne Zellen bestehen, andere unterschiedliche Muster der Vereinigung zu bilden. Streptococcus, zum Beispiel, in Paaren oder Ketten oder Clustern von Zellen wachsen. Es wird allgemein angenommen, dass eine einzelne Kolonie von einer einzigen Zelle durchläuft binäre Spaltung entsteht, allerdings ist diese Annahme nicht für jene Bakterien, die natürlicherweise als Paare, Ketten oder Cluster oder jener Division durch andere Mechanismen gibt wahr. Alternativ kann, wenn zu viele Bakterien beschichtet sind, überlappen dann von Zellen kann auftreten, und erhöhen die Wahrscheinlichkeit von zwei oder mehreren Bakterien, die zu scheinbar eine einzige Kolonie. Um diese Komplikationen zu vermeiden, bei der Beschreibung oder Aufzählung Bakterienkulturen wachsen auf einem festen Medium werden die Kolonien als Kolonie bildende Einheiten (KBE) bezeichnet.
Mit dem Streifen-Platte Verfahren wird ein Gemisch von Zellen über die Oberfläche eines halbfesten, Agar-basierten Nährmedium in einer Petrischale, so dass immer weniger bakteriellen verteiltZellen werden bei weit entfernten Punkten auf der Oberfläche des Mediums aufgebracht und nach Inkubation in Kolonien entwickeln. Der Quadrant Verfahren zur Isolierung von einzelnen Kolonien aus einer Mischung von Zellen wird hier beschrieben.
- Streak-Beschichtung kann mit einer Reihe von Instrumenten (2) durchgeführt werden. Ein Metall-Schleife kann mehrere Male wieder verwendet werden und ist für streifenfreie Beschichtung Routine-Labor-Stämme verwendet. Einweg-Kunststoff-Schleifen sind im Handel erhältlich und werden häufiger bei der Arbeit mit BSL-2-Stämmen in einer biologischen Sicherheitswerkbank verwendet. Viele Forscher lieber mit Einweg-, vor-sterilisierte Holzstäbchen oder flache Zahnstocher für streifenfreie Beschichtung. Diese sind eine preiswerte Alternative zu Einweg-Kunststoff-Schlaufen und kann besonders nützlich sein bei der Arbeit mit einer Probe aus der Umwelt wie Boden, die wahrscheinlich enthält sporenbildende Bakterien.
- Einweg vorsterilisiert Sticks und Zahnstochern muss nicht mit der Bunsenbrenner Do geflammt werdens Vermeidung unnötiger Aerosolisierung sporenbildende Bakterien und Kreuzkontamination von Oberflächen im Labor oder Agar-Platten mit Sporen.
- Etikett um den Rand des Bodens (nicht der Deckel) einer Agar-Platte mit mindestens Ihren Namen, das Datum, die Art des Wachstumsmediums und der Art des Organismus, um auf dem Medium ausplattiert werden.
- Die Platten müssen völlig trocken sein, ohne Kondenswasser am Deckel und vorgewärmten auf Raumtemperatur vor streifenfreie Beschichtung. Wenn die Platten bei 4 ° C gelagert werden, entfernen Sie sie mehrere Stunden oder sogar am Tag zuvor. Breitete sie in kleinen, versetzten Stapeln von nicht mehr als 2-3 Platten und trocknen lassen.
- Die Probe, von der der Streifen-Platte geimpft werden könnte entweder eine Suspension von Zellen in der Brühe oder eine bestehende Kolonie von anderen Agarplatte sein. Um zu beginnen, wird empfohlen, dass nur eine Probe verwendet, um eine einzelne Platte zu beimpfen. Um Zeit und Material zu sparen, kann eine einzelne Platte für mehrere samp verwendet werdenles aber nur, wenn Sie sich bei einer Probe Schlieren auf einer Platte beherrschen.
- Für den ersten Quadranten, wird die Probe aufgenommen und über etwa ein Viertel der Oberfläche des Mediums unter Verwendung eines schnellen, glatt, hin-und herbewegt wird (Feld A in 3) verteilt. Heben Sie die untere Hälfte eines umgedrehten Teller von der Bank. Bewegen der Schleife, Stock, oder Zahnstocher hin und her viele Male über die Agaroberfläche vom Rand zur Mitte der Platte.
- Wenn das Inokulum eine Zellsuspension erhalten eine Öse mit einem Metallring oder 5 bis 10 ul mit einer Mikropipette. Achten Sie darauf, Strudel oder Wirbel die Zellsuspension vor dem Entfernen eines Aliquots für die Beschichtung. Unter aseptischen Bedingungen arbeiten, während Sie diesen Schritt ausführen, flammende nicht nur die Schleife, sondern auch den Rand der Flasche oder Tube vor und nach dem Entfernen des Inokulums. Auch, berühren Sie nicht die Seiten der Tube oder Flasche mit der Schlaufe oder Zylinder der Mikropipette. Wenn das Inokulum Pipettieren, Dosieren Sie die Zellsuspension auf die appropriate Platz auf der Platte dann eine Schleife verwenden, Stick oder Zahnstocher, um ihn über den ersten Quadranten der Platte verteilt.
- Wenn das Inokulum ist eine Kolonie von einer anderen Platte, sanft berühren die Kolonie mit der Schleife, Stick oder Zahnstocher dann breitete sie über den ersten Quadranten der Platte. Achten Sie darauf, das Metall-Schleife vor dem Berühren der Kolonie gekühlt wird. Um sicherzustellen, dass die Schleife nicht zu heiß ist, berühren Sie es leicht auf der Agar-Platte in einem bestimmten Bereich, wo keine Schlieren auftreten. Nur ein paar Zellen aus der Kolonie, nicht die gesamte Kolonie benötigt.
- Bei Verwendung eines Metall-Schleife, schwer es mit Hilfe eines Bunsenbrenners vor der Erteilung des Inokulums für die Platte (Panel B von Abbildung 3). Starten Sie etwa 3-4 cm von der Schleife, mit dem Draht in der Spitze der blauen Kegel, dem heißesten Teil der Flamme. Das Metall sollte sich Red Hot. Bewegen Sie den Draht, so dass die Flamme nähert sich der Schleife. Diese Manipulation verhindert Vernebelung von Bakterien auf der Schleife von der vorherigen Nutzung überlassen.
- Flaming tötet dieBakterien (die aber nicht Sporen) und Konvektionsströme von der erwärmten Luft verhindern, dass andere Verunreinigungen aus der Luft Absetzen auf den Metalldraht während der nachfolgenden Manipulationen.
- Wenn mit einem sterilen Zahnstocher flach, halten Sie das schmale Ende sanft zwischen Daumen und Ringfinger zu einem 10 bis 20 ° Winkel auf das Medium, und nutzen Sie das breite Ende zu streifen die Quadranten. Wenn Sie eine Schleife oder Holzstäbchen, halten Sie ihn wie einen Bleistift unter dem gleichen Winkel. Nicht so hart drücken, dass die Schleife, Stick oder Zahnstocher gräbt sich in den Agar.
- Nach Abschluss des ersten Quadranten, umdrehen und setzen Sie die Platte wieder in Deckel auf der Bank. Entsorgen Sie den Stick oder Zahnstocher oder Re-Flamme das Metall-Schleife, wie in Schritt 4 beschrieben.
- Drehen Sie die Petrischale 90 ° zur Ader im zweiten Quadranten. Heben Sie die untere Hälfte eines umgedrehten Teller von der Bank aus und berühren Sie dann die Schleife, Stick oder Zahnstocher auf den ersten Quadranten in der Nähe des Ende des letzten Streifen. Mit dem Hin-und Her-Muster, über den Las überquerent die Hälfte der Streifen in dem ersten Quadranten dann in der leeren zweiten Quadranten zu verschieben. Umkehren und stellte den Teller zurück in Deckel auf der Bank, sobald der zweiten Quadranten gefüllt ist.
- Die Schleife sollte Stick oder Zahnstocher gehen nie wieder in die erste Hälfte der Streifen in den ersten Quadranten, wo die meisten der ursprünglichen Inokulum wurde hinterlegt.
- Eine neue Kunststoff-Schleife, Holzstäbchen oder Zahnstocher sollte für jeden Quadranten eingesetzt werden.
- Wiederholen Schritt # 6 zweimal für die dritten und vierten Quadranten.
- Achten Sie darauf, der Stick oder Zahnstocher oder Re-Flamme das Metall Schleife zwischen jedem Quadranten zu verfügen.
- Vermeiden Sie gehen in den ersten Quadranten, wenn Streifenbildung dem vierten Quadranten.
- Anschließend wird die Platte umgedreht, so Kondensat, das auf dem Deckel sammelt nicht auf die Kolonien tropfen.
3. Plattengussverfahren Vorgehensweise: Zählung von Bakterienzellen in einer Mischprobe
Diese Methode ist oft verwendend die Anzahl der Mikroorganismen in einer gemischten Probe zu zählen, welche an einen geschmolzenen Agar-Medium vor seiner Verfestigung zugegeben. Das Verfahren führt zu Kolonien gleichmäßig über dem festen Medium, wenn die entsprechenden Proben Verdünnung ausplattiert verteilt wird. Diese Technik wird verwendet, um lebensfähige Platte zählt, in denen die Gesamtzahl der koloniebildenden Einheiten in den Agar und auf der Oberfläche des Agar auf einer einzigen Platte wird aufgelistet durchzuführen. Lebensfähige Keimzahlen den Wissenschaftlern ein standardisiertes Mittel zur Wachstumskurven erzeugen, um die Konzentration der Zellen in der Röhre, aus welcher die Probe plattiert zu berechnen und den Effekt von verschiedenen Umgebungen oder Wachstumsbedingungen auf Bakterienzelle Überleben oder Wachstum zu untersuchen.
- Etikett um den Rand des Bodens (nicht der Deckel) einer sterilen, aber leere Petrischale mit mindestens Ihren Namen, das Datum, die Art des Wachstumsmediums und der Art von Organismus zu dem geschmolzenen Agar-Medium zugesetzt werden.
- Den Verdünnungsfaktor wenn platÝng Reihenverdünnungen, oder eine Reihe von wiederholten Verdünnungen, die in einer systematischen Verringerung der Konzentration der Zellen in der Probe führt. Vorbereiten Reihenverdünnungen ist notwendig, wenn die Anzahl der Zellen in der Probe größer als die Kapazität der Agarplatte, bei dem die statistisch signifikante Bereich von 30 bis 300 KBE. Wenn es mehr als 300 KBE auf einer Platte sind, dann werden die Kolonien wird überfüllt und Überschneidungen.
- Besorgen Sie sich eine Tube mit 18 ml geschmolzenem Agar-Medium.
- Die Agar-Medium sollte in Reagenzgläser verzichtet werden und in einem Autoklaven vorsterilisiert. Am selben Tag für ein Experiment benötigt wird, sollte der Agar in einem Dämpfer für 30 Minuten dann in eine 55 ° C Wasserbad überführt geschmolzen werden. Nur so viel wie für Agar des Experimentes nach Bedarf sollte geschmolzen werden und können nicht wieder verwendet werden kann.
- Zehn Minuten vor dem Gießen Platten sollten die Rohre aus geschmolzenem Agar aus dem 55 ° C warmes Wasserbad zu einer Wärme-Block werden auf den Arbeitsmarkt übertragenAtory Bank bei 48 ° C eingestellt Nachdem der Agar diese Temperatur erreicht, ist sie bereit, zu gießen. Wenn der Agar zu heiß ist, können die Bakterien in der Probe getötet werden. Wenn der Agar ist zu kühl, kann das Medium sein, klumpig einmal verfestigt.
- Erhalten Sie Probe, wobei man entweder eine Bouillonkultur oder eine Suspension von Zellen durch Mischen von Zellen aus einer Kolonie in Puffer oder Salzlösung, hergestellt werden.
- Die Proben können von einer Verdünnungsreihe von einer einzigen Probe abgeleitet werden.
- Probenvolumen zu plattierenden sollte zwischen 0,1 und 1,0 ml betragen.
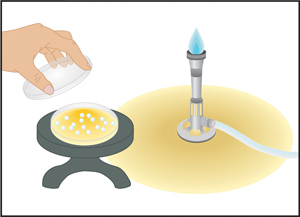
- Öffnen Sie den Deckel des leeren Petrischale, und verzichten Sie Ihre Probe in die Mitte der Platte (Panel A von Abbildung 4). Schließen Sie den Deckel.
- Unter aseptischen Bedingungen arbeiten im Rahmen dieses Verfahrens.
- Verwenden Sie entweder eine serologische Pipette oder Mikropipette Ihrer Probe auf die Platte übertragen. Steuern den Fluss der Probe, damit es nicht spritzt aus der Platte.
- Entfernen Sie den Deckel der Wannee des geschmolzenen Agar, und übergeben Sie den Rand des offenen Rohr durch die Flamme des Bunsenbrenners.
- Öffnen Sie den Deckel der Petrischale mit Ihrer Probe und gießen Sie das Agar-Agar in sorgfältig (Feld B der Abbildung 4). Schließen Sie den Deckel dann mischen die Probe mit dem Agar durch Schwenken der Platte.
- Lassen Sie das Agar zu gründlich, bevor umdrehen, die für die Inkubation zu verfestigen.
4. Spread-Platte Vorgehen: Bildung von diskreten Bakterienkolonien für Plattenzählungen, Anreicherung, Selektion oder Screening
Diese Technik wird typischerweise zu trennen Mikroorganismen in einem kleinen Probenvolumen, die über die Oberfläche einer Agarplatte verteilt, was zur Bildung von diskreten Kolonien gleichmäßig über die Agaroberfläche verteilt, wenn die geeignete Konzentration von Zellen ausplattiert enthalten sind, verwendet. Zusätzlich zur Verwendung dieser Technik für lebensfähige Platte zählt, in denen die Gesamtzahl von koloniebildenden Einheiten auf einem singenle Platte aufgeführt ist und verwendet, um die Konzentration der Zellen in der Röhre, aus welcher die Probe plattiert zu berechnen, wird Excel Plattieren routinemäßig mit der Anreicherung, Selektion und Screening-Experimenten verwendet. Das gewünschte Ergebnis für diese drei Experimenten ist in der Regel die gleichen wie für die Platte zählt, in dem eine Verteilung von diskreten Kolonien bildet über die Oberfläche des Agar. Allerdings ist das Ziel nicht zu gewährleisten, dass alle lebensfähigen Zellen bilden Kolonien. Stattdessen sollten nur diejenigen Zellen innerhalb einer Population, die einen bestimmten Genotyp haben zu wachsen. Die Ausbreitung Platte Prozedur kann sich über die Plattengussverfahren Technik für eine Aufzählung Experiment eingesetzt werden, wenn das Ende Ziel ist es, Kolonien zur weiteren Analyse zugänglich zu isolieren, weil Kolonien wachsen auf der Agar-Oberfläche, während sie eingebettet in den Agar mit dem Plattengussverfahren Verfahren geworden.
Es gibt zwei Strategien hier für die Ausbreitung der Platte beschriebenen Verfahren. Die erste (Methode A) umfasst die Verwendung einer Drehscheibe und Glas oder Metall Rod wie ein Hockeyschläger geprägt. Die zweite (Methode B), bezeichnet als der "Copacabana Verfahren", umfasst Schütteln vorsterilisiert Glasperlen. Beide erleichtern die gleichmäßige Verteilung der Zellen über die Agar-Oberfläche.
Methode A: Spread-Beschichtung mit einer Drehscheibe und Glas-oder Metallstab
- Etikett um den Rand des Bodens (nicht der Deckel) einer Agar-Platte mit mindestens Ihren Namen, das Datum, die Art des Wachstumsmediums und der Art des Organismus, um auf dem Medium ausplattiert werden.
- Den Verdünnungsfaktor wenn Ausplattieren serielle Verdünnungen.
- Die Platten müssen völlig trocken sein, ohne Kondenswasser am Deckel und vorgewärmten auf Raumtemperatur vor der Ausbreitung-Plating. Wenn die Platten bei 4 ° C gelagert werden, entfernen Sie sie mehrere Stunden oder sogar am Tag zuvor. Breitete sie in kleinen, versetzten Stapeln von nicht mehr als 2-3 Platten und trocknen lassen.
- Zentrieren Sie die Platte auf dem Plattenteller (Abbildung 5).
- Erhalten Sier Probe, die eine Bouillonkultur oder eine Suspension von Zellen durch Mischen von Zellen aus einer Kolonie in Puffer oder Kochsalzlösung hergestellt sein sollte.
- Die Proben können von einer Verdünnungsreihe von einer einzigen Probe abgeleitet werden.
- Probenvolumen zu plattierenden sollte zwischen 0,1 und 0,2 ml betragen.
- Öffnen Sie den Deckel der Petrischale, und verzichten Sie Ihre Probe auf die Mitte des Agars. Schließen Sie den Deckel.
- Unter aseptischen Bedingungen arbeiten im Rahmen dieses Verfahrens.
- Verwenden Sie einen Mikropipette Ihrer Probe auf die Platte übertragen. Steuern den Fluss der Probe, damit es nicht spritzt aus der Platte.
- Tauchen des Glasstabs oder Metallstab (auch als Spreizer) in ein Becherglas von 70% (v / v) Ethanol.
- ACHTUNG: Tauchen Sie ein heißes Saling in ein Becherglas von Alkohol.
- Das Ethanol muss nur an das Bodenteil der Spreizer und die ersten Zentimeter des Stiels.
- Abtropfen lassen und überschüssiges Ethanol entzünden, indem es durch die Flamme eines Bunsen-Brenner.
- Die Flamme sollte reisen die Länge der Spreizer und Stammzellen, die in Kontakt mit der Ethanol dann schnell auslöschen kam.
- Sollte das Becherglas mit Ethanol Feuer fangen, keine Panik! Legen Sie eine Glasabdeckung über dem Becher, die sich schnell auslöschen wird das Feuer.
- Öffnen Sie den Deckel der Agarplatte, halten den Deckel in die linke Hand mit Daumen und Zeigefinger. Kühlen Sie den Streuer durch Berühren der Agar entlang der Kante in der Nähe des Randes.
- Berühren Sie nicht die Agar, wo die Zellen wurden hinzugefügt. Das heiße Spreizer tötet die Zellen.
- Mit der linken Hand (bei gedrückter den Deckel des Agar-Platte), drehen Sie den Plattenspieler langsam.
- Obwohl am besten vermieden, wenn man den Deckel muss legte, legen Sie sie verdeckt auf einer desinfizierten Fläche innerhalb des sterilen Bereich des Bunsenbrenners. Mit einem Deckel, der nach oben weist, besteht eine größere Wahrscheinlichkeit einer Kontamination von Bewegungen von Objekten oder Hände, wodurch Luftströmungen, die dazu führenMikroorganismen und Staubpartikel auf der Innenfläche des Deckels steigen.
- Mit der rechten Hand, halten Sie den Streuer sanft auf der Oberfläche des Agars und breitete sich allmählich die Probe gleichmäßig über die gesamte Platte. Bewegen Sie den Streuer hin und her über die Platte als die Drehscheibe dreht.
- Lassen Sie die Probe gründlich zu absorbieren (mindestens 5 Minuten), bevor umdrehen, die für die Inkubation.
Methode B: Spread-Plating mit Glasperlen: Die "Copacabana-Methode"
- Etikett um den Rand des Bodens (nicht der Deckel) einer Agar-Platte mit mindestens Ihren Namen, das Datum, die Art des Wachstumsmediums und der Art des Organismus, um auf dem Medium ausplattiert werden.
- Den Verdünnungsfaktor wenn Ausplattieren serielle Verdünnungen.
- Öffnen Sie den Deckel der Agarplatte, halten den Deckel in die linke Hand mit Daumen und Zeigefinger. Dann öffnen Sie das Glasrohr oder Flasche mit vorsterilisiert glArsch Perlen. Mit der rechten Hand, Flamme der Rand der Tube oder Flasche dann vorsichtig verzichten 10-12 sterilen Glasperlen auf einer Agar-Platte (Abb. 6). Schließen Sie den Deckel der Platte und Flamme der Rand der Tube oder Flasche noch einmal, bevor Sie die Kappe und setzen Sie ihn beiseite.
- Vorsichtig gießen die Perlen aus der Tube oder Flasche ein paar Zentimeter über dem Agar, so dass die Perlen nicht hüpfen aus der Petrischale.
- Verwendung Glasperlen, die 4 mm im Durchmesser sind. Sterilisieren in Glasflaschen oder Teströhrchen im Autoklaven bei 121 ° C auf dem Trocken-Zyklus (Schwerpunkteinstellung) für 30 Minuten.
- Ein Vorteil der Verwendung von Kügelchen statt Spreader ist, dass keine offenen Behälter Ethanol wiederholt flammenden erforderlich sind.
- Öffnen Sie den Deckel des Agar-Platte, und verzichten Sie Ihre Probe auf die Mitte des Agars. Schließen Sie den Deckel.
- Unter aseptischen Bedingungen arbeiten im Rahmen dieses Verfahrens.
- Aliquot von 100 bis 150 ul Probe pro Platte. DiesVolumen erleichtert gleichmäßige Verteilung der Zellen. Verwenden Sie einen Mikropipette Ihrer Probe auf die Platte übertragen. Steuern den Fluss der Probe, damit es nicht spritzt aus der Platte.
- Verteilen Sie die Probe durch leichtes Schütteln der Kügelchen auf der Oberfläche des Agar 6 bis 7. Um sicherzustellen, Zellen gleichmäßig verteilt, verwenden Sie eine horizontale Schüttelbewegung.
- Nicht wirbeln die Perlen oder sonst alle Zellen werden am Ende an der Plattenkante.
- TIPP: Wenn man es richtig macht, hört sich das Verfahren wie "Schütteln Maracas".
- Drehen Sie die Platte 60 ° horizontal dann wieder schütteln 6 bis 7 mal.
- Drehen Sie die Platte 60 ° ein zweites Mal und wieder horizontal zu schütteln. Mittlerweile sollten Sie erreichen sogar die Verteilung der Zellen über die Agar-Oberfläche.
- Wenn Sie fertig Spread-Plating, sollte die Probe absorbiert und die Oberfläche des Agars sollte trocken sein. Gießen Sie die kontaminierten Perlen in einen markierten Sammlung Becherglas mit 10% Chlorbleiche.
- Nichtverwerfen Sie die Perlen in den Müll! Die verwendeten Perlen wird gespült und sterilisiert werden, neu zu sterilisieren sie für wiederholter Nutzung.
- HINWEIS: Wenn der Agar-Oberfläche ist nach wie vor nach dem Schütteln dreimal nass wird, lassen die Platte für einige Minuten sitzen, um an die Flüssigkeit zu erlauben, von der Agar absorbiert werden, dann wiederholen Sie die Schritte 4-6 #, bis der Plattenoberfläche trocken erscheint.
- Anschließend die Platte für die Inkubation.
5. Soft-Agar-Overlay-Verfahren: Bildung von Plaques für Isolierung und Zählung von Phage (Plaque-Assay)
Diese Technik wird allgemein verwendet, um nachzuweisen und zu quantifizieren Bakteriophagen (Phagen), oder bakterielle Viren, die eine Größe im Bereich von 100 bis 200 nm aufweist. Elektronenmikroskop benötigt wird, um einzelne Phagenpartikel zu sehen. Jedoch kann die Gegenwart von infektiösen Phagenpartikeln als Plaques auf einer Agar-Platte (7A) detektiert werden. Phagen können nicht außerhalb ihres Wirtsbakterienzellen replizieren, so Ausbreitung eind Erkennung erfordert Mischen Phagen und Wirtszellen zusammen vor dem Beschichten. Für die Soft-Agar-Overlay-Verfahren, wird ein kleines Volumen, im allgemeinen im Bereich von 50 bis 200 ul ul, eines Phagen Suspension in ein Röhrchen mit etwa 10 8 Bakterien (Wirtszellen), die gleichmäßig in 2,5-3,0 ml dispergiert sind abgegeben weichen (0,5 bis 0,7% [w / v]), geschmolzen Nähragar. Das resultierende Gemisch wird über die Oberfläche einer harten (1,5 bis 1,9% [w / v]) Nähragar-Platte gegossen. Die Platte wird ausreichend, um sicherzustellen, dass die Soft-Agar die gesamte Oberfläche des harten Agar umfasst geschaukelt. Dann wird die Platte auf einer ebenen Fläche angeordnet, bis die obere Agarschicht Zeit gehabt hat, zu verfestigen und anschließend in den Inkubator gestellt werden.
Im Laufe der Zeit eine trübe Suspension von Bakterienzellen, die als Rasen genannten, sichtbar in der Soft-Agar-Medium (7B). Plaques zu bilden, wenn ein Phage infiziert einem der bakteriellen Zellen repliziert in the Zelle, dann lysiert die Zelle freigesetzt zu 100 Nachkommen Phagen (auch bekannt als die Burst-Größe). Die neue Phagenpartikel diffundieren in der Soft-Agar, infizierenden Bakterien in der Umgebung des lysierten Bakterienzellen. Nach mehreren Zyklen von Infektion und Lyse, verschwindet die trübe Bakterienzellsuspension in der Soft-Agar, so dass eine Zone der Lichtung genannt Plaque. Jede Tafel enthält mehr als 10 9 Phagenpartikel, alle genetisch identisch mit dem Original infektiösen Phagenpartikel. Da eine Tafel ergibt sich aus einem einzigen Phagenpartikel, kann die resultierende Anzahl von Plaque bildenden Einheiten (pfu) gezählt werden und eine Konzentration oder Titer, der Phagensuspension berechnet werden kann. Diese Art von Experiment, genannt Plaque-Assay, auch Wissenschaftler ein standardisiertes Verfahren, um in einem Schritt Wachstumskurven zu erzeugen, um Wirtsbereich Spezifität zu untersuchen, und um die Bakterienzellen für genetische Experimente zu transduzieren.
- Bereiten Sie die Indikatorbakterien: einer exponentiell wachsenden Kultur des bakteriellen Wirtsstamm muss für den Soft-Agar-Overlay-Experiment vorbereitet werden. Jeder Host hat seine eigenen Anforderungen an Wachstum. Zwischen 0,3 ml und 0,5 ml einer Suspension von Bakterien vor wird für jede Platte. Wenn eine Flasche Indikatorbakterien hergestellt wird (zB 25 ml), sollte die Kultur in kleineren Mengen (zB 5 ml Aliquots in sterilen Röhrchen mit Schraubverschluss verzichtet) von der Möglichkeit der Kreuzkontamination zu minimieren unterteilt werden. Die Rohre können auf Eis (bei 4 ° C) gehalten werden, bis bereit, in dem Experiment verwendet. Sie aseptisch, um sterile Nährbouillon impfen dann nach den Vorgaben des Bakterienstammes inkubieren. Leftover Kulturen sind am Ende des Tages verworfen werden.
- Bereiten Adsorptionsröhrchen: Zeigen zwei sterile Reaktionsgefäße in einem Rack. Beschriften Sie den Deckel der ersten Röhre "Phagen" und dem Deckel der zweiten Röhre "Control".
- Typischly serielle Verdünnungen von Phagenlysaten vorbereitet und beschichtet in diesem Fall zusätzliche Mikrozentrifugenröhrchen am Rack würde hinzugefügt und markiert mit dem Verdünnungsfaktor.
- Phagen-Stammlösungen sollten frisch aus einzelnen Plaques werden und bei 4 ° C. Um Phagenpartikel disaggregieren, sollten die Materialien bis auf Umgebungstemperatur erwärmt werden, um vor der Durchführung eines Experiments.
- Phage Bestände sollte vorsichtig gehandhabt werden - nicht vortexen oder Pipette kräftig.
- In 50 ul des Phagen Probe zu dem ersten Rohr dann mit 50 ul Phagenpuffer mit dem zweiten Rohr, das als negative Kontrolle für die Plaque-Assay dient.
- Unter aseptischen Bedingungen arbeiten alle Manipulationen von Phagen und Bakterien.
- Als nächstes fügen Sie 500 ul Indikatorbakterien Adsorption an jeden Schlauch.
- Bakterielle Zellen auf dem Boden eines Rohres regeln, wenn sitzt auf Eis oder der Bank für lange Zeiträume. Wenn die Kultur nicht mischened vor dem Entfernen eines Aliquots für das Experiment nicht genug Bakterienzellen Adsorptionsrohr übertragen werden. Es muss eine ausreichende Anzahl von Bakterienkolonien in der Soft-Agar (dh, ein Rasen) zum Nachweis von Plaques gebildet werden. Mit einem Wirbelmischer vorsichtig resuspendieren die Indikator-Bakterien in eine homogene Suspension vor der Übertragung an die Aliquots Adsorptionsröhrchen.
- Mischen Sie die Bakterienzellen und Phagen durch vorsichtiges schnippte die Rohre.
- Inkubieren des Phagen / Bakterien Mischung 15-20 Minuten (nicht länger als 30 Minuten) bei einer geeigneten Temperatur für den Indikator Stamm.
- Bereiten Nährstoff-und Soft-Agar-Agar-Platten hart: Während die Adsorption stattfindet, Platz zwei Soft-Agar-Röhrchen (vorher geschmolzen und gelagert bei 55 ° C) in einem Heizblock bei Ihrem Labortisch bei 46-48 ° C eingestellt
- Das geschmolzene Weichagar Tests müssen die auf die Temperatur des Heizblocks für 10-15 Minuten. Wenn das geschmolzene weichenAgar sitzt länger als 15 Minuten, wird sie beginnen sich zu verfestigen und Klumpen bilden, wenn sie auf dem harten Agar gegossen. Wenn das geschmolzene Weichagar sitzt weniger als 10 Minuten, wird es zu erwärmen, wenn sie dem Phagen / Bakterien zugegeben, töten Wirtszellen vor dem Beschichten. Folglich kann ein Rasen nicht zu bilden, und wenige oder keine Plaques können nachweisbar sein.
- Bereiten Sie zusätzliche Soft-Agar-Röhrchen, wenn Ausplattieren serielle Verdünnungen der Phagen-Lysat.
- Etikett um den Rand des Bodens (nicht der Deckel) von zwei harte Nährstoff-Agar-Platten Platte mit mindestens Ihren Namen, das Datum, die Art der aus dem Nährmedium, und "Phage" oder "Control", die den Adsorptionsröhrchen.
- Fügen Sie zusätzliche Platten mit dem Verdünnungsfaktor, wenn Ausplattieren serielle Verdünnungen bezeichnet.
- Die harten Agarplatten müssen völlig trocken sein, ohne Kondenswasser am Deckel und vorgewärmten auf Raumtemperatur vor der Zugabe des Soft-Agar. Wenn die Platten bei 4 ° C gelagert werden, entfernen Sie sie mehrere Stunden oder sogar das day vor. Breitete sie in kleinen, versetzten Stapeln von nicht mehr als 2-3 Platten und trocknen lassen. Zu hohe Feuchtigkeit in der harten Agarschicht bewirkt Verdünnung der Soft-Agar, so dass Phagen leichter diffundieren durch den Weichagar während der Plaque-Bildung. Folglich wird Plaque-Grösse zu erhöhen.
- Platte Adsorptionsröhrchen ein zu einer Zeit wie folgt: Verwenden Sie einen P1000 Mikropipette zu Überführen Sie den Phagen / Bakterien-Gemisch (sollte etwa 550 ul sein) zu einem Soft-Agar-Röhre dann schnell drehen sie zwischen den Handflächen, den Inhalt zu mischen. Schütteln Sie die Röhre, so dass Luftblasen eingeführt werden. Halten Sie den Schlauch in die linke Hand, öffnen Sie den Deckel eines harten Agar-Platte mit der rechten Hand und sofort gießen Sie den gesamten Inhalt der Tube auf die Oberfläche eines harten Agar-Platte. Vor dem Schließen des Deckels, bewegen Sie die Platte vorsichtig aber rasch auf die geschmolzene Weichagar über die gesamte Oberfläche der Platte verteiltbevor es Zeit hat, sich zu verfestigen. Vermeiden Sie Spritzer des geschmolzenen Weichagar auf den Seiten der Petrischale. Schließen Sie den Deckel.
- Schritt 9 für alle Adsorptionsröhrchen, ein Rohr zu einem Zeitpunkt.
- Legen Sie die Platten auf einer ebenen Fläche und lassen sie ruhig stehen, bis das Soft-Agar verfestigt wird.
- Gewöhnlich 30 Minuten ausreichend.
- Kehren Sie die Platten für die Inkubation.
- Mehrere Platten können gestapelt werden und dann zusammen geklebt invertiert für die Inkubation.
Nach Inkubation können die Platten für Platten, untersucht werden. Die negative Kontrolle sollte nur einen Rasen von Bakterien (keine Löcher indikativ für Plaques). Plaques unterscheiden sich hinsichtlich der Größe, Form und Gesamteindruck. Eine gegebene Phagentyp kann aus einer heterogenen Mischung von Plaques Durchführung, indem Sie die Mitte einer Platte mit einem sterilen Zahnstocher und Übertragen des Inokulums in einem sterilen Mikrozentrifugenröhrchen t isoliert werdenube mit 100 bis 1000 mu l Brühe oder Phagenpuffer. Dieses Lysat kann plattiert mit der oben beschriebenen Verfahrens werden. Mindestens 3 bis 6 aufeinanderfolgenden einzelnen Plaque-Isolierungen sind notwendig, um sicherzustellen, dass eine reine Phagen erhalten wurde. Oft muss das Lysat über einen großen Bereich (10 -1 bis 10 -10) einen Titer, die sich nicht überlappenden Platten produziert auf der Platte verdünnt werden. Die Anzahl hängt von der Größe der Plakette.
6. Replika-Platte Vorgehen: Transfer von Zellen für Screening Mutanten und Auxotrophe
Diese Technik ermöglicht den Vergleich des Zellwachstums auf einer primären Platte zur sekundären Platten, Erzeugen ein Mittel, um Zellen auf einem selektierbaren Phänotyp. Zunächst wird eine primäre oder Master, ist die Platte mit Zellen entweder durch Spread-Plattieren einer Verdünnung, die einzelnen Kolonien produziert oder durch Übertragung auf einem Teller in einem räumlichen Muster durch Gitter Markierungen angegeben geimpft. Sekundäre-Platten, die Medien mit einem Wachstum InhiMonitore oder Medien, die einen bestimmten Nährstoff fehlt, sind mit Zellen von Kolonien auf dem Primär-Platte geimpft. Das räumliche Muster der Kolonien wird zunächst durch Drücken ein Stück Samt an den primären Platte reproduziert. Bakterienzellen Samt halten, weil sie größere Affinität für das Samt als für den Agar haben. Der Abdruck von Zellen auf dem Samt wird dann an mehrere sekundäre Platten mit Zellwachstum sich auf dieselben Muster wie die Kolonie der primären Platte übertragen. Mit anderen Worten, ist es wie mit einem Stempel, die Replikation der Wachstumsmuster von einer Platte zur anderen. Diese Technik ist vorteilhaft, da es eine relativ große Anzahl von Kolonien, die sowohl zu vielen Phänotypen in einem einzigen Experiment untersucht werden können.
- Planen primären Platte: Label um den Rand des Bodens (nicht der Deckel) einer Agar-Platte mit mindestens Ihren Namen, das Datum und die Art der Wachstumsmedium.
- Mark von einem Gitter auf dem Boden der Platte mitmindestens zwei gleichmäßig beabstandeten vertikalen Linien und mindestens zwei gleichmäßig beabstandeten horizontalen Linien. Anzahl der erhaltenen Quadrate.
- Verwenden Sie einen Pre-sterilisierten Zahnstocher, um jedes Quadrat mit einer Zellprobe zu impfen. Für jede Probe, die DAB Mitte des Platzes. Verdecken Sie nicht das gesamte Quadrat mit Zellen (Abbildung 8) oder aber die Probe wird überwuchern, wenn inkubiert und verunreinigen umliegenden Plätzen.
- Jedes Quadrat wird mit einer anderen Probe, die entweder aus Bouillonkulturen oder Kolonien auf einer anderen Platte abgeleitet geimpft werden.
- Umkehren und Inkubation des primären Platte, die verwendet werden, um verschiedene sekundäre Inokulieren werden.
- Beimpfen sekundären Platten: Stapeln Sie die primäre Platte und all die sekundären Platten. Mit einem Marker, um eine Orientierung Markierung auf der Seite der Platten. Achten Sie darauf, die Marke auf der Unterseite jeder Platte, nicht den Deckel.
- Besorgen Sie sich einen sterilen Tuch aus Samt und stelle sie auf den zylindrischen Block (Fbbildung 9). Verriegeln Sie das Tuch aus Samt an Ort und Stelle mit dem Halter. Beachten Sie die Ausrichtung Marke auf den Block.
- Der Block sollte die gleiche Größe wie der Boden einer Petrischale (Durchmesser von 10,2 cm) sein. Es sollte mit einem Sicherungsring gekommen, dass die Klemmen Samt Tuch, um den Block, während Replica-Plating.
- Der Samt Tuch (15,2 x 15,2 cm im Quadrat) müssen im Voraus sterilisiert werden. Stapel 10 oder 12 saubere Quadrate dann wickeln sie in Alufolie dann legen Sie sie im Autoklaven bei 121 ° C auf dem Trocken-Zyklus (Schwerkraft-Einstellung) für 30 Minuten. Bevor Sie sie in einer Replica-Plating Experiment, sicherzustellen, dass sie vollständig trocken sind, indem sie in einem warmen Ofen für mehrere Stunden. Beachten Sie, dass Baumwollsamt Quadrate muss möglicherweise mit Klebeband vor der Sterilisation de-Lintierte.
- Velveteen Quadrate wieder verwendet werden. Nach benutzt Samt Quadrate im Autoklaven dekontaminiert worden sind, sollten sie in klarem Wasser ausspülen und erneut sterilisiert, wie oben beschrieben.
- Der zylindrische Blockk und Schelle sollte zwischen den Anwendungen mit einer kurzen Spülen in 70% (v / v) Ethanol oder 10% Chlorbleiche desinfiziert werden.
- Nehmen Sie den Deckel und invertieren die primäre Platte. Richten Sie die Ausrichtung Markierung auf der Platte mit der Markierung auf dem Block. Senken der Platte, so daß die Oberfläche des Agar in Kontakt mit der Samt Tuch auf den zylindrischen Block ist. Leicht, aber gleichmäßig nach unten drücken mit den Fingerspitzen auf der Rückseite des primären Platte und dann heben Sie vorsichtig die primäre Platte weg aus dem Block. Setzen Sie den Deckel auf dem Teller.
- Der Samt Eindruck von Zellen aus der primären Platte verwendet werden, um etwa 7-8 sekundären Platten vor einem Eindruck mit einer neuen Samt Bedürfnisse einzugehen beimpfen.
- Wiederholen Schritt # 7 mit jedem der sekundären Platten.
- Als positive Kontrolle, sollte die letzte Platte aus der Reihe ein Agar Medium, in dem alle getesteten Stämme wachsen soll sein. So können Sie bestätigen, dass die Zellen können wurden zu allen sekundären pl übertragenZwischenstufen der Serie. Andernfalls kann fehlende Wachstum auf einem bestimmten Prüfmedium unzureichender Zell-Transfer und nicht als Phänotyp des Stammes zugeschrieben werden.
- Um Fehlalarme zu vermeiden, sollten die sekundären Platten aus den am wenigsten zu günstigsten Substrat bestellt werden. Andernfalls könnte Nährstoffen zwischen den Platten ermöglicht den Zellen, auf einer ungünstigen Medium wachsen übertragen werden.
- Kehren Sie die Platten und inkubieren.
- Hinweis: Bei der Inspektion der sekundären Platten für Wachstum, achten Sie darauf, zwischen Wachstum und einem Aufdruck zu unterscheiden. Letzteres ist ein negatives Ergebnis.
7. Bereinigen des Work Space
- Schalten Sie den Bunsenbrenner dann weglegen alle Lieferungen einschließlich Kulturen auf Platten oder in Röhren, extra Medien, und andere Reagenzien.
- Lassen Sie keine alten Kulturen, sei es auf Platten oder in Röhren, um auf dem Labortisch oder in Lagerräumen zu akkumulieren. Diese Proben, die berüchtigten Quellen der Verschmutzung sindwie Formen und unerwünschte Bakterien-Spezies, sollte, sobald sie nicht mehr benötigt werden verworfen.
- Ort kontaminiert geräte (Handschuhe, Pipettenspitzen, Kimwipes), Glas (Tuben, Fläschchen, Flaschen), und gefährliche Abfälle (Bakterienkulturen oder Phagen-Lösungen, verwendet Platten) in die ordnungsgemäße Entsorgung Behälter. Bei der Arbeit mit einem nicht-pathogenen E. coli-Stamm (BSL-1), wird nur nicht-infektiösen gefährlichen Abfällen. Ziehen Sie bei diesen gleichen Verfahren mit pathogenen Organismen (BSL-2 oder höher), ist ansteckend gefährlichen Abfällen. Unabhängig von Biohazard Einstufung, müssen gefährliche Abfälle autoklaviert oder desinfiziert werden, bevor es verworfen wird. Folgen Sie den Richtlinien in der BMBL beschrieben (5 th Ed.) Sowie jene von Ihrem institutionellen Environmental Health and Safety-Abteilung für die sofortige und sachgerechte Entsorgung der biologischen Gefahren während eines Experiments erzeugt zur Verfügung gestellt.
- Reinigen Sie den Arbeitsbereich mit einem Desinfektionsmittel.
- Hände waschengründlich mit antiseptischen Seife und warmem Wasser vor dem Verlassen des Labors.
8. Repräsentative Ergebnisse
Streak-Plate-Technik. Eine Beispielanwendung für streifenfreie Beschichtung wird in Abbildung 1 dargestellt. Dieses Verfahren wird zur Isolierung von Bakterien-Kolonien aus gemischten Zellkulturen verwendet und ist mit Abstand einer der wichtigsten Techniken, um Meister in der Mikrobiologie und Molekulargenetik. Jede Kolonie für eine Population von Zellen, die genetisch identisch sind. Für viele Anwendungen stromabwärtigen ist es unerlässlich, entweder mit einer einzelnen Kolonie oder einem reinen Bakterienkultur durch Inokulieren Medien mit Zellen aus einer einzelnen Kolonie erzeugt starten. Zum Beispiel kann die Morphologie der einzelnen Zellen innerhalb einer Kolonie untersucht unter Verwendung eines Lichtmikroskops werden. Genetischen Identität kann durch Sequenzierung der kleinen ribosomalen Untereinheit-RNA-Gen aus genomischer DNA aus einer Zellkultur mit einer einzelnen Kolonie isoliert begonnen zugeordnet werden.Und metabolische Eigenschaften können durch Unterwerfen Zellen auf verschiedene biochemische und physiologische Untersuchungen beschrieben. Nur durch die Durchführung solcher Experimente mit Reinkulturen kann man sicher sein, von den Eigenschaften zugeschrieben zu einem bestimmten Mikroorganismus. Die Ergebnisse sind nicht von der Möglichkeit, dass die Kultur verunreinigt ist verdeckt. Technische Fehler kann auftreten, wenn die Sterilität des Instruments verwendet, um die Zellen Streifen quer über die Platte wird nicht während des gesamten Verfahrens beibehalten. Vergessen, Flamme eine Schleife oder rufen Sie eine frische Zahnstocher zwischen den Quadranten es schwierig, einzelne Kolonien zu erhalten machen. Einige Bakterienarten können nicht in Reinkultur isoliert werden, da sie abhängig von einer kooperativen Zusammenarbeit mit einem anderen Bakterien-Spezies für bestimmte Anforderungen Wachstum sind. Wird als syntrophs, kann diese Organismen nur unter Co-Kultur Bedingungen gezüchtet werden, so Kolonien (wenn sie gebildet werden) stets von zwei oder mehr Arten bestehen. Eine weitere Herausforderung begegnet im Labor bei der Durchführung derStreak-Plate-Verfahren mit Bakterien aus Umweltproben abgeleitet ist, dass die Zellen das Wachstum Eigenschaften, die von den traditionellen Labor-Stämme wie E. abweichen zeigen coli. Solche Bakterienstämme können Kolonien, die filamentösen sind (im Gegensatz zu engen Cluster von Zellen gegenüber) mit Niederlassungen, die über einen großen Abschnitt von einer Agar-Platte verteilt produzieren, verkalkt und damit feuerfeste das Durchdringen eines Streifen-Platte Instrument, oder umgeben von einer klebrigen Kapsel so dass einzelne Kolonien können nicht erkannt werden. Diese Eigenschaften machen es schwierig, einzelne Kolonien durch den Streifen-Plate-Technik zu reinigen.
Pour-Plate-Technik. Mit dem Pour-Plate-Technik bilden die Kolonien im Agar sowie auf der Oberfläche des Agarmediums wodurch ein bequemes Mittel, um die Anzahl an lebenden Zellen in einer Probe zu zählen. Dieses Verfahren wird in einer Vielzahl von industriellen Anwendungen verwendet. Zum Beispiel ist es für eine Abwasser treatment Anlage, die verantwortlich für die Reinigung bis flüssige Abfälle (z. B. Abwasser-, Run-off aus Gullys), das durch häuslichen, gewerblichen und industriellen Immobilien sowie landwirtschaftliche Praktiken, um Wasserproben nach dem umfassenden Reinigungsprozess zu analysieren ist. Abwasser (Nicht-Trinkwasser) wird in vielfältiger Weise wiederverwendet werden - für die Bewässerung von Non-Food-Pflanzen in der Landwirtschaft, für Hygienespülung in Wohnungen, und in der industriellen Kühltürmen - also muss es frei von chemischen und mikrobiellen Kontamination. Trinkwasser (Trinkwasser) muss nach der EPA-Standards gereinigt werden und wird mit Hilfe von mikrobiologischen Methoden, die Beschichtung Aufzählung der spezifischen menschlichen Pathogenen erlauben. Dargestellt in Abbildung 10 sind Bakterienkolonien, die aus Bakterien-Zellen präsentieren in einer Wasserprobe aus einem öffentlichen Trinkbrunnen gesammelt. Es ist unwahrscheinlich, bakterielle Erreger produziert diese Kolonien angesichts der Reinigungsmaßnahmen für Trinkwasser, jedoch sind alle Mikrobenwo und Verunreinigung durch selbst nicht-pathogenen Stämmen kann nur minimiert werden, nicht vollständig eliminiert werden. Als weiteres Beispiel, muss ein pharmazeutisches Unternehmen, um den Grad der mikrobiellen Kontamination oder Kontamination, eines neuen Arzneimittels während der Produktion, Lagerung und Transport zu beurteilen. Durch Abtasten des Arzneimittels während der verschiedenen Phasen des Prozesses und Plattieren von Proben unter Verwendung des Ausguss-Platte Verfahren, die mikrobielle Belastung, oder die Anzahl der kontaminierenden Bakterien, kann leicht bestimmt werden. Vorsorgliche Massnahmen können dann entwickelt, um minimieren oder zu beseitigen mikrobielle Kontamination sein. Einer der häufigsten technischen Fehler, die bei der Durchführung des Pour-Plate-Technik auftritt, ist unzureichendes Mischen der Probe mit der geschmolzenen Agar verursacht Kolonien zu verklumpen wodurch Platte zählt ungenau. Ein weiterer häufiger Fehler ist Gießen der geschmolzenen Agar, wenn es zu heiß ist, töteten viele der bakteriellen Zellen in der Probe. Dieser Fehler wirkt sich auch die Genauigkeit der Platte zählt geben Zahlen, die unter-stellen the Gesamtzahl von koloniebildenden Einheiten in der Probe.
Spread-Plate-Technik. Der Spread-Plate-Technik ist analog zu der Platte pour-Verfahrens in seiner Nützlichkeit als Mittel, um tragfähige Platte Zählungen durchführen. Da jedoch die Kolonien, die mit der Ausbreitung-Plate-Technik zu bilden gleichmäßig über die Oberfläche des Agarmediums verteilt sind, können Zellen aus einzelnen Kolonien isoliert und in den nachfolgenden experimentelle Manipulationen verwendet werden (zB als Inokulum für einen Streifen-Platte oder eines Bouillonkultur). Drei gemeinsame Anwendungen, bei denen die Ausbreitung-Plate-Technik ist ein wichtiger Bestandteil sind die Anreicherung, Selektion und Screening-Experimenten. In allen drei Anwendungen kann die gewünschten Zelltyp aus dem Gemisch abgetrennt werden und später auf eine beliebige Anzahl von biochemischen, physiologischen oder genetischen Tests unterzogen.
Eine Bereicherung Experiment beinhaltet Plattieren einer Mischkultur auf einem Medium oder Inkubation Platten in eUmgebungsbeanspruchungen das Wachstum dieser Mikroorganismen in der Probe, die die gewünschten metabolischen Eigenschaften Wachstumseigenschaften oder Verhaltensweisen zeigen, zu begünstigen. Diese Strategie nicht hemmt das Wachstum von anderen Organismen, sondern führt zu einer Erhöhung der Zahl der gewünschten Mikroorganismen im Vergleich zu anderen in der Kultur. So sind die Kolonien, die auf einer Anreicherung Platte bilden wahrscheinlich phänotypischen Eigenschaften aufweisen, die den gewünschten Genotyp zu reflektieren. Zum Beispiel wird, wenn Ihr Ziel ist es, Stickstoff-fixierenden Bakterien aus Umweltproben, die eine Mischung von mehr als 1000 verschiedene Bakterienarten zu kultivieren, dann Plattieren der Probe auf einem Stickstoff-defizitären Mediums für jene Bakterien, die diese Verbindung aus der produzieren kann bereichern Atmosphäre mit metabolischen Fähigkeiten durch eine Reihe von Genen, die für die Befestigung erforderlich Stickstoff zur Verfügung gestellt.
Eine Auswahl Experiment wird Plattieren eines gemischten Kultur auf einem Medium, das nur jene Zellen, die con ermöglichtTain ein bestimmtes Gen oder eine Gruppe von Genen zu wachsen. Diese Art von Test ist häufig in molekularbiologischen Labors bei der Transformation Bakterienstämme mit Plasmiden, die Antibiotika-Resistenz-Gene. Wenn Ihr Ziel ist es, nur rekombinanten Zellen, oder solche, die erfolgreich nahm das Plasmid, dann Plattieren der Probe auf einem Medium, das mit einer entsprechenden Konzentration des Antibiotikums wurde ergänzt wird für jene Zellen, die eine Resistenz gegenüber diesem speziellen Medikament auswählen zu kultivieren.
Ein Screening-Experiment wird Plattieren eines gemischten Kultur auf einem Medium, das alle lebensfähigen Zellen wachsen können, wobei jedoch die Zellen mit der gewünschten Genotyp von anderen Zellen basierend auf ihren Phänotyp zu unterscheiden. Wiederum ist diese Art von Experiment gemeinsamen in molekularbiologischen Labors bei der Durchführung Mutagenese-Assays oder Klonieren von Genen in Plasmiden. Ein klassisches Beispiel, wie in 11 gezeigt ist, macht die Verwendung des lacZ-Gens EncoderDing β-Galactosidase; dieses Enzym erlaubt den Zellen zu metabolisieren X-Gal (5-Brom-4-chlor-3-indolyl-β-D-galactopyranosid), ein Substrat Analogon sein natürliches Substrat, Lactose. Spaltung von X-Gal durch β-Galactosidase zu einer unlösliches blaues Produkt. Wenn also ein Medium enthält X-gal und eine Probe mit Zellen entweder mit einem Wildtyp-(funktionellen) oder eine Mutante (nicht funktionsfähig) lacZ-Gen werden auf diesem Medium ausplattiert, dann nach der Inkubation Wildtyp-Zellen, die eine funktionelle lacZ Gen wird als blau-pigmentierte Kolonien erscheinen, während Mutanten-Zellen mit einem nicht-funktionellen lacZ-Gen als unpigmentierte ("weißen") Kolonien erscheinen.
Ein technisches Problem festgestellt am häufigsten, wenn erst zu lernen, wie man die Ausbreitung-Plate-Technik führen wird ungleichmäßige Verteilung der Zellen über die Agar-Oberfläche. Bei der Verwendung eines Plattenspielers und Glasstab, kann die Probe zu schnell, so dass die Kolonien nur in der Nähe der Mitte der Platte zu bilden absorbiert werden. WHenne macht die "Copacabana-Methode", werden die Glasperlen verwirbelt und nicht geschüttelt über die Agar-Oberfläche. Folglich, wachsen zu viele Kolonien entlang des äußeren Randes der Platte. In beiden Fällen wird die resultierende Verteilung der Kolonien nicht nutzen Sie die komplette Fläche zur Verfügung, so können Zellen verklumpen und wachsen in überlappende Kolonien machen Plattenzählungen ungenau oder Unterscheidung von Zelltypen nicht machbar.
Soft-Agar-Overlay-Technik. Ein Verfahren, verwandt mit dem Spread-Plate-Technik verwendet, um Bakterienkolonien zählen können, um tally die Zahl der Phagen verwendet werden. Während zwischen 30 und 300 bakteriellen Zellen über die Agaroberfläche für Keimzahlen (cfu / ml) verteilt sind, sind zwischen 100 und 400 infektiösen Phagen Teilchen mit 10 August-10 September Wirtszellen für die Plaque-Zählungen (pfu / ml) in einer Schicht gemischt Weichagar über die Oberfläche des harten Nährstoff-Agar. Sofern nicht anders gezeigt, ist es im Allgemeinen angenommen, tHat eine einzelne Bakterienzelle teilt sich und sammelt sich eine große Zahl von genetisch identischen Zellen in einem einzigen Cluster bezeichnet eine Kolonie. Wie zuvor beschrieben, ist diese Annahme nicht gültig, wenn die Zellen wachsen in Bündeln (dh, paarweise, Tetraden, Ketten oder Cluster) oder anzeigen Wachstumseigenschaften wie Kapseln, die einzelne Kolonie-Bildung zu verhindern. Eine ähnliche Annahme für die Plaquebildung wird, daß jede Tafel darstellt Aktivität eines einzelnen Phagen. Diese Aussage ist nur wahr, wenn ein Phage ein Bakterium infiziert. Was passiert, wenn mehrere Phagenpartikel ein einzelnes Bakterium infizieren? Dieses Problem betrifft eine wichtige statistische Parameter, die bei der Durchführung von Experimenten mit dem Phagen werden müssen - Multiplizität der Infektion (MOI) - beschreibt das Verhältnis von infektiösen Phagenpartikeln der Anzahl von Wirtszellen in einer Probe. Da einige Zellen adsorbiert mehr als ein Phage, während andere Zellen adsorbiert nur eine oder keine Phagen, sollte eine Population von Wirtszellen bei einer niedrigen MOI (≤ 1) t infizierto Minimierung der Wahrscheinlichkeit, dass eine Zelle mehr als ein Phagenpartikel infiziert werden. Unter Verwendung der Plaque bildende Einheiten (PBE) als funktionelle Definition vermeidet diese Komplikationen bei der Durchführung Plaque zählt, um den Titer eines Phagen-Stammlösung zu berechnen.
Wie in 12 gezeigt, variiert Plaque-Morphologie für verschiedene Phagen. Einige Phagen erzeugen kleine Plaques (Feld A), während andere führen zu großen Plaques (Panel B). Eine Reihe von Variablen beeinflussen Plaquegröße. Es gibt technische Gründe, die zu dieser Variabilität beitragen. Zum Beispiel komplette Medien-und dicken harten Agar Unterstützung Entwicklung von größeren Plaques weil Wirtszellen können Phagen-Wachstum für einen längeren Zeitraum aufrecht zu erhalten. Eine hohe Beschichtungs-Dichte von Wirtszellen (> 10 9 KBE pro Platte) führt zu einer Verringerung der Plaque-Größe. Bei geringeren Konzentrationen von Weichagar wird die Geschwindigkeit der Phagenpartikel Diffusion in den Weichagar zu erhöhen und dadurch die Größe der Plaques. Erinnern wir thbei dieser erhöhten Diffusionsgeschwindigkeit unbeabsichtigt auftreten, wenn die harte Agarplatten nicht ganz trocken sind, dass Kondensation oder überschüssige Feuchtigkeit in die Schüssel verdünnt das Weichagar im Overlay. Diese technische Aufsicht werden inkonsistente Ergebnisse in Bezug auf Plaque-Größe für einen bestimmten Phagen zu produzieren.
Plaque-Grösse ist auch mit einer Reihe von Ereignissen Wirtszelle einschließlich der Effizienz der Adsorption, der Dauer der Latenzzeit (die Zeitspanne von Phagenadsorption der Lyse der Wirtszelle), und der Burst-Größe (die Anzahl von Nachkommen-Freigabe durch Suchbegriffe eine einzelne Infektion). Heterogenes Gemisch von Plaque-Grössen kann beobachtet werden, wenn Phagenpartikel Wirtszellen infizieren in verschiedenen Phasen des Bakterienwachstums werden. Zum Beispiel machen, dass diejenigen, adsorbieren während der frühen exponentiellen Phase größere Plaques mit mehr Nachkommen als die Phagen adsorbieren, dass in späten exponentiellen Phase. Als allgemeine Regel gilt, produzieren lytischen Phagen klare Plaques während lysogenen Phagen Form trübe Plaques. HoWever, produzieren einige lytischen Phagen interessante Muster wie der "Volltreffer" Plaque in 12B gezeigt. Diese klare Plaques werden durch ein trübes Halogen umgeben, weil diese Zellen am Rand der Platte nicht vollständig lysiert werden oder können Phagen resistent gegen eine Infektion. Ein "Volltreffer"-Muster mit gemäßigten Phagen beobachtet wurde, ist eine Plakette mit einer trüben Zentrum durch einen klaren Ring umgeben. Diese Morphologie spiegelt das MOI und die Physiologie der Wirtszelle, die mit Bezug auf die Lyse-Lysogenie Entscheidung. Wenn die Zellen zunächst mit Phagen infiziert, ist die niedrige MOI und Zellen wachsen schnell, weil Nährstoffe reichlich vorhanden sind, gemeinsam das erleichtert lytische Wachstum. Da mehr und mehr Zellen aufgelöst werden, erhöht das Trägheitsmoment und eine klare Formen Plaque. Lysogene in der Mitte der Plakette jedoch weiter wachsen, weil sie immun gegen Lyse, die zu einer klaren Plakette mit einer trüben Zentrum sind.
Die Overlay-Technik kann für Plaque-Assays mit eukaryotischen Viren verändert werden. Indie gleiche Weise Bakteriophagen Form Plaques auf einem Rasen von bakteriellen Zellen in Weichagar bilden eukaryotischen Viren Plaques auf einer Monoschicht von Zellen von einem Gel bedeckt. Eine Monoschicht ist ein Bogen von konfluenten Zellen, die nebeneinander auf der Oberfläche einer Kulturschale, einander zu berühren, aber nicht immer auf voneinander. Zur Durchführung dieser Art von Plaque-Assay werden Aliquots Virus auf empfängliche Monoschichten von eukaryotischen Zellen gegeben. Danach wird die Monoschicht mit einem Agarose-basierte Nährmedium bedeckt - dieses Gel schränkt die Verbreitung von Nachkommen Viren aus infizierten Zellen zu benachbarten Zellen freigesetzt wird in der Monoschicht. Dementsprechend wird eine kugelförmige Fläche, oder Plaque, erzeugt, das Zellen durch Freisetzung von Virionen beschädigt. Um die Visualisierung der Plaques, Farbstoffe, dass Fleck lebenden Zellen in das Zellkulturmedium einen Kontrast zwischen infizierten und nicht infizierten Zellen angewendet werden kann helfen.
Die Soft-Agar-Overlay-Technik wird für Experimente anderer als Plaque-Assays verwendet. Erstens ist es splätzen zu erinnern, dass die harte Nähragar ein Support-Matrix, die das Wachstum von Bakterien erlaubt ist. Zweitens kann der Soft-Agar für die Überlagerung verwendete eine andere Nährstoffzusammensetzung als die harte Agar. Auf diese Weise kann der Soft-Agar als Mittel zur Assay Bakterienstämme für verschiedene Wachstumseigenschaften oder metabolischen Eigenschaften dienen. Zum Beispiel wird die Overlay-Verfahren zu screenen Bakterien für die Fähigkeit, Cellulose (Teather und Holz 1982) abzubauen verwendet. Einzelne Kolonien werden auf einer nicht-selektiven harten Agarmedium dann weich-Agar, enthaltend 0,1% (w / v) Carboxymethylcellulose (CMC) über die Oberfläche des harten Agar gezüchtet wird. Nach der Inkubation werden die Platten mit Flecken, die Visualisierung von Zonen der Lichtung erlaubt um die Kolonien in der Soft-Agar überflutet. Der Löschvorgang ist durch hydrolytische Enzyme von den Bakterien den Abbau der Cellulose in das Medium sekretiert verursacht. In jüngerer Zeit hat die Overlay-Technik wurde verwendet, um Bakterien, die das Wachstum hemmen methanoge erkennennic Archaea im Pansen der Tiere (Gilbert et al. 2010) gefunden. Die bakteriellen Isolate aus Umweltproben auf einer Festplatte Agarnährmedium dann Kolonien werden mit Soft-Agar mit einer Kultur von methanogenen überlagert gewachsen. Nach der Inkubation werden die Platten für die Zonen der Wachstumshemmung um die Kolonien untersucht. Diese Methode identifiziert Bakterienstämme, die Inhibitoren der Methanbakterien in der Soft-Agar zu produzieren.
Die häufigsten technischen Fehler, die mit der Soft-Agar-Overlay-Technik auftreten, werden dem Gießen des geschmolzenen Soft-Agar, wenn es entweder zu heiß oder zu kalt ist. Wenn es zu heiß ist, die Bakterienzellen in dem Medium gemischt wird vor dem Beschichten getötet werden. Wenn es zu kühl ist, wird die Soft-Agar-Agar bildet Klumpen, wenn auf der Festplatte Agar gegossen. In beiden Fällen werden die Ergebnisse nicht eindeutig oder unlesbar am besten.
Replica-Platte Vorgehensweise. Übertragen Kulturen von einer Art von Nährmediumzum anderen, um das Wachstum zu testen wird sehr mühsam, wenn es mehr als nur ein paar Stämme sind. Replika-Plattierung ist ein Verfahren, das gleichzeitige Screening einer großen Anzahl von Mikroorganismen erlaubt. Zum Beispiel, nach Mutagenese eine Kultur von Wildtyp-Zellen, kann man verbreitet-Platte Verdünnungen der Kultur auf Platten mit einzelnen Kolonien zu erhalten. Die primären Platten enthalten ein Medium, das Wachstum aller Zellen unterstützt, einschließlich Wildtyp-prototropher, die alle für das Wachstum notwendig Verbindungen zu synthetisieren, und mutierten Auxotrophe, die eine genetische Mutation in einem Biosyntheseweg wodurch sie nicht auf bestimmte Verbindungen essentiell für das Wachstum synthetisieren tragen. Durch Beschichten der Mischung von Zellen auf einem vollständigen Medium, können die fehlenden Nährstoffe aus der Umgebung entnommen werden. Um zwischen prototropher und Auxotrophen zu unterscheiden, können die Kolonien auf einen Minimal-Medium repliziert werden. Nur prototropher in der Lage zu wachsen. Da das räumliche Muster der primären Platte erhalten bleibt, Comparisauf der sekundären Platte mit der primären Platte ermöglicht die Identifizierung von Mutantenkolonien. Um zu bestimmen, wobei die Verbindung die Mutanten nicht mehr synthetisieren können, können die Kolonien auf Minimalmedium mit spezifischen Verbindungen (z. B. Aminosäuren, Kohlenstoffquellen, Vitamine etc.) ergänzt repliziert werden. Auf diese Weise können Hunderte von Kolonien zur gleichen Zeit mit dem Replika-Platte Prozedur durchmustert werden. Ein technischer Fehler, die auftreten könnten wird mit Agar-Platten, die zu feucht sind, was zu Kolonien zusammen zu schmieren kontaminierende alle Kulturen auf der Platte. Dies führt zu Ergebnissen, die völlig unzuverlässig sind. Ein weiterer technischer Fehler wird zu viel Druck bei der Übertragung von Zellen aus der Samt auf die sekundären Platten. Auch nach dem Inkubieren der sekundären Platten, können die resultierenden Kolonien überlappen Herstellung Wachstumsphänotypen zurückzuführen, um eine Kontamination nicht Auxotrophie.
Nicht alle Wildtyp-Mikroben-Spezies sind prototropher, so die Replik-pEnde der Prozedur kann verwendet werden, um gleichzeitig zu screenen verschiedenen Wildtyp-Stämme charakteristisch für Wachstum Anforderungen werden. Wie in Abbildung 13 dargestellt, "Tupfen" von Zellen aus vier verschiedenen Pseudomonas Bakterienstämme wurden in zweifacher Ausfertigung auf einem Gitter versehenen Platte, die komplette Medien genannt YTA überzogen (Abb. A). Die Stämme wurden dann auf drei sekundären Platten repliziert (Abbildung B, C und D) der Minimal-Medium (MSA) mit einer anderen Kohlenstoffquelle (Acetamid, Lactose und Glycin, jeweils) ergänzt zusammengesetzt. Die Ergebnisse zeigen, dass zwei der vier Pseudomonas-Stämme (P. aeruginosa und P. stutzeri) unfähig, wachsen auf diesen drei Kohlenstoffquellen sind. Als Kontrolle wurden die Stämme auf eine vierte Platte mit YTA Medium vermehrt, um zu bestätigen Zellen während des gesamten Verfahrens übertragen wurden. Da alle vier Stämme auf der Platte wachsen YTA Kontrolle zeigten die Defizite auf das Wachstum der letzten drei Platten in der Serie sind zuverlässig. Die Replik-Plattierung Ergebnisse sind in Tabelle 1 aufgeführt. Ein Fehler häufig gemacht wird, interpretieren einen Abdruck des Wachstums auf einem sekundären Platte als positives Ergebnis. Vergleichen Sie beispielsweise den Phänotyp von P. aeruginosa dem von P. stutzeri auf MSA + acetamid (Panel B). Diese zeigt einen Abdruck des Wachstums, der ein negatives Ergebnis ist, und kann auftreten, wenn Nährstoffe von der vorherigen Platte mit den Stammzellen übertragen werden. Kein Wachstum neuer Zellen tritt auf, weil die fehlenden Nährstoffe nicht zur Verfügung stehen, um Nachkommenschaft Zellen. Es ist leicht, einen Abdruck mit dem tatsächlichen Wachstum zu verwirren. Wenn Sie Zweifel haben, soll der Versuch wiederholt unter Verwendung einer alternativen Methode, wie z. B. Streifen-Plating-Zellen aus der primären Platte auf sekundäre Medien werden.
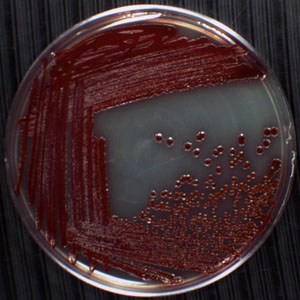
Abbildung 1. Beispiel für einzelne Kolonien auf einem Teller. Die rosafarbene Kugeln in der Nähe der Mitte der Platte sind Kolonien von Serratia marcescens, ein Gram-negativen, stäbchenförmigen Proteobakterium in der Familie Enterobacteriaceae. Aufgrund seiner Vorliebe für feuchte Umgebungen wird diese Mikroorganismen häufig gefunden wachsenden in den Ecken von Badewannen, Waschbecken in Becken, in Fugenmörtel, und auf Duschvorhänge. S. marcescens ist leicht zu erkennen, da sie eine rötliche Pigment namens Prodigiosin produziert. Die Kolonien auf dieser Platte wurden unter Verwendung des Streifen-Plate-Technik, mit Einzelkolonien in dieser vierten Quadranten nach Inkubation bei 30 ° C für 24 Stunden. Die anderen drei Quadranten für konfluenten Wachstum der Zellen auf der Agaroberfläche in überlappende Kolonien entwickelten abgeschieden.

Abbildung 2. Instrumente für Streak-Plate-Technik verwendet. Von oben nach unten, dargestellt sind Zahnstocher (abgeflachte nicht rund), eine Drahtschlinge, eine Einweg-Kunststoff-Schleife, und Holzstöcken. Zahnstocher sind in der Regel übertragenzu einem kleinen Becherglas mit dem breiten Ende nach unten, dann mit Folie abgedeckt, wenn autoklaviert, um vor sterilisieren zu bedienen. Holzstäbe sind mit 18 mm Reagenzgläser anschließend autoklaviert, um vor dem Gebrauch sterilisiert werden übertragen.
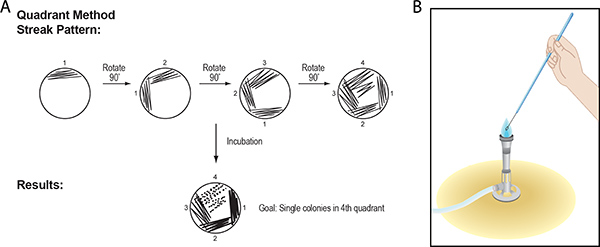
Abbildung 3. (A) Streak-Plate-Technik unter Verwendung des Quadranten-Methode. Ein Pre-sterilisierten Schleife wird Stick oder Zahnstocher benutzt, um die Probe über ein Viertel der Agar-Oberfläche mit einer schnellen, glatten, Hin-und-vierten Gang von der Felge auf die Mitte der Platte verteilt. Diese Aktion wird für jeden der vier Quadranten der Platte wiederholt. Nach der Inkubation wird das Zellwachstum auf dem Weg des Instruments verwendet werden, um die Zellen auf der Platte ablagern. Mechanische Trennung von Zellen in einer Mischprobe mit Hilfe dieser Technik sollten in einzelnen Kolonien im vierten Quadranten führen (siehe Abbildung 1 für ein Beispiel). Einzelne Kolonien werden als Kolonie-bildende Einheiten bezeichnet (cfu). (B) Wenn ein Metall-Schleife für streifenfreie Beschichtung verwendet wird, muss es sterilisiert mit der Flamme eines Bunsenbrenners vor mit dem Inokulum oder der Agar-Medium in Kontakt treten. Daran erinnern, dass der heißeste Teil der Flamme die Spitze des blauen Kegels ist. Halten Sie den Handgriff des Instruments, platzieren Sie den Draht in die Flamme ca. 3-4 cm von der Schleife. Lassen Sie es lange genug für den Draht zu Red Hot geworden. Bewegen Sie den Draht, so dass die Flamme nähert sich der Schleife. Achten Sie darauf, das Metall-Schleife vor dem Berühren des Inokulums gekühlt wird.

Abbildung 4. Pour-Plate-Technik. (A) Ein kleines Volumen der Probe (zwischen 0,1 bis 1,0 ml) wird unter aseptischen Bedingungen in eine leere, aber sterilen Petrischale mit der Verwendung einer 5,0 ml serologische Pipette abgegeben. (B) geschmolzen Agar äquilibriert auf eine Temperatur von etwa 48 ° C wird dann in der Schale mit der Probe gegossen. Nach dem Schließen des Deckels wird die Platte vorsichtig geschwenkt, um die Probe zu mischen undgeschmolzenen Agar. Der Agar darf für etwa 30 Minuten Platten werden dann für die Inkubation invertiert zu verfestigen.
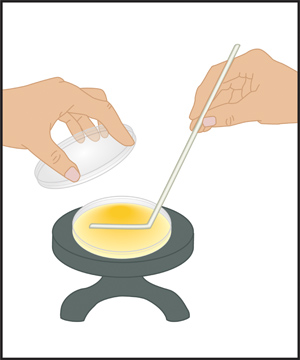
Abbildung 5. Spread-Plate-Technik mit einer Drehscheibe und Glas-Spreader. Nachdem der Agar Platte auf einem Drehteller angeordnet ist, ein kleines Volumen der Probe (0,1 bis 0,2 ml) wird unter aseptischen Bedingungen auf die Mitte der Platte mit einer Mikropipette verzichtet. Der Streuer ist durch Eintauchen in ein Becherglas von Ethanol und übergeben Sie ihr dann durch die Flamme des Bunsenbrenners, um überschüssiges Ethanol entzünden sterilisiert. Vor Kontakt mit der Probe, sollte der Streuer durch Berühren der Agar in der Nähe des Randes der Platte gekühlt werden. Der Streuer ist sanft hin und her durch die Probe über die Platte bewegt wird, während die Drehscheibe langsam dreht. Diese Aktion ermöglicht es schrittweise, aber auch die Verbreitung der Probe auf der Agar-Oberfläche. Nach dem Schließen des Deckels, sollte die Platte auf der Tischplatte u eingestellt werdenndisturbed für mindestens 5 Minuten, damit sich die Probe vollständig zu absorbieren, in den Agar vor dem Umdrehen der Platte für die Inkubation.
6. Spread-Plate-Technik mit Glasperlen (Copacabana Verfahren). Glasperlen, die in einem Autoklaven sterilisiert werden vorab auf der Oberfläche einer Agar-Platte sitzt auf der Tischplatte gegossen. Ein kleines Volumen der Probe (100 bis 150 ul) wird unter aseptischen Bedingungen auf die Mitte des Agars mit einer Mikropipette verzichtet. Mit der Deckel der Platte geschlossen, eine horizontale Schüttelbewegung wird verwendet, um sanft bewegen die Perlen hin und her über die Platte 6-7 mal, die Verbreitung der Probe. Diese Aktion wird nach Drehen der Platte 60 ° wiederholt. Die Schüttelbewegung wird ein drittes Mal mit einem anderen 60 ° Drehung wiederholt. Nachdem die Probe vollständig in den Agar-Medium absorbiert wird, werden die Kügelchen aus in ein Becherglas mit 10% Bleichmittel gegossen. DiePlatten werden dann zur Inkubation invertiert.
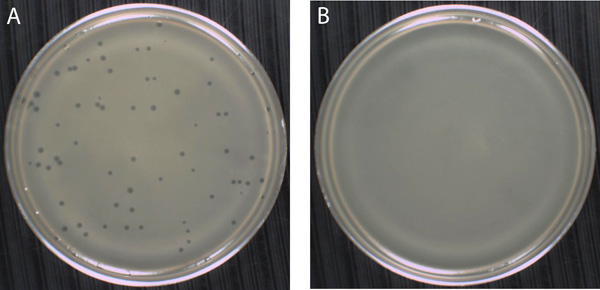
7. Soft-Agar-Overlay-Verfahren zur Isolierung und Quantifizierung von Phagen an die Bildung von Plaques (auch als Plaque-Assay) bezogen. (A) Die Anwesenheit des Phagen können als Zonen Clearing, oder Plaques nachgewiesen werden, auf einer konfluenten bakteriellen Suspension von Kolonien, die in der Soft-Agar. Phage T4 ist eine bösartige, doppelsträngigen DNA-Phagen, der Host, Escherichia coli, wodurch die Wirtszellen zu lysieren und lassen Nachkommen Phagen infiziert. Nach mehreren Runden der Infektion und Lyse, die benachbarte E. coli-Zellen in der unmittelbaren Umgebung des ursprünglichen infizierten Wirtszelle verlassen verschwindet eine Plakette mit Milliarden von T4-Phagen Partikel. Phage T4 erzeugt Plaques, die etwa 1 mm im Durchmesser sind. In diesem Experiment wurden 200 ul einer 10 -5 Verdünnung einer 2 x 10 8 pfu / ml Lager des Phagen T4wurde mit etwa 300 ul E. gemischt coli-Zellen als Indikator einer exponentiell wachsenden, belüfteten Kultur bei 37 ° C vorbereitet Sowohl die Phagen und die Bakterien wurden auf ein EHA Weichagar Rohr, gemischt, dann auf die Oberfläche eines harten EHA Agarplatte gegossen zugegeben. Beachten Sie, dass es nicht notwendig war, damit Phagen und Bakterien zu adsorbieren, bevor sie mit einem Überzug in diesem Fall. Nachdem die Weichagar zu verfestigen für 20 Minuten ungestört, wurden die Platten umgedreht und bei 37 ° C für 24 Stunden inkubiert. (B) Da der infizierenden Phagen Teilchen, Bakterienwachstum entsteht eine trübe Suspension von Zellen in der Soft-Agar, in denen diskrete Kolonien nicht sichtbar sind. Stattdessen E. eine gerade Rasen von Bakterienzellen, in diesem Fall coli, Formulare über den gesamten Soft-Agar-Schicht.
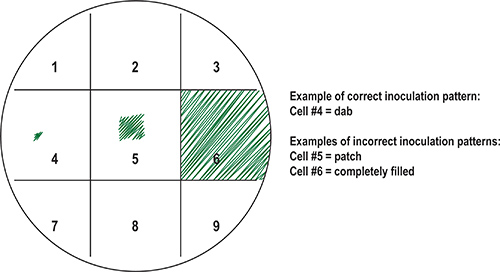
Abbildung 8. Herstellung von primären Platte (Master) mit bakteriellen Proben. Um Proben organisiert zu halten, kann die Unterseite der Platte in ein Raster markiert werden und die daraus resultierenden Plätze nummeriert. Jede Probe kann auf einen Platz am Gitter zugeordnet werden. Gezeigt werden Beispiele für richtige Impfung gegen falsche Muster. Idealerweise wird eine kleine Anzahl von Zellen in die Mitte des Quadrats mit einem sterilen Inokulation Werkzeug, wie ein Zahnstocher "DAB" die Probe (Zelle # 4) übertragen. Gemeinsame Inokulation Fehler, wie in Zelle Nr. 5 ("Patch") und Zelle # 6 ("fill") dargestellt, durch einem übermäßigen Wachstum von bakteriellen Proben nach Inkubation folglich kontaminierenden angrenzenden Feldern.
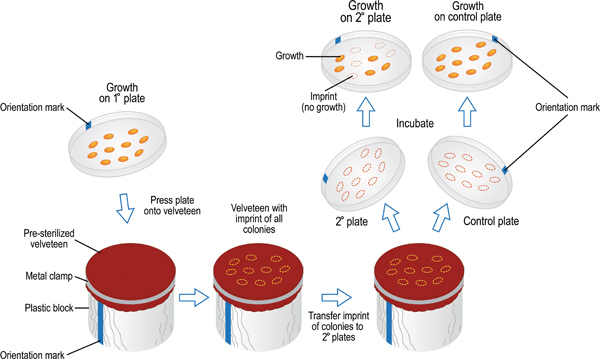
Abbildung 9. Replica-Plate-Technik verwendet, um Zellen von der Grundschule zur weiterführenden Platten für die Phänotyp-Bildschirme zu übertragen. Die Markierung auf der primären Platte wird mit der Markierung auf dem Samt bedeckt Block dann Lowe ausgerichtet Rot, damit der Agar-Oberfläche, um das Tuch zu kontaktieren. Die Zellen werden von der Platte auf dem Samt, indem Sie leicht übertragen, sondern gleichmäßig nach unten drücken auf der primären Platte mit den Fingerspitzen. Diese Aktion wird Eindruck zu der Zellproben auf der Samt in der gleichen räumlichen Muster der primären Platte. Das gleiche Verfahren wird verwendet, um Zellen aus dem Samt zu einem sekundären Platte übertragen. So viele wie 7-8 sekundären Platten können angeimpft mit dem gleichen primären Platte Eindruck auf den Samt werden. Die letzte Platte von der Samt geimpft sollte als eine positive Kontrolle zu dienen. Es sollte ein Medium, das Wachstum aller getesteten Stämme unterstützt, dass für eine ausreichende Zell-Transfer während der gesamten Serie von Platten aufgetreten sein. Nach der Inkubation kann die sekundäre Platten kontrolliert und für das Wachstum im Vergleich zu keiner Wachstum erzielt werden. Somit können mehrere Bakterienstämme gleichzeitig auf mehreren Wachstumsmedien gescreent werden in einem einzigen Experiment.
_upload/3064/3064fig10.jpg "/>
Abbildung 10. Beispiel durch Verwendung von pour-Plate-Technik. Eine 1,0 ml Probe von Wasser aus einem öffentlichen Trinkbrunnen gesammelt wurde in eine sterile leere Petrischale verzichtet. Dann schmolz aber gekühlten YTA wurde in die Schale mit der Probe gefüllt. Der Agar enthielt auch 100 pg / ml Cycloheximid, um das Wachstum von Hefen und Schimmelpilzen, die bereits im Wasser vorhandenen Probe darf zu verhindern. Nach dem Schwenken zu mischen, wurde die Platte auf einer ebenen Fläche gesetzt und das Agar durfte vollständig erstarren. Die Platte wurde bei 37 ° C für 48 Stunden inkubiert. Dargestellt ist das Ergebnis dieses Experiments. Beachten Sie den Unterschied im Aussehen der Oberfläche Kolonien, die großen und kreisförmigen sind in Form, im Vergleich zu Sub-Surface-Kolonien, die sehr klein sind und unregelmäßig zu gestalten, da die erstarrte Medium hemmt Kolonie Verbreitung in Subsahara-Oberfläche.

Abbildung 11. </ Strong> Beispiel durch Verwendung von Spread-Plate-Technik. Die "Copacabana Verfahren" wurde verwendet, um Platte eine Mischung aus E. coli-Zellen für ein Screening-Experiment. In diesem Fall wird das Wachstumsmedium (LB) X-Gal enthält, so dass diese Zellen exprimieren ein funktionelles β-Galactosidase-Enzym blaue Kolonien, während diese Zellen mit einer Mutation in den lacZ-Gens und somit nicht in der Lage, um eine funktionelle β-Galactosidase-Enzym Form auszudrücken weiße Kolonien. Oft als "blau / weiß Bildschirm" genannt, können die zwei Arten von Kolonien leicht voneinander werden auf der gleichen Platte zu unterscheiden.

Abbildung 12. Beispiel durch Plaque-Assay mit Soft-Agar-Overlay-Technik. Es zeigen Plaques auf der Wirtsstamm Mycobacterium smegmatis MC 2 155 (ATCC 700.084) von zwei unterschiedlichen pH ausgebildetAltersklassen: (A) Mykobakteriophagen Zerstörer und (B) Mykobakteriophagen MSSS. Diese Phagen wurden von Studenten im Praktikum UCLA MIMG 103L im Frühjahr 2010 getrennt. M. smegmatis ist ein nicht-pathogenen Actinobakteriums und gehört zu einer Familie der Mykobakterien, die ein paar Erregern, die zu schweren Krankheiten wie Tuberkulose (M. tuberculosis, M. africanum, M. bovis) und Lepra (M. leprae) verursacht umfasst. Etwa 50 ul einer 10 -2 Verdünnung von einem Zerstörer und 10 -3 Verdünnung von MSSS wurden jeweils mit 500 ul M. inkubiert smegmatis für 20 Minuten bei 37 ° C, dann mit MBTA (Weichagar) vermischt und auf MHA (hart Agar) Platten. Nachdem die Weichagar zu verfestigen für 20 Minuten ungestört, wurden die Platten umgedreht und bei 37 ° C für 48 Stunden inkubiert. Beachten Sie die unterschiedlichen Morphologien Plaque von den einzelnen Phagen produziert. Zerstörer (A) bildet kleine (mittlerer Durchmesser von etwa 1 mm), klar Plaques charakteristischen eines lytischen Phagenwährend MSSS (B) entwickelt große "Volltreffer" Plaques mit klaren Zentren von trübem Hof (mittleren Durchmesser von ca. 3,2 mm) umgeben ist. Die trübe Ring kann aus Bakterien, die resistent gegen eine Infektion Phagen verträglich sind. Dieses Muster unterscheidet sich von der durch lysogenen Phagen, die trüben Plaques gebildet.
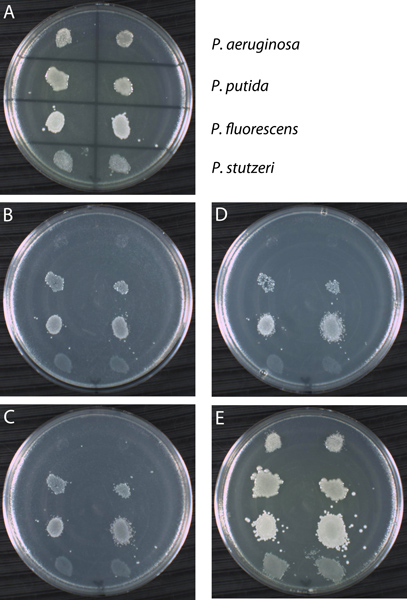
Abbildung 13. Beispiel durch Verwendung von Nachbau-Plate Verfahren. Acetamid, Lactose und Glycin: Vier Pseudomonas-Stämme (P. aeruginosa, P. putida, P. fluorescens und P. stutzeri) wurden im Duplikat für das Wachstum auf drei verschiedenen C-Quellen getestet. (A) der primären Platte ist ein Vollmedium (YTA) mit den vier Stämme wie angegeben inokuliert. Nach der Inkubation bei 30 ° C für 24 Stunden wachsen alle vier Stämme auf YTA. Der primäre Platte wurde auf Replika-Platte auf einem Minimalmedium mich eingesetztPalladium (MSA) mit einem einzigen Kohlenstoffquelle ergänzt: acetamid (B), Lactose (C) und Glycin (D). Die letzte Platte in der Serie war eine positive Kontrolle YTA Platte (E). Wie gezeigt, zeigen die Stämme variable Wachstumsmuster nach der Inkubation auf die sekundären Platten. Beachten Sie, dass es manchmal schwierig ist, zwischen Wachstum und einem Aufdruck von Zellen zu unterscheiden. So vergleichen Sie das Impressum von P. erzeugt stutzeri auf den drei Platten zu MSA kein Wachstum auf demselben drei Platten von P. aeruginosa. Beides sind negative Ergebnisse im Vergleich zu den Wachstumsmuster von P. ausgestellt putida und P. fluorescens. Allerdings wachsen alle Stämme auf die positive Bestätigung Steuerplatte Zellen wurden für alle sekundären Platten in der Reihe überführt. Die Ergebnisse dieses Experiments sind in Tabelle 1 aufgeführt.
| YTA (Primären) | MSA + acetamid | MSA + Laktose | MSA + Glycin | YTA (Kontrolle) | |
| P. aeruginosa | + | - | - | - | + |
| P. putida | + | + | + | + | + |
| P. fluorescens | + | + | + | + | + |
| P. stutzeri | + | - | - | - | + |
Tabelle 1. Zusammenfassung der Replika-Plattierung Ergebnisse. Wachstum angegeben als Pluszeichen (+) und ohne Wachstum als Minuszeichen (-). YTA ist ein komplettes Medium (Trypton Hefe-Agar) und MSA ist ein Minimal-Medium (minimal Salze Agar). Die Platten wurden mit MSA einziger Kohlenstoffquelle ergänzt, wie angegeben.
Diskussion
Kultivierung von Mikroorganismen umfasst eine Reihe von Beschichtungsverfahren, die alle, dass aseptischen Technik in der Manipulation von Zellen und Medien aufrechterhalten werden müssen. Fünf verschiedene Verfahren wurden in diesem Protokoll beschrieben. Obwohl diese Plattierungstechniken routinemäßig verwendet, um Bakterien und Phagen zu manipulieren, können sie auch zu Säugetierzellkulturen und eukaryotischen Mikroorganismen üblicherweise in der molekularen Genetik, wie Hefe (dh, Saccharomyces cerevisiae, Candida albicans, Schizosaccharomyces pombe), Algen und Protozoen angewendet werden ( dh, Volvox, Chlamydomonas, Amöben, Paramecium) und Nematoden (z. B. Caenorhabditis elegans). Es gibt auch zahlreiche (und noch ausgefeiltere) Variationen von jeder Plating-Verfahren in Abhängigkeit von der experimentellen Ziel oder Organismus unter-Studie. Somit ist es wichtig, nicht nur die am besten geeignete Technik für ein bestimmtes Experiment oder Ziel-Mikroorganismus, sondern auch, um eine maßgeschneiderte Methodik, tHat die experimentellen Ergebnisse entsprechend richtet sich das Forschungsinteresse Frage oder ein Problem.
Einige der wichtigsten aktuellen Anwendungen der Beschichtung Techniken in diesem Protokoll diskutiert beinhalten technologische Fortschritte, die High-Throughput-Screening-Ergebnisse für Wirkstoffforschung und Experimente führen. Verwenden Sie z. B. Genom-Sequenzierung Zentren der "Copacabana-Methode" für Spread-Plating Klon-Bibliotheken, die sich E. coli-Zellen mit Plasmiden, die DNA-Fragmente aus dem Genom eines Mikroorganismus transformiert. Weil Dutzende von großen Platten (so genannte Bioassay Trays) auf einmal bereit sind, wird eine automatisierte Schüttler verwendet, um die Glasperlen für den gesamten Stapel von Schalen zu schütteln. Darüber hinaus bei der Auswahl von Kolonien von diesen Platten nach der Inkubation wird ein Roboter-Kolonie-Picker verwendet, um Zellen von entsprechenden Kolonien als Inokulum für LB-Medium in 384-Well Mikroplatten zu sammeln. Für diese High-Throughput-Screening-Assay, die verfahrensrechtlichen Grundsätze des Spread-pEnde der Technik gelten, sondern Technologie ermöglicht verschiedene Schritte zu automatisieren und so skaliert, dass eine große Anzahl von Proben gleichzeitig und innerhalb eines kurzen Zeitrahmens analysiert werden zu können.
Biotechnologie-und Pharmaunternehmen investieren erhebliche Ressourcen in die Entwicklung von High-Throughput-Technologien für die grundlegenden Techniken in der Mikrobiologie und Molekulargenetik. Zum Beispiel gibt es Mehrkanal-Mikroliterpipetten, um die Lautstärke Transfers für bis zu 8 oder 12 Proben auf einmal durchzuführen. Es gibt sogar Roboter-Arbeitsstationen, die Manöver eine 96-Kanal-Pipette Kopf! Diese Bemühungen betreffen multidisziplinäre Teams von Wissenschaftlern, Biologen, die Paarung methodische Kompetenz besitzen, mit Ingenieuren und Programmierern, die die Instrumentierung notwendig, um die mechanischen Abläufe mit den Versuchen verbundenen Aufgaben entwickeln können. Unabhängig von der Anwendung der Forschung ist es das Ziel von Unternehmen bei der Entwicklung dieser Technologien teilten das gleiche - Labor automatisierenry Prozesse, Tools, Systeme und Instrumente, so dass sie weniger arbeitsintensiv und effizienter.
Offenlegungen
Ich habe nichts zu offenbaren.
Danksagungen
Besonderer Dank geht an Cori Sanders bei Iroc Designs zur Vorbereitung von Illustrationen und Kris Reddi und Bhairav Shah an der UCLA für den Aufbau und die Unterstützung der Probe Kulturen mit Zahlen. Finanzierung für dieses Projekt wurde von HHMI (HHMI Grant Nr. 52006944) zur Verfügung gestellt.
Materialien
| Name | Company | Catalog Number | Comments | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
1. Yeast Tryptone Agar (YTA)
Autoclave at 121°C for 20 minutes to sterilize. Store at 4°C. If preparing tubes for the pour-plate procedure, allow the agar to cool to ~55°C then add 2.0 ml of 50 mg/ml cycloheximide. Aseptically dispense 18.0 ml of the melted agar per 18 mm tube then store at 4°C. The agar will solidify and will need to be melted in a steamer or microwave prior to use. 2. Minimal Salts Agar (MSA) + 0.1% (w/v) carbon source
Autoclave at 121°C for 20 minutes to sterilize. Store at 4°C. * Carbon sources used for experiments presented in Figure 13 include acetamide, lactose, and glycine. ** Trace salts solution is prepared in 0.1 N HCl as follows. It is added to the base before sterilization (autoclave at 121°C for 20 minutes).
3. EHA soft agar (0.65 % w/v)
Autoclave at 121°C for 20 minutes to sterilize. Store at 4°C. 4. EHA hard agar (1.2% w/v)
Autoclave at 121°C for 20 minutes to sterilize. Store at 4°C. 5. 1X Middlebrook Top Agar (MBTA soft agar, 0.5% w/v)
Melt 50 ml of 2XTA and allow it to cool to ~55°C. Using aseptic technique, add the CaCl2 and 7H9 broth to the melted agar. Aseptically dispense 4.5 ml of the mixture per 13 mm tube and store in a 55°C incubator ≤7 days. Cooling MBTA to room temperature or 4°C will cause the CaCl2 to precipitate out of solution. * 100 mM CaCl2. stock must be stored at room temperature to prevent CaCl2 from precipitating out of solution. ** 7H9 liquid medium: Neat
Mix the base with water then add the glycerol while stirring. Autoclave at 121°C for 20 minutes to sterilize. Store at 4°C. *** 2X Middlebrook Top Agar (2XTA, 1.0% w/v)
Autoclave at 121°C for 20 minutes to sterilize. Dispense 50 ml aliquots into 100 ml bottles and store at 4°C. 6. Middlebrook 7H10 Agar Plates (MHA hard agar, 1.9% w/v)
Mix the agar base with water then add the glycerol while stirring. Heat the solution to boiling then stir for one minute to completely dissolve the base powder. Autoclave at 121°C for 20 minutes to sterilize. Allow the agar to cool to ~55°C then aseptically add the following reagents:
* AD supplement
Filter-sterilize this solution; do not autoclave. Store at 4°C. ** Filter-sterilize and store these solutions at 4°C for ≤60 days. 7. LB agar (1.5% w/v) + X-Gal (60 μg/ml)
Autoclave at 121°C for 20 minutes to sterilize. Allow the agar to cool to ~55°C then aseptically add 3.0 ml of 20 mg/ml X-Gal solution. Freshly prepare X-gal stock by dissolving 400 mg X-Gal in 20 ml dimethylformamide (DMF). Table of specific reagents:
Table of specific equipment:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Referenzen
- Barker, K. At the Bench: A Laboratory Navigator. , Cold Spring Harbor Laboratory Press. Cold Spring Harbor, New York. (1998).
- US Department of Health and Human Services (DHHS). Biosafety in Microbiological And Biomedical Laboratories (BMBL). , 5th, U.S. Government Printing Office. Washington DC. Available from: http://www.cdc.gov/biosafety/publications/bmbl5/index.htm (2009).
- Dulbecco, R., Vogt, M. Some problems of animal virology as studied by the plaque technique. Cold Spring Harbor Symp. Quant. Biol. 18, 273-279 (1953).
- Fields, B. N., Knipe, D. M., Chanock, R. M., Hirsch, M. S., Melnick, J. L., Monath, T. P., Roizman, B. Fields Virology. 1, 2nd, Raven Press. New York, NY. (1991).
- Gilbert, R. A., Ouwerkerk, D., Zhang, L. H., Klieve, A. V. Cooperative Research Center for Beef Genetic Technologies. In vitro detection and primary cultivation of bacteria producing materials inhibitory to ruminal methanogens. Journal of Microbiological Methods. 80, 217-218 (2010).
- Jett, B. D., Hatter, K. L., Huycke, M. M., Gilmore, M. S. Simplified agar plate method for quantifying viable bacteria. BioTechniques. 23, 648-650 (1997).
- Karam, J. D. Molecular Biology of Bacteriophage T4. , ASM Press. Washington, DC. (1994).
- Knipe, D. M., Howley, P. M., Griffin, D. E., Lamb, R. A., Martin, M. A., Roizman, B., Straus, S. E. Fundamental Virology. , 4th, Lippincott, Williams, and Wilkins. Philadelphia, PA. (2001).
- Lederberg, J., Lederberg, E. M. Replica plating and indirect selection of bacterial mutants. J. Bacteriol. 63, 399-406 (1952).
- Miller, J. H. A Short Course in Bacterial Genetics: A Laboratory Manual and Handbook for Escherichia coli and related bacteria. , Cold Spring Harbor Laboratory Press. Plainview, NY. (1992).
- Mills, K. V., Gareau, J. R., Garcia, A. M. Low-cost modification to the Copacabana method for spreading transformation mixtures. BioTechniques. 39, 188(2005).
- Jordan, T., et al. RESEARCH: National Genomics Research Initiative Phage Resource Laboratory Manual. , Howard Hughes Medical Institute. (2008).
- Sambrook, J., Russell, D. W. Molecular Cloning - A Laboratory Manual. , 3rd, Cold Spring Harbor Laboratory Press. Cold Spring Harbor, New York. (2001).
- Sanders, E. R., Miller, J. H. I. Microbiologist: A Discovery-based Course in Microbial Ecology and Molecular Evolution. , ASM Press. Washington, DC. (2010).
- Slonczewski, J. L., Foster, J. W. Microbiology - An Evolving Science. , 2nd, W.W. Norton & Co., Inc. New York, NY. (2011).
- Teather, R. M., Wood, P. J. Use of Congo Red-Polysaccharide Interactions in Enumeration and Characterization of Cellulolytic Bacteria from the Bovine Rumen. Applied and Environmental Microbiology. 43, 777-780 (1982).
- Wise, K. Preparing Spread Plates Protocols. , Available from: http://www.microbelibrary.org/index.php/component/resource/laboratory-test/3085-preparing-spread-plates-protocols (2006).
- Worthington, M., Luo, R. Q., Pelo, J. Copacabana method for spreading E. coli and yeast colonies. BioTechniques. 30, 738-742 (2001).
- American Type Culture Collection (ATCC). , Available from: http://www.atcc.org/ (2012).
- EPA Microbiology Home Page. , Available from: http://www.epa.gov/nerlcwww/index.html (2012).
- The GENE Project: Laboratory Methods and Materials. , Available from: http://www.phys.ksu.edu/gene/g1.html (2012).
- Joint Genome Institute (JGI) Genome Sequencing Protocols: Library Plating Protocol. , Available from: http://www.jgi.doe.gov/sequencing/protocols/prots_production.html (2012).
- Microbial Genetics (maintained by Stanley Maloy at UCSD). , Available from: http://www.sci.sdsu.edu/~smaloy/MicrobialGenetics (2012).
- Organization of American States (OAS) Department of Sustainable Development (DSD). , Available from: http://www.oas.org/DSD/publications/Unit/oea59e/ch26.htm (2012).
- Public Health Agency of Canada: Material Safety Data Sheets (MSDS) for Infectious Substances. , Available from: http://www.phac-aspc.gc.ca/msds-ftss (2012).
- United States Environmental Protection Agency (EPA) Office of Water. , Available from: http://water.epa.gov (2012).
- American Society for Microbiology (ASM)'s Curriculum Recommendations Microbiology Majors Program. , Available from: http://www.asm.org/asm/index.php/unknown/asm-s-curriculum-recommendations-microbiology-majors-program.html (2012).
- Introductory Course in Microbiology. , Available from: http://www.asm.org/asm/index.php/unknown/asm-s-curriculum-recommendations-introductory-course-in-microbiology.html (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten