Method Article
Assépticas Técnicas de Laboratório: Métodos Plating
Neste Artigo
Resumo
Quando se trabalha com meios de comunicação e dos reagentes utilizados para os microorganismos de cultura, uma técnica asséptica deve ser praticado para assegurar a contaminação é minimizado. Uma variedade de métodos de revestimento são rotineiramente utilizados para isolar, propagar ou bactérias enumerar e de fagos, os quais incorporam os procedimentos que mantêm a esterilidade de materiais experimentais.
Resumo
Microrganismos estão presentes em todas as superfícies inanimadas criação de fontes de contaminação ubíquos possível no laboratório. Experimental sucesso depende da capacidade de um cientista para esterilizar superfícies de trabalho e equipamentos, bem como evitar o contato de instrumentos esterilizados e soluções com superfícies não-estéreis. Aqui apresentamos os passos para métodos de revestimento vários rotineiramente utilizadas em laboratório para isolar, propagar, ou enumerar microorganismos, como bactérias e fagos. Todos os cinco métodos incorporar uma técnica asséptica, ou procedimentos que mantêm a esterilidade de materiais experimentais. Procedimentos descritos incluem (1) raia excesso de regulamentação culturas bacterianas para isolar colônias de solteiro, (2) derramar excesso de regulamentação e (3) difusão-plating para enumerar viáveis colônias bacterianas, (4) overlays ágar mole para isolar fago e placas de enumerar, e ( 5) réplica de placas para transferir células de uma placa para a outra em um padrão idêntico espacial. Estes procedimentos podem ser executados em tele bancada de laboratório, desde que envolvem não patogénicas estirpes de microrganismos (Nível de Biossegurança 1, BSL-1). Se estiver trabalhando com BSL-2 organismos e, em seguida estas manipulações devem ocorrer em uma cabine de segurança biológica. Consulte a edição mais atual da Biossegurança em Laboratórios Microbiológicos e Biomédicos (BMBL), bem como Fichas de Dados de Segurança (MSDS) para substâncias infecciosas para determinar a classificação de risco biológico, bem como as precauções de segurança e instalações de confinamento necessárias para o microrganismo em questão. Cepas bacterianas e estoques fago pode ser obtido a partir de pesquisadores de investigação, empresas e coleções particulares mantidos por organizações como a American Type Culture Collection (ATCC). Recomenda-se que não patogénicos-estirpes ser utilizado quando a aprendizagem dos métodos de revestimento diferentes. Seguindo os procedimentos descritos neste protocolo, o aluno deve ser capaz de:
- Realizar procedimentos de chapeamento sem contaminatimídia ng.
- Isolar simples colônias de bactérias pelo método de raia-plating.
- Utilizar métodos de verter de revestimento e propagação de revestimento a fim de determinar a concentração de bactérias.
- Realizar sobreposições agar mole quando se trabalha com fago.
- Transferir as células bacterianas de uma placa para a outra utilizando o procedimento de réplica de placas.
- Dada uma tarefa experimental, selecione o método de plaqueamento apropriado.
Protocolo
1. Prepare um espaço de trabalho seguro e estéril
- Estar familiarizado com todas as regras de laboratório e precauções de segurança a serem tomadas quando se trabalha com microorganismos. Independentemente da classificação de risco biológico, todos os materiais que entram em contacto com microrganismos são considerados resíduos infectantes e devem ser descontaminados antes do descarte. Siga as orientações de segurança, em conformidade com os fornecidos pela Saúde Ambiental institucional e departamentos de segurança, a criação de lixeiras apropriadas para a eliminação imediata e adequada de materiais potencialmente contaminados (riscos biológicos).
- Esterilizar todos os instrumentos, soluções e meios de comunicação antes de usá-los para procedimentos de chapeamento.
- Limpar todos os materiais bagunçando sua área de trabalho na bancada do laboratório.
- Limpar a área de trabalho com desinfectante para minimizar a contaminação possível.
- Configurar um bico de Bunsen e trabalhar devagar, com cuidado e, deliberadamente, dentro da área de campo estéril criado pelo updrapé da chama.
- Se estiver trabalhando com BSL-2 organismos, configure seu espaço de trabalho em um gabinete de Biossegurança. Um queimador de Bunsen não pode ser usado no interior do armário, porque o calor da chama interrompe o fluxo de ar essenciais para a sua funcionalidade.
- Organizar todos os suprimentos necessários para o procedimento na bancada do laboratório perto do campo estéril. Certifique-se de todos os materiais sejam devidamente rotulados. Organizar a área de trabalho para maximizar a eficiência de trabalho e evitar movimentos desnecessários irá minimizar o tempo de exposição de materiais experimentais aos contaminantes aerotransportados.
- Coloque o bico de Bunsen à sua direita no banco.
- Coloque placas de agar ou placas de Petri à sua esquerda.
- Dispor culturas de células, tubos, frascos, garrafas, no centro da bancada.
- Solte as tampas de tubos, frascos e garrafas para que eles possam ser facilmente abertas com uma das mãos durante as manipulações subsequentes.
- Lave bem as mãos com sabonete anti-séptico e quenteágua antes de manusear microrganismos.
2. Procedimento Streak Placa: Isolamento de colónias de bactérias usando o método Quadrante
O procedimento raia placa foi projetada para isolar culturas puras de bactérias, ou colônias, a partir de populações mistas por separação mecânica simples. Colónias individuais são compostas de milhões de células que crescem em um cluster sobre ou dentro de uma placa de ágar (Figura 1). Uma colónia, ao contrário de uma única célula, é visível a olho nu. Em teoria, todas as células de uma colónia são derivadas de uma única bactéria depositada inicialmente na placa e, portanto, são referidos como um clone, ou aglomerado de células geneticamente idênticos.
- As bactérias existem numa variedade de formas e tamanhos. Por exemplo, células de Escherichia coli individuais são em forma de bastonete com um comprimento médio de 2 micra e largura de 0,5 micron, enquanto as células de Streptococcus são esféricas com um diâmetro médio de 1 m. Algumas bactérias (such como E. coli) existem como células únicas, enquanto outros formam padrões distintos de associação. Streptococcus, por exemplo, crescer em pares ou em cadeias forma ou grupos de células. Supõe-se geralmente que uma única colônia surge a partir de uma única célula passando por fissão binária, no entanto, essa premissa não é verdadeira para as bactérias que naturalmente existem em pares, correntes, ou clusters, ou que dividir por outros mecanismos. Alternativamente, se as bactérias são muitas chapeado, em seguida, se sobrepõem de células pode ocorrer e aumentar a probabilidade de duas ou mais bactérias que dão origem a que parece ser uma única colónia. Para evitar estas complicações quando se descreve ou enumerando culturas bacterianas que crescem em meio sólido, as colónias são referidos como unidades formadoras de colónias (CFU).
Com o procedimento raia-placa, uma mistura de células é espalhado sobre a superfície de um semi-sólido, meio de agar nutriente baseado numa placa de Petri de tal modo que cada vez menos bacterianaas células são depositados nos pontos de amplamente separadas sobre a superfície do meio e, após incubação, desenvolver-se colónias. O método quadrante para isolar colónias únicas a partir de uma mistura de células será descrito aqui.
- Streak-plating pode ser realizado com um número de diferentes instrumentos (Figura 2). Um anel de metal pode ser re-utilizada várias vezes e é utilizado para raia chapeamento-estirpes laboratoriais de rotina. Descartáveis lacetes de plástico estão disponíveis comercialmente e são utilizados mais frequentemente quando se trabalha com NBS-2 estirpes num armário de segurança biológica. Muitos cientistas preferem usar descartáveis, pré-esterilizadas varas de madeira ou palitos planos para raia-plating. Estes são uma alternativa barata para circuitos de plástico descartáveis e pode ser especialmente útil quando se trabalha com uma amostra do ambiente, tais como o solo que provavelmente contém bactérias formadoras de esporos.
- Descartáveis pré-esterilizados paus e palitos não precisa ser inflamado com o bico de Bunsen quis prevenção de dispersão em aerossol desnecessária de bactérias formadoras de esporos e de contaminação cruzada das superfícies de laboratório ou placas de ágar com esporos.
- Rótulo em volta da borda do fundo (não a tampa) de uma placa de agar com pelo menos o seu nome, a data, o tipo de meio de crescimento, e do tipo de organismo a ser revestida sobre o meio.
- As placas devem estar completamente seco sem condensação na tampa e pré-aquecido até à temperatura ambiente antes da raia-chapeamento. Se as placas são armazenadas a 4 ° C, removê-los várias horas ou mesmo no dia anterior. Espalhe-os em pequenas pilhas escalonados de não mais de 2-3 pratos e deixe secar.
- A amostra a partir do qual a faixa de placa será inoculado poderia ser tanto uma suspensão de células em caldo ou uma colónia existente a partir de uma outra placa de ágar. Para começar, recomenda-se que apenas uma amostra ser usada para inocular uma placa única. Para poupar tempo e materiais, uma placa única pode ser usado para samp múltiplales, mas só quando você se tornar proficientes em estrias uma amostra em um prato.
- Para o primeiro quadrante, a amostra é apanhada e espalhado sobre cerca de um quarto da superfície do meio usando um rápido, suave, o movimento de vai-e-vem (Painel A da Figura 3). Levantar a metade inferior de uma placa invertida do banco. Mover o laço, vara, ou um palito frente e para trás diversas vezes ao longo da superfície de agar a partir do aro para o centro da placa.
- Se o inoculo é uma suspensão de células, obter uma ansa cheia com um anel de metal ou 5 a 10 uL com uma micropipeta. Certifique-se de redemoinho ou vórtice da suspensão celular antes de remover uma alíquota para revestimento. Use uma técnica asséptica durante a realização deste passo, à chama não só circuito, mas também o aro da garrafa ou do tubo antes e após a remoção do inoculo. Além disso, não tocar os lados do tubo ou frasco com o laço ou cano da micropipeta. Se pipetagem do inóculo, dispense a suspensão da célula para a appropriate lugar sobre a placa, em seguida, utilizar um circuito vara, ou palito para espalhar sobre o primeiro quadrante da placa.
- Se o inoculo é uma colónia de outro prato, tocar suavemente a colónia com o laço, vara ou palito, em seguida, espalhá-lo ao longo do primeiro quadrante da placa. Certifique-se o anel de metal é resfriado antes de tocar a colônia. Para garantir o loop não é muito quente, tocar-lhe levemente na placa de ágar-ágar em uma área designada onde não há estrias irá ocorrer. Apenas algumas poucas células são necessários a partir da colônia, e não toda a colônia.
- Se utilizando um anel de metal, chama-lo usando um bico de Bunsen antes de obter o inoculo para a placa (Painel B da Figura 3). Começar cerca de 3-4 polegadas a partir do circuito, com o fio na ponta do cone azul, a parte mais quente da chama. O metal deve tornar-se vermelho quente. Mover o fio de modo a chama se aproxima do loop. Esta manipulação impede aerossolização de bactérias que restam no circuito do uso anterior.
- Flaming mata obactérias (embora nem esporos) e correntes de convecção do ar aquecido impedir outros contaminantes do ar a partir de sedimentação para o fio de metal durante as manipulações subsequentes.
- Se estiver usando um palito estéril plana, segure a extremidade mais estreita suavemente entre o polegar eo dedo anular em um ângulo de 10 ° a 20 para o meio, e utilizar a grande final a raia os quadrantes. Se estiver usando um loop ou bastão de madeira, segure-o como um lápis no mesmo ângulo. Não pressione com tanta força que as escavações do laço da vara, ou palito no agar.
- Depois de completar o primeiro quadrante, inverter e colocou a placa traseira em tampa no banco. Descarte o pau ou um palito ou re-chama o anel de metal, tal como descrito no passo # 4.
- Ligue a placa de Petri de 90 ° a raia segundo quadrante. Levantar a metade inferior de uma placa invertida a partir do banco, em seguida, tocar o lacete vara, ou palito de dentes para o primeiro quadrante perto do fim do traço última. Usando a parte traseira e padrão de luz, atravesse a lasmeia t das estrias no primeiro quadrante então mover-se em vazio o quadrante segundo. Inverter e estabelecer a placa traseira em tampa no banco uma vez que o segundo quadrante está cheio.
- O loop, bastão ou palito nunca deve voltar para a primeira metade das faixas no primeiro quadrante, onde a maioria do inóculo original foi depositada.
- Um novo ciclo de plástico stick, de madeira ou de palito de dentes deve ser usado para cada quadrante.
- Repita a etapa # 6 duas vezes para os terceiro e quarto quadrantes.
- Certifique-se de dispor do bastão ou palito ou re-chama o anel de metal entre cada quadrante.
- Evite ir para o primeiro quadrante estrias quando o quarto quadrante.
- Incubar a placa de cabeça para baixo de modo a condensação que se acumula sobre a tampa não escorre sobre as colónias.
3. Despeje Procedimento Placa: Enumeração de células bacterianas em uma amostra mista
Este método é frequentemente usard para contar o número de microorganismos numa amostra misto, que é adicionado a um meio de ágar fundido antes da sua solidificação. O processo resulta em colónias distribuídos uniformemente por todo o meio sólido quando a diluição da amostra adequada é revestida. Esta técnica é usada para realizar a contagem da placa viáveis, em que o número total de unidades formadoras de colónias dentro do agar e na superfície do agar em uma única chapa é enumerados. As contagens viáveis fornecer cientistas um meio padronizado para gerar curvas de crescimento, para calcular a concentração de células no tubo a partir da qual a amostra foi revestida, e para investigar o efeito de vários ambientes ou condições de crescimento sobre a sobrevivência da célula bacteriana ou taxa de crescimento.
- Rótulo em volta da borda do fundo (não a tampa) de uma placa de Petri estéril mas vazio com pelo menos o seu nome, a data, o tipo de meio de crescimento, e do tipo de organismo a ser adicionado ao meio de agar fundido.
- Inclua o fator de diluição se platção diluições em série, ou uma série de diluições repetidas, o que resulta numa redução sistemática da concentração de células na amostra. Preparação diluições em série é necessário se o número de células na amostra ultrapassar a capacidade da placa de agar, em que o intervalo é estatisticamente significativa de 30-300 cfu. Se houver mais do que 300 CFU sobre uma placa, em seguida, as colónias será cheia e de sobreposição.
- Obter um tubo contendo 18 ml de meio de agar fundido.
- O meio de ágar deve ser dispensada em tubos de ensaio e pré-esterilizados em autoclave. No mesmo dia em que é necessário para uma experiência, o agar deve ser fundida num vapor durante 30 minutos, em seguida, transferidos para um banho de água 55 ° C. Apenas ágar, tanto quanto é necessário para a experiência deve ser derretido em que não pode ser re-utilizado.
- Dez minutos antes de vazar placas, os tubos de ágar fundido deve ser transferido a partir do banho de água 55 ° C para um bloco de calor no trabalhobanco Atory fixada em 48 ° C. Uma vez que o agar atinge esta temperatura, está pronto para verter. Se o agar é muito quente, as bactérias na amostra pode ser morto. Se o agar é muito legal, o meio pode ser irregular, uma vez solidificado.
- Obter a sua amostra, o qual deve ser tanto uma cultura de caldo ou de uma suspensão de células produzidas por células de mistura a partir de uma colónia em tampão ou soro fisiológico.
- As amostras podem ser derivadas de uma série de diluições de uma única amostra.
- Volume de amostra a ser revestida deve estar entre 0,1 e 1,0 ml.
- Abrir a tampa da placa de Petri vazia, e dispensar a sua amostra para o meio da placa (Painel A da Figura 4). Feche a tampa.
- Use uma técnica asséptica durante todo este procedimento.
- Use uma pipeta sorológica ou uma micropipeta para transferir a amostra para o prato. Controlar o fluxo da amostra de modo que não espirrar para fora da placa.
- Retire a tampa da banheirae do agar fundido, e passar o aro do tubo aberto através da chama do queimador de Bunsen.
- Abrir a tampa da placa de Petri contendo a amostra e derramar o ágar-ágar em cuidadosamente (Painel B da Figura 4). Fechar a tampa, em seguida, misturar a amostra com o ágar rodando suavemente o prato.
- Permitir que o agar solidificar antes de completamente invertendo a placa para a incubação.
4. Processo de espalhamento em placa: Formação de discretos Colônias de contagens, o enriquecimento, seleção ou triagem
Esta técnica é tipicamente usado para microorganismos separados contidos dentro de um pequeno volume de amostra, que é espalhado sobre a superfície de uma placa de ágar, resultando na formação de colónias discretas, distribuídos uniformemente em toda a superfície do ágar quando a concentração apropriada de células é revestida. Para além de utilizar esta técnica para contagens de placas viáveis, em que o número total de unidades formadoras de colónias em um canteplaca le é enumerado e usada para calcular a concentração de células no tubo a partir da qual a amostra foi revestida, a dispersão de revestimento-se rotineiramente utilizados em experiências de enriquecimento, selecção e triagem. O resultado desejado para estas três experiências é geralmente a mesma que para as contagens de placas, em que uma distribuição de colónias discretas formam em toda a superfície do ágar. No entanto, o objetivo não é garantir que todas as células viáveis formar colônias. Em vez disso, apenas as células dentro de uma população que têm um genótipo particular deve crescer. O procedimento de placa propagação podem ser empregues em relação ao método da placa de fluidez de uma experiência de enumeração se o objectivo final é isolar colónias para posterior análise, porque colónias crescer acessivelmente sobre a superfície do ágar enquanto que eles se tornam incorporado no agar com o procedimento placa de fluidez.
Existem duas estratégias descritas aqui para o procedimento de placa propagação. O (Método A) primeiro envolve o uso de uma mesa giratória e de vidro ou de metal rod em forma de taco de hóquei. A segunda (método B), referido como o "Método de Copacabana", envolve a agitação contas pré-esterilizados de vidro. Ambos facilitar ainda propagação de células em toda a superfície do ágar.
Método A: Spread revestimento com uma plataforma giratória e de vidro ou barra de metal
- Rótulo em volta da borda do fundo (não a tampa) de uma placa de agar com pelo menos o seu nome, a data, o tipo de meio de crescimento, e do tipo de organismo a ser revestida sobre o meio.
- Inclua o fator de diluição se plaqueamento diluições seriadas.
- As placas devem estar completamente seco sem condensação na tampa e pré-aquecido até à temperatura ambiente antes de propagação de revestimento. Se as placas são armazenadas a 4 ° C, removê-los várias horas ou mesmo no dia anterior. Espalhe-os em pequenas pilhas escalonados de não mais de 2-3 pratos e deixe secar.
- Centro de chapa do sobre a mesa giratória (Figura 5).
- Obter vocêr amostra, o que deve ser uma cultura de caldo ou de uma suspensão de células produzidas por células de mistura a partir de uma colónia em tampão ou soro fisiológico.
- As amostras podem ser derivadas de uma série de diluições de uma única amostra.
- Volume de amostra a ser revestida deve estar entre 0,1 e 0,2 ml.
- Abrir a tampa da placa de Petri, e dispensar a sua amostra para o centro do ágar. Feche a tampa.
- Use uma técnica asséptica durante todo este procedimento.
- Usar uma micropipeta para transferir a sua amostra para a chapa. Controlar o fluxo da amostra de modo que não espirrar para fora da placa.
- Mergulhar o vareta de vidro ou de metal haste (também chamado um espalhador) para um copo de 70% (v / v) de etanol.
- CUIDADO: Nunca mergulhe uma espátula quente em um recipiente com álcool.
- O etanol deve apenas toque na parte inferior do espalhador e da primeira polegada da haste.
- Escorra e inflamar excesso de etanol, passando-a através da chama de um Bunsen queimador.
- A chama deve percorrer o comprimento do tronco distribuidor e que entrou em contato com o etanol, em seguida, rapidamente extinguir.
- Se o copo de pegar fogo o etanol, não entre em pânico! Colocar uma tampa de vidro sobre o copo, o qual irá rapidamente extinguir o fogo.
- Abra a tampa da placa de ágar, segurando a tampa em sua mão esquerda com o polegar eo dedo indicador. Arrefece-se a espalhador por tocá-lo para o ágar ao longo da borda próxima do aro.
- Não toque no agar, onde as células foram adicionadas. O espalhador quente irá matar as células.
- Com a mão esquerda (enquanto ainda está segurando a tampa da placa de ágar), girar o prato lentamente.
- Apesar de ser evitados, se você deve colocar a tampa para baixo, coloque-o de bruços sobre uma superfície desinfetada no campo estéril do bico de Bunsen. Com uma tampa que fica voltada para cima, há uma maior chance de contaminação dos movimentos de objetos ou as mãos, criando correntes de ar que causammicroorganismos e partículas de poeira para descer para a superfície interior da tampa.
- Com a mão direita, segurar o espalhador suavemente sobre a superfície do agar e gradualmente se espalhou a amostra uniformemente sobre toda a placa. Mova o distribuidor e para trás ao longo da placa como o prato está girando.
- Deixar a amostra absorver completamente (pelo menos 5 minutos), antes de invertendo a placa para a incubação.
Método B: Spread revestimento com esferas de vidro: o "Método Copacabana"
- Rótulo em volta da borda do fundo (não a tampa) de uma placa de agar com pelo menos o seu nome, a data, o tipo de meio de crescimento, e do tipo de organismo a ser revestida sobre o meio.
- Inclua o fator de diluição se plaqueamento diluições seriadas.
- Abra a tampa da placa de ágar, segurando a tampa em sua mão esquerda com o polegar eo dedo indicador. Em seguida, abra o tubo de vidro ou garrafa contendo pré-esterilizado glcontas bunda. Com o seu lado direito chama, o aro do tubo ou frasco, então, cuidadosamente dispensar 10-12 esferas de vidro estéril para uma placa de ágar (Figura 6). Fechar a tampa da placa, e chama a borda do tubo ou frasco, uma vez mais antes de substituir a tampa e definindo-la de lado.
- Verta cuidadosamente as esferas para fora do tubo ou frasco de alguns centímetros acima do ágar de modo que os grânulos não saltar para fora da placa de Petri.
- Use pérolas de vidro que são 4 mm de diâmetro. Esterilizá-los em garrafas de vidro ou tubos de ensaio na autoclave a 121 ° C no ciclo seco (configuração gravidade) durante 30 minutos.
- Uma vantagem da utilização de grânulos, em vez de um espalhador é que nenhum recipientes abertos de etanol são necessários para flamejante repetido.
- Abrir a tampa da placa de ágar, e dispensar a sua amostra para o centro do ágar. Feche a tampa.
- Use uma técnica asséptica durante todo este procedimento.
- Alíquota de 100 uL a 150 de amostra por placa. Estevolume irá facilitar ainda propagação de células. Usar uma micropipeta para transferir a sua amostra para a chapa. Controlar o fluxo da amostra de modo que não espirrar para fora da placa.
- Espalhar a amostra agitando os grânulos através da superfície do agar de 6 a 7 vezes. Para garantir células se espalham de maneira uniforme, use um movimento horizontal de tremer.
- Não agite as contas ou então todas as células vai acabar na borda da placa.
- DICA: Se feito corretamente, o procedimento soa como "maracas tremor".
- Girar a placa de 60 °, em seguida, horizontalmente agitar novamente 6 a 7 vezes.
- Gire a placa 60 ° uma segunda vez e horizontalmente agitar novamente. Até agora, você deve atingir um espalhamento uniforme de células em toda a superfície do ágar.
- Quando terminar propagação de revestimento, a amostra deve ser absorvido e da superfície do ágar deve estar seco. Deitar fora as contas contaminadas em um copo coleção marcada contendo cloro 10%.
- Nãorejeitar as contas no lixo! As pérolas utilizadas serão lavadas e autoclavado, re-esterilizá-los para o uso de repetição.
- NOTA: Se a superfície do ágar é ainda molhado após agitação por três vezes, permitem que o prato de se sentar durante vários minutos para permitir ao líquido a ser absorvido pelo ágar, em seguida, repetir os passos 4-6 # até que a superfície da placa aparece seco.
- Inverter a placa para a incubação.
5. Procedimento Overlay macio Agar: formação de placas para isolamento e enumeração de Phage (ensaio de placa)
Esta técnica é normalmente usado para detectar e quantificar bacteriófago (fago), ou vírus bacterianos que variam em tamanho de 100 a 200 nm. Um microscópio eletrônico é necessário para ver partículas de fago individuais. No entanto, a presença de partículas de fago infecciosas pode ser detectado como placas sobre uma placa de ágar (Figura 7A). Fagos não pode replicar fora das suas células hospedeiras bacterianas, de modo a propagação umadetecção d exige fagos mistura e células hospedeiras juntos antes do revestimento. Para o procedimento de gelose de cobertura macio, um pequeno volume, geralmente na gama de 50 uL a 200 uL, de uma suspensão de fagos é dispensado para um tubo contendo cerca de 10 8 bactérias (células hospedeiras), que estão uniformemente dispersos em 2,5-3,0 ml de mole (0,5 a 0,7% [w / v]), ágar nutriente derretido. A mistura resultante é vertida sobre a superfície de um disco rígido (1,5 a 1,9% [w / v]) em placa de ágar nutriente. A placa é agitada suficientemente para assegurar que o agar mole cobre toda a superfície do agar rígido. Em seguida, a placa é colocada sobre uma superfície plana até que a camada de agar superior tem tido tempo para solidificar e, subsequentemente, pode ser colocada na incubadora.
Ao longo do tempo, uma suspensão turva de células bacterianas, referido como um relvado, torna-se visível ao longo do meio de agar mole (Figura 7B). As placas formar se um fago infecta uma das células bacterianas, replica dentro thcélula e, em seguida, lisa a célula liberando cerca de 100 fagos progênie (aka, o tamanho da explosão). As partículas fágicas novos difundir no agar mole, infectando as bactérias na área que circunda o celular lisada bacteriana. Após vários ciclos de infecção e de lise, a suspensão de células turva bacteriana no agar mole desaparece, deixando uma zona de compensação chamado uma placa. Cada placa contém mais de 10 partículas fágicas 9, todos os geneticamente idênticos para a partícula de fago inicial infeccioso. Porque uma placa surge a partir de uma partícula de fago único, o número resultante de unidades formadoras de placa (pfu) pode ser contado e da concentração original, ou titulação, da suspensão de fagos pode ser calculado. Este tipo de experiência, chamado um ensaio de placa, também fornece um meio cientistas padronizado para gerar curvas de um passo de crescimento, para investigar a especificidade de gama de hospedeiros, e para transduzir células bacterianas para experiências genéticas.
- Prepare as bactérias indicadoras: Uma cultura em crescimento exponencial da estirpe hospedeira bacteriana precisa ser preparado para o experimento macio gelose de cobertura. Cada host tem seus requisitos próprios de crescimento. Entre 0,3 ml e 0,5 ml de uma suspensão de bactérias indicadoras é necessária para cada placa. Se um balão de bactérias indicadoras é preparado (por exemplo, 25 ml), a cultura deve ser dividida em alíquotas menores (por exemplo, alíquotas de 5 ml dispensadas em estéreis com tampa de rosca tubos) para minimizar a possibilidade de contaminação cruzada. Os tubos podem ser mantidos em gelo (a 4 ° C) até à sua utilização no ensaio. Use uma técnica asséptica para inocular caldo nutriente estéril, em seguida, incubar acordo com as especificações da estirpe bacteriana. Culturas Leftover deve ser descartada no final do dia.
- Prepare os tubos de adsorção: Coloque dois microtubos estéreis em um rack. Etiquete a tampa do primeiro tubo "Phage" ea tampa do segundo tubo de "Control".
- Típicodiluições seriadas de ly lisados fágicos são preparados e plaqueadas em cujo caso os tubos de microcentrífuga adicionais seria adicionado à cremalheira e rotuladas com o factor de diluição.
- Estoques de fagos deve ser feita fresco a partir de placas individuais e armazenadas a 4 ° C. Para desagregar as partículas de fagos, os estoques deve ser aquecido até à temperatura ambiente antes de se realizar uma experiência.
- Estoques de fagos devem ser manuseados com cuidado - não vortex ou pipetar vigorosamente.
- Adicionar 50 ul da sua amostra de fagos para o primeiro tubo, em seguida, adicionar 50 ul de tampão de fagos para o segundo tubo, que vai servir como um controlo negativo para o ensaio de placa.
- Use uma técnica asséptica em todas as manipulações de fagos e bactérias.
- Em seguida adicionar 500 uL de bactérias indicadoras a cada tubo de adsorção.
- As células bacterianas irá assentar no fundo de um tubo de se sentar em gelo ou a bancada durante longos períodos de tempo. Se a cultura não é misturared antes da remoção de uma aliquota para a experiência, não o suficiente células bacterianas serão transferidos para o tubo de adsorção. Tem de haver um número suficiente de colónias bacterianas formadas no agar mole (ou seja, um relvado) para a detecção de placas. Utilizar um misturador de vórtice para suavemente re-suspender as bactérias indicadoras numa suspensão homogénea antes de transferir alíquotas para os tubos de adsorção.
- Misturar as células bacterianas e de fagos por suavemente flicking os tubos.
- Incubar o fago / bactérias mistura durante 15-20 minutos (não mais do que 30 minutos) a uma temperatura adequada para a estirpe indicador.
- Preparar agar mole de nutrientes e placas de agar rígidos: Enquanto adsorção está ocorrendo, o local de dois tubos de agar mole (previamente fundidos e armazenadas a 55 ° C) em um bloco de aquecimento a sua bancada de laboratório fixado em 46-48 ° C.
- O ágar macio derretido deve ser equilibrada com a temperatura do bloco de aquecimento durante 10-15 minutos. Se o macio derretidaágar senta mais de 15 minutos, ele começa a solidificar e formar grumos quando derramado sobre o ágar rígido. Se o agar macio derretido está a menos de 10 minutos, será demasiado quente quando adicionado à phage / mistura de bactérias, matando as células hospedeiras antes de plaqueamento. Por conseguinte, um relvado não podem formar e placas poucas ou nenhuma pode ser detectável.
- Preparar adicionais tubos agar mole se plaqueamento diluições em série do lisado de fago.
- Rótulo em volta da borda do fundo (não a tampa) de duas rígido placa de ágar nutriente placas com pelo menos o seu nome, a data, o tipo de meio de crescimento, e "Phage" ou "Control" correspondente aos tubos de adsorção.
- Incluir placas adicionais marcados com o fator de diluição se plaqueamento diluições seriadas.
- As placas de agar rígidos deve estar completamente seco sem condensação na tampa e pré-aquecido até à temperatura ambiente antes da adição do ágar mole. Se as placas são armazenadas a 4 ° C, removê-los de várias horas ou até mesmo o day antes. Espalhe-os em pequenas pilhas escalonados de não mais de 2-3 pratos e deixe secar. Humidade excessiva na camada de agar rígido irá causar a diluição do agar mole, permitindo que o fago de se difundir mais facilmente através do ágar mole durante a formação da placa. Consequentemente, o tamanho da placa irá aumentar.
- Placa de adsorção tubos um de cada vez como se segue: Use uma micropipeta para P1000 assepticamente transferir o fago / bactérias mistura (deve ser de aproximadamente 550 ul) para um tubo de agar mole, em seguida, rapidamente girá-lo entre as palmas das suas mãos para misturar os conteúdos. Não agite o tubo de modo que as bolhas de ar são introduzidas. Segurando o tubo em sua mão esquerda, abra a tampa de uma placa de agar duro com a mão direita e imediatamente despeje todo o conteúdo do tubo sobre a superfície de uma placa de agar rígido. Antes de fechar a tampa, agitar a placa suavemente, mas rapidamente para espalhar o agar macio derretido sobre toda a superfície da placaantes que tenha tempo para solidificar. Evitar salpicos do ágar mole derretido para os lados da placa de Petri. Feche a tampa.
- Repita a etapa # 9 para todos os tubos de adsorção, um tubo de cada vez.
- Coloque as placas em uma superfície plana e permitir-lhes ficar imóveis até que o ágar mole é solidificado.
- Normalmente 30 minutos é suficiente.
- Inverta as placas durante a incubação.
- Múltiplas placas podem ser empilhadas e coladas então invertido para a incubação.
Após a incubação, as placas podem ser inspeccionado para placas. O controle negativo deve ter apenas um gramado de bactérias (sem furos de placas indicativas). As placas variar em termos de aparência tamanho, forma e global. Um tipo de fago dada pode ser isolado a partir de uma mistura heterogénea de placas por cuidadosamente perfuração no centro de uma placa com um palito estéril e transferindo o inoculo para uma t microcentrífuga esterilizadoUbe contendo 100-1000 uL de caldo ou tampão fago. Este lisado pode ser revestida usando o mesmo procedimento descrito acima. Pelo menos 3 a 6 sucessivas único-placa isolamentos são necessárias para assegurar que um fago puro foi obtido. Muitas vezes, o lisado deve ser diluído por um grande número (10 -1 a 10 -10) para encontrar um título que produz não sobrepostos placas sobre uma placa. O número varia, dependendo do tamanho da placa.
6. Replica Procedimento Placa: transferência de células mutantes para rastreio e auxotrofos
Esta técnica permite a comparação do crescimento das células em uma placa de primário para placas secundárias, gerando um meio para as células de tela para um fenótipo seleccionável. Primeiro, um primário, ou mestre, placa é inoculada com células, quer por difusão-plaqueamento uma diluição que produz colónias únicas ou transferindo-os para uma placa em um padrão espacial especificada por marcações de grade. Placas secundárias contendo mídia com crescimento inhibos credores ou meios que não tenha um nutriente particular, são inoculados com células de colónias na placa primária. O padrão espacial de colónias é reproduzido primeiro premindo um pedaço de veludo para a placa principal. As células bacterianas aderir ao veludo, porque eles têm uma maior afinidade para o veludo do que para o ágar. O cunho de células na veludo, em seguida, é transferido para placas de múltiplos secundários com o crescimento de células que reflecte o padrão de colónia mesma que a da placa primária. Em outras palavras, é como ter um selo de borracha, a replicação do padrão de crescimento a partir de uma placa para a outra. Esta técnica é vantajoso porque permite que um número relativamente grande de colónias a serem rastreados, simultaneamente, para muitos fenótipos em uma única experiência.
- Preparar placa primária: Label em torno do bordo do fundo (não a tampa) de uma placa de agar com pelo menos o seu nome, a data, e do tipo de meio de crescimento.
- Marcar uma grelha no fundo da placa compelo menos duas linhas verticais igualmente espaçados e pelo menos dois igualmente espaçados linhas horizontais. Número dos quadrados resultantes.
- Use um palito pré-esterilizado para inocular cada quadrado com uma amostra de células. Para cada amostra, dab o centro da praça. Não cubra toda a praça com células (Figura 8) ou então a amostra overgrow quando incubada e contaminar praças vizinhas.
- Cada quadrado irá ser inoculados com uma amostra diferente, derivado quer a partir de culturas de caldo ou colónias sobre uma outra placa.
- Inverta a placa e incubar primário, que será usado para inocular vários meios secundários.
- Inocular as placas secundárias: Pilha a placa principal e todas as placas secundárias. Com um marcador, fazer uma marca de orientação no lado das placas. Assegure-se a marca está no lado inferior de cada placa, e não a tampa.
- Obter um pano de veludo estéril e coloque-o no bloco cilíndrico (Figura 9). Bloquear o pano de veludo no lugar com o titular. Observe a marca de orientação sobre o bloco.
- O bloco deve ser o mesmo tamanho que o fundo de uma placa de Petri (10,2 cm de diâmetro). Deve vir com um anel de travamento que grampos de pano a veludo para o bloco, enquanto placas de réplicas.
- O pano de veludo (15,2 x 15,2 centímetros quadrados) devem ser pré-esterilizado. Empilhar 10 ou 12 quadrados limpas, em seguida, envolvê-los em folha de alumínio, em seguida, colocá-los na autoclave a 121 ° C no ciclo seco (configuração gravidade) durante 30 minutos. Antes de usá-los em um experimento de replicação em placa, certifique-se que estão completamente secos, colocando-os em um forno quente por várias horas. Note que praças veludo pode precisar de ser de-linted com fita adesiva antes da esterilização.
- Quadrados Velveteen pode ser re-utilizado. Depois de quadrados de veludo utilizados foram descontaminados na autoclave, que deve ser lavado em água pura e re-esterilizado, tal como descrito acima.
- O bloco cilíndricok eo grampo deve ser desinfectada entre utilizações, com um breve lavagem em 70% (v / v) de etanol ou lixívia de cloro de 10%.
- Remover a tampa e inverter a placa primária. Alinhe a marca de orientação sobre a placa com a marca do bloco. Baixar a placa de modo a que a superfície do ágar está em contacto com o tecido de veludo sobre o bloco cilíndrico. Levemente, mas igualmente pressionar para baixo com as pontas dos dedos na parte de trás da placa principal e, em seguida, retire cuidadosamente a placa primária fora do bloco. Colocar a tampa sobre a placa.
- A impressão de veludo de células a partir da placa primária pode ser usado para inocular placas de cerca de 7-8 secundário antes uma impressão com um necessidades de veludo novos a serem feitas.
- Repita a etapa # 7 com cada uma das placas secundárias.
- Como controlo positivo, a última placa na série deve ser um meio de agar em que todas as estirpes testadas devem crescer. Desta forma, você pode confirmar que as células foram transferidas para todas as pl secundáriaates na série. Caso contrário, a falta de crescimento sobre um meio de ensaio particular pode ser atribuída a transferência de células insuficiente, em vez de um fenótipo da estirpe.
- Para evitar falsos positivos, as placas secundárias devem ser ordenados do menos para o substrato mais favorável. Caso contrário, os nutrientes podem ser transferidos entre as placas permitindo que as células a crescer em meio desfavorável.
- Inverter as placas e incubar.
- Nota: Ao inspecionar as placas secundárias para o crescimento, certifique-se de distinguir entre o crescimento ea uma impressão. O último é um resultado negativo.
7. Limpando o Espaço de Trabalho
- Desligue o bico de Bunsen, em seguida, pôr de lado todos os suprimentos, incluindo culturas em placas ou em tubos, meios de extras, e outros reagentes.
- Não permita que antigas culturas, seja em chapas ou em tubos, para acumular na bancada do laboratório ou em áreas de armazenamento. Estas amostras, que são fontes notórias de contaminaçãotais como moldes e indesejados espécies bacterianas, devem ser descartados logo que não são mais necessários.
- Lugar contaminado material de laboratório (luvas, ponteiras e kimwipes), vidro (tubos, frascos, garrafas), e resíduos perigosos (culturas bacterianas ou soluções de fagos, placas usadas) no receptáculo eliminação adequada. Ao trabalhar com um E. não-patogênica coli estirpe (BSL-1), só os resíduos perigosos não infecciosa é gerado. Ao realizar os mesmos procedimentos com os organismos patogénicos (BSL-2 ou superior), resíduos perigosos infecciosa é gerado. Independentemente da classificação de risco biológico, resíduos perigosos devem ser autoclavados ou desinfetados antes de ser descartado. Siga as orientações descritas na BMBL (5 ª ed.), Bem como as fornecidas pelo seu institucional Saúde Ambiental e do departamento de Segurança para a eliminação imediata e adequada de riscos biológicos gerados durante um experimento.
- Limpe a área de trabalho com desinfetante.
- Lavar as mãoscom sabão anti-séptico e água morna antes de deixar o laboratório.
8. Os resultados representativos
Streak-placa Technique. Uma aplicação de amostra para plaqueamento raia é mostrado na Figura 1. Este procedimento é usado para isolar colônias de bactérias a partir de culturas de células mistas e é de longe uma das técnicas mais importantes para mestre em microbiologia e genética molecular. Cada colónia representa uma população de células que são geneticamente idênticos. Para muitas aplicações a jusante é imperativo para iniciar com qualquer colónia um único ou uma cultura pura bacteriana gerada pela inoculação de meios de comunicação com as células a partir de uma única colónia. Por exemplo, a morfologia das células individuais dentro de uma colónia pode ser inspeccionado usando um microscópio de luz. Identidade genética pode ser atribuído a sequenciação do gene da subunidade pequena de RNA ribossomal a partir de DNA genómico isolado a partir de uma cultura de células começou com uma única colónia.E características metabólicas pode ser descrito submetendo as células a vários ensaios bioquímicos e fisiológicos. Apenas através da realização de tais experiências com culturas puras podem ter a certeza de uma das propriedades atribuídas a um microorganismo particular. Os resultados não são obscurecidos por a possibilidade de que a cultura é contaminada. Erros técnicos pode ocorrer se a esterilidade do instrumento utilizado para raia as células ao longo da placa não é mantido durante todo o procedimento. Esquecer-se de chama um laço ou recuperar um palito fresca entre quadrantes tornar difícil a obtenção de colónias únicas. Algumas espécies bacterianas não pode ser isolado em cultura pura como eles são dependentes de uma associação de cooperação com outras espécies bacterianas para certos requisitos de crescimento. Referida como syntrophs, estes organismos podem apenas ser cultivadas sob condições de co-cultura, de modo colónias (se formou) sempre será constituída por duas ou mais espécies. Um outro desafio encontrado no laboratório durante a execução doraia-placa procedimento com bactérias derivadas a partir de amostras ambientais é que as células apresentam características de crescimento que se desviam a partir de estirpes laboratoriais tradicionais, tais como E. coli. Tais estirpes bacterianas podem produzir colónias que são filamentosa (em oposição a aglomerados apertados de células) com ramificações que se espalham sobre uma grande parte de uma placa de ágar, calcificada e, assim, refractário à penetração por um instrumento raia-placa, ou rodeado por uma cápsula pegajoso de modo que as colónias individuais não podem ser discernidas. Estas características tornam difícil de purificar colónias únicas pela técnica raia-placa.
Pour-placa Technique. Com a técnica de fluidez-placa, as colónias formar dentro do agar, bem como sobre a superfície do meio de ágar proporcionando assim um meio conveniente para contar o número de células viáveis em uma amostra. Este procedimento é utilizado numa variedade de aplicações industriais. Por exemplo, é crítico para um Tre águas residuaisatment planta, que é responsável pela limpeza de resíduos líquidos (por exemplo, esgoto, escoamento de bueiros) gerado pelas propriedades domésticas, comerciais e industriais, bem como práticas agrícolas, para analisar amostras de água após o processo de purificação extensiva. Água residuária tratada (água não potável) é reutilizada em uma variedade de formas - para a irrigação de culturas não alimentares na agricultura, para uma descarga sanitária em residências e em torres de resfriamento industriais - por isso deve estar livre de contaminação química e microbiana. A água potável (água potável) deve ser purificado de acordo com os padrões da EPA e é testado usando métodos de revestimento microbiológicos que permitem que a enumeração de agentes patogénicos humanos específicos. Mostrada na Figura 10 são colónias bacterianas resultantes de células de bactérias presentes em uma amostra de água recolhida a partir de uma fonte de abastecimento público. É improvável patógenos bacterianos produzidos nessas colônias, dadas as medidas de purificação de água potável, no entanto, os micróbios são cadaonde e contaminação por mesmo não patogénicas estirpes podem ser apenas minimizada, não inteiramente eliminada. Como outro exemplo, uma companhia farmacêutica tem de avaliar o grau de contaminação microbiana, ou materiais biológicos, de um novo medicamento durante a produção, armazenamento e transporte. Por amostragem do fármaco durante as várias fases do processo e as amostras de revestimento utilizando o procedimento de fluidez-placa, a carga microbiana, ou o número de bactérias contaminantes, podem ser prontamente determinada. Medidas de precaução, em seguida, podem ser concebidas para minimizar ou eliminar a contaminação microbiana. Um dos erros mais comuns técnicas que ocorre ao executar a técnica de Pour Plate é insuficiente mistura da amostra com as colônias de agar derretido fazendo com que se aglutinarem tornando contagens impreciso. Outro erro freqüente é derramar o agar derretido quando está muito quente, matando muitas das células bacterianas na amostra. Este erro também irá afetar a precisão das contagens dando números que ª sub-representaçãoe número total de unidades formadoras de colónias na amostra.
Propagação-placa Technique. A técnica de espalhamento em placa é análogo ao procedimento de fluidez-placa na sua utilidade como um meio para realizar contagens de placas viáveis. No entanto, porque as colónias que formam utilizando a técnica de espalhamento em placa-se uniformemente distribuído por toda a superfície do meio de ágar, as células de colónias individuais podem ser isolados e utilizados em subsequentes manipulações experimentais (por exemplo, como o inoculo para uma faixa de placa ou de um caldo de cultura). Três aplicações comuns em que a técnica de espalhamento em placa-é um componente importante são as experiências de selecção de enriquecimento, e de triagem. Em todos os três aplicações, o tipo de célula desejado pode ser separado da mistura e depois submetido a qualquer número de testes bioquímicos, fisiológicos, ou genética.
Um experimento de enriquecimento envolve plaqueamento uma cultura mista em meio ou em placas de incubação eAMBIENTE condições que favorecem o crescimento desses microrganismos no interior da amostra que demonstram as propriedades metabólicas desejados, características de crescimento, ou comportamentos. Esta estratégia não inibe o crescimento de outros organismos, mas os resultados em um aumento no número de microorganismos relativo desejado para os outros na cultura. Assim, as colónias que formam sobre uma placa de enriquecimento provável exibem propriedades fenotípicas que reflectem o genótipo desejado. Por exemplo, se o seu objectivo é cultivar bactérias fixadoras de azoto a partir de uma amostra ambiental contendo uma mistura de mais de 1000 diferentes espécies bacterianas, em seguida, a amostra plaqueamento em meio deficiente em azoto irá enriquecer para estas bactérias que podem produzir este composto a partir do atmosfera usando as capacidades metabólicas fornecidos por um conjunto de genes necessários para a fixação de nitrogênio.
Um experimento de seleção envolve plaqueamento uma cultura mista em um meio que permite que apenas as células que conTain um gene particular, ou conjunto de genes para crescer. Este tipo de experiência é comum em laboratórios de biologia molecular ao transformar estirpes bacterianas com plasmídeos contendo os genes de resistência a antibióticos. Se seu objetivo é cultivar apenas as células recombinantes, ou aqueles que conseguem assumiu o plasmídeo, em seguida, chapeamento da amostra em um meio que foi complementada com uma concentração adequada do antibiótico vai selecionar para as células que apresentam uma resistência a esta droga particular.
Uma experiência de triagem envolve plaqueamento uma cultura mista em um meio que permite que todas as células viáveis para crescer, no entanto, as células com o genótipo desejado pode ser distinguida de outras células com base na sua fenótipo. Novamente, este tipo de experiência é comum em laboratórios de biologia molecular ao realizar ensaios de mutagénese ou genes em plasmídeos de clonagem. Um exemplo clássico, como mostrado na Figura 11, faz uso do gene lacZ ENCODing β-galactosidase; esta enzima permite que as células para metabolizar X-Gal (5-bromo-4-cloro-3-indolil-β-D-galactopiranósido), um análogo de substrato do seu substrato natural, lactose. A clivagem de X-Gal por β-galactosidase resulta em um produto insolúvel azul. Assim, se um meio contém X-gal, e uma amostra contendo células quer com um gene lacZ de tipo selvagem (funcional) ou mutante (não-funcional) são plaqueadas neste meio, em seguida, após incubação células do tipo selvagem que abrigam um lacZ funcional gene aparecerá como azul-pigmentadas colônias, enquanto as células mutantes com um gene lacZ não-funcional aparecerá como sem pigmento ("branco") colônias.
Um problema técnico encontrado com mais freqüência quando primeiro aprender a executar a técnica de espalhamento em placa está irregular propagação de células em toda a superfície do ágar. Quando se utiliza uma mesa giratória e vareta de vidro, a amostra pode ser absorvido demasiado rapidamente de tal modo que as colónias formar apenas perto do centro da placa. Wgalinha fazendo o "Método Copacabana", as pérolas de vidro são rodadas em vez de agitado em toda a superfície do ágar. Consequentemente, muitas colónias crescer ao longo da borda exterior da placa. Em qualquer caso, a distribuição resultante de colónias não tirar proveito da área de superfície disponível para as células completas podem se aglutinarem e crescer em colónias sobrepostas fazendo contagens de placas impreciso ou distinção de tipos de células inviável.
Técnica Overlay macio Agar. Um procedimento semelhante à técnica de espalhamento em placa-usado para contar as colónias bacterianas pode ser usado para contar o número de fagos. Considerando que entre 30 e 300 células bacterianas são espalhadas sobre a superfície do ágar para contagem de placas (CFU / ml), entre 100 e 400 partículas fágicas infecciosas são misturados com 10 8 a 10 9 células hospedeiras para a contagem de placa (ufp / ml) dentro de uma camada de agar mole espalhar sobre a superfície de ágar nutriente rígido. A não ser que demonstrado o contrário, é geralmente assumido tcélula um chapéu único bacteriana divide e acumula um grande número de células geneticamente idênticas em um único cluster chamada de colônia. Como discutido anteriormente, esta hipótese não é válida quando as células crescem em cachos (ou seja, pares, tétrades, cadeias ou clusters) ou exibir características de crescimento, tais como cápsulas que impedem a formação única colônia. Uma suposição semelhante é feita para a formação de placa, em que cada placa representa a actividade de um fago único. Esta afirmação é verdadeira apenas se um fago infecta uma bactéria. O que acontece se várias partículas de fago infectar uma única bactéria? Este problema relaciona-se com um parâmetro importante estatística que deve ser considerado quando se realizar as experiências com o fago - multiplicidade de infecção (MOI) - descrevendo a proporção de partículas de fago infecciosas para o número de células hospedeiras numa amostra. Porque alguns adsorver células mais do que um fago enquanto que outras células adsorver fago apenas uma ou nenhuma, uma população de células hospedeiras deve ser infectadas a uma MOI de baixo (≤ 1) tó minimizar a probabilidade de que uma célula será infectado por mais do que uma partícula de fago. Empregando a unidade de placa de conformação (pfu) como uma definição funcional evita estas complicações ao realizar a contagem de placa para calcular o título de um estoque de fago.
Como mostrado na Figura 12, a morfologia da placa varia para fago diferente. Alguns fagos gerar pequenas placas (painel A), enquanto outros dão origem a grandes placas (painel B). Um certo número de variáveis que afectam o tamanho de placa. Há razões técnicas que contribuem para essa variabilidade. Por exemplo, meio completo e desenvolvimento de suporte rígido espessa de placas de agar maiores porque as células hospedeiras pode sustentar o crescimento de fagos por um longo período de tempo. Uma densidade de plaqueamento elevado de células hospedeiras (> 10 9 ufc por placa) irá causar uma redução no tamanho da placa. Usando concentrações mais baixas de agar mole irá aumentar a taxa de difusão de partículas de fagos no agar mole e assim aumentar o tamanho das placas. Lembre-tha esta taxa de difusão aumento pode ocorrer acidentalmente se as placas de ágar rígidos não são completamente secas, tais humidade que a condensação ou em excesso no prato dilui o agar mole na cobertura. Esta supervisão técnica irá produzir resultados inconsistentes com relação ao tamanho da placa para um fago particular.
Tamanho da placa também está relacionado com um número de eventos de células hospedeiras, incluindo a eficiência de adsorção, a duração do período de latência (o tempo de duração do fago adsorção à lise da célula hospedeira), eo tamanho de rebentamento (o número de descendentes libertado por uma única infecção). Uma mistura heterogénea de tamanhos de placa pode ser observado se partículas de fago infectar células hospedeiras de diferentes fases de crescimento bacteriano. Por exemplo, aqueles que adsorver durante fase exponencial de crescimento fazer placas maiores com fago mais progenitura do que aqueles que adsorvem na fase exponencial tardia. Como regra geral, fago lítico produzir placas claras enquanto lisogénico fágicas placas turvas formulário. Hoto, alguns fago lítico produzir padrões interessantes, como o "olho de boi" placa mostrada na figura 12B. Estas placas claras estão rodeados por um halo turva porque essas células na borda da placa não são totalmente lisadas ou podem ser resistentes aos fagos infecção. Um "olho de boi" padrão observado com fago temperado é uma placa com um centro turva cercado por um anel claro. Esta morfologia reflecte a MOI e da fisiologia da célula hospedeira com respeito à decisão de lise lisogenia. Quando as células são infectadas com o primeiro fago, o MOI é baixa e as células crescem rapidamente porque os nutrientes são abundantes; juntos o que facilita o crescimento lítico. Como mais e mais células são lisadas, o MOI aumenta e forma-se uma placa claras. Lysogens no centro da placa, no entanto, continuam a crescer, porque eles são imunes a lise dando origem a uma placa transparente com um centro turva.
A técnica de sobreposição pode ser modificada para ensaios de placa com vírus eucarióticos. Emda mesma maneira placas de bacteriófagos forma sobre um tapete de células bacterianas em agar mole, vírus eucarióticos formar placas sobre uma monocamada de células cobertos por um gel. Uma monocamada confluente é uma folha de células em crescimento lado a lado sobre a superfície de um prato de cultura, tocam um no outro, mas não crescente no topo de uma outra. Para levar a cabo este tipo de ensaio de placa, alíquotas de vírus são adicionados a monocamadas sensíveis de células eucarióticas. Em seguida, a monocamada é coberta com um meio de agarose baseado nutriente - este gel restringe a propagação de vírus de progenitura libertados de células infectadas para as células adjacentes na monocamada. Por conseguinte, uma área esférica, ou placa, é produzido que contém as células danificadas por libertação de viriões. Para ajudar a visualização das placas, corantes que as células vivas mancha pode ser aplicado para a cultura de células fornecendo contraste entre as células infectadas e não infectadas.
A técnica de sobreposição soft-agar é utilizado para outras experiências do que ensaios de placa. Primeiro, é significant lembrar que o ágar nutriente é difícil uma matriz de suporte que permite o crescimento de bactérias. Em segundo lugar, o agar macio usado para a sobreposição pode ter uma composição diferente do que o nutriente ágar rígido. Desta forma, o agar macio-pode servir como um meio para estirpes ensaio bacteriano de várias características de crescimento ou propriedades metabólicas. Por exemplo, a técnica de sobreposição é usado para as bactérias de tela para a capacidade de degradar a celulose (Teather e Wood, 1982). Colónias únicas são cultivadas em meio de ágar não selectivo rígido, em seguida, macio-ágar contendo 0,1% (w / v) carboximetil celulose (CMC) é espalhada sobre a superfície do ágar rígido. Após a incubação, as placas são inundado com mancha que permite a visualização de zonas de compensação à volta das colónias no ágar macio. A compensação é causada por enzimas hidrolíticas secretadas pelas bactérias quebrar a celulose no meio. Mais recentemente, a técnica de sobreposição tem sido utilizado para detecção de bactérias que inibem o crescimento de methanogeArchaea nic encontrado no rúmen de animais (Gilbert et al. de 2010). As bactérias isoladas de amostras ambientais são cultivadas em um meio difícil de ágar nutriente, então colônias são sobrepostos com soft-agar contendo uma cultura de microrganismos metanogênicos. Após a incubação, as placas são inspeccionadas para zonas de inibição de crescimento à volta das colónias. Este método identifica estirpes bacterianas que produzem inibidores das metanógenos no agar mole.
Os erros mais comuns técnicos que ocorrem com a técnica de sobreposição de soft-agar estão chegando a derreter-ágar macio ou quando está muito quente ou muito fria. Se for muito quente, as células bacterianas misturado no meio serão mortos antes de plaqueamento. Se for demasiado fria, em seguida, o agar macio-irá formar grumos quando derramado sobre o ágar rígido. Em ambos os casos, os resultados vão ser ambígua ou ilegível na melhor das hipóteses.
Replica placa-Processo. Transferir culturas a partir de um tipo de meio nutrientepara o outro para testar requisitos de crescimento torna-se bastante laborioso se houver mais do que apenas algumas estirpes. Plaqueamento de réplica é um método que permite a triagem simultânea de um grande número de microorganismos. Por exemplo, após mutagenizing uma cultura de células do tipo selvagem, uma pode espalhar-placa diluições da cultura para obtenção de placas com colónias únicas. As placas primários contêm um meio que suporta o crescimento de todas as células, incluindo tipo selvagem prototrophs, que sintetizam todos os compostos necessários para o crescimento, e auxotrofos mutantes, que transportam uma mutação genética numa via biossintética tornando-os incapazes de sintetizar compostos particulares essenciais para o crescimento. Através do plaqueamento da mistura de células em um meio completo, os nutrientes em falta pode ser tomada a partir do meio ambiente. Para distinguir entre prototrophs e auxotrofos, as colónias podem ser replicados para um meio mínimo. Apenas prototrophs será capaz de crescer. Porque o padrão espacial da placa principal é preservada, comparisem da placa secundária com a placa primária permite a identificação de colónias mutantes. Para determinar quais os mutantes composto já não são capazes de sintetizar, as colónias podem ser replicados em suporte mínimo suplementado com compostos específicos (por exemplo, aminoácidos, fontes de carbono, vitaminas, etc.) Desta forma, centenas de colónias podem ser rastreados, ao mesmo tempo utilizando o procedimento de réplica-placa. Um erro técnico que poderia ocorrer se utilizando placas de ágar que são demasiado molhado, causando colónias para esfregaço juntos contaminando todas as culturas sobre a placa. Isso produz resultados que são inteiramente confiáveis. Outra técnica de erro é a aplicação de pressão demasiado quando da transferência de células a partir do veludo para as placas secundárias. Mais uma vez, após incubação das placas secundárias, as colónias resultantes podem sobrepor-se a produção de fenótipos de crescimento atribuídos a contaminação, em vez de auxotrofia.
Nem todas as espécies do tipo selvagem microbianos são prototrophs, de modo a réplica-pprocedimento tarde pode ser usado para simultaneamente tela diferentes estirpes do tipo selvagem para os requisitos de crescimento característicos. Como mostrado na Figura 13, "dabs" de células provenientes de quatro diferentes estirpes de Pseudomonas bacterianas foram plaqueadas em duplicado em uma placa de grade marcado contendo meio completo chamado YTA (painel A). As estirpes foram então replicado em três placas secundárias (painéis B, C e D), composto de meio mínimo (MSA), suplementado com uma fonte de carbono diferente (acetamida, lactose, e glicina, respectivamente). Os resultados demonstram que dois dos quatro estirpes de Pseudomonas (P. aeruginosa e P. stutzeri) são incapazes de crescer sobre estas três fontes de carbono. Como um controlo, as estirpes foram replicados para uma placa de quarto com meio YTA para confirmar as células foram transferidas durante todo o procedimento. Uma vez que todas as quatro estirpes de crescer na placa de controlo YTA, as deficiências de crescimento expostos nas anteriores três placas na série são fiáveis. Os resultados réplica de placas-estão tabulados na Tabela 1. Um erro comumente cometido é interpretar uma marca de crescimento em um prato secundário, um resultado positivo. Por exemplo, compare o fenótipo de P. aeruginosa para que de P. stutzeri em MSA + acetamida (painel B). Este último apresenta uma marca de crescimento, que é um resultado negativo, e pode ocorrer se nutrientes a partir da placa anterior são transferidos com as células-mãe. No crescimento de novas células ocorre porque os nutrientes em falta não estão disponíveis para as células descendentes. É fácil confundir uma marca com um crescimento real. Em caso de dúvida, o experimento deverá ser repetido utilizando um método alternativo, como raia de revestimento de células da placa primária em meios secundários.
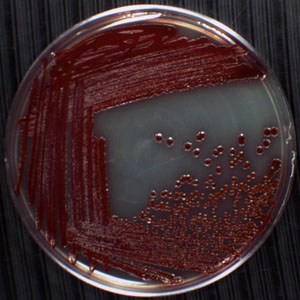
Figura 1. Exemplo de colónias únicas em uma placa. As esferas-de-rosa, perto do centro da placa são colónias de Serratia marcescens, uma bactéria Gram negativa, Proteobacterium de forma cilíndrica na família Enterobacteriaceae. Devido à sua preferência por ambientes úmidos, este microrganismo é comumente encontrada crescendo nos cantos de banheiras, nas bacias de escoamento, na argamassa, azulejos e em cortinas de chuveiro. S. marcescens é fácil de reconhecer, uma vez que produz um pigmento vermelho chamado prodigiosina. As colónias sobre esta placa foram gerados utilizando a técnica de raia-placa, com colónias únicas que aparecem na incubação quarto quadrante seguindo a 30 ° C durante 24 horas. Os outros três quadrantes mostram crescimento confluente em que as células depositada sobre a superfície do ágar desenvolvido em colónias que se sobrepõem.

Instrumentos Figura 2. Usado para raia placa técnica. De cima para baixo, mostra são palitos (achatados, não volta), um laço de arame, um laço de plástico descartável, e varas de madeira. Palitos são tipicamente transferidospara uma proveta de vidro de pequeno com a extremidade larga para baixo, em seguida, coberta com uma folha de quando autoclavadas a esterilizar antes da utilização. Varas de madeira são transferidos para 18 tubos de ensaio mm então autoclavada para esterilizar antes da utilização.
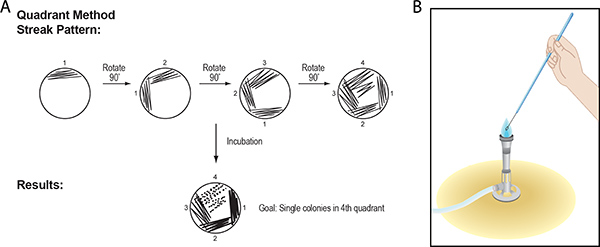
Figura 3. (A) a técnica Streak-placa usando o método de quadrante. Um ciclo de pré-esterilizado, vara ou palito é usado para espalhar a amostra através de um quarto da superfície do agar com um rápido, suave, o movimento de vai-e-quarto a partir do aro para o centro da placa. Esta acção é repetida para cada um dos quatro quadrantes da placa. Após a incubação, o crescimento celular aparece ao longo do caminho do instrumento utilizado para depositar as células na placa. A separação mecânica das células de uma amostra mista usando esta técnica deve resultar em colónias únicas no quarto quadrante (ver Figura 1 para um exemplo). Colónias únicas são referidos como unidades formadoras de colónias (ufc). (B) Quando um anel de metal é utilizado para raia-chapeamento, ele deve ser esterilizados usando a chama de um bico de Bunsen antes do contacto com o inoculo ou o meio de agar. Lembre-se que a parte mais quente da chama é a ponta do cone azul. Segurando o identificador do aparelho, colocar o fio na chama aproximadamente 3-4 polegadas a partir do ciclo. Deixe o tempo suficiente para o fio para se tornar vermelho quente. Mover o fio de modo a chama se aproxima do loop. Certifique-se o anel de metal é resfriado antes de tocar o inóculo.

Figura 4. Técnica Pour-placa. (A) Um pequeno volume de amostra (entre 0,1 e 1,0 ml) é dispensada assepticamente em um prato de Petri vazia, mas estéril com o uso de um 5,0 ml serológica pipeta. (B) de agar derretido equilibrada a uma temperatura de aproximadamente 48 ° C é então vertida em placa de Petri com a amostra. Depois de fechar a tampa, a placa é agitado suavemente para misturar a amostra eagar fundido. O ágar é deixada solidificar durante cerca de 30 minutos, em seguida, as placas são invertidos para a incubação.
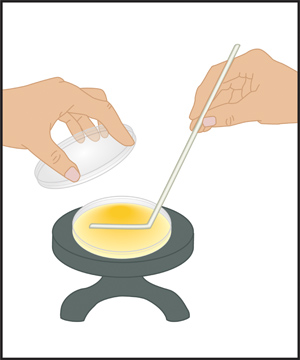
Figura 5. De espalhamento em placa-técnica com um gira-discos e espalhador de vidro. Depois de a placa de agar é colocada sobre uma mesa giratória, um pequeno volume de amostra (0,1 a 0,2 ml) é dispensada assepticamente para o centro da placa utilizando uma micropipeta. O difusor é esterilizada por mergulhando-o num copo de etanol, em seguida, passando-o através da chama do queimador de Bunsen para inflamar o etanol em excesso. Antes de fazer contacto com a amostra, o espalhador deve ser arrefecido por tocá-lo para o ágar perto da borda da placa. O difusor é levemente deslocado para trás e para frente através da amostra através da placa, enquanto o prato está lentamente girando. Esta acção permite gradual, mas um espalhamento uniforme da amostra através da superfície de agar. Depois de fechar a tampa, a placa deve ser definido no u bancadandisturbed durante pelo menos 5 minutos para permitir que a amostra para absorver completamente para dentro do ágar antes de invertendo a placa para a incubação.
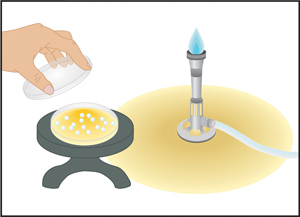
Figura 6. De espalhamento em placa-técnica com pérolas de vidro (Método Copacabana). Pérolas de vidro que foram pré-esterilizados em autoclave são vertidos sobre a superfície de uma placa de agar sentado sobre o topo da bancada. Um pequeno volume de amostra (100 a 150 uL) é dispensada assepticamente para o centro do agar usando uma micropipeta. Com a tampa da placa de fechada, um movimento horizontal agitação é utilizado para mover suavemente os grânulos frente e para trás ao longo da placa de 6 a 7 vezes, espalhando-se a amostra. Esta acção é repetida após a rotação do prato 60 °. O movimento agitação é repetido uma terceira vez na sequência de uma outra rotação de 60 °. Uma vez que a amostra foi completamente absorvida para o meio de agar, as esferas são decantado para um copo contendo lixívia a 10%. Oplacas, em seguida, são invertidos para a incubação.
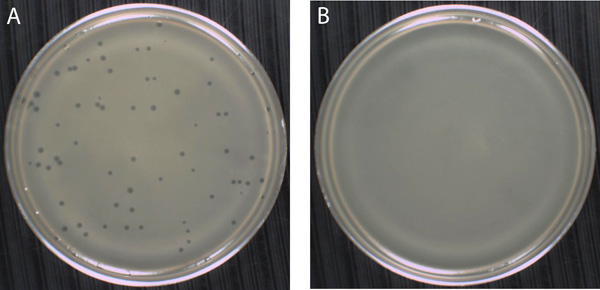
Figura 7. Soft-ágar técnica de sobreposição usado para isolar e enumerar fago com base na formação de placas (também chamado um ensaio de placa). (A) A presença de fagos pode ser detectado como zonas de compensação, ou placas, com uma suspensão confluente de colónias de bactérias que crescem no ágar macio. Fago T4 é uma virulenta, fago DNA de cadeia dupla que infecta seu hospedeiro, Escherichia coli, fazendo com que as células hospedeiras para lisar e libertar fago descendência. Após várias rodadas de infecção e lise, o E. vizinho células de Escherichia na área imediata ao redor da célula infectada host original desaparece, deixando uma placa contendo bilhões de partículas de fago T4. Fago T4 produz placas que são aproximadamente 1 mm de diâmetro. Nesta experiência, 200 uL de uma diluição de 10 -5 uma 2 x 10 8 estoque pfu / ml de fago T4foi misturada com cerca de 300 uL de E. células indicadoras coli preparado como um crescimento exponencial cultura, gaseificado, a 37 ° C. Tanto o fago e bactérias foram adicionados a um tubo de agar macio EHA, misturado, em seguida, vertida sobre a superfície de uma placa de agar EHA rígido. Note-se que não era necessário para permitir que o fago e bactérias para adsorver antes do plaqueamento neste caso. Depois de permitir que o agar macio para solidificar sem ser perturbado por 20 minutos, as placas foram invertidas e incubadas a 37 ° C durante 24 horas. (B) Na ausência de infectar as partículas de fagos, os resultados crescimento bacteriano em uma suspensão turva de células no agar mole em que as colónias discretas não são visíveis. Em vez disso, um relvado mesmo de células bacterianas, neste caso E. coli, as formas em toda a camada de ágar inteiro moles.
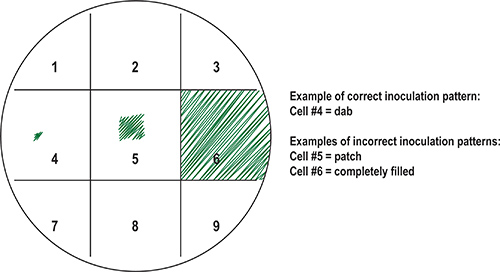
Figura 8. Preparação de placa primária (principal) com amostras bacterianas. Para manter as amostras organizados, o fundo da placa pode ser marcado para uma grelha de quadrados e resultantes numerados. Cada amostra pode ser atribuído um quadrado na grelha. São mostrados exemplos de padrões de inoculação correctas em relação incorrecta. Idealmente, um pequeno número de células são transferidas para o centro do quadrado usando uma ferramenta de inoculação estéril tal como um palito de dentes para "dab" a amostra (célula # 4). Erros comuns de inoculação, como aqueles mostrados na célula # 5 ("patch") e celular # 6 ("fill"), resultado no crescimento de amostras de bactérias após a incubação, consequentemente contaminando quadrados adjacentes.
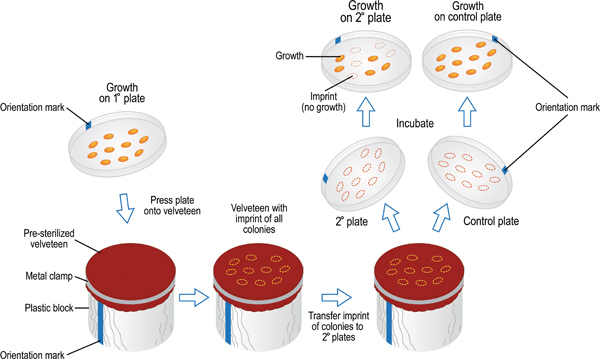
A Figura 9. Réplica-placa técnica utilizada para transferir células de primário para placas secundárias para telas de fenótipo. A marca na placa de primário é alinhada com a marca sobre o bloco de veludo coberto, em seguida, Lowe vermelho para permitir que a superfície do ágar para contacto com o tecido. As células são transferidas a partir da placa para o veludo levemente mas uniformemente pressionando para baixo sobre a placa primária com as pontas dos dedos. Esta acção irá deixar uma marca das amostras de células sobre o veludo no mesmo padrão espacial, a placa principal. O mesmo procedimento é utilizado para transferir as células a partir do veludo para uma placa secundária. Tal como muitos como 7-8 placas secundárias podem ser inoculados com a impressão mesma placa primário sobre a veludo. A última placa inoculada a partir do veludo deve servir como um controlo positivo. Deve ser um meio que suporta o crescimento de todas as cepas testadas, garantindo a transferência de células suficiente ocorreu durante toda a série de placas. Após a incubação, as placas secundárias podem ser inspeccionadas e pontuados para crescimento versus nenhum crescimento. Assim, múltiplas estirpes bacterianas podem ser rastreados em simultâneo em meios de crescimento em vários uma única experiência.
_upload/3064/3064fig10.jpg "/>
Figura 10. Exemplo de resultado usando Pour Plate-técnica. Uma amostra de 1,0 ml de água coletada de um bebedouro público foi dispensado em uma placa de Petri esterilizada vazia. Em seguida, derreteu, mas YTA arrefecida foi vertida em o prato com a amostra. O ágar também continha 100 ug / ml cicloheximida para prevenir o crescimento de bolores e leveduras que possam ter estado presentes na amostra de água. Após agitação suave a mistura, a placa foi colocada sobre uma superfície plana e agar foi deixada a solidificar completamente. A placa foi incubada a 37 ° C durante 48 horas. É mostrado o resultado desta experiência. Note-se a diferença na aparência de colónias de superfície, que são grandes e de forma circular, versus superfície sub-colónias, que são muito pequenos e de forma irregular moldar porque o meio solidificado inibe colónia propagação em sub-superficial.

Figura 11. </ Strong> Exemplo de resultado usando espalhamento em placa-técnica. O "Método Copacabana" foi usado para placa de uma mistura de células de E. coli para uma experiência de triagem. Neste caso, o meio de crescimento (LB) contém X-Gal, de modo que esses células que expressam um funcional β-galactosidase colónias enzima formar o azul, enquanto as células com uma mutação no gene lacZ e, assim, incapaz de expressar uma forma enzima funcional β-galactosidase colónias brancas. Muitas vezes referida como uma "tela azul / branco", os dois tipos de colônias podem ser facilmente distinguidos um do outro no mesmo prato.

Figura 12. Exemplo de resultado de ensaio de placa usando soft-agar técnica de sobreposição. Mostrado são placas formadas na estirpe hospedeira Mycobacterium smegmatis mc 2 155 (ATCC 700.084) por dois pH diferentesidades: (A) Mycobacteriophage Destroyers e MSSS (B) Mycobacteriophage. Estes fagos foram isolados por alunos do curso de laboratório da UCLA MIMG 103L na Primavera de 2010. M. smegmatis é um Actinobacterium não patogénico e pertence a uma família de micobactérias que inclui um patógenos poucos conhecidos por causar doenças graves, tais como a tuberculose (M. tuberculosis, M. africanum, M. bovis) e da lepra (M. leprae). Cerca de 50 ul de uma diluição de 10 -2 Destroyers e uma diluição de 10 -3 MSSS cada foram incubadas com 500 uL de M. smegmatis durante 20 minutos a 37 ° C, em seguida, misturado com MBTA (agar mole) e vertida sobre MHA (ágar rígido) placas. Depois de permitir que o agar macio para solidificar sem ser perturbado por 20 minutos, as placas foram invertidas e incubadas a 37 ° C durante 48 horas. Note as morfologias de placa distintos produzidos por cada fago. Destruidores (A) forma pequena (diâmetro médio de cerca de 1 mm), placas claras característica de um fago líticoenquanto MSSS (B) desenvolve o "olho de boi" grandes placas com centros claros rodeadas por um halo turvo (diâmetro médio de aproximadamente 3,2 mm). O anel nebuloso pode ser constituída por bactérias que são resistentes aos fagos infecção. Este padrão é distinto do que se formou por lisogénico fago, que produzem placas turvas.
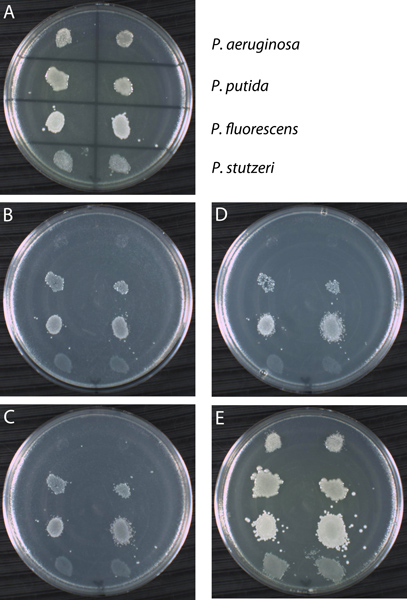
Figura 13. Exemplo de resultado usando réplica da placa-procedimento. Quatro estirpes de Pseudomonas (P. aeruginosa, P. putida, P. fluorescens, e P. stutzeri) foram testados em duplicado para crescimento em três diferentes fontes de carbono: acetamida, lactose e glicina. (A) A placa principal é um meio completo (YTA) inoculadas com as quatro estirpes, como indicado. Após incubação a 30 ° C durante 24 horas, todas as quatro estirpes de crescer em YTA. A placa primária foi utilizado para placa réplica para mínima mesódio (MSA), suplementado com uma única fonte de carbono: acetamida (B), lactose (C), e glicina (D). A última placa da série era um controlo positivo YTA placa (E). Como mostrado, as estirpes mostram padrões de crescimento seguinte variável de incubação sobre as placas secundárias. Note-se que é por vezes difícil distinguir entre o crescimento e uma marca de células. Por exemplo, compare o cunho gerado por P. stutzeri sobre as três placas de MSA para nenhum crescimento em mesmas três placas por P. aeruginosa. Ambos são resultados negativos em comparação com os padrões de crescimento exibido por P. putida e P. fluorescens. No entanto, todas as estirpes que crescem sobre as células de controlo positivo de placas confirmando foram transferidos para todas as placas secundárias na série. Os resultados desta experiência estão tabulados na Tabela 1.
| YTA (Primário) | MSA + acetamida | MSA + lactose | MSA + glicina | YTA (Controle) | |
| P. aeruginosa | + | - | - | - | + |
| P. putida | + | + | + | + | + |
| P. fluorescens | + | + | + | + | + |
| P. stutzeri | + | - | - | - | + |
Tabela 1. Resumo dos resultados do chapeamento de réplica. Crescimento indicado como sinal de adição (+) e nenhum crescimento representado como sinal de menos (-). YTA é um meio completo (ágar de triptona de levedura) e MSA é um meio mínimo (ágar sais mínimos). As placas foram suplementadas com MSA uma única fonte de carbono, como indicado.
Discussão
Microrganismos de cultura envolve um certo número de métodos de revestimento, todas as quais requerem que uma técnica asséptica ser mantida durante toda a manipulação de células e meios de comunicação. Cinco diferentes procedimentos foram descritos neste protocolo. Embora estas técnicas de revestimento são rotineiramente usado para manipular as bactérias e os fagos, eles também podem ser aplicados a cultura de células de mamíferos e de microrganismos eucarióticos comumente utilizados em genética molecular, tais como levedura (ie, Saccharomyces cerevisiae, Cândida albicans, Schizosaccharomyces pombe), algas e protozoários ( isto é, Volvox, Chlamydomonas, Amoeba, Paramecium), e nemátodos (isto é, Caenorhabditis elegans). Há também numerosas (e ainda mais sofisticados) variações de cada método de plaqueamento, dependendo do objectivo experimental ou organismo em estudo. Assim, é importante não só seleccionar a técnica mais apropriada para um microorganismo experiência ou determinado alvo, mas também para adaptar o t tal metodologiachapéu dos resultados experimentais adequadamente abordar a questão ou problema de pesquisa.
Alguns dos aplicativos mais atuais das técnicas de revestimento discutidas neste protocolo envolvem avanços tecnológicos que produzem alto rendimento resultados para experiências de descoberta de triagem e de drogas. Por exemplo, os centros de seqüenciamento do genoma usar o "Método de Copacabana" para bibliotecas propagação chapeamento-clone, que são E. células de Escherichia transformada com os plasmídeos que contêm fragmentos de DNA derivadas do genoma de um microorganismo. Devido dezenas de grandes placas (chamado tabuleiros de bioensaio) são preparados de uma vez, um agitador de placas automatizado é usado para agitar as pérolas de vidro para todo o lote de bandejas. Além disso, ao seleccionar colónias destas placas após a incubação, um seleccionador de colónia robótico é utilizado para recolher células a partir de colónias adequadas como o inoculo para caldo LB em 384 poços de microplacas. Para este ensaio de rastreio de alto rendimento, os princípios processuais da propagação p-técnica tarde aplica, mas a tecnologia permite vários passos para ser automatizado e dimensionadas para permitir um grande número de amostras a serem analisadas simultaneamente e dentro de um curto espaço de tempo.
Empresas farmacêuticas e de biotecnologia investem recursos consideráveis no desenvolvimento de tecnologia de alto rendimento para as técnicas mais básicas de microbiologia e genética molecular. Por exemplo, existem multi-canal micropipetas de realizar transferências de volume até 8 ou 12 amostras de uma vez. Há ainda são estações de trabalho robótico que manobra uma 96-canal cabeça pipeta! Estes esforços envolvem equipes multidisciplinares de cientistas, biólogos emparelhamento que possuem experiência metodológica com os engenheiros e programadores de computador que podem desenvolver a instrumentação necessária para executar as operações mecânicas associadas com os experimentos. Independentemente da aplicação de pesquisa, o objetivo compartilhado por empresas que desenvolvem essas tecnologias é o mesmo - para automatizar laboratóriosry processos, ferramentas, sistemas e instrumentos, tornando-os menos trabalhoso e mais eficiente.
Divulgações
Não tenho nada para revelar.
Agradecimentos
Agradecimentos especiais a Cori Sanders em projetos da IROC para a preparação de ilustrações e Kris Reddi e Shah Bhairav na UCLA para a criação de culturas de amostras e auxiliando com números. O financiamento para este projeto foi fornecido pelo HHMI (HHMI Grant No. 52006944).
Materiais
1. Yeast Tryptone Agar (YTA)
| Name | Company | Catalog Number | Comments |
| Yeast extract | 2.0 g | ||
| Tryptone | 10.0 g | ||
| Agar | 15.0 g | ||
| Distilled water | up to 1000.0 ml | ||
| pH 7.0 |
Autoclave at 121°C for 20 minutes to sterilize. Store at 4°C.
If preparing tubes for the pour-plate procedure, allow the agar to cool to ~55°C then add 2.0 ml of 50 mg/ml cycloheximide. Aseptically dispense 18.0 ml of the melted agar per 18 mm tube then store at 4°C. The agar will solidify and will need to be melted in a steamer or microwave prior to use.
2. Minimal Salts Agar (MSA) + 0.1% (w/v) carbon source
| NH4Cl | 1.0 g |
| NH2HPO4•2H2O | 2.14 g |
| KH2PO4 | 1.09 g |
| MgSO4•7H2O | 0.2 g |
| Carbon source* | 1.0 g |
| Trace salts solution** | 10.0 ml |
| Agar | 15.0 g |
| Distilled water | up to 1000.0 ml |
| pH 7.0 |
Autoclave at 121°C for 20 minutes to sterilize. Store at 4°C.
* Carbon sources used for experiments presented in Figure 13 include acetamide, lactose, and glycine.
** Trace salts solution is prepared in 0.1 N HCl as follows. It is added to the base before sterilization (autoclave at 121°C for 20 minutes).
| FeSO4•7H2O | 300.0 mg |
| MnCl2•4H2O | 180.0 mg |
| Co(NO3)2•6H2O | 130.0 mg |
| ZnSO4.7H2O | 40.0 mg |
| H2MoO4 | 20.0 mg |
| CuSO4•5H2O | 1.0 mg |
| CaCl2 | 1000.0 mg |
| HCl (0.1 N) | up to 1000.0 ml |
3. EHA soft agar (0.65 % w/v)
| Agar | 6.5 g | |
| Tryptone | 13.0 g | |
| NaCl | 8.0 g | |
| Na Citrate•2H2O | 2.0 g | |
| Glucose | 3.0 g | |
| Distilled water | up to 1000.0 ml |
Autoclave at 121°C for 20 minutes to sterilize. Store at 4°C.
4. EHA hard agar (1.2% w/v)
| Agar | 12.0 g |
| Tryptone | 13.0 g |
| NaCl | 8.0 g |
| Na Citrate•2H2O | 2.0 g |
| Glucose | 3.0 g |
| Distilled water | up to 1000.0 ml |
Autoclave at 121°C for 20 minutes to sterilize. Store at 4°C.
5. 1X Middlebrook Top Agar (MBTA soft agar, 0.5% w/v)
| 100 mM CaCl2 stock* | 1 ml |
| 7H9 liquid medium: Neat ** | 50 ml |
| 2XTA *** | 50 ml |
Melt 50 ml of 2XTA and allow it to cool to ~55°C. Using aseptic technique, add the CaCl2 and 7H9 broth to the melted agar. Aseptically dispense 4.5 ml of the mixture per 13 mm tube and store in a 55°C incubator ≤7 days. Cooling MBTA to room temperature or 4°C will cause the CaCl2 to precipitate out of solution.
* 100 mM CaCl2. stock must be stored at room temperature to prevent CaCl2 from precipitating out of solution.
** 7H9 liquid medium: Neat
| 7H9 broth base | 4.7 g |
| 40% glycerol stock | 5 ml |
| Distilled water | up to 900.0 ml |
Mix the base with water then add the glycerol while stirring. Autoclave at 121°C for 20 minutes to sterilize. Store at 4°C.
*** 2X Middlebrook Top Agar (2XTA, 1.0% w/v)
| 7H9 broth base | 4.7 g |
| Agar | 1.0 g |
| Distilled water | up to 1000.0 ml |
Autoclave at 121°C for 20 minutes to sterilize. Dispense 50 ml aliquots into 100 ml bottles and store at 4°C.
6. Middlebrook 7H10 Agar Plates (MHA hard agar, 1.9% w/v)
| 7H10 agar base | 19.0 g |
| 40% glycerol stock | 12.5 ml |
| Distilled water | 887.5 ml |
Mix the agar base with water then add the glycerol while stirring. Heat the solution to boiling then stir for one minute to completely dissolve the base powder. Autoclave at 121°C for 20 minutes to sterilize. Allow the agar to cool to ~55°C then aseptically add the following reagents:
| AD supplement (pre-warmed to 37°C)* | 100 ml |
| 50 mg/ml Carbenicillin ** | 1.0 ml |
| 10 mg/ml Cycloheximide ** | 1.0 ml |
* AD supplement
| NaCl > | 17 g > |
| Albumin (Fraction V)> | 100 g> |
| Dextrose (D-Glucose)> | 40 g> |
| Distilled water> | up to 2000.0 ml> |
Filter-sterilize this solution; do not autoclave. Store at 4°C.
** Filter-sterilize and store these solutions at 4°C for ≤60 days.
7. LB agar (1.5% w/v) + X-Gal (60 μg/ml)
| Tryptone | 10.0 g |
| Yeast extract | 5.0 g |
| NaCl | 10.0 g |
| Agar | 15.0 g |
| Distilled water | up to 1000.0 ml |
| pH 7.5 at 25°C |
Autoclave at 121°C for 20 minutes to sterilize. Allow the agar to cool to ~55°C then aseptically add 3.0 ml of 20 mg/ml X-Gal solution. Freshly prepare X-gal stock by dissolving 400 mg X-Gal in 20 ml dimethylformamide (DMF).
Table of specific reagents:
| Name of the reagent | Company | Catalogue number |
| Yeast extract | Becton Dickenson | 212750 |
| Tryptone | Becton Dickenson | 211705 |
| Agar | Becton Dickenson | 214030 |
| NH4Cl | Acros Organics | 123340010 |
| NH2HPO4•2H2O | Sigma-Aldrich | 30435 |
| KH2PO4 | Fisher Scientific | BP 303-500 |
| MgSO4•7H2O | Sigma-Aldrich | 230391 |
| Acetamide | Sigma-Aldrich | A-0500 |
| Lactose | Fisher Scientific | L6-500 |
| Glycine | Sigma-Aldrich | G-7126 |
| FeSO4•7H2O | Sigma-Aldrich | F8048 |
| MnCl2•4H2O | Sigma-Aldrich | M-3634 |
| Co(NO3)2•6H2O | Sigma-Aldrich | 230375 |
| ZnSO4.7H2O | Sigma-Aldrich | Z4750 |
| H2MoO4 | Acros Organics | 213621000 |
| CuSO4•5H2O | Sigma-Aldrich | 209198 |
| CaCl2 | Sigma-Aldrich | C1016 |
| HCl | Fisher Scientific | A144-212 |
| Cycloheximide | Sigma | 038K1561 |
| Carbenicillin | Cellgro | 46-100-RG |
| NaCl | Fisher | S271-1 |
| Na Citrate•2H2O | Fisher | S279-500 |
| Glucose (Dextrose) | BD | 215530 |
| 7H9 broth base | BD | 271310 |
| 7H10 agar base | BD | 262710 |
| Glycerol | Shelton | IB15760 |
| Albumin (Fraction V) | Fisher | S71907 |
| X-Gal (5-bromo-4-chloro-3-indolyl-β-D-galactopyranoside) | Teknova | X1205 |
| Dimethylformamide (DMF) | Sigma | D4551 |
| Ethanol | Fisher | CDA19 |
| Chlorine Bleach | Chlorox | 02490/06644884 |
| CiDecon (disinfectant) | Decon Laboratories, Inc. | 8504 |
Table of specific equipment:
| Name of equipment | Company | Catalogue number | Experiment |
| Metal loops | American Educational Products | S17352 | Streak plating |
| Disposable plastic loops | Fisher | 22-363-602 | Streak plating |
| Wooden sticks | Fisher | 23-400-104 | Streak plating |
| Flat Toothpicks | American Educational Products | S67859 | Streak plating |
| Turn tables | Fisher | 08-758Q | Spread plating |
| Glass rods | Bellco Glass | NC9004380 | Spread plating |
| 4 mm glass beads | Fisher | 11-312B | Spread plating |
| Velveteen cloth | Bel-Art Products | 09-718-2 | Replica plating |
| Cylindrical block | Bel-Art Products | 09-718-1 | Replica plating |
Referências
- Barker, K. . At the Bench: A Laboratory Navigator. , (1998).
- US Department of Health and Human Services (DHHS). . Biosafety in Microbiological And Biomedical Laboratories (BMBL). , (2009).
- Dulbecco, R., Vogt, M. Some problems of animal virology as studied by the plaque technique. Cold Spring Harbor Symp. Quant. Biol. 18, 273-279 (1953).
- Fields, B. N., Knipe, D. M., Chanock, R. M., Hirsch, M. S., Melnick, J. L., Monath, T. P., Roizman, B. . Fields Virology. 1, (1991).
- Gilbert, R. A., Ouwerkerk, D., Zhang, L. H., Klieve, A. V. Cooperative Research Center for Beef Genetic Technologies. In vitro detection and primary cultivation of bacteria producing materials inhibitory to ruminal methanogens. Journal of Microbiological Methods. 80, 217-218 (2010).
- Jett, B. D., Hatter, K. L., Huycke, M. M., Gilmore, M. S. Simplified agar plate method for quantifying viable bacteria. BioTechniques. 23, 648-650 (1997).
- Karam, J. D. . Molecular Biology of Bacteriophage T4. , (1994).
- Knipe, D. M., Howley, P. M., Griffin, D. E., Lamb, R. A., Martin, M. A., Roizman, B., Straus, S. E. . Fundamental Virology. , (2001).
- Lederberg, J., Lederberg, E. M. Replica plating and indirect selection of bacterial mutants. J. Bacteriol. 63, 399-406 (1952).
- Miller, J. H. . A Short Course in Bacterial Genetics: A Laboratory Manual and Handbook for Escherichia coli and related bacteria. , (1992).
- Mills, K. V., Gareau, J. R., Garcia, A. M. Low-cost modification to the Copacabana method for spreading transformation mixtures. BioTechniques. 39, 188 (2005).
- Jordan, T., et al. . RESEARCH: National Genomics Research Initiative Phage Resource Laboratory Manual. , (2008).
- Sambrook, J., Russell, D. W. . Molecular Cloning - A Laboratory Manual. , (2001).
- Sanders, E. R., Miller, J. H. I. . Microbiologist: A Discovery-based Course in Microbial Ecology and Molecular Evolution. , (2010).
- Slonczewski, J. L., Foster, J. W. . Microbiology - An Evolving Science. , (2011).
- Teather, R. M., Wood, P. J. Use of Congo Red-Polysaccharide Interactions in Enumeration and Characterization of Cellulolytic Bacteria from the Bovine Rumen. Applied and Environmental Microbiology. 43, 777-780 (1982).
- Wise, K. . Preparing Spread Plates Protocols. , (2006).
- Worthington, M., Luo, R. Q., Pelo, J. Copacabana method for spreading E. coli and yeast colonies. BioTechniques. 30, 738-742 (2001).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados