Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
RNAi-Screening auf postembryonalen Phänotypen in Identifizieren C elegans
In diesem Artikel
Zusammenfassung
Wir beschreiben eine Methode, um sensibilisiert postembryonalen Regulatoren der Protein-Expression und Lokalisation in identifizieren C elegans Mit Hilfe eines RNAi-basierter Genom-Bildschirm und einen integrierten Transgen, die eine funktionelle, fluoreszenzmarkierten Protein exprimiert.
Zusammenfassung
C. elegans has proven to be a valuable model system for the discovery and functional characterization of many genes and gene pathways1. More sophisticated tools and resources for studies in this system are facilitating continued discovery of genes with more subtle phenotypes or roles.
Here we present a generalized protocol we adapted for identifying C. elegans genes with postembryonic phenotypes of interest using RNAi2. This procedure is easily modified to assay the phenotype of choice, whether by light or fluorescence optics on a dissecting or compound microscope. This screening protocol capitalizes on the physical assets of the organism and molecular tools the C. elegans research community has produced. As an example, we demonstrate the use of an integrated transgene that expresses a fluorescent product in an RNAi screen to identify genes required for the normal localization of this product in late stage larvae and adults. First, we used a commercially available genomic RNAi library with full-length cDNA inserts. This library facilitates the rapid identification of multiple candidates by RNAi reduction of the candidate gene product. Second, we generated an integrated transgene that expresses our fluorecently tagged protein of interest in an RNAi-sensitive background. Third, by exposing hatched animals to RNAi, this screen permits identification of gene products that have a vital embryonic role that would otherwise mask a post-embryonic role in regulating the protein of interest. Lastly, this screen uses a compound microscope equipped for single cell resolution.
Protokoll
1. Screening-Stamm Bau
Die sorgfältige Gestaltung des Screening-Stamm ist entscheidend für den Erfolg des Bildschirms und wurde an anderer Stelle 3 beschrieben. Für einige Forscher, ist mit einer Belastung, die ein sichtbares Produkt drückt aus einem Transgen für das Experiment benötigt. Viele Stämme mit integrierten Transgenen sind bei den einzelnen Forschern oder CGC. Wenn eine transgene Sorte für den Bildschirm erforderlich ist, aber nicht verfügbar ist, dann kann es unter Verwendung eines veröffentlichten Verfahren wie Bombardement 4, UV / TMP 5 oder 6 Mos Transposon-Insertion werden. Um unsere Protein von Interesse sichtbar zu machen, fügten wir das gfp-codierende Sequenz im Rahmen mit dem reifen cDNA-Sequenz (wir haben DZ-1-Sequenz). Da diese GFP-Fusionsprotein ist nicht sichtbar, durch Binokular, verwendeten wir ein Koinjektions Marker, der sichtbar war und hatte keinen Einfluss auf die Anzeige von unserem Protein von Interesse (TTX-3P :: RFP , für einen Überblick über einige andere Standard-Marker, siehe 7). Wir haben dann erstellt ein integriertes Transgen aus dem extrachromosomalen Array. Wir fanden, dass Bombardierung des Transgens ergab einen niedrigen Kopienzahl integrierter Linie, dass weder rettete den Seziermikroskop Phänotyp noch produziert sichtbaren Ebenen der GFP-markierten Produkt (Beifuß und Gumienny, unveröffentlicht). UV / TMP Integration eines mit mehreren Kopien extrachromosomalen Array ursprünglich durch Injektion 8,9 gemacht ergab sichtbar, Rettung Ebenen des transgenen Produkts (Abbildung 3A). Western-Blot mit anti-GFP Antikörper bestätigt, dass das Transgen (Allel Namen texIs100) ausgedrückt wird und korrekt (Beifuß und Gumienny, unveröffentlicht) verarbeitet. Integrierte Transgene sollte fünfmal outcrossed werden, um überflüssige Hintergrund-Mutationen unabhängig von der Quelle zu entfernen.
Für unseren Bildschirm, wollten wir das einzige Protein von Interesse, um die transgene, markierte Form zu sein. Daher entfernten wir die funktionalen endogenen gEn durch die Einführung einer Loss-of-function Mutante DBL-1-Allel.
Schließlich haben wir sensibilisiert der Stamm auf die Auswirkungen der RNAi. Die RNAi wird von der Bibliothek, für viele Gene, erzeugen eine stärkere Reduktion der Gen-Produkt, wenn die Tiere eine Mutation, die die Tiere sensibilisiert für die Auswirkungen von RNAi 10,11 enthalten Das Gewebe (e) von Interesse sollten bei der Auswahl eines geeigneten werden RNAi Sensibilisierung Hintergrund 11. Wir nutzten die kanonische RRF-3 (pk1426) Allel zu unserem Stamm-Screening machen. RRF-3 ist eine RNA-gerichtete RNA-Polymerase (RdRP) Homolog, die normalerweise inhibiert somatischen RNAi 10. Mutationen in anderen Genen wie RNAi hypersensitizing eri-1 oder eri-1-lin 15b kann stattdessen verwendet werden, um die Wirksamkeit der RNAi 11-13 erhöhen.
Das Screening-Stamm haben wir den Genotyp RRF-3 (pk1426); texIs100; DBL-1 (NK3).
2. Auswahl und Vorbereitung derRNAi-Bibliothek
Im Handel erhältliche C elegans cDNA-Bibliotheken repräsentieren etwa 55% oder 87% der vorhergesagten Gene in C. elegans einzeln (Vidal Labor oder Ahringer Labor Bibliotheken, jeweils). Einzelne Klone sind käuflich zu erwerben (Open Biosystems, Geneservice, Ltd.) Wir haben uns für die ORF-RNAi-Bibliothek, die von der Labor Vidal (Open Biosystems) gebaut, weil seine Klone sind meist voller Länge cDNAs kloniert und Gateways bereit für nachgelagerte Anwendungen (Invitrogen Corporation, Carlsbad, CA). Das Originalbild Ahringer Labor genomischen cDNA-Bibliothek enthält cDNA-Fragmente, die nicht so weit nützlich stromabwärtigen Charakterisierungsexperimenten 14,15. Beide Bibliotheken verwenden einen Vektor, der zwei T7-Promotoren flankieren den Einsatz (Abbildung 1) enthält. Die Konstrukte werden in bakterielle Stamm HT115, die T7-Polymerase exprimiert nach Induktion durch Isopropyl-β-D-thiogalactopyranosid (IPTG) gezüchtet. Bakterien enthalten, Bibliothek Klone werden mit IPTG induziert, umdsRNA zu produzieren (siehe Schritt 3.4).
Duplizieren Sie die gesamte Bibliothek nach Erhalt und verwenden Sie das Duplikat für alle Experimente. Das Original und Duplikat Bibliotheken sollten in verschiedenen -80 ° C Tiefkühltruhen, die an elektrischen Leitungen zu trennen sind, gespeichert werden.
3. Herstellung von Bakterien mit Bibliothek Klone
- (Innerhalb von zwei Monaten des Experiments) Streak Bakterien aus ausgewählten Fütterung Bibliothek Klone, sowie positiven und negativen Kontrollen, beschriftet auf LB-Carbenicillin / Tetracyclin-Platten (wir wachsen 8 Kulturen pro 100 mm Platte) und Inkubation bei 37 ° C über Nacht ( > 16 Stunden) (Abbildung 1). Für jeden 24-Well-Platte verwenden wir zwei Kontrollen. Die positive Kontrolle, bli-4 (K04F10.4), produziert eine dosisabhängige postembryonalen pheontype 16,17. Die negative Kontrolle, C06C3.5, eine vorhergesagte Pseudogen ( WormBase.org ), die keine beobachteten Phänotypen (Beifuß und Gumienny, unveröffentlicht) bewirkt.
- (Innerhalb von zwei Monaten des Experiments, vorzugsweise früher) Bereiten 24-Loch-Nematoden Wachstumsmedium (NGM)-Platten mit 25 bis 50 mu g / ml Carbenicillin und 1 mM IPTG. Lagerung bei 4 ° C.
- (Tag 1) für jeden Klon, impfen 2 ml LB-Medium, das 50 ug / ml Carbenicillin mit einer einzelnen Kolonie von der Platte gestreift (en) (in Schritt 3.1) unter Verwendung von Standard steriler Technik. Bei 37 ° C über Nacht (> 16 Stunden) in einem Shaker Set, um 220 bis 240 Umdrehungen pro Minute (Abbildung 1). Die Lösung sollte nach 16 Stunden trübe.
- (Tag 2) Am nächsten Morgen, zu veranlassen doppelsträngige RNA (dsRNA)-Produktion durch Hinzufügen von IPTG zu den Kulturen zu einer Endkonzentration von 1 mM. Inkubieren Kulturen bei 37 ° C, 220 - 240 Upm, für weitere 4 - 5 Stunden. Dieser Schritt sorgt für eine konsistente und stabile RNAi-induzierten Knockdown von Gen-Produkt, als allein auf Grundlage der IPTG in den Agar der NGM-Platte (die in Schritt 3.5).
- Spot 30 ul jeder induzierte bakterielle culture in separate Vertiefungen einer 24-Well-Platte mit NGM RNAi von 25 bis 50 mu g / ml Carbenicillin und 1 mM IPTG (Abbildung 1, siehe Abbildung 4 für die Vorlage verwendet, um RNAi-Experimente verfolgen). Setzen Platten, aufgedeckt, in einer sterilen Werkbank für 20 Minuten oder bis die bakteriellen Flecken trocken sind. Nicht übertrocknen. Übertrocknung bewirkt, dass die Kanten der Agar zu ziehen weg von der Platte und Tiere wird unwiederbringlich, wenn sie kriechen in die resultierenden Spalten.
4. Herstellung von Nematoden
Beginnen Sie mit einem inszenierten Bevölkerung von Nematoden. Staging Tiere verringert die Wahrscheinlichkeit, dass die Unterschiede zwischen der Kontrolle und den experimentellen RNAi-Studien beobachtet einfach sind aufgrund von Unterschieden im Entwicklungsstand der Tiere (wie auch immer, wenn der RNAi führt zu einer Entwicklungsverzögerung, sollte dies durch die Kontrollperson festgestellt werden (Abbildung 2)) . Darüber hinaus beginnen die RNAi-Experiment mit L1-Larven vermeidet potenzielle Störfaktoren Auswirkungen der embryonale Letalität durch RNAi von Gendas kann es auch spielen postembryonalen Rollen von Interesse.
Staging wird durch die erste Bleichen eines Mixed-Bühne Bevölkerung der Screening-Stamm, der nur Eierschalen-geschützten Embryonen überleben erreicht. Diese Stufen Tiere innerhalb von 12 Stunden bei 20 ° C oder etwa 18 Stunden bei 16 ° C 18. Um mehr dicht Bühne Tiere, Tiere Verhaftung im ersten Larvenstadium (L1), indem Embryonen schlüpfen in M9-Medium ohne Nahrung. Starved L1 Tieren für Lebensmittel Lebenslauf Wachstum aus dem gleichen Ausgangsmaterial Alter von 19 Jahren gelegt.
- (Tag 1) Benutzen Sie drei 100 mm-Platten der Fed, saubere Tiere, die Screening-Stamm graviden Erwachsenen und Embryonen enthalten. Waschen Sie Tiere aus Platten mit sterilen M9 durch Pipettieren Flüssigkeit über der Plattenoberfläche sanft auf Tiere und Embryonen zu lockern. Übertragen Sie die Wäsche in ein steriles 15 ml Röhrchen mit Schraubverschluss.
- Wenn das Waschergebnis ist größer als 3,5 ml, Spin-down der Tiere (~ 3000 rpm für 30 Sekunden) und nehmen Flüssigkeit auf 3,5 ml. Wenn das Waschergebnis ist lESS als 3,5 ml, H 2 0 oder M9 mit dem Rohr auf 3,5 ml Gesamtvolumen.
- Tragen Sie geeignete Schutzkleidung (Kittel, Handschuhe und Schutzbrille) beim Umgang mit Chemikalien. Mischen Sie 0,5 ml 5 N NaOH mit 1 ml frisches Bleichmittel (5% Natriumhypochlorit, weniger als 1 Jahr alt). Fügen Sie diese Mischung auf die 3,5 ml Nematoden zu waschen. Starten einer Zeit Hochzählen. Dieser Vorgang sollte nicht länger als 10 Minuten. Wenn ja, dann Embryonen wird gefährdet und Ertrag reduziert werden kann, oder das Bleichmittel ist alt und muss ersetzt werden.
- Vortex das Rohr für etwa 10 Sekunden. Wiederholen der Verwirbelungskammer alle zwei Minuten für 4 bis 10 Minuten. Nach jedem Vortexen, beobachten Sie die Lösung unter einem Binokular für Tierkadaver.
- Wenn Embryonen werden freigesetzt und Erwachsene sind fragmentiert (6 - 8 Minuten), Pellet die Embryonen, die durch das Drehen des Rohres in einer Tischzentrifuge (~ 3000 rpm, 30 Sekunden).
- Saugen so viel von der Überstand wie möglich, ohne das Pellet. Seien Sie vorsichtig, nichtgierig.
- In steriler M9 auf ~ 10ml. Vortex kurz oder gut schütteln, um das Pellet zu resuspendieren.
- Drehen Sie den Überstand und wieder. Wenn der Geruch von Bleichmittel in das Rohr erfasst werden kann, wiederholen gründlich wie nötig.
- Resuspendieren Embryonen in 10 bis 15 ml steriler M9 und Transfer zu einem kleinen (25 ml) Erlenmeyerkolben (für Belüftung). Schütteln Sie bei der richtigen Temperatur für den Stamm und Experiment (15 - 25 ° C) über Nacht (> 16 Stunden bei 20 ° C). In dieser Zeit werden die Tiere alle schlüpfen und Arrest in der L1 Diapause.
- 1 ml M9 (~ 3000 min, 30 sek.) - (Tag 2) nach unten L1 Stadium Tiere in einem 15 ml-Tube und resuspendieren in 0,5 spinnen. Platz 5 ul Lösung auf einer separaten Platte und zählen die Anzahl der Tiere. Nehmen Sie die Einstellung der Lautstärke, wenn nötig, zu 30 Würmer / 3 ergeben - 10 l Lösung.
- Spot etwa 30 Hypochlorit synchronisiert L1 Stufe Tiere in der Bakterien in jeder Vertiefung der vorbereiteten 24-Well-Platte. Zu viele more als 30 Tieren pro Vertiefung kann in der Bevölkerung hungern vor dem Erreichen Reife führen. Lassen Sie die Platte trocken mit dem Deckel schief. Setzen Sie den Deckel und befestigen Sie ihn mit einem Gummiband. Die Platte umdrehen und legen Sie sie bei der entsprechenden Temperatur für die Zeit benötigt, um an der gewünschten Phase zu erzielen. Weil RRF-3 (pk1426) ist eine temperaturempfindliche Allel wurden unsere Screening-Belastung bei 15 Tieren angewachsen - 17 ° C für drei bis vier Tagen zu beobachten, L4 zum adulten Stadium Tiere (Abbildung 1).
5. Die Beobachtung der Nematoden
(Tag 5) Sobald die Nematoden auf die gewünschte Stufe (2A) sind gewachsen, bestätigen, dass die positiven und negativen Kontrollen die erwarteten Phänotypen erzeugen mit Hilfe eines Binokular. Wir verwenden bli-4 als Kontrolle für RNAi Wirksamkeit, da es dosisabhängig post-embryonale Defekte erzeugt, die von Blasen Erwachsene (mild RNAi-Phänotyp, nicht gezeigt), um entwicklungspolitisch verhaftet, winzigen Larven (stark, eRWARTETE RNAi-Phänotyp) (Abbildung 2B). Dann beobachten die Tiere in jeder Versuchsgruppe auch mit einem Binokular und beachten Sie, offensichtlich anormalen Phänotypen durch RNAi (Abbildung 2C) induziert. Nachdem die Kontrollen bestätigt werden und grobe Phänotypen festgestellt, Leinwand, die RNAi-Experimente für Phänotypen von Interesse. Wir verwenden ein zusammengesetztes Mikroskop mit Fluoreszenz und einem 63x Objektiv, mindestens fünf Tiere von jeder RNAi Experiment beobachten ausgestattet, beginnend mit Kontrollen (Abbildung 3).
- Bereiten Sie einen Schieber (Standard 75 x 25 mm), indem Sie einen 4% Agar-Pad (4% Agar in H 2 O). Um eine gleichmäßige Dicke von Agar-Pad zu gewährleisten, legen Sie einen sauberen Objektträger aus Glas zwischen zwei Abstandshaltern (jeweils Abstandshalter ist aus einem Objektträger mit zwei Schichten von Labor auf denen sich bereits gemacht). Pipette etwa 100 ul (zwei bis drei Tropfen aus Glas Pasteur-Pipette) aus geschmolzenem 4% Agar auf der Mitte des sauberen Objektträger. Bilden eine Agar-Pad durch Abdecken des geschmolzenen Agar Verwendung eines zusätzlichen Dia schnell, aber vorsichtig oben auf den Abstandshaltern einem platziertennd der geschmolzenen Agar. Setzen Sie das Agar-Pad durch leichtes Verschieben der Glas-Objektträger auseinander, so dass das Pad Einhaltung einer Folie.
- Setzen Sie ein ~ 5 ul Tropfen Anästhetikum (Levamisol oder Natriumazid, zum Beispiel) auf dem Agar-Pad. Berg 8 bis 12 Tiere in der Narkose. Cover mit einem 22 x 22 mm # 1.5 Deckglas (oder die Dicke des Deckglases am besten für das zusammengesetzte Mikroskop geeignet) und beobachten Tiere mit einem zusammengesetzten Mikroskop.
- Rekord-Gen, Anzahl der Tiere beobachtet und Phänotyp Ergebnisse für jede RNAi-Experiment. Für einen Bildschirm, und verwenden einen Standard-Template (siehe zB Abbildung 5).
- Überprüfen der RNAi-Phänotyp hergestellt durch ein anderes, sekundären Phänotyp, wenn möglich, und indem das Experiment. (Zum Beispiel, das gezeigte Beispiel Bildschirm Veränderung der GFP-markierten DBL-1 Körperregion, wie einem primären Screen und Körpergröße als zweites Display 20 verwendet.) Bakterien können aus dem gleichen Streifen (bei 4 ° C) bis zu verwendenden zwei Monate. Ältere Streifen können reiter in schlechtem Wachstum in flüssigen Übernacht-Kulturen und sollte zuerst ausgestrichen werden.
- Sequenz an zumindest einem Ende des cDNA-Inserts zu bestätigen, dass der Einsatz der Bibliothek Bezeichnung (Ahringer Bibliothek hat eine Zuverlässigkeit Analyse durchgeführt, siehe entspricht http://biocompute.bmi.ac.cn/CelRNAi/ ) 21. 5'-AGCGAGTCAGTGAGCGAG-3 'und 5'-GTAAAACGACGGCCAGT-3' (M13f20 Primer): Empfohlene Grundierungen (für Einsätze von beiden Bibliothek) auf Websites im L4440-abgeleiteten Vektor und Flanke der Insertionsstelle zu glühen.
Mit dieser Methode kann eine einzelne Person vernünftigerweise zu inszenieren und zu beobachten zwei vor vier Sätze von Experimenten jede Woche. So inszeniert Tiere am Montag und Dienstag zu beobachten Donnerstag und Freitag werden jeweils wieder und kann am Freitag und Samstag ausgetragen für Montag und Dienstag Beobachtung. Abhängig von der Phänotyp (en) gescreent jeden Tag kann ein Satz einer 24-Well-Platte umfassen, durch die Verbindung Mikroskopie oder mehr, wenn der Phänotyp schnell identifiziert wird. Somit kann mindestens 88 verschiedene RNAi-Experimente in einer Woche beobachtet werden, unter Berücksichtigung der positiven und negativen Kontrollen und von Screening. Ein Binokular Bildschirm könnte viel schneller durchgeführt werden, ohne die Notwendigkeit für die Montage Tiere Folien für die Anzeige. Mehrere Personen können auch ein Labor erhöhen den Durchsatz durch die Staffelung der Termine und / oder die Verwendung mehrerer Mikroskope. Ein alternatives Verfahren zum Züchten von Tieren in einem dünnen Film aus Agar und Übertragen ein Stück von dem Agar mit Tieren direkt zu einer Folie für die Anzeige kann Zeit sparen. Diese Variante wurde erfolgreich für das Screening von männlichen Schwanz Anomalien bei 400-facher Vergrößerung 22 verwendet.
6. Repräsentative Ergebnisse
Beispiele für normale und Lokalisierung von GFP-markierten DBL-1 sind in 3 gezeigt. Normale Expression von GFP-markierten DBL-1 umfasst Bauchmark Zellkörper und eine Reihe von Tränenpünktchen (Abbildung 3A). DBL-1 is stark abgeschwächt, wenn die Tiere gefüttert werden, die RNA verhindert DBL-1 mRNA-Translation (Abbildung 3B). dbl-1 (RNAi) produziert auch kleine Tiere, die ein zweites Display in diesem Beispiel (Daten nicht gezeigt). Gene, die DBL-1 Lokalisation beeinflussen werden leicht durch RNAi unter Verwendung eines Stammes für diesen Bildschirm (Abbildung 3C) entworfen identifiziert.

Abbildung 1. Screen-Schema zu identifizieren extrazellulären Regulatoren der DBL-1-Signalisierung. Bakterien der Fütterung Bibliothek über Nacht gezüchtet mit IPTG induziert, und bei 37 ° C für weitere 4 Stunden, damit die Bakterien doppelsträngige RNA (dsRNA) zu erzeugen. 30 ul der induzierten Bakterienkultur pro Vertiefung auf eine 24-Well-NGM (Nematoden Wachstumsmedium), enthaltend 25 ug / ml Carbenicillin und 1 mM IPTG entdeckt und man ließ sie in einer sterilen Werkbank trocknen. Wir inszenieren erste Larvenstadium (L1) Larven, indem Hypochlorit-behandelten EMBRYos schlüpfen in Medien ohne Nahrung, die eine Diapause L1 (Arrested Development) induziert. Etwa 30 synchronisiert L1 Stadium Tiere werden auf jeder Vertiefung, welche Bakterien aus der RNAi-Bibliothek, die Tiere inszeniert, um das Wachstum wieder aufnehmen und die dsRNA durch die Bakterien verbrauchen 23 erstellt erlaubt überzogen. Nach 72 h bei 15 ° C, die jungen erwachsenen Würmer sind für eine sichtbare Phänotyp gescreent werden. In diesem Alter sind die Körpergröße Mängel offensichtlich und Fluoreszenz ist hell aus texIs100 Transgenexpression.
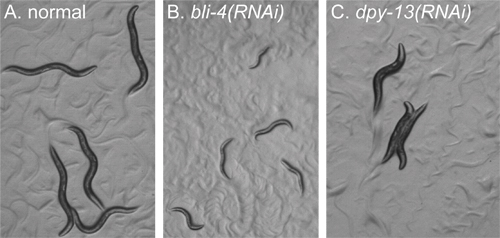
Abbildung 2. Beispiele für Phänotypen vor Screening zu identifizieren. Alle Bilder von Tieren in einem Teller gut wurden mit der gleichen Vergrößerung unter Verwendung eines Präpariermikroskops genommen. Die Tiere wurden alle etwa 72 Stunden nach dem Ausplattieren wie ausgehungerte L1-Larven abgebildet. Alle Bilder wurden identisch behandelt. A) RNAi eines Gens, das keine groben morphologischen Fehler gibt. Tiere erscheinen Wildtyp. B) RNAi von bli-4. Tiere zeigen verhaftet Entwicklung, und sind winzig im Vergleich zu den Tieren in Panel A. C) RNAi von dpy-13. Die Tiere sind auf der gleichen Stufe der Entwicklung, wie Tiere in Feld A, sondern zeigen eine "pummelig" Körper Morphologie.
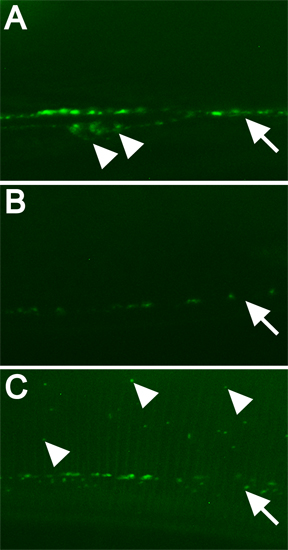
Abbildung 3. Beispiele von fluoreszenzmarkierten Protein und wie RNAi-spezifischer Gene verändert Lokalisierung Muster. Alle Bilder wurden bei 630x Vergrößerung mit Spinning-Disk-konfokale Mikroskopie, 5 Sek. genommen. Exposition. Balken = 10 um. Alle Bilder wurden identisch behandelt. Offene Pfeilspitzen zeigen Zellkörper. Pfeile kennzeichnen Linie der Tränenpünktchen. Gefüllt Pfeilspitzen zeigen einige aberrant lokalisierte GFP-markierten DBL-1. A) RNAi-fed Pseudogen C06C3.5 ("Wildtyp"). B) DBL-1 (RNAi)-Steuerung. C) RNAi eines Gens für normale Lokalisierung von GFP-markierten DBL-1 erforderlich.
Abbildung 4. Vorlage beispielsweise zum Verfolgen RNAi-Experimente (Bibliothek Klone) in 24-Well-Platten. Diese Vorlage kann verwendet werden, um eine permanente Aufzeichnung der Versuche, im Gegensatz zu direkten Markieren Platten zu erstellen.
Abbildung 5. Vorlage Beispiel für die Aufzeichnung RNAi-Phänotypen. Erweitern Sie bei Bedarf.
Diskussion
Die RNAi-Screening hier vorgestellte Methode ermöglicht eine schnelle und sensitive Analyse von Gen-Produkten für einen normalen (oder transgene) postembryonalen Phänotyp erforderlich. Das gezeigte Beispiel ist ein Bildschirm für die Gene in der subzellulären Lokalisation eines fluoreszenzmarkierten Proteins beteiligt. Allerdings ist dieses Protokoll modifiziert werden, um Gene, die anderen postembryonalen Phänotypen von Interesse zu identifizieren.
Diese Methode nutzt eine Kandidateng...
Offenlegungen
Wir haben nichts zu offenbaren.
Danksagungen
Die Autoren danken Herrn Dr. Rick Padgett (Waksman Institut, Rutgers University, NJ) für das Geschenk des DBL-1 cDNA und Dr. Christopher Rongo (Waksman Institut, Rutgers University, NJ) für eine Injektion Marker danken. Dr. Barth Grants Labor durchgeführt, die das Gen für die Bombardierung Pistole mit geringer Kopienzahl Integration des GFP-markierten DBL-1-Konstrukt. Die René Garcia Labor leistete technische Hilfe bei der Erstellung der texIs100. Die René Garcia, Robyn Lints und Hongmin Qin Laboratorien zur Verfügung gestellt produktive Beratung. Diese Arbeit wurde vom Start-up-Mitteln aus dem TAMHSC Department of Molecular and Cellular Medicine finanziert. Die Verbindung Umfang und Spinning-Disk-konfokale wurden mit Mitteln der Abteilung und der TAMHSC College of Medicine Dekanat zur Verfügung gestellt gekauft.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare |
| NGM-Agar | Nematoden Wachstumsmedium | IPM Scientific, Inc | Kann nach der NGM-Agar-Protokoll 25 vorbereitet werden |
| M9 Medium | 22mm KH 2 PO 4, 42mm Na 2 HPO 4, 86mm NaCl, 1 mM MgSO 4 | 26 | |
| Agar-Agar | EMD Chemicals Inc. | 1.01614.1000 | 2% in Wasser für NGM Platten. 4% in Wasser für Objektträger-Pads (Autoklav zunächst Mikrowelle und danach zu schmelzen). |
| Bacto Peptone | Becton Dickinson - Difco CP | 211677 | 0,25% |
| IPTG | Research Products International Corp | I56000-5.0 | 1 mM Endkonzentration |
| Carbenicillin | Research Products International Corp | C46000-5.0 | 50 ug / ml Gebrauchsverdünnung |
| LB-Nährlösung Lennox | Becton Dickinson - Difco CP | 240230 | 20 g / Liter |
| Tetracyclin | Sigma | 268054 | 12,5 pg / ml Gebrauchsverdünnung |
| Natriumhypochlorit | Jede Marke | 5% Haushaltsbleiche | Verwenden Sie frische Bleiche. |
| Natriumhydroxid | Beliebige Marke | CAS 1310-73-2 | 5 N Lager |
| M9-Medium | Wormlab Rezept-Buch | http://130.15.90.245/wormlab_recipe_book.htm # Commonlab | 26 |
| Levamisol | Sigma | 31742 | 100 pM - 1 mM Gebrauchsverdünnung |
| Natriumazid | Fisher Scientific | S227 | 10 mM in M9 Gebrauchsverdünnung |
| 24-Well-Platte | Greiner Bio-One | 662160 | VWR Distributor |
| Objektträger | Jede Marke | 75 x 25 x 1 mm | |
| Mikroskop Deckgläser | Jede Marke | 22 x 22 mm No.1.5 | Verwenden Sie die Stärke durch das Mikroskop Hersteller empfohlen wird. |
| zusammengesetztes Mikroskop | Carl Zeiss, Inc. | A1m | Verwendung Ziele und Filter können die Bedürfnisse des Experiments lassen. |
| Medien Pumpe | Manostat Varistaltic Pumpe | Kate Modell # 72-620-000 | Verwenden Sie Schläuche und die passenden Einstellungen für die Maschine |
Referenzen
- Giacomotto, J., Segalat, L. High-throughput screening and small animal models, where are we. Br. J. Pharmacol. 160, 204-216 (2010).
- Lamitina, T. Functional genomic approaches in C. elegans. Methods in Molecular Biology. 351, 127-138 (2006).
- Boutros, M., Ahringer, J. The art and design of genetic screens: RNA interference. Nat. Rev. Genet. 9, 554-566 (2008).
- Praitis, V., Casey, E., Collar, D., Austin, J. Creation of low-copy integrated transgenic lines in Caenorhabditis elegans. Genetics. 157, 1217-1226 (2001).
- Yandell, M. D., Edgar, L. G., Wood, W. B. Trimethylpsoralen induces small deletion mutations in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 91, 1381-1385 (1994).
- Frokjaer-Jensen, C. Single-copy insertion of transgenes in Caenorhabditis elegans. Nature Genetics. 40, 1375-1383 (2008).
- Giordano-Santini, R., Dupuy, D. Selectable genetic markers for nematode transgenesis. Cell. Mol. Life. Sci. , (2011).
- Berkowitz, L. A., Knight, A. L., Caldwell, G. A., Caldwell, K. A. Generation of Stable Transgenic C. elegans Using Microinjection. J. Vis. Exp. (18), e833 (2008).
- Mello, C., Fire, A. DNA transformation. Methods in Cell Biology. 48, 451-482 (1995).
- Simmer, F. Loss of the putative RNA-directed RNA polymerase RRF-3 makes C. elegans hypersensitive to RNAi. Curr. Biol. 12, 1317-1319 (2002).
- Zhuang, J. J., Hunter, C. P. Tissue-specificity of Caenorhabditis elegans enhanced RNAi mutants. Genetics. , (2011).
- Samuelson, A. V., Klimczak, R. R., Thompson, D. B., Carr, C. E., Ruvkun, G. Identification of Caenorhabditis elegans genes regulating longevity using enhanced RNAi-sensitive strains. Cold Spring Harbor Symposia on Quantitative Biology. 72, 489-497 (2007).
- Wang, D. Somatic misexpression of germline P granules and enhanced RNA interference in retinoblastoma pathway mutants. Nature. 436, 593-597 (2005).
- Fraser, A. G. Functional genomic analysis of C. elegans chromosome I by systematic RNA interference. Nature. 408, 325-330 (2000).
- Kamath, R. S. Systematic functional analysis of the Caenorhabditis elegans genome using RNAi. Nature. 421, 231-237 (2003).
- Peters, K., McDowall, J., Rose, A. M. Mutations in the bli-4 (I) locus of Caenorhabditis elegans disrupt both adult cuticle and early larval development. Genetics. , 129-195 (1991).
- Thacker, C., Peters, K., Srayko, M., Rose, A. M. The bli-4 locus of Caenorhabditis elegans encodes structurally distinct kex2/subtilisin-like endoproteases essential for early development and adult morphology. Genes & Development. 9, 956-971 (1995).
- Byerly, L., Cassada, R. C., Russell, R. L. The life cycle of the nematode Caenorhabditis elegans. I. Wild-type growth and reproduction. Dev. Biol. 51, 23-33 (1976).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Savage-Dunn, C. Genetic screen for small body size mutants in C. elegans reveals many TGFbeta pathway components. Genesis. 35, 239-247 (2003).
- Qu, W. Reliability analysis of the Ahringer Caenorhabditis elegans RNAi feeding library: a guide for genome-wide screens. BMC Genomics. 12, 1471-2164 (2011).
- Nelson, M. D. A Bow-Tie Genetic Architecture for Morphogenesis Suggested by a Genome-Wide RNAi Screen in Caenorhabditis elegans. PLoS Genetics. 7, e1002010 (2011).
- Timmons, L., Fire, A. Specific interference by ingested dsRNA. Nature. 395, 854 (1998).
- Sarin, S., Prabhu, S., O'Meara, M. M., Pe'er, I., Hobert, O. Caenorhabditis elegans mutant allele identification by whole-genome sequencing. Nature Methods. 5, 865-867 (2008).
- Lewis, J. A., Fleming, J. T. Basic culture methods. Methods in Cell Biology. 48, 3-29 (1995).
- Brenner, S. The genetics of Caenorhabditis elegans. Genetics. 77, 71-94 (1974).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten