Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Dépistage ARNi pour identifier les phénotypes postembryonnaires dans C. elegans
Dans cet article
Résumé
Nous décrivons une méthode pour identifier des régulateurs sensibilisés postembryonnaires de l'expression des protéines et la localisation dans C. elegans L'aide d'un écran de génomique ARNi-sol et un transgène intégré qui exprime une fonctionnelle, la protéine fluorescente étiqueté.
Résumé
C. elegans s'est avéré être un système de modèle précieux pour la découverte et la caractérisation fonctionnelle des gènes de nombreuses voies et 1 gène. Des outils plus sophistiqués et les ressources pour les études dans ce système sont faciliter la découverte continue de gènes présentant des phénotypes plus subtils ou des rôles.
Ici, nous présentons un protocole généralisé, nous avons adapté pour identifier C. gènes elegans avec des phénotypes postembryonnaires d'intérêt en utilisant le phénomène 2. Cette procédure peut être facilement modifié pour doser le phénotype de choix, que ce soit par l'optique de lumière ou de la fluorescence sur un microscope à dissection ou composé. Ce protocole de dépistage capitalise sur les actifs physiques de l'organisme et des outils moléculaires de la C. elegans communauté de la recherche a produit. A titre d'exemple, nous démontrons l'utilisation d'un transgène intégré qui exprime un produit fluorescent dans un écran ARNi pour identifier les gènes nécessaires à la localisation normale de cetteproduit chez les larves et les adultes stade tardif. Tout d'abord, nous avons utilisé un génomique disponible dans le commerce bibliothèque ARNi avec pleine longueur inserts d'ADNc. Cette bibliothèque facilite l'identification rapide de plusieurs candidats par réduction ARNi du produit du gène candidat. Deuxièmement, nous avons généré un transgène intégré qui exprime notre protéine fluorecently marqué de l'intérêt dans un contexte sensible ARNi. Troisièmement, à exposer les animaux éclos à l'ARNi, cet écran permet l'identification de produits géniques qui ont un rôle vital embryonnaires qui seraient autrement masquer un rôle post-embryonnaire dans la régulation de la protéine d'intérêt. Enfin, cet écran utilise un microscope composé doté pour la résolution de cellule unique.
Protocole
1. Construction de la souche de dépistage
La conception soignée de la souche de dépistage est essentiel pour la réussite de l'écran et a été décrite ailleurs 3. Pour certains chercheurs, en utilisant une souche qui exprime un produit visible à partir d'un transgène est nécessaire pour l'expérience. De nombreuses souches hébergeant des transgènes intégrés sont disponibles auprès des chercheurs de la CCG ou une personne. Si une souche transgénique est nécessaire pour l'écran, mais n'est pas disponible, alors il peut être généré à l'aide d'une méthode publiée comme bombardements 4, UV / TMP 5, ou insertion d'un transposon Mos 6. Afin de visualiser notre protéine d'intérêt, nous avons inséré la séquence codante gfp dans le cadre avec la séquence mature ADNc (nous avons utilisé DBL-1 séquence). Parce que cette protéine de fusion GFP n'est pas visible en disséquant la portée, nous avons utilisé un marqueur co-injection qui était visible et n'a pas d'incidence sur la visualisation de notre protéine d'intérêt (ttx-3p :: DP , pour un examen de plusieurs autres marqueurs classiques, voir 7). Nous avons ensuite créé un transgène intégré à partir du tableau extrachromosomique. Nous avons constaté que le bombardement du transgène a donné un faible nombre de copies le numéro de ligne intégré que ni sauvé le phénotype microscope à dissection, ni produit des niveaux visibles de produit GFP-tagged (Beifuss et Gumienny, non publié). UV / TMP intégration d'un réseau de plusieurs copies extrachromosomique à l'origine par injection 8,9 cédé visible, les niveaux de sauvetage produit du transgène (figure 3A). Western blot avec anticorps anti-GFP a confirmé que le transgène (allèle texIs100 nom) est exprimé et traité correctement (Beifuss et Gumienny, non publié). Transgènes intégrés devrait être outcrossed cinq fois pour éliminer les mutations de fond étrangers indépendamment de la source.
Pour notre écran, nous voulions que la seule protéine d'intérêt à l'transgénique, sous forme étiqueté. Par conséquent, nous avons enlevé le g fonctionnelle endogèneène en introduisant une perte de fonction mutante DBL-1 allèle.
Enfin, nous sensibilisé la souche aux effets de l'ARNi. Le RNAi de la bibliothèque, pour de nombreux gènes, de produire une réduction plus sévère dans le produit du gène si les animaux contiennent une mutation qui sensibilise les animaux aux effets de l'ARNi 10,11 Le tissu (s) d'intérêt doit être envisagée lorsque le choix d'un appropriée ARNi sensibilisation de fond 11. Nous avons utilisé l'allèle canonique far-3 (pk1426) pour faire de notre souche de dépistage. RRF-3 est un ARN-polymérase dirigée vers l'ARN (PRDR) homologue qui inhibe normalement somatiques ARNi 10. Des mutations dans les gènes ARNi autres hypersensitizing comme eri-1 ou eri-1 lin-15b peut être utilisé à la place pour accroître l'efficacité de l'ARNi 11-13.
La souche de dépistage nous avons faite a le génotype far-3 (pk1426); texIs100; dbl-1 (NK3).
2. Choix et préparation duARNi bibliothèque
Disponible dans le commerce C. bibliothèques d'ADNc elegans représentent environ 55% ou 87% des gènes prédits dans C. elegans individuellement (Vidal laboratoire ou Ahringer bibliothèques de laboratoire, respectivement). Des clones individuels sont disponibles à l'achat (Open Biosystems, Geneservice, Ltd). Nous avons choisi la bibliothèque ORF-ARNi construit par le laboratoire de Vidal (Open Biosystems) parce que ses clones sont la plupart du temps sur toute la longueur de passerelle ADNc cloné et prêt pour les applications en aval (Invitrogen Corporation, Carlsbad, CA). La bibliothèque d'origine Ahringer laboratoire génomique ADNc contient des fragments d'ADNc qui ne sont pas aussi largement utile pour les expériences de caractérisation aval 14,15. Les deux bibliothèques utilisent un vecteur qui contient deux promoteurs T7 flanquant l'insert (figure 1). Les constructions sont cultivées dans la souche bactérienne HT115, qui exprime la T7 polymerase lors de l'induction par l'isopropyl-β-D-thiogalactopyranoside (IPTG). Les bactéries contenant des clones de la banque sont induites à l'IPTG àproduire des ARNdb (voir l'étape 3.4).
Dupliquer toute la bibliothèque lors de la réception et l'utilisation du double pour toutes les expériences. Les bibliothèques d'origine et en double doit être stockée dans différentes -80 ° C congélateurs qui sont connectés à séparer les lignes électriques.
3. Préparation des bactéries avec des clones de bibliothèque
- (Dans les deux mois de l'expérience), les bactéries Streak de certains clones de la banque d'alimentation, ainsi que des contrôles positifs et négatifs, sur étiquetés LB-carbénicilline / tétracycline plaques (nous grandissons 8 cultures pour 100 plaque de mm) et incuber à 37 ° C pendant la nuit ( > 16 heures) (Figure 1). Pour chaque plaque de 24 puits, nous utilisons deux contrôles. Le contrôle positif, bli-4 (K04F10.4), produit une dose-dépendante post-embryonnaire pheontype 16,17. Le contrôle négatif, C06C3.5, est un pseudogène prédit ( WormBase.org ) qui ne provoque aucun phénotypes observés (Beifuss et Gumienny, non publié).
- (Dans les deux mois de l'expérience, de préférence plus tôt) Préparer 24 ainsi nématodes milieu de croissance (NGM) des plaques contenant 25 à 50 pg / ml et 1 mM IPTG carbénicilline. Conserver à 4 ° C.
- (Jour 1) Pour chaque clone, inoculer 2 ml contenant du milieu LB 50 pg / ml de carbénicilline avec une seule colonie de la plaque striée (s) (à l'étape 3.1) en utilisant la norme technique stérile. Incuber à 37 ° C pendant la nuit (> 16 heures) dans un ensemble agitateur à 220 - 240 rpm (Figure 1). La solution devrait être nuageux après 16 heures.
- (Jour 2) Le lendemain matin, induire l'ARN double brin (ARNdb) la production par l'ajout d'IPTG à la culture à une concentration finale de 1 mM. Incuber les cultures à 37 ° C, 220 - 240 rpm, pour un 4 supplémentaires - 5 heures. Cette étape garantit une approche plus cohérente et robuste ARNi induite par effet de choc du produit du gène de compter uniquement sur l'IPTG dans la gélose de la plaque de NGM (à l'étape 3.5).
- Spot de 30 ul de chaque cu induits bactérienneslture dans des puits séparés d'une plaque de 24 puits NGM ARNi contenant 25 - 50 pg / ml et 1 mM IPTG carbénicilline (Figure 1, voir la figure 4 pour le modèle utilisé pour suivre les expériences de RNAi). Mettre les plaques, à découvert, dans une hotte à flux stérile pendant 20 minutes ou jusqu'à ce que les taches bactériennes sont à sec. Ne pas trop sécher. Surséchage provoque les bords de la gélose à se détacher de la plaque et les animaux sera irrémédiable si elles pénètrent dans les fissures qui en résultent.
4. Préparation de nématodes
Commencez avec une population mise en scène de nématodes. Mise en scène des animaux diminue la chance que les différences observées entre la commande et les essais expérimentaux ARNi sont tout simplement dues à des différences dans le stade de développement des animaux (toutefois, si le RNAi entraîne un retard de développement, ceci doit être noté par l'examinateur (Figure 2)) . En outre, à partir de l'expérience ARNi avec des larves L1 évite les éventuels effets confondants de létalité embryonnaire par ARNi des genes qui peuvent également jouer un rôle postembryonnaires d'intérêt.
Mise en attente est réalisée en commençant par une population mixte de blanchiment-étape de la souche de dépistage, qui ne coquille-protégées embryons survivent. Cette stades animaux à l'intérieur d'environ 12 heures à 20 ° C ou environ 18 heures à 16 ° C 18. Pour plus étroitement les animaux de scène, des animaux d'arrêt dans le premier stade larvaire (L1) en laissant trappe embryons dans un milieu M9 sans nourriture. Animaux affamés L1 mis sur la croissance CV nourriture de la même âge à partir du 19.
- (Jour 1) Utilisez trois plaques de 100 mm de nourris, propres animaux de contrainte de dépistage qui contiennent les adultes gravides et des embryons. Laver les animaux à partir de plaques en utilisant stérile M9 par pipetage liquide à travers la surface de la plaque en douceur pour desserrer les animaux et les embryons. Transférer le lavage dans une tube de 15 ml stérile avec bouchon à vis.
- Si le lavage est supérieure à 3,5 ml, ralentit les animaux (~ 3000 rpm pendant 30 secondes) et enlever le liquide à 3,5 ml. Si le lavage est lesse à 3,5 ml, ajouter H 2 0 ou M9 au tube à totaliser 3,5 ml.
- Porter un équipement de protection (blouse de laboratoire, des gants et des lunettes) lors de la manipulation des produits chimiques. Mélanger 0,5 ml NaOH 5 N avec 1 ml de javellisant douce (hypochlorite de sodium à 5%, moins de 1 an). Ajouter ce mélange au lavage 3,5 ml de nématodes. Démarrer un compte à rebours vers le haut. Ce processus ne devrait pas prendre plus de 10 minutes. Si c'est le cas, les embryons seront en danger et le rendement peut être réduit, ou l'eau de Javel est vieux et doit être remplacé.
- Vortexer le tube pendant environ 10 secondes. Répétez l'vortex toutes les deux minutes pour 4 à 10 minutes. Après chaque vortex, d'observer la solution sous un microscope à dissection pour les carcasses d'animaux.
- Lorsque les embryons sont libérés et adultes sont fragmentés (6 - 8 minutes), granulés les embryons en faisant tourner le tube dans une centrifugeuse de table (~ 3000 rpm, 30 secondes).
- Aspirer le plus de surnageant possible sans perturber le culot. Soyez prudent, ne pasgourmand.
- Ajouter stérile M9 ~ 10ml. Brièvement Vortex ou bien agiter pour remettre en suspension le culot.
- Spin et retirer le surnageant à nouveau. Si l'odeur d'eau de Javel peut être détectée dans le tube, répéter le rinçage si nécessaire.
- Embryons Remettre en suspension dans 10 à 15 ml stérile M9 et le transfert à un petit (25 ml) Erlenmeyer (pour l'aération). Agiter à la température appropriée pour la souche et de l'expérience (15 - 25 ° C) pendant une nuit (plus de 16 heures à 20 ° C). En ce moment, les animaux seront tous éclore et à l'arrestation dans la diapause L1.
- (Jour 2) Isoler les animaux L1 scène dans un tube de 15 ml et remettre en suspension dans 0,5 à 1 ml M9 (~ 3000 rpm, 30 sec.). Placez une solution à 5 pi sur une plaque séparée et compter le nombre d'animaux. Procéder à des ajustements du volume, si nécessaire, pour donner 30 vers / 3 - une solution à 10 pi.
- Spot environ 30 hypochlorite synchronisés animaux stade L1 sur les bactéries présentes dans chaque puits de la préparation de 24 puits de plaque. Un trop grand nombre more de 30 animaux par puits peut entraîner la population affamée avant d'atteindre la maturité. Laissez la plaque sèche à la guingois couvercles. Replacez le couvercle et le fixer avec une bande de caoutchouc. Retourner la plaque et placez-le à la température appropriée pour le temps nécessaire pour marquer au stade souhaité. Parce que far-3 (pk1426) est un allèle sensible à la température, nos animaux de contrainte de dépistage ont été cultivées à 15 à 17 ° C pendant trois à quatre jours pour observer les animaux L4 stade adulte (figure 1).
5. Observation des nématodes
(Jour 5) Une fois que les nématodes ont grandi à l'étape désirée (figure 2A), confirment que les contrôles positifs et négatifs de produire les phénotypes attendus en utilisant un microscope à dissection. Nous utilisons bli-4 comme un contrôle de l'efficacité de RNAi, parce qu'elle produit dose-dépendante de post-embryonnaires défauts qui vont de la légère (adultes ampoules phénotype ARNi, non représenté) pour développemental arrêtés, minuscules larves (forte, eESCOMPTES ARNi phénotype) (figure 2B). Ensuite, observer les animaux dans chaque puits expérimental en utilisant un microscope à dissection et notes évidentes phénotypes anormaux induits par l'ARNi (figure 2C). Après les contrôles sont confirmés et les phénotypes bruts noté, filtrer les expériences de RNAi pour les phénotypes d'intérêt. Nous utilisons un microscope composé équipé de fluorescence et un objectif 63x pour observer au moins cinq animaux de chaque expérience ARNi, à commencer par les contrôles (Figure 3).
- Préparer une diapositive (standard 75 x 25 mm) en faisant un tampon agar 4% (4% agar dans H 2 O). Pour assurer une épaisseur uniforme de coussin agar, placer une lame de verre propre entre deux entretoises (pièce d'écartement est fait d'une lame de microscope avec deux couches de ruban de laboratoire sur l'). Introduire à la pipette environ 100 pi (deux à trois gouttes d'une pipette Pasteur en verre) de 4% agar fondu sur le centre de la lame de verre propre. Former un pad agar agar en couvrant la fusion en utilisant un chariot supplémentaire placé rapidement mais doucement sur le dessus des entretoises unee l'agar fondu. Exposer le pad agar en glissant doucement les lames de verre en dehors, laissant le pavé adhérant à une diapositive.
- Placez une goutte ~ 5 ul d'anesthésique (lévamisole ou l'azoture de sodium, par exemple) sur le pavé agar. Mont 8 - 12 animaux dans l'anesthésie. Couvrir avec un 22 x 22 mm # 1.5 lamelle (ou l'épaisseur de la lamelle la plus adaptée au microscope composé) et observer les animaux en utilisant un microscope composé.
- Les résultats de gènes d'enregistrement, nombre d'animaux observés, et le phénotype de chaque expérience ARNi. Pour un écran, fabriquer et utiliser un modèle standard (voir par exemple, la figure 5).
- Vérifiez le phénotype de RNAi-produit par un autre phénotype, secondaire, si possible, et en répétant l'expérience. (Par exemple, l'écran exemple illustré utilisé altération de la GFP-tagged DBL-1 la localisation comme un écran primaire et la taille du corps comme un écran secondaire 20.) Les bactéries peuvent être utilisés à partir de la strie même (conservé à 4 ° C) pour un maximum de deux mois. Les seniors stries peut result de la croissance pauvre en liquides cultures de la nuit et devrait être restreaked premier.
- Séquence d'au moins une extrémité de l'insert d'ADNc pour confirmer que l'insert correspond à l'étiquette de bibliothèque (la bibliothèque Ahringer a eu une analyse de fiabilité effectués, voir http://biocompute.bmi.ac.cn/CelRNAi/ ) 21. Primaires recommandés (pour les insertions de la bibliothèque soit) recuit sur les sites du L4440-vecteur dérivé et le flanc du site d'insertion: 5'-AGCGAGTCAGTGAGCGAG-3 'et 5'-GTAAAACGACGGCCAGT-3' (amorce M13f20).
En utilisant cette méthode, une seule personne peut raisonnablement mettre en scène et d'observer deux à quatre séries d'expériences chaque semaine. Par exemple, les animaux mis en scène le lundi et le mardi on peut observer jeudi et vendredi, respectivement, et peut de nouveau être mis en scène le vendredi et le samedi pour le lundi et le mardi observation. Selon le phénotype (s) projeté chaque jour, un ensemble peut comprendre une plaque de 24 puits par le composé microscopie ou plus si le phénotype est rapidement identifié. Ainsi, au moins 88 différentes expériences de RNAi peut être observée dans une semaine, tenant compte des contrôles positifs et négatifs et le taux de dépistage. Un écran champ de dissection pourrait être réalisée beaucoup plus rapidement, sans qu'il soit nécessaire pour le montage de diapositives sur les animaux pour le visionnement. Plusieurs personnes pourraient aussi augmenter le débit d'un laboratoire par les horaires stupéfiantes et / ou utilisant les microscopes multiples. Une méthode alternative pour animaux en croissance sur un film mince d'agar et le transfert d'une tranche de la gélose contenant les animaux directement à une diapositive pour la visualisation peut gagner du temps. Cette variation a été utilisé avec succès pour le dépistage des anomalies queue du mâle à 400x de grossissement 22.
6. Les résultats représentatifs
Exemples de localisation normale et altérée de la GFP-tagged DBL-1 sont présentés dans la figure 3. L'expression normale de la GFP-tagged DBL-1 comprend ventrales des nerfs de la moelle des organismes cellulaires et une rangée de punctae (figure 3A). DBL-1 is fortement atténué lorsque les animaux sont nourris d'ARN qui empêche la traduction DBL-1 ARNm (figure 3B). dbl-1 (ARNi) produit également de petits animaux, l'écran secondaire dans cet exemple (données non présentées). Les gènes qui affectent la localisation DBL-1 sont facilement identifiés par l'ARNi en utilisant une souche conçu pour cet écran (figure 3C).

Figure 1. Régime d'écran pour identifier des régulateurs extracellulaires de DBL-1 de signalisation. Les bactéries de la bibliothèque d'alimentation sont cultivés pendant une nuit, induite par l'IPTG, et incubées à 37 ° C pendant 4 heures supplémentaires pour permettre aux bactéries pour produire des ARN double brin (ARNdb). 30 pl de la culture bactérienne induite est repéré par puits sur un 24-ainsi NGM (milieu de croissance des nématodes) plaque contenant 25 pg / ml et 1 mM IPTG carbénicilline et laissé à sécher sous une hotte à flux stérile. Nous première étape stade larvaire (L1) en laissant les larves d'hypochlorite de traité EMBRyo éclosent dans les médias sans nourriture, ce qui induit une diapause L1 (développement arrêté). Environ 30 animaux synchronisés stade L1 sont étalées sur chaque bactérie et contenant de la bibliothèque ARNi, qui permet en scène des animaux pour relancer la croissance et de consommer l'ARNdb créé par les 23 bactéries. Après 72 heures à 15 ° C, les vers adultes jeunes sont sélectionnés pour un phénotype visible. À cet âge, les défauts de la taille du corps sont évidents et la fluorescence est clair à partir texIs100 expression du transgène.
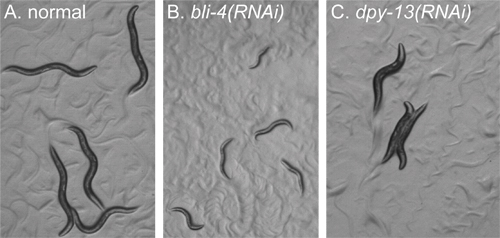
Figure 2. Exemples de phénotypes pour identifier avant le dépistage. Toutes les images d'animaux dans une plaque de puits ont été prises au même grossissement en utilisant un microscope à dissection. Les animaux ont été tous les imagé environ 72 heures après le placage sous forme de larves L1 affamé. Toutes les images ont été traitées de manière identique. A) ARNi d'un gène qui ne donne pas de défauts morphologiques brutes. Animaux apparaissent de type sauvage. B) RNAi des bli-4. Animaux afficher l'arrêt du développement, et sont infimes par rapport aux animaux dans le panneau A. C) ARNi de dpy-13. Les animaux sont au même stade de développement comme des animaux dans le panneau A, mais présentent une morphologie "plutôt moche" corps.
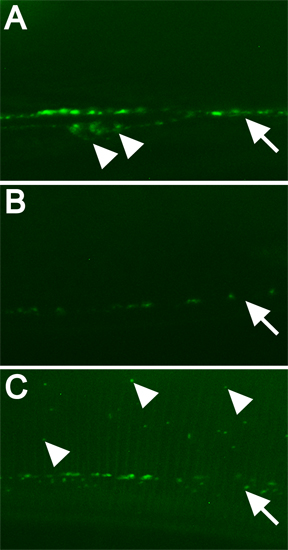
Figure 3. Des exemples de protéines marquées par fluorescence et la façon dont l'ARNi des gènes spécifiques modifie modèle de localisation. Toutes les images ont été prises à un grossissement de 630x avec la microscopie confocale disque en rotation, 5 sec. l'exposition. Échelle = 10 um. Toutes les images ont été traitées de manière identique. Pointes de flèches ouvertes indiquent les corps cellulaires. Les flèches marquent la ligne de punctae. Pointes de flèches remplies indiquent une certaine GFP-tagged aberrante localisée DBL-1. A) ARNi nourris pseudogène C06C3.5 ("wild-type"). B) dbl-1 (ARNi) de contrôle. C) ARNi d'un gène nécessaire pour la localisation normale de la GFP-tagged DBL-1.
Figure 4. Par exemple modèle pour le suivi des expériences de RNAi (clones de la banque) dans des plaques 24 puits. Ce modèle peut être utilisé pour créer un enregistrement permanent des expériences, à la différence directement l'étiquetage des plaques.
Figure 5. Par exemple modèle pour les phénotypes ARNi enregistrement. Développer, au besoin.
Discussion
La méthode de dépistage RNAi présentée ici permet une analyse rapide et sensible des produits des gènes nécessaires à la normale (ou transgéniques) phénotype postembryonnaire. L'exemple montré est un écran de gènes impliqués dans la localisation subcellulaire d'une protéine fluorescente étiqueté. Toutefois, ce protocole peut être modifié pour identifier les gènes qui affectent d'autres phénotypes postembryonnaires d'intérêt.
Cette méthode tire parti d&#...
Déclarations de divulgation
Nous n'avons rien à communiquer.
Remerciements
Les auteurs tiennent à remercier le Dr Rick Padgett (Waksman Institute, l'Université Rutgers, New Jersey) pour le don de la DBL-1 d'ADNc et le Dr Christopher Rongo (Waksman Institute, l'Université Rutgers, New Jersey) pour un marqueur d'injection. Le laboratoire du Dr Barth Grant effectué le bombardement canon à gènes pour l'intégration du nombre de copies bas de la GFP-tagged DBL-1 construit. Le laboratoire de René Garcia fourni une assistance technique lors de la création de texIs100. Le Garcia René, Lints Robyn, et Hongmin Qin laboratoires ont fourni des conseils productifs. Ce travail a été financé par fonds de démarrage du ministère TAMHSC de médecine moléculaire et cellulaire. Le champ d'application composé et confocale disque de rotation ont été achetés avec des fonds fournis par le département et le Collège TAMHSC du Bureau médecine du doyen.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue | Commentaires |
| Agar NGM | Milieu de croissance causée par le nématode | IPM Scientific, Inc | Peut être préparé en suivant le protocole agar NGM 25 |
| M9 moyen | 22mm KH 2 PO 4, 42mm Na 2 HPO 4, 86mm NaCl, 1 mM MgSO 4 | 26 | |
| Agar-Agar | EMD Chemicals Inc | 1.01614.1000 | 2% dans de l'eau pour les plaques NGM. 4% dans de l'eau pour les plaquettes de lames de microscope (autoclave initialement et micro-ondes pour faire fondre par la suite). |
| Bacto Peptone | Becton Dickinson - Difco CP | 211677 | 0,25% |
| IPTG | Recherche International Products Corp | I56000-5.0 | Une concentration de 1 mM final |
| carbénicilline | Recherche International Products Corp | C46000-5.0 | 50 pg / ml de dilution de travail |
| LB Broth Lennox | Becton Dickinson - Difco CP | 240230 | 20 g / litre |
| tétracycline | Sigma | 268054 | 12,5 ug / ml de dilution de travail |
| l'hypochlorite de sodium | Toute marque | Eau de javel à 5% | Utilisez l'eau de Javel fraîche. |
| l'hydroxyde de sodium | Toutes les marques | CAS 1310-73-2 | 5 stock N |
| Milieu M9 | Livre de recettes Wormlab | http://130.15.90.245/wormlab_recipe_book.htm # Commonlab | 26 |
| lévamisol | Sigma | 31742 | 100 uM - 1 mM de dilution de travail |
| azoture de sodium | Fisher Scientific | S227 | 10 mM dans M9 travail de dilution |
| 24 et la plaque | Greiner Bio-One | 662160 | Distributeur VWR |
| des lames de microscope | Toute marque | 75 x 25 x 1 mm | |
| lamelles de microscope | Toute marque | 22 x 22 mm No.1.5 | Utilisez l'épaisseur recommandée par le fabricant de microscope. |
| microscope composé | Carl Zeiss, Inc | A1m | Utilisez objectifs et des filtres pour répondre aux besoins de l'expérience. |
| pompe médias | Manostat Varistaltic pompe | Kate modèle # 72-620-000 | Utiliser un tube et les paramètres appropriés pour la machine |
Références
- Giacomotto, J., Segalat, L. High-throughput screening and small animal models, where are we. Br. J. Pharmacol. 160, 204-216 (2010).
- Lamitina, T. Functional genomic approaches in C. elegans. Methods in Molecular Biology. 351, 127-138 (2006).
- Boutros, M., Ahringer, J. The art and design of genetic screens: RNA interference. Nat. Rev. Genet. 9, 554-566 (2008).
- Praitis, V., Casey, E., Collar, D., Austin, J. Creation of low-copy integrated transgenic lines in Caenorhabditis elegans. Genetics. 157, 1217-1226 (2001).
- Yandell, M. D., Edgar, L. G., Wood, W. B. Trimethylpsoralen induces small deletion mutations in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 91, 1381-1385 (1994).
- Frokjaer-Jensen, C. Single-copy insertion of transgenes in Caenorhabditis elegans. Nature Genetics. 40, 1375-1383 (2008).
- Giordano-Santini, R., Dupuy, D. Selectable genetic markers for nematode transgenesis. Cell. Mol. Life. Sci. , (2011).
- Berkowitz, L. A., Knight, A. L., Caldwell, G. A., Caldwell, K. A. Generation of Stable Transgenic C. elegans Using Microinjection. J. Vis. Exp. (18), e833 (2008).
- Mello, C., Fire, A. DNA transformation. Methods in Cell Biology. 48, 451-482 (1995).
- Simmer, F. Loss of the putative RNA-directed RNA polymerase RRF-3 makes C. elegans hypersensitive to RNAi. Curr. Biol. 12, 1317-1319 (2002).
- Zhuang, J. J., Hunter, C. P. Tissue-specificity of Caenorhabditis elegans enhanced RNAi mutants. Genetics. , (2011).
- Samuelson, A. V., Klimczak, R. R., Thompson, D. B., Carr, C. E., Ruvkun, G. Identification of Caenorhabditis elegans genes regulating longevity using enhanced RNAi-sensitive strains. Cold Spring Harbor Symposia on Quantitative Biology. 72, 489-497 (2007).
- Wang, D. Somatic misexpression of germline P granules and enhanced RNA interference in retinoblastoma pathway mutants. Nature. 436, 593-597 (2005).
- Fraser, A. G. Functional genomic analysis of C. elegans chromosome I by systematic RNA interference. Nature. 408, 325-330 (2000).
- Kamath, R. S. Systematic functional analysis of the Caenorhabditis elegans genome using RNAi. Nature. 421, 231-237 (2003).
- Peters, K., McDowall, J., Rose, A. M. Mutations in the bli-4 (I) locus of Caenorhabditis elegans disrupt both adult cuticle and early larval development. Genetics. , 129-195 (1991).
- Thacker, C., Peters, K., Srayko, M., Rose, A. M. The bli-4 locus of Caenorhabditis elegans encodes structurally distinct kex2/subtilisin-like endoproteases essential for early development and adult morphology. Genes & Development. 9, 956-971 (1995).
- Byerly, L., Cassada, R. C., Russell, R. L. The life cycle of the nematode Caenorhabditis elegans. I. Wild-type growth and reproduction. Dev. Biol. 51, 23-33 (1976).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Savage-Dunn, C. Genetic screen for small body size mutants in C. elegans reveals many TGFbeta pathway components. Genesis. 35, 239-247 (2003).
- Qu, W. Reliability analysis of the Ahringer Caenorhabditis elegans RNAi feeding library: a guide for genome-wide screens. BMC Genomics. 12, 1471-2164 (2011).
- Nelson, M. D. A Bow-Tie Genetic Architecture for Morphogenesis Suggested by a Genome-Wide RNAi Screen in Caenorhabditis elegans. PLoS Genetics. 7, e1002010 (2011).
- Timmons, L., Fire, A. Specific interference by ingested dsRNA. Nature. 395, 854 (1998).
- Sarin, S., Prabhu, S., O'Meara, M. M., Pe'er, I., Hobert, O. Caenorhabditis elegans mutant allele identification by whole-genome sequencing. Nature Methods. 5, 865-867 (2008).
- Lewis, J. A., Fleming, J. T. Basic culture methods. Methods in Cell Biology. 48, 3-29 (1995).
- Brenner, S. The genetics of Caenorhabditis elegans. Genetics. 77, 71-94 (1974).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon