È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
RNAi di screening per identificare fenotipi postembrionale in C. elegans
In questo articolo
Riepilogo
Descriviamo un metodo per identificare i regolatori sensibilizzati postembrionale di espressione proteica e la localizzazione in C. elegans Utilizzando un RNAi-based schermo genomica e un transgene integrato che esprime una funzionale, la proteina fluorescente tag.
Abstract
C. elegans ha dimostrato di essere un sistema modello utile per la scoperta e caratterizzazione funzionale di molti geni e vie del gene 1. Strumenti più sofisticati e le risorse per gli studi in questo sistema sono facilitare la scoperta continua di geni con fenotipi più sottili o ruoli.
Qui vi presentiamo un protocollo generalizzato abbiamo adattato per l'identificazione C. geni elegans con fenotipi postembrionale di interesse utilizzando RNAi 2. Questa procedura viene modificato facilmente per il fenotipo di dosaggio scelta, sia dalla luce ottiche o fluorescenza su un microscopio da dissezione o composto. Questo protocollo di screening sfrutta le risorse fisiche dell'organismo e gli strumenti molecolari del C. elegans comunità di ricerca ha prodotto. Come esempio, si dimostra l'utilizzo di un transgene integrato che esprime un prodotto fluorescente in una schermata RNAi per identificare i geni necessari per la localizzazione normale di questaprodotto in fase avanzata le larve e adulti. In primo luogo, abbiamo utilizzato un commercialmente disponibile libreria genomica RNAi con inserti di cDNA a lunghezza completa. Questa libreria facilita la rapida identificazione di candidati multiple riduzione RNAi del prodotto genico candidato. In secondo luogo, abbiamo generato un transgene integrato che esprime la nostra proteina fluorecently tag di interesse in un RNAi sensibile background. In terzo luogo, esponendo animali tratteggiati per RNAi, questo schermo consentire l'identificazione di prodotti genici che hanno un ruolo fondamentale embrionale che altrimenti mascherare un post-embrionale ruolo nel regolare la proteina di interesse. Infine, questo schermo utilizza un microscopio composto attrezzato per la risoluzione di singola cella.
Protocollo
1. Screening ceppo di costruzione
L'accurata progettazione del ceppo screening è critico per il successo dello schermo ed è stato descritto altrove 3. Per alcuni ricercatori, utilizzando un ceppo che esprime un prodotto visibile da un transgene è necessario per l'esperimento. Molti ceppi ospitano transgeni integrati sono disponibili dai ricercatori CGC o individuale. Se un ceppo transgenico è necessario per lo schermo, ma non è disponibile, allora esso può essere generato utilizzando un metodo pubblicato come bombardamento 4, UV / TMP 5, o Mos trasposone inserimento 6. Per visualizzare la proteina di interesse, si inserisce il GFP-sequenza codificante in frame con la sequenza matura cDNA (abbiamo usato dp sequenza-1). Poiché questa proteina di fusione GFP non è visibile da dissezione campo di applicazione, abbiamo usato un marker coiniezione che era visibile e non ha influenzato la visione della nostra proteina di interesse (TTX-3p :: rfp , per una rassegna di diversi altri indicatori standard, vedere 7). Abbiamo poi creato un transgene integrato dalla matrice extracromosomico. Abbiamo scoperto che il bombardamento del transgene ha prodotto una copia a basso numero di linea integrata che non ha salvato il fenotipo microscopio da dissezione, né prodotto livelli visibili di prodotto GFP-tagged (Beifuss e Gumienny, inedito). UV / TMP integrazione di una matrice di copia multipla extracromosomico originariamente per iniezione 8,9 prodotto visibile, il salvataggio livelli di prodotto transgene (Figura 3A). Western blot con anticorpo anti-GFP ha confermato che il transgene (allele nome texIs100) viene espresso ed elaborato correttamente (Beifuss e Gumienny, inedito). Transgeni integrati dovrebbero essere outcrossed cinque volte a rimuovere le mutazioni di fondo estranei indipendentemente dalla sorgente.
Per il nostro schermo, abbiamo voluto la proteina di interesse solo per il transgenico, form tag. Pertanto, abbiamo rimosso la g funzionale endogenoene con l'introduzione di una perdita di funzione mutante DBL-1 allele.
Infine, si sensibilizzati il ceppo agli effetti di RNAi. La RNAi dalla biblioteca, per molti geni, producono una riduzione più grave prodotto del gene se gli animali contengono una mutazione che sensibilizza gli animali agli effetti della RNAi 10,11 Il tessuto (s) di interesse deve essere considerato nella scelta di un appropriato RNAi sensibilizzazione sfondo 11. Abbiamo usato l'allele canonica RRF-3 (pk1426) per rendere il nostro sforzo screening. RRF-3 è una RNA polimerasi RNA-directed (RdRp) omologo che normalmente inibisce somatica RNAi 10. Mutazioni in altri geni RNAi hypersensitizing come eri-1 o ERI-1 lin-15B può essere utilizzato invece per aumentare l'efficacia della RNAi 11-13.
Il ceppo di screening che abbiamo fatto ha il genotipo RRF-3 (pk1426); texIs100; dbl-1 (NK3).
2. Scelta e preparazione unoRNAi library
Disponibile in commercio C. elegans librerie di cDNA rappresentano circa il 55% o 87% dei geni predetti in C. elegans individualmente (Vidal laboratorio o Ahringer biblioteche di laboratorio, rispettivamente). Cloni individuali sono disponibili per l'acquisto (Open Biosystems, Geneservice, Ltd.). Abbiamo scelto la ORF-RNAi biblioteca costruita dal Vidal laboratorio (Open Biosystems) perché i suoi cloni sono prevalentemente a lunghezza intera Gateway cDNA clonato e pronto per applicazioni a valle (Invitrogen Corporation, Carlsbad, CA). L'originale Ahringer libreria genomica laboratorio cDNA contiene frammenti di cDNA che non sono utili come ampiamente per esperimenti di caratterizzazione valle 14,15. Entrambe le librerie utilizzare un vettore che contiene due promotori T7 fiancheggiano l'inserto (Figura 1). I costrutti sono coltivate in ceppo batterico HT115, che esprime polimerasi T7 dopo induzione da isopropil-β-D-tiogalattopiranoside (IPTG). I batteri contenenti cloni della libreria vengono indotte con IPTG perprodurre dsRNA (vedere punto 3.4).
Duplicare l'intera libreria al momento del ricevimento e utilizzare il duplicato per tutti gli esperimenti. Le librerie originali e duplicato devono essere conservati in vari -80 ° C congelatori che sono collegati per separare le linee elettriche.
3. Preparazione di batteri con cloni libreria
- (Entro due mesi dell'esperimento) batteri Streak da cloni selezionati biblioteca di alimentazione, così come controlli positivi e negativi, su etichetta LB-carbenicillina / tetraciclina piastre coltiviamo 8 culture per piastra da 100 mm) e incubare a 37 ° C per una notte ( > 16 ore) (Figura 1). Per ogni piastra da 24 pozzetti, usiamo due controlli. Il controllo positivo, bli-4 (K04F10.4), produce una dose-dipendente postembrionale pheontype 16,17. Il controllo negativo, C06C3.5, è previsto uno pseudogene ( WormBase.org ) che non provoca fenotipi osservati (Beifuss e Gumienny, inedito).
- (Entro due mesi dell'esperimento, preferibilmente prima) Preparare 24 e nematodi crescita di medio (NGM) piastre contenenti 25 - 50 mcg / ml carbenicillina e 1mM IPTG. Conservare a 4 ° C.
- (Giorno 1) Per ogni clone, inoculare 2 mezzi LB ml contenenti 50 mg / ml carbenicillina con una singola colonia dalla piastra striata (s) (in fase 3.1) con standard di tecnica sterile. Incubare a 37 ° C per una notte (> 16 ore) in un insieme shaker a 220-240 rpm (Figura 1). La soluzione deve essere torbida dopo 16 ore.
- (Giorno 2) La mattina successiva, inducono il doppio filamento di RNA (dsRNA) la produzione con l'aggiunta di IPTG alle culture ad una concentrazione finale di 1 mM. Incubare colture a 37 ° C, 220 - 240 rpm, per ulteriori 4 - 5 ore. Questa fase assicura una più coerente e robusto RNAi indotta knockdown del prodotto del gene che fare affidamento esclusivamente sulla IPTG nel agar della piastra NGM (al punto 3.5).
- Spot 30 pl di ciascuna cu batteriche indottelture nei pozzetti separati di una piastra da 24 pozzetti NGM RNAi contenente 25 - 50 mg / ml carbenicillina e 1mM IPTG (Figura 1, si veda la Figura 4 per il modello utilizzato per monitorare gli esperimenti di RNAi). Mettere le piastre, scoperto, in una cappa a flusso sterile per 20 minuti o fino a quando le macchie batteriche sono asciutti. Non asciugare eccessivamente. Secchino troppo fa sì che i bordi della agar a tirare fuori dalla piastra e gli animali sarà irrimediabile se strisciano nelle fessure che ne derivano.
4. Preparazione di nematodi
Inizia con una popolazione messa in scena di nematodi. Messa in scena gli animali diminuisce la possibilità che le differenze osservate tra il controllo e le prove sperimentali di RNAi sono semplicemente dovuti a differenze di fase dello sviluppo degli animali (tuttavia, se l'RNAi provoca un ritardo dello sviluppo, questo dovrebbe essere rilevato dal screener (Figura 2)) . Inoltre, di iniziare l'esperimento RNAi con larve L1 evita potenziali effetti confondenti di letalità embrionale da RNAi di genes che possono anche svolgere un ruolo postembrionale di interesse.
Staging si ottiene prima sbiancamento mista fase popolazione del ceppo screening, che solo a guscio d'uovo protetti da embrioni sopravvivere. Questo stage animali con un'approssimazione di circa 12 ore a 20 ° C o circa 18 ore a 16 ° C 18. Per gli animali più strettamente teatrali, animali arresto nel primo stadio larvale (L1) lasciando portellone embrioni in terreno M9 senza cibo. L1 animali affamati posta sulla crescita degli alimenti dal curriculum della stessa età di partenza 19.
- (Giorno 1) Utilizzare tre piastre 100 mm di animali nutriti, puliti ceppo di screening che contengono gli adulti gravide ed embrioni. Lavare gli animali da piastre con sterile M9 da Pipette liquido sulla superficie di allentare leggermente la piastrina di animali ed embrioni. Trasferire il lavaggio in una provetta sterile 15 ml con tappo a vite.
- Se il lavaggio è superiore a 3,5 ml, spin down gli animali (~ 3000 rpm per 30 secondi) e rimuovere il liquido a 3,5 ml. Se il lavaggio è Less di 3,5 ml, aggiungere H 2 0 o M9 al tubo al totale 3,5 ml.
- Indossare indumenti protettivi (camice, guanti e occhiali), mentre manipolazione di sostanze chimiche. Mescolare 0,5 ml NaOH 5 N con 1 ml di candeggina fresca (ipoclorito di sodio al 5%, meno di 1 anno di età). Aggiungere questa miscela al lavaggio nematode 3,5 ml. Avviare un timer che conta up. Questo processo non dovrebbe richiedere più di 10 minuti. Se lo fa, allora embrioni saranno in pericolo e la resa può essere ridotto, o la candeggina è vecchio e deve essere sostituito.
- Vortex la provetta per circa 10 secondi. Ripetere il vortex ogni due minuti per 4 a 10 minuti. Dopo ogni vortex, osservare la soluzione in un ambito di dissezione per le carcasse di animali.
- Quando gli embrioni vengono rilasciati e gli adulti sono frammentate (6 - 8 minuti), pellet gli embrioni, facendo girare il tubo in una centrifuga da tavolo (~ 3000 rpm, 30 secondi).
- Aspirare tanto del supernatante possibile senza disturbare il pellet. Attenzione, nonavidi.
- Aggiungi sterile M9 a ~ 10ml. Vortex brevemente o agitare bene per risospendere il pellet.
- Spin e rimuovere il surnatante di nuovo. Se l'odore di candeggina può essere rilevata nel tubo, come necessario ripetere risciacquo.
- Embrioni Risospendere in 10 - 15 ml sterile M9 e trasferimento in un piccolo (25 ml) Beuta (per l'aerazione). Agitare a temperatura adeguata per il ceppo e dell'esperimento (15 - 25 ° C) per una notte (> 16 ore a 20 ° C). In questo tempo, gli animali saranno tutti schiudono e l'arresto in diapausa L1.
- (Giorno 2) Spin premuto L1 animali stadio in un tubo da 15 ml e risospendere in 0,5 - 1 ml M9 (~ 3000 rpm, 30 sec.). Mettere 5 ul di soluzione su una piastra separata e contare il numero di animali. Eseguire le regolazioni del volume, se necessario, a cedere 30 vermi / 3 - 10 ul di soluzione.
- Spot circa 30 ipoclorito sincronizzati animali L1 stadio sui batteri in ciascun pozzetto della preparato piastra da 24 pozzetti. Troppi mori di 30 animali per ben può comportare la popolazione affamata prima della maturità. Lasciate che il monodisco a secco con il traverso coperchi. Sostituire il coperchio e fissarlo con un elastico. Capovolgere la piastra e posizionarlo a temperatura appropriata per il tempo necessario per segnare in fase desiderata. Poiché RRF-3 (pk1426) è un allele sensibile alla temperatura, gli animali ceppo di screening sono state coltivate a 15 - 17 ° C per tre o quattro giorni per osservare L4 agli animali stadio adulto (Figura 1).
5. Osservazione dei nematodi
(Giorno 5) Una volta che i nematodi sono cresciuti allo stadio desiderato (Figura 2A), confermano che i controlli positivi e negativi produrre i fenotipi attesi utilizzando un microscopio da dissezione. Usiamo bli-4 come un controllo per l'efficacia RNAi, perché produce dose-dipendenti post-embrionali difetti che vanno dagli adulti vesciche (lieve fenotipo RNAi, non mostrato) al evolutivi arrestato, piccole larve (forte, expected RNAi fenotipo) (Figura 2B). Poi osservare gli animali in ciascun sperimentale pozzetto usando un microscopio da dissezione e note fenotipi evidenti anormali indotta da RNAi (Figura 2C). Dopo i controlli sono confermati e fenotipi lordi notato, schermare gli esperimenti di RNAi per fenotipi di interesse. Usiamo un microscopio a fluorescenza e dotato di un obiettivo 63x per osservare almeno cinque animali da esperimento RNAi ciascuna, cominciando con controlli (Figura 3).
- Preparare una slitta (standard di 75 x 25 mm) facendo una pad agar 4% (4% agar in H 2 O). Per garantire uno spessore uniforme di pad agar, posizionare un vetrino di vetro pulito tra due distanziali (ciascun distanziatore è fatto di un vetrino da microscopio con due strati di nastro di laboratorio su di esso). Pipettare circa 100 pl (due o tre gocce di una pipetta Pasteur bicchiere) di agar fuso 4% sul centro del vetrino pulito. Forma un tampone agar coprendo l'agar fuso con un vetrino supplementare collocato rapidamente ma delicatamente sulla sommità di uno dei distanzialind l'agar fuso. Esporre il pad agar facendo scorrere delicatamente i vetrini a parte, lasciando il pad che aderisce a una diapositiva.
- Mettere una goccia ~ 5 microlitri di anestetico (levamisole o azide di sodio, per esempio) sul pad agar. Monte 8 - 12 animali l'anestetico. Coprire con un 22 x 22 mm # 1,5 coprioggetto (o lo spessore del vetrino più adatto per il microscopio composto) e osservare gli animali utilizzando un microscopio composto.
- Risultati Record gene, il numero di animali osservati, e fenotipo di ciascun esperimento RNAi. Per uno schermo, realizzare e utilizzare un modello standard (vedi esempio, la Figura 5).
- Verifica RNAi-fenotipo prodotta da un altro, fenotipo secondario, se possibile, e ripetendo l'esperimento. (Ad esempio, lo schermo utilizzato nell'esempio illustrato alterazione di GFP-tag DBL-1 localizzazione come schermo primario e le dimensioni del corpo come uno schermo secondario 20.) Batteri possono essere utilizzati dalla striscia stessa (conservato a 4 ° C) fino a due mesi. Anziani striature possono rISULTATO in scarsa crescita in liquidi culture durante la notte e dovrebbe essere restreaked prima.
- Sequenza almeno una estremità dell'inserto di cDNA per confermare che l'inserto corrisponde all'etichetta libreria (libreria Ahringer ha avuto una analisi di affidabilità effettuata; vedere http://biocompute.bmi.ac.cn/CelRNAi/ ) 21. Primer consigliati (per gli inserimenti da entrambe le librerie) ricottura sui siti della L4440-derived vettore e fiancheggiano il sito di inserimento: AGCGAGTCAGTGAGCGAG-5'-3 'e 5'-GTAAAACGACGGCCAGT-3' (M13f20 primer).
Utilizzando questo metodo, una sola persona può ragionevolmente mettere in scena e osservare da due a quattro serie di esperimenti di ogni settimana. Per esempio, gli animali in scena il Lunedi e Martedì possono essere osservati Giovedi e Venerdì, rispettivamente, e può essere nuovamente in scena il Venerdì e Sabato per Lunedi e Martedì osservazione. A seconda del fenotipo (s) proiettato ogni giorno, una serie può comprendere una piastra da 24 pozzetti composto da microscopia o più se il fenotipo è presto identificato. Così, almeno 88 diversi esperimenti RNAi può essere osservata in una settimana, tenendo conto controlli positivi e negativi e tasso di screening. Uno schermo portata dissezione può essere eseguito molto più veloce, senza la necessità di montaggio animali su vetrini per la visualizzazione. Più persone potrebbero anche aumentare la velocità di un laboratorio da parte orari impressionanti e / o utilizzando microscopi multipli. Un metodo alternativo per la crescita di animali su una pellicola sottile di agar e trasferire una fetta di agar contenente animali direttamente su un vetrino per la visualizzazione può risparmiare tempo. Questa variante è stata usata con successo per lo screening di anomalie coda maschi a 400x di ingrandimento 22.
6. Risultati rappresentativi
Esempi di localizzazione normale e alterata di GFP-tag DBL-1 sono mostrati in Figura 3. Normale espressione di GFP-tag DBL-1 include ventrali nervose corpi cellulari cavo e una fila di punctae (Figura 3A). DBL-1 is fortemente attenuato quando animali sono alimentati RNA che impedisce DBL-1 mRNA traduzione (Figura 3B). dbl-1 (RNAi) produce anche piccoli animali, lo schermo secondario in questo esempio (dati non mostrati). I geni che influiscono DBL-1 localizzazione sono facilmente identificabili da RNAi utilizzando un ceppo progettato per questo schermo (Figura 3C).

Figura 1. Schema di schermo per identificare regolatori extracellulari di DBL-1 segnalazione. I batteri della libreria di alimentazione vengono coltivati per una notte, indotta con IPTG, e incubate a 37 ° C per altre 4 ore per permettere ai batteri di produrre RNA a doppio filamento (dsRNA). 30 microlitri della coltura batterica indotta è macchiato per pozzetto su un 24-ben NGM (crescita media nematode) piastra contenente 25 ug / ml carbenicillina e IPTG 1 mM e lasciate asciugare in una cappa a flusso sterile. Ci primo stadio larvale stadio (L1) larve lasciando ipoclorito trattato embryo si schiudono in media senza cibo, che induce una diapausa L1 (arresto di sviluppo). Circa 30 animali sincronizzati fase L1 sono placcati su ciascun pozzetto contenente i batteri dalla libreria RNAi, che permette in scena gli animali di riprendere la crescita e di consumare la dsRNA creato dai batteri 23. Dopo 72 ore a 15 ° C, i vermi adulti giovani sono sottoposti a screening per un fenotipo visibile. A questa età, i difetti sono evidenti dimensioni del corpo e la fluorescenza è luminoso da texIs100 espressione del transgene.
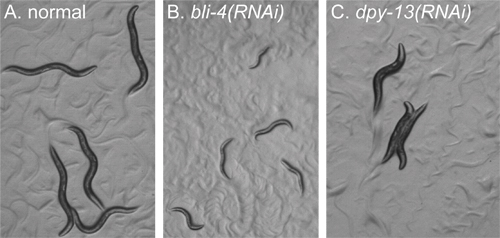
Figura 2. Esempi di fenotipi per identificare prima del controllo. Tutte le immagini di animali in una piastra pozzetto sono state adottate allo stesso ingrandimento utilizzando un microscopio da dissezione. Gli animali sono stati tutti ripresi circa 72 ore dopo la placcatura come larve L1 affamata. Tutte le immagini sono state trattate in modo identico. A) RNAi di un gene che non dà difetti morfologici grossolani. Gli animali appaiono di tipo selvatico. B) RNAi di bli-4. Animali visualizzare arresto di sviluppo, e sono molto piccoli rispetto agli animali nel pannello A. C) RNAi di DPY-13. Gli animali sono allo stesso stadio di sviluppo come animali nel pannello A, ma visualizzare una morfologia "losca" corpo.
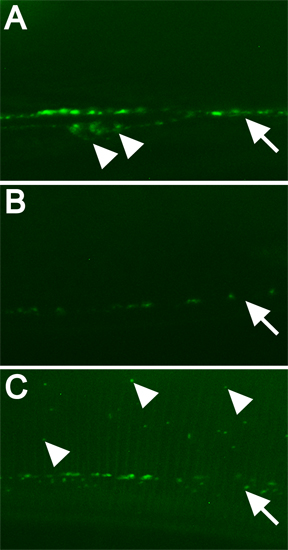
Figura 3. Esempi di proteine fluorescenti con tag e come RNAi di geni specifici altera modello di localizzazione. Tutte le immagini sono state scattate con un ingrandimento 630x disco rotante con la microscopia confocale, 5 sec. esposizione. Scala bar = 10 micron. Tutte le immagini sono state trattate in modo identico. Open frecce indicano corpi cellulari. Frecce segnare linea di punctae. Frecce piene indicano alcuni GFP-tagged aberrante localizzato DBL-1. A) RNAi-fed pseudogene C06C3.5 ("wild-type"). B) dbl-1 (RNAi) di controllo. C) RNAi di un gene necessario per la localizzazione normale di GFP-tagged DBL-1.
Figura 4. Esempio di modello per il monitoraggio esperimenti di RNAi (cloni della libreria) in piastre da 24 pozzetti. Questo modello può essere utilizzato per creare una registrazione permanente degli esperimenti, a differenza direttamente etichettatura piastre.
Figura 5. Esempio di modello per la registrazione di fenotipi RNAi. Espandere, se necessario.
Discussione
Il metodo di screening RNAi qui presentata consente un'analisi sensibile e rapido di prodotti genici necessari per un normale (o transgenico) fenotipo postembrionale. L'esempio illustrato è uno schermo per i geni coinvolti nella localizzazione subcellulare di una proteina fluorescente etichettato. Tuttavia, questo protocollo può essere modificata per identificare geni che interessano altri fenotipi postembrionale di interesse.
Questo metodo si avvale di un approccio del gene candid...
Divulgazioni
Non abbiamo nulla da rivelare.
Riconoscimenti
Gli autori desiderano ringraziare il Dr. Rick Padgett (Waksman Institute, Rutgers University, NJ) per il dono della DBL-1 cDNA e il Dr. Christopher Rongo (Waksman Institute, Rutgers University, NJ) per un marker di iniezione. Laboratorio del Dr. Barth Grant effettuato il bombardamento pistola gene per l'integrazione a basso numero di copie della GFP-tagged DBL-1 costrutto. Il laboratorio di René Garcia ha fornito assistenza tecnica durante la creazione del texIs100. Il René Garcia, Lint Robyn, e Hongmin Qin laboratori fornito consulenza produttivo. Questo lavoro è stato finanziato da fondi di start-up del Dipartimento TAMHSC di Medicina Molecolare e Cellulare. L'ambito composto e confocale disco rotante sono stati acquistati con fondi messi a disposizione dal dipartimento e il Collegio TAMHSC dell'Ufficio Medicina della Dean.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | Comments |
| NGM Agar | Nematode crescita media | IPM Scientific, Inc | Può essere preparato secondo il protocollo NGM agar 25 |
| M9 Media | 22mm KH 2 PO 4, 42mm Na 2 HPO 4, 86mm NaCl, 1 mM MgSO 4 | 26 | |
| Agar-Agar | EMD Chemicals Inc. | 1.01614.1000 | 2% in acqua per lastre NGM. 4% in acqua per pastiglie vetrino da microscopio (inizialmente in autoclave e forno a microonde per sciogliere successivamente). |
| Bacto Peptone | Becton Dickinson - Difco CP | 211677 | 0,25% |
| IPTG | Research Products International Corp. | I56000-5.0 | Una concentrazione di 1 mM finale |
| carbenicillina | Research Products International Corp. | C46000-5.0 | 50 ug / ml di lavoro diluizione |
| LB Broth Lennox | Becton Dickinson - Difco CP | 240230 | 20 g / litro |
| tetraciclina | Sigma | 268054 | 12,5 ug / ml di lavoro diluizione |
| ipoclorito di sodio | Qualsiasi marchio | Candeggina al 5% | Usa candeggina fresca. |
| idrossido di sodio | Qualsiasi Marca | CAS 1310-73-2 | 5 N magazzino |
| M9 media | Wormlab Ricettario | http://130.15.90.245/wormlab_recipe_book.htm # Commonlab | 26 |
| Levamisol | Sigma | 31742 | 100 pM - diluizione 1 mM di lavoro |
| di sodio azide | Fisher Scientific | S227 | 10 mM in diluizione M9 lavoro |
| Piastra da 24 pozzetti | Greiner Bio-One | 662160 | VWR distributore |
| vetrini | Qualsiasi marchio | 75 x 25 x 1 mm | |
| microscopio vetrini coprioggetto | Qualsiasi marchio | 22 x 22 mm No.1.5 | Utilizzare lo spessore raccomandato dal produttore microscopio. |
| microscopio composto | Carl Zeiss, Inc. | A1M | Utilizzare gli obiettivi e filtri per soddisfare le esigenze dell'esperimento. |
| mezzi di pompa | Pressostato Varistaltic pompa | Kate modello # 72-620-000 | Utilizzare tubi e le impostazioni appropriate per la macchina |
Riferimenti
- Giacomotto, J., Segalat, L. High-throughput screening and small animal models, where are we. Br. J. Pharmacol. 160, 204-216 (2010).
- Lamitina, T. Functional genomic approaches in C. elegans. Methods in Molecular Biology. 351, 127-138 (2006).
- Boutros, M., Ahringer, J. The art and design of genetic screens: RNA interference. Nat. Rev. Genet. 9, 554-566 (2008).
- Praitis, V., Casey, E., Collar, D., Austin, J. Creation of low-copy integrated transgenic lines in Caenorhabditis elegans. Genetics. 157, 1217-1226 (2001).
- Yandell, M. D., Edgar, L. G., Wood, W. B. Trimethylpsoralen induces small deletion mutations in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 91, 1381-1385 (1994).
- Frokjaer-Jensen, C. Single-copy insertion of transgenes in Caenorhabditis elegans. Nature Genetics. 40, 1375-1383 (2008).
- Giordano-Santini, R., Dupuy, D. Selectable genetic markers for nematode transgenesis. Cell. Mol. Life. Sci. , (2011).
- Berkowitz, L. A., Knight, A. L., Caldwell, G. A., Caldwell, K. A. Generation of Stable Transgenic C. elegans Using Microinjection. J. Vis. Exp. (18), e833 (2008).
- Mello, C., Fire, A. DNA transformation. Methods in Cell Biology. 48, 451-482 (1995).
- Simmer, F. Loss of the putative RNA-directed RNA polymerase RRF-3 makes C. elegans hypersensitive to RNAi. Curr. Biol. 12, 1317-1319 (2002).
- Zhuang, J. J., Hunter, C. P. Tissue-specificity of Caenorhabditis elegans enhanced RNAi mutants. Genetics. , (2011).
- Samuelson, A. V., Klimczak, R. R., Thompson, D. B., Carr, C. E., Ruvkun, G. Identification of Caenorhabditis elegans genes regulating longevity using enhanced RNAi-sensitive strains. Cold Spring Harbor Symposia on Quantitative Biology. 72, 489-497 (2007).
- Wang, D. Somatic misexpression of germline P granules and enhanced RNA interference in retinoblastoma pathway mutants. Nature. 436, 593-597 (2005).
- Fraser, A. G. Functional genomic analysis of C. elegans chromosome I by systematic RNA interference. Nature. 408, 325-330 (2000).
- Kamath, R. S. Systematic functional analysis of the Caenorhabditis elegans genome using RNAi. Nature. 421, 231-237 (2003).
- Peters, K., McDowall, J., Rose, A. M. Mutations in the bli-4 (I) locus of Caenorhabditis elegans disrupt both adult cuticle and early larval development. Genetics. , 129-195 (1991).
- Thacker, C., Peters, K., Srayko, M., Rose, A. M. The bli-4 locus of Caenorhabditis elegans encodes structurally distinct kex2/subtilisin-like endoproteases essential for early development and adult morphology. Genes & Development. 9, 956-971 (1995).
- Byerly, L., Cassada, R. C., Russell, R. L. The life cycle of the nematode Caenorhabditis elegans. I. Wild-type growth and reproduction. Dev. Biol. 51, 23-33 (1976).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Savage-Dunn, C. Genetic screen for small body size mutants in C. elegans reveals many TGFbeta pathway components. Genesis. 35, 239-247 (2003).
- Qu, W. Reliability analysis of the Ahringer Caenorhabditis elegans RNAi feeding library: a guide for genome-wide screens. BMC Genomics. 12, 1471-2164 (2011).
- Nelson, M. D. A Bow-Tie Genetic Architecture for Morphogenesis Suggested by a Genome-Wide RNAi Screen in Caenorhabditis elegans. PLoS Genetics. 7, e1002010 (2011).
- Timmons, L., Fire, A. Specific interference by ingested dsRNA. Nature. 395, 854 (1998).
- Sarin, S., Prabhu, S., O'Meara, M. M., Pe'er, I., Hobert, O. Caenorhabditis elegans mutant allele identification by whole-genome sequencing. Nature Methods. 5, 865-867 (2008).
- Lewis, J. A., Fleming, J. T. Basic culture methods. Methods in Cell Biology. 48, 3-29 (1995).
- Brenner, S. The genetics of Caenorhabditis elegans. Genetics. 77, 71-94 (1974).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon