Method Article
Generierung von Dispersed präsomitischen Mesoderm Zellkulturen für Imaging der Zebrafisch-Segmentierung Uhr in Einzelzellen
In diesem Artikel
Zusammenfassung
Somitogenese ist eine rhythmische Entwicklungsprozess, die räumlich Muster die Körperachse von Wirbeltierembryonen. Bisher haben wir transgenen Zebrafischlinien, die fluoreszierenden Reporter nutzen, um die zyklischen Gene, die dieser Prozess vorangetrieben beobachten. Hier wir Kultur verteilt Zellen aus diesen Linien und Bild ihre Schwingungen über die Zeit in vitro.
Zusammenfassung
Segmentierung ist ein periodischer und sequentielle morphogenetischen Prozess bei Wirbeltieren. Diese rhythmische Bildung von Blöcken aus Gewebe genannt Somiten entlang der Körperachse ist Nachweis einer genetischen Oszillator Strukturieren des sich entwickelnden Embryos. Im Zebrafisch wird die intrazelluläre Uhr fahren Segmentierung der Mitglieder des Her / Hes Transkriptionsfaktor-Familie in den negativen Rückkopplungsschleifen organisiert zusammen. Wir haben kürzlich erzeugten transgenen fluoreszierenden Reporterlinien für die zyklische Gen HER1, die die räumlich-zeitliche Muster der Schwingungen in präsomitischen Mesoderm (PSM) zu rekapitulieren. Mit diesen Zeilen, ein in vitro-Kultursystem, die Echtzeit-Analyse der Segmentierung Uhr Schwingungen innerhalb einzelner, isolierter PSM Zellen erlaubt entwickelten wir. Durch das Entfernen von PSM Gewebe von transgenen Embryos und dann Dispergieren Zellen aus schwing Regionen auf Glasboden Gerichte erzielten wir Kulturen geeignet für Zeitraffer-Bildgebung der Fluoreszenzsignal voneinzelnen Taktzellen. Dieser Ansatz bietet eine experimentelle und konzeptuelle Rahmen für die direkte Manipulation der Segmentierung Uhr mit beispiellosen Einzelzellen-Auflösung, so dass seine zellautonome und Tissue Level-Eigenschaften zu unterscheiden und seziert werden.
Einleitung
Die periodische Bildung von Segmenten entlang der Wirbeltierkörperachse oder Somitogenese, ist ein Beweis für eine räumliche und zeitliche Oszillator im sich entwickelnden Embryo. Die bevorzugte Mechanismus Steuerung Somitogenese ist konzeptionell von einem "Takt-und Wellenfront"-Modell ein, bei dem die "Uhr", bestehend aus zellulären Oszillatoren jetzt vermutlich intrazellulär durch die rhythmische Expression einer Reihe von Genen zyklischen 2 angetrieben werden, hakt die Bildung beschrieben Somiten von präsomitischen Mesoderm (PSM). Da der Embryo entwickelt, eine Reifung "Wellenfront" in der PSM bewegt sich zusammen mit der Geweberückschritte in Richtung der hinteren, Verlangsamung und verhaften zellulären Oszillatoren, wie es geht 3. Gemeinsam wird diese raumzeitlich dynamischen System die Segmentierung Uhr bezeichnet. Aktuelle Ansätze zur Segmentierung Uhr Spannweite drei zunehmenden Organisation der genetischen Oszillator in einzelnen Zellen auf lokale Kopplung th studierenan tritt zwischen Zellen und schließlich die weltweite Regulierung der Positionsinformationen in der kollektiven PSM Gewebe 4.
Frühere Studien legen nahe, die zellautonome Segmentierung Oszillator im Zebrafisch besteht aus Genen und Proteinprodukte aus der ihr / hes factorfamily Transkription, die gedacht werden, um eine negative Rückkopplungsschleife über Transkriptionsrepression 5-7 bilden. Die Delta / Notch-Signalwegs synchronisiert Schwingungen zwischen Nachbarzellen und reguliert die Tarifperiode der Bevölkerung 10.8. In der Schwanzknospe erzeugt Fgf Signalmoleküle scheinen einen Gradienten über die Zebrafisch-PSM zu bauen, und dabei die Hypothese aufgestellt, um zu verlangsamen und zu verhaften oszillierenden Zellen in der vorderen 11 beitragen. Bis jetzt haben die funktionellen Rollen jedes dieser Moleküle in Somitogenese durch genetische Mutation, Morpholino-Injektion, Hitzeschock-Überexpression und-Antagonist Medikament TREA suchttment Uhr von Komponenten und Signalisierung zwischen Zellen 5,7,10,12. Mit Hilfe dieser Störungen hat Segmentierung Uhr-Funktion von Gewebe-Level-Beschreibungen der Somitendefekten und den Verlust der gleichmäßigen Schwingungen in der Expression von Genen wie zyklische her1, her7 und Delta abgeleitet worden C. Doch fast alle diese Daten sind von Embryonen fixiert und nicht zu Veränderungen, was für das dynamische Funktion der Segmentierung Uhr sind genau zu erfassen. In jüngerer Zeit hat mehr Embryo Zeitraffer-Bildgebung die ersten Mutanten mit veränderter Oszillatorperiode offenbart, sondern diese Beobachtungen wurden auch bei der Gewebeniveau 7,13 gemacht. So wurde die Hypothese aufgestellt Verhalten der Zelle autonomen Oszillators während Somitogenese nicht beobachtet.
Statische Schnappschüsse von Somitogenese ein unvollständiges Bild, denn von Natur aus, wird der Prozess durch ein schwingfähiges System angetrieben. Frühere Arbeiten in Maus-und Küken-Zellen zeigten, dass das Niveau der Niederschriftund Protein-Aufstieg und Fall, aber Abtasten eines ca. 2 h Schwingung alle 30 oder 45 min beschränkt notwendigerweise die gesammelten Daten und damit die Schlussfolgerungen, die gezogen werden können 14,15. Studium der anderen biologischen Oszillatoren, vor allem, circadiane Uhren, hat sich von inszenierten Messungen von Gen-und Proteinexpression um Echtzeit-Überwachung mit Fluoreszenz-und Biolumineszenz Reporter 16,17 bewegt. Diese Instrumente sind wichtig für den Nachweis Uhr Eigenschaften von Einzelzellen 18. Ein Reporter der Biolumineszenz Hes1 zyklischen Gen wurde entwickelt und kurz in einzelnen Zellen der Maus PSM 19 aus. Die Durchschnittsperiode und der Varianz wurden für eine kleine Anzahl von Zellen berechnet wird, zeigt, dass Schwingungen bestehen für mehrere Zyklen in vitro. Allerdings haben diese Studien nicht quantitativ anzugehen, die Stabilität und Robustheit der Oszillator-Frequenz und Amplitude, ob Zellen können spontan betreten oder verlassen Schwingungen,und wie die Zellen behalten ihre Phasenbeziehungen. Zusätzlich können die Effekte von Signalmolekülen in der Embryos auf dem Zell-autonome Uhr wurden nicht direkt getestet. Folglich sind diese grundlegenden Eigenschaften der einzelnen Zelle Oszillator bleiben völlig unbekannt.
Wir haben vor kurzem transgene Fische Linien mit BAC Rekombination 20 bis Venus (YFP) Fluoreszenz-Reporter von 30 her1 Ausdruck fahren entwickelt. Solche Leitungen nutzen die regulatorischen Signale des intakten chromosomalen Locus in die sie eingebettet sind, und wiederholen die zeitliche Dynamik und räumlichen Muster der HER1. Dieser Durchbruch ermöglicht die Echtzeit-Überwachung der Gen-Expression in sich entwickelnden Zebrafischembryos in vivo. Um die grundlegenden Eigenschaften von Zell-autonome Schwingungen und wie solche Ausdruck wird über die Zeit geregelt zu studieren, haben wir vor kurzem eine zuverlässige Methode, um von PSM-Zellen in vitro zu isolieren und zu Rekord. Diese Protocol beschreibt, wie wir unsere Reporter transgenen Linien verwendet werden, um verteilte Zellkulturen, aus denen wir die Schwingungen des Zebrafisch-Segmentierung Uhr in einzelne Zellen zu charakterisieren generieren. Wir können damit noch offenen Fragen anzugehen in dem Feld, das nicht zugänglich mit statischen oder Gewebe-Level-Analyse waren, als auch direkt manipulieren die Segmentierung Uhr auf Einzelzellebene mit Signalmolekülen und Inhibitoren.
Protokoll
1. Bevor Dissection
- Am Tag vor der Dissektion erhalten Embryonen aus einer incross der Zebrafisch-Paare heterozygot für das transgene Allel.
- Heben Embryonen in E3-Medium ohne Methylenblau bei 28 o C bis Schild Stufe (6 Stunden nach der Befruchtung).
- Transfer-Embryonen in E3-Medium ohne Methylenblau bis 20 o CO / N. Bei 20 o C, werden Embryonen 1 h pro Somiten bilden, sobald sie Schwanzknospen-Stadium zu erreichen. Nach 17-20 h O / N bei 20 ° C, sollte Embryonen im 5-8 Somiten sein inszenieren am folgenden Morgen zu Beginn der Dissektion Protokoll.
- Assemble-Tools und Reagenzien für die Präparation erforderlich.
- Feuerpolierte Glaspipette für die Übertragung von Embryonen und Gewebe
- Feinen Pinzette zur Entfernung von Chorion aus Embryonen
- Sylgard-beschichtete 35-mm-Schale für die Präparation - Gießen Sylgard Polymer in eine 35 mm-Schale und Heilung O / N in einem 37 ° C Machen Sie das auch mit Dissektioneine Nadelspitze auf ein kleines Volumen des ausgehärteten Polymer zu entfernen. Das Gericht kann gereinigt und anschließend wiederverwendet werden.
- Geschärfte, abgeflacht Wolframdraht-Tools für PSM Gewebemanipulation
- Mikro-Skalpell zum Schneiden gewünschten Stücke von PSM zu Kultur
- 35-mm-Kunststoffschale für Trypsin Inkubation
- L15-Medium mit 10% fötalem Rinderserum
- 0,25% Trypsin / EDTA-Lösung
- Sigmacoted Gel-loading Tipps für die Verbreitung
- Petrischalen aus Kunststoff E3-Medium ohne Methylenblau.
- Decken Sie Glasbodenschale mit Bild Fibronectin1 Substrat (10 ug / ml in PBS). Lassen Gericht auf der Bank zu beschichten beim Präparieren.
- Verwenden Stereoskop mit geeigneten Fluoreszenzfiltern zu identifizieren und zu sortieren positiven transgenen Embryonen (5-8 Somitenstadium).
- Identifizieren transgenen Embryos durch die Untersuchung der präsomitischen Mesoderm (PSM) für YFP Expression unter Fluoreszenzkanal (Abbildung 1A). Fluoreszenz-shoULD unsichtbar im Bereich der letzten Somiten zum Schwanzknospen. Wählen Embryonen mit hellsten Signal; 25% der Nachkommen sollten homozygoten Embryonen mit 2 Kopien des Transgens sein. Die Anzahl der identifizierten Embryonen erforderlich ist je nach Experiment basiert. Ein Stück wird seziert Schwanzknospen ergeben 1000 Zellen, im Durchschnitt. Normalerweise sind ein paar zusätzliche positive Embryonen nützlich, wenn Fehler während der Präparation gemacht.
- Verwenden Durchlicht als Positionsbezug, jede Autofluoreszenz zu unterscheiden, vor allem in den Dotterzelle, aus dem Signal.
- Übertragen positive Embryonen zu einem separaten Kunststoffpetrischale mit E3-Medium ohne Methylenblau.
2. PSM Dissection und Zerstreuung
- Bereiten Embryonen für die Präparation.
- Unter einem Dissektionsmikroskop, mit feinen Pinzette vorsichtig entfernen das Chorion von jedem Embryo in der E3-Medium. Achten Sie darauf, um den Embryo zu beschädigen oder stören die Dotterzelle.
- Füllen Sylgard-beschichtete Sezieren Gericht mit L15-Medium mit Serum. Mit einer Pinzette oder einem anderen flachen Werkzeug, entfernen Sie alle Luftblasen von der Oberfläche des Sylgard.
- Übertragen dechorionated Embryonen mit der feuerpolierten Glaspipette auf die Dissektion Gericht.
- Mit den Drahtwerkzeuge, verschieben Sie alle Embryonen auf der einen Seite der Sezieren Gericht.
- Dissect PSM von einer einzigen Embryo.
- Orient ein einzelner Embryo auf seiner lateralen Seite in der kleinen und in der Sylgard Schicht innerhalb der Sezieren Gericht (Abbildung 1B) gemacht.
- Mit der Mikro-Skalpell, Schnitt durch den Embryo und Dotterzelle knapp vor den Hinterhirn und durch die ventralen Pol des Embryos (1B, gestrichelte rote Linie).
- Entfernen Sie die vordere Stück des Embryos aus dem Brunnen, bewegen sie weg auf die Seite der Schale, und dann die Draht Werkzeuge, um aus dem hinteren Abschnitt einschließlich der PSM weg kratzen restlichen Dotterzelle Körnchen.
- Sobald das Eigelb wurde abgekratzt, glätten und richten Sie die PSM mit dem vorderen Ende nach vom Experimentator und die hintere in Richtung des Experimentators (Abbildung 1C).
- Wenn die dünne Schicht Ektoderm hat sich nicht von dieser Stelle gezogen wird, verwenden Sie die Draht Werkzeuge, um sie zu schälen weg von der Spitze des PSM Gewebe.
- Mit der Mikro-Skalpell die Schwanzknospe, die hintersten Spitze des PSM über das Ende der Chorda, weg vom Rest des Gewebes (1C, gestrichelte rote Linie). Hinweis: andere Gewebeteile von der PSM beispiels anterioren PSM näher an der zuletzt gebildeten Somiten, können auch in Kultur genommen werden, abhängig von den experimentellen Frage.
- Bewegen Sie den Schwanzknospen Stück in eine Ecke der Schale von der Sektionsbereich. Verwenden Sie die Draht Werkzeuge, um jeglichen Schmutz und unerwünschten Embryo Gewebe aus dem Seziersaal Feld zu löschen.
- Wiederholen Sie mit dem nächsten Embryo.
- Pool Schwanzknospen Stücke aus mehreren Embryos, je nachdem, wie viele Zellen für das Experiment erforderlich ist. Im Durchschnitt ein einziges Stück Schwanzknospen Gewebe ergibt 1000 Zellen.
- Füllen Sie leere 35-mm-Kunststoffschale mit einem kleinen Volumen von Trypsin-EDTA.
- Mit der feuerpolierten Glas Pipette Schwanzknospen Stücke aus Sezieren Gericht in Schale mit Trypsin / EDTA. Schwanzknospen Stücke inkubieren in Trypsin / EDTA für 20 Minuten bei RT.
- Während das Gewebe wird in Trypsin, entfernen Sie die Fibronectin1 Lösung aus dem Glasboden-Imaging-Gericht.
- Mit MilliQ Wasser waschen die Lösung aus Glas 3 mal. Saugen Sie jeden Wasch zu entfernen und sicherzustellen, dass das Gericht vollständig trocken ist.
- Dispersionsschwanzknospen Stücke in Medium für Bildgebung.
- Je 100 ul der L15-Medium mit Serum in bildgebenden Gericht.
- Mit einem Gel-beschichteten Spitze, entfernen Sie die Stücke aus der Schwanzknospe Trypsin / EDTA in einem möglichst kleinen Volumen wie möglich.
- Pipettieren Schwanzknospen-Stücke in das Medium in derBild Gericht. Pipettieren Sie die Stücke nach oben und unten mehrmals, um sie auseinander brechen und Zellen aussetzen im Medium. Achten Sie darauf, um Luftblasen zu einzuführen. Check für Zellklumpen unter dem Mikroskop und verteilen mehr als nötig.
- Lassen Sie verteilten Zellen in der Suspension auf die Fibronectin1-beschichtete Glas für 20 min bei RT begleichen.
- Hinzufügen eines kleinen Volumens von Zusatz L15-Medium mit Serum zu den Zellen vor Beginn der Bildgebung. Seien Sie vorsichtig, nicht zu ständiger Zellen zu zerstören.
- Angesichts der experimentellen Frage, fügen Sie keine zusätzliche oder medikamentöse Behandlungen auf die Kultur.
3. Imaging von Dispersed PSM Zellen
- Richten Sie Bild Gericht Temperaturkammer auf der Zeitraffer-Imaging-Mikroskop. Eingestellt Warner Kammer auf die gewünschte Temperatur für das Experiment. Lassen Gericht für mindestens 30 min ins Gleichgewicht vor Beginn der Bildaufnahme. Crosscheck Temperatur mit externen Temperaturfühler in die Schale gelegt, falls erforderlich. HINWEIS: Aufgrund derdie Beziehung zwischen Temperatur und Somitogenese Rate 21 stabile Temperaturregelung ist für genaue Messungen der Periode, in einzelnen PSM-Zellen.
- Erwerben Sie Testbilder in der Fluoreszenzkanal, um zu überprüfen, dass die Belichtungszeit und Gain bieten einen großen dynamischen Bereich von Intensitäten ohne Sättigung zu guten Rauschpegel zu gewährleisten. Ein typisches Fluoreszenzbild von dispergierten Zellen aus unseren Linien erzeugt mit diesem Protokoll erfassten erfordert 400 ms und 40 ms für einen Durchlichtbild mit einem EMCCD Kamera, die bei einer EM-Gewinn von 85. Außerdem Pre-Amp Gain und Auslesegeschwindigkeit aus der Kamera sind auch wichtig, um das Signal über Lärm zu maximieren.
- Mit dem Durchlichtkanal, wählen Sie Felder von Zellen für die Zeitraffer-Akquisition.
- Führen Zeitraffer-Akquisition-Protokoll für die gewünschte Länge der Zeit.
- Erwerben Sie ein Durchlicht-und Fluoreszenzbild einer pro Feld. HINWEIS: Stellen Sie ein Intervall zwischen den Anschaffungs rounds, die zeitliche Dynamik ohne Foto-Bleichen über erweiterte Bildgebung oder Induktion Toxizität in den Zellen erfassen wird. Dieses Protokoll verwendet eine 2 Minuten-Intervall.
- Überprüfen Zeitraffer-Set-up gelegentlich während der Aufnahme, um sicherzustellen, dass die Zellen im Fokus bleiben, keine Software-oder Hardware-Fehler, etc.
4. Image Processing der erworbenen Zeitraffer-Filme
- Offen für eine Filmdatei erworben Feld in einer Bildverarbeitungs-Software. HINWEIS: Fidschi wurde für die gesamte Verarbeitung in diesem Protokoll verwendet.
- Split Durchlicht-und Fluoreszenzbilder von einem Feld in zwei Stapeln von Bildern.
- Track eine einzelne Zelle in der Durchlichtkanal.
- Legen einen kreis ROI (Region von Interesse), die um die ausgewählte Zelle in dem ersten Rahmen in der übertragenen Lichtkanal. HINWEIS:.. Nur Zellen, die ein gesund sind am Ende der Aufnahme, zwei haben nicht bewegen außerhalb des Feldes, und 3 nicht in Kontakt mit anderen ce kommen.lls werden nachverfolgt.
- Speichern Sie eine ROI alle paar Frames an den ROI-Manager. Verfolgen Zelle, bis zum letzten Frame.
- Messen Intensität über die gespeicherten ROIs auf die Fluoreszenz-Kanal.
- Wählen Sie das Fluoreszenz-Stack und mit dem gespeicherten ROIs, verwenden Sie die benutzerdefinierte Kreis Interpolator-Plug-in-und Makro, um die Intensität des Raupen Zelle über die Zeit zu messen.
- Überprüfen Sie die Ausgabe Spur von der Makro. Wenn die Spur qualitativ erfasst Merkmale der Fluoreszenz Zeitraffer, exportieren Sie die Werte in eine Excel-Arbeitsblatt.
- Speichern Sie die ROI-Liste für die Zelle.
- Wiederholen Verfolgung in tragenen Kanal und Messen der Fluoreszenzkanal für andere Zellen in dem Feld.
- Wiederholen Sie alle Schritte für weitere Felder aus dem Experiment.
Ergebnisse
Dieses Protokoll produziert von lebensfähigen Kulturen, verteilt, einzelne PSM Zellen für Zeitraffer-Bildgebung der Fluoreszenzsignal (Abbildung 2). Unsere Transgen erzeugt einen Reporter, dessen Kreislauf von Produktion und Abbau erfolgt mit ähnlicher Dynamik zu dem endogenen Gen-und Protein im Embryo, in der Größenordnung von einer halben Stunde. Aufgrund seiner schnellen Umsatz, sollte die YFP-Signal in Einzelzellen schnell erkannt, um die Bleich zu minimieren und mit hoher Zeitauflösung, um die Funktionen der einzelnen Schwingungszyklus zu erfassen. Auch angesichts der relativen Dunkelheit des Signals, Kultur-und Erwerbsbedingungen sind sorgfältig abgestimmt, um empfindliche und robuste Ergebnisse zu gewährleisten. . 1 Ein ECM-Substrat für die Beschichtung der Glasbodenkulturschalen verwendet: Wir haben die folgenden Faktoren bei der Erzeugung optimal PSM Zellkulturen für die Bildgebung wichtig gefunden. 2. Zugabe von Serum auf die Präparation und Abbildungsmedium. 3. Präparation von PSM identifiziert Embryonen nach der Paarung hete genommenrozygous Paare, deren Nachkommen zwei Kopien des Transgens potenziell tragen. 4. Bilderfassung in einem optimalen Vergrößerungsbereich, mit einer höheren NA Ziel, effiziente Erfassung des Fluoreszenzsignals zu gewährleisten. 5. Beleuchtung mit einem Festkörperlichtquelle, um Intensitätsschwankungen, die Hintergrundgeräusche beitragen könnten, minimiert. 6. Signaldetektion mit einem hochempfindlichen EM-CCD-Kamera, um die Signalauslese maximieren.
Suboptimale Kulturen Zellen, die nicht in der gesamten Aufnahme bleiben Sie gesund gerundet und enthalten. Wir vermuten, dass unsere ECM-Substrat, ein Fragment von Zebrafisch fibronectin1 hält Zellen in einem undifferenzierten PSM-ähnlichen Zustand, im Vergleich zu anderen häufig verwendeten Substraten wie Poly-Lysin oder Laminin. Andere Substrate, die wir getestet verursacht Zellen auf dem Glas und der Verlust von Schwingungsfluoreszenzsignal im Verlauf der Aufzeichnung zu glätten. Wir fanden auch, dass die Zugabe von Serum in dem Medium während der Dissektion Streuung,und die Aufnahme war nicht nur wichtig, um das Trypsin für die Dissoziation verwendet zu stillen, sondern auch erhöhte Fluoreszenzintensität über Hintergrund, wahrscheinlich aufgrund der verbesserten Zelllebensfähigkeit. Um eine optimale Signalerfassung von einzelnen Zellen zu gewährleisten verwendeten wir eine 40x-Objektiv speziell für Fluoreszenz-Bildgebung (Zeiss-Plan NeoFluor Serie) entwickelt mit einer hohen NA. Wir fanden auch, dass ein Festkörper-Lichtquelle stabiler als herkömmliche Beleuchtung Quecksilberlampen, die kritisch zu Hintergrundschwankungen, die zu verrauschten Bildern beitragen minimieren. Diese Modifikationen sind wichtig, um robuste Ergebnisse zu gewährleisten.
Unter Verwendung dieses Protokolls erwarten wir Kulturen, in denen die Mehrzahl der Zellen in einem bestimmten Bereich an einem gewissen Punkt während der Aufzeichnung fluoreszierend. Wir finden, dass fluoreszierende Zellen typischerweise während der Aufnahmen bleiben gerundet und sind manchmal ziemlich beweglich. Einige Zellen, einschließlich fluoreszierenden Zellen können während der Aufnahme apoptotisch. Diese Zellen werden von jedem ein ausgeschlossennalyse. Wir Zellen, die in Kontakt mit anderen Zellen kommen auch ausschließen oder verschieben außerhalb des Sichtfeld. Im Durchschnitt sehen wir eine Reduktion von 12% der Zellzahl pro Halbbild bis zum Ende der 10 h nach Aufnahme durch Zählen der Anzahl von gesunden Zellen in der übertragenen Lichtkanal gemessen. Dieser Verlust beinhaltet sowohl Zelltod und Zellen, die aus dem Feld bewegt haben. Angesichts dieser Vorbehalte können wir im Durchschnitt, Track 5 fluoreszierenden Zellen pro Feld, das lebensfähig und sichtbar bleiben, und in der Regel erfassen 6 Felder pro Zustand. Zum Beispiel wird ein Experiment mit vier Bedingungen haben insgesamt 24 Feldern erworben und in der Regel etwa 30 verfolgt Zellen pro Zustand. Unter Standardaufnahmebedingungen mit L15-Medium mit 10% fötalem Rinderserum, finden wir, daß PSM-Zellen (n = 101 Zellen aus 4 Experimentwiederholungen) zwischen 2 und 7 Spitzen erzeugen, wobei der Mittelwert und Standardabweichung von 3 ± 1 Spitzen (2 -3 Zyklen). Die mittlere Spitzenzahl, sowie die 25%-Perzentil, 2 Spitzen und das 75%-Perzentil is drei Spitzen. Mit unserem semi-automatische Nachverfolgung und Analyse können wir schnell Fluoreszenzintensität über die Zeit zu generieren Spuren für einzelne PSM-Zellen mit einem in 2C gezeigt Vertreter Zelle Spur. Diese Ausgangs Spuren können dann verwendet werden, um quantitative Messungen der Eigenschaften der Schwingungs PSM Zellen, wie Frequenz, Amplitude, Anzahl der Zyklen und Zeitsteuerung von Spitzen herzustellen.
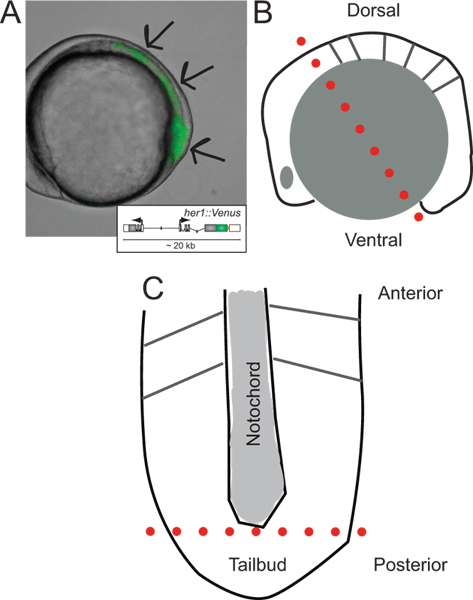
Abbildung 1. Identifizierung von transgenen Embryos und Schaltpläne der Dissektion. (A) Seitenansicht eines transgenen Zebrafischembryo YFP Fluoreszenz ausdrücken bei ~ 5-Somiten-Stadium. Bild ist eine Überlagerung von Durchlicht-und Fluoreszenzkanal. Pfeile zeigen Bereiche des Signals ausgehend von der Spitze der Schwanzknospen, während PSM Richtung l ast Somiten gebildet. Inset zeigt eine schematische Darstellung des Reporter-Transgen (für weitere Informationen über die Entwicklung und in-vivo-Verhalten finden Sie Soroldoni et al. 30. (B) Schematische Darstellung der Seitenansicht eines Embryos vor der ersten bei der Präparation geschnitten. Punktierte rote Linie zeigt den ersten Schnitt über die Hinterhirn gemacht, obwohl das Eigelb Zelle direkt hinter der Schwanzknospe. (C) Schematische Darstellung der abgeflachten PSM nach der Entfernung des Dotterkörnchen und Epidermis, entlang der anterior-posterioren Achse ausgerichtet. punktierte rote Linie zeigt den zweiten Schnitt, um die Spitze zu entfernen der Schwanzknospe für Zerstreuung und Kultur. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
07fig2.jpg "/>
Abbildung 2. Repräsentative Bilder und Spuren aus einer Einzelzelle in einem verteilten PSM Kultur. (A) Montage von Durchlichtbilder aus einer einzigen Zelle PSM in einem 6-h Zeitraffer-Aufnahme. Beachten Sie, dass die Zelle weiterhin gerundet und gesund im Verlauf der Aufzeichnung. (B) entsprechende Montage der Fluoreszenzbilder von einer einzelnen Zelle PSM. Intensitätsspitzen der Zelle im Verlauf der Aufzeichnung gezählt. (C) Durchschnittsintensität über die Zeit von einem ROI auf dieser Zelle angeordnet gemessen. Die Werte werden von jedem Bild aus einem Video mit einer Bildrate von einem pro 2 min mit dem Kreis Interpolator-Plug-in und benutzerdefinierte Makros in Fidschi geschrieben übernommen. Peaks in der Intensität sind wieder in der gesamten Spur nummeriert. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Diskussion
Um einen zellulären Prozess, der im Laufe der embryonalen Entwicklung stattfindet, zu studieren, Biologen verwenden typischerweise einen Ansatz im Kontext des gesamten Embryos. Jedoch zu verstehen, wie eine einzelne Zelle in einem Entwicklungszeitrahmen verhält, ist eine Methode, zu prüfen und zu stören einzelnen Zellen isoliert auch sehr nützlich. Durch die Erzeugung verteilt PSM Zellkulturen unter Verwendung von transgenen Zebrafischembryonen haben wir jetzt ein Werkzeug, um die Zelle autonome Natur des genetischen Schwingungen im Segmentierung Uhr in einer quantitativ direkt studieren. Es ist möglich, die Dynamik der Schwingungen im Fluoreszenzreporter in Hunderten von Zellen, die unter einer Vielzahl von Bedingungen zu messen.
Der Beobachtungszeitraum von Einzelzellen in Kultur ist länger als die Periode Somitogenese im intakten Embryo. Wir beobachten, dass eine intakte PSM Explantat in Kultur zeigt auch langsamer Schwingungen als das intakte Embryo (Daten nicht gezeigt), was darauf hindeutet that die in isolierten Zellen beobachtet längeren Zeitraum ist nicht einfach bei einem Schaden aus Zerstreuung. Eine variable Periode und Amplitude wird in den meisten unserer Single-Cell-Zeitreihen beobachtet. Die Quelle dieser Variabilität ist nicht bekannt, sollte aber wichtige Details über Tempo-Schaltungen machen die Segmentierung der Uhr zeigen.
Mit dieser Methode können wir die genetischen Komponenten der Segmentierung auf der zellulären Ebene, und Fragen, die herausfordernd sind, um in der ganzen Embryo untersuchen zu studieren. Beispielsweise durch kontrollierte Zugabe von bekannten Signalmoleküle in den sich entwickelnden Embryo in unseren Kulturen zu finden, können wir ihre Auswirkungen auf die einzelnen PSM zellulären Oszillators in einer robusten und reproduzierbaren Assay zu prüfen. Unsere PSM Kultursystem öffnet die Tür zu einer strengen Bewertung, welche Faktoren allein oder in Kombination zu fördern Schwingungen in diesen Zellen, welche Faktoren diese Schwingungen zu verhindern, und um das Zusammenspiel zwischen solchen Molekülen zu testen. Mit diesen Werkzeugen in der Hand, wollen wirbewerten bestehenden Modelle der Somitogenese veröffentlicht, die auf Gewebe-Level-Daten, sowie die Nutzung Ergebnisse in einzelnen Zellen basieren, um Vorhersagen, die in der ganzen Embryo getestet werden können, zu erzeugen.
Nach unserem Wissen ist dies der erste Zebrafisch primären Zellkultur-Protokoll für akute Zeitraffer-Aufnahme der Fluoreszenz in Einzelzellen; Weiterentwicklung und Verfeinerung dieses Protokolls ist kein Zweifel möglich ist. Andere Protokolle verwenden oft Zebrafischembryonen in stabilen Zelllinien, die mit Reporter transfiziert und für die Langzeitabbildungs 27-29 verwendet werden kann, zu erzeugen. Während stabile Linien sind nützlich für die Abbildung ein Prozess, der nicht für die Entwicklung Timing, wie der circadianen Uhr gebunden ist, ist die Frage der embryonalen Segmentierung erfordert sofortige Bildgebung während Zellen noch in den oszillierenden, Stammvater Staat. Sobald Zellen stoppen Schwing, nehmen sie eine differenzierte Zelle Schicksal, und als im Gewebe, würde sich in ein Somiten integriert werden. Es ist possible dass mit dem richtigen Faktor oder in vitro vorhanden Faktoren, die wir PSM-wie Zebrafisch-Zellkulturlinien, die Schwingungs bleiben würde, so dass deutlich mehr Zeitbeobachtungen, mehrere aufeinander folgende Störungen oder Hochdurchsatz-Screening erzeugen könnte.
Wir erwarten, dass diese Verfahren zur Herstellung von Primärkulturen von dispergierten Zellen für Zeitabbildungs ist gut geeignet für die Untersuchung von jedem zellulären Prozess, der in einer Entwicklungszeitrahmen, die nicht zugänglich Zebrafisch unter Verwendung stabiler Zelllinien auftritt. Die Isolierung der verschiedenen Zelltypen auf unterschiedliche Entwicklungsstadien aus den entsprechenden transgenen Linien Reporter, entweder über Dissektion oder über FACS nach embryonalen Dissoziation, könnte die Ausgangszellen zu liefern. Einige Optimierung der Kulturbedingungen, die embryonalen Ursprungs der Zellen geführt wird, kann erforderlich sein. Durch die Kombination dieser flexiblen und sensiblen Protokoll mit der schnell wachsenden Sammlung von transgenen ZebrafischLinien, hoffen wir, ein in vitro-Ansatz der Entwicklungsbiologie, die komplementär zu klassischen genetischen und embryologischen Methoden ist, zu erleichtern.
Offenlegungen
Autor Beiträge:
ABW entwickelt und verfeinert, die Verbreitung, Kultur-, Imaging-und Zell Tracking-Protokolle. DS erzeugt der transgenen Linien und überwachte die Gestaltung der Zeitraffer-Fluoreszenzmikroskop in diesem Protokoll verwendet. AO Pionier ersten Proof-of-principle Experimente zu distanzieren und Bild PSM-Zellen in der Kultur. JS schrieb den Kreis Interpolator Plugin-Tool verwendet werden, um in Fidschi Fluoreszenzintensität von Zeitraffer-Filmen messen. ABW und ACO schrieb das Manuskript.
Danksagungen
Diese Arbeit wurde durch ein EMBO-Langzeit-Stipendiums (ABW) unterstützt, ein National Science Foundation International Postdoctoral Research Fellowship (ABW), die Max-Planck-Gesellschaft (ABW, DS, JS, ACO), ein DIGS-BB-Stipendium (AO), und der Europäische Forschungsrat im Rahmen der Europäischen Gemeinschaften Siebten Rahmenprogramms STG-207634 (DS, ACO). Wir danken Ravi Desai für hilfreiche Kommentare zum Manuskript. Wir möchten auch die MPI-CBG-Protein-Expression Anlage für die Herstellung des Zebrafisch Fibronectin1 Fragment, dem Fisch den zuständigen Personen MPI-CBG für die Pflege und Wartung unserer Fisch-Linien und des MPI-CBG Lichtmikroskopie-Anlage für die Bildgebung Unterstützung danken.
Materialien
| Name | Company | Catalog Number | Comments |
| Epifluorescence microscope | Olympus | Model: SZX16 | |
| Epifluorescence microscope | X-cite Illumination | Series: 120Q | |
| Dissection microscope | Olympus | Model: SZX12 | |
| Fine forceps no. 55 | Fine Science Tools | 11295-51 | |
| Glass transfer pipettes | Assistent | 567/2 | |
| 35 mm plastic petri dishes | Greiner | 627102 | |
| 60 mm plastic petri dishes | Greiner | 628102 | |
| Sylgard polymer | SASCO | 266727 | |
| Manipulation tools | Made in-house | For description of manipulation tools Ref. 22 | |
| Microsurgical knife | World Precision Instruments | 500249 | |
| L15 medium | Invitrogen | 57322 | |
| Penicillin/streptomyocin | PAA | P11-010 | |
| Fetal bovine serum | Invitrogen | 257322 | |
| 0.05% trypsin / 0.02% EDTA | PAA | L11-004 | |
| Gel loading tips | Fisher Scientific | 253188 | |
| Sigmacote | Sigma Aldrich | 254589 | |
| Micropipette set | Gilson International | F167300 | |
| Zebrafish Fibronectin1 70 kD fragment | MPI-CBG protein facility | Generated in-house from construct based on previously published work Ref. 23-25 | |
| Glass bottom imaging dishes | Mattek (single well) | P35G-1.5-14-C | |
| Glass bottom imaging dishes | Greiner (CellView -multi-well) | 262502 | |
| E3 medium without methylene blue | Made in-house | From The Zebrafish Book, 5th Ed. Ref. 26 | |
| Plastic transfer pipettes | Ratiolab | 260011 | |
| Warner heating/cooling chamber | Warner Instruments | TC-324B/344B | |
| EM-CCD camera | Andor | Model: iXOn 888 | |
| Wide-field fluorescence microscope with Venus filter set | Zeiss | Model: Axiovert 200M | |
| Wide-field fluorescence microscope with Venus filter set | NeoFluor 40x, NA 0.75 | ||
| Wide-field fluorescence microscope with Venus filter set | Lumencor Light Engine | Model: Spectra X | |
| Wide-field fluorescence microscope with Venus filter set | BrightLine HC 575/15 | F39-575 |
Referenzen
- Cooke, J., Zeeman, E. C. A clock and wavefront model for control of the number of repeated structures during animal morphogenesis. J Theor Biol. 58, 455-476 (1976).
- Oates, A. C., Morelli, L. G., Ares, S. Patterning embryos with oscillations: structure, function and dynamics of the vertebrate segmentation clock. Development. 139, 625-639 (2012).
- Dequeant, M. L., Pourquie, O. Segmental patterning of the vertebrate embryonic axis. Nat Rev Genet. 9, 370-382 (2008).
- Oates, A. C., Gorfinkiel, N., Gonzalez-Gaitan, M., Heisenberg, C. P. Quantitative approaches in developmental biology. Nat Rev Genet. 10, 517-530 (2009).
- Oates, A. C., Ho, R. K. Hairy/E(spl)-related (Her) genes are central components of the segmentation oscillator and display redundancy with the Delta/Notch signaling pathway in the formation of anterior segmental boundaries in the zebrafish. Development. 129, 2929-2946 (2002).
- Lewis, J. Autoinhibition with transcriptional delay: a simple mechanism for the zebrafish somitogenesis oscillator. Curr Biol. 13, 1398-1408 (2003).
- Schroter, C., Oates, A. C. Segment Number and Axial Identity in a Segmentation Clock Period Mutant. Curr Biol. 20, 1254-1258 (2010).
- Riedel-Kruse, I. H., Muller, C., Oates, A. C. Synchrony dynamics during initiation, failure, and rescue of the segmentation clock. Science. 317, 1911-1915 (2007).
- Ozbudak, E. M., Lewis, J. Notch signalling synchronizes the zebrafish segmentation clock but is not needed to create somite boundaries. PLoS Genet. 4, e15 (2008).
- Herrgen, L., et al. Intercellular Coupling Regulates the Period of the Segmentation Clock. Curr Biol. 20, 1244-1253 (2010).
- Sawada, A., et al. Fgf/MAPK signalling is a crucial positional cue in somite boundary formation. Development. 128, 4873-4880 (2001).
- Giudicelli, F., Ozbudak, E. M., Wright, G. J., Lewis, J. Setting the tempo in development: an investigation of the zebrafish somite clock mechanism. PLoS Biol. 5, e150 (2007).
- Herrgen, L., Schroter, C., Bajard, L., Oates, A. C. Multiple embryo time-lapse imaging of zebrafish development. Methods Mol Biol. 546, 243-254 (2009).
- Hirata, H., et al. Oscillatory expression of the bHLH factor Hes1 regulated by a negative feedback loop. Science. 298, 840-843 (2002).
- Maroto, M., Dale, J. K., Dequeant, M. L., Petit, A. C., Pourquie, O. Synchronised cycling gene oscillations in presomitic mesoderm cells require cell-cell contact. Int J Dev Biol. 49, 309-315 (2005).
- Reppert, S. M., Weaver, D. R. Coordination of circadian timing in mammals. Nature. 418, 935-941 (2002).
- Welsh, D. K., Imaizumi, T., Kay, S. A. Real-time reporting of circadian-regulated gene expression by luciferase imaging in plants and mammalian cells. Methods Enzymol. 393, 269-288 (2005).
- Soroldoni, D., Oates, A. C. Live transgenic reporters of the vertebrate embryo's Segmentation Clock. Curr Opin Genet Dev. 21, 600-605 (2011).
- Masamizu, Y., et al. Real-time imaging of the somite segmentation clock: revelation of unstable oscillators in the individual presomitic mesoderm cells. Proc Natl Acad Sci USA. 103, 1313-1318 (2006).
- Soroldoni, D., Hogan, B. M., Oates, A. C. Simple and efficient transgenesis with meganuclease constructs in zebrafish. Methods in molecular biology. 546, 117-130 (2009).
- Schroter, C., et al. Dynamics of zebrafish somitogenesis. Dev Dyn. 237, 545-553 (2008).
- Picker, A., Roellig, D., Pourquie, O., Oates, A. C., Brand, M. Tissue micromanipulation in zebrafish embryos. Methods in molecular biology. 546, 153-172 (2009).
- Mould, A. P., et al. Identification of multiple integrin beta1 homologs in zebrafish (Danio rerio). BMC Cell Biol. 7, 24 (2006).
- Mould, A. P., Koper, E. J., Byron, A., Zahn, G., Humphries, M. J. Mapping the ligand-binding pocket of integrin alpha5beta1 using a gain-of-function approach. Biochem J. 424, 179-189 (2009).
- Zhao, Q., Liu, X., Collodi, P. Identification and characterization of a novel fibronectin in zebrafish. Exp Cell Res. 268, 211-219 (2001).
- Westerfield, M. In The zebrafish book : a guide for the laboratory use of zebrafish (Brachydanio rerio). , (1993).
- Vallone, D., Santoriello, C., Gondi, S. B., Foulkes, N. S. Basic protocols for zebrafish cell lines: maintenance and transfection. Methods Mol Biol. 362, 429-441 (2007).
- Carr, A. J., Whitmore, D. Imaging of single light-responsive clock cells reveals fluctuating free-running periods. Nat Cell Biol. 7, 319-321 (2005).
- Whitmore, D., Foulkes, N. S., Sassone-Corsi, P. Light acts directly on organs and cells in culture to set the vertebrate circadian clock. Nature. 404, 87-91 (2000).
- Soroldoni, D., et al. A Doppler effect in embryonic pattern formation. Science. 345, 222-225 (1126).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten