Method Article
Hochdurchsatz-, Automated Extraktion von DNA und RNA aus klinischen Proben mit TruTip Technologie Gemeinsames Liquid Handling Robots
In diesem Artikel
Zusammenfassung
TruTip ist eine einfache Extraktion von Nukleinsäuren Technologie, bei der eine poröse, monolithische Bindematrix in eine Pipettenspitze eingesetzt ist. Folglich ist die Probenvorbereitung Format kompatibel mit den meisten Liquid Handling Geräte, und kann für viele mittel-bis High-Throughput-klinische Anwendungen und Probe-Typen verwendet werden.
Zusammenfassung
TruTip ist eine einfache Extraktion von Nukleinsäuren Technologie, bei der eine poröse, monolithische Bindematrix in eine Pipettenspitze eingesetzt ist. Die Geometrie des Monolithen können nach bestimmten Pipettenspitzen Bereich im Volumen von 1,0 bis 5,0 ml angepasst werden. Die große Porosität des Monolithen ermöglicht viskosen oder komplexen Proben zu leicht durch sie hindurch mit minimalem Fluidik Gegendruck. Bi-direktionale Flow maximiert Verweilzeit zwischen dem Monolith und Probe und ermöglicht große Probenvolumina innerhalb eines einzigen TruTip verarbeitet werden. Die grundlegenden Schritte, unabhängig von Probenvolumen oder TruTip Geometrie gehören Zell-Lyse, Nukleinsäure-Bindung an den inneren Poren des TruTip Monolith, Waschen entfernt ungebundenen Bestandteile der Probe und Lysepuffern und Eluieren gereinigt und konzentriert Nukleinsäuren in einem geeigneten Puffer. Die Attribute und Anpassungsfähigkeit der TruTip sind in drei automatischen klinischen Probe Verarbeitung Protokolle unter Verwendung einer Eppendorf epMotion 5070 gezeigt, Hamilton STAR und STARPLUS Liquid-Handling-Roboter, einschließlich RNA-Isolation aus Nasopharynx absaugen, genomische DNA-Isolierung aus Vollblut und fetalen DNA-Extraktion und Anreicherung von großen Mengen im mütterlichen Plasma (jeweils).
Einleitung
Nukleinsäure-Aufreinigung notwendig ist für die meisten molekularen Diagnostik, Forschung nur zu verwenden, und Life-Science-Anwendungen. Verschiedene Ansätze wurden seit der Zeit der Phenol / Chloroform Extraktionen entstanden, von denen viele auf dem grundlegenden Prinzip der Nukleinsäure-Bindung an Silica in der Gegenwart chaotroper Salze 1 basiert. Die Extraktion wurde gestrafft und automatisiert durch den Einsatz von verschiedenen Perlen-und Membran-basierte Formate, mit Spin Filter, magnetische Kügelchen und verwandte Ansätze dominieren die Life Sciences-Industrie (siehe Beispiele in 2-13). Während wirksam, haben Partikel und Membranen Einschränkungen bekannt, wann mit herausfordernden klinischen Matrizen konfrontiert. Zum Beispiel, Membranen und Kügelchen Säulen auf-konform sind, haben kleine Porengrößen (also hohen Gegendrücken) und erfordern irgendeine Art von Unterstützung, um durch eine Zentrifuge oder Vakuum verarbeitet werden. Die physikalischen Eigenschaften von Membranen und Kügelchen Spalten einesignifikante Strömungswiderstand, der die Art der Proben, die effizient, ohne zu verstopfen die Verbrauchsmaterialien und / oder die Gesamtmenge (Eingang) Probenvolumen, die uni-direktional verarbeitet durch den Strömungsweg können verarbeitet werden begrenzt. Umgekehrt muß magnetischen Teilchen in der Probe durch Rühren verteilt. Die Notwendigkeit, die homogen verteilen magnetischen Teilchen in einer Lösung schränkt die insgesamt eingesetzten Probenmenge, die mit vielen magnetischen Kügelchen Verbrauchsmaterialien verarbeitet werden können. Klinische Probe Attribute (wie Viskosität oder Komplexität) zu ineffizienten Magnetpartikel-Konzentration auf der Seite eines Rohres oder Stabes führen. Darüber hinaus können Silica Feinstaub brechen der Perlen während der Extraktion, verlieren ihre Magnetisierung und verunreinigen das letzte Probe.
Die TruTip Technologie wurde entwickelt, um einige dieser Nukleinsäure-Probe Verarbeitung Auflagen und Beschränkungen 14 zu überwinden. Durch die Einbettung eines porösen Monolithen innerhalb einerPipettenspitze, Fluidik Gegendruck abgesenkt, die Ablaufsteuerung ermöglicht durch Vakuum (dh Pipette Aspiration). Diese Funktion ermöglicht die Extraktion und Instrumentierung benötigt, um Nukleinsäuren aus schwierigen Probe Typen zu reinigen, um stark vereinfacht werden (Abbildung 1). Die Geometrie und die Porosität des Monolithen wird hergestellt, um ein Verstopfen zu minimieren, während die Dicke des Monolithen ausreichende Nukleinsäure Bindungskapazität für Probenvolumina im Bereich von 1,0 bis 5,0 ml. Durchfluss in beide Richtungen während Probe Aufnahme und Abgabe ermöglicht längere Verweilzeiten zwischen der Probenlösung und der Monolith für eine effiziente Bindung Nukleinsäuregewinnung und Elution und ermöglicht relativ große Probenvolumina verarbeitet, dass mehr als die Volumenkapazität der Pipettenspitze selbst sein. Wir bereits berichtet die Entwicklung und Anwendung eines manuellen TruTip Verfahren zur Reinigung von Influenza-RNA aus Nasen-Rachen-Proben unter Verwendung eines Single-oder Multi-Channel-Rainin Pipette 15. Equivalent Extraktionseffizienzen wurden zwischen automatisierten und manuellen QIAcube TruTip Methoden bei 10 6 Genkopien Influenza A pro ml Nasen-Rachen absaugen erhalten. Das Ziel dieser Studie war es, mittel-und High-Throughput-, automatisierte TruTip Nukleinsäurereinigung Verfahren zur Nasen-Rachen absaugen (NPA) und anderen klinisch relevanten Probenarten mit Liquid-Handling-Roboter häufig in Bezug klinischen Laboratorien gefunden zu demonstrieren.
Protokoll
Drei automatisierte Extraktion TruTip Protokolle werden hier beschrieben und gezeigt: 1) mit mittlerem Durchsatz RNA Extraktion aus NPA auf einem Eppendorf epMotion 5070, 2) mit hohem Durchsatz genomischen DNA-Extraktion aus geringen Mengen von Vollblut auf der Hamilton STAR und 3) selektive Isolierung und Anreicherung der fetalen DNA fragmentiert von großen Mengen im mütterlichen Plasma auf der Hamilton STARPLUS. Automatisierte Skripte sind von Akonni verfügbar und nur High-Level-Programm Deskriptoren finden Sie hier. Die Extraktion und Elution Reagenzien werden automatisch aus Schüttgut Reagenz Mulden bis zu 96-Well-Platten durch das automatisierte Script (n) abgegeben, bevor die klinische Proben verarbeitet werden.
1. Automatisierte RNA Extraktion aus Nasopharyngeal Aspirate
Die zuvor beschriebene manuelle Methode TruTip 15 ist jetzt angepasst, um auf einem Eppendorf epMotion 5070 Liquid Handling-Roboter laufen, mit einem großen Poren TruTip Matrix in 1.0 ml Eppendorf Rohr eingebettettte Tipps, a 2 ml Deep-Well-Platte (USA Scientific), Akonni TruTip Extraktionsreagenzien und NPA als Sample-Typ. Die epMotion 5070 Liquid Handling Roboter hält bis zu 8 Spitzen gleichzeitig, so dass eine automatisierte Baseline-Protokoll ist für 8 parallele Extraktionen beschrieben. Jedoch können bis zu 24 Proben in einem einzigen Programm in einer Tief-Well-96-Probenplatte verarbeitet werden. Eine separate epMotion Programm ist verfügbar (und erforderlich), um 16 oder 24 Proben verarbeiten. Das Protokoll unten beschrieben ist für einen 8 Probe automatisiertes Skript.
Setup-
- Bringen Nasen-Rachen-Proben auf RT vor Beginn der Extraktion.
- Je 100 ul Nasen-Rachen absaugen plus 150 ul Nuklease-freies Wasser in die Säule 1 der Probe Platte (Abbildung 2A).
- Legen Sie die Probe Platte in Position B1 auf der epMotion Arbeitstisch (2B).
- Platz Pipettenspitzen TruTips und 30 ml Reagenz Mulden auf ihre jeweiligen epMotion Worktable Positionen (Abbildung 2B).
- Öffnen Sie die Eppendorf epBlue Software, wählen Sie die Datei, indem Akonni Run für 8 Proben zur Verfügung gestellt, und laden Sie die Methode, indem Sie die RUN-Taste auf der Registerkarte RUN.
- Unter Level Sensor-Einstellungen, wählen Levels und Tipps, und klicken Sie auf die Schaltfläche Ausführen.
- Geben Sie die Probenmenge in die Software und klicken Sie auf RUN.
- Die epMotion Skript wird den Benutzer auffordern, Extraktion und Elution Reagenzien zu den Reagenziengefäßen an Position B2 auf dem Arbeitstisch liegt hinzuzufügen. Fügen Sie die empfohlenen Mengen von jedem Reagenz zur jeweiligen Trog. Für 8 Proben, sind die minimalen Reagenzienvolumina:
| Reagenz | Volumen (ml) | Trough Position |
| 95% Ethanol | 3.5 | 2 |
| Waschpuffer D | 9.0 | 3 |
| Waschpuffer E | 9.0 | 4 |
| Elution Buffer A2 | 1.3 | 5 |
| Lyse und Binding Buffer D | 11.0 | 6 |
- Eingang der Reagenzienvolumina in der Tabelle durch die epMotion Software während der Eingabeaufforderung aus Schritt 8 oben dargestellt. Der Eingangswert sollte die tatsächliche Volumen des Puffers durch den Benutzer in jedem entsprechenden Reagenzbehälter abgegeben wird, und muss größer als oder gleich der Mindestmengen oben erwähnt. Falsche Einträge Volumen könnte in falsche Volumina durch die epMotion Hardware in jedes Röhrchen oder auch geliefert in der 96-Well-Platte (n) führen.
Automatisiertes Programm
- Wählen Sie RUN, um die automatisierte Methode zu starten. Die automatisierte Skript wird durch die folgenden Schritte ohne Eingreifen des Benutzers zu verschieben:
Probenlyse und Reagenz Distribution:- Dosieren 375 ul Lysis Buffer D in Spalte 1 und mischen Sie für 10Zyklen (apirate + verzichten = 1 Zyklus). Dieser Schritt beginnt die Lyse Inkubation Prozess, während die übrigen Reagenzien aliquotiert werden.
- Dosieren 1,6 ml Wash Buffer D in Spalte 2.
- Dosieren 1,6 ml Waschpuffer E in Spalte 3.
- Geben Sie 50 ul Elutionspuffer A in Spalte 4 wählen.
- Pause für 6 min, um die 10 min Probeninkubation in Lysispuffer vervollständigen D.
- In 375 ul Ethanol zu jeder Vertiefung in Spalte 1, Mischen jeder Probe mit Ethanol bis 10 Pipettierzyklen.
- Last 8 TruTips von Position A2 auf dem Arbeitstisch, und beginnen die Extraktion in Abbildung 1 dargestellt.
- Aspirieren und Abgeben sample / Lysepuffer / Ethanol-Gemisch von Spalte 1 der Probenplatte für sieben Zyklen, um die Nukleinsäure an die TruTip Monolith binden. Obwohl Probe durch den TruTip können variieren (aufgrund der Unterschiede in der klinischen Probe Viskosität), war Nukleinsäure Ausbeute für diese Probe nichtt durch Streumengenänderung betroffen. Optionen zur Verbesserung der Probendurchsatz in der Diskussion beschrieben.
- Bewegen TruTips zu Probenteller Spalte 2 und Zyklus Wash Buffer D 5x über den Monolithen, um restliche Lysepuffer und Probenmatrix entfernen.
- Bewegen TruTips zu Probenplatte Spalte 3 und Zyklus Waschpuffer E 5x über den Monolithen, um Proteine und andere Verunreinigungen von der gebundenen Nukleinsäure zu entfernen.
- Bewegen TruTips dem leeren Reagenzreservoir Position 1 (in Arbeitstisch Lage B2) und Zyklus 80x (mit einer schnellen Durchfluss), um den Monolith trocknen. Es ist wichtig, gründlich trocknen TruTip als Restlösemittel in eluierten Nukleinsäure-Präparationen negativ beeinflussen Enzyme wie Reverse Transkriptase und Taq DNA-Polymerase.
- Bewegen TruTips zu Probenteller Spalte 4 und Zyklus 5x in Elutionspuffer A. Das extrahierte und gereinigte Nukleinsäure ist nun in Elutionspuffer in Probenteller Spalte 4 Brunnen.
- Eject TruTips in die epMotion Abfalleimer.
- Wenn das Programm fertig ist, entfernen Sie manuell die Probe Platte aus dem Gerät und übertragen Sie die gereinigte Nukleinsäure, neue Rohre für die langfristige Lagerung oder die weitere Verwendung. Erweiterte epMotion Benutzer können Anweisungen an den Run-Datei in den eluierten Proben in getrennten Lagerung Rohre oder PCR-Platten übertragen, falls gewünscht.
Das Programm für die insgesamt 16 Proben Schritte 10.1 bis 10.13 wiederholen mit Probenteller Spalten 5-8. Für die 24-sample-Programm Schritte 10.1 bis 10.13 wiederholt werden 2x mit Probenteller Spalten 5-8 und 9-12, beziehungsweise.
2. 96-well Genomic DNA Extraktion aus Vollblut
Ein Hamilton STAR Liquid-Handling-Roboter wird zur automatisierten Extraktion von 96 Proben gleichzeitig demonstrieren aus Vollblut. Die Hamilton STAR unterscheidet sich von der epMotion System, dass eine optionale Heizung / Schüttler Einheit ist auf dem Deck, was wichtig ist für die enzymatische Verdauung von bestimmten klinischen verfügbarcal Matrices, wie Vollblut. Da das System mit einem 96-Kanal-Pipette Kopf angebracht werden kann, ist eine separate 96-Well-Platte für jedes der TruTip Schritte und Reagenzien.
Setup-
- Schalten Sie den STAR Instrument und Computer.
- Öffnen Sie die Hamilton Run-Control-Software.
- Öffnen Sie die Datei, indem Akonni Run für 96 Proben.
- Platz Laborgeräte auf die STAR Deck wie in Abbildung 3 dargestellt.
- Dosieren Reagenzien in die entsprechenden Mulden (Volumen bezeichnen die mindestens erforderlich, um 96 Proben verarbeiten):
| Reagenz | Volumen (ml) | Trough Position |
| Lyse und Binding Buffer F | 75 | 5 |
| 95% Ethanol | 100 | 6 |
| Waschpuffer J | 175 | 7 |
| WaschenBuffer K | 175 | 8 |
| Elution Buffer A2 | 12 | 9 |
| Proteinase K (20 mg ml -1) | 8 | 15 |
6. Die Proben auf RT Gleichgewicht.
7. Legen Sie die Probenröhrchen in dem Probenträger Racks (deck Position 4 in Abbildung 3). Ort Probe 1 in der Rückseite der hinteren linken Träger und bewegen sich nacheinander jeden Träger mit der Probe 96 Ende in der vorderen rechten Position.
Automatisiertes Programm
- Wählen Sie die PLAY-Taste in der oberen linken Ecke des Run-Datei Fenster und geben Sie die Anzahl der Proben bearbeitet. Die automatisierte Skript wird dann durch die folgenden Schritte ohne Eingreifen des Benutzers zu verschieben:
Probenlyse und Reagenz Aufteilung- Übertragen Sie 200 ul von jeder Probe Rohr mit dem Inkubationsplatte an Position 14 auf der erasser / Rüttel-Modul (Abbildung 3).
- Dosieren 80 ul Proteinase K in jeder Probe auch der Inkubationszeit Platte.
- Dosieren 600 ul Lysis Buffer F in jeder Vertiefung der Inkubationsplatte.
- Mischen Sie die Lösung für 10 Zyklen, und dann Inkubation für 20 min bei 70 ° C und 500 Umdrehungen pro Minute. Die 70 ° C eingestellten Temperatur ergibt sich eine ~ 60 ° C Temperatur der Probe innerhalb des tiefen Well-Platte, die im Bereich von Proteinase-K-Aktivität ist. Während die Proben Inkubation werden, setzt die Liquid-Handling-System, das durch den Verzicht auf Reagenzien in die entsprechenden Platten und Brunnen:
- 100 ul Elutionspuffer A in jede Vertiefung der Deepwellplatte an Position 13.
- 800 ul Ethanol in jeder Vertiefung der Deepwellplatte an Position 10.
- 1,6 ml Wash Buffer K in jede Vertiefung der Deepwellplatte an Position 12.
- 1,6 ml Waschpuffer J in jede Vertiefung der Deepwellplatte an Position 11.
- Nach 20 min Inkubation wird die Probe aus dem Gemisch Inkubationsschale zur Deepwellplatte an Position 10 überführt und gemischt bis 12 Pipettierzyklen.
- Eject Reagenz Tipps in den Papierkorb.
Gewinnung
Dieser Teil des gDNA Blut Verfahren ist sehr ähnlich zu dem epMotion Influenza-Protokoll, mit Ausnahme der Zusammensetzung der Wasch-Reagenzien und Taktzahlen. Die Hamilton TruTips Spitzen sind schwarz, so dass die Strömung von Flüssigkeiten durch die TruTip ist nicht sichtbar.
- Legen Sie 96 TruTips von Deck 3-Stellung.
- Aspirieren und Abgeben der Probe / Lysepuffer / Ethanol-Gemisch in der Position 10 für 10 Zyklen Nukleinsäuren an die TruTip Monolith binden.
- Bewegen TruTips bis 11 und Zyklus Wash Buffer J 5x über den Monolithen zu positionieren, um das restliche Lysepuffer und Probenmatrix entfernen.
- Bewegen TruTips bis 12 und Zyklus Waschpuffer K 5x, Proteine und andere Verunreinigungen von der gebundenen Nukleinsäure Entnahmestellung.
- Schalten Sie die TruTip 40x mit hoher Geschwindigkeit an der Luft trocknen.
- Bewegen TruTips auf Position 13 und Zyklus 5x in Elutionspuffer A2. Das extrahierte und gereinigte Nukleinsäure befindet sich nun im Elutionspuffer im tiefen Well-Platte.
- Eject TruTips in den Abfalleimer.
Wenn das Programm fertig ist, entfernen Sie die Elution Plate aus dem Instrument und übertragen die extrahierten Proben in die entsprechenden Rohre für die Lagerung oder nachgelagerte Anwendungen.
3. DNA-Extraktion aus Large Volume Plasmaproben
Die Hamilton STARPLUS Instrument wird verwendet, um eine automatische Protokoll zur Extraktion frei zirkulierende fötale DNA aus 5 ml mütterlichen Plasma zeigen. Die STARPLUS System unterstützt zwei automatisierte Pipettenkanal Arme, eine mit 8 x 5 ml-Kanäle und einen mit 8 x 1 ml Kanäle. Diese Arme können parallel für gestaffelte Verarbeitung arbeiten in Chargen von je 8 Proben. Eine 5 ml TruTip ist für die anfängliche l verwendetarge-Volumen-Extraktion und eine 1 ml TruTip zur Größe Trennung und weitere Konzentration der extrahierten Nukleinsäure verwendet.
Set-up
- Schalten Sie den STARPLUS Instrument und Computer.
- Öffnen Sie die Hamilton Run-Control-Software.
- Öffnen Sie die Datei, indem Akonni Run für 8 großvolumigen Plasma-Proben zur Verfügung gestellt.
- Platz Laborgeräte auf die STARPLUS Deck wie in der Abbildung 4 dargestellt.
- Dosieren Reagenzien in die entsprechenden Behälter:
| Reagenz | Volumen (ml) | Trough Position |
| CN-W1 | 17. | 5A |
| CN-W2 | 17. | 5B |
| CN-W4 | 21. | 5C |
| Proteinase K (20 mg ml -1) | 5 | 6A |
| EBB | 17. | 6B |
| EBA2 | 5 | 6C |
| CN-W3 | 5 | 6D |
| CN-B2 | 5 | 6E |
| CN-B3 | 5 | 6F |
| CN-L1 | 52 | 7 |
| CN-B1 | 175 | 8 |
- Erlauben Probe auf RT Gleichgewicht.
- Legen Sie die Probenröhrchen in dem Probenträger Racks (deck Position 3 in Abbildung 4). Probe 1 Platz im Fond und bewegen nacheinander in Richtung der Vorderseite des Decks.
Automatisiertes Programm
Wegen der großen Eingang Probenvolumen muß Lyse und Homogenisierung Schritte von der Hamilton STARPLUS Instrument durchgeführt werden in einem Wasserbad. Steps Benutzereingriff im automatisierten Protokoll sind mit einem Sternchen (*) am beginnin angegebeng des Satzes, und fett gedruckt.
Probenlyse und Reagenz Distribution: Die Probe wird mit Proteinase K und Lyse-Puffer, um die Probe zu homogenisieren und lassen Sie die DNA.
- Wählen Sie die PLAY-Taste in der oberen linken Ecke des Run-Datei Fenster. Geben Sie die Anzahl der Proben verarbeitet werden, den Standort von Pipettenspitzen auf dem Deck, und die Lage der TruTips auf dem Deck.
- Die automatisierte Skript fügt 615 ul Proteinase K, 5 ml Plasma und 6,2 ml Lysispuffer CN-L1 zu je 50 ml konischen Röhrchen, und dann PAUSE.
- * Entfernen Sie die 50 ml konische Röhrchen aus der Probe Deck, Vortex für 30 Sekunden bei hoher Geschwindigkeit, und Inkubation off-line für 30 min bei 60 ° C im Wasserbad oder Heizblock. Nachdem die konische Röhrchen aus dem Hamilton-Deck entfernt werden, setzen Sie das automatisiertes Skript weiterhin Abgabe Reagenzien in ihren jeweiligen Platten und Brunnen (4B und 4C):
- 2 ml CN-W1 zu jedem anderen gut in Position 9 Spalte 1.
- 2 ml CN-W2 zu jedem anderen gut in Position 9 Spalte 2.
- 2 ml CN-W4 zu jedem anderen gut in Position 9 Spalte 3.
- 250 ul EBA2 zu jedem anderen und in Position 9 Spalten 4 und 5.
- 495 ul CN-B2 zu jedem anderen gut in Position 10 Spalte 1.
- 1 ml EBB zu jedem anderen gut in Position 10 Spalte 2.
- 500 ul CN-W3 zu jedem anderen gut in Position 10 Spalte 3.
- 500 ul CN-W4 zu jedem anderen gut in Position 10 Spalte 4 wählen.
- 50 ul EBA2 zu jedem anderen gut in Position 10 Spalte 5.
Da die 5 ml Kanäle sind zu breit, um benachbarten Vertiefungen für jede Probe, die automatisiertes Programm verzichtet Reagenzien in jedem anderen auch der tiefen Well-Platte in Position Deck 9 zu verwenden. Der mechanische Arm für die Kanäle 1 ml verwendet erfordert auch den Einsatz von jedem anderen auch in Reagenz Platten für die Ausgrenzung und Konzentration steps des Protokolls.
Nach Abgabe Reagenzien, wird das Programm PAUSE.
- * Nach der 30 min, 60 ° C Inkubation, legen Sie die 50 ml konischen Röhrchen auf Eis für 5 min.
- * Bringen Sie die 50 ml konische Röhrchen in ihre ursprüngliche Position innerhalb der Probe Gepäckträger an Deck Position 4, und der Wiederaufnahme des automatisierten Skript.
- In 12 ml Binding Buffer CN-B1 zu jeder Probe und mischen Rohr 10x.
Large Volume Extraction: 5 ml TruTips werden zum Extrahieren von Gesamt-DNA aus dem Plasma lysierten Probe verwendet.
- Pick-up 5 ml TruTips von Position 2 für die großvolumige Nukleinsäure-Extraktion.
- Zyklus die Probe mixture15 Mal von 50 ml konischen Röhrchen, beginnend an der Unterseite des Rohres und Bewegen 3 mm höher nach jedem Pipettierzyklus. Dieser Schritt verbindet die gesamte Nukleinsäure in das TruTip Monolith.
- Bewegen TruTips den Deep Well Platten in Position 6 Spalte 1 und cycle 1x in Wash Buffer CN-W1.
- Bewegen TruTips bis 9 Spalte 2 und 1x Zyklus in Wash CN-W2 positionieren.
- Bewegen TruTips bis 9 Spalte 3 und Zyklus 2x in Wash CN-W4 positionieren.
- Bewegen TruTips bis 9 Spalte 4 und Zyklus 40x bei hoher Geschwindigkeit zu positionieren Bindungsmatrix trocknen.
- Bewegen TruTips bis 9 Spalte 5 und 10x Zyklus, um die gebundenen Nukleinsäuren von der 5 ml TruTips eluieren positionieren. Dies ist großvolumigen Elution Nr. 1.
- Bewegen TruTips bis Spalte 6, und wiederholen Sie den Schritt mit der zweiten Teilmenge von Elutionspuffer. Dies ist großvolumigen Elution # 2.
- Übertragen Elution Nr. 2 in Position 9 Spalte 5, um es mit Elution Nr. 1 zu kombinieren, und entsorgen Sie die TruTips.
Ausschluss und Konzentration: Die DNA mit hohem Molekulargewicht ausgewählt ist aus der extrahierten Probe entfernt, und die restlichen DNA wird isoliert und konzentriert.
- In Kombination Elutionsmittel von Schritt 22 bis 10 Spalte 1 zu positionieren und gründlich mischen 10x.
- Pick up 1 ml Trutips von Position 13 und Zyklus 20x, die DNA mit hohem Molekulargewicht zu dem Monolithen zu binden.
- Bewegen Sie die TruTips bis 10 Spalte 2 und Zyklus 5x positionieren, um die Spitze spülen und entfernen DNA mit hohem Molekulargewicht. Die TruTips zurückgehalten werden und zurück in der Spitze Rack auf Position 13 platziert.
- Mit Reagenz Tipps von Position 12, fügen Sie 575 ul Binding Buffer CN-B3 auf die Probe in Position 10 Spalte 1 und Mix 10x.
- Pick-up die TruTips von Schritt 25 zurück auf 10 Spalte 1 und Zyklus 20x um die verbleibende DNA aus der Probe binden an die 1 ml TruTip positionieren.
- Bewegen TruTips bis 10 Spalte 4 und Zyklus 1x Wash in CN-W3 positionieren, um alle verbleibenden Inhibitoren zu entfernen.
- Bewegen Sie die TruTips bis 10 Spalte 5 und 1x Zyklus in Wash CN-W4 positionieren, um restliche Salze aus CN-W3 spülen.
- Heben TruTips über Position 10 Spalte 5 und Zyklus Luft durch die Spitzen 35x den Monolithen trocknen.
- Bewegen TruTips bis 10 Spalte 6 und Zyklus 10x in EBA2 positionieren, um das gereinigte, Größe-Auswahl eluierenTed und konzentrierte Nukleinsäure.
- Verwerfen TruTips.
- Übertragen Sie die eluierte Probe aus Spalte 6 in 1,5 ml Röhrchen in Position 11. Extrahiert Proben sind bereit für die Lagerung oder Weiterverarbeitung.
Ergebnisse
Echtzeit-PCR-Daten für Influenza RNA Extraktion aus NPA sind in 5A gezeigt. Geschätzte Nukleinsäure-Extraktion Effizienz ist vergleichbar zu den Ergebnissen mit der manuellen Version des Protokolls 15 erhalten. Eine lineare Abhängigkeit der durchschnittlichen C t-Werte beobachtet wird zwischen 10 4 und 10 6 ml -1 Genkopien Influenza geändert (R 2 = 0,99 und 0,98 für Influenza A und B bezeichnet), mit Standardabweichungen der durchschnittlichen C T-Werte von weniger als 1 Zyklus. Das Fehlen einer Kreuzkontamination mit der epMotion System ist in Abbildung 5B, wo 12 positive Proben, die NPA 10 6 ml -1 Genkopien NPA mit 12 Puffer Rohlinge durchsetzt demonstriert. Die durchschnittliche C t für die positiven Kontrollen war 30,16 ± 0,14, und alle Puffer Rohlinge waren negativ. Die gesamte Verarbeitung Probe beträgt 16, 28 und 40 min für 8, 16 und 24 Proben.Da eine typische klinische Nasen-Rachen absaugen oder Tupfer enthält 10 7 Genkopien ml -1 (vorausgesetzt 1000 Virionen pro KID 50 16 und> 10 4 TCID 50 ml -1 Influenza 17), wird die automatisierte epMotion Protokoll voraussichtlich auf eine Mehrheit wirksam von klinischen Proben NPA.
Da der Bereich der molekularen Tests auf humane genomische DNA durchgeführt wird, ist das primäre Ziel der Nukleinsäure-Extraktion aus Vollblut in schadstofffreier, hochmolekulare genomische DNA zu erzeugen. Die automatisierte Protokoll für 96 Proben innerhalb 1 h, was eine Verbesserung gegenüber anderen automatisierten Systemen (zB Promega MagneSil = 90 min und Qiagen QIAamp DNA Blood BioRobot MDx System = 2,5 h). 6A zeigt die UV / Vis-Absorption Profile für abgeschlossen 45 positive Blutproben gleichzeitig mit 45 Reagenzleerwerte auf der Hamilton STAR-Protokoll verarbeitet, mit einer durchschnittlichen A 260/280 Verhältnis von 1,96 und die durchschnittliche A 260/230 Verhältnis von 1,93. Ein A 260/280 Verhältnis zwischen 1,7-2,0 und A 260/230 Verhältnis> 1,7 sind in der Regel sehr bezeichnend für reine DNA, frei von restlichem Salze, Proteine oder Lösungsmittel, und akzeptabel für die meisten nachgeschalteten molekularen Anwendungen. Die 1%-igen Agarosegel in 6B zeigt, dass die resultierende gDNA mit hohem Molekulargewicht (> 24 kb) mit minimaler Scherung. Die durchschnittliche Nukleinsäure Ausbeute aus dem vollen Satz von 45 positiven Proben beträgt 5,26 ± 0,46 ug menschlichen DNA pro 200 ul Vollblut, basierend auf dem Life Technologies Quantifiler Menschliche DNA Quantifizierung Kit. Kreuzkontamination Studien ähnlich wie in 5B gezeigt sind, wurden mit Puffer Negativkontrolle Rohlinge mit der positiven Proben vermischt und extrahiert parallel durchgeführt, alle Rohlinge wurden erneut negativ durch PCR (nicht gezeigt). Das gereinigte gDNA eignet sich auch für zahlreiche andere PCR-Analysen (nicht gezeigt).
Ergebnisse in Echtzeit aus acht Replikatproben einer gepoolten mütterlichen Plasma Probe mit dem großvolumigen TruTip Verfahren verarbeitet werden in 7 gezeigt. Der vollständige Protokoll (einschließlich off-line Proteinase K Inkubation) wird in etwa 2,5 Stunden, ähnlich dem Qiagen Handbuch zirkulierenden Nukleinsäuren Kit (keine vergleichbaren automatisierten Extraktion Kits sind noch nicht verfügbar) beendet. Die durchschnittlichen C t-Werte über alle Wiederholungen sind 34,58 ± 0,66 und 29,76 ± 0,50 für fetale männlich (CHY) und insgesamt (CH1) DNA verbunden, die sehr gute Reproduzierbarkeit des automatisierten Extraktionsverfahren demonstriert. Die Konzentration an fötaler DNA in der Gesamt-DNA-Pool (der Genom-Äquivalente) wird auf der Grundlage fit Punktanalyse Vergleich mit Standards berechnet, wobei die resultierende durchschnittliche% fötaler DNA in allen Proben von 2,8%. Die tatsächliche% fetalen DNA für diese Probe ist unbekannt, weil Proben vor der Durchführung der Extraktion wurden gepoolt. Fetal DNA-Zusammensetzungin nicht-gepoolten Plasma-Proben wurde mit Hilfe der TruTip Verfahren sind in der Regel 1,5-fach höher im Vergleich zu den Ergebnissen mit einem Qiagen Circulating Nucleic Acids Kit (nicht gezeigt).
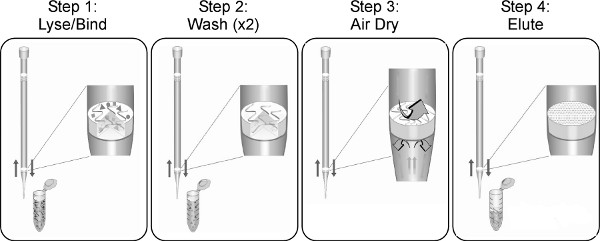
Abbildung 1. Die TruTip Extraktion und Workflow, unabhängig von der Liquid-Handling-System. Andere Probenvorbereitung und Liquid-Handling Schritte können in automatisierte Routinen abhängig von den Fähigkeiten des jeweiligen Instruments und Liquid-Handling-Software eingearbeitet werden.
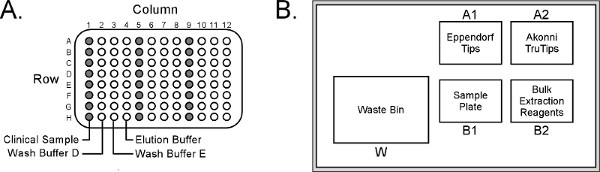
Abbildung 2. (A) Eppendorf epMotion 5070 Probenteller Layout. (B) Arrangement von Reagenzien / ConsumaBles auf dem Arbeitstisch. Die Probe Platte kann konfiguriert werden für bis zu 24 Proben (Spalten 1, 5 bzw. 9), obwohl die epMotion nur bearbeiten, werden maximal 8 Proben gleichzeitig. Klicken Sie hier, um eine größere Abbildung anzuzeigen .
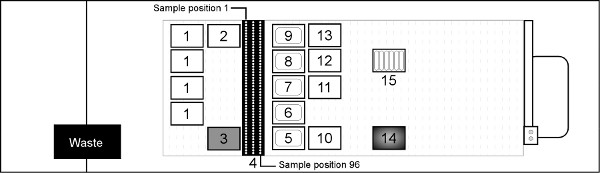
Abbildung 3. Hamilton STAR Deckslayout zur Reinigung von genomischer DNA aus Vollblut (nicht maßstabsgetreu) Deck Position 1 = 1 ml Hamilton gefiltert Tipps;. 2 = 1 ml Hamilton nicht gefilterten Tipps, 3 = Akonni / Hamilton 1 ml LPT 2 mm TruTips; 4 = Eingang Blutprobe Trägern (Blutentnahme-oder Mikrozentrifugengefäße); 5-9 = 290 ml Reagenz Tröge für Lysis Buffer F, Ethanol, Waschpuffer J, K Wash Buffer und Elution Buffer A2, bzw., 10 = 96 dEEP gut Binding Platte; 11 = 96 Tiefbrunnen waschen J; 12 = 96 Tiefbrunnen waschen K; 13 = 96 Tiefbrunnen Elution Platte; 14 = Hamilton HHS2 Heizung / Shaker mit Nunc 96 Tiefbrunnen Inkubationsplatte; 15 = 50 ml Reagenz Trog Zusatz von Proteinase K. Klicke hier, um eine größere Abbildung anzuzeigen .
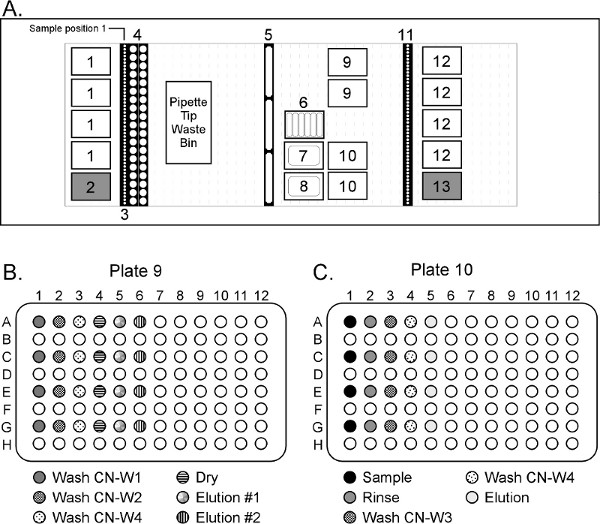
Abbildung 4. Hamilton STARPLUS Deckslayout zur Reinigung von DNA aus großvolumigen Plasma-Proben (nicht maßstabsgetreu). Das System ist mit 8 x 5 ml Kanälen und 8 x 1 ml Kanäle (nicht in der Deck-Layout dargestellt) ausgestattet. Deck Position 1 = Hamilton 4 ml filtriert Tipps, 2 = Akonni / Hamilton 5 ml TruTips; 3 = Quelle Plasmaproben, 4 = 50 ml konische Röhrchen, 5 = 120 ml Reagenz Tröge mit CN-W1, W2 und CN-CN-W4 Reagengen; 6 = Low-Volume-Reagenz Tröge mit Proteinase K, CN-B2, CN-B3, EBA2, EBB und CN-W3 Reagenzien; 7 = 290 ml Reagenz Trog mit CN-L1 Reagenz, 8 = 290 ml Reagenz Trog mit CN B1-Reagenz, 9 = 96 Deep Well Platten für Schritt 1, 10 = 96 Deep Well Platten für Schritt 2; 11 = Probenträger für gereinigten Endprodukt; 12 = 1 ml Hamilton ungefilterten Tipps; 13 = Akonni / Hamilton 1 ml LPT 4 mm TruTips. Klicke hier, um eine größere Abbildung anzuzeigen .
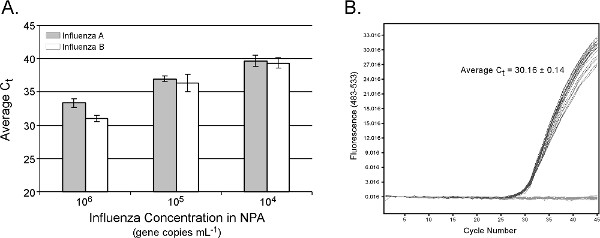
Abbildung 5. (A). Echtzeit-PCR-Ergebnisse von automatisierten TruTip Extraktion von Influenza-Virus ergänzt in Nasen-Rachen absaugen (NPA). Eingang NPA volume = 100 ul, Elutionsvolumen = 50 ul. Ergebnisse sind der Durchschnitt von 3 replizieren extractions von 5 verschiedene Hintergründe NPA (n = 15) pro Verdünnung Ebene und Grippe vorbei. qPCR wurde auf dem LightCycler ® 480 System mit Assaybedingungen zuvor beschrieben 15 durchgeführt. (B) Keine Kreuzkontamination wird erkannt, wenn 12 positive Proben ohne NPA-Template-Kontrollen und vorbehaltlich der automatisierten Extraktion durchsetzt sind. Klicken Sie hier, um eine größere Abbildung anzuzeigen .
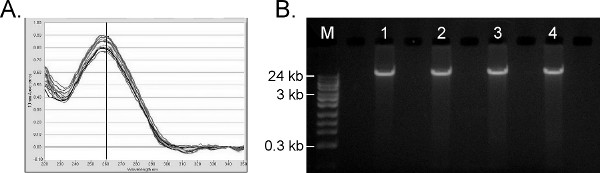
Abbildung 6. Ergebnisse von menschlicher genomischer DNA Extraktion aus Vollblut. A) UV-Vis-Spuren aus einem ND-1000 (ThermoFisher) von 10 zufällig ausgewählten repliziert. B) 1% Agarosegel von TruTip gereinigte gDNA. M = Fisher 24 kb Max DNA Ladder. Die Bahnen 1 - 4 = ~ 100 ng gereinigte gDNA aus vier zufällig ausgewählte repliziert. Klicken Sie hier, um eine größere Abbildung anzuzeigen .
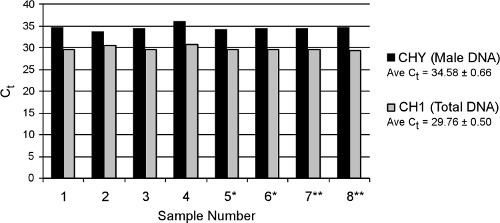
Abbildung 7. Echtzeit-PCR-Ergebnisse aus acht replizieren TruTip Extraktionen frei zirkulierender DNA aus Plasma. Proben durch Sternchen (* oder **) bezeichnet wurden an unterschiedlichen Tagen extrahiert. CHY quantifiziert männlichen fetalen DNA quantifiziert und CH1 Gesamt-DNA vorhanden ist (Mutter und Kind). qPCR wurde auf einem LightCycler 480 System (Roche) mit bisher veröffentlichten Tests Targeting CHY und CH1 18 durchgeführt.
Diskussion
Die Einfachheit des TruTip Konzept und Workflow (Abbildung 1) machen es leicht angepasst, automatisierte, effiziente und effektive für eine Reihe von klinischen Probenmatrices, Eingang Probenvolumina und Liquid-Handling-Systeme. Es sollte jedoch erkannt werden, dass jede klinische Probe einzigartig ist, und eine mit der nächsten in der Viskosität, Partikel, Schleim, Oberflächenverunreinigungen, mikrobiellen und / oder menschlichen genetischen Hintergründen variieren. Angesichts erwarteten Variationen in der klinischen Probe Zusammensetzung und Verwendungen von einer automatisierten Probenvorbereitung TruTip Protokoll, kann es daher notwendig sein, bestimmte Schritte in einer TruTip Verfahren zu modifizieren, um die gewünschten Ergebnisse für eine bestimmte Art Probe zu erreichen. Unabhängig von der Art der Probe enthalten jedoch TruTip Parameter, die haben in der Regel den größten Einfluss auf Nukleinsäure Reinheit und / oder Verwertung:
- Beispiel Mischen und Homogenisieren mit Lysepuffer (und Alkohol). Während TruTips eine erneute habenlativ große Porengröße Probenhomogenisierung und Verflüssigung ist sehr wichtig für eine effiziente Lyse und nachfolgende Bindung Schritte zum TruTip Monolith. Mit einer homogenen und gut Flüssiggas Lysat können Proben durch die TruTip mit höheren Flussraten, die die gesamte Probe Bearbeitungszeit reduziert bewegen. Das große Volumen Plasma-Protokoll zeigt das Potenzial für die Nutzer, um gründlich zu homogenisieren und zu verflüssigen schwierigen Proben (on-line oder off-line), trotz der großen Eingang Probenvolumen.
- Flussraten. Langsamer Flussraten während Nukleinsäurebindung oder Elution der Regel in höheren Ausbeuten führen Nukleinsäure, wenn auch auf Kosten der gesamten Bearbeitungszeit. Langsamer Strömungsgeschwindigkeiten kann auch die Ausdehnung der DNA Scherung.
- Zyklus-Nummern. Die optimale Anzahl der Aspiration und verzichten Zyklen abhängig ist Probe Typ, total Probenvolumen und Strömungsgeschwindigkeiten. Schritt 1 in 1 ist typischerweise der Punkt, an dem Zyklus numbers (und Strömungsgeschwindigkeiten) kann einige empirische Optimierung, mit Proben wie Nasen-Rachen absaugen (Abb. 5), die eine der schwierigeren Lysate auf Grund des Angebotes von NPA Viskositäten von verschiedenen Patienten zu optimieren.
- Trocknen. Vollständige Trocknung der TruTip Monolith ist zwingend notwendig, um das restliche organische Lösungsmittel aus co-eluiert mit der gereinigten Nukleinsäure-Probe und hemmende Folgeprozesse oder Tests zu verhindern. Da TruTip nicht über Zentrifugieren oder Vakuumfiltration getrocknet, ist es wichtig, sowohl die Strömungsgeschwindigkeit und Taktzahlen während der Trocknungsstufe zu maximieren. Manchmal gibt es ein Restrisiko Tropfen Waschlösung auf der Endstation der TruTip nach der Trocknung Zyklen abgeschlossen sind. Die Hamilton Roboter hat die Fähigkeit, eine "Tip Touch" auf der Seite der Vertiefung, um die Tröpfchen Freilegung, wodurch eine lösungsmittelfreie Elution. Die epMotion System nicht über diese Funktion, sondern eine der TruTip Terminus in el Vorspülenution Puffers programmiert ist, den gleichen Effekt zu erzielen.
Da die Geometrie Pipettenspitze Material und Befestigungsart der Roboter-Arm-Kanal einzigartig für jedes Instrument Hersteller sind, ist eine andere TruTip Konstrukt für jede Liquid-Handling-System erforderlich. Die TruTip Monolith Abmessungen (Durchmesser, Dicke und Porengröße) haben mit Nukleinsäure-bindende Kapazität (und Elution Wirkungsgrade) korrelieren, ist wie bei jedem Festphasenextraktion Technik erwartet. Während dicke (> 4 mm) Matrizen zu einer 1 ml TruTip auf Nukleinsäure-bindende Kapazität für großvolumige Proben zu erhöhen und / oder die Bindung auszugleichen Kapazität über spezifische TruTip Formate eingebettet sein kann, gibt es einen Kompromiss zwischen TruTip Dicke und Flussraten während die anfängliche Bindung Schritt (in Gegenwart von Rohlysaten). Daher ist es manchmal vorteilhaft, größeren Durchmesser Monolithen in größerem Volumen Pipettenspitzen einzubetten für die ersten Schritte eines automatisierten Protokoll (zB </ Em> die 5 ml Hamilton / Akonni TruTips für großvolumige Extraktionen). Angesichts der spezifischen TruTip Konfigurationen durch die Hersteller von Liquid Handling-Roboter diktiert, jedoch haben wir nicht unbedingt erwarten TruTip Nukleinsäure Renditen identisch sein über Liquid Handling-Plattformen verschiedener Hersteller, oder über verschiedene TruTip Größen.
Klinische Proben (per Definition) enthalten erhebliche Mengen an humaner genomischer DNA, wenn sie aus einer normalerweise sterilen Websites (zB Rückenmarksflüssigkeit) erworben werden. Manchmal ist die humane genomische DNA gewünscht wird (wie in 6), während in anderen Anwendungen der menschlichen DNA stellt einen unerwünschten Hintergrund Genom (wie bei Figur 5). Die Anwesenheit von Hintergrund-DNA ist in der Regel unproblematisch, solange die Gesamtmenge von Nucleinsäure in der Probe nicht mehr als die Bindungskapazität des Monolithen und die Hintergrund-DNA als Träger zu dienen, wenn das gewünschte Ziel-NukleinsäureSäure ist in Spuren vorhanden. Das Ziel des hochvolumigen Plasmaextraktion Protokoll (Fig. 7) zu isolieren (fragmentierten) fötale DNA in Gegenwart einer 10-20 fachen Überschuß der mütterlichen DNA, die ähnlich wie die Probenvorbereitung Ziel Infektionskrankheit Tests ist, außer dass die Sequenzen hohe Kongruenz und kann nur durch sehr spezifische molekulare Tests und / oder Größe Diskriminierung unterscheiden. In diesem Fall wird die gesamte Umlauf-DNA isoliert mit einer 5 ml TruTip und anschließende hochmolekularen und niedermolekularen fetalen DNA durch getrennt nachfolgende Bindung und Elution mit einem 1 ml TruTip durch Veränderung der Bindung Pufferbedingungen. Selektive Größe Trennung und Anreicherung von Ziel-Nukleinsäuren auf ihre Bindung und Elution Eigenschaften auf eine Silica-Monolith basiert, ist ein deutlich anderen Wirkmechanismus als durch Membranen oder Größenausschluss-Spin-Säulen erreicht. Größe Trennung und Anreicherung von mikrobieller DNA aus humanen genomischen DNA kann eine seinccomplished in zukünftigen Anwendungen über Customizing TruTip verbindlich und Elutionspuffer.
Die automatisierten Protokolle gezeigt betonen die Nützlichkeit der TruTip Monolith selbst zum Verarbeiten diverse klinische Proben, und wie sie für große Mengen und spezifischen Liquid-Handling-Roboter angepasst werden. Die optimierte Methoden in der Regel schneller Extraktion Protokolle im Vergleich zu anderen automatisierten Systemen führen. Die Einfachheit des TruTip Technologie, aber auch bietet einige Kostenvorteile für die Interessenten in den Kauf eines neuen, automatisierten Aufreinigung von Nukleinsäure-System, weil die primären Hardware für die Automatisierung TruTip Verfahren erforderlich ist die Pipettenkanal Arm selbst eher als magnetische Stäbe, Vakuum-Systeme oder On-Board-Zentrifugen. Verwendung Fertigspritze Reagenz Platten können auch zu einer Verminderung Raum und erforderlichen Verbrauchsmaterialien und die doppelte Durchsatz pro Lauf. Minimierung Deck mit TruTip Protokolle ermöglicht auch fortgeschrittene Anwender zu integrieren stromaufwärts oder downstream automatisierte Prozesse mit dem TruTip. Zum Beispiel Hamiltons easyBlood Lösung zur Fraktionierung Vollblut können mit dem automatisierten TruTip Extraktionsverfahren, die deutlich straffen würde Bio-Banking-Prozesse integriert werden. Post-Extraktionsverfahren wie Nukleinsäure Quantifizierung, Normalisierung, PCR Set-up, oder DNA-Sequenzierung sind auch leicht mit TruTip auf den größeren Liquid-Handling-Plattformen integriert.
Offenlegungen
Die Autoren sind Mitarbeiter von Akonni Biosystems, Inc., die Materialien, die in diesem Artikel verwendet zu produzieren.
Freier Zugang und Produktion dieses Artikels wird durch Akonni Biosystems, Inc. gesponsert
Danksagungen
Teile dieser Arbeit wurden durch die National Institutes of Health (NIH) unter dem Förderkennzeichen 44 R AI072784 unterstützt. Wir danken Dr. Kirsten St. George, Sara B. Griesemer, Daryl Lamson und Amy Dean des Labors für Viruskrankheiten, Wadsworth Center in New York State Dept of Health für quantifiziert Influenza-Virionen und Zugang zu klinisch validiert Influenza Echtzeit PCR-Assays.
Materialien
| Name | Company | Catalog Number | Comments |
| Reagent/Material | |||
| TruTip Influenza Extraction Kit (EPM TruTips) |  Akonni Biosystems, Inc. Akonni Biosystems, Inc. | 300-11120 | |
| 95% Ethanol | Acros Organics/ThermoFisher Scientific | AC615110040 | |
| 99% Acetone | Sigma-Aldrich | 270725-4L | |
| DEPC-treated water | Life Technologies | AM9906 | |
| Reagent Reservoir, 30 ml | Eppendorf | 960050100 | |
| Deep well plate 96/2,000 μl | USA Scientific | 30502302 | |
| epT.I.P.S. Motion Filtertips, 1,000 μl | Eppendorf | 960050100 | |
| Equipment | |||
| epMotion 5070 System | Eppendorf | 5070 000.000 | |
| Dispensing tool TM1000-8 | 960001061 | ||
| Reservoir rack | 960002148 | ||
| Table 1. Reagents and equipment for automated RNA extraction from NPA. | |||
| Reagent/Material | |||
| TruTip gDNA Blood Extraction Kit (Hamilton TruTips) |  Akonni Biosystems, Inc. Akonni Biosystems, Inc. | 300-20341 | |
| 95% ethanol | Acros Organics/ThermoFisher Scientific | AC615110040 | |
| Proteinase K | AMRESCO LLC | E195 | |
| 1 ml Hamilton filtered CO-RE 96 tip rack | Hamilton Robotics, Inc. | 235905 | |
| 1 ml Hamilton non-filtered CO-RE 96 tip rack | Hamilton Robotics, Inc. | 235904 | |
| 50 ml Reagent Trough | Hamilton Robotics, Inc. | 187297 | |
| Deep Well 2 ml plate | USA Scientific | 1896-2800 | |
| Nunc 96 DWP-2 ml | Thermofisher | 27874 | |
| Reagent Trough | Fisher | 14-222-412 | |
| Equipment | |||
| Hamilton STAR System | Hamilton Robotics, Inc. | 173027 | |
| 1 ml Independent Pipette Channels / Modular Arm | Hamilton Robotics, Inc. | 173081/173050 | |
| 1 ml 96-channel head | Hamilton Robotics, Inc. | 199090 | |
| Tip Carriers | Hamilton Robotics, Inc. | 182085 | |
| Sample Carriers/Inserts | Hamilton Robotics, Inc. | 173400/182238 | |
| Plate Carriers | Hamilton Robotics, Inc. | 182090 | |
| Multiflex Carrier | Hamilton Robotics, Inc. | 188039 | |
| HHS2 Heater Shaker Unit | Hamilton Robotics, Inc. | 199033 | |
| Rack Carrier | Hamilton Robotics, Inc. | 188047 | |
| Table 2. Regents and equipment for 96-well genomic DNA extraction from whole blood. | |||
| Reagent/Material | |||
| TruTip R+D Circulating DNA Extraction Kit (Hamilton TruTips) |  Akonni Biosystems, Inc. Akonni Biosystems, Inc. | Call to inquire | |
| 100% ethanol | Sigma-Aldrich | 459828-1L | |
| Isopropanol | Acros Organics/ThermoFisher Scientific | AC327270010 | |
| Proteinase K | AMRESCO LLC | E195 | |
| Filtered 4 ml Tips | Hamilton Robotics, Inc. | 184022 | |
| Unfiltered 1 ml Tips | Hamilton Robotics, Inc. | 235939 | |
| 96-Deep Well Plates | USA Scientific | 1896-2800 | |
| 50 ml Conical Tubes | Corning/ThermoFisher Scientific | 05-526B | |
| 50 ml Reagent Troughs | Hamilton Robotics, Inc. | 187297 | |
| 120 ml Reagent Troughs | Hamilton Robotics, Inc. | 182703 | |
| Large Volume 96-Pos Reagent Troughs | ThermoFisher Scientific | 14-222-412 | |
| Equipment | |||
| STARplus Autoload Workstation Base / Deck Module | Hamilton Robotics, Inc. | 173025/190012 | |
| 1 ml Independent Pipette Channels / Arm | Hamilton Robotics, Inc. | 173081/173052 | |
| 5 ml Independent Channel / Modular Arm | Hamilton Robotics, Inc. | 184090/173050 | |
| Plate Carriers | Hamilton Robotics, Inc. | 182090 | |
| Multiflex Carrier | Hamilton Robotics, Inc. | 188039 | |
| Rack Carrier for 50 ml Reagent Troughs | Hamilton Robotics, Inc. | 188047 | |
| 120 ml Reagent trough carrier | Hamilton Robotics, Inc. | 185290 | |
| Tip Carriers | Hamilton Robotics, Inc. | 182085 | |
| 50 ml Tube Carriers | Hamilton Robotics, Inc. | 182245 | |
| 24 Position Sample Carriers | Hamilton Robotics, Inc. | 173400 | |
| 32 Position Sample Carrier | Hamilton Robotics, Inc. | 173410 | |
| Table 3. Reagents and equipment for large volume DNA extraction from plasma. | |||
Referenzen
- Boom, R., Sol, C. J. A., et al. Rapid and simple method for purification of nucleic acids. J. Clin. Microbiol. 28, 495-503 (1990).
- Baker, M. P., Mitchell, A., et al. Isolation of genomic DNA from blood using a novel filter-based DNA purification technology. BioTechniques. 31, 142-145 (2001).
- Sinclair, B. To bead or not to bead: applications of magnetic bead technology. The Scientist. 12, 17-24 (1998).
- Dederich, D. A., Okwuonu, G., et al. Glass bead purification of plasmid template DNA for high throughput sequencing of mammalian genomes. Nucl. Acids Res. 30, e32 (2002).
- Levison, P. R., Badger, S. E., et al. Recent developments of magnetic beads for use in nucleic acid purification. J. Chromatogr. A. 816, 107-111 (1998).
- Hourfar, M. K., Schmidt, M., Seifried, E., Roth, W. K. Evaluation of an automated high-volume extraction method for viral nucleic acids in comparison to a manual procedure with preceding enrichment. Vox Sang. 89, 71-76 (2005).
- Perelle, S., Cavellini, L., et al. Use of a robotic RNA purification protocol based on the NucliSens easyMAG for real-time RT-PCR detection of hepatitis A virus in bottled water. J. Virol. Methods. 157, 80-83 (2009).
- Riemann, K., Adamzik, M., et al. Comparison of manual and automated nucleic acid extraction from whole-blood samples. J. Clin. Lab. Anal. 21, 244-248 (2007).
- Hukari, K. W., Shultz, M., Isely, N., Milson, R., West, J. A. A completely automated sample preparation instrument and consumable device for isolation and purification of nucleic acids. J. Lab. Autom. 16, 355-365 (2011).
- Kessler, H., Mühlbauer, G. Fully automated nucleic acid extraction. MagNA Pure LC. Clin. Chem. 47, 1124-1126 (2001).
- Miller, S., Seet, H., Khan, Y., Wright, C., Nadarajah, R. Comparison of QIAGEN automated nucleic acid extraction methods for CMV quantitative PCR testing. Am. J. Clin. Pathol. 133, 558-563 (2010).
- Dundas, N., Leos, N. K., Mitui, M., Revell, P., Rogers, B. B. Comparison of automated nucleic acid extraction methods with manual extraction. J. Mol. Diagn. 10, 311-316 (2008).
- Kruhøffer, M., Voss, T., et al. Evaluation of the QIAsymphony SP workstation for magnetic particle-based nucleic acid purification from different sample types for demanding downstream applications. J. Lab. Autom. 15, 41-51 (2010).
- Belgrader, P. Apparatus, system, and method for purifying nucleic acids. United States Patent. , (2010).
- Chandler, D. P., Griesemer, S. B., et al. Rapid, simple influenza RNA extraction from nasopharyngeal samples. J. Virol. Methods. 183, 8-13 (2012).
- Chan, K. H., Lai, S. T., et al. Analytical sensitivity of rapid influenza antigen detection tests for swine-origin influenza virus (H1N1). J. Clin. Virol. 45, 205-207 (2009).
- Chan, K. H., Lama, S. Y., et al. Comparative analytical sensitivities of six rapid influenza A antigen detection test kits for detection of influenza A. subtypes H1N1, H3N2 and. 38, 169-171 (2007).
- Fan, H. C., Blumenfeld, Y. J., Chitkara, U., Hudgins, L., Quake, S. R. Noninvasive diagnosis of fetal aneuploidy by shotgun sequencing DNA from maternal blood. Proc. Natl. Acad. Sci. USA. 105, 16266-16271 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten