Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Erzeugung, Verstärkung und Titration von rekombinanten Respiratory Syncytial Viren
In diesem Artikel
Zusammenfassung
Wir beschreiben eine Methode zur Erzeugung und Verstärkung genetisch modifiziert respiratory syncytial Viren (RSV) und eine optimierte Plaque-Assay für RSV. Wir veranschaulichen dieses Protokolls durch die Schaffung von zwei rekombinanter Viren, die Quantifizierung der RSV-Replikation ermöglichen bzw. live-Analyse des RSV Einschlusskörperchen und Einschlusskörperchen-assoziierten Granulat Dynamik.
Zusammenfassung
Der Einsatz von rekombinanten Viren ist entscheidend in der Basis- oder angewandte Virologie geworden. Reverse Genetik ist nachgewiesen worden, werden eine extrem leistungsfähige Technologie, sowohl virale Replikationsmechanismen zu entschlüsseln und zu studieren antivirale Medikamente oder Impfstoffe Entwicklungsplattform vorsehen. Den Bau und die Manipulation von einem reverse genetische System für einen negativ-Strang RNA-Virus wie ein respiratory syncytial Virus (RSV), allerdings bleibt zart und erfordert spezielles Know-how. Der RSV-Genom ist eine Single-Strang, negativen Sinn RNA von ca. 15 kb, die als Vorlage für die virale RNA-Replikation und Transkription dient. Unser reverse Genetik-System nutzt eine cDNA-Kopie des RSV lange Belastung Humangenoms (HRSV). Diese cDNA sowie cDNAs Codierung virale Proteine der komplexen Polymerase (L, P, N und M2-1) befinden sich in einzelnen Expressionsvektoren unter T7 Polymerase Steuersequenzen. Die Transfektion dieser Elemente in BSR-T7/5 Zellen, die stabil T7 Polymerase ausdrücken, ermöglicht die zytoplasmatischen Replikation und Transkription der rekombinanten RSV zu gentechnisch veränderten Virionen. Eine neue RSV, die an der Zelloberfläche und in der Kultur Überstand des BSRT7/5 vorliegt, ist versammelt, um menschliche HEp-2 Zellen für virale Verstärkung zu infizieren. Zwei oder drei Runden der Verstärkung sind erforderlich, um virale Bestände mit 1 x 106 bis 1 x 107 Plaque-bildenden Einheiten (PFU) / mL. Methoden für die optimale Ernte, Einfrieren und Titration der viralen Bestände sind hier ausführlich beschrieben. Wir illustrieren das Protokoll hier vorgestellten durch die Schaffung von zwei rekombinanter Viren bzw. Ausdruck freies grünes fluoreszierendes Protein (GFP) (RSV-GFP) oder viralen M2-1 mit GFP (RSV-M2-1-GFP) verschmolzen. Wir zeigen, wie mithilfe von RSV-GFP um RSV-Replikation und der RSV-M2-1-GFP viralen Strukturen sowie virale Proteindynamik in lebenden Zellen visualisieren mit Videomikroskopie Techniken zu quantifizieren.
Einleitung
Menschlichen RSV ist die führende Ursache der Hospitalisierung für akute Infektion der Atemwege in Säuglinge weltweit1. RSV ist darüber hinaus mit einer erheblichen Krankheitslast bei Erwachsenen vergleichbar mit Grippe, mit die meisten Krankenhausaufenthalt und Sterblichkeit in der ältere2Belastung verbunden. Gibt es keine Impfstoffe oder spezifische antivirale Medikamente noch gegen RSV, aber vielversprechende neue Medikamente sind in Entwicklung3,4. Die Komplexität und die Schwere der Techniken der Quantifizierung der RSV Multiplikation erschweren die Suche nach antivirale Medikamente oder Impfstoffe trotz erheblicher Anstrengungen. Die Quantifizierung der RSV Multiplikation in-vitro-basiert im Allgemeinen auf teure, mühsame und zeitraubende Methoden bestehen vor allem in der Analyse des zytopathischen Effekts durch Plaque-Reduzierung, Mikroskopie, Immunostaining Assays, quantitative (qRT) Reverse-Transkriptase-Polymerase-Kettenreaktion (PCR) und Enzym-linked Immunosorbentprobe Tests zu bestimmen. Viren mit modifizierten Genomen und mit dem Ausdruck Reporter-Gene, wie die Codierung für die GFP, eignen sich eher für solche Vorführungen. Gekoppelt an die Verwendung von automatisierten Platte Leser, machen Reporter-gen tragenden rekombinante Viren diese Assays für Standardisierung und Hochdurchsatz-Zwecke besser geeignet.
RSV ist eine behüllte, nonsegmented negativen Sinne RNA-Virus, das gehört zur Gattung der Pneumoviridae Familie, Ordnung Mononegavirales5 Orthopneumovirus . Der RSV-Genom ist eine Single-Strang, negativen Sinn RNA von ca. 15 kb, enthält eine forensisches Region an den 3' und 5' Extremitäten genannt Führer und Anhänger und 10 transkriptionelle Einheiten 11 Proteine codieren. Die Gene sind wie folgt sortiert: 3'-NS1, NS2, N, P, M, SH, G, F, M2 (M2-1 und M2-2 Proteine kodieren) und L-5 ". Die genomische RNA ist fest Nucleoprotein N. Using Encapsidated genomische RNA als Vorlage verpackt, virale RNA-abhängige RNA-Polymerase (Trichothecene) Transkription und Replikation der viralen RNA zu gewährleisten. Virale Trichothecene besteht aus der großen Protein L trägt die Nukleotid-Polymeraseaktivität schlechthin, seine obligatorische Cofaktor Phosphoprotein P und das M2-1-Protein, das Funktionen wie eine virale Transkription Faktor6. In infizierten Zellen induziert RSV die Bildung von zytoplasmatischen Einschlüssen Einschlusskörperchen (IBs) genannt. Morphologisch ähnliche zytoplasmatischen Einschlüssen sind für mehrere Mononegavirales7,8,9,10beobachtet worden. Neuere Untersuchungen zu den Tollwut-Virus, vesikuläre Stomatitis-Virus (VSV) und Ebola-Virus RSV hat gezeigt, dass virale RNA-Synthese tritt in IBs, die somit als virale Fabriken8,9,11, angesehen werden kann 12. Virus Fabriken konzentrieren die RNA und virale Proteine für virale RNA-Synthese benötigt und enthalten auch zelluläre Proteine13,14,15,16, 17. IBs weisen eine funktionale Subkompartiment genannt IB-assoziierten Granulat (IBAGs), die sich darauf konzentrieren die neu dazu im Entstehen begriffenen viralen mRNA zusammen mit dem M2-1-Protein. Die genomische RNA und die L, P und N werden in IBAGs nicht erkannt. IBAGs sind kleine dynamische kugelförmigen Strukturen im Inneren IBs, die die Eigenschaften des flüssigen Organellen12aufweisen. Trotz der zentralen Rolle von IBs in virale Vermehrung ist sehr wenig über Natur, interne Struktur, Entstehung und Betrieb dieser viralen Fabriken bekannt.
Der Ausdruck des Genoms des Poliovirus aus einer cDNA aktiviert die Produktion des ersten ansteckende virale Klons in 198118. Für negative einzelsträngigen RNA-Viren fand es nicht bis 1994 die Produktion eines ersten Tollwut-Virus nach Transfektion von Plasmiden in Zellen19 statt. Das erste Plasmid-basierte reverse genetische System für RSV erschien 199520. Reverse Genetik führten zu großen Fortschritten auf dem Gebiet der Virologie. Die Möglichkeit der Einführung von spezifischen Änderungen in das virale Genom hat kritische Einblicke in die Replikation und Pathogenese von RNA-Viren zur Verfügung gestellt. Diese Technologie hat die Entwicklung von Impfstoffen auch erheblich erleichtert, indem es spezifische Dämpfung durch gezielte Reihe von Modifikationen erlaubt. Genom Änderungen erlauben eine schnelle Quantifizierung der viralen Vermehrung erheblich verbessert die antivirale Screening und Untersuchung von ihrer Wirkungsweise.
Obwohl zuvor beschrieben, bleibt die Erlangung genetisch veränderten RSV zart. Hier zeigen wir ein Protokoll erstelle ich zwei Arten von rekombinanten HRSV, RSV-GFP oder RSV-M2-1-GFP bzw. zum Ausdruck zu bringen. In diesem Protokoll beschreiben wir die Transfektion Voraussetzungen für die neue rekombinanten Viren, sowie ihre Verstärkung zu viralen Aktien mit hoher Titer, geeignet für reproduzierbare Experimente zu retten. Der Bau der reversen Genetik Vektoren ist per se nicht beschrieben. Wir beschreiben Methoden für die optimale ernten und Einfrieren von viralen Bestände. Die genaueste Methode, infektiöse Viruspartikel zu quantifizieren bleibt Plaque-Assay. Zellen sind infiziert mit seriellen Verdünnungen der analysierten Suspension und inkubiert mit einem Overlay, das verbietet die Verbreitung von freien Viruspartikel im Überstand. Unter solchen Bedingungen wird der Virus nur zusammenhängende Zellen, aus denen eine "Tafel" für jedes erste infektiöse Partikel infizieren. In der konventionellen RSV-Titration-Assay sind Gedenktafeln von Immunostaining aufgedeckt und unter mikroskopischer Beobachtung gezählt. Diese Methode ist teuer und zeitaufwändig. Hier haben wir ein sehr einfaches Protokoll für eine RSV-Plaque-Assay mikrokristalline Cellulose verwenden, die es die Bildung von Plaques, die mit bloßem Auge sichtbar ermöglicht. Wir zeigen, wie RSV-GFP Maßnahme RSV Replikation und damit zum verwendet werden kann die Auswirkungen der antiviralen Medikamenten zu quantifizieren. Kombination von reverse Genetik und live-imaging-Technologie, zeigen wir Ihnen wie RSV-M2-1-GFP ermöglicht es Wissenschaftlern, M2-1 in lebenden Zellen zu visualisieren und die Dynamik der intrazellulären viralen Strukturen wie IBs zu folgen.
Protokoll
1. materielle Vorbereitung
- Zelle Medien zu kaufen (reduzierte Serum Medien, minimale unerlässlich [MEM] 10 x MEM und Dulbecco modifizierten Adlers Medium [DMEM]), Transfection Reagens und mikrokristalline Cellulose (siehe Tabelle der Materialien).
- Erhalten folgenden Vektoren für reverse Genetik: die genomische Vector(s) und die Codierung des N-Proteins und der Polymerase komplexe Proteine Expressionsvektoren. Die genomischen Vektoren enthalten das volle cDNA Genom der RSV-GLP (p-RSV-GFP) und der RSV-M2-1GFP (p-RSV-M2-1GFP) stromabwärts von Bakteriophagen T7-RNA-Polymerase (T7 Pol) Veranstalter. Die Expressionsvektoren (bezeichnet als p-N, p -P-p-L und p-M2-1) enthalten die kodierenden Sequenz von N, P, L oder M2-1 flussabwärts die T7-Pol (siehe Rincheval Et Al.12 und Rameix-Welti Et Al.21 Details bezüglich der Plasmid-Konstrukte).
- Bereiten Sie die Medien für eine Zellkultur in einer sterilen Umgebung und für die Transfektion und Infektion. Verwendung DMEM mit 2 mM L-Glutamin ergänzt mit 10 % fetalen Kälberserum (FCS), 1.000 Einheiten/mL Penicillin und 1 mg/mL Streptomycin (oder ohne Antibiotika) und MEM mit 2 mM L-Glutamin ergänzt mit 0 %, 2 % oder 10 % FCS, 1.000 Einheiten/mL Penicillin und 1 mg/mL Streptomycin, bezeichnet als "komplette Medium" in das folgende Protokoll.
- Erhalten Sie BSRT7/522 Zellen zu und Bestände in kompletten Medium mit 10 % Dimethyl Sulfoxid (DMSO) bei 1 bis 2 x 106 Zellen/mL ergänzt. Erhalten Sie die Zelle-Bestände in flüssigem Stickstoff. Erhalten Sie HEp-2 Zellen. Kultur-BSRT7/5-Zellen im kompletten DMEM und HEp-2 Zellen in komplette MEM bei 37 ° C und 5 % CO2 in einer sterilen Umgebung.
- Bereiten Sie eine 10 X RSV Erhaltung Lösung (0,5 M HEPES und 1 M MgSO4 [pH 7.5] in Wasser) in einer sterilen Umgebung.
- Erhalten Sie ein umgekehrtes Fluoreszenzmikroskop kompatibel mit GFP-Fluoreszenz-Messungen und kompatibel mit live imaging, wenn es notwendig ist, um eine Infektion zu überwachen ist. Erhalten Sie einen Mikrotestplatte Reader kompatibel mit GFP-Fluoreszenz-Messungen für die Quantifizierung der RSV-GFP-Replikation.
(2) Rettung und erste Passage der rekombinanten Virus
Hinweis: Führen Sie die folgenden Schritte in einer sterilen Umgebung, mit einer Klasse II Sicherheitsschrank.
- Machen Sie am Vortag Transfection eine Aussetzung der Zelllinie BSRT7/5 bei 5 x 105 Zellen/mL in kompletten Medium. Verteilen Sie 2 mL Zellsuspension pro Bohrloch in einer 6-Well-Platte. Bereiten Sie einen Brunnen pro Virus, das gerettet werden soll und eine weitere für negativ-Kontrolle. Inkubieren Sie die Platte bei 37 ° C und 5 % CO2. Überprüfen Sie, dass die Zellen an der Mündung eine 80 %-90 % am nächsten Tag.
- Fixierung der reversen Genetik Vektoren (aus Schritt 1.2) p-RSV-GFP und p-RSV-M2-1-GFP, sowie p-N, p -P-p-L und p-M2-1. Mix für jeden Virus, retten, 1 µg der p-N und p -P, 0,5 µg p-L, 0,25 µg p-M2-1 und 1,25 µg p-RSV (GFP oder M2-1 GLP) in einem Rohr.
Hinweis: Verschiedenen Expressionsvektoren für M2-1, N, P und L können verwendet werden; jedoch ist das Verhältnis zwischen den Proteinen einzuhalten. Führen Sie die negative-Kontrolle den p-RSV-Vektor durch ein leerer Vektor ersetzen. -
Weiter zur Transfektion, nach der Transfektion Reagens Hersteller-Protokolls (siehe Tabelle der Materialien).
- Die gemischten Vektoren 250 µL reduzierte Serum Medium hinzufügen. Verdünnen Sie in einem anderen Gefäß 10 µL des Transfection Reagens in 250 µL reduzierte Serum-Medium. Sanft Wirbel sowohl Rohre und warten Sie 5 min. Mix den Inhalt beider Röhren und warten Sie 20 min bei Raumtemperatur.
- Spülen Sie die BSRT7/5-Zellen mit 1 mL reduzierte Serum Medium und verteilen Sie 1,5 mL MEM mit 10 % FCS ohne Antibiotika pro Bohrloch zu. Falls erforderlich, brüten Sie bei 37 ° C und 5 % CO2 bis die Inkubation in Schritt 2.3.1 beschriebenen abgeschlossen ist.
- Fügen Sie 500 µL der Transfektion Mischung im Schritt 2.3.1 auf eine gut vorbereitet, wenn die 20 min Inkubationszeit vorbei ist. Legen Sie die Zellen in den Inkubator bei 37 ° C und 5 % CO2 für 3 Tage. Ändern Sie nicht das Nährmedium der Zellen während der Transfektion.
- Beobachten, GFP-Fluoreszenz (Anregung bei 488 nm) und Emission bei 515 – 535 nm unter einem invertierten Fluoreszenzmikroskop mit 20 X Vergrößerung 1 X pro Tag, um die Effizienz der Rettung zu überwachen, über die GFP filtern.
- Am zweiten Tag nach der Transfektion Samen Sie die Zellen für den ersten Durchgang der geretteten Viren. Bereiten Sie eine Aussetzung der HEp-2 Zellen bei 5 x 105 Zellen/mL in kompletten Medium. 2 mL Zellsuspension pro Bohrloch in einer 6-Well-Platte (einen Brunnen pro Virus Rettungs-und eine Negativkontrolle) zu verteilen.
- Am dritten Tag der Transfektion, kratzen Zellen in jede Vertiefung der transfizierten BSRT7/5-6-Well-Platte verwenden verschiedenen Schaber für jedes gut. Übertragen Sie jedes gut Inhalte (Zellen und überstand) zu einem steril 2 mL Microcentrifuge Schlauch. Vortex jedes Rohr kräftig für mindestens 30 s, um die geretteten Virus aus der Zelle Membranen freisetzen.
Hinweis: Dies entspricht 0 (P0) Durchgang des geretteten Virus (Abbildung 1). -
Verwenden Sie die frischen virale P0 Aussetzung die erste Verstärkung der geretteten Viren durchführen.
- Entfernen Sie das Kulturmedium von HEp-2-6-Well-Platte am Vortag ausgesät (siehe Punkt 2.5) und 500 µL der P0 Suspension (aus Schritt 2,6) pro Bohrloch schnell hinzufügen. Platzieren Sie die HEp-2-Platte bei 37 ° C auf eine Wippe Rocker für weiche Agitation für 2 h.
- Entfernen Sie und entsorgen Sie die 500 µL des Inokulums und 2 mL MEM mit 2 % FCS. 3 Tage inkubieren Sie die Platte bei 37 ° C und 5 % CO2 . Dies erzeugt den ersten Durchgang (P1) der geretteten Viren (Abbildung 1).
- Fügen Sie 1/10 des Volumens des 10 X RSV Erhaltung Lösung (0,5 M HEPES und 1 M MgSO4 [pH 7.5]) in die restlichen P0-Suspension (aus Schritt 2.6). Wirbel der Mikroröhrchen energisch für 5 s und aliquoten beschriftet den Inhalt in kryogenen Röhren mit alkoholbeständigem Tags. Tauchen Sie der Rohre für mindestens 1 h in Alkohol bei-80 ° C vorgekühlt und bei-80 ° c lagern
- Titrieren P0-Lager (siehe Punkt 2.6) von jedem geretteten Virus (siehe Abschnitt 4 für die mikrokristalline Cellulose-Titration).
- Beachten Sie, dass GFP Fluoreszenz (Anregung bei 488 nm) und Emission bei 515 – 535 nm der HEp-2 Zellen infiziert mit P0 Aussetzung unter einem invertierten Fluoreszenzmikroskop mit 20 X Vergrößerung 1 X pro Tag, um die Infektion zu überwachen. Das Aussehen der kleinen Syncytia unter dem Hellfeld-Mikroskop beobachten und Zell-Ablösung der RSV zytopathogenen Effekts (CPE) widerspiegelt (siehe Abbildung 2).
- Beachten Sie, dass die Rettung gescheitert ist, wenn weder die Fluoreszenz als auch die CPE nach 2 – 3 Tagen sichtbar ist.
- Holen Sie den ersten Durchgang (P1) am Tag 3 oder 4 wie unter Punkt 2.6 beschrieben. Kurz gesagt, kratzen Sie die Zellen, sammeln Sie die Zellen und überstand zusammen, Wirbel, die Erhaltung-Lösung wie unter Schritt 2.8, aliquoten Probe hinzufügen und Einfrieren.
- Titrieren Sie (siehe Abschnitt 4 für die Titration Assay) und verstärken Sie den ersten Durchgang (siehe Abschnitt 3 für die Amplifikation) zu.
3. Verstärkung der geretteten Viren
Hinweis: Das folgende Protokoll beschreibt die Verstärkung der geretteten Viren in einem 75 cm2 Kolben. Passen Sie die Flasche auf den benötigten Mengen und die erforderlichen Multiplizität der Infektion (MOI). Tabelle 1 zeigt Bände für verschiedene Fläschchen. Führen Sie die folgenden Schritte in einer sterilen Umgebung in einer Klasse II Sicherheitsschrank.
- Bereiten Sie eine Aussetzung der HEp-2 Zellen bei 5 x 105 Zellen/mL in kompletten Medium, am Tag vor der Verstärkung. Verteilen Sie 15 mL Zellsuspension pro 75 cm2 Fläschchen und inkubieren Sie die Fläschchen bei 37 ° C und 5 % CO2. Bereiten Sie eine Flasche pro Virus zu verstärken.
- Am Tag nach dem Beginn der Inkubation, überprüfen Sie, dass die Zellen 80 – 100 % Zusammenfluss.
- Verdünnen Sie die virale Suspension aus Schritt 2.12 in MEM ohne FCS eine 3 mL Suspension bei 50.000 PFU/mL (entsprechend einer MOI von 0,01 PFU/Zelle) zu erhalten.
- Entfernen Sie das Medium und fügen Sie schnell die virale 3 mL-Suspension hinzu. Legen Sie die Flasche bei 37 ° C auf eine Wippe Rocker für weiche Agitation für 2 h.
- Entfernen Sie und entsorgen Sie das Inokulum und 15 mL MEM mit 2 % FCS. 2 – 4 Tage bei 37 ° C und 5 % CO2 inkubieren.
- Überprüfen Sie die Zellmorphologie und GFP-Fluoreszenz (Anregung bei 488 nm) und Emission bei 515 – 535 nm unter einem invertierten Fluoreszenzmikroskop mit 20 X Vergrößerung um den richtigen Zeitpunkt zu ernten, die Viren zu schätzen. Beachten Sie, dass dies in der Regel bei 50 – 80 % der HEp-2 Zelle Schicht aufgrund der RSV-CPE getrennt wird, die zwischen 48 und 72 h Postinfection (PI) auftritt (siehe Abbildung 3).
- Kratzen Sie die Zellen, die mit einer Zelle Schaber. Sammeln Sie die Zellen und der Überstand zusammen und übertragen Sie sie auf eine 50 mL Zentrifugenröhrchen.
- Fügen Sie 1/10 des Volumens des 10 x RSV Erhaltung Lösung (0,5 M HEPES und 1 M MgSO4 [pH 7.5]). Wirbel der Rohre kräftig für 5 s und klären Sie die Aussetzung durch eine 5 min Zentrifugation bei 200 X g.
- Übertragen des Überstands in ein 50 mL-Tube. Vortex kurz und aliquoten Suspension in kryogenen Röhren mit alkoholbeständigem Tags gekennzeichnet. Tauchen Sie der Rohre in vorgekühlte-80 ° C Alkohol für mindestens 1 h und bei-80 ° c lagern
- Freigeben eines Aliquoten zu titrieren die virale Aussetzung (siehe Abschnitt 4).
(4) Plakette Titration Assay
- Bereiten Sie 12-Well Platten für die Titration am Tag vor der Titration-Assay durchgeführt wird (sechs Brunnen werden benötigt, um eine Tube Virus Titrieren). Samen der Brunnen mit 1 mL HEp-2 Zellen bei 5 x 105 Zellen/mL in kompletten Medium.
-
Am nächsten Tag bereiten Sie eine sterile mikrokristalline Cellulose-Suspension (2,4 % [w/V] im Wasser) (siehe Tabelle der Materialien).
- Zerstreuen Sie 2,4 g mikrokristalline Cellulose Pulver in 100 mL destilliertem Wasser, mit einem standard Magnetrührer bis zur vollständigen Auflösung des Pulvers (in der Regel 4 – 12 h). Autoklaven die Aussetzung bei 121 ° C für 20 min und bei Raumtemperatur vor der Verwendung zu speichern.
Hinweis: Unter solchen Bedingungen ist die Federung für 1 Jahr stabil. - Nach dem Öffnen der Lösung in einer sterilen Umgebung, bewahren Sie es bei 4 ° C für 6 Monate. Mischen Sie die Suspension vor Gebrauch (per Hand zu schütteln oder vortexen) immer um sicherzustellen, dass sie homogen ist.
- Zerstreuen Sie 2,4 g mikrokristalline Cellulose Pulver in 100 mL destilliertem Wasser, mit einem standard Magnetrührer bis zur vollständigen Auflösung des Pulvers (in der Regel 4 – 12 h). Autoklaven die Aussetzung bei 121 ° C für 20 min und bei Raumtemperatur vor der Verwendung zu speichern.
- Bereiten Sie die 2 x MEM in einer sterilen Umgebung. Verdünnen Sie kommerzielle MEM 10 X mit sterilem Wasser und L-Glutamin, 1.000 Einheiten/mL Penicillin und 1 mg/mL Streptomycin hinzufügen. Die Verdünnung kräftig schütteln und bei 4 ° c lagern
Hinweis: Führen Sie Schritte 4.4 – 4.10 in einer sterilen Umgebung mit einer Klasse II Sicherheitsschrank. - Bereiten Sie sechs Röhren 900 µL MEM ohne FCS pro Virus titriert werden (die Titration Röhren). Tauen Sie das Virus Aliquote Vortex Rohr sie energisch für 5 s und Transfer 100 µL, die erste Titration.
- Führen Sie eine zehnfache Verdünnung 6 X wie folgt. Fügen Sie 100 μL des Virus zu 900 μl Medium in der ersten Röhre setzen Sie die Kappe auf dem Rohr und dessen Inhalt durch Vortexen mischen, für ein paar Sekunden. Ändern Sie die Spitze auf die Pipette, 900 µL Medium in der zweiten Röhre 100 µL der ersten Verdünnung hinzu, setzen Sie die Kappe auf dem Rohr und Wirbel. Wiederholen Sie den Vorgang bis das sechste Rohr.
Hinweis: Es ist sehr wichtig, die Tipp für jede Verdünnung zu ändern. - Schreiben Sie das Virus Namen und die Falte Verdünnungen auf HEp-2-12-Well-Platten. Fügen Sie eine Markierung um die Platte und Deckel passen, weil sie getrennt werden können, während der Färbung (Schritt 4,9). Entfernen Sie das Medium aus den Platten und verteilen Sie 400 µL einer Verdünnung pro Bohrloch zu. Inkubieren Sie die Platten bei 37 ° C für 2 h, Virus Adsorption.
Hinweis: Ändern Sie die Pipettenspitze zwischen jedem Inokulum oder gehen Sie vom niedrigsten zum höchsten Konzentration mit der gleichen Spitze. Impfen Sie eine limitierte Serie von Platten (1-2) gleichzeitig, um die Zellen trocknen zu vermeiden. -
Bereiten Sie die mikrokristalline Cellulose-Overlay während der Virus-Adsorption (Magistralrezeptur). Um 100 mL Overlay zu erhalten, mischen Sie 10 mL Suspension von 2,4 % mikrokristalline Cellulose, 10 mL 2 x MEM und 80 mL MEM mit 2 % FCS.
- Stellen Sie den pH-Wert des 2 x MEM auf rund 7,2 mit einer sterilen Natrium Bikarbonat-Lösung bei 7,5 %, nach der Farbindikator. Mikrokristalline Cellulose-Aufhängung und die MEM und mischen Sie kräftig.
- Am Ende der 2 h Inkubation geben Sie 2 bis 3 mL-Overlay in jedes 12-Well-Platten ohne das Inokulum zu entfernen. Achten Sie darauf, um die Kontamination der benachbarten Brunnen mit hohen virale Titer Befallsdruck zu vermeiden. 6 Tage inkubieren Sie die Platte bei 37 ° C und 5 % CO2 . Bewegen Sie die Platte nicht und bewegen Sie den Inkubator nicht während der Inkubation.
-
Fahren Sie fort, die Zellen mit Kristallviolett-Lösung (8 % Kristallviolett [V/V], 2 % Formaldehyd [V/V], und 20 % Ethanol [V/V] in Wasser) zu beflecken.
- Schützen Sie die Arbeitsfläche der Biosicherheit Schrank mit einem Blatt (das Kristallviolett Farben stark Oberflächen).
- Schütteln Sie die Platten zum Ausziehen der mikrokristalline Cellulose-Overlay. Die Überstände entfernen und waschen Sie die Zellen 2 X mit 1 x Phosphat-gepufferte Kochsalzlösung (PBS). Behandeln Sie die Platten eins nach dem anderen um die Zellen trocknen zu vermeiden. Geben Sie 1 – 2 mL Kristallviolett-Lösung hinzu und warten Sie 10-15 Minuten entfernen Sie die Lösung, die für das anschließende Platte beflecken wiederverwendet werden können.
- Tauchen Sie die Platten und Deckel in frischem Bleichmittel für ein paar Sekunden und dann waschen Sie sie gründlich mit Leitungswasser. Beachten Sie, dass die Platten und Abdeckungen durch das Bleichmittel dekontaminiert werden.
- Die Platten und Deckel auf Küchenpapier legen und trocknen lassen. Trocknen Sie die Platten bei einer Umgebungstemperatur nach Wasser spülen und bei Raumtemperatur lagern. Für längere Lagerung (Monate), halten die Platten geschützt vor Licht, die Farbe zu schützen. Beachten Sie, dass wenn die Zellen ihre Färbung verlieren, sie wieder mit Kristallviolett gefärbt werden können.
- Virus-Titer zu berechnen. Zählen Sie die Plaques in den Vertiefungen der Trockenplatten, die mit dem bloßen Auge sichtbar sind. Überprüfen Sie, dass die Zahl der Plaketten für die verschiedenen Verdünnungen kohärente (Faktor 10 zwischen jeder Verdünnung). Wählen Sie den Brunnen, an dem die Plaketten die am einfachsten sind zu zählen. Die Zahl der Plaketten gegen das Inokulum Volumen und die Verdünnung zu beurteilen.
Hinweis: Auf das Beispiel in Abbildung 4sind 21 Plaketten an die 10-5 Verdünnung gezählt. Diese entsprechen einem Titer von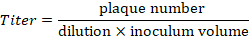
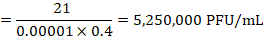
(5) die Verwendung von HRSV-GFP rekombinanten Virus Virusreplikation in Zellen mit kleinen interferierende RNA oder Virostatika behandelt zu überwachen
Hinweis: Führen Sie alle Schritte mit Ausnahme von 5.1 und 5.2.5 in einer sterilen Umgebung mit einer Klasse II Sicherheitsschrank.
-
Überwachung des Effekts der zellulären Gen-silencing auf RSV-Multiplikation
Hinweis: Die Transfektion Protokoll hängt das Reagenz (siehe Tabelle der Materialien).- Die GFP-Messung 96-Well Platten vorbereiten. Zwei Tage vor der Assay für gegebene kleine interferierende RNA (SiRNA), bereiten Sie eine Lösung der reduzierte Serum-Medien mit SiRNA in einer Konzentration von 100 nM und einer SiRNA Transfection Reagens verdünnt bei 1/500. Inkubieren Sie die Lösung für 30 min bei Raumtemperatur.
- Fügen Sie 25 µL der Lösung in die Vertiefungen der Platte in 5.1.1 (in dreifacher Ausfertigung) vorbereitet. Samen der Brunnen mit 75 µL aus einer Suspension von A549 Zellen 4 x 105 Zellen/ml in kompletten Medium ohne Antibiotika, um eine endgültige Zellkonzentration von 3 x 105 Zellen/mL zu erhalten. Inkubieren Sie die Platte für 48 h bei 37 ° C und 5 % CO2.
Hinweis: Die endgültige SiRNA-Konzentration ist 25 nM und die endgültige Transfection Reagens Lautstärke ist 0,5 µL/Well). - Wie folgt infizieren Sie die Zellen. Entfernen Sie das Medium aus dem Brunnen. Hinzufügen von 100 µL der RSV-GFP Aussetzung bei 50.000 PFU/mL und inkubieren Sie für 2 h bei 37 ° C und 5 % CO2. Entfernen Sie die virale Aussetzung zu und fügen Sie 100 µL DMEM mit 2 % FCS und ohne Phenol Rot hinzu. Inkubieren Sie die Platte bei 37 ° C und 5 % CO2.
- Bei 24 h und 48 h p.i., Messung der Fluoreszenz, legen Sie mithilfe eines Spectrofluorometer auf Anregung und Emission Wellenlängen von 488 und 520 nm bzw. (Fluoreszenz ist relative Fluoreszenz-Einheiten ausgedrückt). Verwenden Sie noninfected A549 Zellen als Standards für Fluoreszenz und Hintergrund.
Hinweis: Die Zellen müssen mit 4 % Paraformaldehyd (PFA) behoben werden, bevor Sie messen sie ohne das Cover der Platte.
-
Bewertung von der Droge-Hemmung mit RSV-GFP
- Die GFP-Messung 96-Well Platten vorbereiten. Samen Sie am Vortag der Assay die Brunnen mit 100 µL einer Suspension von HEp-2 Zellen bei 5 x 105 Zellen/mL in kompletten Medium ohne Phenol rot.
- Bereiten Sie eine serielle Verdünnung von den getesteten Medikamente (AZ4316 in diesem Beispiel) in MEM mit 2 % FCS und Antibiotika (50 µL pro Well) ergänzt. Bereiten Sie eine virale Suspension bei 10.000 PFU/mL in MEM ohne Stromazellen vaskulären Faktor (SVF) und Phenol-rot (50 µL pro Well).
- Entfernen Sie das Medium aus der 96-Well-HEp-2 Teller und 50 µL der Droge Suspension und 50 µL der viralen Suspension (in dreifacher Ausfertigung) hinzufügen. Durchführen Sie eine mock-Infektion als ein Steuerelement parallel.
Hinweis: Medikament Verdünnung und virale Aussetzung können gemischt werden, bevor Sie sie auf die Zelle hinzufügen, oder sie können nacheinander hinzugefügt werden. - Inkubieren Sie die Platte für 48 h bei 37 ° C und 5 % CO2.
- Messung der Fluoreszenz, mit einem Spectrofluorometer, wie in Schritt 5.1.4 beschrieben. Verwenden Sie Mock-infizierten HEp-2 Zellen als Standards für Fluoreszenz-Hintergrund-Niveaus.
6. Charakterisierung der M2-1 Lokalisierung In Vivo mit dem RSV-M2-1-GFP rekombinanten Virus
Hinweis: Führen Sie Schritte 6.1 und 6.2 in einer sterilen Umgebung, mit einer Klasse II Sicherheitsschrank.
- Bereiten Sie eine Aussetzung der HEp-2 Zellen bei 5 x 105 Zellen/mL in kompletten Medium. Samen 1,5 mL Zellsuspension in einem 35 mm Petrischale permeationsfähigen zu CO2 und angepasst für live-imaging.
- Führen Sie die Infektion am Tag nach der Aussaat mit dem RSV-M2-1-GFP-Virus bei MOI 1, wie in 3.3 – 3.5 beschrieben (entfernen Sie das Medium, 500 µL zu 1 mL Inokulum, und die Probe bei 37 ° C inkubieren, während sanft schütteln für 2 h; das Inokulum zu entfernen und hinzufügen 1,5 mL MEM mit 2 % FCS). Die gewünschte Zeit (IBs startet erscheinen ab 10 h p.i.) inkubieren Sie die Zellen bei 37 ° C und 5 % CO2 .
- Heizen Sie die Inkubation Kammer von einem inversen Mikroskop mit 40 X 100 X Zielen bei 37 ° C, vor dem Einsetzen der Petrischale mit infizierten Zellen auf der Bühne ausgestattet. Öffnen Sie die CO2 -Versorgung und Fokus Stabilisierung warten.
- Durchführen Sie Bildgebung mit GFP-kompatible Filter, unter einer niedrigen Intensität und Bild Erregerfrequenz (von 1 bis 0,1 Bild pro min) zur Minimierung von Phototoxizität.
Ergebnisse
In dieser Arbeit beschrieben, ein detailliertes Protokoll zur Herstellung rekombinanter RSV-Viren mit dem Ausdruck eines fluoreszierenden Proteins (Abbildung 2). Im pRSV-GFP wurde das GFP-gen zwischen dem P und M gen eingeführt, wie für die Kirsche gen zuvor veröffentlichte Arbeit21beschrieben. In pRSV-M2-1-GFP das M2-gen war unangetastet gelassen und ein zusätzliches Gen Kodierung für M2-1-GFP zwischen SH und G Gene
Diskussion
Hier präsentieren wir eine Methode zur Rettung des rekombinanten RSV aus fünf Plasmide und ihre Verstärkung. Die Fähigkeit zu manipulieren, das Genom der Viren revolutioniert Virologie-Forschung, um Mutationen zu testen und ein zusätzliches Gen oder ein tagged viral Protein ausdrücken. Der RSV haben wir beschrieben und als ein Beispiel in diesem Artikel ist ein Virus mit dem Ausdruck ein Reportergen, RSV-GLP (unveröffentlicht) und drückt eine M2-1-Protein mit einem GLP-Tag12verschmolzen. R...
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Die Autoren danken Dr. Qin Yu von AstraZeneca R & D Boston, MA, USA, für die Bereitstellung der AZD4316 Droge. Die Autoren sind dankbar für die Cymages-Plattform für den Zugriff auf die ScanR Olympus Mikroskop, das durch unterstützt wurde aus der Region Ile-de-France (DIM eine Gesundheit) gewährt. Die Autoren erkennen Unterstützung von INSERM und der Universität Versailles Saint-Quentin.
Materialien
| Name | Company | Catalog Number | Comments |
| 35 mm µ dish for live cell imaging | Ibidi | 81156 | |
| A549 | ATCC | ATCC CCL-185 | |
| Avicel RC-591 | FMC BioPolymer | Avicel RC-591 | Technical and other information on Avicels is available at http://www.fmcbiopolymer.com. Store at room temperature. Protocol in step 4 is optimized for this reagent. |
| BSRT7/5 | not commercially available | See reference 22. Buchholz et al. 1999 | |
| Crystal violet solution | Sigma | HT90132 | |
| Fluorescence microscope for observations | Olympus | IX73 Olympus microscope | |
| Fluorescence microscope for videomicroscopy | Olympus | ScanR Olympus microscope | |
| HEp-2 | ATCC | ATCC CCL-23 | |
| HEPES ≥99.5% | Sigma | H3375 | |
| L-Glutamine (200 mM) | ThermoFisher Scientific | 25030024 | |
| LIPOFECTAMINE 2000 REAGENT | ThermoFisher Scientific | 11668019 | Protocol in step 2.3. is optimized for this reagent. |
| MEM (10x), no glutamine | ThermoFisher Scientific | 11430030 | |
| MEM, GlutaMAX Supplement | ThermoFisher Scientific | 41090-028 | |
| MgSO4 ReagentPlus, ≥99.5% | Sigma | M7506 | |
| Opti-MEM I Reduced Serum Medium | ThermoFisher Scientific | 51985-026 | |
| Paraformaldehyde Aqueous Solution, 32%, EM Grade | Electron Microscopy Sciences | 15714 | |
| Penicillin-Streptomycin (10,000 U/mL) | ThermoFisher Scientific | 15140122 | |
| Plasmids | not commercially available | See reference 21. Rameix-Welti et al. 2014 | |
| See Saw Rocker | VWR | 444-0341 | |
| Si RNA GAPDH | Dharmacon | ON-TARGETplus siRNA D-001810-10-05 | SMARTpool and 3 of 4 individual siRNAs designed by Dharmacon. |
| Si RNA IMPDH2 | Dharmacon | ON-TARGETplus siRNA IMPDH2 Pool- Human L-004330-00-0005 | SMARTpool of 4 individual siRNAs designed by Dharmacon. Individual references and sequences J-004330-06: GGAAAGUUGCCCAUUGUAA; J-004330-07: GCACGGCGCUUUGGUGUUC; J-004330-08: AAGGGUCAAUCCACAAAUU; J-004330-09: GGUAUGGGUUCUCUCGAUG; |
| Si RNA RSV N | Dharmacon | ON-TARGETplus custom siRNA | UUCAGAAGAACUAGAGGCUAU and UUUCAUAAAUUCACUGGGUUA |
| SiRNA NT | Dharmacon | ON-TARGETplus Non-targeting Pool | |
| SiRNA transfection reagent | Dharmacon | DharmaFECT 1 Ref: T-2001-03 | Protocol in steps 5.1.and 5.1.2 are optimized for this reagent. |
| Sodium Bicarbonate 7.5% solution | ThermoFisher Scientific | 25080094 | |
| Spectrofluorometer | Tecan | Tecan infinite M200PRO |
Referenzen
- Shi, T., et al. Global, regional, and national disease burden estimates of acute lower respiratory infections due to respiratory syncytial virus in young children in 2015: a systematic review and modelling study. The Lancet. 390 (10098), 946-958 (2017).
- Falsey, A. R., Hennessey, P. A., Formica, M. A., Cox, C., Walsh, E. E. Respiratory Syncytial Virus Infection in Elderly and High-Risk Adults. The New England Journal of Medicine. 352 (17), 1749-1759 (2005).
- DeVincenzo, J. P., et al. Activity of Oral ALS-008176 in a Respiratory Syncytial Virus Challenge Study. The New England Journal of Medicine. 373 (21), 2048-2058 (2015).
- DeVincenzo, J. P., et al. Oral GS-5806 Activity in a Respiratory Syncytial Virus Challenge Study. The New England Journal of Medicine. 371 (8), 711-722 (2014).
- Afonso, C. L., et al. Taxonomy of the order Mononegavirales: update 2016. Archives of Virology. 161 (8), 2351-2360 (2016).
- Collins, P. L., Hill, M. G., Cristina, J., Grosfeld, H. Transcription elongation factor of respiratory syncytial virus, a nonsegmented negative-strand RNA virus. Proceedings of the National Academy of Sciences of the United States of America. 93 (1), 81-85 (1996).
- Hoenen, T., et al. Inclusion bodies are a site of ebolavirus replication. Journal of Virology. 86 (21), 11779-11788 (2012).
- Heinrich, B. S., Cureton, D. K., Rahmeh, A. A., Whelan, S. P. Protein expression redirects vesicular stomatitis virus RNA synthesis to cytoplasmic inclusions. PLoS Pathogens. 6 (6), e1000958 (2010).
- Lahaye, X., et al. Functional Characterization of Negri Bodies (NBs) in Rabies Virus-Infected Cells: Evidence that NBs Are Sites of Viral Transcription and Replication. Journal of Virology. 83 (16), 7948-7958 (2009).
- Kolesnikova, L., Mühlberger, E., Ryabchikova, E., Becker, S. Ultrastructural organization of recombinant Marburg virus nucleoprotein: comparison with Marburg virus inclusions. Journal of Virology. 74 (8), 3899-3904 (2000).
- Dolnik, O., Stevermann, L., Kolesnikova, L., Becker, S. Marburg virus inclusions: A virus-induced microcompartment and interface to multivesicular bodies and the late endosomal compartment. European Journal of Cell Biology. 94 (7-9), 323-331 (2015).
- Rincheval, V., et al. Functional organization of cytoplasmic inclusion bodies in cells infected by respiratory syncytial virus. Nature Communications. 8 (1), 563 (2017).
- Santangelo, P. J., Bao, G. Dynamics of filamentous viral RNPs prior to egress. Nucleic Acids Research. 35 (11), 3602-3611 (2007).
- Lifland, A. W., et al. Human Respiratory Syncytial Virus Nucleoprotein and Inclusion Bodies Antagonize the Innate Immune Response Mediated by MDA5 and MAVS. Journal of Virology. 86 (15), 8245-8258 (2012).
- Garcia, J., Garcia-Barreno, B., Vivo, A., Melero, J. A. Cytoplasmic inclusions of respiratory syncytial virus-infected cells: formation of inclusion bodies in transfected cells that coexpress the nucleoprotein, the phosphoprotein, and the 22K protein. Virology. 195 (1), 243-247 (1993).
- Brown, G., et al. Evidence for an association between heat shock protein 70 and the respiratory syncytial virus polymerase complex within lipid-raft membranes during virus infection. Virology. 338 (1), 69-80 (2005).
- Radhakrishnan, A., et al. Protein analysis of purified respiratory syncytial virus particles reveals an important role for heat shock protein 90 in virus particle assembly. Molecular & Cellular Proteomics. 9 (9), 1829-1848 (2010).
- Racaniello, V. R., Baltimore, D. Cloned poliovirus complementary DNA is infectious in mammalian cells. Science. 214 (4523), 916-919 (1981).
- Schnell, M. J., Mebatsion, T., Conzelmann, K. K. Infectious rabies viruses from cloned cDNA. The EMBO Journal. 13 (18), 4195-4203 (1994).
- Collins, P. L., et al. Production of infectious human respiratory syncytial virus from cloned cDNA confirms an essential role for the transcription elongation factor from the 5' proximal open reading frame of the M2 mRNA in gene expression and provides a capability for vaccine. Proceedings of the National Academy of Sciences of the United States of America. 92 (25), 11563-11567 (1995).
- Rameix-Welti, M. -. A., et al. Visualizing the replication of respiratory syncytial virus in cells and in living mice. Nature Communications. 5, 5104 (2014).
- Buchholz, U. J., Finke, S., Conzelmann, K. K. Generation of bovine respiratory syncytial virus (BRSV) from cDNA: BRSV NS2 is not essential for virus replication in tissue culture, and the human RSV leader region acts as a functional BRSV genome promoter. Journal of Virology. 73 (1), 251-259 (1999).
- Cianci, C., Meanwell, N., Krystal, M. Antiviral activity and molecular mechanism of an orally active respiratory syncytial virus fusion inhibitor. Journal of Antimicrobial Chemotherapy. 55 (3), 289-292 (2005).
- Derscheid, R. J., et al. Human respiratory syncytial virus memphis 37 grown in HEp-2 cells causes more severe disease in lambs than virus grown in vero cells. Viruses. 5 (11), 2881-2897 (2013).
- McKimm-Breschkin, J. L. A simplified plaque assay for respiratory syncytial virus - direct visualization of plaques without immunostaining. Journal of Virological Methods. 120, 113-117 (2004).
- Matrosovich, M., Matrosovich, T., Garten, W., Klenk, D. New low-viscosity overlay medium for viral plaque assays. Virology Journal. 7, 1-7 (2006).
- Novina, C. D., Sharp, P. A. The RNAi revolution. Nature. 430 (6996), 161-164 (2004).
- Sintchak, M. D., Nimmesgern, E. The structure of inosine 5'-monophosphate dehydrogenase and the design of novel inhibitors. Immunopharmacology. 47 (2-3), 163-184 (2000).
- Beaucourt, S., Vignuzzi, M. Ribavirin: A drug active against many viruses with multiple effects on virus replication and propagation. Molecular basis of ribavirin resistance. Current Opinion in Virology. 8, 10-15 (2014).
- Hruska, J. F., Bernstein, J. M., Douglas, R. G., Hall, C. B. Effects of Ribavirin on Respiratory Syncytial Virus in vitro. Antimicrobial Agents and Chemotherapy. 17 (5), 770-775 (1980).
- Simões, E. A. F., et al. Past, Present and Future Approaches to the Prevention and Treatment of Respiratory Syncytial Virus Infection in Children. Infectious Diseases and Therapy. 7 (1), 87-120 (2018).
- Alvarez, R., et al. RNA interference-mediated silencing of the respiratory syncytial virus nucleocapsid defines a potent antiviral strategy. Antimicrobial Agents and Chemotherapy. 53 (9), 3952-3962 (2009).
- DeVincenzo, J., et al. A randomized, double-blind, placebo-comtrolled study of an RNAi-based therapy directed against respiratory syncytial virus. Proceedings of the National Academy of Sciences of the United States of America. 107 (19), 8800-8805 (2010).
- Zhou, Y., et al. High-throughput screening of a CRISPR/Cas9 library for functional genomics in human cells. Nature. 509 (7501), 487-491 (2014).
- Nikolic, J., et al. Negri bodies are viral factories with properties of liquid organelles. Nature Communications. 8 (1), 58 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten