È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Generazione, amplificazione e titolazione del virus sinciziale respiratorio ricombinante
In questo articolo
Riepilogo
Descriviamo un metodo per la generazione e amplificando geneticamente modificate virus sinciziale respiratorio (ad) e un saggio della placca ottimizzato per ad. Illustriamo questo protocollo creando due virus ricombinanti che rispettivamente permettono di quantificazione della replica di RSV e live analisi dei corpi di inclusione di RSV e dinamiche di granuli di corpi di inclusione-collegata.
Abstract
L'uso di virus ricombinanti è diventato cruciale in virologia di base o applicata. Genetica inversa ha dimostrata di essere una tecnologia estremamente potente, sia per decifrare i meccanismi di replicazione virale e per studiare farmaci antivirali o fornire la piattaforma di sviluppo per i vaccini. La costruzione e la manipolazione di un sistema di genetico inverso per un negativo-strand RNA virus come un virus respiratorio sinciziale (RSV), tuttavia, rimane delicate e richiede particolare know-how. Il genoma di RSV è un RNA single-strand, senso negativo di circa 15 kb che serve come modello per sia virale RNA replicazione e trascrizione. Il nostro sistema di genetica inversa utilizza una copia del cDNA del genoma di ceppo lunga RSV (HRSV). Questo cDNA, come pure di cDNA codificanti proteine virali della polimerasi complessa (L, P, N e M2-1), vengono inseriti in vettori di espressione individuale sotto sequenze di controllo della polimerasi di T7. La transfezione di questi elementi in cellule di BSR-T7/5, che esprimono stabilmente della polimerasi T7, consente la replica citoplasmatica e trascrizione di RSV ricombinante, dando origine a virioni geneticamente. Una nuova RSV, che è presente alla superficie delle cellule e nel surnatante cultura di BSRT7/5, si è riunita per infettare cellule HEp-2 per l'amplificazione virale. Due o tre cicli di amplificazione sono necessari per ottenere stock virali contenenti 1 x 106 a 1 x 107 unità formanti placca (PFU) / mL. Metodi per l'ottimale raccolta, congelamento e la titolazione degli stock virali sono descritti qui in dettaglio. Illustriamo il protocollo presentato qui creando due virus ricombinante rispettivamente esprimendo gratis proteina fluorescente verde (GFP) (RSV-GFP) o virale M2-1 fuso a GFP (RSV-M2-1-GFP). Vi mostriamo come utilizzare RSV-GFP per quantificare RSV replica e la RSV-M2-1-GFP per visualizzare strutture virali, come pure le dinamiche di proteina virale in cellule vive, utilizzando tecniche di microscopia dei video.
Introduzione
RSV umana è la principale causa di ricovero in ospedale per infezione acuta delle vie respiratorie nei neonati nel mondo1. Inoltre, RSV è associato a un onere notevole malattia negli adulti paragonabili all'influenza, con la maggior parte dell'ospedalizzazione e l'onere di mortalità in anziani2. Non esistono vaccini o farmaci antivirali specifici disponibili ancora contro RSV, ma promettendo nuovi farmaci sono in sviluppo3,4. La complessità e la pesantezza delle tecniche di quantificazione di moltiplicazione RSV ostacolare la ricerca di antivirali o vaccini nonostante i notevoli sforzi attuali. La quantificazione della moltiplicazione di RSV in vitro è generalmente basata su metodi laboriosi, lunghi e costosi, che consistono principalmente nell'analisi dell'effetto citopatico da microscopia, immunostaining, riduzione della placca saggi, quantitative trascrittasi inversa (qRT)-reazione a catena della polimerasi (PCR) ed enzima-collegata dell'immunosorbente test di analisi. Virus con genomi modificati e che esprimono i geni reporter, come quelli codificanti per la GFP, sono più adatti per tali proiezioni. Accoppiato all'uso di lettori di micropiastre automatizzati, virus ricombinante di reporter gene-trasportare può rendere queste analisi più adatta per scopi di alto-rendimento e standardizzazione.
RSV è un involucro,-senso virus a RNA che appartiene al genere di Orthopneumovirus di Pneumoviridae famiglia, ordine Mononegavirales5. Il genoma di RSV è un RNA single-strand, senso negativo di circa 15 kb, che contiene una regione non codificante alle estremità 3' e 5' chiamato Leader e Trailer e 10 unità trascrizionale codifica 11 proteine. I geni vengono ordinati come segue: 3'-NS1, NS2, N, P, M, SH, G, F, M2 (codifica per le proteine M2-1 e M2-2) e L-5'. il RNA genomico è strettamente confezionato dalla nucleoproteina N. Using il RNA genomico incapsidato come un modello, in modo virale RNA polimerasi RNA-dipendente (RdRp) trascrizione e replicazione del RNA virale. RdRp virale è composta da grande proteina L, che trasporta l'attività della polimerasi del nucleotide per sé, suo cofattore obbligatorio la fosfoproteina P e la proteina M2-1 che funziona come una trascrizione virale fattore6. Nelle cellule infette, RSV induce la formazione di inclusioni citoplasmiche chiamati corpi di inclusione (IBs). Le inclusioni citoplasmiche morfologicamente simili sono state osservate per diversi Mononegavirales7,8,9,10. Recenti studi sul virus della rabbia, virus della stomatite vescicolare (VSV), il virus di Ebola e RSV ha mostrato che la sintesi di RNA virale si presenta in IBs, che può dunque essere considerata come fabbriche virali8,9,11, 12. le fabbriche di virus concentrare il RNA e proteine virali, necessarie per la sintesi del RNA virale e contengono anche proteine cellulari13,14,15,16, 17. IBs esibiscono un subcompartment funzionale chiamato granuli IB-collegato (IBAGs), che si concentrano il recentemente sintetizzato mRNA virale nascente insieme con la proteina M2-1. il RNA genomico e la L, P e N non vengono rilevati in IBAGs. IBAGs sono piccole strutture sferiche dinamici all'interno di IBs che presentano le proprietà di organelli liquido12. Nonostante il ruolo centrale di IBs in moltiplicazione virale, pochissimo è conosciuto circa la natura, struttura interna, formazione e funzionamento di queste fabbriche virali.
L'espressione del genoma di un poliovirus da un cDNA ha permesso la produzione del primo clone virale infettiva nel 198118. Per singolo filamento negativo virus del RNA, non era fino al 1994 che la produzione di un primo virus di rabbia dopo trasfezione di plasmidi in cellule19 ha avuto luogo. Il sistema genetico inverso basato su plasmide primo per RSV è stato pubblicato nel 199520. Genetica inversa hanno portato a grandi progressi nel campo della virologia. La possibilità di introdurre modifiche specifiche nel genoma virale ha fornito le comprensioni critiche nella replica e nella patogenesi del virus del RNA. Questa tecnologia inoltre notevolmente ha facilitato lo sviluppo di vaccini permettendo attenuazione specifici attraverso una serie mirata di modifiche. Modifiche di genoma permettendo una rapida quantificazione della moltiplicazione virale notevolmente migliorato lo screening antivirale e studio della loro modalità di azione.
Anche se descritto in precedenza, ottenendo geneticamente ad rimane delicato. Qui, dettagliamo un protocollo per creare due tipi di HRSV ricombinante, che esprimono rispettivamente RSV-GFP o RSV-M2-1-GFP. In questo protocollo, descriviamo le condizioni di transfezione necessarie per salvare i nuovi virus ricombinanti, come pure la loro amplificazione per ottenere stock virali con alto titolo, adatto per sperimentazioni riproducibile. La costruzione di vettori di genetica inversa di per sé non è descritto qui. Descriviamo i metodi per la raccolta ottimale e il blocco di stock virali. Il metodo più preciso per quantificare le particelle virali infettive rimane saggio della placca. Le cellule sono infettate con diluizioni seriali della sospensione analizzata e incubate con un overlay che vieta la diffusione di particelle virali libere nel surnatante. In tali condizioni, il virus infetterà solo celle contigue, formando una "targa" per ogni particella infettiva. Nell'analisi della titolazione RSV convenzionale, le piastre sono rivelate immunostaining e conteggiate sotto osservazione microscopica. Questo metodo è costoso e richiede tempo. Qui abbiamo descritto un protocollo molto semplice per un saggio della placca di RSV mediante sovrapposizione di cellulosa microcristallina che consente la formazione di placche visibili ad occhio nudo. Vi mostriamo come RSV-GFP può essere utilizzato per misura RSV replica e, quindi, di quantificare l'impatto di antivirali. Combinazione di genetica inversa e diretta tecnologia di imaging, dimostriamo come RSV-M2-1-GFP permette agli scienziati di visualizzare M2-1 in cellule vive e di seguire la dinamica di strutture virali intracellulari, come IBs.
Protocollo
1. materiale preparazione
- Acquisto di media delle cellule (siero ridotto mezzi di comunicazione, minimo essenziale [MEM], 10 x MEM e Dulbecco s modified esente Eagle [DMEM]), reagente di transfezione e cellulosa microcristallina (Vedi Tabella materiali).
- Ottenere i seguenti vettori per genetica inversa: la genomica tutt'e i vettori di espressione che codifica la proteina di N e le proteine del complesso della polimerasi. I vettori genomici contengono il genoma completo del cDNA della RSV-GFP (GFP-RSV-p) e della RSV-M2-1GFP (p-RSV-M2-1GFP) a valle dal promotore del batteriofago T7 RNA polimerasi (T7 pol). I vettori di espressione (designato come p-N, p -P, p-L e p-M2-1) contengono la sequenza di codificazione di N, P, L o M2-1 a valle il pol T7 (Vedi Rincheval et al.12 e Rameix-Welti et al.21 per i dettagli riguardanti i costrutti di plasmide).
- Preparare i supporti per una coltura cellulare in un ambiente sterile e per la transfezione e infezione. Uso DMEM con 2 mM L-Glutammina completati con 10% siero fetale di vitello (FCS), 1.000 unità/mL di penicillina e streptomicina 1 mg/mL (o senza antibiotici) e MEM con 2 mM L-Glutammina completati con 0%, 2% o 10% FCS, 1.000 unità/mL di penicillina e 1 mg/mL streptomicina, designata come "medie completa" il seguente protocollo.
- Ottenere cellule22 BSRT7/5 e fanno scorte in completa supplementato con 10% di dimetilsolfossido (DMSO) a 1-2 x 106 cellule/mL. Conservare le scorte delle cellule in azoto liquido. Ottenere cellule HEp-2. Cellule di BSRT7/5 di cultura in DMEM completo e cellule HEp-2 in MEM completa a 37 ° C e 5% di CO2 in un ambiente sterile.
- Preparare una soluzione di conservazione 10x RSV (0,5 M HEPES e 1m MgSO4 [pH 7.5] in acqua) in un ambiente sterile.
- Ottenere un microscopio a fluorescenza invertito compatibile con misure di fluorescenza di GFP e compatibile con live imaging se è necessario monitorare un'infezione. Ottenere un lettore di micropiastre compatibile con misure di fluorescenza di GFP per la quantificazione di replica RSV-GFP.
2. salvataggio e primo passaggio dei Virus ricombinante
Nota: Eseguire le seguenti operazioni in un ambiente sterile, utilizzando una classe di sicurezza II gabinetto.
- Il giorno prima di transfezione, fare una sospensione della linea cellulare BSRT7/5 a 5 x 105 cellule/mL in mezzo completo. Distribuire 2 mL della sospensione di cellule per pozzetto di una piastra a 6 pozzetti. Preparare un pozzetto al virus che sta per essere salvato e un ulteriore pozzetto per il controllo negativo. Incubare la piastra a 37 ° C e 5% CO2. Verificare che le cellule siano in una confluenza di 80 – 90% il giorno successivo.
- Sbloccare la genetica inversa vettori (dal punto 1.2) p-RSV-GFP e p-RSV-M2-1-GFP, così come p-N, p -P, p-L e p-M2-1. Mix, per ogni virus di salvataggio, 1 µ g di p-N e p -P, 0,5 µ g di p-L, 0,25 µ g di p-M2-1 e 1.25 µ g di p-RSV (GFP o GFP M2-1) in un tubo.
Nota: Possono essere utilizzati diversi vettori per N, P, L e M2-1; Tuttavia, il rapporto tra le proteine deve essere mantenuto. Eseguire il controllo negativo sostituendo il vettore p-RSV con un vettore vuoto. -
Procedere alla transfezione, seguendo il protocollo del produttore del reagente di transfezione (Vedi tabella materiali).
- Aggiungere 250 µ l di siero riduttore terreno per i vettori misti. In un altro tubo, diluire 10 µ l di reagente di transfezione in 250 µ l di siero riduttore terreno. Vortice delicatamente tubi e aspettare per 5 min, miscela il contenuto di entrambe tubi e aspettare 20 minuti a temperatura ambiente.
- Sciacquare le cellule BSRT7/5 con 1 mL di siero riduttore terreno e distribuire 1,5 mL di MEM con 10% FCS senza antibiotici per pozzetto. Se necessario, incubare a 37 ° C e 5% CO2 finché non viene completata l'incubazione descritto al punto 2.3.1.
- Aggiungere 500 µ l della miscela transfezione preparata al punto 2.3.1 ad un pozzo, quando il tempo di incubazione di 20 min. Posizionare le cellule nell'incubatore a 37 ° C e 5% CO2 per 3 giorni. Non modificare il terreno di coltura delle cellule durante la transfezione.
- Osservare la fluorescenza di GFP (eccitazione a 488 nm) e emissione a 515 – 535 nm sotto un microscopio a fluorescenza invertito a 20 ingrandimenti 1 x al giorno per monitorare l'efficienza di salvataggio, utilizzando la GFP filtrare.
- Il secondo giorno dopo la trasfezione, le cellule per il primo passaggio dei virus hanno salvato del seme. Preparare una sospensione di cellule HEp-2 a 5 x 105 cellule/mL in terreno completo. Distribuire 2 mL della sospensione di cellule per pozzetto di una piastra a 6 pozzetti (un pozzetto al virus di salvataggio e un controllo negativo).
- Il terzo giorno di transfezione, gratta e Vinci cellule in ciascun pozzetto della piastra 6 pozzetti BSRT7/5 transfettata, utilizzando un raschietto differente per ciascun pozzetto. Trasferire ogni contenuto ben (cellule e surnatante) in una provetta da microcentrifuga sterile mL 2. Vortexare ciascuna provetta vigorosamente per almeno 30 s per rilasciare il virus hanno salvato dalle membrane cellulari.
Nota: Questo corrisponde al passaggio 0 (P0) del virus hanno salvato (Figura 1). -
Utilizzare la sospensione virale fresca di P0 per eseguire il primo amplificazione dei virus hanno salvato.
- Rimuovere il terreno di coltura dalla piastra a 6 pozzetti HEp-2 teste di serie il giorno prima (Vedi punto 2.5) e rapidamente aggiungere 500 µ l della sospensione per pozzetto P0 (dal punto 2.6). Posizionare la piastra di HEp-2 a 37 ° C su un rocker di altalena per agitazione morbido per 2 h.
- Rimuovere e scartare i 500 µ l di inoculo e aggiungere 2 mL di MEM con 2% di FCS. Incubare la piastra a 37 ° C e 5% CO2 per 3 giorni. Questo produrrà il primo passaggio (P1) dei virus hanno salvato (Figura 1).
- Aggiungere 1/10 del volume di soluzione di conservazione di RSV x 10 (0,5 M HEPES e 1m MgSO4 [pH 7.5]) nella sospensione rimanente P0 (dal punto 2.6). Vortice microprovette vigorosamente per 5 s e aliquota il contenuto nei tubi criogenici etichettati con tag resistente all'alcool. Immergere le provette per almeno 1 h in alcool preraffreddata a-80 ° C e conservarli a-80 ° C.
- Titolo lo stock di P0 (Vedi punto 2.6) di ogni virus hanno salvato (vedere sezione 4 per la titolazione di cellulosa microcristallina).
- Osservare la fluorescenza di GFP (eccitazione a 488 nm) e emissione a 515 – 535 nm delle cellule HEp-2 infettati con la sospensione di P0 sotto un microscopio a fluorescenza invertito a 20 ingrandimenti 1 x al giorno per controllare l'infezione. Osservare sotto un microscopio a campo chiaro l'aspetto di piccoli sincizi e distacco che riflette l'effetto citopatogeno RSV (CPE) di cella (Vedi Figura 2).
- Si noti che il salvataggio non è riuscita se fluorescenza né CPE è visibile dopo 2 – 3 giorni.
- Raccogliere il primo passaggio (P1) al giorno 3 o 4 come descritto al punto 2.6. In breve, raschiare le cellule, raccogliere le cellule e il surnatante insieme, vortice, aggiungere la soluzione di conservazione come descritto al punto 2.8, aliquota del campione e congelarlo.
- Titolo (vedere sezione 4 per il dosaggio di titolazione) e amplificare il primo passaggio (vedere la sezione 3 per l'amplificazione).
3. amplificazione dei virus hanno salvato
Nota: Il seguente protocollo descrive l'amplificazione dei virus salvati in una boccetta di2 cm 75. Adattare le dimensioni della muffola per il volume necessario e la necessaria molteplicità di infezione (MOI). La tabella 1 indica volumi per Boccette differenti. Eseguire le seguenti operazioni in un ambiente sterile in un armadietto di sicurezza II classe.
- Preparare una sospensione di cellule HEp-2 a 5 x 105 cellule/mL in terreno completo, il giorno prima l'amplificazione. Distribuire 15 mL della sospensione delle cellule per fiaschetta2 75 cm e incubare i matracci a 37 ° C e 5% CO2. Preparare una boccetta al virus per amplificare.
- Il giorno dopo l'inizio dell'incubazione, verifica che le cellule sono 80% – 100% confluenti.
- Diluire la sospensione virale dal punto 2.12 in MEM senza FCS per ottenere una sospensione di 3 mL alle 50.000 PFU/mL (corrispondente a un MOI di 0.01 PFU/cella).
- Rimuovere il mezzo e di aggiungere rapidamente il 3 mL di sospensione virale. Posizionare il pallone a 37 ° C su un rocker di altalena per agitazione morbido per 2 h.
- Rimuovere e scartare l'inoculo e aggiungere 15 mL di MEM con 2% di FCS. Incubare a 37 ° C e 5% CO2 per 2 – 4 giorni.
- Controllare la morfologia delle cellule e la fluorescenza di GFP (eccitazione a 488 nm) e emissione a 515 – 535 nm sotto un microscopio a fluorescenza invertito a 20 ingrandimenti al fine di stimare il momento giusto per raccogliere i virus. Si noti che questo è di solito quando 50% – 80% dello strato di cellule HEp-2 è staccato a causa del CPE di RSV che si verifica tra 48 e 72 h postinfection (p.i.) (Vedi Figura 3).
- Raschiare tutte le celle utilizzando un raschietto di cella. Raccogliere insieme sia le cellule e il surnatante e trasferirli in una provetta da centrifuga 50 mL.
- Aggiungere 1/10 del volume di 10 x soluzione di conservazione RSV (0,5 M HEPES e 1m MgSO4 [pH 7.5]). Vortice i tubi energicamente per 5 s e chiarire la sospensione di una centrifugazione di 5 min a 200 x g.
- Trasferire il surnatante in una provetta 50 mL. Vortex brevemente e aliquota della sospensione in provette criogeniche etichettati con tag resistente all'alcool. Immergere le provette in alcool preraffreddato-80 ° C per almeno 1 h e conservarli a-80 ° C.
- Sblocca una delle aliquote a titolare la sospensione virale (Vedi sezione 4).
4. placca titolazione dosaggio
- Preparare 12-pozzetti piastre per la titolazione il giorno prima che venga eseguito il test di titolazione (sei pozzi saranno necessario titolo uno tubo di virus). I pozzetti con 1 mL di cellule HEp-2 a 5 x 105 cellule/mL in sostanza completa del seme.
-
Il giorno successivo, preparare una sospensione sterile cellulosa microcristallina (2,4% [p/v] in acqua) (Vedi tabella materiali).
- Disperdere 2,4 g di polvere di cellulosa microcristallina in 100 mL di acqua distillata, utilizzando un agitatore magnetico standard, fino al completo scioglimento della polvere (solitamente 4 – 12 h). Autoclave la sospensione a 121 ° C per 20 min e conservarla a temperatura ambiente prima dell'uso.
Nota: In tali condizioni, la sospensione è stabile per 1 anno. - Dopo aver aperto la soluzione in un ambiente sterile, conservarlo a 4 ° C per 6 mesi. Mescolare sempre la sospensione prima dell'uso (con mano tremante o Vortex) per assicurarsi che sia omogeneo.
- Disperdere 2,4 g di polvere di cellulosa microcristallina in 100 mL di acqua distillata, utilizzando un agitatore magnetico standard, fino al completo scioglimento della polvere (solitamente 4 – 12 h). Autoclave la sospensione a 121 ° C per 20 min e conservarla a temperatura ambiente prima dell'uso.
- Preparare il 2 x MEM in un ambiente sterile. Diluire commerciale MEM 10 x con acqua sterile e aggiungere L-glutamina, 1.000 unità/mL di penicillina e streptomicina 1 mg/mL. Agitare energicamente la diluizione e conservarla a 4 ° C.
Nota: Eseguire passaggi 4.4 – 4.10 in un ambiente sterile utilizzando una classe di sicurezza II gabinetto. - Preparare sei provette contenenti 900 µ l di MEM senza FCS per virus da dosare (i tubi di titolazione). Scongelare le aliquote di virus, vortexare vigorosamente per 5 s e trasferire 100 µ l alla titolazione prima provetta.
- Eseguire una diluizione dieci volte 6 x, come indicato di seguito. Aggiungere 100 μL di virus a 900 μL di mezzo nel primo tubo, mettere il tappo sul tubo e mescolare il contenuto nel Vortex per pochi secondi. Cambiare la punta sulla pipetta, aggiungere 100 µ l della diluizione prima a 900 µ l di terreno nel secondo tubo, mettere il tappo sul tubo e vortice. Ripetere la procedura fino a quando il tubo di Sesto.
Nota: È molto importante cambiare la punta per ogni diluizione. - Scrivere il virus nome e le diluizioni di piega sulle piastre 12-pozzetti HEp-2. Aggiungere un segno per abbinare la piastra e la sua copertura, perché possono essere separate durante la colorazione (passo 4.9). Rimuovere il mezzo delle placche e distribuire 400 µ l di una diluizione per pozzetto. Incubare le piastre a 37 ° C per 2 h, per adsorbimento del virus.
Nota: Cambiare la punta della pipetta tra ciascun inoculo o procedere dalla più bassa alla più alta concentrazione con la stessa punta. Inoculare una serie limitata di piastre (1 o 2) contemporaneamente per evitare le celle di essiccazione. -
Preparare la sovrapposizione di cellulosa microcristallina durante l'adsorbimento del virus (preparazione estemporanea). Per ottenere 100 mL di sovrapposizione, mescolare 10 mL di sospensione 2,4% cellulosa microcristallina, 10 mL di 2x MEM e 80 mL di MEM con 2% di FCS.
- Regolare il pH del 2 x MEM per circa 7,2 con una soluzione di bicarbonato di sodio sterile al 7,5%, seguono l'indicatore di colore. Aggiungere la sospensione di cellulosa microcristallina e la MEM e mescolare energicamente.
- Alla fine dell'incubazione 2h, aggiungere 2 o 3 mL di sovrapposizione in tutti i pozzetti delle piastre 12-pozzetti senza rimuovere l'inoculo. Fare attenzione a evitare la contaminazione dei pozzetti adiacenti con Inoculanti da alto titolo virale. Incubare la piastra a 37 ° C e 5% CO2 per 6 giorni. Non spostare la piastra e non spostare l'incubatore durante l'incubazione.
-
Procedere a macchiare le celle, utilizzando la soluzione al cristalvioletto (8% cristallo viola [v/v], 2% formaldeide [v/v] e [v/v] etanolo al 20% in acqua).
- Proteggere la superficie di lavoro della cappa di biosicurezza con un foglio (la viola di cristallo colori fortemente superfici).
- Agitare delicatamente le piastre a decollare la sovrapposizione di cellulosa microcristallina. I surnatanti di rimuovere e lavare le cellule 2 x con 1x tampone fosfato salino (PBS). Gestire i piatti uno per uno per evitare le celle di essiccazione. Aggiungere 1 – 2 mL della soluzione di cristalvioletto e attendere 10 – 15 min. Rimuovi la soluzione, che può essere riutilizzata per successiva piastra di colorazione.
- Immergere i piatti e coperchi in candeggina fresco per pochi secondi e, quindi, lavarle accuratamente con acqua del rubinetto. Si noti che le piastre e le coperture sono decontaminate dalla candeggina.
- Mettere i piatti e coperchi su carta assorbente e lasciarli asciugare. Dopo il risciacquo con acqua, asciugare le piastre a temperatura ambiente e conservarle a temperatura ambiente. Per lunghi periodi di stoccaggio (mesi), tenere le piastre al riparo dalla luce per proteggere il colore. Si noti che se le cellule perdono la loro colorazione, può essere macchiati nuovamente con cristallo viola.
- Calcolare i titoli del virus. Contare le placche nei pozzetti delle piastre asciutte, che sono visibili ad occhio nudo. Verifica che il numero delle placche delle diluizioni differenti è coerente (fattore 10 tra ogni diluizione). Scegliere il bene su cui le targhe sono il metodo più semplice per contare. Valutare il numero delle placche contro il volume di inoculo e la diluizione.
Nota: Nell'esempio riportato in Figura 4, 21 placche sono contate con la diluizione 10-5 . Questi corrispondono a un titolo di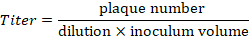
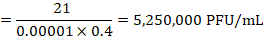
5. l'uso di Virus ricombinanti HRSV-GFP per monitorare la replica virale nelle cellule trattate con piccolo RNA interferente o antivirali
Nota: Eseguire tutti i passaggi tranne 5.1 e 5.2.5 in un ambiente sterile utilizzando una classe di sicurezza II gabinetto.
-
L'effetto di silenziamento genico cellulare sulla moltiplicazione di RSV di monitoraggio
Nota: Il protocollo di transfezione dipende il reagente (Vedi Tabella materiali).- Preparare piastre da 96 pozzetti per la misurazione di GFP. Due giorni prima del saggio, per dato piccolo RNA interferente (siRNA), preparare una soluzione di supporto riduttore del siero contenente siRNA ad una concentrazione di 100 nM e un reagente di transfezione di siRNA diluito a 1/500. Incubare la soluzione a temperatura ambiente per 30 minuti.
- Aggiungere 25 µ l della soluzione ai pozzetti della piastra preparato in 5.1.1 (in triplice copia). Seme i pozzetti con 75 µ l di una sospensione di cellule A549 a 4 x 105 cellule/mL in terreno completo senza antibiotici per ottenere una concentrazione finale delle cellule di 3 x 105 cellule/mL. Incubare la piastra per 48 h a 37 ° C e 5% CO2.
Nota: La concentrazione finale siRNA è di 25 nM e il volume di reagente di transfezione finale è 0,5 µ l/pozzetto). - Infettare le cellule come segue. Rimuovere il supporto dai pozzi. Aggiungere 100 µ l della sospensione di RSV-GFP a 50.000 PFU/mL e incubare per 2 ore a 37 ° C e 5% CO2. Rimuovere la sospensione virale e aggiungere 100 µ l di DMEM con 2% FCS e senza rosso fenolo. Incubare la piastra a 37 ° C e 5% CO2.
- Alle h 24 e 48 h p.i., misurare la fluorescenza, utilizzando una Spettrofluorimetro impostata su lunghezze d'onda di eccitazione e di emissione di 488 e 520 nm, rispettivamente (fluorescenza è espressa in unità di fluorescenza relativa). Utilizzare non infette cellule A549 come standard per i livelli di fluorescenza e sfondo.
Nota: Le cellule hanno bisogno di essere fissato con paraformaldeide al 4% (PFA) prima di misurare la loro senza il coperchio della piastra.
-
Valutazione dell'inibizione della droga usando RSV-GFP
- Preparare piastre da 96 pozzetti per la misurazione di GFP. Il giorno prima del saggio, del seme i pozzetti con 100 µ l di una sospensione di cellule HEp-2 a 5 x 105 cellule/mL in terreno completo senza rosso fenolo.
- Preparare una diluizione seriale della testata (AZ4316 in questo esempio) la droga in MEM completata con 2% FCS e antibiotici (50 µ l per pozzetto). Preparare una sospensione virale a 10.000 PFU/mL in MEM senza fattore vascolare stromal (SVF) e senza rosso fenolo (50 µ l per pozzetto).
- Rimuovere il mezzo da 96 pozzetti HEp-2 piastra e aggiungere 50 µ l della sospensione di droga e 50 µ l della sospensione virale (in triplice copia). Eseguire un'infezione fittizia in parallelo come un controllo.
Nota: La diluizione di droga e sospensione virale possono essere mescolati prima della loro aggiunta sulle cellule, o possono essere aggiunti in sequenza. - Incubare la piastra per 48 h a 37 ° C e 5% CO2.
- Misurare la fluorescenza, utilizzando una spettrofluorimetro come descritto al punto 5.1.4. Utilizzare mock-infettato cellule HEp-2 come standard per livelli di fluorescenza di fondo.
6. caratterizzazione di M2-1 localizzazione In Vivo con il Virus ricombinante RSV-M2-1-GFP
Nota: Eseguire i passaggi 6.1 e 6.2 in un ambiente sterile, utilizzando una classe di sicurezza II gabinetto.
- Preparare una sospensione di cellule HEp-2 a 5 x 105 cellule/mL in terreno completo. Seme di 1,5 mL di sospensione cellulare in un 35 mm di Petri permeante di CO2 e adattato per live imaging.
- Eseguire l'infezione il giorno dopo la semina con il virus RSV-M2-1-GFP a MOI 1, come descritto nei passaggi 3.3 – 3,5 (rimuovere il mezzo, aggiungere 500 µ l ad 1 mL di inoculo e incubare il campione a 37 ° C agitando delicatamente per 2 h; rimuovere l'inoculo e aggiungere 1,5 mL di MEM con 2 % FCS). Incubare le cellule a 37 ° C e 5% CO2 per il tempo desiderato (IBs inizieranno ad apparire da 10 h p.i.).
- Preriscaldare la camera di incubazione di un microscopio invertito dotato di 40 x 100 obiettivi x a 37 ° C, prima di mettere la capsula di Petri contenente le cellule infette sul palco. Aprire l'alimentazione di CO2 e attendere per la stabilizzazione di messa a fuoco.
- Eseguire imaging con filtri di GFP-compatibile, sotto una frequenza di eccitazione di bassa intensità e immagine (da 1 a 0,1 immagine al minuto) per ridurre al minimo la fototossicità.
Risultati
In questo lavoro, abbiamo descritto un protocollo dettagliato per produrre ricombinanti virus RSV che esprime una proteina fluorescente (Figura 2). OSPRO-GFP, il gene GFP è stato introdotto tra i geni P e M, come descritto per il gene della ciliegio in lavoro precedentemente pubblicato21. In OSPRO-M2-1-GFP, il gene di M2 è stato lasciato intatto e un ulteriore gene che codifica per M2-1-GFP è stata inserita tra i geni
Discussione
Qui presentiamo un metodo di salvataggio del ricombinante ad da cinque plasmidi e la loro amplificazione. La capacità di manipolare il genoma del virus ha rivoluzionato la ricerca virologia per testare le mutazioni ed esprimere un ulteriore gene o una proteina virale taggata. La RSV abbiamo descritto e utilizzato come esempio in questo articolo è un virus che esprime un gene reporter, RSV-GFP (non pubblicati) ed esprime una proteina M2-1 fuso a un tag GFP12. Salvataggio di RSV è impegnativo e r...
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Gli autori ringraziano il Dr. Qin Yu da AstraZeneca R & D Boston, MA, USA, per la fornitura di droga AZD4316. Gli autori sono grati alla piattaforma Cymages per l'accesso al microscopio ScanR Olympus, che è stata sostenuta da sovvenzioni dalla regione Ile-de-France (DIM ONE HEALTH). Gli autori riconoscono supporto da INSERM e l'Università di Versailles Saint-Quentin.
Materiali
| Name | Company | Catalog Number | Comments |
| 35 mm µ dish for live cell imaging | Ibidi | 81156 | |
| A549 | ATCC | ATCC CCL-185 | |
| Avicel RC-591 | FMC BioPolymer | Avicel RC-591 | Technical and other information on Avicels is available at http://www.fmcbiopolymer.com. Store at room temperature. Protocol in step 4 is optimized for this reagent. |
| BSRT7/5 | not commercially available | See reference 22. Buchholz et al. 1999 | |
| Crystal violet solution | Sigma | HT90132 | |
| Fluorescence microscope for observations | Olympus | IX73 Olympus microscope | |
| Fluorescence microscope for videomicroscopy | Olympus | ScanR Olympus microscope | |
| HEp-2 | ATCC | ATCC CCL-23 | |
| HEPES ≥99.5% | Sigma | H3375 | |
| L-Glutamine (200 mM) | ThermoFisher Scientific | 25030024 | |
| LIPOFECTAMINE 2000 REAGENT | ThermoFisher Scientific | 11668019 | Protocol in step 2.3. is optimized for this reagent. |
| MEM (10x), no glutamine | ThermoFisher Scientific | 11430030 | |
| MEM, GlutaMAX Supplement | ThermoFisher Scientific | 41090-028 | |
| MgSO4 ReagentPlus, ≥99.5% | Sigma | M7506 | |
| Opti-MEM I Reduced Serum Medium | ThermoFisher Scientific | 51985-026 | |
| Paraformaldehyde Aqueous Solution, 32%, EM Grade | Electron Microscopy Sciences | 15714 | |
| Penicillin-Streptomycin (10,000 U/mL) | ThermoFisher Scientific | 15140122 | |
| Plasmids | not commercially available | See reference 21. Rameix-Welti et al. 2014 | |
| See Saw Rocker | VWR | 444-0341 | |
| Si RNA GAPDH | Dharmacon | ON-TARGETplus siRNA D-001810-10-05 | SMARTpool and 3 of 4 individual siRNAs designed by Dharmacon. |
| Si RNA IMPDH2 | Dharmacon | ON-TARGETplus siRNA IMPDH2 Pool- Human L-004330-00-0005 | SMARTpool of 4 individual siRNAs designed by Dharmacon. Individual references and sequences J-004330-06: GGAAAGUUGCCCAUUGUAA; J-004330-07: GCACGGCGCUUUGGUGUUC; J-004330-08: AAGGGUCAAUCCACAAAUU; J-004330-09: GGUAUGGGUUCUCUCGAUG; |
| Si RNA RSV N | Dharmacon | ON-TARGETplus custom siRNA | UUCAGAAGAACUAGAGGCUAU and UUUCAUAAAUUCACUGGGUUA |
| SiRNA NT | Dharmacon | ON-TARGETplus Non-targeting Pool | |
| SiRNA transfection reagent | Dharmacon | DharmaFECT 1 Ref: T-2001-03 | Protocol in steps 5.1.and 5.1.2 are optimized for this reagent. |
| Sodium Bicarbonate 7.5% solution | ThermoFisher Scientific | 25080094 | |
| Spectrofluorometer | Tecan | Tecan infinite M200PRO |
Riferimenti
- Shi, T., et al. Global, regional, and national disease burden estimates of acute lower respiratory infections due to respiratory syncytial virus in young children in 2015: a systematic review and modelling study. The Lancet. 390 (10098), 946-958 (2017).
- Falsey, A. R., Hennessey, P. A., Formica, M. A., Cox, C., Walsh, E. E. Respiratory Syncytial Virus Infection in Elderly and High-Risk Adults. The New England Journal of Medicine. 352 (17), 1749-1759 (2005).
- DeVincenzo, J. P., et al. Activity of Oral ALS-008176 in a Respiratory Syncytial Virus Challenge Study. The New England Journal of Medicine. 373 (21), 2048-2058 (2015).
- DeVincenzo, J. P., et al. Oral GS-5806 Activity in a Respiratory Syncytial Virus Challenge Study. The New England Journal of Medicine. 371 (8), 711-722 (2014).
- Afonso, C. L., et al. Taxonomy of the order Mononegavirales: update 2016. Archives of Virology. 161 (8), 2351-2360 (2016).
- Collins, P. L., Hill, M. G., Cristina, J., Grosfeld, H. Transcription elongation factor of respiratory syncytial virus, a nonsegmented negative-strand RNA virus. Proceedings of the National Academy of Sciences of the United States of America. 93 (1), 81-85 (1996).
- Hoenen, T., et al. Inclusion bodies are a site of ebolavirus replication. Journal of Virology. 86 (21), 11779-11788 (2012).
- Heinrich, B. S., Cureton, D. K., Rahmeh, A. A., Whelan, S. P. Protein expression redirects vesicular stomatitis virus RNA synthesis to cytoplasmic inclusions. PLoS Pathogens. 6 (6), e1000958 (2010).
- Lahaye, X., et al. Functional Characterization of Negri Bodies (NBs) in Rabies Virus-Infected Cells: Evidence that NBs Are Sites of Viral Transcription and Replication. Journal of Virology. 83 (16), 7948-7958 (2009).
- Kolesnikova, L., Mühlberger, E., Ryabchikova, E., Becker, S. Ultrastructural organization of recombinant Marburg virus nucleoprotein: comparison with Marburg virus inclusions. Journal of Virology. 74 (8), 3899-3904 (2000).
- Dolnik, O., Stevermann, L., Kolesnikova, L., Becker, S. Marburg virus inclusions: A virus-induced microcompartment and interface to multivesicular bodies and the late endosomal compartment. European Journal of Cell Biology. 94 (7-9), 323-331 (2015).
- Rincheval, V., et al. Functional organization of cytoplasmic inclusion bodies in cells infected by respiratory syncytial virus. Nature Communications. 8 (1), 563 (2017).
- Santangelo, P. J., Bao, G. Dynamics of filamentous viral RNPs prior to egress. Nucleic Acids Research. 35 (11), 3602-3611 (2007).
- Lifland, A. W., et al. Human Respiratory Syncytial Virus Nucleoprotein and Inclusion Bodies Antagonize the Innate Immune Response Mediated by MDA5 and MAVS. Journal of Virology. 86 (15), 8245-8258 (2012).
- Garcia, J., Garcia-Barreno, B., Vivo, A., Melero, J. A. Cytoplasmic inclusions of respiratory syncytial virus-infected cells: formation of inclusion bodies in transfected cells that coexpress the nucleoprotein, the phosphoprotein, and the 22K protein. Virology. 195 (1), 243-247 (1993).
- Brown, G., et al. Evidence for an association between heat shock protein 70 and the respiratory syncytial virus polymerase complex within lipid-raft membranes during virus infection. Virology. 338 (1), 69-80 (2005).
- Radhakrishnan, A., et al. Protein analysis of purified respiratory syncytial virus particles reveals an important role for heat shock protein 90 in virus particle assembly. Molecular & Cellular Proteomics. 9 (9), 1829-1848 (2010).
- Racaniello, V. R., Baltimore, D. Cloned poliovirus complementary DNA is infectious in mammalian cells. Science. 214 (4523), 916-919 (1981).
- Schnell, M. J., Mebatsion, T., Conzelmann, K. K. Infectious rabies viruses from cloned cDNA. The EMBO Journal. 13 (18), 4195-4203 (1994).
- Collins, P. L., et al. Production of infectious human respiratory syncytial virus from cloned cDNA confirms an essential role for the transcription elongation factor from the 5' proximal open reading frame of the M2 mRNA in gene expression and provides a capability for vaccine. Proceedings of the National Academy of Sciences of the United States of America. 92 (25), 11563-11567 (1995).
- Rameix-Welti, M. -. A., et al. Visualizing the replication of respiratory syncytial virus in cells and in living mice. Nature Communications. 5, 5104 (2014).
- Buchholz, U. J., Finke, S., Conzelmann, K. K. Generation of bovine respiratory syncytial virus (BRSV) from cDNA: BRSV NS2 is not essential for virus replication in tissue culture, and the human RSV leader region acts as a functional BRSV genome promoter. Journal of Virology. 73 (1), 251-259 (1999).
- Cianci, C., Meanwell, N., Krystal, M. Antiviral activity and molecular mechanism of an orally active respiratory syncytial virus fusion inhibitor. Journal of Antimicrobial Chemotherapy. 55 (3), 289-292 (2005).
- Derscheid, R. J., et al. Human respiratory syncytial virus memphis 37 grown in HEp-2 cells causes more severe disease in lambs than virus grown in vero cells. Viruses. 5 (11), 2881-2897 (2013).
- McKimm-Breschkin, J. L. A simplified plaque assay for respiratory syncytial virus - direct visualization of plaques without immunostaining. Journal of Virological Methods. 120, 113-117 (2004).
- Matrosovich, M., Matrosovich, T., Garten, W., Klenk, D. New low-viscosity overlay medium for viral plaque assays. Virology Journal. 7, 1-7 (2006).
- Novina, C. D., Sharp, P. A. The RNAi revolution. Nature. 430 (6996), 161-164 (2004).
- Sintchak, M. D., Nimmesgern, E. The structure of inosine 5'-monophosphate dehydrogenase and the design of novel inhibitors. Immunopharmacology. 47 (2-3), 163-184 (2000).
- Beaucourt, S., Vignuzzi, M. Ribavirin: A drug active against many viruses with multiple effects on virus replication and propagation. Molecular basis of ribavirin resistance. Current Opinion in Virology. 8, 10-15 (2014).
- Hruska, J. F., Bernstein, J. M., Douglas, R. G., Hall, C. B. Effects of Ribavirin on Respiratory Syncytial Virus in vitro. Antimicrobial Agents and Chemotherapy. 17 (5), 770-775 (1980).
- Simões, E. A. F., et al. Past, Present and Future Approaches to the Prevention and Treatment of Respiratory Syncytial Virus Infection in Children. Infectious Diseases and Therapy. 7 (1), 87-120 (2018).
- Alvarez, R., et al. RNA interference-mediated silencing of the respiratory syncytial virus nucleocapsid defines a potent antiviral strategy. Antimicrobial Agents and Chemotherapy. 53 (9), 3952-3962 (2009).
- DeVincenzo, J., et al. A randomized, double-blind, placebo-comtrolled study of an RNAi-based therapy directed against respiratory syncytial virus. Proceedings of the National Academy of Sciences of the United States of America. 107 (19), 8800-8805 (2010).
- Zhou, Y., et al. High-throughput screening of a CRISPR/Cas9 library for functional genomics in human cells. Nature. 509 (7501), 487-491 (2014).
- Nikolic, J., et al. Negri bodies are viral factories with properties of liquid organelles. Nature Communications. 8 (1), 58 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon