É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Geração, amplificação e titulação do vírus sincicial respiratórios recombinantes
Neste Artigo
Resumo
Nós descrevemos um método para gerar e amplificar geneticamente modificado vírus sincicial respiratórios (RSVs) e um ensaio de placa otimizado para RSVs. Ilustramos este protocolo criando dois vírus recombinantes que respectivamente permitem quantificação de replicação RSV e análise de corpos de inclusão RSV e grânulos associada a corpos de inclusão dinâmica de viver.
Resumo
O uso de vírus recombinantes tornou-se crucial em virologia básica ou aplicada. Genética reversa tem sido provada para ser uma tecnologia extremamente poderosa, tanto para decifrar os mecanismos de replicação viral e para estudar antivirais ou fornecer a plataforma de desenvolvimento de vacinas. A construção e manipulação de um sistema de genética reversa para um negativo-vertente vírus RNA como um vírus sincicial respiratório (VSR), no entanto, continua a ser delicados e requer conhecimentos especiais. O genoma da RSV é um single-strand, negativo-sentido RNA de cerca de 15 kb, o que serve como um modelo para replicação do RNA viral e transcrição. Nosso sistema de genética reversa utiliza uma cópia de cDNA do genoma humano de longa estirpe de RSV (HRSV). Este cDNA, bem como a codificação de proteínas virais do complexo da polimerase de cDNAs (L, P, N e M2-1), são colocados em vetores de expressão individual sob sequências de controle do polymerase T7. O transfection desses elementos nas células de BSR-T7/5, que expressam estàvel polymerase T7, permite a replicação citoplasmática e transcrição de recombinação RSV, dando origem a virions geneticamente modificados. Uma nova RSV, que está presente na superfície da célula e no sobrenadante de cultura de BSRT7/5, está reunida para infectar células humanas de HEp-2 para amplificação viral. Duas ou três rodadas de amplificação são necessárias para obter ações virais contendo 1 x 106 a 1 x 107 unidades formadoras de placa (PFU) / mL. Métodos para o ideal da colheita, congelamento e titulação de estoques virais são descritos aqui em detalhes. Ilustramos o protocolo aqui apresentado, criando dois vírus recombinantes expressando respectivamente livre proteína verde fluorescente (GFP) (RSV-GFP) ou viral M2-1 fundida a GFP (RSV-M2-1-GFP). Mostramos como usar RSV-GFP para quantificar a replicação RSV e a RSV-M2-1-GFP para visualizar estruturas virais, bem como a dinâmica da proteína viral em células vivas, usando técnicas de vídeo microscopia.
Introdução
RSV humana é a principal causa de hospitalização por infecção aguda do trato respiratório em crianças em todo o mundo1. Além disso, a RSV é associado com um fardo de doença substancial em adultos comparáveis à gripe, com a maioria da hospitalização e carga de mortalidade no idoso2. Não existem vacinas ou específicas antivirais disponíveis ainda contra RSV, mas prometendo novas drogas estão em desenvolvimento3,4. A complexidade e o peso das técnicas de quantificação de multiplicação RSV impedem a busca de antivirais ou vacinas, apesar dos esforços consideráveis atuais. A quantificação de multiplicação RSV in vitro geralmente é baseada em métodos trabalhosos, demorados e caros, que consistem principalmente na análise do efeito citopático por microscopia, imunocoloração, redução de placa bacteriana, ensaios, quantitativos transcriptase reversa (qRT)-reação em cadeia da polimerase (PCR) e testes de ensaio enzima-lig da imunoabsorção. Vírus com genomas modificadas e expressar genes repórter, tais como aqueles que codifica para o GFP, são mais adequados para tais projeções. Juntamente com a utilização de leitores de placa automatizado, vírus recombinantes gene portadores de repórter podem fazer estes ensaios mais adequados para fins de padronização e alta produtividade.
RSV é um vírus de RNA sentido negativo envelopado, conjuntos que pertence ao gênero Orthopneumovirus do Pneumoviridae familiar, ordem Mononegavirales5. O genoma da RSV é um single-strand, negativo-sentido RNA de cerca de 15 kb, que contém uma região não-codificante as extremidades 3' e 5' chamado Leader e Trailer e 10 unidades transcricionais codificação 11 proteínas. Os genes estão ordenados da seguinte forma: 3'-NS1, NS2, N, P, M, SH, G, F, M2 (codificação para proteínas M2-1 e M2-2) e L-5'. O RNA genômico é firmemente embalado pela cadeia s. Using o RNA genômico encapsidated como um modelo, viral RNA-dependente do RNA polimerase (RdRp) garantirá a transcrição e replicação do RNA viral. RdRp viral é composto da proteína grande L que exerce a atividade de polimerase nucleotídeo por si, seu cofator obrigatório o phosphoprotein P e a proteína M2-1, que funciona como uma transcrição viral fator6. Em células infectadas, RSV induz a formação de inclusões citoplasmáticas, chamado de corpos de inclusão (IBs). Inclusões citoplasmáticas morfologicamente semelhantes têm sido observadas por várias Mononegavirales7,8,9,10. Estudos recentes sobre o vírus da raiva, vírus da estomatite vesicular (VSV), vírus Ebola e RSV mostrou que a síntese de RNA viral ocorre na IBs, que assim podem ser consideradas como fábricas viral8,9,11, 12. as fábricas de vírus concentrar o RNA e as proteínas virais necessárias para a síntese de RNA viral e também contêm proteínas celulares13,14,15,16, 17. SII exibem um subcompartment funcional chamado grânulos de IB-associado (IBAGs), que concentram o recém synthetized nascente mRNA viral juntamente com a proteína M2-1. O genoma RNA e a L, P e N não são detectadas em IBAGs. IBAGs são pequenas estruturas esféricas dinâmicas dentro IBs que exibem as propriedades do líquido organelas12. Apesar do papel central da IBs na multiplicação viral, muito pouco é conhecido sobre a natureza, estrutura interna, formação e operação destas fábricas viral.
A expressão do genoma de um vírus de um cDNA permitiu a produção do primeiro clone infeccioso viral em 198118. Para single-stranded vírus de RNA negativos, não foi até 1994 que a produção de um vírus de raiva primeira seguindo transfection de plasmídeos em células19 teve lugar. O primeiro baseado em plasmídeo reverso sistema genético para RSV foi publicado em 1995,20. Genética reversa têm levado a grandes avanços no campo da virologia. A possibilidade de introduzir modificações específicas no genoma viral forneceu insights críticos sobre a replicação e a patogênese dos vírus de RNA. Esta tecnologia também muito tem facilitado o desenvolvimento de vacinas permitindo atenuação específica através de uma série específica de modificações. Modificações de genoma, permitindo uma rápida quantificação de multiplicação viral grandemente melhoraram a triagem antiviral e estudo do seu modo de ação.
Embora anteriormente descrito, obter RSVs geneticamente modificados continua a ser delicado. Aqui, detalhamos um protocolo para criar dois tipos de HRSV recombinante, respectivamente, expressando RSV-GFP ou RSV-M2-1-GFP. Neste protocolo, descrevemos as condições de transfeccao necessárias para resgatar os novos vírus recombinantes, bem como sua amplificação para obter ações virais com título elevado, apropriado para experimentações reprodutíveis. A construção de vetores a genética reversa por si não está descrita aqui. Descrevemos os métodos de colheita ideal e congelamento de estoques virais. O método mais preciso para quantificar as partículas virais infecciosas permanece o ensaio da chapa. Células são infectadas com diluições em série da suspensão analisada e incubadas com uma sobreposição que proíbe a difusão de partículas virais livres no sobrenadante. Em tais condições, o vírus só irá infectar células contíguas, formando uma "placa" para cada partícula infecciosa inicial. No ensaio de titulação convencional de RSV, placas são reveladas por imunocoloração e contadas sob observação microscópica. Este método é caro e demorado. Aqui descrevemos um protocolo muito simples para um ensaio de placa RSV usando sobreposição de celulose microcristalina que permite a formação de placas visíveis a olho nu. Mostramos como RSV-GFP pode ser usado para replicação de RSV medida e, assim, para quantificar o impacto de antivirais. Combinação genética reversa e tecnologia de imagem ao vivo, demonstramos como RSV-M2-1-GFP permite aos cientistas para visualizar M2-1 em células vivas e a seguir a dinâmica das estruturas virais intracelulares, tais como IBs.
Protocolo
1. material preparação
- Compra de mídia de célula (soro reduzida mídia, mínimo essencial [MEM], 10 x MEM e Dulbecco modificada do meio da águia [DMEM]), reagente de transfeccao e celulose microcristalina (ver Tabela de materiais).
- Obter os seguintes vetores para genética reversa: o vector(s) genômica e os vetores de expressão de codificação da proteína N e as proteínas complexas de polimerase. Os genômicos vetores contêm o genoma completo do cDNA da RSV-GFP (p-RSV-GFP) e de 1GFP-RSV-M2 (p-RSV-M2-1GFP) a jusante do promotor do bacteriófago T7 RNA polimerase (T7 pol). Os vetores de expressão (designado como p-N, p -P, p-L e p-M2-1) contêm a sequência de codificação de N, P, L ou M2-1 a jusante a pol T7 (ver Rincheval et al.12 e Rameix-Welti et al.21 para obter detalhes sobre as construções de plasmídeo).
- Prepare a mídia para uma cultura de células em um ambiente estéril e a transfeccao e infecção. Uso DMEM com 2 mM L-glutamina suplementado com 10% de soro fetal bezerro (FCS), 1.000 unidades/mL penicilina e estreptomicina 1 mg/mL (ou sem antibióticos) e MEM com 2 mM L-glutamina suplementado com 0%, 2% ou 10% FCS, 1.000 unidades/mL penicilina e 1 mg/mL estreptomicina, designada como "meio completo" no seguinte protocolo.
- Obter células de22 BSRT7/5 e faça reservas em completo suplementado com 10% dimetilsulfóxido (DMSO) em 1 a 2 x 106 células/mL. Conserve os estoques de célula em nitrogênio líquido. Obter células HEp-2. BSRT7/5 células de cultura em DMEM completa e células HEp-2 em MEM completa em 37 ° C e 5% CO2 em um ambiente estéril.
- Prepare-se 10 x RSV conservação solução (HEPES de 0,5 M e 1 M MgSO4 [pH 7.5] em água) em um ambiente estéril.
- Obter um microscópio de fluorescência invertido, compatível com as medidas de fluorescência de GFP e compatível com imagens ao vivo, se é necessário monitorar uma infecção. Obter um leitor de microplacas compatível com medições de fluorescência as boas práticas agrícolas para a quantificação de replicação RSV-GFP.
2. o resgate e a primeira passagem do vírus recombinante
Nota: Execute todas as etapas a seguir em um ambiente estéril, usando uma classe de segurança II.
- O dia antes de transfeccao, faça uma suspensão da linha celular BSRT7/5 a 5 x 105 células/mL em meio completo. Distribua 2 mL de suspensão de células por poço em uma placa de 6. Prepare um poço por vírus que vão ser resgatado e um bem adicional para controle negativo. Incube a placa a 37 ° C e 5% de CO2. Verifique se as pilhas são uma confluência de 80 – 90% no dia seguinte.
- Descongelar a genética reversa vetores (da etapa 1.2) p-RSV-GFP e p-RSV-M2-1-GFP, assim como o p-N, p -P, p-L e p-M2-1. Mistura, para cada vírus resgatar, 1 µ g de p-N e p -P, 0,5 µ g de p-L, 0,25 µ g de p-M2-1 e 1,25 µ g de p-RSV (GFP ou GFP M2-1) em um tubo.
Nota: Vetores de expressão diferente para N, P, L e M2-1 podem ser utilizados; no entanto, a relação entre as proteínas deve ser mantida. Execute o controlo negativo, substituindo o vetor p-RSV com um vetor vazio. -
Prossiga para transfeccao, seguindo o protocolo do fabricante reagente de transfeccao (ver tabela de materiais).
- Adicione 250 µ l de meio de soro reduzida para os vetores mistos. Em outro tubo, dilua 10 µ l do reagente de transfeccao em 250 µ l de meio de soro reduzida. Suavemente o vórtice tubos e espere 5 min. misturar o conteúdo dos dois tubos e esperar 20 min à temperatura ambiente.
- Enxagúe as células de BSRT7/5 com 1 mL de meio de soro reduzida e distribuir 1,5 mL de MEM com FCS 10% sem antibióticos por bem. Se necessário, incube a 37 ° C e 5% de CO2 , até que seja concluída a incubação descrita no passo 2.3.1.
- Adicione 500 µ l do mix do transfection preparado na etapa 2.3.1 para um poço quando acabou o tempo de incubação de 20 min. Coloca as células em incubação a 37 ° C e 5% de CO2 por 3 dias. Não altere o meio de cultura das células durante o transfection.
- Observar a fluorescência de GFP (excitação em 488 nm) e emissões em 515 – 535 nm sob um microscópio de fluorescência invertido na ampliação de 20 x 1 x por dia para monitorar a eficiência de resgate, usando o GFP filtrar.
- No segundo dia após o transfeccao, sementes das células para a primeira passagem dos vírus resgatados. Prepare uma suspensão de células HEp-2 a 5 x 105 células/mL em meio completo. Distribua 2 mL de suspensão de células por poço em uma placa de 6 (um bem por vírus para resgate e um controle negativo).
- No terceiro dia de transfeccao, células zero em cada poço da placa transfectada 6-bem BSRT7/5, com uma espátula de diferente para cada poço. Transferência de cada conteúdo bem (células e sobrenadante) em um tubo de microcentrifuga estéril de 2 mL. Tubo de vórtice cada vigorosamente durante pelo menos 30 s para liberar o vírus resgatado de membranas celulares.
Nota: Isso corresponde a passagem 0 (P0) do vírus resgatado (Figura 1). -
Use a suspensão de P0 viral fresca para executar a primeira amplificação dos vírus resgatados.
- Remover o meio de cultura da placa HEp-2 6-bem semeado no dia anterior (consulte a etapa 2.5) e rapidamente adicionar 500 µ l de suspensão de P0 (da etapa 2.6) por poço. Coloque a placa HEp-2 a 37 ° C em um roqueiro gangorra para suave agitação por 2 h.
- Remova e descarte a 500 µ l de inóculo e adicionar 2 mL de MEM com FCS 2%. Incube a placa a 37 ° C e 5% de CO2 por 3 dias. Isto produzirá a primeira passagem (P1) dos vírus resgatados (Figura 1).
- Adicione 1/10 do volume da solução de conservação da RSV do x do 10 (HEPES de 0,5 M e 1 M MgSO4 [pH 7.5]) para a suspensão de P0 restante (da etapa 2.6). Vórtice microtubos vigorosamente por 5 s e alíquota o conteúdo em tubos criogênicos rotulado com tags resistente ao álcool. Imergir os tubos pelo menos 1 h em álcool pré-resfriado a-80 ° C e armazená-los a-80 ° C.
- Titula-se o estoque de P0 (ver passo 2.6) de cada vírus resgatados (ver secção 4 para a titulação de celulose microcristalina).
- Observe a fluorescência de GFP (excitação em 488 nm) e emissões em 515 – 535 nm das células HEp-2 infectado com a suspensão de P0 sob um microscópio de fluorescência invertido na ampliação de 20 x 1 x por dia para monitorar a infecção. Observar ao microscópio brightfield a aparência de pequenas citoplasma e desprendimento que reflecte o efeito GENECLASS RSV (CPE) da pilha (ver Figura 2).
- Observe que o resgate falhou se fluorescência nem CPE estiver visível após 2-3 dias.
- Colete a primeira passagem (P1) no dia 3 ou 4 conforme descrito na etapa 2.6. Em breve, raspar as células, coletar as células e o sobrenadante junto, vórtice-los, adicionar a solução de conservação, conforme descrito na etapa 2.8, alíquota da amostra e congelá-lo.
- Titula-se (ver seção 4 para o ensaio de titulação) e amplificar a primeira passagem (veja a seção 3 para a amplificação).
3. amplificação dos vírus resgatados
Nota: O protocolo seguinte descreve a amplificação dos vírus resgatados num balão de2 de 75 cm. Adapte o tamanho do balão para o volume necessário e a multiplicidade exigida de infecção (MOI). A tabela 1 indica volumes para frascos diferentes. Execute todas as etapas a seguir em um ambiente estéril em uma classe de segurança II.
- Prepare uma suspensão de células HEp-2 a 5 x 105 células/mL em meio completo, o dia antes da amplificação. Distribuir 15 mL da suspensão celular por balão2 de 75 cm e incubar os frascos a 37 ° C e 5% de CO2. Prepare um frasco por vírus para amplificar.
- O dia após o início da incubação, verifique se as células estão 80%-100% confluente.
- Dilua a suspensão viral da etapa 2.12 em MEM sem FCS para obter uma suspensão de 3 mL em 50.000 PFU/mL (correspondente a um MOI de 0,01 PFU/célula).
- Remover o meio e adicionar rapidamente a suspensão viral de 3 mL. Coloca o balão a 37 ° C em um roqueiro gangorra para suave agitação por 2 h.
- Remova e descarte o inóculo e adicionar 15 mL de MEM com FCS 2%. Incube a 37 ° C e 5% de CO2 para 2 – 4 dias.
- Verificar a morfologia celular e fluorescência de GFP (excitação em 488 nm) e emissões em 515 – 535 nm sob um microscópio de fluorescência invertido na ampliação de 20 x para estimar o tempo certo para colher os vírus. Note que isto é normalmente quando 50 – 80% da camada de células HEp-2 é desanexado devido a CPE RSV que ocorre entre 48 e 72 h postinfection (PI) (ver Figura 3).
- Raspe todas as células com uma espátula de célula. Coletar as células e o sobrenadante juntos e transfira para um tubo de centrífuga de 50 mL.
- Adicione 1/10 do volume dos 10 x solução de conservação RSV (0,5 M de HEPES e 1m MgSO4 [pH 7.5]). Vórtice os tubos vigorosamente por 5 s e clarificar a suspensão por uma centrifugação de 5 min a 200 x g.
- Transferi o sobrenadante para um tubo de 50 mL. Vórtice brevemente e alíquota a suspensão em tubos criogênicos rotulado com tags resistente ao álcool. Imergir os tubos em álcool pré-resfriado-80 ° C durante pelo menos 1 h e armazená-los a-80 ° C.
- Descongele dentre as alíquotas para dosear a suspensão viral (ver secção 4).
4. placa ensaio de titulação
- Prepare-se 12-poços chapas à titulação no dia antes do ensaio de titulação é realizado (seis poços deverão titula-se um tubo de vírus). As sementes dos poços com 1 mL de células HEp-2 a 5 x 105 células/mL em meio completo.
-
No dia seguinte, prepare uma suspensão de celulose microcristalina estéril (2,4% [w/v] em água) (ver tabela de materiais).
- Disperse a 2,4 g de pó de celulose microcristalina em 100 mL de água destilada, utilizando um agitador magnético padrão, até completa dissolução do pó (geralmente 4-12 h). Autoclave a suspensão a 121 ° C por 20 min e armazená-lo em temperatura ambiente antes do uso.
Nota: Sob tais condições, a suspensão é estável por 1 ano. - Depois de abrir a solução em um ambiente estéril, armazená-lo em 4 ° C por 6 meses. Misture sempre a suspensão antes de usar (por mão tremendo ou num Vortex) para certificar-se que é homogênea.
- Disperse a 2,4 g de pó de celulose microcristalina em 100 mL de água destilada, utilizando um agitador magnético padrão, até completa dissolução do pó (geralmente 4-12 h). Autoclave a suspensão a 121 ° C por 20 min e armazená-lo em temperatura ambiente antes do uso.
- Preparar o 2 x MEM em um ambiente estéril. Diluir o MEM comercial 10 x com água estéril e adicionar L-glutamina, 1.000 unidades/mL penicilina e estreptomicina 1 mg/mL. Agitar vigorosamente a diluição e armazená-lo em 4 ° C.
Nota: Execute etapas 4.4 – 4.10 em um ambiente estéril, usando uma classe de segurança II. - Prepare-se seis tubos contendo 900 µ l de MEM sem FCS por vírus para ser titulado (os tubos de titulação). Descongelar as alíquotas de vírus, tubo de vórtice-los vigorosamente durante 5 s e a transferência de 100 µ l a primeira titulação.
- Realizar uma diluição dez vezes 6 x, como se segue. Adicione 100 μL de vírus para 900 μL do meio no primeiro tubo, coloque a tampa do tubo e misturar seu conteúdo num Vortex durante alguns segundos. Mudar a ponta da pipeta, adicionar 100 µ l da primeira diluição a 900 µ l de meio no segundo tubo, colocar a tampa no tubo e vórtice. Repita o procedimento até o sexto tubo.
Nota: É muito importante mudar a ponta para cada diluição. - Escreva o vírus nome e as diluições sobre as placas de 12-poços de HEp-2. Adicione uma marca para coincidir com a placa e a tampa, porque eles podem ser separados durante coloração (etapa 4.9). Retire o meio as chapas e distribuir 400 µ l da diluição por bem. Incube as placas a 37 ° C por 2h, para a adsorção do vírus.
Nota: Mudar a ponta da pipeta entre cada inóculo ou proceder do menor ao maior concentração com a mesma dica. Inocule uma série limitada de placas (1 a 2) simultaneamente para evitar as células de secagem. -
Prepare a sobreposição de celulose microcristalina durante a adsorção do vírus (preparação extemporânea). Para obter 100 mL de sobreposição, misture 10 mL de suspensão microcristalina de 2,4%, 10 mL de 2 x MEM e 80 mL de MEM com FCS 2%.
- Ajustar o pH da 2 x MEM para cerca de 7,2 com uma solução de bicarbonato de sódio estéril em 7,5%, após o indicador de cor. Adicione a suspensão de celulose microcristalina e o MEM e misture vigorosamente.
- No final da incubação a 2 h, adicione 2 a 3 mL de sobreposição para cada poço das placas 12-poços sem remover o inóculo. Tenha cuidado para evitar a contaminação dos poços adjacentes com inoculo de alto título viral. Incube a placa a 37 ° C e 5% de CO2 por 6 dias. Não mova a placa e não mova a incubadora durante a incubação.
-
Prossiga para manchar as células, usando solução de cristal violeta (violeta de cristal de 8% [v/v], 2% de formaldeído [v/v] e etanol 20% [v/v] na água).
- Protege a superfície de trabalho da Biossegurança do armário com uma folha (a violeta cristal cores fortemente superfícies).
- Agite as placas para tirar o decalque de celulose microcristalina. Remover os sobrenadantes e lavar as células 2 x com 1x tampão fosfato salino (PBS). Lidar com as placas uma a uma, para evitar as células de secagem. Adicionar 1 a 2 mL da solução de cristal violeta e esperar 10-15min. remover a solução, que pode ser reutilizada para coloração de placa subsequente.
- Mergulhe as placas e tampas em lixívia fresca por alguns segundos e, em seguida, lave-as com água da torneira. Note que as placas e tampas são descontaminadas pelo alvejante.
- Colocar as placas e tampas em papel toalha e deixe-os secar. Seguida a água, secar as placas a uma temperatura ambiente e armazená-los em temperatura ambiente. Para longos períodos de armazenamento (meses), manter as placas protegidas da luz para proteger a cor. Note que se as células perdem sua coloração, eles podem ser manchados novamente com violeta de cristal.
- Calcule a concentração de vírus. Conte as placas dos poços das placas secas, que são visíveis a olho nu. Verifique se o número de placas de diferentes diluições é coerente (fator 10 entre cada diluição). Escolha o bem sobre o qual as placas são mais fáceis de contar. Avalie o número de placas versus o volume de inóculo e a diluição.
Nota: No exemplo da Figura 4, 21 placas são contadas com a diluição de 10-5 . Estas correspondem a um título de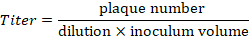
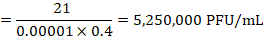
5. o uso de HRSV-GFP vírus recombinante para monitorar a replicação Viral em células tratadas com RNA de interferência pequeno ou antivirais
Nota: Execute todas as etapas, exceto 5.1 e 5.2.5 em um ambiente estéril, usando uma classe de segurança II.
-
O efeito de silenciamento na multiplicação RSV do gene celular de monitoramento
Nota: O protocolo do transfection depende do reagente (ver Tabela de materiais).- Prepare-se para a medição de GFP placas de 96 poços. Dois dias antes do ensaio, para dado RNA de interferência pequeno (siRNA), preparar uma solução de mídia reduzida soro contendo siRNA em uma concentração de 100 nM e um reagente de transfeccao siRNA diluído a 1/500. Incube a solução por 30 min à temperatura ambiente.
- Adicione 25 µ l da solução aos poços da placa preparada em 5.1.1 (em triplicado). Os poços com 75 µ l de uma suspensão de células A549 4 x 105 células/ml em meio completo sem antibióticos para obter uma concentração final de células de 3 x 105 células/mL de semente. Incube a placa por 48 h a 37 ° C e 5% de CO2.
Nota: A concentração final de siRNA é 25 nM e o volume de reagente de transfeccao final é 0,5 µ l/poço). - Infectar as células como segue. Remova o meio dos poços. Adicione 100 µ l de suspensão de RSV-GFP em 50.000 PFU/mL e incubar durante 2 h a 37 ° C e 5% de CO2. Remover a suspensão viral e adicionar 100 µ l de DMEM com 2% de FCS e sem vermelho de fenol. Incube a placa a 37 ° C e 5% de CO2.
- Em 24 h e 48 h p.i., medir a fluorescência, utilizar um spectrofluorometer definido para comprimentos de onda de excitação e emissão de 488 e 520 nm, respectivamente (fluorescência é expresso em unidades de fluorescência relativo). Use infectados A549 células como normas para níveis de fluorescência e plano de fundo.
Nota: As células precisam ser corrigidos com paraformaldeído 4% (PFA) antes de medi-los sem a tampa da placa.
-
Avaliação da inibição da droga usando RSV-GFP
- Prepare-se para a medição de GFP placas de 96 poços. O dia antes do ensaio, as sementes dos poços com 100 µ l de uma suspensão de células HEp-2 a 5 x 105 células/mL em meio completo sem vermelho de fenol.
- Preparar uma diluição serial do testado droga (AZ4316 neste exemplo) na MEM complementado com FCS 2% e antibióticos (50 μL por poço). Prepare uma suspensão viral em 10.000 PFU/mL em MEM sem fator vascular do estroma (SVF) e sem vermelho de fenol (50 μL por poço).
- Remover o meio do HEp-2 de 96 poços da placa e adicionar 50 µ l de suspensão de drogas e 50 µ l da suspensão viral (em triplicado). Execute uma simulação infecção em paralelo como um controle.
Nota: A diluição de drogas e suspensão viral podem ser misturados antes de adicioná-los sobre as células, ou eles podem ser adicionados em sequência. - Incube a placa por 48 h a 37 ° C e 5% de CO2.
- Medir a fluorescência, utilizando um spectrofluorometer conforme descrito na etapa 5.1.4. Use mock-infectado as células HEp-2 como normas para os níveis de fundo de fluorescência.
6. Caracterização da localização M2-1 In Vivo com o vírus recombinante RSV-M2-1-GFP
Nota: Execute os passos 6.1 e 6.2 em um ambiente estéril, usando uma classe de segurança II.
- Prepare uma suspensão de células HEp-2 a 5 x 105 células/mL em meio completo. Semente de 1,5 mL de suspensão de célula em uma 35 mm placa de Petri permeant de CO2 e adaptados para vive de imagem.
- Realizar a infecção, o dia após a semeadura com o vírus RSV-M2-1-GFP em MOI 1, conforme descrito nas etapas 3.3 – 3.5 (remover o meio, adicione 500 µ l a 1 mL de inóculo e incubar a amostra a 37 ° C, agitando suavemente por 2h; remover o inóculo e adicione 1,5 mL de MEM com 2 % FCS). Incube as celulas em 37 ° C e 5% de CO2 para o tempo desejado (IBs começarão aparecer de 10 h p.i.).
- Pré-aqueça a câmara de incubação de um microscópio invertido equipado com 40 x 100 objectivos x a 37 ° C, antes de colocar a placa de Petri contendo as células infectadas no palco. Abra a CO2 entregas e aguardar estabilização do foco.
- Execute a imagem com filtros compatíveis com as boas práticas agrícolas, sob uma frequência de excitação baixa intensidade e imagem (de 1 a 0,1 imagem / min) para minimizar a fototoxicidade.
Resultados
Neste trabalho, descrevemos um protocolo detalhado para produzir recombinantes vírus RSV, expressando uma proteína fluorescente (Figura 2). Em pRSV-GFP, o gene GFP foi introduzido entre os genes P e M, conforme descrito para o gene cereja em trabalho anteriormente publicado21. No pRSV-M2-1-GFP, o gene M2 foi deixado intocado e um adicional gene que codifica para M2-1-GFP foi inserido entre os genes SH e G12. A...
Discussão
Aqui nós apresentamos um método de resgate de recombinante RSVs de cinco plasmídeos e sua amplificação. A capacidade de manipular o genoma do vírus tem revolucionado a pesquisa de virologia para testar as mutações e expressar um gene adicional ou uma proteína viral marcada. A RSV temos descrito e usado como exemplo neste artigo é um vírus que está expressando um gene repórter, o RSV-GFP (não publicado) e expressa uma proteína M2-1 fundida a uma marca GFP12. Resgate de RSV é desafia...
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Os autores agradecer Dr. Qin Yu da AstraZeneca R & D Boston, MA, EUA, para fornecer a droga AZD4316. Os autores são gratos à plataforma Cymages para acessar o microscópio ScanR Olympus, que foi apoiado por concede da região de Ile-de-France (uma só saúde DIM). Os autores reconhecem apoio do INSERM e a Universidade de Versailles Saint-Quentin.
Materiais
| Name | Company | Catalog Number | Comments |
| 35 mm µ dish for live cell imaging | Ibidi | 81156 | |
| A549 | ATCC | ATCC CCL-185 | |
| Avicel RC-591 | FMC BioPolymer | Avicel RC-591 | Technical and other information on Avicels is available at http://www.fmcbiopolymer.com. Store at room temperature. Protocol in step 4 is optimized for this reagent. |
| BSRT7/5 | not commercially available | See reference 22. Buchholz et al. 1999 | |
| Crystal violet solution | Sigma | HT90132 | |
| Fluorescence microscope for observations | Olympus | IX73 Olympus microscope | |
| Fluorescence microscope for videomicroscopy | Olympus | ScanR Olympus microscope | |
| HEp-2 | ATCC | ATCC CCL-23 | |
| HEPES ≥99.5% | Sigma | H3375 | |
| L-Glutamine (200 mM) | ThermoFisher Scientific | 25030024 | |
| LIPOFECTAMINE 2000 REAGENT | ThermoFisher Scientific | 11668019 | Protocol in step 2.3. is optimized for this reagent. |
| MEM (10x), no glutamine | ThermoFisher Scientific | 11430030 | |
| MEM, GlutaMAX Supplement | ThermoFisher Scientific | 41090-028 | |
| MgSO4 ReagentPlus, ≥99.5% | Sigma | M7506 | |
| Opti-MEM I Reduced Serum Medium | ThermoFisher Scientific | 51985-026 | |
| Paraformaldehyde Aqueous Solution, 32%, EM Grade | Electron Microscopy Sciences | 15714 | |
| Penicillin-Streptomycin (10,000 U/mL) | ThermoFisher Scientific | 15140122 | |
| Plasmids | not commercially available | See reference 21. Rameix-Welti et al. 2014 | |
| See Saw Rocker | VWR | 444-0341 | |
| Si RNA GAPDH | Dharmacon | ON-TARGETplus siRNA D-001810-10-05 | SMARTpool and 3 of 4 individual siRNAs designed by Dharmacon. |
| Si RNA IMPDH2 | Dharmacon | ON-TARGETplus siRNA IMPDH2 Pool- Human L-004330-00-0005 | SMARTpool of 4 individual siRNAs designed by Dharmacon. Individual references and sequences J-004330-06: GGAAAGUUGCCCAUUGUAA; J-004330-07: GCACGGCGCUUUGGUGUUC; J-004330-08: AAGGGUCAAUCCACAAAUU; J-004330-09: GGUAUGGGUUCUCUCGAUG; |
| Si RNA RSV N | Dharmacon | ON-TARGETplus custom siRNA | UUCAGAAGAACUAGAGGCUAU and UUUCAUAAAUUCACUGGGUUA |
| SiRNA NT | Dharmacon | ON-TARGETplus Non-targeting Pool | |
| SiRNA transfection reagent | Dharmacon | DharmaFECT 1 Ref: T-2001-03 | Protocol in steps 5.1.and 5.1.2 are optimized for this reagent. |
| Sodium Bicarbonate 7.5% solution | ThermoFisher Scientific | 25080094 | |
| Spectrofluorometer | Tecan | Tecan infinite M200PRO |
Referências
- Shi, T., et al. Global, regional, and national disease burden estimates of acute lower respiratory infections due to respiratory syncytial virus in young children in 2015: a systematic review and modelling study. The Lancet. 390 (10098), 946-958 (2017).
- Falsey, A. R., Hennessey, P. A., Formica, M. A., Cox, C., Walsh, E. E. Respiratory Syncytial Virus Infection in Elderly and High-Risk Adults. The New England Journal of Medicine. 352 (17), 1749-1759 (2005).
- DeVincenzo, J. P., et al. Activity of Oral ALS-008176 in a Respiratory Syncytial Virus Challenge Study. The New England Journal of Medicine. 373 (21), 2048-2058 (2015).
- DeVincenzo, J. P., et al. Oral GS-5806 Activity in a Respiratory Syncytial Virus Challenge Study. The New England Journal of Medicine. 371 (8), 711-722 (2014).
- Afonso, C. L., et al. Taxonomy of the order Mononegavirales: update 2016. Archives of Virology. 161 (8), 2351-2360 (2016).
- Collins, P. L., Hill, M. G., Cristina, J., Grosfeld, H. Transcription elongation factor of respiratory syncytial virus, a nonsegmented negative-strand RNA virus. Proceedings of the National Academy of Sciences of the United States of America. 93 (1), 81-85 (1996).
- Hoenen, T., et al. Inclusion bodies are a site of ebolavirus replication. Journal of Virology. 86 (21), 11779-11788 (2012).
- Heinrich, B. S., Cureton, D. K., Rahmeh, A. A., Whelan, S. P. Protein expression redirects vesicular stomatitis virus RNA synthesis to cytoplasmic inclusions. PLoS Pathogens. 6 (6), e1000958 (2010).
- Lahaye, X., et al. Functional Characterization of Negri Bodies (NBs) in Rabies Virus-Infected Cells: Evidence that NBs Are Sites of Viral Transcription and Replication. Journal of Virology. 83 (16), 7948-7958 (2009).
- Kolesnikova, L., Mühlberger, E., Ryabchikova, E., Becker, S. Ultrastructural organization of recombinant Marburg virus nucleoprotein: comparison with Marburg virus inclusions. Journal of Virology. 74 (8), 3899-3904 (2000).
- Dolnik, O., Stevermann, L., Kolesnikova, L., Becker, S. Marburg virus inclusions: A virus-induced microcompartment and interface to multivesicular bodies and the late endosomal compartment. European Journal of Cell Biology. 94 (7-9), 323-331 (2015).
- Rincheval, V., et al. Functional organization of cytoplasmic inclusion bodies in cells infected by respiratory syncytial virus. Nature Communications. 8 (1), 563 (2017).
- Santangelo, P. J., Bao, G. Dynamics of filamentous viral RNPs prior to egress. Nucleic Acids Research. 35 (11), 3602-3611 (2007).
- Lifland, A. W., et al. Human Respiratory Syncytial Virus Nucleoprotein and Inclusion Bodies Antagonize the Innate Immune Response Mediated by MDA5 and MAVS. Journal of Virology. 86 (15), 8245-8258 (2012).
- Garcia, J., Garcia-Barreno, B., Vivo, A., Melero, J. A. Cytoplasmic inclusions of respiratory syncytial virus-infected cells: formation of inclusion bodies in transfected cells that coexpress the nucleoprotein, the phosphoprotein, and the 22K protein. Virology. 195 (1), 243-247 (1993).
- Brown, G., et al. Evidence for an association between heat shock protein 70 and the respiratory syncytial virus polymerase complex within lipid-raft membranes during virus infection. Virology. 338 (1), 69-80 (2005).
- Radhakrishnan, A., et al. Protein analysis of purified respiratory syncytial virus particles reveals an important role for heat shock protein 90 in virus particle assembly. Molecular & Cellular Proteomics. 9 (9), 1829-1848 (2010).
- Racaniello, V. R., Baltimore, D. Cloned poliovirus complementary DNA is infectious in mammalian cells. Science. 214 (4523), 916-919 (1981).
- Schnell, M. J., Mebatsion, T., Conzelmann, K. K. Infectious rabies viruses from cloned cDNA. The EMBO Journal. 13 (18), 4195-4203 (1994).
- Collins, P. L., et al. Production of infectious human respiratory syncytial virus from cloned cDNA confirms an essential role for the transcription elongation factor from the 5' proximal open reading frame of the M2 mRNA in gene expression and provides a capability for vaccine. Proceedings of the National Academy of Sciences of the United States of America. 92 (25), 11563-11567 (1995).
- Rameix-Welti, M. -. A., et al. Visualizing the replication of respiratory syncytial virus in cells and in living mice. Nature Communications. 5, 5104 (2014).
- Buchholz, U. J., Finke, S., Conzelmann, K. K. Generation of bovine respiratory syncytial virus (BRSV) from cDNA: BRSV NS2 is not essential for virus replication in tissue culture, and the human RSV leader region acts as a functional BRSV genome promoter. Journal of Virology. 73 (1), 251-259 (1999).
- Cianci, C., Meanwell, N., Krystal, M. Antiviral activity and molecular mechanism of an orally active respiratory syncytial virus fusion inhibitor. Journal of Antimicrobial Chemotherapy. 55 (3), 289-292 (2005).
- Derscheid, R. J., et al. Human respiratory syncytial virus memphis 37 grown in HEp-2 cells causes more severe disease in lambs than virus grown in vero cells. Viruses. 5 (11), 2881-2897 (2013).
- McKimm-Breschkin, J. L. A simplified plaque assay for respiratory syncytial virus - direct visualization of plaques without immunostaining. Journal of Virological Methods. 120, 113-117 (2004).
- Matrosovich, M., Matrosovich, T., Garten, W., Klenk, D. New low-viscosity overlay medium for viral plaque assays. Virology Journal. 7, 1-7 (2006).
- Novina, C. D., Sharp, P. A. The RNAi revolution. Nature. 430 (6996), 161-164 (2004).
- Sintchak, M. D., Nimmesgern, E. The structure of inosine 5'-monophosphate dehydrogenase and the design of novel inhibitors. Immunopharmacology. 47 (2-3), 163-184 (2000).
- Beaucourt, S., Vignuzzi, M. Ribavirin: A drug active against many viruses with multiple effects on virus replication and propagation. Molecular basis of ribavirin resistance. Current Opinion in Virology. 8, 10-15 (2014).
- Hruska, J. F., Bernstein, J. M., Douglas, R. G., Hall, C. B. Effects of Ribavirin on Respiratory Syncytial Virus in vitro. Antimicrobial Agents and Chemotherapy. 17 (5), 770-775 (1980).
- Simões, E. A. F., et al. Past, Present and Future Approaches to the Prevention and Treatment of Respiratory Syncytial Virus Infection in Children. Infectious Diseases and Therapy. 7 (1), 87-120 (2018).
- Alvarez, R., et al. RNA interference-mediated silencing of the respiratory syncytial virus nucleocapsid defines a potent antiviral strategy. Antimicrobial Agents and Chemotherapy. 53 (9), 3952-3962 (2009).
- DeVincenzo, J., et al. A randomized, double-blind, placebo-comtrolled study of an RNAi-based therapy directed against respiratory syncytial virus. Proceedings of the National Academy of Sciences of the United States of America. 107 (19), 8800-8805 (2010).
- Zhou, Y., et al. High-throughput screening of a CRISPR/Cas9 library for functional genomics in human cells. Nature. 509 (7501), 487-491 (2014).
- Nikolic, J., et al. Negri bodies are viral factories with properties of liquid organelles. Nature Communications. 8 (1), 58 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados