Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Direkte Messung des KDM1A Target Engagements mit Chemoprobe-basierten Immunoassays
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Protokoll zur Messung des KDM1A-Zielengagements in einer menschlichen oder tierischen Zelle, Gewebe oder Blutproben vor, die mit KDM1A-Hemmern behandelt werden. Das Protokoll verwendet Chemoprobe-Tagging des freien KDM1A-Enzyms und direkte Quantifizierung des Zielbegebungsmittels mittels chemoprobebasierter Immunoassays und kann in präklinischen und klinischen Studien eingesetzt werden.
Zusammenfassung
Die Bewertung des Zielengagements, definiert als die Wechselwirkung eines Arzneimittels mit dem Protein, für das es entwickelt wurde, ist eine Grundvoraussetzung für die Interpretation der biologischen Aktivität jeder Verbindung in der Arzneimittelentwicklung oder in Grundlagenforschungsprojekten. In der Epigenetik wird das Zielengagement am häufigsten durch die Analyse von Proxy-Markern bewertet, anstatt die Vereinigung der Verbindung zum Ziel zu messen. Zu den nachgelagerten biologischen Auslesungen, die analysiert wurden, gehören die Histonmarkmodulation oder Genexpressionsänderungen. KDM1A ist eine Lysindemethylase, die Methylgruppen aus mono- und dimethyliertem H3K4 entfernt, eine Modifikation, die mit dem Silencing der Genexpression verbunden ist. Die Modulation der Proxy-Marker hängt vom Zelltyp und der Funktion der genetischen Ausgestaltung der untersuchten Zellen ab, was die Interpretation und den Cross-Case-Vergleich recht schwierig machen kann. Um diese Probleme zu umgehen, wird ein vielseitiges Protokoll vorgestellt, um die Dosiseffekte und die Dynamik des direkten KDM1A-Zielengagements zu bewerten. Der beschriebene Test nutzt eine KDM1A-Chemoprobe, um hemmungsloses Enzym zu erfassen und zu quantifizieren, kann ohne genetische Veränderung breit auf Zellen oder Gewebeproben angewendet werden, verfügt über ein ausgezeichnetes Nachweisfenster und kann sowohl für die Grundlagenforschung verwendet werden. und Analyse klinischer Proben.
Einleitung
Lysin-spezifische demethylase 1 (KDM1A)1 ist eine Demethylase, die an der Kontrolle der Gentranskription beteiligt ist. Dieses Protein hat sich als Kandidat pharmakologisches Ziel2 in der Onkologie herauskristallisiert; einschließlich Akute myeloische Leukämie3 (AML), Myelodysplasie-Syndrom (MDS)4, Myelofibrose (MF)5,6, Kleinzelllungenkrebs (SCLC)7; bei Sichelzellerkrankungen (SCD)8,9, und bei Erkrankungen des zentralen Nervensystems einschließlich Alzheimer (AD), Multiple Sklerose (MS); und in Aggression10.
Die meisten KDM1A-hemmenden Verbindungen in der klinischen Entwicklung sind Cyclopropylamin-Derivate und hemmen das Protein durch kovalente Bindung an seinen Flavin-Adenin-Dinukleotid(FAD)-Kofaktor11. Die Hemmung von KDM1A induziert Veränderungen der Genexpression, aber diese Veränderungen variieren enorm zwischen Geweben, Zelltypen oder Krankheitsfällen. Hemmung von KDM1A ändert auch Histonzeichen12, aber diese Veränderungen werden in der Regel lokal an einer bestimmten Stelle im Genom produziert, und sind wieder, hoch Gewebe und zellspezifisch.
Das Protokoll wurde entwickelt, um das KDM1A-Zielengagement in biologischen Proben direkt zu messen und wurde für die Verwendung mit Cyclopropylamin-abgeleiteten Inhibitoren optimiert. Der Test basiert auf der ELISA-Technologie und analysiert parallel, Total und Free (d.h. ungebunden durch Inhibitor) KDM1A in einem nativen Proteinextrakt aus einer biologischen Probe in einem Solid-Phase-Assay. In einem ersten Schritt wird die biologische Probe in Gegenwart der biotinylierten KDM1A-selektiven Chemoprobe OG-88113,14, abgeleitet aus dem selektiven KDM1A-Inhibitor ORY-1001 (Iadademstat), einem potenten Inhibitor von KDM1A Entwicklung zur Behandlung onkologischer Erkrankungen. Die Chemoprobe hat einen IC50 für KDM1A von 120 nM und enthält eine FAD-Bindungsart, die mit einem biotinylierten Polyethylenglykol (PEG)-Schwanz verbunden ist. Die Chemoprobe bindet ausschließlich an die freie KDM1A, nicht aber an den inhibitorgebundenen KDM1A in der Probe. Nach der Chemoprobebindung werden die KDM1A, die Komplexe in der Probe enthält, auf Mikrotiterplatten mit Streptavidin-beschichteter Oberfläche erfasst, um freie KDM1A zu bestimmen, oder auf Platten, die mit einem monoklonalen Anti-KDM1A-Capture-Antikörper beschichtet sind, um die Gesamt-KDM1A zu bestimmen. Nach dem Waschen werden beide Platten mit einem Anti-KDM1A-Detektionsantikörper inkubiert, erneut gewaschen und mit einem sekundären HRP-konjugierten Esel-Antikaninchen-IgG-Antikörper zur Detektion mit einem lumineszierenden Substrat und zur Quantifizierung durch Messung relativer Lichteinheiten (RLU) in einem Luminometer (Abbildung 1).
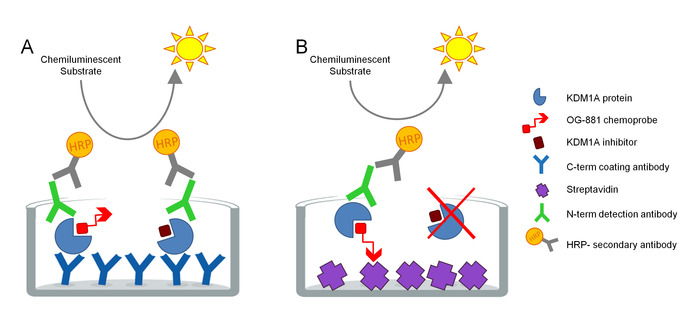
Abbildung 1. Schema des ELISA-Enzyms verknüpftchemontierten Immunabsorptions-Assays für KDM1A-Zielengagement: A) Bestimmung der Gesamt-KDM1A mit Sandwich-ELISA und B) Bestimmung des freien KDM1A mit Chemoprobe ELISA. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
In beiden ELISA-Platten ist eine Standardkurve enthalten, um die Linearität jedes Assays zu überprüfen. Die Bestimmung des KDM1A-Zielengagements in jeder Probe wird dann als relativer Wert zur Vordosis- oder Fahrzeugprobe berechnet.
Access restricted. Please log in or start a trial to view this content.
Protokoll
Blutproben wurden vom Instituto de Investigacion Biomédica Sant Pau Biobank nach spanischem Recht (Real Decreto de Biobancos 1716/2011) und der Genehmigung der lokalen Ethikkommissionen entnommen. Die Untersuchungen mit tierischem Gewebe wurden in Übereinstimmung mit den institutionellen Leitlinien für die Pflege und Verwendung von Labortieren (Richtlinie 86/609/EWG des Rates der Europäischen Gemeinschaften) durchgeführt, die vom Ethikausschuss für Tierversuche auf der PRAAL-PCB.
1. Herstellung biologischer Proben für den Test.
VORSICHT: Dieses Protokoll beinhaltet die Manipulation biologischer Proben, die dem Blutpathogenstandard (OSHA) (29 CFR 1910.1030), der Richtlinie 2000/54/EG des Europäischen Parlaments und der Vom 18. September 2000 oder gleichwertigen Verordnungen. Darüber hinaus können die biologischen Proben Spuren biologisch aktiver chemischer Prüfstoffe enthalten, und das Protokoll kann eine weitere Manipulation solcher Verbindungen beinhalten. Überprüfen Sie das Sicherheitsdatenblatt (SDS) der verbindungen, die vor Beginn des Experiments verwendet wurden, und beachten Sie streng alle im Forschungszentrum festgelegten Sicherheitsmaßnahmen, einschließlich der Verwendung einer angemessenen persönlichen Schutzausrüstung (PSA). Tragen Sie während des Experiments die richtige Schutzkleidung und verwenden Sie die richtige Abschirmung. Rückstände in den entsprechenden Abfallbehältern (biologische/zytotoxische Abfälle) entsorgen.
HINWEIS: Dieses Protokoll beginnt mit Zellen oder Proben von Probanden, die mit einem KDM1A-Hemmer behandelt wurden, und deren unbehandelten oder fahrzeug-/placebobehandelten Kontrollen3.
- Mit Fahrzeug- oder KDM1A-Inhibitor in vitro behandelte Zellen
- Für die in Suspension angebauten Zellen, wie 10 ml Kulturen, übertragen Sie die Suspensionen in saubere 15 ml konische Rohre und gehen Sie auf 1.1.3.
- Für die anhaftenden Zellen (in 75 cm2 Kolben angebaut) das Medium aus dem Kolben entfernen und mit 4 ml PBS kurz waschen. Lösen Sie die Zellen von ihren Gefäßen mit 1,5 ml 0,5% Trypsin-EDTA während 2 - 5 min (Die Trypsinisierungsbedingungen können variieren, folgen Sie den Empfehlungen des Anbieters für die Zelllinie), fügen Sie 4 ml PBS hinzu und übertragen Sie die Zellen in saubere 15 ml konische Röhren.
- Legen Sie die Rohre in eine Tischzentrifuge ein und sammeln Sie die Zellen durch Zentrifugation für 5 min bei 400 x g bei 4 °C. Entfernen Sie den Überstand, setzen Sie das Pellet in 1 ml PBS, das mit einer Mikropipette abgegeben wird, wieder ab und übertragen Sie die Suspension in ein 1,5 ml Mikrozentrifugenrohr.
- Legen Sie die Proben in eine Mikrorohrzentrifuge und zentrifugieren Sie sie für 5 min bei 400 x g bei 4 °C. Entfernen Sie die PBS durch Aspiration mit einer Mikropipette und entweder halten Sie die Pellets auf dem Eis und gehen Sie zu Schritt 2; oder die Pellets auf Trockeneis einfrieren und bei -80 °C bis Schritt 3 aufbewahren.
- Proben von Probanden oder Tieren, die mit Fahrzeug/Placebo oder KDM1A-Hemmer behandelt wurden
- Gewebe: Schneiden Sie das Gewebe mit einem Skalpell in kleine, ca. 1 cm3 Stück. Die Gewebeteile in Flüssigkeit N2 in einem Dewar-Behälter einfrieren und bei -80 °C bis Schritt 3 aufbewahren.
- Polymorphe mononukleäre Blutkörperchen (PBMCs): Verdünnen Sie 10 ml frischesBlut (Prozessmaximum 2 h nach Blutentnahme), die in K 2-EDTA-Röhrchen mit 2 PbS-Volumen in einem 50 ml konischen Röhrchen gesammelt werden. Isolieren Sie die PBMCs aus Blut mit kommerziell erhaltenen PBMC-Trennröhrchen gemäß den Anweisungen des Herstellers. Pellets auf dem Eis aufbewahren und mit Schritt 3.2 fortfahren; oder die Pellets auf Trockeneis einfrieren und bei -80 °C bis Schritt 3 aufbewahren.
HINWEIS: Ein Nasszellpellet von 20 bis 50 l enthält je nach Zellgröße 1 x 107 Zellen. Ein nasses PBMC-Pellet aus 10 ml gesundem menschlichen Blut hat ein Volumen von 20 l und enthält 1 x 107 PBMCs. Gewebe oder Zellpellets können bei -80 oC für bis zu 6 Monate gelagert werden.
2. Lösungsvorbereitung
- Bereiten Sie die Arbeitslösung OG-881 vor: Nehmen Sie aus dem 4 °C-Kühlschrank eine 10-L-Einweg-Aliquot der 20 mM Biotinyton-Lösung OG-881 und lassen Sie sie 10 min bei Raumtemperatur (RT). Bereiten Sie die 2-Ml-Arbeitslösung durch serielle Verdünnung der OG-881-Stoffsolut vor. Ion in PBS, mit einer Mikropipette mit Filterspitzen und Wechsel der Spitze zwischen den verschiedenen Verdünnungsschritten.
- 10x Protease-Inhibitor vorbereiten: 1 Tablette in 1 ml PBS in einem Mikrozentrifugenrohr auflösen.
- Bereiten Sie das gewünschte Volumen des 1x Zelllysepuffers mit 25 nM OG-881 Chemoprobe vor. Für jede ml 100 l kommerziell erhaltenen 10x Zelllysepuffer, 150 l 10x Protease-Hemmer, 12,5 l mit 2 'M OG-881 und 737,5 'L des Typ 1 Doppeldestilliertes Wasser mischen.
- Optional bereiten Sie das gewünschte Volumen von 1x Zelllysepuffer vor, jedoch mit 25 nM ORY-1001 anstelle von OG-881 wie in Schritt 2.3. Weniger potente Inhibitoren können verwendet werden, können aber höhere Konzentrationen erfordern, für den Einsatz in der Positivkontrolle mit 100%iger Hemmung (siehe Schritt 3.5).
HINWEIS: Ergreifen Sie geeignete Maßnahmen, um eine unbeabsichtigte Kontamination von Lösungen oder Proben mit den Inhibitor-Lagerlösungen OG-881 oder KDM1A zu vermeiden. Um das gewünschte Volumen des 1x Zelllysepuffers mit 25 nM OG-881 zu berechnen, gehen Sie davon aus, dass 400 l pro 40 mg pulverisiertes Gewebe oder 200 l pro Nasspellet von 107 Zellen erforderlich sind.
3. Native Proteinextraktion
- Aus Geweben:
- Mit einem Mörtel und auf Trockeneis gekühltem Stößel einen 1 cm3 Würfel gefrorenen Gewebes pulverisieren und homogenisieren. Aliquot die Proben in Einweg-Fläschchen mit einem Wert von 40 mg Gewebepulver, vermeiden Sie das Auftauen zu jeder Zeit. Fahren Sie mit Schritt 3.1.2 fort. zur sofortigen Verarbeitung oder Lagerung bei -80 °C.
- 40 mg pulverisiertes Gewebe in 400 l 1x Zelllysepuffer mit 25 nM OG-881, Wirbel für 10 s, aufheben und die Probe mindestens fünfmal durch eine 18-Spur stumpfe Spritzennadel zwingen, bis eine Lyse des Gewebes erreicht ist und eine trübe hellgelbe bis orange Suspension Erhalten. Vermeiden Sie Blasenbildung.
- Weiter zu Schritt 3.3
- Aus Zellpellets (PBMCs und Zelllinien):
- Setzen Sie ein Pellet von 1 x 107 Zellen in 200 l 1x Zelllysepuffer mit 25 nM OG-881 wieder auf. Wirbeln Sie die Proben kurz und halten Sie sie auf Eis für 5 min.
- Sonicate die Proben in einem Beschallungsgerät mit 3 Impulsen von je 20 s bei 45 kHz; 20 s zwischen den Impulsen auf Eis legen.
HINWEIS: Sobald die biologischen Proben im 1x Zelllysepuffer resuspendiert wurden, halten Sie sie während des restlichen Prozesses auf dem Eis.
- Die Proben zusätzlich 5 min auf Eis halten, kurz wirbeln und die Proben 10 min bei 14 000 x g in einer vorgekühlten Zentrifuge bei 4 °C zentrifugieren.
- Mit einer 1 ml Mikropipette die Überräube in frische 1,5 ml Mikrozentrifugenrohre übertragen und während 2 h auf dem Eis lassen. Fahren Sie mit Schritt 4 fort.
- Optional kann eine positive Steuerung zur Simulation eines Zielengagements von 100 % wie folgt vorbereitet werden:
- Das Zellpellet oder Pulvergewebe aus einem Fahrzeug oder einer unbehandelten (Prädosis)-Probe im erforderlichen Volumen des 1x Zelllysepuffers mit 25 nM ORY-1001 wieder aufsetzen und wie in Schritt 3.1 bis 3.3 beschrieben verarbeiten.
- Die Überräube der Positivkontrolle in frische 1,5 ml Mikrozentrifugenrohre übertragen und 1 h auf Eis lassen. ORY-1001 hemmt absichtlich die KDM1A und blockiert die Chemoprobebindung.
- Fügen Sie dem Positivkontroll-Überstand (Volumen für die positivkontrollierbare Austragung einer 40 mg-Gewebeprobe) bzw. 2,5 l mit 2 M OG-881 Arbeitslösung (Volumen für die positive Kontrolle, das aus einer 10 7-Zell-Probe erzeugt wird) 5 l l von 2 M OG-881 hinzuzufügen, um die gleiche OG-881-Konzentration wie die anderen Proben und lassen Sie auf Eis während 2 h. Fahren Sie mit Schritt 4 fort.
4. Quantifizierung des nativen Proteins mit Bradford-Assay
- Verdünnen Sie das kommerziell beschaffte Bradford Protein Assay Reagenz 5 Mal mit H2O Typ 1 doppelt destilliertem Wasser. Berechnen Sie das Volumen des Reagenzes, das für die Gesamtmenge der Proben und Standards erforderlich ist (1 ml pro Probe oder Standard + 5 ml Überschussvolumen).
- Für die Standardkurve Bovine Serum Albumin (BSA) bereiten Sie ein Mikrozentrifugenrohr mit 1 ml verdünnter Bradford Protein Assay Lösung (leer) und sieben Mikrozentrifugenröhren mit 995 l der verdünnten Bradford Protein Assay-Lösung vor. Fügen Sie jedem der 7 Mikrozentrifugenrohre jeweils 5 L der BSA-Normen (Konzentration von 125 bis 2.000 g/ml) hinzu und mischen Sie sie, indem Sie die Rohre mehrmals sanft invertiert. 5 min bei RT inkubieren.
- Übertragen Sie die verdünnten Standards auf Küvetten und lesen Sie die OD der Rohlings- und Bovine Serum Albumin Standard-Proben in einem Spektralphotometer bei 280 nm.
- Für die biologischen Proben bereiten Sie ein Mikrozentrifugenrohr mit 1 ml verdünnter Bradford Protein Assay Lösung (leer) und ebenso viele Mikrozentrifugenröhrchen mit 999 l des verdünnten Bradford Protein Assay Reagenz als Proben vor, die quantifiziert werden müssen. Mit einer automatischen P2-Mikropipette, fügen Sie 1 l nativen Proteinextrakt in Schritt 3 zu jedem Mikrozentrifugenrohr vorbereitet und mischen, indem Sie die Rohre vorsichtig mehrmals invertieren. Inkubieren Sie die Proben 5 min bei RT.
- Übertragen Sie die Volumina auf Küvetten und lesen Sie die OD der Proben in einem Spektralphotometer bei 280 nm.
- Vorzugsweise gehen Sie sofort mit Schritt 5 fort. Alternativ können Sie die nativen Proteinextrakte bei -80 °C bis Schritt 5 aufbewahren. Vermeiden Sie Tauwetterzyklen einfrieren.
5. Lumineszierende ELISAs für die vollständige und freie KDM1A-Bestimmung
HINWEIS: Halten Sie die Labortemperatur konstant bei 23-24 °C (RT).
- Beschichtung von Mikrotiterplatten mit Capture KDM1A Antikörper oder Streptavidin
- KDM1A ELISA insgesamt: Bereiten Sie für jede Platte 10 ml KDM1A-Capture-Antikörper auf eine Endkonzentration von 2 g/ml in PBS vor. Übertragen Sie 100 l in jede Bohrung der Platte.
- Kostenloser KDM1A ELISA: Für jede Platte 10 ml Streptavidin bei 10 g/ml in PBS vorbereiten. Übertragen Sie 100 l in jede Bohrung der Platte.
- Die Total- und Free KDM1A ELISA-Platten mit Klebefolie top-sealieren und die Platten über Nacht bei 4 °C im Kühlschrank bebrüten.
- Waschen und Blockieren der Platten
- Nehmen Sie die Teller aus dem Kühlschrank und lassen Sie sie vor Gebrauch ca. 45 min bei RT ausdemieren.
- Bereiten Sie 1.000 ml Waschpuffer (0,1% Tween in PBS) und 50 ml Sperrpuffer (1% BSA in PBS) pro Platte vor.
- Waschen Sie die Platten 3 mal mit Waschpuffer. Tippen Sie in diesem und den nachfolgenden Schritten nach jedem Waschschritt auf die Platte auf Papiertücher, um die Restlösung zu entfernen.
- Fügen Sie 200 l Blockierpuffer pro Brunnen auf beide Platten, top-seal beide Platten mit einem Klebefilm und inkubieren 2 h bei RT.
- Biologische Probenvorbereitung
- Verdünnen Sie die nativen Proteinextrakte, die am Ende von Schritt 3 erhalten wurden, mit PBS auf die entsprechende Konzentration. Die empfohlene Konzentration variiert in Abhängigkeit von der KDM1A-Expression in der biologischen Probe. Beispiele für geeignete Bereiche sind (1) Zellpellets: 0,5 - 10 g pro Brunnen. (2) PBMCs: 5 - 30 g pro Brunnen. (3) Pulverisiertes Gewebe (Gehirn, Lunge, Haut): 20 - 100 g pro Brunnen. Bewahren Sie die Proben während der Vorbereitung auf dem Eis auf. Führen Sie nach Möglichkeit technische Dreifachstichprobenanalysen durch.
- Vorbereiten einer Standardkurve mit humanem rKDM1A:
- Zur Vorbereitung der KDM1A-Standard-Arbeitslösung das entsprechende Volumen von rKDM1A für eine Endkonzentration von 25 pg/l zu pipenieren, 75 l von 2 'M OG-881 hinzuzufügen und mit 1 x PBS zu einem Gesamtvolumen von 6 ml in einem 15 ml Falkenrohr zu vervollständigen. Halten Sie die KDM1A Standard-Arbeitslösung während 1 h auf Eis und mischen Sie die Lösung vorsichtig, indem Sie das 15 ml Falkenrohr mehrmals alle 20 min inumlegen.
- Bereiten Sie die KDM1A Standard-Verdünnungsserie in 1,5 ml Mikrozentrifugenrohren nach Tabelle 1 (Standardvorbereitung) in Volumen vor, genug für die dreifache Analyse von zwei 96 Brunnenmikrotiterplatten.
| STANDARD-SERIE | KDM1A Standard-Arbeitslösung (L) | |
| (pg KDM1A/well) | PBS (L) | |
| 2500 für C-* | 800 | - |
| 2500 | 800 | - |
| 1750 | 560 | 240 |
| 1250 | 400 | 400 |
| 750 | 240 | 560 |
| 250 | 80 | 720 |
| 25 | 8 | 792 |
| 0 | 0 | 800 |
| notiz: | ||
| (1) Das volumen, das von jeder Verdünnung vorbereitet wird, reicht aus, um in dreifacher Ausführung 2 Platten assay zu laufen. | ||
| (2) Der empfohlene Bereich liegt zwischen 2,5 und 5.000 pg / | ||
| * Für die Negativkontrolle C-, ohne KDM1A Detektionsantikörper | ||
Tabelle 1: Standardvorbereitung. Zur Herstellung der Standard-Serie von KDM1A-Protein, Pipette die angegebenen Volumen der KDM1A Standard Arbeitslösung und PBS in acht ordnungsgemäß beschriftete 1,5 ml Mikrozentrifugenrohre.
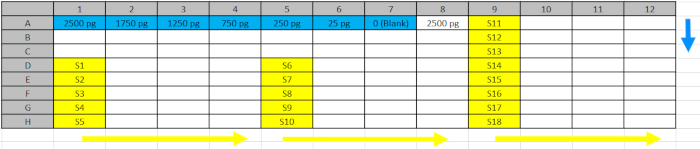
Tabelle 2: Tiefbrunnenplattendesign. Normen (blau) und Proben (gelb) ab Schritt 5.4.2. wurden in die reflektierten Positionen der Tiefenbrunnenplatte geleitet, um das Laden in die ELISA-Platten in Richtung der blauen (Standard) und gelben (Proben) Pfeile zu erleichtern. Bitte klicken Sie hier, um diese Datei herunterzuladen.
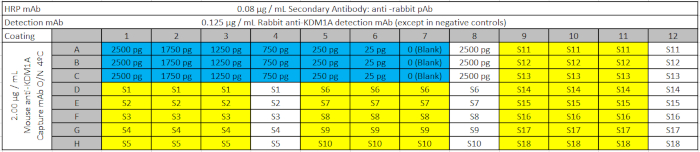
Tabelle 3: ELISA-Plattendesign. Die Assayplatte enthält die Standardkurve mit abnehmenden Mengen des rekombinanten KDM1A-Ziels (in blau); die biologischen Proben (S) in gelb; und entsprechende Negativkontrollen (enthalten die Proben, aber nicht den primären Detektionsantikörper) in Weiß, die von der Deep Well Plate auf die ELISA-Platten geladen werden. Der Rohling (0 in der Standardkurve) enthält alle Erfassungs- und Detektionsreagenzien, aber keine Probe. Bitte klicken Sie hier, um diese Datei herunterzuladen.
- Elisa
- (Fortsetzung von Schritt 5.2.4.) Nach 2 h Inkubation den Sperrpuffer entsorgen und die Platten mit Waschpuffer waschen.
- Übertragen Sie die entsprechend verdünnten Proben (native Proteinextrakte und Standardkurve aus Schritt 5.3.) auf einen gekühlten 96 Tiefbrunnenspeicherblock nach der in Tabelle 2 (Deep Well Plate Design) dargestellten Plattenverteilung.
- Halten Sie diesen Block auf Eis, bis sie 100 l Probe / gut in den Total und Free ELISA Platten nach der Insetable 3 (ELISA Plate Design) Pipettieren.
- 1 h bei RT inkubieren, die Proben entsorgen und die Platten 5 Mal mit Waschpuffer waschen.
- Bereiten Sie 20 ml Kaninchen Anti-KDM1A-Detektionsantikörper bei 0,125 g/ml in Blockierpuffer vor, fügen Sie 100 l pro Brunnen in jede Platte des Assays, außer in Brunnen, die den Negativkontrollen C- entsprechen. Die Platte versiegeln und 1 h bei RT inkubieren.
- Entsorgen Sie die Detektions-Antikörperlösung und waschen Sie die Platten 6 Mal mit Waschpuffer.
- 25 ml sekundärer Ziegen-Anti-Kaninchen-Antikörper HRP auf eine Verdünnung von 1:5.000 im Sperrpuffer vorbereiten, 100 l pro Brunnen zu den Mikrotiterplatten hinzufügen; und 1 h bei RT inkubieren.
-
Chemilumineszenz-Erkennung
- 30 min vor dem Ende von Schritt 5.4.7. und unter weichen Lichtbedingungen zu gleichen Teilen von Luminol-Enhancer und Peroxidlösung (10,5 ml: 10,5 ml, für 2 Platten) in eine Bernsteinflasche mischen und bei RT belassen.
HINWEIS: Bewahren Sie die Luminol Working Solution in einer Bernsteinflasche auf und vermeiden Sie eine längere Exposition gegenüber intensivem Licht. Die kurzfristige Exposition gegenüber typischer Laborbeleuchtung schadet der Arbeitslösung nicht. - Mindestens 20 min vor der Messung der Lumineszenz, schalten Sie den Mikroplattenleser bei 25 °C ein und richten Sie Auslesungen auf 1.000 ms Integrationszeit und 150 ms Einsendezeit ein. Parametereinstellungen können eine Optimierung in Funktion des Instruments erfordern.
- Nach 1 h Inkubation in Schritt 5.4.7. die sekundäre Antikörperlösung entsorgen und die Platten 6 Mal mit Waschpuffer waschen.
- Pipette 100 l pro Bohrung der Luminol Arbeitslösung (Chemiluminescent Substrate), die in Schritt 5.5.1 hergestellt wird. Pipette sehr langsam und vermeiden Blasenbildung. Verwenden Sie einen Timer, um die Zeit zwischen dem Zusatz der Lösung und der Lumineszenzmessung der Platten zu steuern und diese Zeit konstant zu halten, um eine gute Inter-Assay-Reproduzierbarkeit zu erreichen.
- Versiegeln Sie die Platten und die Zentrifuge bei RT für 45 s in einer Plattenzentrifuge auf 500 x g, um restliche Blasen zu beseitigen. Inkubieren Sie die Platten für 1 min auf einem Plattenschütter bei 100 Rpm.
- Legen Sie die Platte in den Vorleser ein und lassen Sie sie 3 min liegen, um die Temperatur bei 25 °C (ohne Klebefolie) zu stabilisieren. Beginnen Sie immer mit der Kostenlosen ELISA-Platte.
- Lesen Sie die relativen Lumineszenzeinheiten (RLU) jedes ELISA-Platten-Assays (frei und gesamt KDM1A).
- Speichern und kopieren Sie die Raw RLU-Werte aus den Excel-Dateien für Raw Data, um die Ergebnisse weiter zu analysieren.
- 30 min vor dem Ende von Schritt 5.4.7. und unter weichen Lichtbedingungen zu gleichen Teilen von Luminol-Enhancer und Peroxidlösung (10,5 ml: 10,5 ml, für 2 Platten) in eine Bernsteinflasche mischen und bei RT belassen.
6. Berechnung des Zielengagements
- Berechnen Sie in einer Tabellenkalkulationssoftware die RLU Free- und RLU-Gesamtwerte der Proben SX und Referenzproben REF (unbehandelt, Fahrzeug- oder Vordosisprobe) aus ihren technischen Replikationsrohdaten, wie unten beschrieben:
- Geben Sie die einzelnen Raw RLUi Total und Raw RLUi Freie Daten aus Rohlingen, Standardkurven, Negativkontrollen C- und biologische Proben (SX und REF) in das Analysedatenblatt ein (z.B. Excel). Geben Sie außerdem die Beträge (in pg) von KDM1A aus der Standardkurve in das Datenblatt ein.
- Berechnen Sie die Raw-Mittel-RLU, Standardabweichungen und Variationskoeffizienten CVRLU aus den einzelnen Raw Total- und Raw Free RLUi-Daten für jeden technischen Replikationsdatenpunkt.
- Ausreißereliniminierung anwenden (Beispiel für Triplicates): Für jeden einzelnen Raw RLU Total und Raw RLU Freier Datenpunkt RLUi aus einem technischen dreifachen Datenpunkt, Anwenden von Grubbs-Kriterien beim Cv für die Dreifache > 0,15 und Ablehnen einzelner verdächtiger Raw RLU-Werte wann
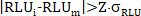 ,
,
wobei Z = 1,148 für n = 3 und 90 % Konfidenzintervall (CI). - Wenn die Ausreißereliniminierung angewendet wurde, berechnen Sie die Raw-Mittel-RLU, die Standardabweichung vonRLU und CVRLU von den nicht abgelehnten (nr) Raw RLUi Total- und Raw RLUi Free-Werten für jeden Datenpunkt neu.
- Anwenden der Hintergrundkorrektur: Berechnen Sie die durchschnittlichen RLU-Freien und RLU-Gesamtwerte für jede Standardstichprobe und jedes Beispiel SX und Referenzbeispiel REF wie:


- Grafische Darstellung der Daten wie folgt:
- Zeichnen Sie die RLU-Freien und RLU-Gesamtwerte (Y-Achse) relativ zu ihrer Probenidentifikation (X-Achse) in einem Balkendiagramm.
- Zeichnen Sie auch die RLU-Werte (Y-Achse) der Standards in einem Streudiagramm relativ zu ihrer Menge an rKDM1A-Protein (X-Achse) für Freie und Gesamtmessungen sowie die entsprechenden Linientrendlinien und berechnen die r2 (das Quadrat der linearen Korrelationskoeffizienten) Werte.
- Berechnen des Zielengagements (TE); d. h. der Anteil der KDM1A, der durch den KDM1A-Inhibitor in jeder Probe SX relativ zu einer Referenzprobe REF (unbehandelte, Fahrzeug- oder Vordosisprobe) gebunden ist, wie folgt:
- Berechnen Sie das Verhältnis R des mittleren RLU-Freien zu Gesamtwerten für die SX- und REF-Samples wie:

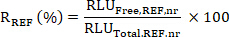
- Berechnen Sie dann das Zielengagement (TE) der Stichprobe SX wie:
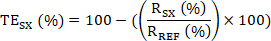
Optional: (1) Wenn N biologische Replikationsexperimente durchgeführt wurden, jede mit n technischen Replikationen; berechnen Sie zuerst den TESX für die technischen Replikationssätze. Anschließend berechnen Sie die mittleren TE-, SD- und CV-Werte für den biologischen Replikationssatz.
- Berechnen Sie das Verhältnis R des mittleren RLU-Freien zu Gesamtwerten für die SX- und REF-Samples wie:
- Überarbeiten, ob die Kriterien für die Assayakzeptanz erfüllt sind: Überprüfen Sie, ob (1) der Assay-Hintergrund akzeptabel ist und der mittlere Leerwert < 0,05 x 107 RLU; (2) die Probe-Autolumineszenz fehlt und die RLUs der Negativkontrollen C- liegen unterhalb der unteren Quantifizierungsgrenze (LLOQ = Mean Blank + 10x SD); (3) die rKDM1A-Standardkurve ist linear und r2 x 0,98; (4) Die biologischen Proben weisen RLU-Werte auf, die in den dynamischen und linearen Bereich des Assays fallen, d. h. zwischen LLOQ und 2.500 pg/well.
HINWEIS: Schritte 6.1. bis 6.4. in einem Berechnungsdatenblatt leicht automatisiert werden. - Exportieren Sie die TE-Daten in eine Open-Source- oder kommerziell erworbene Statistiksoftware der Wahl für die grafische Darstellung der TE-Werte und zusätzliche statistische Auswertungen.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Die Linearität der Total- und Free KDM1A-Bestimmung.
Eine Standard-Serie wurde wie in Schritt 5.3.2. beschrieben unter Verwendung von 0 bis 2500 pg des humanen rekombinanten KDM1A-Enzyms in voller Länge hergestellt. Die RLU-Werte von Total und Free rKDM1A wurden bewertet, um die Linearität zu überprüfen (Abbildung 2A und 2B). Die Daten werden als Mittelwert aus...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Das hier vorgestellte Protokoll wurde entwickelt, um das KDM1A-Zielengagement direkt mit einem neuartigen KDM1A Chemoprobe Capture-basierten ELISA zu messen. Die Methode wurde an kultivierten menschlichen Zelllinien und Ex-vivo-Proben von Mensch, Ratte und Maus und Pavian (einschließlich PBMCs, Lunge, Gehirn, Haut, Tumoren) validiert, kann aber leicht auf andere Arten angewendet werden, bei denen die KDM1A-Antikörper Epitope und katalytische Mitte konserviert werden. Da OG-881 eine aktivitätsbasierte Chemoprobe ist, i...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autorin Tamara Maes ist geschäftsführende Direktorin und Aktionärin, und die Autoren Cristina Mascara und Raquel Ruiz Rodriguez sind Mitarbeiter von Oryzon Genomics S.A. Oryzon Genomics S.A. entwickelt KDM1A-Inhibitoren und hält Patente für Verbindungen und Methoden in diesem Artikel.
Danksagungen
Diese Studie wurde von Oryzon Genomics finanziert. S.A., Hoffman-La Roche, teilweise unterstützt durch das CIIP-20152001 und RETOS Kollaborationsprogramm RTC-2015-3332-1.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 0,05% Trypsin-EDTA (1X) | Thermo Scientific | #25300-062 | |
| 10 X Protease Inhibitor Tablets | Roche | #11836153001 | |
| 96 deep well storage block | VWR | #734-1679 | |
| 96 well ELISA plates | Nunc | #436110 | |
| Adhesive black Film | Perkin Elmer | #6050173 | |
| Adhesive transparent Film | VWR | #60941-062 | |
| Biotinylated KDM1A probe OG-881 | Oryzon Genomics S.A. | NA | |
| Bovine Serum Albumin | Sigma | # 3117057001 | |
| Bovine Serum Albumin Standard | Thermo Scientific | #23208) | |
| Bradford Protein Assay | BioRad | #500-0001 | |
| Cell lysis buffer 10X | Cell Signaling | #9803 | |
| Centrifuge for 96- well plates | Hettich | Rotina 420R | |
| Flask | Thermo Scientific | #156499 | |
| Full length, enzymatically active human Recombinant LSD1 / KDM1A | Active Motif | #31426 | |
| Graphpad Prism 5 Project | GraphPad Software | NA | |
| Luminol-Enhacer and Peroxide Solution (Chemiluminescent Substrate) | Thermo Scientific | #37074 | |
| Micro Centrifuge | Eppendorf | 5415 R | |
| Microplate reader Infinite 200-Tecan | Tecan | Infinite 200 | |
| Mouse monoclonal capture antibody Anti-KDM1A (N-terminal epitope) | Abcam | #ab53269 | |
| Needle G18 gauge blunt | BD | #303129 | |
| ORY-1001 (iadademstat) | Oryzon Genomics S.A. | NA | |
| PBMC separation tubes 10 ml | Greiner bio-one | #163288 | |
| PBMC separation tubes 50 ml | Greiner bio-one | #227288 | |
| PBS 1x | Sigma | #D8537 | |
| Plate shaker | Heidolph Instruments | Rotamax 120 | |
| Polysorbate 20 | Sigma | #P7949 | |
| Rabbit monoclonal detection antibody Anti-KDM1A (C-terminal epitope) | Cell Signaling | #672184BF-100 | |
| Secondary antibody Peroxidase-conjugated Donkey Anti-rabbit IgG | Thermo Scientific | #31458 | |
| Spectrophotometer cuvette 1.5 | Deltalab | #302100 | |
| Spectrophotometer for cuvette | GE Healthcare | GeneQuant 1300 | |
| Streptavidin | Promega | #Z704A | |
| Syringe | BD | #303172 | |
| Type 1 ultrapure water | Millipore | Milli-Q Advantage A10 | |
| Ultrasonic cleaner | VWR | USC200T |
Referenzen
- Shi, Y. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1. Cell. 119 (7), 941-953 (2004).
- Maiques-Diaz, A., Somervaille, T. C. LSD1: biologic roles and therapeutic targeting. Epigenomics. 8 (8), 1103-1116 (2016).
- Maes, T. ORY-1001, a Potent and Selective Covalent KDM1A Inhibitor, for the Treatment of Acute Leukemia. Cancer Cell. 33 (3), 495-511 (2018).
- Sugino, N. A novel LSD1 inhibitor NCD38 ameliorates MDS-related leukemia with complex karyotype by attenuating leukemia programs via activating super-enhancers. Leukemia. 31 (11), 2303-2314 (2017).
- Kleppe, M., Shank, K., Efthymia, P., Riehnhoff, H., Levine, R. L. Lysine-Specific Histone Demethylase, LSD1, (KDM1A) As a Novel Therapeutic Target in Myeloproliferative Neoplasms. Blood. 126, 601(2015).
- Jutzi, J. S., et al. LSD1 Inhibition Prolongs Survival in Mouse Models of MPN by Selectively Targeting the Disease Clone. HemaSphere. 2 (3), 54(2018).
- Mohammad, H. P. DNA Hypomethylation Signature Predicts Antitumor Activity of LSD1 Inhibitors in SCLC. Cancer Cell. 28 (1), 57-69 (2015).
- Rivers, A., et al. RN-1, a potent and selective lysine-specific demethylase 1 inhibitor, increases γ-globin expression, F reticulocytes, and F cells in a sickle cell disease mouse model. Experimental Hematology. 43 (7), 546-553 (2015).
- Rivers, A. Oral administration of the LSD1 inhibitor ORY-3001 increases fetal hemoglobin in sickle cell mice and baboons. Experimental Hematology. 67, 60-64 (2018).
- Buesa, C., et al. The dual LSD1/MAO-B inhibitor ORY-2001 prevents the development of the memory deficit in samp8 mice through induction of neuronal plasticity and reduction of neuroinflammation. Alzheimer’s & Dementia. 11 (7), P905(2015).
- Schmidt, D. M., McCafferty, D. G. Trans-2-Phenylcyclopropylamine is a mechanism-based inactivator of the histone demethylase LSD1. Biochemistry. 46 (14), 4408-4416 (2007).
- Forneris, F., Binda, C., Vanoni, M. A., Battaglioli, E., Mattevi, A. Human histone demethylase LSD1 reads the histone code. Journal of Biological Chemistry. 280 (50), 41360-41365 (2005).
- Oryzon Genomics, Methods to determine kdm1a target engagement and chemoprobes useful therefor. , WO2017158136 (2016).
- Mascaró, C., Ortega, A., Carceller, E., Rruiz Rodriguez, R., Cicero, F., Lunardi, S., Yu, L., Hilbert, M., Maes, T. Chemoprobe-based assays of histone lysine demethylase 1A target occupation enable in vivo pharmacokinetics and -dynamics studies of KDM1A inhibitors. Journal of Biological Chemistry. , In Press (2019).
- Rodriguez-Suarez, R. Development of Homogeneous Nonradioactive Methyltransferase and Demethylase Assays Targeting Histone H3 Lysine 4. Journal of Biomolecular Screening. 17 (1), 49-58 (2011).
- Lynch, J. T., Cockerill, M. J., Hitchin, J. R., Wiseman, D. H., Somervaille, T. C. CD86 expression as a surrogate cellular biomarker for pharmacological inhibition of the histone demethylase lysine-specific demethylase 1. Analytical Biochemistry. 442 (1), 104-106 (2013).
- Schulz-Fincke, J. Structure-activity studies on N-Substituted tranylcypromine derivatives lead to selective inhibitors of lysine specific demethylase 1 (LSD1) and potent inducers of leukemic cell differentiation. European Journal of Medicinal Chemistry. 144, 52-67 (2018).
- Ishii, T., et al. CETSA quantitatively verifies in vivo target engagement of novel RIPK1 inhibitors in various biospecimens. Scientific Report. 7, 13000(2017).
- Maes, T. ORY-2001: An Epigenetic drug for the treatment of cognition defects in Alzheimer’s disease and other neurodegenerative disorders. Alzheimer’s & Dementia. 12 (7), P1192(2017).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten