Method Article
Gezielte Studien mit serial Block Face und Focused Ion Beam Scan Electron Microscopy
In diesem Artikel
Zusammenfassung
Hier präsentieren wir ein Protokoll zur effizienten Kombination von serieller Blockfläche und fokussierter Ionenstrahlelektronenmikroskopie für einen Interessenbereich. Dies ermöglicht eine effiziente Suche in drei Dimensionen und das Auffinden seltener Ereignisse in einem großen Sichtfeld.
Zusammenfassung
Dieses Protokoll ermöglicht eine effiziente und effektive Abbildung von Zell- oder Gewebeproben in drei Dimensionen auf der Auflösungsebene der Elektronenmikroskopie. Die Elektronenmikroskopie (EM) ist seit vielen Jahren eine von Natur aus zweidimensionale Technik. Mit dem Aufkommen von seriellen Rasterelektronenmikroskop-Bildgebungstechniken (Volumen EM), die entweder ein integriertes Mikrotom oder einen fokussierten Ionenstrahl verwenden, um eingebettete Gewebe zu schneiden und dann zu betrachten, wird die dritte Dimension leicht zugänglich. Die serielle Blockflächenelektronenmikroskopie (SBF-SEM) verwendet ein in der SEM-Kammer eingeschlossenes Ultramikrotom. Es hat die Fähigkeit, große Proben (1.000 x 1.000 m) zu verarbeiten und große Sichtfelder bei kleinen X,Y-Pixelgrößen abzubilden, ist aber in der Z-Dimension durch das Diamantmesser begrenzt. Fokussierter Ionenstrahl SEM (FIB-SEM) ist in 3D-Auflösung nicht begrenzt (isotrope Voxel von 5 nm sind erreichbar), aber das Sichtfeld ist viel begrenzter. Dieses Protokoll veranschaulicht einen Workflow zum Kombinieren der beiden Techniken, um einzelne Interessengebiete (ROIs) in einem großen Feld zu finden und dann das anschließende Zielvolumen mit hoher isotroper Voxelauflösung abzubilden. Die Vorbereitung fester Zellen oder Gewebe ist für Volumen-EM-Techniken aufgrund der zusätzlichen Kontraste, die für eine effiziente Signalerzeugung in der SEM-Bildgebung erforderlich sind, anspruchsvoller. Solche Protokolle sind zeitaufwändig und arbeitsintensiv. Dieses Protokoll beinhaltet auch eine mikrowellengestützte Gewebeverarbeitung, die das Eindringen von Reagenzien erleichtert, wodurch die für das Verarbeitungsprotokoll benötigte Zeit von Tagen auf Stunden reduziert wird.
Einleitung
Dieses Protokoll beschreibt einen Workflow für die effiziente Ausrichtung der hochauflösenden, dreidimensionalen Elektronenmikroskopie (EM) auf eine bestimmte Interessenregion (ROI). Seit seinen Anfängen in den 1930er Jahren ist EM eine im Wesentlichen zweidimensionale Technik. Die ersten veröffentlichten Bilder waren von ganzen Geweben oder Zellen, aber das gab bald Abschnitten, die von Hand mit einem Ultramikrotom geschnitten und mit einem Transmissionselektronenmikroskop (TEM) abgebildet wurden. TEM produziert sehr hochauflösende Mikrographien, bei denen selbst kleinste Zellstrukturen deutlich erkennbar sind. Die dünne Sektion, die notwendig ist, damit das Gewebe vom Elektronenstrahl abgebildet wird, machte die Informationen in der Z-Dimension jedoch minimal. Da es sich bei Zellen um dreidimensionale Strukturen handelt, mussten Wechselwirkungen zwischen Zellstrukturen und Zelloberflächen aus begrenzten Daten abgeleitet werden. Dies erhöhte das Potenzial für Fehlinterpretationen, insbesondere in komplexen Strukturen. Einigen Mikroscopisten gelang es, genauere 3D-Strukturen durch serial-sectioning-Zellen und Gewebe zu erhalten und sie dann sorgfältig aus einzelnen TEM-Bildern zu rekonstruieren1. Dies war ein sehr arbeitsintensiver Prozess und vor dem Aufkommen der digitalen Bildgebung und Computer-Rendering waren die Ergebnisse auch schwer zu visualisieren. In den letzten Jahren wurden zwei Techniken eingeführt, die kollektiv als Volumenelektronenmikroskopie (Volumen EM)2 bekannt geworden sind, die EM in drei Dimensionen für mehr Laboratorien zugänglich gemacht haben.
Die Idee, einen Stapel von Bildern aus einem eingebetteten Block in einem Elektronenmikroskop zu erhalten, kann bis 1981 zurückverfolgt werden, als Steve Leighton und Alan Kuzirian ein Miniaturmikrotome bauten und es in die Kammer eines Rasterelektronenmikroskops3 (SBF-SEM) legten. . Dieser Prototyp wurde schließlich 23 Jahre später von Denk und Horstmann4 kopiert und verbessert und anschließend kommerzialisiert. Etwa zur gleichen Zeit wurden biologische Wissenschaftler auf eine andere Technologie aufmerksam, die vor allem in der Materialwissenschaft verwendet wird, der fokussierte Ionenstrahl. Diese Technik verwendet einen Ionenstrahl irgendeiner Art (Gallium, Plasma), um eine sehr kleine Menge an Oberflächenmaterial aus einer Probe (FIB-SEM)5zu entfernen. Beide Techniken verwenden Schnitte, gefolgt von Bildgebung, die eine Reihe von Bildern bereitstellt, die zu einem X-, Y-, Z-Stack kombiniert werden können. Beide Techniken liefern 3D-Informationen, jedoch in unterschiedlichen Auflösungsskalen. SBF-SEM wird durch die physikalischen Eigenschaften des Diamantmessers auf Scheiben von nicht dünner als 50 nm für lange serielle Bildgebungsläufe beschränkt; Die schnittige Probenblockgröße ist jedoch groß, bis zu 1 mm x 1 mm x 1 mm. Durch das große digitale Erfassungsformat des rückgestreuten Elektronendetektors (32k x 32k Pixel), der ein Signal von der Blockfläche empfängt können Bildpixelgrößen von bis zu 1 nm betragen. Dies führt zu nicht-isotropen Voxeln, bei denen die X,Y-Dimension häufig kleiner als die Z ist. Aufgrund der Präzision des Ionenstrahls hat FIB-SEM die Möglichkeit, Bilder mit isotropen Voxeln zu sammeln. Die Gesamtfläche, die abgebildet werden kann, ist jedoch recht klein. Eine zusammenfassende Tabelle mit verschiedenen Beispielen und Bänden, die mit den beiden Techniken abgebildet sind, wurde zuvor veröffentlicht3.
Die Gewebevorbereitung für Volumen EM ist schwieriger als bei Standard-TEM oder SEM, da Proben gebeizt werden müssen, um eine ausreichende Signalerzeugung im SEM zu gewährleisten. Häufig müssen Färbungen nicht nur für den jeweiligen Gewebetyp, sondern auch für die Zugabe optimiert werden. im Gegensatz zu bestimmten zellulären Strukturen, um die Identifizierung und Rekonstruktion zu erleichtern. Das hier verwendete Protokoll basiert auf dem NCMIR-Standard6. Zusätzliche Färbung bedeutet in der Regel zusätzliche Protokollschritte. Daher müssen für Volumen EM Standardprotokolle erweitert werden, um sicherzustellen, dass genügend Zeit für Reagenzien ist, um in die Probe einzudringen. Mikrowellenunterstützte Verarbeitung kann die Zeit für die Färbung von Stunden auf Minuten reduzieren und macht Volumen EM Probe Vorbereitung effizienter7. Diese Methode gilt für alle Zell- und Gewebetypen8 und für Forschungsfragen, bei denen die Inhomogenität des Gewebes die Probenahme eines bestimmten Bereichs wesentlich macht9.
Sobald ein Datenstapel abgerufen ist, kann er ausgerichtet und die Strukturen von Interesse von den übrigen Daten segmentiert und in 3D modelliert werden. Obwohl die Automatisierung der Bildgebung viele Teile des Gewebes relativ einfach gemacht hat, ist der Prozess der digitalen Rekonstruktion und Visualisierung der Daten eine zeitaufwändige Aufgabe. Software zu diesem Zweck ist noch nicht integriert oder vollautomatisiert. Da ein Großteil der frühen Arbeiten mit Volumen EM auf die Neurowissenschaften ausgerichtet war, sind die Techniken zur Färbung und digitalen Segmentierung von Strukturen wie Axonen im Vergleich zu anderen Zellen und Organellen ziemlich weit fortgeschritten. Während die Literatur über andere nicht-neuronale Gewebe schnell wächst, erfordern nichtlineare oder unregelmäßige Strukturen mehr manuelle Eingabe.
Die Verwendung von SBF-SEM und FIB-SEM ist ein nützlicher Ansatz für die Ausrichtung und Abbildung spezifischer, nicht homogener Gewebestrukturen mit hoher Auflösung in 3D. Die Kombination mit einer Mikrowellen-gestützten Gewebeverarbeitung, die die Zeit für die Probenvorbereitung erheblich verringert. Zusammen wird dieser Workflow die Erzeugung hochauflösender isotroper Voxel-Bild-Datensätze von feinen Strukturen zu einem effizienten und schnelleren Prozess machen.
Protokoll
1. Probenfixierung und -verarbeitung für die Elektronenmikroskopie
- Fix Sämlinge von Arabidopsis thaliana auf Agarplatten in 0,5% Paraformaldehyd, 2,5% Glutaraldehyd in 0,1 M Phosphatpuffer (PB) pH 6,8 für 2 h bei Raumtemperatur (RT).
VORSICHT: Aldehyde sind reizende und korrosive und haben krebserregendes, erbgutveränderndes und teratogenes Potenzial. Alle Lösungen müssen mit entsprechender Schutzausrüstung und einer Dunstabzugshaube behandelt werden. - In Schritt 1.1 gewachsene Wurzelspitzen der Pflanze schneiden und 2-3 Spitzen in 0,5 ml-Rohre geben, die bei 4 °C über Nacht das gleiche Fixativ enthalten.
ANMERKUNG: Das Volumen dieser und aller Lösungen in den verbleibenden Schritten wird durch das Probenvolumen bestimmt; ein Mindestverhältnis von Probe zu Lösung ist 10:1. Proben, die in jeder Dimension größer als 1 mm sind, sind schwer zu färben, so dass die Arbeit mit größeren Gewebeblöcken schwieriger ist. Nicht alle Gewebe haben die gleichen Eigenschaften; Pflanzenblätter und Stiele können z. B. schwer zu färben sein. Wenn größere Proben oder schwierige Gewebetypen gewünscht werden, muss die Optimierung der Probenverarbeitung durchgeführt werden, bevor sie zur Datenerfassung übergehen kann. - Bereiten Sie die Thiocarbohydrazid (TCH) Lösung vor, die vor Schritt 1.7 frisch und verfügbar benötigt wird. 0,1 g Thiocarbohydrazid auf 10 ml doppelt destilliertes Wasser (ddH2O) geben und im Ofen für 1 h auf 60 °C auflösen. Filtern Sie vor der Anwendung die TCH-Lösung mit einem 0,22 m Spritzenfilter.
- Fixativ aus den Rohren entfernen und mit 0,1 M PB pH 6.8 ersetzen. Legen Sie die Rohre bei 100 U/min auf einen Orbital-Schütteltisch und waschen Sie 10 min. Wiederholen Sie die Wäsche mit frischem PBS 5 Mal.
- Post fixieren Sie die Wurzelspitzen, indem Sie PB durch 2% Osmiumtetroxid (OsO4) und 0,2% Rutheniumrot in 0,1 M PB pH 6,8 ersetzen. Setzen Sie die Schläuche in die Mikrowelle mit den Deckeln geöffnet und starten Sie Programm 9 (Tabelle 1).
VORSICHT: Osmium ist bei Einnahme extrem gefährlich, bei Einatmen sehr gefährlich und bei Hautkontakt gefährlich. Behandeln Sie immer mit entsprechender Schutzausrüstung und in einer Dunstabzugshaube.
HINWEIS: Während des gesamten Protokolls sind die Deckel der Röhren während der Mikrowellenschritte immer geöffnet. - Waschen Sie die Wurzelspitzen zweimal mit ddH2O für jeweils 5 min auf der Tischplatte. Für die dritte und vierte ddH2O Waschprogramm 15 auf der Mikrowelle verwenden (Tabelle 1). Nach den ersten 40 Sekunden ddH2O waschen, Proben aus der Mikrowelle nehmen und den Puffer durch frische ddH2O ersetzen. Legen Sie die Proben wieder in die Mikrowelle und setzen Sie das Programm fort.
HINWEIS: Die Mikrowelle schlägt Alarm, wenn der Puffer aktualisiert werden muss. Stellen Sie sicher, dass der Deckel für die Vakuumkammer jedes Mal korrekt ausgetauscht wird. - Inkubationsproben in zuvor vorbereiteter TCH-Lösung bei RT für 2 min auf der Bank und für die weitere Inkubation verwenden Sie das Mikrowellenprogramm 8 (Tabelle 1). Wechseln Sie die Lösung nicht zwischen Bank und Mikrowelle.
- Waschen Sie Proben, wie in Schritt 1.6 beschrieben.
- Proben in 1% OsO4 in ddH2O für Mikrowellenprogramm 9 (Tabelle 1) platzieren
- Waschen Sie Proben, wie in Schritt 1.6 beschrieben.
- Inkubationsproben in 1% Uranylacetat in ddH2O mit Mikrowellenprogramm 16 (Tabelle 1).
VORSICHT: Uranylacetat ist giftig, reizend und hat krebserregendes, erbgutveränderndes und teratogenes Potenzial. Behandeln Sie immer mit entsprechender Schutzausrüstung. - Waschen Sie Proben, wie in Schritt 1.6 beschrieben.
- Bereiten Sie die Lead-Lösung von Walton für den Einsatz in Schritt 1.14 vor. Machen Sie zunächst eine Lagerlösung aus L-Assigsäure, indem Sie 0,998 g L-Assigsäure zu 250 ml ddH2O hinzufügen und den pH-Wert auf 3,8 mit 1 M KOH einstellen. Als nächstes 0,066 g Bleinitrat in 10 ml L-Aspartinsäure-Stammlösung auflösen und pH auf 5,5 einstellen. Lassen Sie die Lösung 30 min bei 60 °C im Ofen.
HINWEIS: Es sollten keine Fällungsfällungen entstehen. - Proben in Waltons Bleilösung 30 min im Ofen bei 60 °C inkubieren.
- Waschen Sie Proben, wie in Schritt 1.6 beschrieben.
- Dehydrieren Sie Proben in EtOH in abgestuften Schritten von 50%, 70%, 90% in ddH2O und dann 2x in 100% EtOH. Verwenden Sie Mikrowellenprogramm 10 (Tabelle 1) und die Mikrowelle wird Benutzer alle 40 s auffordern, die Lösung durch den nächsten EtOH-Schritt zu ersetzen. Dies ist der letzte Schritt in der Mikrowelle.
- Weitere Dehydrierung in 100% Propylenoxid 2x für je 10 min bei RT auf der Bank, ersetzen die Lösung zwischen den Stufen.
HINWEIS: Propylenoxid kann einige Kunststoffe wie Polystyrole auflösen; Verwenden Sie für diesen Schritt entweder Glasfläschchen oder testen Sie Kunststoffe auf Beständigkeit.
VORSICHT: Propylenoxid ist leicht entzündlich. Behandeln Sie immer mit entsprechender Schutzausrüstung und in einer Dunstabzugshaube. - Beginnen Sie mit der Infiltration von Wurzelspitzen, indem Sie 50% Spurrharz in Propylenoxid (min 2 h) inkubieren.
VORSICHT: Die Harzkomponenten von Spurr sind Reizstoffe. Behandeln Sie immer mit entsprechender Schutzausrüstung und in einer Dunstabzugshaube. - Ersetzen Sie die Lösung durch 100% Spurr's und lassen Sie sie über Nacht bei RT.
- Wechseln Sie 2 mal in frisches 100% Spurr-Harz (min 2 h Inkubationen).
- Proben in eine Einbettform geben, wieder frisches 100% Spurr-Harz enthalten und in einem Ofen bei 65 °C für 36 bis 48 h polymerisieren.
HINWEIS: Die verwendete Einbettform hängt von der Art des Gewebes und dem für die Bildgebung verwendeten Ansatz ab. Hier wurde eine flache Silikoneinbettform verwendet (Abbildung 1A).
2. Vorbereiten eingebetteter Proben für die Bildgebung
- Proben aus dem Ofen nehmen, Harz aus der Einbettform entfernen (Abbildung 1B).
- Mit einer Rasierklinge die Probe grob auf einen Block von maximal 0,5 mm x 0,5 mm x 0,5 mm (Abbildung 1C)trimmen.
HINWEIS: Um eine Aufladung in SBF-SEM zu verhindern, ist es wichtig, so viel blankes Harz wie möglich zu schneiden und die Probe so flach/dünn wie möglich zu machen. Im Idealfall enthalten alle Seiten des Blocks bereits Gewebe, aber vor allem muss die Seite, die am Metallstift befestigt wird (siehe Schritt 2.3), exponiertes Gewebe enthalten, so dass das Gewebe in direktem Kontakt mit dem leitfähigen Metall ist. - Entfernen Sie die Probe aus dem Fremdharz und befestigen Sie sie mit leitfähigem Epoxidharz an einem Metallstift (Abbildung 1D), um sicherzustellen, dass ein Teil des Gewebes den Metallstift berührt. Das Epoxid über Nacht im Ofen bei 65 °C aushärten lassen.
HINWEIS: Stellen Sie sicher, dass das Sample in der Mitte des Stifts positioniert ist, da die Bewegung der Bühne im SB-SEM begrenzt ist. Das Entfernen der sehr kleinen harzummantelten Probe aus zusätzlichem Harz kann schwierig sein, da die kleine Probe eine Tendenz hat, wegzufliegen, wenn sie abgelöst wird. Eine einfache und effektive Lösung hierfür besteht darin, die Probe mit einem Blatt Paraffinfolie abzudecken, wie im Zusatzfilm der Referenz1gezeigt. - Befestigen Sie den Träger im Halter für das Ultramikrotom. Verwenden Sie eine Rasierklinge, um überschüssiges Epoxid zu entfernen und verwenden Sie das Ultramikrotom und ein Diamantmesser, um das Gesicht und die Seiten des Blocks zu glätten und eine Pyramide zu bilden. Stellen Sie sicher, dass zumindest ein Teil des Gewebes bereits auf der Blockfläche freigelegt ist.
HINWEIS: Der zusätzliche Schritt der Verwendung eines Diamantmessers ist optional, aber er erleichtert die Annäherung des resultierenden Blocks im SBF-SEM, da der Schatten des Messers auf der Blockfläche klarer ist, wodurch die Schätzung des Abstands zwischen Messer und Blockfläche leichter zu bestimmen ist. - Legen Sie den getrimmten Probenblock in den Sputerlackturm und beschichten Sie die Probe mit einer dünnen Schicht (2 x 5 nm) Platin (Pt).
HINWEIS: Das Platin auf der Blockfläche wird während des Anflugs im SBF-SEM abgeschnitten (siehe unten), aber das Platin an den Seiten der Pyramide sorgt für zusätzliche Leitfähigkeit. In diesem Beispiel wurde die Probe mit Platin beschichtet, aber Gold oder Gold/ Palladium ist auch wirksam; Die Beschichtung mit Gold führte jedoch zu erhöhtem Schmutz auf der Blockfläche während eines Bildlaufs.
3. Bildgebung im SBF-SEM
- Setzen Sie den Träger in das SBF-SEM-Mikroskop ein und bringen Sie das Messer in die Nähe der Probenoberfläche. Mit dem Diamantmesser den oberen Teil der Probe abschneiden, so dass die Pt-Schicht bereits entfernt wurde und zumindest ein Teil des Gewebes freigelegt ist.
HINWEIS: Da dieser Prozess für jedes SBF-SEM-Mikroskop unterschiedlich ist, wird hier nicht jeder Schritt angegeben. Solange die Probenoberfläche frei von Pt ist und zur Bildgebung bereit ist, sind die nächsten Schritte möglich. - Beginnen Sie mit der Bildgebung mit niedriger Auflösung und kurzen Verweilzeiten, um sich einen Überblick über die Probe zu verschaffen und eine Interessenregion zu finden (Abbildung 1E).
HINWEIS: Hier wurden 512 x 512 Pixel und 1 s Verweilzeit für das schnelle Scannen und Positionieren der Bühne verwendet und 2.000 x 2.000 Pixel mit 1 s Verweilzeit wurden für die Optimierung des Bildfensters und die Einstellung des Fokus verwendet. - Erfassen Sie mit einer Beschleunigungsspannung von 1,5-2,0 kV bei einem Strom von 80 x 100 pA ein Bild des Gewebes.
HINWEIS: Das hier gezeigte Beispiel wurde auf einem Hochvakuumsystem abgebildet, bei dem der Strahlstrom angepasst werden muss, um die Ladung zu minimieren, die sehr stichprobenabhängig ist. In der Regel ist der Elektronenstrahl auf 1,5 bis 2,0 kV eingestellt, aber dies wird stichprobenabhängig sein. Bei höheren Vergrößerungen (in der Regel > 10.000x) ist das Harz zu stark vom Strahl betroffen, um eine glatte Schnittung zu gewährleisten, so dass typischerweise die Pixelgröße auf 8 x 20 nm bei einer Bildgröße von 8.000 x 10.000 x 8.000 x 10.000 Pixeln mit einer entsprechenden Vergrößerung von 430 – 1.400x und Feldgrößen von 64 x 64 mm bzw. 200 x 200 mm. - Bestimmen Sie einen Bereich von Interesse und entscheiden Sie, wie viele Abschnitte benötigt werden, um das Volumen des Interesses abzudecken und starten Sie den Bildlauf, indem Sie den rückgestreuten Elektronendetektor verwenden.
HINWEIS: In dem hier vorgestellten Beispiel wurden 500 Abschnitte von 80 nm mit 10 nm Pixeln und 10.000 x 10.000 Pixel-Bildern (Verweilzeit 1 s) abgebildet. Das Mikroskop wurde auf 1,6 kV und 100 pA eingestellt. Im Allgemeinen hängt die Anzahl der Abschnitte von der Stichprobe und der Größe des ROI ab und kann zwischen 100 s und 1.000 s aufeinander folgenden Abschnitten variieren. Das resultierende Dataset besteht aus einzelnen Bildern jedes Abschnitts. Diese Bilder müssen in einen 3D-Stack konvertiert werden.
4. SBF-SEM Datenverarbeitung
- Wählen Sie mit Fidschi die Datei-Import-Bildsequenz aus, und suchen Sie den Bildstapel, um die Bilder zu laden. Abhängig von der Größe des Datasets aktivieren Sie das Kontrollkästchen "Virtueller Stapel verwenden".
HINWEIS: Wenn das Dataset wirklich groß ist, konvertieren Sie es zuerst in 8 Bit (falls bei 16 Bit gesammelt) und behältern Sie die Daten ggf. so lange, bis sie eine praktikabele Größe haben. - Mit der Wiedergabeschaltfläche am unteren Rand des Bildes scrollen Sie durch das Dataset, um zu sehen, ob der Imaging-Lauf erfolgreich war. Prüfen Sie auf typische bildgebende Artefakte für SBF-SEM, wie Abschnitte, die vom Messer auf der Blockfläche fallen, Aufladen in Bereichen aus blankem Harz, Schneiden von Artefakten des Messers (horizontale Linien auf dem Bild).
- Mit dem Befehl Bildeigenschaften passen Sie die Pixelgröße und Voxeltiefe (d. h. Schnittdicke) während der Ausführung verwendet. Wenn die Daten bereits lagergebunden sind, berücksichtigen Sie dies.
- Verwenden des Befehls plugins-registration-linear stack alignment with SIFT, um die Daten zu registrieren.
HINWEIS: Die Registrierung von SBF-SEM-Daten ist erforderlich, da es während der Bildgebung aufgrund des Ladens oder Driftens der Probe zu leichten Probenbewegungen kommt. Da dies nur eine minimale Bewegung in XY ist, ist nur eine Übersetzung erforderlich. - Überprüfen Sie die Ausrichtung, indem Sie durch das Dataset scrollen, und wenn OK den Befehl speichern im Dateimenü verwenden, um das ausgerichtete Dataset als 3D-tif-Datei zu speichern.
- Analysieren Sie das Dataset sorgfältig, um festzustellen, ob der ROI enthalten ist, und enthalten Sie die Informationen, die für die biologische Frage erforderlich sind. Wählen Sie auf dem letzten Bild des Stapels (= die aktuelle Blockfläche) einen neuen ROI für die FIB-SEM-Bildgebung aus.
HINWEIS: Wenn es keine gute Region für DIE FIB-SEM-Bildgebung auf der aktuellen Blockfläche gibt, können weitere Abschnitte aus dem Block (der sich noch im SBF-SEM befindet) geschnitten werden, bis ein ROI erscheint. Es gibt eine Grenze für das Volumen, das mit dem FIB abgebildet werden kann. Der ROI auf dem SBF-SEM-Bild kann in X,Y von 30-40 x 15-20 'm maximal sein.
5. Bildgebung im FIB-SEM
- Nehmen Sie Übersichtsbilder des Beispiels im SBF-SEM, idealerweise mit einer oder mehreren Rändern der Probe, die dann im FIB-SEM erkennbar sind (Abbildung 2A,C).
HINWEIS: In diesem Beispiel wurden die SBF-SEM-Übersichtsbilder mit einer Pixelgröße von 10 nm, 8.000 x 8.000 Pixeln bei 1 s Verweilzeit aufgenommen. Das Mikroskop wurde noch auf 1,6 kV und 100 pA eingestellt. - Entfernen Sie die Probe aus dem SBF-SEM und legen Sie sie in den Sputterlacker. Beschichten Sie die Probe mit 20 nm Platin für die FIB-SEM-Bildgebung.
- Belastungsprobe in FIB-SEM und mit Sekundärelektronendetektor bei 15 kV, 1 nA lokalisieren den ROI im SBF-SEM auf der Blockfläche identifiziert (Abbildung 2B,D).
HINWEIS: Die Bildgebung bei 15 kV ist notwendig, um die Platinbeschichtung zu durchschauen. - Bringen Sie den ROI auf der Probe in den Koinzidenzpunkt der FIB- und SEM-Strahlen, indem Sie die Bühne bewegen und kippen (Abbildung 3A).
HINWEIS: Die FIB-Säule ist in der Regel unter einem Winkel montiert (Abbildung 3A). Das bedeutet, dass jede Probe so geneigt werden muss, dass die zu bebilderte und zu steilende Fläche parallel zum FIB-Strahl positioniert wird. Die zu bebilderte Oberfläche ist nun in Bezug auf den SEM-Strahl geneigt und ein Gewebegraben muss entfernt werden, bevor das SEM den ROI abbilden kann (Abbildung3E) - Mit dem FIB-Strahl- und Gaseinspritzsystem eine 1 mm-Schutzschicht aus Platin auf der Oberfläche über dem ROI ablegen (Abbildung 3C). Als nächstes, mit einem niedrigen Frässtrom (50-100 pA), fräsen feine Linien in die Platin-Abscheidung für Auto-Fokussierung und 3D-Tracking während der Bildgebung lauf (Abbildung 3D). Mit der FIB-Säule und dem Kohlenstoffgasinjektor diese Leitungen mit Kohlenstoffabscheidung abdecken.
HINWEIS: Die Größe des ROI entspricht hier der Größe des ROI auf dem SBF-SEM-Bild und die maximale Größe kann somit 30-40 x 15-20 m betragen. In diesem Beispiel wurde ein ROI von 17 x 8 m ababgebildet. Zum Schutz der Linien und zur Schaffung eines schwarz-weißen Kontrasts zwischen Kohlenstoff und Platin, der sich ideal für die Automatische Fokussierung eignet, ist eine Kohlenstoffablagerung erforderlich. Die fräsenden Ströme, die für jeden Schritt verwendet werden, finden Sie in Tabelle 2. - Mit einem hohen Frässtrom fräsen Sie einen Graben von 30 m vor dem ROI und schaffen so die Bildfläche für den SEM-Strahl (Abbildung 3E).
HINWEIS: Der FIB-Strahl ist von Natur aus destruktiv, noch mehr bei hohen Strömen. Achten Sie darauf, die Bildgebung bei hohen Strömen auf ein Minimum zu beschränken und bei niedriger Vergrößerung und schnellen Scangeschwindigkeiten ein Bild zu erhalten, um ein Schmelzen des Harzes am ROI zu vermeiden. - Glätten Sie die Bildfläche mit einem Frässtrom, der näher an dem Strom liegt, der während des Bildlaufs verwendet wird. Beenden Sie das Polieren, wenn alle Autofokus- und 3D-Tracking-Markierungen auf der Bildfläche deutlich sichtbar sind (Abbildung 3F). Dem Fortschritt der FIB kann gefolgt werden, indem die Oberfläche mit der EM (mit einem zurückgestreuter Elektronendetektor) beim Polieren abgestellt wird.
- Bestimmen Sie den Bereich, der auf der neu erstellten Fläche abgebildet werden soll, und legen Sie die Bildparameter fest. Stellen Sie sicher, dass der Elektronenstrahl auf die Oberfläche fokussiert ist, legen Sie Helligkeit und Kontrast fest und legen Sie die Pixelgröße und schnittdicke fest. Halten Sie die Bildzeit unter 1 Minute, indem Sie die Verweilzeit und den Liniendurchschnitt anpassen.
HINWEIS: Es ist wichtig, eine Niedrige Spannung für die Bildgebung mit dem Elektronenstrahl zu verwenden, um sicherzustellen, dass nur die Oberfläche des Blocks abgebildet wird (d. h., dass keine Elektronen aus tiefer in die Probe abgebildet werden). Dies geschieht, indem die Spannung unter 2 kV gehalten wird und ein rückseitiges, gestreutes Elektronendetektor mit einer Netzspannung verwendet wird, so dass nur hochenergetische Elektronen abgebildet werden können. In diesem Beispiel wurde der Elektronenstrahl auf 1,5 kV und 1 nA mit einer Netzspannung von 1,2 kV auf dem hinteren gestreuten Elektronendetektor eingestellt. Auch hier wurde eine Pixelgröße von 5 nm mit 5nm Abschnitten verwendet, um einen Datensatz mit isotropen Voxeln zu ergeben. Bei einer Verweilzeit von 6,5 s und einem Liniendurchschnitt von 1,0 wurde eine Fläche von 17 x 10 m abgebildet. - Legen Sie die Fenster für die automatische Optimierung und 3D-Verfolgung fest, indem Sie die gleiche Pixelgröße, den gleichen Verweilzeit- und Liniendurchschnitt verwenden, wie sie für die Bildgebung verwendet werden.
HINWEIS: Dieser Prozess unterscheidet sich für verschiedene Systeme, daher wird nur der Schritt erwähnt, ohne die verschiedenen Aktionen anzugeben. - Starten Sie den Bildvorgang und überwachen Sie die Stabilität des Prozesses während der ersten 50 bis 100 Abschnitte. Sobald das System reibungslos läuft, verlassen Sie den Raum und stellen Sie sicher, dass es so wenig Störungen wie möglich im Raum gibt.
HINWEIS: Die Dauer des Durchlaufs und die Anzahl der Abschnitte hängen von der Größe des ROI und der Schnittdicke ab. In FIB-SEM ist die Z-Achse eigentlich die Höhe der ROI-Box auf dem SBF-SEM-Bild (max. 15 x 20 m; Abbildung 3E). - Registrieren Sie die FIB-SEM-Daten auf die gleiche Weise wie oben beschrieben für die SBF-SEM-Daten.
Ergebnisse
Bilder aus dem SBF-SEM geben einen Überblick über das Gewebe und geben Einblicke in die räumliche Ausrichtung von Zellen und interzellulären Verbindungen (Abbildung 4A). Die nachfolgende FIB-SEM-Bildgebung in einer neuen Region, die in der Regel eine nach der Inspektion des SBF-SEM-Laufs ermittelte Region ist, fügt hochauflösende Details bestimmter Zellen und/oder Strukturen hinzu (Abbildung 4B).
Abbildung 4C ,D zeigen den Unterschied in der Wiedergabe der nicht-isotropen Voxel der SBF-SEM-Daten (Abbildung 4C) und der isotropen VOXel FIB-SEM-Daten (Abbildung 4D). Die in SBF-SEM verwendete z-Dicke bedeutet, dass das Rendering die Abschnitte deutlich anzeigt, was zu einem "Treppeneffekt" auf der Oberfläche führt. In den FIB-SEM-Daten sorgen die 5 nm-Abschnitte dafür, dass das Rendering deutlich glatter erscheint und einzelne Abschnitte vollständig in die Oberfläche einfließen.
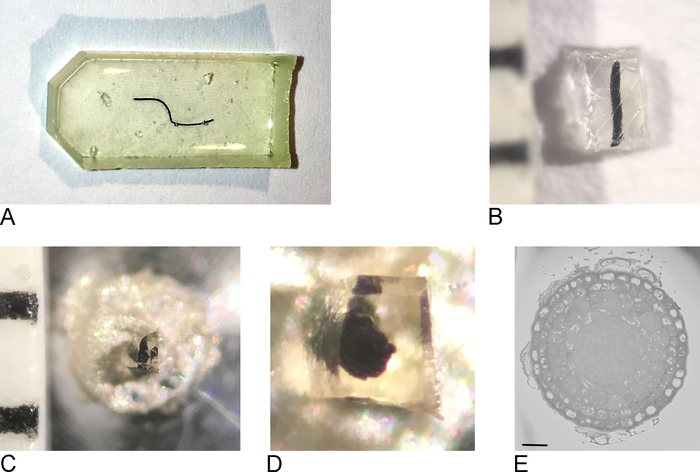
Abbildung 1: Erstellung der Blockfläche aus einer eingebetteten Harzprobe. (A) Eine in Harz eingebettete Wurzelspitze. (B) Mit einer Rasierklinge wird das überschüssige Harz weggetrimmt, bis ein Block von 0,5 mm2 übrig bleibt. (C,D) Der getrimmte Block wird auf einen Metallstift geklebt und nach einer Nacht im Ofen werden die Seiten des Blocks getrimmt und die Oberfläche mit einem Diamantmesser mit einem Ultramikrotom geglättet. (E) Innerhalb des SBF-SEM ist das Sample so ausgerichtet, dass Blockface und ROI erkannt werden können, Skalenstange = 20 m. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
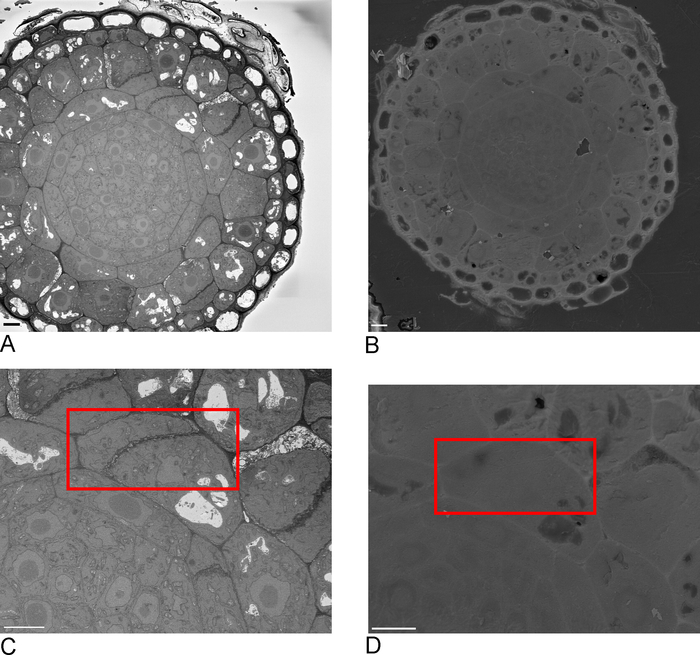
Abbildung 2: Korrelation zwischen SBF-SEM und FIB-SEM. Übersichtsbilder der Blockfläche mit dem SBF-SEM (A) und dem FIB-SEM (B), Skalenbalken = 5 m (C,D) Zoom auf den ROI. Rotes Kästchen umgibt die Region, die mit FIB-SEM, Skalenleiste = 5 m abgebildet werden soll. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
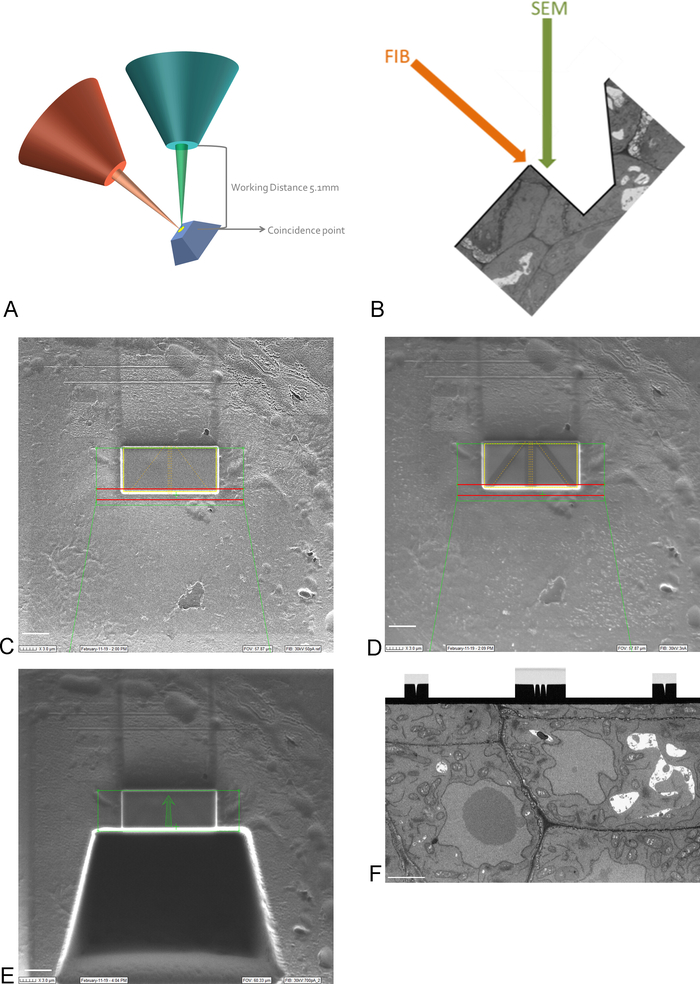
Abbildung 3: FIB-SEM-Schema und Vorbereitungsschritte. (A) Schema mit der Ausrichtung des FIB-Strahls, des SEM-Strahls und der Probe. Die Probe muss bis zum Koinzidenzpunkt der FIB- und SEM-Strahlen positioniert werden, um auf derselben Region fräsen und abbilden zu können. (B) Schematische Zeichnung des Grabens, der für die SEM-Bildgebung der von der FIB entfernten Abschnitte benötigt wird. (C) Bild mit dem FIB-Strahl aufgenommen, der Platinabscheidung auf dem ROI zeigt, Skalenbalken 5 m. (D) Bild mit dem FIB-Strahl aufgenommen, das die Für den Autofokus und die 3D-Verfolgung verwendete Linien zeigt. Die Linien in der Mitte werden für die automatische Fokussierung verwendet und die äußeren Linien bieten 3D-Tracking. Die Kohlenstoffablagerung auf den Linien liefert den notwendigen Kontrast (Platin vs Kohlenstoff), um diese Aufgaben auszuführen, Scalebar 5 m. (E) Bild, das mit dem FIB-Strahl nach dem Fräsen des Grabens aufgenommen wurde, Skalenstange 5 m. (F) Bild mit dem SEM-Strahl, der die Bereich von Interesse, der während des FIB-SEM-Laufs abgebildet wurde, Skalenbalken 2 m. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
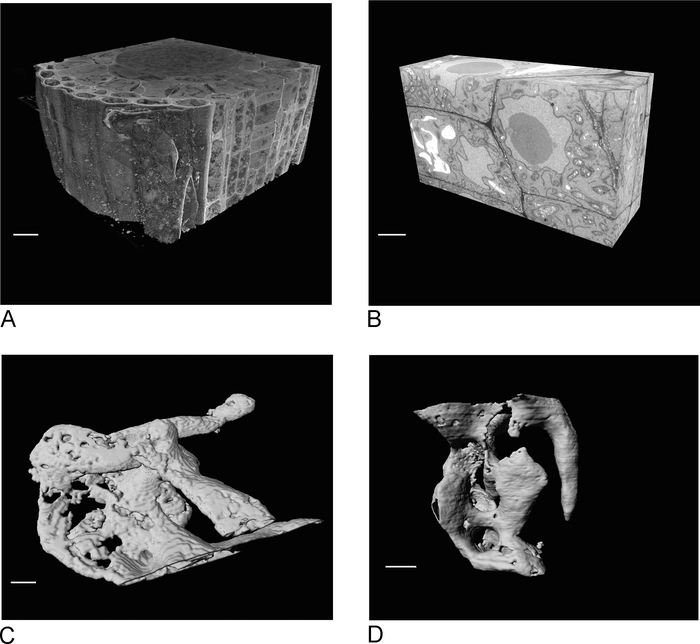
Abbildung 4: SBF-SEM- und FIB-SEM-Ergebnisse vor und nach der Segmentierung. (A) 3D-Ansicht des SBF-SEM-Datensatzes (100 x 100 x 40 m, Maßstabsbalken = 10 m), (B) 3D-Ansicht des FIB-SEM-Datasets (17 x 10 x 5,4 m, Maßstabsbalken = 2 m), (C) Gerenderte Vakuolen, segmentiert aus SBF-SEM-Daten durch Schwellenwertung, Skalenbalken = 2 m D. Gerenderte FIB-SEM-Daten durch Schwellenwerte, Skalenbalken = 2 m. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Protokoll für Mikrowellenverarbeitung | |||||||||
| programmieren # | beschreibung | Benutzeraufforderung (ein/aus) | Zeit (st:min:sec) | Leistung (Watt) | Temperatur (°C) | Ladekühler (aus/auto/on) | Vakuum-/Bubbler-Pumpe (aus/bubb/vac-Zyklus/vakuum ein/vap) | Steady Temp | |
| Pumpe (ein/aus) | Temperatur (°C) | ||||||||
| 8 | Tch | weg | 0:01:00 | 150 | 50 | weg | VAKUUM-Zyklus | auf | 30 |
| weg | 0:01:00 | 0 | 50 | weg | VAKUUM-Zyklus | auf | 30 | ||
| weg | 0:01:00 | 150 | 50 | weg | VAKUUM-Zyklus | auf | 30 | ||
| 9 | Osmium | weg | 0:02:00 | 100 | 50 | weg | VAKUUM-Zyklus | auf | 30 |
| weg | 0:02:00 | 0 | 50 | weg | VAKUUM-Zyklus | auf | 30 | ||
| weg | 0:02:00 | 100 | 50 | weg | VAKUUM-Zyklus | auf | 30 | ||
| weg | 0:02:00 | 0 | 50 | weg | VAKUUM-Zyklus | auf | 30 | ||
| weg | 0:02:00 | 100 | 50 | weg | VAKUUM-Zyklus | auf | 30 | ||
| 10 | 50% EtOH | auf | 0:00:40 | 150 | 50 | weg | weg | auf | 30 |
| 70% EtOH | auf | 0:00:40 | 150 | 50 | weg | weg | auf | 30 | |
| 90% EtOH | auf | 0:00:40 | 150 | 50 | weg | weg | auf | 30 | |
| 100% EtOH | auf | 0:00:40 | 150 | 50 | weg | weg | auf | 30 | |
| 100% EtOH | auf | 0:00:40 | 150 | 50 | weg | weg | auf | 30 | |
| 15 | 0.1M CACODYLATE | auf | 0:00:40 | 250 | 50 | weg | VAKUUM-Zyklus | auf | 30 |
| 0.1M CACODYLATE | auf | 0:00:40 | 250 | 50 | weg | VAKUUM-Zyklus | auf | 30 | |
| 15 | ddH2O | auf | 0:00:40 | 250 | 50 | weg | VAKUUM-Zyklus | auf | 30 |
| ddH2O | auf | 0:00:40 | 250 | 50 | weg | VAKUUM-Zyklus | auf | 30 | |
| 16 | Uranylacetat | weg | 0:01:00 | 150 | 50 | weg | VAKUUM-Zyklus | auf | 30 |
| weg | 0:01:00 | 0 | 50 | weg | VAKUUM-Zyklus | auf | 30 | ||
| weg | 0:01:00 | 150 | 50 | weg | VAKUUM-Zyklus | auf | 30 | ||
| weg | 0:01:00 | 0 | 50 | weg | VAKUUM-Zyklus | auf | 30 | ||
| weg | 0:01:00 | 150 | 50 | weg | VAKUUM-Zyklus | auf | 30 | ||
| weg | 0:01:00 | 0 | 50 | weg | VAKUUM-Zyklus | auf | 30 | ||
| weg | 0:01:00 | 150 | 50 | weg | VAKUUM-Zyklus | auf | 30 | ||
Tabelle 1. Detailliertes Protokoll für die Mikrowellenverarbeitung.
| schritt | gegenwärtig | Geschätzte Zeit |
| Deposition Platinum | 3n A | 10-15 Minuten |
| Fräsen von Autotune- und Tracking-Marken | 50-100 pA | 4-6 Minuten |
| Deposition Carbon | 3 nA | 5-10 Minuten |
| Fräsen grober Graben | 15-30 nA | 30-50 Minuten |
| Polierfläche | 1.5-3 nA | 15-20 Minuten |
| Imaging Run | 700 pA-1,5 nA | Stunden-Tage |
Tabelle 2. FIB-Fräsströme für die Probenvorbereitung und Bildgebung
Diskussion
Volumenelektronenmikroskopie ist anspruchsvoller und zeitaufwändiger als herkömmlichesemoder SEM oder TEM. Da Gewebe oder Zellen en bloc gefärbt werden müssen, müssen die Verarbeitungsschritte lang genug sein, um das Eindringen von Reagenzien in der gesamten Probe zu gewährleisten. Die Verwendung von Mikrowellenenergie zur Erleichterung der Penetration ermöglicht eine kürzere, effizientere Verarbeitung und verbessert die Färbung. Da die EM-Vorbereitung viel strenger ist als für die Leichte Mikroskopie, müssen alle Lösungen und Reagenzien streng qualitätskontrolliert werden. Veränderungen des pH-Werts, der Tonizität, der Verwendung von unreinen Reagenzien und der Einführung von Verunreinigungen aufgrund schlechter Technik können alle schädliche Auswirkungen auf das endgültige Bild haben.
Volume EM erfordert auch individuell zugeschnittene Protokolle für jeden einzelnen Probentyp. Säugetiergewebe unterschiedlicher Art: Pflanzen, Einzelzellen wie Hefe, Trypanosomen, C. elegansusw., alle benötigen ihre eigenen Variationen, um optimale Ergebnisse zu erzielen. Die Fixierung und Färbung muss so gestaltet sein, dass die strukturelle Integrität erhalten bleibt und die Probe so nah wie möglich an ihrer In-vivo-Morphologie bleibt. Die Fixierung von Geweben bei physiologischer Temperatur, pH-Wert und Tonizität ist entscheidend, um die Probe so lebensecht wie möglich zu machen. Hochdruck-Gefrieren (HPF) von Proben kann helfen, eine lebensähnlichere Situation zu erhalten (oder vielleicht nur andere Artefakte zu liefern), aber für andere als Zellen und sehr dünne Gewebe wird HPF scheitern, da Glaseis nur in kleinen Mengen erzeugt werden kann. Daher ist für viele Fragen chemische Fixierung die einzige Option. Unabhängig davon, ob es sich bei der Fixierung um HPF oder chemisch handelt, müssen die strukturellen Ergebnisse bei jedem EM-Experiment sorgfältig mit ähnlichen Ergebnissen aus der Bildgebung von lebenden Zellen oder Geweben verglichen werden, um zu sehen, ob sie konsistent sind. Die Färbung muss auch unter Berücksichtigung der spezifischen Frage optimiert werden, die beantwortet werden muss, und des Protokolls, das für die Visualisierung der digitalen Bilder verwendet wird.
Sowohl ein SBF-SEM- als auch ein FIB-System in unmittelbarer Nähe zu haben, ist in vielen Experimenten ein großer Vorteil. Das große Sichtfeld und die hohe X,Y-Auflösung von SBF-SEM machen das Finden einzelner Strukturen/Zellen/Ereignisse einfach und bieten eine gesamträumliche Orientierung der Zellen in Geweben. Darüber hinaus ist seine Fähigkeit, die Bildgebung durch eine Probe in Z zu ermöglichen, sehr leistungsstark; Rekonstruktionen, die feine geometrische Details erfordern, können jedoch aufgrund der nicht-isotropen Voxel, die sie erzeugt, mit dieser Technik fehlschlagen oder Artefakte erzeugen. Die FIB ist durch die Physik des Prozesses auf ein kleineres Bildgebungsfeld beschränkt, aber ihre 3D-Auflösung ist ausreichend für sehr genaue Rekonstruktionen. Die Kombination der beiden Techniken ist einfach, da Proben ohne weitere Verarbeitung oder Vorbereitung von SBF-SEM auf FIB umgestellt werden können. Wir erkennen an, dass die Verwendung des SBF-SEM für die Suche nach einem Bestimmten Bereich eine sehr teure Verwendung eines viel leistungsfähigeren Tools ist. Die Möglichkeit, die neue Blockfläche sofort zu sehen und festzustellen, ob der ROI erreicht wurde, ist jedoch ein großer Vorteil. Darüber hinaus können die Alternativen zur Verwendung von seriellen halbdünnen (0,5 m) LM-Abschnitten kleine Strukturen entfernen, bevor sie erkannt werden, und die Inspektion eines Blocks mit einzelnen TEM-Abschnitten, die geschnitten, auf ein Gitter gelegt und dann in einem ebenso teuren TEM betrachtet werden müssen, ist nicht effizient wie die vorgestellte Methode.
Da viele Programme zum Segmentieren und Rendern der Daten vorhanden sind und die Anforderungen einer bestimmten Struktur möglicherweise nicht am besten von einer einzelnen Anwendung bedient werden können, kann kein Standardworkflow vorgeschlagen werden. Einige einfache Strukturen können mit einem Schwellenwertalgorithmus segmentiert werden, wenn sie innerhalb sehr schmaler Graustufenwerte liegen. Neuronale Strukturen können mit einem Programm wie Ilastik11 halbautomatisch segmentiert werden, aber es wird weniger nützlich für zufälligere oder komplexer geformte Organellen wie ER sein. Microscopy Image Browser ist ein sehr flexibles Programm, das EM-Daten ausrichten, segmentieren und rendern kann, aber eine erhebliche Benutzerinteraktion erfordert12. In der Regel wird die Zeit, die für die digitale Visualisierung der Ergebnisse benötigt wird, die Zeit für die Vorbereitung der Probe und die Bildgebung erheblich überschreiten.
Volumen-EM-Techniken haben die dritte Dimension für die ultrastrukturelle Analyse geöffnet. Andere Methoden zur Erlangung von 3D EM haben Einschränkungen in ihrem Volumen (TEM-Tomographie) oder ihrer Effizienz (SerieLLtem). Obwohl EM-Techniken zum größten Teil zu komplex und kostspielig sind, um in einzelnen Laboratorien eingesetzt zu werden, ist die Zahl der gemeinsamen Kerneinrichtungen, die sie anbieten, gestiegen und die Anzahl der erfolgreich abgebildeten Probentypen hat rapide zugenommen. Für diejenigen mit einer bestimmten Frage und einem bestimmten Gewebe ist es wahrscheinlich, dass jemand in der Lage sein wird, Ratschläge und Anweisungen über seine Vorbereitung und Bildgebung anzubieten. Volumen-EM-Geräte können verbessert werden, um die Kapazität zur Handhabung größerer Proben im SBF-SEM und die Fähigkeit, größere ROIs mit der FIB zu fräsen, einzubeziehen. Software, die in der Lage ist, Strukturen von Interesse auf eine automatisiertere Weise zu segmentieren, wird den Prozess der Analyse der Daten erheblich vereinfachen und Verbesserungen in der Rechengeschwindigkeit werden die dafür benötigte Zeit reduzieren. Trotz seiner aktuellen Einschränkungen ist Volume EM immer noch ein nützliches Werkzeug und die Kombination von SBF-SEM und FIB-SEM bietet einen effizienten Workflow, um seltene Ereignisse zu identifizieren und sie mit hoher Auflösung zu bebildern.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Die Ausrüstung für Volumen EM wurde durch einen großzügigen Zuschuss der Regierung von Flandern zur Verfügung gestellt.
Materialien
| Name | Company | Catalog Number | Comments |
| 3View 2XP | Gatan | NA | In chamber ultramicrotome for SBFI |
| Cacodylate buffer 0.2M solution | EM Sciences | 11652 | |
| Conductive epoxy resin (circuit works) | RS components | 496-265 | |
| Diatome Histo 4.0mm diamond knife | EM Sciences | 40-HIS | |
| Digitizing tablet | Wacom | DTV-1200W | No longer available |
| Eppendorf tubes | Eppendorf | 0030 120.094 | |
| Flat Embedding Mold | EM Sciences | 70900 | |
| Gluteraldehyde 25% solution | EM Sciences | 16220 | |
| High MW Weight Tannic Acid | EM Sciences | 21700 | |
| Lead Citrate | Sigma-Aldrich | 22861 | |
| NaCl | Sigma-Aldrich | 746398 | |
| Osmium Tetroxide 4% solution | EM Sciences | 19170 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | |
| Pelco Biowave Pro + | Ted Pella | 36700 | |
| Potassium Ferrocyanide | Sigma-Aldrich | P3289 | |
| Quorum Q150T ES sputter coater | Quorum Technologies | 27645 | |
| Reichert-Jung Ultracut ultramicrotome | NA | NA | No longer available |
| Sodium Cacodylate 0.2M | EM Sciences | 11653 | |
| Spurrs Resin kit | EM Sciences | 14300 | |
| Uranyl Acetate | EM Sciences | 22400 |
Referenzen
- Linberg, K. A., Fisher, S. K. An ultrastructural study of interplexiform cell synapses in the human retina. Journal of Comparative Neurology. 243, 561-576 (1986).
- Peddie, C. J., Collinson, L. M. Exploring the third dimension: volume electron microscopy comes of age. Micron. 61, 9-19 (2014).
- Leighton, S. SEM images of block faces, cut by a miniature microtome within the SEM – A technical note. Scanning Electron Microscopy. (pt 2), 71-76 (1981).
- Denk, W., Horstmann, H. Serial block-face scanning electron microscopy to reconstruct three dimensional tissue nanostructure. PLoS Biology. 11, 329 (2004).
- Heymann, J. A., Hayles, M., Gestmann, I., Giannuzzi, L. A., Lich, B., Subramaniam, S. Site specific 3D imaging of cells and tissues with a dual beam microscope. Journal of Structural Biology. 155 (1), 63-73 (2006).
- . NCMIR Methods for 3D EM: A new protocol for preparation of biological specimens for serial block face scanning electron microscopy. V7_01_10 Available from: https://ncmir.ucsd.edu/sbem-protocol (2019)
- Giberson, R. T., Austin, R. L., Charlesworth, J., Adamson, G., Herrera, G. A. Microwave and digital imaging technology reduce turnaround times for diagnostic electron microscopy. Ultrastructural Pathology. 27 (3), 187-196 (2003).
- Kremer, A., et al. Developing 3D EM in a broad biological context. Journal of Microscopy. 259 (2), 80-96 (2015).
- Vanslembrouck, B., Kremer, A., Pavie, B., van Roy, F., Lippens, S., van Hengel, J. Three-dimensional reconstruction of the intercalated disc including the intercellular junctions by applying volume scanning electron microscopy. Journal of Histochemistry and Cell Biology. 149, 479-490 (2018).
- Russel, M. R., et al. 3D correlative light and electron microscopy of cultured cells using serial blockface scanning electron microscopy. Journal of Cell Science. 130 (1), 278-291 (2017).
- Sommer, C., Strähle, C., Köthe, U., Hamprecht, F. A. ilastik: Interactive Learning and Segmentation Toolkit. Eighth IEEE International Symposium on Biomedical Imaging (ISBI). Proceedings. , 230-233 (2011).
- Belevich, I., Joensuu, M., Kumar, D., Vihinen, H., Jokitalo, E. Microscopy Image Browser: A Platform for Segmentation and Analysis of Multidimensional Datasets. PLoS Biology. 14 (1), e1002340 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten