Method Article
Einschaltstige Bereicherung einer TAP-Tagged-Histon-Deacetylase des Filamentous Fungus Aspergillus nidulans für Enzymatische Aktivität Assay
In diesem Artikel
Zusammenfassung
Die Histondeaketylasen der Klasse 1 (HDACs) wie RpdA haben als potenzielle Zielscheibe zur Behandlung von Pilzinfektionen an Bedeutung gewonnen. Hier stellen wir ein Protokoll zur spezifischen Anreicherung von TAP-getagenem RpdA in Kombination mit einem HDAC-Aktivitätstest vor, der in vitro Wirksamkeitsprüfung von Histon-Deacetylase-Inhibitoren ermöglicht.
Zusammenfassung
Die Histondeacetylasen der Klasse 1 (HDACs) wie RpdA haben als potenzielle Ziele für die Behandlung von Pilzinfektionen und für den Genomabbau von Pilzsekundenkataboliten an Bedeutung gewonnen. Das Inhibitor-Screening erfordert jedoch gereinigte Enzymaktivitäten. Da Diaketylasen der Klasse 1 ihre Funktion als Multiprotein-Komplexe ausüben, sind sie in der Regel nicht aktiv, wenn sie als einzelne Polypeptide in Bakterien ausgedrückt werden. Daher müssen endogene Komplexe isoliert werden, die bei der Anwendung konventioneller Techniken wie Ionenaustausch und Größenausschlusschromatographie aufwändig und zeitaufwendig sind. Die Tandem-Affinitätsreinigung wurde als Werkzeug entwickelt, um Multiproteinkomplexe aus Zellen zu veranzeln und erwies sich damit als ideal für die Isolierung endogener Enzyme. Hier stellen wir Ihnen ein detailliertes Protokoll zur einstufigen Anreicherung von aktiven RpdA Komplexen über den ersten Reinigungsschritt von C-Tagg-RpdA von Aspergillus nidulanszur Verfügung. Die gereinigten Komplexe können dann für das anschließende Inhibitor-Screening mit einem Deacetylase-Test verwendet werden. Die Proteinanreicherung zusammen mit dem enzymatischen Aktivitätsuntergang kann innerhalb von zwei Tagen abgeschlossen werden.
Einleitung
Histone Deacetylases (HDACs) sind Zn2 +-abhängige hydrolytische Enzyme, die in der Lage sind, Acetylgruppen aus Lysin-Rückständen von Histonen und anderen Proteinen zu entfernen. Basierend auf der Sequenzähnlichkeit werden HDACs in mehrere Klassen1gruppiert. Vor kurzem hat sich gezeigt, dass die Pilzklasse 1 HDAC RpdA, ein Ortholog der Bäckerhefe(Saccharomyces cerevisiae) Rpd3p, für den opportunistischen Pilzpathogen Aspergillus fumigatus2 unverzichtbar ist. Deshalb hat RpdA als potenzielles Ziel für die Behandlung von Pilzinfektionen an Bedeutung gewonnen. Die Beurteilung der Deacetylase-Aktivität in vitro ist wichtig für die Charakterisierung von enzymatischen Eigenschaften und ermöglicht es, die Wirksamkeit neuartiger Substanzen für die Entwicklung von Inhibitoren zu bestimmen. Obwohl rekombinante Ausdruck einer Codon-optimierten Version des menschlichen HDAC1 in Escherichia coli wurde vorkurzem 3berichtet, scheiterten Versuche, in voller Länge RpdA in diesem Host auszudrücken 4. Da die Pilzklasse 1 HDACs wie RpdA und HosA ihre Funktion als Multiprotein-Komplexe ausüben, ist es außerdem sinnvoll, einheimische endogene Komplexe für enzymatische Inhibitorstudien zu verwenden. Aufgrund hemmender Faktoren und des Vorhandenseins verschiedener HDACs in Pilzlysaten ist die in ganzen Proteinextrakten gemessene katalytische Aktivität jedoch relativ gering und kann nicht einzelnen Enzymen zugeordnet werden. Darüber hinaus identifizierten frühere Studien an den filamentösen Pilzen A . nidulans eine Klasse 2 HDAC, HdaA, als vorherrschende Deacetylase in chromatographischen Bruchteilen von Pilzextrakten. Daher sind mehrere konventionelle chromatographische Reinigungsschritte erforderlich, um nicht-HdaA-Aktivität von Pilzstämmen zu trennen4. Die Einführung der Tandemaffeinigung (TAP) in A. nidulans5 hat die Bereicherung spezifischer Deacetylase-Aktivitäten erheblich erleichtert. Das ursprüngliche TAP-Tag besteht aus zwei Proteina-A-Domains und einem Calmodulin-bindenden Peptid (CBP), das durch eine Tabulattech-Virus-Protease (TEV) Spaltstelle 6 getrennt ist. Dies ermöglicht die närrische Reinigung und Eliution von markierten Proteinen einschließlich ihrer Interaktionspartner7. Bei der Verwendung der angereicherten Proteine für Aktivitätsuntersuchungen ist die milde Eule unter den einheimischen Bedingungen durch Protease-Spaltung ein wichtiges Merkmal der TAP-Tag-Reinigung. Ein GFP-markiertes Protein zum Beispiel kann auch durch immobilisierte Antikörper angereichert werden, kann aber unter einheimischen Bedingungen nicht ausgelotet werden.
Hier stellen wir ein detailliertes Protokoll für die einstufige Anreicherung von aktiven RpdA-Komplexen über den ersten Reinigungsschritt von C-Tagg-RpdA von A. nidulans (IgG-Sparte und TEV-Sparte) für das anschließende Inhibitor-Screening zur Verfügung, das eine Histon-Deacetylase Assay. Die Affinitätsbereicherung war, wie sich herausstellte, auf nur einen Reinigungsschritt beschränkt, auch weil die enzymatische Aktivität nach einer zweistufigen TAP-Reinigung im Vergleich zur IgG-Reinigung allein signifikant reduziert wurde.
Dennoch sollte das eingeführte Protokoll auch für die Anreicherung anderer markierter Enzyme gelten, die an Chromatin-Regulierung beteiligt sind, wie Acetyltransferasen, Methyltransferasen und Thrithylasen. Durch das Anhängen des zweiten Reinigungsschrittes des TAP-Protokolls können Proteine, die mit den markierten Ködern gleichgesetzt werden, als komplexe Partner von (neuartigen) enzymatischen Komplexen betrachtet werden.
Protokoll
1. Anbau von A. nidulans
- Inoculum-Vorbereitung
Achtung: Alle Schritte außer der Inkubation sollten unter einem laminaren Fließschrank erfolgen.- Die TAP-Belastung (z.B. TIB32.12) aus dem Glycerinbestand (-80 ° C) auf Glucose/Xylose minimales Medium (GXMM; Pro Liter: 10,0 g Glukose, 0,5 g Xylose, 10 ml 1 M di-ammonium-Tartrat-Lösung, 20 ml von 50 × Salzlösung [pro Liter: 26.0 g KCl, 26.0 g MgSO4 · 7 H2O, 76.0 g KH2PO4, 1 mL Chloroform], 1 mL von 1.000 × Hutner es Spurenelementen8) inklusive 1,5% (w/v) agar und den benötigten Ergänzungen, wie von Todd et al. 20079beschrieben. 2 – 3 Tage bei 37 ° C in die Inkubate.
NOTE: TIB32.1 enthält eine rpdA:: TAP Fusion, die von einem xylose-induzierbaren Promoter, XylP(p) 10, gesteuert wird. Aus diesem Grund ist xylose in dem obigen Rezept enthalten. Omit xylose, wenn die Stämme wachsen und Konstrukte ausdrücken, die nicht unter XylP(p)-Kontrolle stehen. - Bereiten Sie ein steriles 1,5 mL Zentrifugenrohr mit 1,5 mL steriler Nadelaufhängungslösung vor (CSS; 0,9% (w/v) NaCl, 0,01% (v/v) Polysorbate 80).
- Eine Einweg-Impfung in CSS tragen, Konidien von der Platte aus Schritt 1.1.1 abkratzen und in der Röhre von Schritt 1.1.2 aussetzen.
- Übertragen Sie 150 μL der Nadelaufhängung auf zehn 25 cm 2 Zellkulturflaschen mit Entlüftungskappe, die GXMM agar inklusive Ergänzungen und 200 μL CSS enthält.
- Die Nadelaufhängung in den Flaschen mit einer sterilen Impfschleife verteilen und die Flaschen 2 – 3 Tage bei 37 ° C inkubieren.
- Um die Konidien zu ernten, verwenden Sie 10 mL CSS pro Flasche. Die Lösung in eine Flasche gießen, die Flasche mit dem mitgelieferten Schraubverschluss fest verschließen und die Flasche kräftig schütteln.
- Nach dem Benetzen der Pilzfläche, komplett abkratzen Restkonidien mit einer sterilen Impfschleife.
- Geben Sie Kidien durch 40 μm Zellstrainer, die auf ein steriles 50 mL-Zentrifugenrohr gelegt werden, und sammeln Sie die Aufhängung von fünf Flaschen in einem Rohr.
- Zentrifuge die Röhre mit 1.000 × g für 10 min.
- Decant das Supernatant und rettet das Pellet in 10 mL CSS pro Rohr.
- Sammeln Sie beide Aufhängungen in einem Rohr, spülen Sie das leere Rohr mit 40 mL CSS, fügen Sie die Aufhängung, und Zentrifuge, wie in Schritt 1.1.9 beschrieben.
- Decant das Supernatant und rettet das Nadelstichchen in 4 mL CSS.
- Bereiten Sie zwei serielle 1:50 Verdünnungen der Nadelaufhängung vor und bestimmen Sie die Anzahl der Konidien in der resultierenden 1:2, 500-verdünnten Aufhängung mit einer Zählkammer wie beschrieben 11.
- Die TAP-Belastung (z.B. TIB32.12) aus dem Glycerinbestand (-80 ° C) auf Glucose/Xylose minimales Medium (GXMM; Pro Liter: 10,0 g Glukose, 0,5 g Xylose, 10 ml 1 M di-ammonium-Tartrat-Lösung, 20 ml von 50 × Salzlösung [pro Liter: 26.0 g KCl, 26.0 g MgSO4 · 7 H2O, 76.0 g KH2PO4, 1 mL Chloroform], 1 mL von 1.000 × Hutner es Spurenelementen8) inklusive 1,5% (w/v) agar und den benötigten Ergänzungen, wie von Todd et al. 20079beschrieben. 2 – 3 Tage bei 37 ° C in die Inkubate.
- Wachstum und Ernte von Myzelien
- Bei der Arbeit unter einem laminaren Fließschrank, impfen 4 – 6 Ein-Liter-kegelförmige Flaschen mit jeweils 250 ml GXMM einschließlich entsprechender Ergänzungen in einer Dichte von 5 × 10 conidia/mL und Inkubat bei 180 U/min bei 37 ° C für 14 – 16 h.
- Käsetuch in einen Trichter auf eine Flasche legen und die Myzelie durch das Tuch filtern. Kurz mit deionisiertem Wasser waschen.
- Entfernen Sie so viel Feuchtigkeit wie möglich, indem Sie die Myzelie, die in der Käseplatte gefangen ist, zwischen den Händen und dann Papiertüchern drücken.
- Die getrocknete Myzelie als flache Bleche mit einem Schraubdeckel auf einen Kunststoffbecher übertragen und mit flüssigem Stickstoff einfrieren. Damit ist ein hohes Flächenvolumen für den folgenden Lyophililierungsprozess gewährleistet.
- Bewahren Sie die gefrorene Myzelie bei-80 ° C vor der Lyophilisierung auf.
Hinweis: Das Protokoll kann hier angehalten werden. - Lyophilize die Myzelie über Nacht.
- Stoppen Sie den Gefriertrocknungsprozess, wenn die Temperatur der Myzelie konstant bleibt (18 – 24 h). Die Becher entfernen und sofort mit den mitgelieferten Schraubverschlüssen verschließen.
Hinweis: Bei dicht verschlossenen Myzelien kann bei RT mehrere Wochen lang lophilisierte Myzelien gelagert werden.
2. Einstufige Anreicherung von TAP-getaggtem HDAC (angepasst von Bayram et al . 2012)12
- Vorbereitung von Puffern und Lösungen
Hinweis: 2 Mercaptoethanol (EtSH) und Protease-Inhibitoren direkt vor der Anwendung zu Puffern hinzufügen. Filtern Sie alle Puffer, die für die Chromatographie verwendet werden, durch 0,22 μm Nitrocellulose-Membranen, um die Einführung von Verunreinigungen/Verunreinigungen in das Chromatographie-Harz zu vermeiden. Die Anweisungen in den folgenden Schritten beziehen sich auf die Erstellung von 1 L pro Puffer. Puffer bei 4 ° C aufbewahren.- Extraktionspuffer (B250): 250 mM NaCl, 100 mM Tris-HCl pH 7.5 (RT), 0,1% (v/v) TX-100, 5 mM EtSH.
- Auflösen Sie 12,35 g Tris-HCl, 2,62 g Tris (freie Basis) und 13,2 g NaCl in 800 mL deionisiertes Wasser und fügen Sie 10 ml einer 10% (v/v) TX-100-Lösung hinzu.
- Überprüfen Sie pH bei RT und passen Sie sich bei Bedarf mit NaOH oder HCl [5 M] an pH 7,5 an.
- Machen Sie bis zu 1 L und filtern Sie durch eine 0,22 μm Nitrocellulose-Membran.
- Fügen Sie 35 μL EtSH pro 100 mL Puffer (5 mM Endkonzentration) direkt vor der Verwendung hinzu.
- Waschpuffer 250 (WB250): 250 mM NaCl, 40 mM Tris-HCl pH 8.0 (RT), 0,1% (v/v) TX-100, 5 mM EtSH.
- Bereiten Sie WB250 wie beschrieben für B250 (2.1.1) vor, aber mit 3,59 g Tris-HCl und 2,08 g Tris (freie Basis).
- Waschpuffer 150 (WB150): 150 mM NaCl, 40 mM Tris-HCl pH 8.0 (RT), 0,1% (v/v) TX-100, 5 mM EtSH.
- Bereiten Sie WB150 vor, wie für B250 (2.1.1) beschrieben, aber mit 3,59 g Tris-HCl, 2,08 g Tris (freie Basis) und 8,77 g NaCl.
- TEV Gleichgewichtspuffer (TEB): 150 mM NaCl, 40 mM Tris-HCl pH 8.0 (RT), 0,5 mM EDTA, 0,1% (v/v) TX-100,5 mM EtSH.
- Wie WB150, aber fügen Sie EDTA auf 0,5 mM endgültige Konzentration.
- TEV Spaltpuffer (TCB): 150 mM NaCl, 40 mM Tris-HCl pH 8.0 (RT), 0,5 mM EDTA, 0,1% (v/v) TX-100, 10% (v/v) Glycerin, 5 mM EtSH.
- Bereiten Sie sich wie in Schritt 2.1.1 mit 3,59 g Tris-HCl, 2,08 g Tris (freie Basis) und 8,77 g NaCl vor und fügen Sie 100 ml Glycerin hinzu, bevor Sie das Volumen auf 1 L einstellen.
- 50 × Protease-Inhibitor-Cocktail: Lösen Sie eine Tablette eines kommerziell erhältlichen Cocktails aus Protease-Inhibitoren in 1 ml Wasser auf und lagern Sie bei-20 ° C.
- TBS-T: 50 mM Tris-HCl pH 7.6 (RT), 0,9% (w/v) NaCl, 0,1% (v/v) Polysorbat 20.
- Bereiten Sie sich wie in Schritt 2.1.1 beschrieben vor. Verwenden Sie 6,06 g Tris-HCl, 1,40 g Tris (freie Basis), 9 g NaCl und 1 ml Polysorbate 20.
- 5 × LSB (Laemmli-Musterpuffer): 315 mM Tris-HCl pH 6.8 (RT), 25% (v/v) EtSH, 50% (v/v) Glycerol, 10% (w/v) SDS, 0,05% (w/v) bromophenol blau.
- Extraktionspuffer (B250): 250 mM NaCl, 100 mM Tris-HCl pH 7.5 (RT), 0,1% (v/v) TX-100, 5 mM EtSH.
- Zubereitung von Proteinextrakt
- 1,5 g lyophilisierende Myzelie und eine Schleifkugel in das Schleifgefäß einer Kugelmühle geben.
NOTE: Auch frische Myzelien können verwendet werden. Die Myzelie vor dem Einfrieren möglichst gründlich trocknenund die Schleifgefäße in flüssigem Stickstoff zum Schleifen frischer Myzelien vorkühlen. - Mycelia bei 25 Hz für 30 s zu pulverisieren.
Hinweis: In Fällen, in denen keine Schleifmaschine zur Verfügung steht, schleifen Sie Myzelien mit Mörtel und Pestle zu einem Pulver, wie es von Bayram et al beschrieben wird. 12. - Mit Myzelpulver auf ein 15 ml Zentrifugenrohr übertragen.
- Kippen Sie das Zentrifugenrohr mit gemahlener Myzelie, um eine nachträgliche Vermischung der Myzelie mit Puffer zu ermöglichen.
- Fügen Sie 6 mL eiskalten B250 inklusive 1 × Protease-Inhibitor-Cocktail pro Gramm Myzelpulver hinzu und mischen Sie mit einem kleinen Spachtel, bis eine vollständige Homogenisierung des Rohextrakts erreicht ist. Wenn Sie frische Myzelien verwenden, verweisen Sie auf Bayram et al. 12, um das richtige Biomasse-bis Förderpufferverhältnis zu erreichen.
- Halten Sie die Röhre auf Eis für 5 min.
- Legen Sie das Rohr und ein Balance-Rohr in eine Zentrifuge und drehen Sie bei 40.000 × g für "20 min bei 4 ° C." Führen Sie während der Zentrifugation das Gleichgewicht von IgG-Harz (Schritt 2.3) durch.
- Entfernen Sie nach der Zentrifugation 10 μL des Supernatants für die SDS-PAGE-Analyse. Legen Sie die Probe in ein 1,5 mL-Rohr mit 40 μL Wasser und 12,5 μL von 5 × LSB; In Abbildung1 als "Ex" bezeichnet.
- Entfernen Sie das Supernatant (gereinigtes Lysat) vorsichtig mit einer serologischen Pipette und übertragen Sie auf die Säule, die die gleichgesättigten IgG-Perlen (step2.3) enthält, und schließen Sie sich mit der mitgelieferten Endkappe fest.
- 1,5 g lyophilisierende Myzelie und eine Schleifkugel in das Schleifgefäß einer Kugelmühle geben.
- Gleichgewichtung von IgG-Harz (durchgeführt während des Schrittes 2.2.7)
- Bereiten Sie eine 10 ml Einweg-Chromatographie-Säule und pipet 300 μL gut resuspended IgG Harz (50% Gülle) in die Spalte. Füllen Sie die Spalte auf 10 mL mit B250 und lassen Sie den Puffer durch die Schwerkraft durchströmen.
- Fügen Sie 1 mL B250 inklusive 1 × Protease-Inhibitor-Cocktail hinzu und lassen Sie durchströmen. Stecken Sie die Unterseite der Spalte.
- Batch-Reinigung von TAP-getagkendem HDAC
- Inkubieren Sie die Chromatografiesäule mit den gleichgewichtlichen Perlen und dem gereinigten Lysat aus Schritt 2.2.9 auf einem Drehmischer bei 10 U/min bei 4 ° C für 2 – 4 h.
- Nach der Stapelbindung die Kappe entfernen und die Säule unten öffnen, um den Durchflussvorgang zu sammeln.
- Nehmen Sie eine Probe für die SDS-PAGE-Analyse, wie sie in Schritt 2.2.8 beschrieben wird; In Abbildung1 als "FT" bezeichnet.
- Waschen Sie Perlen mit 10 ml WB250. Verwenden Sie zunächst 1 mL Puffer, um eingeklemmte Perlen mit einem Pipettor aus der Säulenkappe zu entfernen und diese Aufhängung in einem Spül auf das geräste Harz zu übertragen, um die Wiedergeburtung der Perlen zu ermöglichen. Dann füllen Sie die Säule nach oben, schließen Sie mit einer Stapelkappe und verbinden Sie sich mit einer Peristaltikpumpe. Stellen Sie eine Durchflussrate von ca. 1 – 5 mL/min ein. Verhindern Sie, dass das Harz trocken läuft.
- Wiederholungsschritt 2.4.4 dreimal für insgesamt vier Waschbären mit WB250.
- Mehrperlen dreimal mit 10 ml TEB waschen, wie in Schritt 2.4.4 beschrieben.
- Schließen Sie die Chromatografie-Säule an der Unterseite, erneuern Sie die IgG-Perlen in 1 mL TCB , und fügen Sie 20 ° L von 50 × Protease-Inhibitor-Cocktail sowie 10 μL TEV (~ 1 mg/mL Lager, S219V Mutant-Variante im eigenen Haus produziert).
- Kappen Sie die Säule und inkubieren Sie auf einem Drehmischer bei 10 U/min bei 4 ° C über Nacht, um die Proteinkomplexe, die über den markierten HDAC gebunden sind, zu beseitigen.
Hinweis: Alternativ kann TEV bei erhöhter Temperatur (16 – 25 ° C) durchgeführt werden, was die Reaktionszeit reduziert, aber das Risiko eines Eiweißabbaus erhöht. - Am nächsten Tag öffnen Sie die Säule und sammeln Sie das Eluat in einem 2 mL Zentrifuge Rohr. Verwenden Sie 0,7 ml TCB, um Perlen von der Kappe zu entfernen und die Wand der Säule zu spülen.
- Legen Sie die Säule aus dem vorigen Schritt auf das offene 2-mL-Rohr, das selbst in einem 50 mL-Zentrifugenrohr platziert ist.
- Übertragen Sie diese Montage in eine Tischzentrifuge und drehen Sie mit 300 × g für 2 min. Das ist das TEV-Eluat.
Hinweis: Bei der Tandemaffinitätsreinigung verwenden Sie das TEV-Eluat als Eingang für den Kalmodulin-Affinitätsschritt. Sie können auch das TEV-Eluat aufteilen und einen Teil für die Feststellung von HDAC-Aktivitäten und den zweiten Teil verwenden, um die TAP-Reinigung abzuschließen. - 50 μL aus dem TEV-Eluat entfernen und 12,5 μL von 5 × LSB für SDS-PAGE (in den Abbildungen 1 und 2"TE" genannt) hinzufügen und die restlichen Eluate auf Eis halten.
- Um die Wirksamkeit der Protease-Elasution zu beurteilen, fügen Sie 2 mL von 5% (v/v) Essigsäure und Inkubat bei RT für 5 min. Auch hier wird das erste mL verwendet, um das Harz wiederzubeleben.
- Sammeln Sie das Säure-Eluat und nehmen Sie wieder 50 μL Probe und fügen Sie 12,5 μL von 5 × LSB (in Abbildung1 als "AE" bezeichnet). LSB wird gelb, wenn es der sauren Lösung hinzugefügt wird. Um die Säure zu neutralisieren, fügen Sie 10 M NaOH in Schritten von 1 μL hinzu und mischen Sie gut, bis die Farbe wieder blau wird.
- Das IgG-Harz mit TBS-T wieder ausgleichen, um die Säure zu neutralisieren. Bewahren Sie das Harz in TBS-T/20% (v/v) Ethanol bei 4 ° C für die Wiederverwendung mit dem gleichen markierten Protein auf.
- Lagerung von Lösungsfraktionen
- Das Elutieren in ~ 100 μL-Braktionen ausrichten, um mehrere Gefrier-Twetterzyklen zu vermeiden.
- Aliquots in flüssigem Stickstoff einfrieren und bei-80 ° C halten.
Hinweis: Auf diese Weise gespeicherte Proben werden monatelang stabil sein, ohne enzymatische Aktivität zu verlieren.
3. Analyse der Reinigung durch SDS-PAGE und Westernblotting
- Verwenden Sie Standard-Protokolle zum Gießen von SDS-Polyacrylamid-Gelen oder verwenden Sie Fertiggel13. Denature-Gel-Proben aus dem vorigen Abschnitt bei 95 ° C für 5 min, Zentrifuge bei "15.000 × g für 5 min" und Last auf 12% Gele. Empfohlene Ladevolumen sind in der Legende von Abbildung1 angegeben. Elektrophorese die Proben in 1 × Tris-Glycine SDS-PAGE laufen Puffer bei 180 V-Konstante für 60 – 70 min, bis die bromophenolblaue Markierung des SDS-PAGE-Ladepuffers beginnt, aus dem Gel zu wandern.
- Verwenden Sie Standard-Protokolle für die Silberfärbung der Gele. Verwenden Sie zum Beispiel das Protokoll von Blum et al. 1987 14, das ist auch mit der MS-Analyse kompatibel.
- Verwenden Sie Standard-Protokolle für Western blotting15. Für die Generierung von repräsentativen Ergebnissen wurde unten ein handelsübliches Blottiersystem eingesetzt.
- Probenblasen mit handelsüblichen Anti-Kalmodubinungsproteinen (Anti-CBP) Antikörper in 5% (w/v) Milchpulver in TBS-T bei 4 ° C über Nacht.
Hinweis: Anti-CBP-Antikörper richtet sich gegen den Teil des TAP-Tags, der nach der TEV-Spaltung noch vorhanden ist. - Verwenden Sie Standard-Protokolle für die Erkennung und Entwicklung von Blots. So wurden zum Beispiel Anti-Kaninasen IgG-alkalische Phosphatase Konjugat und ein BCIP/NBT Farbentwicklungssubstrat für die Generierung von repräsentativen Ergebnissen verwendet.
4. Deacetylase-Test mit in vitro [3H] acetat-beschrifteten Hühner-Renticulocyt-Histonen (nach Trojer et al. 2003)4
- Siehe das Protokoll von Kölle et al. 199816 für die Zubereitung von [3H] acetat-beschrifteten Hühnerreticulozyten-Histonen.
- Pro Assay-Zustand drei 1,5 mL Zentrifugenröhren auf Eis für Messungen in Triplikaten. Bereiten Sie auch drei Rohre für die puffere Hintergrundkontrolle vor.
- 25 μL WB150 in jedes Rohr geben und 25 μL des TEV-Eluats von Schritt 2.4.11 hinzufügen und auf Eis halten.
- Einen Rohrinkubator auf 25 ° C vorheizen.
- Verwenden Sie 15 s Intervalle (Stoppuhr) für die Zugabe von 10 μL von beschrifteten Histonen [1,5 mg/mL] zu jedem Rohr.
- Bevor Sie mit dem Test beginnen, nehmen Sie 10 μL der [3H] aketatären Hühnerhistones auf und starten Sie die Stoppuhr.
- Fünf Sekunden nach dem Start die beschrifteten Histone dazugeben, die Röhre fest schließen, Wirbel ziehen und in den Inkubator geben.
- Verwenden Sie 15 s Intervalle für den Zusatz von 10 μL von beschrifteten Histonen und gehen Sie wie im vorherigen Schritt beschrieben.
- Nach 60 Minuten Inkubation 50 μL Stopp-Lösung (1 M HCl/0,4 M Essigsäure) in jedes Rohr in 15 s Intervallen hinzufügen; Wirbel sofort. Nach dem Zusatz der sauren Lösung ist der Assay-Mix stabil und kann bis zum nächsten Schritt bei RT gehalten werden.
- Fügen Sie jedem Rohr 800 μL Ethylacetat hinzu, um die freigesetzte [3H] Essigsäure zu extrahieren.
- Schließen Sie die Rohre und Wirbel jedes Rohr für 5 s fest.
- Legen Sie das Rohr in eine Mikrozentrifuge und drehen Sie sich bei 10 Minuten bei RT um 10.000 × g .
- In der Zwischenzeit eine Szintillation vial pro Assay-Probe vorbereiten und 3 mL Szintillation Cocktail für hydrophobe Proben hinzufügen.
- Nach der Zentrifugation (Schritt 2.12) 600 μL der oberen organischen Phase vorsichtig auf die vorbereiteten Szintillationsrohre übertragen und die Rohre fest verschließen.
- Messen Sie die Radioaktivität, die der HDAC-Aktivität in einem flüssigen Szintillationszähler entspricht.
Ergebnisse
Ein typisches Ergebnis der vorgestellten Einstufenanreicherung von RpdA ist in Abbildung 1 ("IgG" genannt) zu sehen. Der Vollständigkeit halber haben wir auch Durchfluss-und Ellenbänder ("CFT" und "CE") aufgenommen, die den zweiten Reinigungsschritt durch ein Calmodulin-Harz ("CaM") illustrieren, wie es12beschrieben wurde. Das ausgezeigte silberbefleckte Gel (A) zeigt deutlich die Wirksamkeit des ersten Affinitätsschrittes, der bei der Tandemreinigung noch weiter erhöht wird. Die meisten prominenten Proteine, die im Proteinextrakt und im Durchströmen vorhanden sind, sind jedoch bereits im TEV-Eluate erschöpft. (TE) Es ist wichtig zu beachten, dass die TEV-Ellenbänder im Vergleich zu Extrakt und gt;100 Durchströmen × konzentriert sind. Die Sternchen markieren den markierten RpdA (vgl. Panel B). Die berechnete MW von RpdA inklusive CBP (RpdA: CBP) beträgt 82 kDa, das Protein wandert jedoch bei einem deutlich höheren scheinbaren Molekülgewicht von ca. 120 kDa. Dieses Phänomen wurde bereits beobachtet und kann den spezifischen Eigenschaften seines C-terminus 4,17zugeordnet werden. Der Immunoblot (B) zeigt starke Signale, die bei ca. 120 kDa abwandern, entsprechend CBP-getaggtem RpdA (RpdA:: CBP) im TEV-Eluat ("TE"), dem Kalmodulin-Durchströmen ("CFT"), und Eluate ("CE"). Im Säure-Eluat ("AE") ist ein zweites Signal mit einem etwas größeren MW zu sehen. Dies entspricht dem Anteil der an das IgG-Harz gebundenen RpdA, der nicht durch TEV-Senkung freigesetzt wurde. Der Größenunterschied entspricht 16 kDa des Proteins Eine Wiederholung von ungespaltertem RpdA:: TAP. Wie zu erwarten, konnten keine Bänder durch den Anti-CBP-Antikörper in den Wild-Typ-Kontrollfraktionen (Tafel B, "Wildtyp") erkannt werden.
Die Ergebnisse eines repräsentativen Deacetylase-Aktivitätsuntersuchungen mit dem spezifischen HDAC-Inhibitor-Trichostatin A (TSA) werden in Abbildung2 dargestellt. Dieser Test wurde ursprünglich für die Pflanzen 18 entwickelt und wurde auch für die Hemmung von Mambitor-Screening gegen Säugetierdiakete19,20verwendet. Die für den Test verwendeten Histone wurden beschriftet und wie beschrieben 16. Es wird gezeigt, wie sich die Konzentrationen von TSA auf die katalytische Aktivität des angereicherten RpdA-Komplexes ("TE ± TSA") auswirken. Die Empfindlichkeit der Aktivität bestätigt, dass die gemessenen cpM-Werte tatsächlich auf RpdA zurückzuführen sind und nicht durch unspezifische Protease-Aktivität. Dies ist eine wichtige Beobachtung, da sie darauf hinweist, dass TEV, das bei einer recht hohen Konzentration vorhanden ist, nicht in den HDAC-Aktivitäts-Test eingreift. Um die gemessene HDAC-Aktivität RpdA zuzuordnen, wurde eine Wild-Typ-Steuerung als Negativsteuerung ("Co") eingesetzt. Erwartungsgemäß wurde im TEV-Eluat des wilden Typs nur marginale HDAC-Aktivität (ca. 5-10% der RpdA-angereicherten Bruchteile) nachgewiesen. Interessanterweise wird die HDAC-Aktivität nach dem zweiten Affinitätsreinigungsschritt ("CE") im Vergleich zum TEV-Eluat deutlich reduziert.
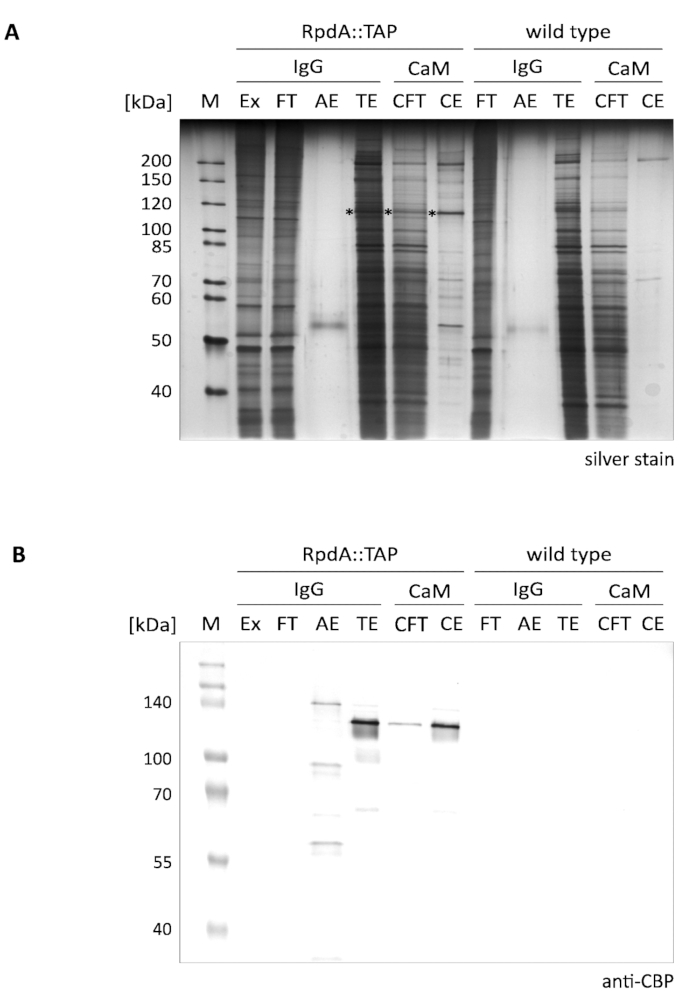
Bild 1. Tandem Affinity purificationation of TAP-tagged RpdA. Es werden ein silberfarbenes 10% SDS-Polyacrylamid-Gel (A) und ein westlicher Schandfleck mit dem Anti-CBP-Antikörper (B) gezeigt. Die Spurenkennzeichnung und die belasteten Mengen sind wie folgt: "M": 2 °L 1:10 verdünnter unbefleckter Proteinmarker (Silberfleck), 3,5 °L prägefärbter Proteinmarker (westlicher Schlot); "Ex": Proteinextraktprobe, wie sie in Schritt 2.2.8 (2 -L 1:10 Verdünnung, 5 -L) hergestellt wird; "FT": IgG-Harzdurchlaufprobe, wie sie in Schritt 2.4.3 (2 -L von 1:10 Verdünnung, 5 °L) vorbereitet ist; "AE": Säure Eluate des Schrittes 2.4.14 (10 -, 10 -L); "TE": TEV Eyelution von TEV Spalt, Schritt 2.4.12 (10-L, 10 -L); "CFT": Kalmodulin durchströmen (20 μL, 20 μL); "Ce": Calmodulin eluate (10 μL, 10 μL). Die Größe der ausgewählten Marker-Proteine ist auf der linken Seite der Paneele angegeben. Die in Klammern angegebenen Mengen entsprechen Probenbelastungen für Silberfleck bzw. Westernschlitz. Sternchen im silberbefleckten Gel deuten auf das RpdA Fusionsprotein hin. Der Immunoblot (B) wurde mit alkalischer Phosphatase mit dem Farbentwicklungssystem BCIP/NBT nachgewiesen. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
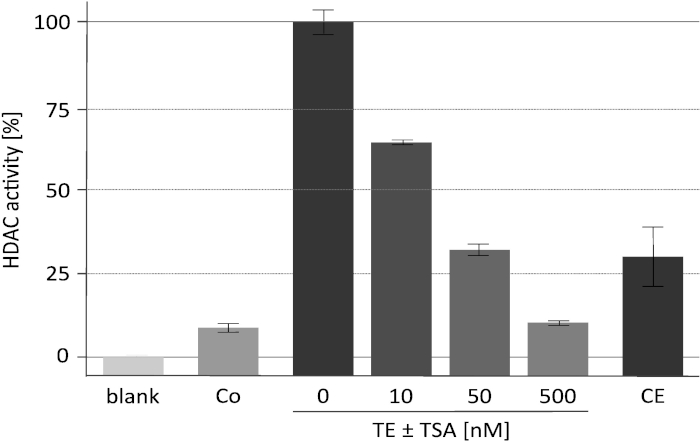
Bild 2. HDAC-Aktivitätsuntergang unter zunehmender Konzentration von Trichostatin A. Die Wirksamkeit der RpdA-Hemmung wurde mit 25 μL affinity-gereinigtem rekombiniertem RpdA ("TE") und 25 μL von 0,10,50 und 500 nM des HDAC-Inhibitors TSA getestet, der im RPMI-1640-Medium verdünnt ist. RPMI wurde verwendet, um die Hintergrundaktivität ("blank") zu beurteilen. Die Aktivitäten des Endversorgers nach dem zweiten Kalmodulin-Affinitätsschritt ("CE") und einer unbetagschten Sorte nach dem ersten Affinitätsreinigungsschritt (Negativkontrolle, "Co") werden angezeigt. Aktivitäten werden als Prozent der angereicherten RpdA ohne TSA (100%, "TE") gezeigt. Fehlerbalken geben die Standardabweichung von drei Replikaten an. Diese Zahl wurde von Bauer et al . 20162 modifiziert. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Diskussion
Dieses Protokoll beschreibt eine einstufige Anreicherung eines TAP-getaggten BATP-markierten BOP-Klasse-1-FHDAC aus dem filamentösen Pilz A . nidulans zur Beurteilung der In-vitro-Deacetylase-Aktivität. Das TAP-Tag wurde ursprünglich in Bäckerhefe zur Identifizierung von Eiweiß-Protein-Interaktionspartnern des markierten Proteins6 eingeführt. Anschließend wurde der Tag für seinen Einsatz in A. nidulans5für den Einsatz in A. nidulans 5 kodon-optimiert. Hier stellen wir ein geradliniges Schritt-für-Schritt-Protokoll für die Anwendung des ersten Affinitätsreinigungsschrittes der TAP-Strategie zur einstufigen Anreicherung der Klasse 1 HDAC RpdA zur Verfügung. Der zweite Affinitätsreinigungsschritt erhöht deutlich den Reinigungsgrad, was besonders wichtig für die Identifizierung von basteninteragierenden Proteinen ist. Dennoch wird nur der erste Schritt für spätere Aktivitätstests empfohlen, da der Mangel an signifikanter Kontamination nach dem ersten Schritt durch ein Kontrollexperiment mit einem wilden Stamm bestätigt wurde. Darüber hinaus ist die Aktivität nach einstufiger Anreicherung im Vergleich zum vollständigen TAP deutlich höher. Neben RpdA2 wurdedieses Protokoll auch erfolgreich zur Reinigung von A. nidulans-Komplexen der zweiten Klasse 1 HDAC HosA21eingesetzt.
Aufgrund unserer Beobachtungen, dass die Pilzklasse 1 HDACs stabile Komplexe4bilden, ist es uns gelungen, das Protokoll von Bayram et al . 2012 12zu ändern, das die Grundlage dieser Methode darstellt. Dennoch müssen einige kritische Schritte erwähnt werden. Die Herstellung hochkonzentrierter Proteinextrakte, um nahezu physiologische Bedingungen zu gewährleisten, ist für komplexe Stabilität entscheidend. Daher ist es wichtig, die gegebenen Empfehlungen bezüglich des Pufferverhältnisses zur Biomass/Extraktion zu berücksichtigen. In dieser Hinsicht ist es auch wichtig, gut gemahlen feine Myzelpulver zu verwenden, um eine ordnungsgemäße Extraktion zu gewährleisten. Hier ist der Einsatz einer Schleifmaschine eindeutig von Vorteil. Wie im Protokollabschnitt erwähnt, lohnt es sich, den TSV-Kaltsaal für 1-2 h bei Raumtemperatur auszuprobieren, um die Reinigung zu beschleunigen. Dies wurde für RpdA getestet, ohne schädliche Auswirkungen auf die Stabilität zu beobachten (unveröffentlichte Beobachtung, Bauer I, 2018). Darüber hinaus ist der Austausch von NP40 (im Originalprotokoll) mit TX-100 möglicherweise nicht für andere Proteinkomplexe geeignet. Bei der Verwendung dieser Methode zur Reinigung anderer TAP-markierter Proteine sollte man sich auch auf das Bayram-Protokoll beziehen, das eine Reihe wertvoller Hinweise enthält, die für eine ausreichende Reinigung anderer Proteinkomplexe 12 hilfreich seinkönnten.
Neben der hier beschriebenen TAP-Methode werden häufig auch andere Affinitäts-Tags und-Techniken zur einstufigen Anreicherung von Proteinen von filamentösen Pilzen verwendet, darunter His-und GFP-Tags. Da die HDACs der Klasse 1 jedoch in der Regel als hochmolekulare Komplexe funktionstüchtig sind, sind einheimische Elitionsbedingungen Voraussetzung für die Anreicherung katalytisch aktiver HDACs. Wichtig ist, dass viele andere Affinitätsreinigungen unter ungünstigen Bedingungen durchgeführt werden. So ist beispielsweise die Anreicherung von HDACs über GFP-Falle, die auf der Anti-Gen-Antikörper-Interaktion basiert, aufgrund der sauren Elitionsbedingungen, die die Proteinprotein-Interaktion von Harzverbindungen stören, nicht geeignet. Darüber hinaus führten Versuche, His-tagged RpdA durch Metallchelat-Affinität Chromatographie22zu reinigen, zu einem erheblichen Verlust der katalytischen Aktivität während des Reinigungsverfahrens, obwohl Imidazol anstelle von niedrigen pH-Bedingungen für die Lösung verwendet wurde ( Unveröffentlichte Daten, Bauer, I, 2010).
Eine Einschränkung des beschriebenen enzymatischen Assay-Protokolls ist die Verwendung des radioaktiven Substrats. Allerdings wurden auch 23, 24 Tests auf Fluoreszenzbasis entwickelt und sind kommerziell erhältlich. Diese Assays werden in gut gezeigten Platten durchgeführt und eignen sich somit auch für das Hochdurchsatz-Screening von HDAC-Inhibitoren. In diesem Fall wäre eine gehobene Verfahrensweise erforderlich.
Mögliche anstehende Anwendungen dieses Protokolls umfassen die Anreicherung von spezifischen Teil-Komplexen von DGB 1-Festplatten, um ihre spezifischen physiologischen Rollen zu beurteilen und/oder Unterschiede in ihrer Anfälligkeit für HDAC-Inhibitoren. Bei der Festlegung der beschriebenen Methode für andere Enzyme wird dringend empfohlen, ein negatives Kontrollexperiment mit einem nicht tagkenden Stamm durchzuführen. Dies sichert die Spezifität der gemessenen Enzymaktivitäten und würde eine Verunreinigung durch unspezifisch gebundene Enzyme aufdecken.
Die hier beschriebenen Reinigungs-und Aktivitätsuntersuchen können innerhalb von zwei Tagen durchgeführt werden und die angereicherten Enzyme sind mindestens mehrere Monate stabil, wenn sie in Aliquots bei-80 ° C gelagert werden. Zusammenfassend bietet dieses Protokoll eine relativ einfache und kostengünstige Möglichkeit, um die HDAC-Komplexe der Klasse 1 zur Aktivitätsmessung und Bestimmung der Inhibitor-und Wirkungskraft zu erreichen.
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Wir danken Petra Merschak, Abteilung Molekularbiologie (Biozentrum, Medizinische Universität Innsbruck), für ihre Hilfe und Unterstützung bei diesem Manuskript. Darüber hinaus möchten wir uns bei den Gutachtern für ihre wertvollen Kommentare bedanken.
Diese Arbeit wurde vom Wissenschaftsfonds (P24803 an SG und P21087 zu GB) und von der intramural Fördermittel (MUI Start, ST201405031 bis IB) finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| 10 x SDS-PAGE running buffer | Novex | ||
| 2-mercaptoethanol (EtSH) | Roth | 4227 | |
| 25 cm2 cell culture flasks with vent cap | Sarstedt | 833910002 | For spore production |
| 47 mm vacuum filtration unit | Roth | EYA7.1 | |
| AccuFLEX LSC-8000 | HITACHI | – | Scintillation counter |
| Acetic acid | Roth | 7332 | |
| Anti-Calmodulin Binding Protein Epitope Tag Antibody | Millipore | 07-482 | Used at 1:1333 dilution |
| Anti-Rabbit IgG (whole molecule)–Alkaline Phosphatase (AP) antibody | Sigma-Aldrich | A3687 | |
| Ball mill | Retsch | 207450001 | Mixer Mill MM 400 |
| BCIP/NBT | Promega | S3771 | Color development substrate for AP |
| Cell strainer | Greiner | 542040 | |
| Cheese cloth for harvesting mycelia | BioRen | H0028 | Topfentuch |
| Dimethylsulfoxid (DMSO) | Roth | 4720 | |
| EDTA | Prolabo | 20309.296 | |
| Ethyl acetate | Scharlau | Ac0155 | |
| Freeze Dryer | LABCONCO | 7400030 | FreeZone Triad |
| Glycerol | Roth | 3783 | |
| HCl | Roth | 4625 | |
| IgG resin | GE Healthcare | 17-0969-01 | IgG Sepharose 6 Fast Flow |
| Inoculation loops | VWR | 612-2498 | |
| KOH | Merck | 5033 | |
| Laminar flow cabinet | Thermo Scientific | – | Hera Safe KS |
| Mixed Cellulose Esters Membrane Filters | Millipore | GSWP04700 | |
| NaCl | Roth | 3957 | |
| NaOH | Roth | 6771 | |
| Neubauer counting chamber improved | Roth | T728 | |
| Novex gel system | Thermo Scientific | For SDS-PAGE | |
| Novex Tris-glycine SDS running buffer (10X) | Thermo Scientific | LC2675 | Running buffer for SDS-PAGE |
| peqGold protein-marker II | VWR | 27-2010P | Protein ladder used for silver stain |
| Peristaltic Pump P-1 | GE Healthcare | 18111091 | |
| Pipette controller | Brand | 26302 | accu-jet pro |
| Poly-Prep chromatography columns | Bio-Rad | 731-1550 | |
| ProSieve QuadColor protein marker | Biozym | 193837 | Prestained protein ladder used for western blot |
| Protease inhibitor cocktail tablets | Sigma-Aldrich | 11873580001 | cOmplete, EDTA-free |
| Rotary mixer | ELMI | – | Intelli-Mixer RM-2 S |
| Rotiszint eco plus | Roth | 0016 | |
| RPMI-1640 | Sigma-Aldrich | R6504 | |
| Scintillation vials | Greiner | 619080 | |
| Sorvall Lynx 4000 | Thermo Scientific | ||
| Thermomixer comfort | Eppendorf | ||
| TIB32.1 | A. nidulans rpdA::TAP strain. Genotype: alcA(p)::rpdA; veA1; argB2; yA2; pIB32::argB; ArgB+; PyrG+ | ||
| Trans-Blot Turbo RTA Midi Nitrocellulose Transfer Kit | Bio-Rad | 1704271 | |
| Trans-Blot Turbo Transfer System | Bio-Rad | 1704150 | |
| Trichostatin A (TSA) | Sigma-Aldrich | T8552 | 5 mM stock in DMSO |
| Tris (free base) | Serva | 37190 | |
| Tris-HCl | Roth | 9090 | |
| Polysorbate 20 | Roth | 9127 | Tween 20 |
| Polysorbate 80 | Sigma-Aldrich | P1754 | Tween 80 |
| TX-100 | Acros Organics | 215682500 | Triton X-100, Octoxynol-9 detergent |
Referenzen
- Gregoretti, I. V., Lee, Y. -. M., Goodson, H. V. Molecular evolution of the histone deacetylase family: functional implications of phylogenetic analysis. Journal of Molecular Biology. 338 (1), 17-31 (2004).
- Bauer, I., et al. A Class 1 Histone Deacetylase with Potential as an Antifungal Target. mBio. 7 (6), (2016).
- Stefan, A., et al. Purification of active recombinant human histone deacetylase 1 (HDAC1) overexpressed in Escherichia coli. Biotechnology Letters. 40 (9-10), 1355-1363 (2018).
- Trojer, P., et al. Histone deacetylases in fungi: novel members, new facts. Nucleic Acids Research. 31 (14), 3971-3981 (2003).
- Bayram, &. #. 2. 1. 4. ;., et al. VelB/VeA/LaeA complex coordinates light signal with fungal development and secondary metabolism. Science. 320 (5882), 1504-1506 (2008).
- Puig, O., et al. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24 (3), 218-229 (2001).
- Rigaut, G., et al. A generic protein purification method for protein complex characterization and proteome exploration. Nature Biotechnology. 17 (10), 1030-1032 (1999).
- Hill, T. W., Kafer, E. Improved protocols for Aspergillus minimal medium: trace element and minimal medium salt stock solutions. Fungal Genetics Reports. 48, (2001).
- Todd, R. B., Davis, M. A., Hynes, M. J. Genetic manipulation of Aspergillus nidulans: meiotic progeny for genetic analysis and strain construction. Nature Protocols. 2 (4), 811-821 (2007).
- Zadra, I., Abt, B., Parson, W., Haas, H. xylP promoter-based expression system and its use for antisense downregulation of the Penicillium chrysogenum nitrogen regulator NRE. Applied and Environmental Microbiology. 66 (11), 4810-4816 (2000).
- Stolz, D. J., et al. Histological Quantification to Determine Lung Fungal Burden in Experimental Aspergillosis. Journal of Visualized Experiments. (133), (2018).
- Bayram, &. #. 2. 1. 4. ;., et al. Identification of protein complexes from filamentous fungi with tandem affinity purification. Methods in Molecular Biology. 944, 191-205 (2012).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Blum, H., Beier, H., Gross, H. J. Improved silver staining of plant proteins, RNA and DNA in polyacrylamide gels. Electrophoresis. 8 (2), 93-99 (1987).
- Towbin, H., Staehelin, T., Gordon, J. Electrophoretic transfer of proteins from polyacrylamide gels to nitrocellulose sheets: procedure and some applications. Biotechnology. 24, 145-149 (1992).
- Kölle, D., et al. Biochemical methods for analysis of histone deacetylases. Methods. 15 (4), 323-331 (1998).
- Tribus, M., et al. A novel motif in fungal class 1 histone deacetylases is essential for growth and development of Aspergillus. Molecular Biology of the Cell. 21 (2), 345-353 (2010).
- Sendra, R., Rodrigo, I., Salvador, M. L., Franco, L. Characterization of pea histone deacetylases. Plant Molecular Biology. 11 (6), 857-866 (1988).
- Valente, S., et al. 1,3,4-Oxadiazole-containing histone deacetylase inhibitors: anticancer activities in cancer cells. Journal of Medicinal Chemistry. 57 (14), 6259-6265 (2014).
- Mai, A., et al. Synthesis and biological properties of novel, uracil-containing histone deacetylase inhibitors. Journal of Medicinal Chemistry. 49 (20), 6046-6056 (2006).
- Pidroni, A., et al. A Class 1 Histone Deacetylase as Major Regulator of Secondary Metabolite Production in Aspergillus nidulans. Frontiers in Microbiology. 9, 2212 (2018).
- Porath, J., Carlsson, J., Olsson, I., Belfrage, G. Metal chelate affinity chromatography, a new approach to protein fractionation. Nature. 258 (5536), 598-599 (1975).
- Wegener, D., Wirsching, F., Riester, D., Schwienhorst, A. A fluorogenic histone deacetylase assay well suited for high-throughput activity screening. Chemistry & Biology. 10 (1), 61-68 (2003).
- Peng, L., Yuan, Z., Seto, E. Histone deacetylase activity assay. Methods in Molecular Biology. 1288, 95-108 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten