Method Article
Mit Zapfen angereicherte Kulturen von der Netzhaut von Hühnerembryonen zur Untersuchung zellulärer Interaktionen von Stäbchen zu Zapfen
In diesem Artikel
Zusammenfassung
Wir beschreiben eine Methode zur Gewinnung von Primärkulturen von Zapfenphotorezeptoren aus der Netzhaut von Hühnerembryonen und deren Verwendung für ein High-Content-Screening.
Zusammenfassung
Das menschliche Tagessehen beruht auf der Funktion von Zapfenphotorezeptoren im Zentrum der Netzhaut, der Fovea. Patienten, die an der häufigsten Form der vererbten Netzhautdegeneration, Retinitis pigmentosa, leiden, verlieren die Nachtsicht aufgrund des mutationsbedingten Verlustes von Stäbchenphotorezeptoren, ein Phänomen, dem ein fortschreitender Funktionsverlust und der Tod von Zapfen folgen, was zur Erblindung führt. Genetiker haben viele Gene mit Mutationen identifiziert, die diese Krankheit verursachen, aber die ersten identifizierten Mutationen stellten die Mechanismen der sekundären Zapfendegeneration in Frage und wie eine dominante Mutation im Rhodopsin-Gen, das für das ausschließlich in Stäbchen exprimierte visuelle Pigment kodiert, eine Zapfendegeneration auslösen kann.
Dieses Ergebnis von Transplantationen in einem genetischen Modell der Krankheit führte zum Konzept der Zellinteraktionen zwischen Stäbchen und Zapfen und der autonomen Degeneration von Zapfen in allen genetischen Formen der Retinitis pigmentosa.
Zapfen machen 5% aller Photorezeptoren beim Menschen und nur 3% bei der Maus aus, so dass ihre Untersuchung bei diesen Arten schwierig ist, aber Zapfen übertreffen Die Stäbchen bei Vogelarten. Wir haben 96-Well-Platten angepasst, um Netzhautvorläufer aus der Netzhaut von Hühnerembryonen im Stadium 29 ihrer Entwicklung zu kulturieren. In diesen Primärkulturen machen Zapfen nach in vitro Differenzierung 80% der Zellen aus. Die Zellen degenerieren über einen Zeitraum von einer Woche in Abwesenheit von Serum. Hier beschreiben wir die Methoden und deren Standardisierung.
Dieses mit Kegel angereicherte Kultursystem wurde verwendet, um den Epithel-abgeleiteten Zapfenlebensfähigkeitsfaktor (EdCVF) durch hochgehaltiges Screening einer retinalen pigmentierten Epithel-normalisierten cDNA-Bibliothek von Ratten zu identifizieren. Rekombinantes EdCVF verhindert die Degeneration der Zapfen.
Einleitung
Die Netzhaut von Wirbeltierarten ist dual, mit Stäbchenphotorezeptoren für das Sehen bei schwachem Licht und Zapfenphotorezeptoren für Tageslicht, Farbe und Sehschärfe. Die Sehschärfe von Primaten beruht auf einer Region im Zentrum der Netzhaut, der Fovea, die mit Zapfen angereichert ist, aber insgesamt machen Zapfen nur 5% aller Photorezeptoren aus. Folglich ist die Analyse der Zapfen in der Netzhaut von Primaten und insbesondere die Kultur der Zapfen technisch schwierig. Alle anderen Säugetierarten haben keine Fovea und der Prozentsatz der Zapfen ist niedrig für Nagetiere, die am häufigsten in der Netzhautforschung verwendet werden. Dies ist bei Vogelarten nicht der Fall, bei denen Zapfen die Netzhaut dieser gut sehenden Vogelarten dominieren. Dinosaurier, die das Ökosystem dominiert haben, als die Säugetiere zum ersten Mal während der Evolution auftauchten, sind der phylogenetische Ursprung der Vögel1. Als Folge einer solchen Konkurrenz zwischen Dinosauriern und frühen Säugetieren sind die Säugetiere meist nachtaktiv mit Netzhäuten, die von Stäbchen dominiert werden. Erst später im Laufe der Evolution wurde das tagaktive Sehen einiger Säugetierarten, zu denen Primaten gehören, zu einem evolutionären Vorteil. Dennoch bleibt die Ahnenzeit als Atavismus des nächtlichen Engpasses in der Evolution des Säugetiersehens2,3.
Bei der Untersuchung der Netzhautzelldifferenzierung zeigten Adler und Hatlee, dass Photorezeptoren etwa 70% der retinalen differenzierten Zellen in Kulturen ausmachen, die am Embryonaltag (ED) 6 oder Stadium 294von Hühnern stammen. Aufgrund der Prävalenz von Zapfen in der Netzhaut von Hühnern wurden Kulturen von Netzhautzellen aus ED6-Hühnerembryonen als mit Zapfen angereicherte Kulturen entwickelt5.
Die Bedeutung der kegelvermittelten Sehschärfe für den Menschen ist eine Binsenweisheit. Menschen, die von genetischen oder alternden Krankheiten betroffen sind, die die Zapfenfunktion verändern, sind stark behindert. Dies hat eine sehr große Anzahl von Studien über vererbte Netzhautdegenerationen (IRD) mit dem Ziel gefördert, Behandlungen für diese blinden Krankheiten zu finden6,7. Der erste Erfolg, der mit einem rekombinanten adeno-assoziierten Vektor (AAV) zur Therapie einer schweren Form der IRD Leber kongenitalen Amaurose (LCA) erzielt wurde, ist ein Proof of Concept für die Gentherapie8. Die Identifizierung der Gene, deren Mutationen IRD auslösen, eröffnet die Möglichkeit, diese Krankheiten durch Gentherapie zu heilen. Dennoch resultieren diese Krankheiten aus Mutationen in mehr als 200 verschiedenen Genen9. Selbst bei autosomal-rezessiven Formen der IRD, wenn die Wiedereinführung der normalen Kopie des morbiden Gens die Sehfunktion wiederherstellen könnte, begünstigen die wirtschaftlichen Kosten jeder einzelnen Entwicklung die am weitesten verbreiteten zum Nachteil der weniger häufigen und derjenigen, für die der genetische Ursprung unbekannt bleibt. Diese Tatsache veranlasste die Forscher, über allgemeinere Therapien nachzudenken. Der apoptotische Zelltod erschien als ein häufiger Weg und ein therapeutisches Ziel dieser Krankheiten, die durch die Degeneration von Photorezeptoren fortschreiten, einschließlich autosomal-dominanter Formen10,11. Die Erfolge eines solchen Ansatzes fehlen jedoch. Für die häufigste Form von IRD, Retinitis pigmentosa (RP), ist der gemeinsame Weg der sekundäre Funktionsverlust, gefolgt von der Degeneration von Zapfen12,13. Die Verhinderung des Verlustes der Zapfenfunktion bewahrt die zentrale Sicht der Fovea unabhängig von den ursächlichen Mutationen14.
Im Frühstadium der RP löst der Verlust von Stäbchen eine Verringerung der Expression des stäbchenabgeleiteten Zapfenlebensfähigkeitsfaktors (RdCVF) aus, der durch das Nukleoredoxin-ähnliche 1(NXNL1)-Genkodiert wird, das die Stoffwechsel- und Redoxsignalisierung zwischen Stäbchen und Zapfen unterbricht15. Die Verabreichung eines rekombinanten AAV, das die beiden Produkte des NXNL1-Gens, den trophischen Faktor RdCVF und das Thioredoxin-Enzym RdCVFL, kodiert, könnte theoretisch den Verlust des Zapfensehens bei allen genetischen Formen von RP16verhindern. Wir haben gezeigt, dass das NXNL1-Genprodukt, RdCVFL, in mit Hühnerkegeln angereicherten Kulturen exprimiert wird17 und wo es eine schützende Rolle spielt18. RdCVF und das NXNL1-Gen wurden durch high-content Screening einer retinalen cDNA-Bibliothek identifiziert, wobei das Überleben von Zellen aus einer mit Zapfen angereicherten Kultur als Auslesung19verwendet wurde. Wir haben das Äquivalent von 210.000 einzelnen Klonen der Bibliothek mit 8 parallelen Tests für jeden Klon überprüft. Dies stellt eine sehr große Anzahl von Tests dar, die einen einfachen Zugang zum biologischen Material, den Netzhäuten von Hühnerembryonen, erfordern. Wir fanden heraus, dass es relativ einfach war, wöchentlich embryonierte Hühnereier zu erhalten, da sie für die Agroindustrie für Legehennen und fleischproduzierende Hühner weit verbreitet sind. Nach sorgfältiger Standardisierung der mit Kegeln angereicherten Kulturen bietet das System eine einfache, robuste und reproduzierbare Möglichkeit, Tausende von Molekülen auf ihre Fähigkeit zu testen, die Lebensfähigkeit des Kegels zu erhalten. Diese Zellen sind auch für genetische Manipulationen20 anfällig, die für die Untersuchung der Signaltransduktion und für biochemische Analysen21,22,23von Vorteil sind.
Netzhautforscher haben alternative Methoden wie die Verwendung der Zapfenzelllinie 661W24,25,26entwickelt. Dennoch bleibt die Identität dieser Zelllinie umstritten27,28. Die 661W-Zellen wurden aus Netzhauttumoren einer transgenen Mauslinie geklont, die das SV40-Große-T-Antigen unter Kontrolle des humanen Interphotorezeptor-Retinol-bindenden Proteinpromotors exprimiert. SV40 großes T-Antigen vermittelt zelluläre Transformation und Immortalisierung. Folglich muss der signalweg, der mit 661W-Zellen identifiziert wurde, in den Kontext einer transformierten und immortalisierten Zelllinie gemeldet werden, die sich in vielerlei Hinsicht von Zapfen in situ unterscheidet. In dieser Hinsicht besteht das mit Dem Zapfen angereicherte Kultursystem aus primären Neuronen, den Zapfen, die physiologisch relevanter sind.
Während es möglich ist, eine reine Kultur von Photorezeptoren durch Vibratomschnitt der Netzhaut der Maus zu erhalten, macht der sehr geringe Prozentsatz an Zapfen in der äußeren Netzhaut von Nagetieren diesen Ansatz ungeeignet für die Herstellung von kegelangereicherten Kulturen29. Die Schweine-Netzhaut enthält keine Fovea, sondern hat eine Region namens Area Centralis, die sehr reich an Zapfen30ist. Der hohe Anteil an Zapfen in den Netzhaut-Tagelnagetieren, wie Arvicanthis ansorgei und Psammomys obsesus31,32, bietet eine mögliche Lösung, erfordert aber die Zucht solcher exotischen Arten. Erwachsene Schweineaugen, die von lokalen Schlachthöfen gesammelt wurden, können verwendet werden, um eine Mischkultur von Stäbchen und Zapfen herzustellen, die zur Untersuchung des Überlebens von Photorezeptoren verwendet wurden33. Eine elegante Lösung besteht darin, Zapfen aus der Schweinehaut durch Schwenken mit Erdnussagglutinin (PNA) -Lektin vorzureinigen, das selektiv an Zapfen bindet34. Dennoch ist diese Methode aufgrund ihrer Komplexität schwer in großem Maßstab umzusetzen.
Human induced pluripotent stem cells (iPS) bietet den vielversprechendsten Ansatz, um eine Zapfen-Photorezeptorzellpopulation zu erhalten, die für die Netzhauttransplantation verwendet werden kann, aber auch an zapfenangereicherte Kultur angepasst werden kann35,36. Da für Stab-Photorezeptoren37der Transkriptionsfaktor NRL benötigt wird, hat die Nrl-/- Maus eine Netzhaut, die von kurzwelligen Zapfen (S-Zapfen) dominiert wird. Die Inaktivierung könnte verwendet werden, um S-Kegel-angereicherte Präparate durch Differenzierung des Menschen von iPS38,39herzustellen. Ein weiterer möglicher Ansatz ist die Förderung der Zapfendifferenzierung mit Schilddrüsenhormonsignalisierung40. Während neuartige Methoden zur Herstellung kegelangereicherter Kulturen aus menschlichem iPS entstehen, bieten Hühnerembryonen eine derzeit bewährte Methode19.
Die mit Dem Zapfen angereicherte Kultur war maßgeblich an der Identifizierung von RdCVF durch Expression Cloning19 beteiligt. Dieses System wurde auch erfolgreich eingesetzt, um zu zeigen, dass RdCVF die Glukoseaufnahme und ihren Stoffwechsel durch aerobe Glykolyse stimuliert22. Darüber hinaus wurde eine mit Kegeln angereicherte Kultur verwendet, um die schützende Rolle von RdCVFL, dem zweiten Produkt des NXNL1-Gens 23,zu validieren. In jüngerer Zeit wurde dieses System verwendet, um die Existenz von schützenden Molekülen zu demonstrieren, die von retinalen pigmentierten Epithelzellen abgesondert werden, die mit OTX241transduziert wurden.
Protokoll
Das Protokoll wurde vom Komitee für die Ethik der Tierversuche der Universität Pierre und Marie Curie und dem französischen Forschungsministerium genehmigt (Genehmigungsnummer: APAFIS#1028 2015070211275177). Die Tierversuche wurden unter folgender Genehmigung durchgeführt: "Certificat d'autorisation d'expérimenter sur les animaux vertébrés A-75-1863. Préfecture de Police de Paris (9. November 2011 - 8. November 2016)".
1. Inkubation befruchteter Eier
- Sammeln Sie wöchentlich befruchtete Eier (Stamm I 657, rotes Etikett), die auf natürliche Weise in einer industriellen Brüterei gewonnen werden.
- Halten Sie die befruchteten Eier bei 17 ° C (ihre biologische Null) im Labor, nachdem sie von der Henne "gelegt" wurden.
- Für jede Kultur sieben befruchtete Eier für 24 Stunden bei 20 ° C und dann 136 Stunden bei 37 ° C mit intermittierender Umkehrung der Neigung (eine progressive Bewegung für 2 Stunden von einer Seite zu ihrem Gegenteil, ein 4-Stunden-Zyklus) der Eier in einer befeuchteten Kammer.
2. Rückgewinnung der Hühnerembryonen
- Waschen Sie die Oberfläche der sieben Eier mit Desinfektionsmittel (z. B. Pursept A express).
- Um die Eierschale zu brechen, machen Sie ein Loch in der Oberseite der Schale mit einer großen geraden Zange. Dann schneiden Sie die Schale, um den Hut als weich gekochtes Ei aus dem Ei zu entfernen.
- Extrahieren Sie vorsichtig jeden Embryo aus der Eierschale mit einer gekrümmten Zinnen und geben Sie ihn dann in eine Petrischale mit steriler Phosphatpuffersalzlösung (PBS), die zuvor auf 37 °C erhitzt wurde. Entfernen Sie vorsichtig die Hülle, die die Embryonen umgibt (Chorion oder Chorioallanto-Membran).
- Überprüfen Sie das Entwicklungsstadium jedes Embryos durch visuellen Vergleich mit Hamburger und Hamilton42.
- Wählen Sie zwei Embryonen im 29. Entwicklungsstadium aus (Abbildung 1). Die Flügel beugen sich an den Ellbogen. Der Kragen sticht sichtbar hervor. Der Gesetzentwurf ist prominenter als in der 28. Phase.
- Die Augen dieser ausgewählten Embryonen zu enukleieren und in CO2-unabhängigesMedium zu übertragen (Life-Technologien).
ACHTUNG: Es ist sehr wichtig, dass sich der Embryo im Stadium 29 und nicht im Stadium 28 oder 30 befindet (Abbildung 1); Deshalb ist es notwendig, mindestens 7 Eier zu inkubieren, auch wenn nur zwei schließlich verwendet werden. Der Chorion ist sehr dünn, so schwer zu unterscheiden, aber er ist sehr nah am Embryo, so dass er entfernt werden muss, ohne den Embryo zu berühren.
3. Dissektion der Netzhaut
- Enthaupten und entkernen Sie die ausgewählten Embryonen mit einer gekrümmten Zette.
- Übertragen Sie die vier Augen in ein CO2-unabhängigesMedium. Dieses Medium enthält 0,9 mMCaCl2 und 0,65 mMMgCl2.
- Positionieren Sie das Auge mit der Hornhaut nach unten, dem Sehnerv gegenüber dem Experimentator. Bohren Sie mit zwei geraden Zetten ein Loch in den Sehnerv.
- Setzen Sie einen Ast jeder Zette zwischen die Netzhaut und das Pigmentepithel ein (Abbildung 2). Ziehen Sie an jeder Zette und drehen Sie das Auge, um das Epithel von der Netzhaut zu lösen. Entfernen Sie die Hornhaut, gefolgt von der Linse und dem Glaskörper.
- Übertragen Sie die vier Netzhäute in eine Petrischale, die Ringers Medium bei pH 7,2 enthält.
VORSICHT: Stellen Sie sicher, dass nur die Netzhaut übrig bleibt und entfernen Sie alle Spuren von Glaskörper und verbleibendem retinalem Pigmentepithel.
4. Vorbereitung der Netzhautzellsuspension
- Schneiden Sie die vier Netzhäute mit zwei geraden Zangen in sehr kleine Stücke.
- Waschen Sie die Netzhautstücke zweimal mit Ringers Medium.
- Nach dem zweiten Waschen mit dem Medium von Ringer die Netzhautstücke auf den Boden der Röhre fallen lassen und das Medium des Ringers entfernen. Die Netzhautstücke 20 Minuten lang bei 37 °C mit einer Trypsinlösung (0,25% w/v) behandeln.
- Verteilen Sie die Lösung nach 10 Minuten durch sukzessives Absaugen. Entladung mit einer Pasteur-Pipette und Überprüfung auf Dissoziation der Netzhautstücke. Stoppen Sie die Reaktion, indem Sie Nährmedien hinzufügen, die mit 10% inaktiviertem Fötalkalbserum ergänzt sind.
- Inkubieren Sie die Zellsuspension mit 0,05 mg DNase I. Dissoziieren Sie die Zellcluster und die DNA durch sukzessives Absaugen und Entladen mit einer Pasteur-Pipette unmittelbar nach Zugabe der DNase.
- Waschen Sie die Netzhautzellsuspension zweimal mit einem chemisch definierten Kulturmedium (CDCM): ein gleiches Volumen von Dulbeccos Modified Eagle Medium und M199-Medien, ergänzt mit 100 μg/ml Linolsäure/BSA, 0,86 μM Insulin, 0,07 μM Transferrin, 2,0 μM Progesteron, 0,28 μM Prostaglandin, 0,29 μM Na2SeO3,182 μM Putrescin, 3 mM Taurin, 4,7 μM Cytidin 5′-Diphosphocholin, 2,7 μM Cytidin 5′-Diphosphoethanolamin, 0,55 μM Hydrocortison, 0,03 μM Triiodothyronin, 1 mM Natriumpyruvat und 20 μM Gentamycin.
5. Netzhautzellaussaat
- Zwei schwarze 96-Well-Kulturplatten mit transparentem Boden für 2 Stunden bei 37 °C mit Poly-L-Lysin bei 32,25 μg/cm2behandeln.
- Spülen Sie diese Platten zweimal mit dem M199-Kulturmedium aus. Das Zellpellet wird in 1 ml CDCM resuspendiert.
- Zu einem Aliquot von 10 μL der Zellsuspension Trypanblau hinzufügen, um die lebenden Zellen zu färben. Die Zellsuspensionsprobe wird in ein Hämozytometer (Zellzählkammer von Malassez) gegeben.
- Zählen Sie unter einem Mikroskop die gefärbten Zellen für vier Reihen des Hämozytometers (dh 40 Quadrate) und berechnen Sie dann die durchschnittliche Zellzahl für eine Zeile. Berechnen Sie die Konzentration der Zellen der Suspension nach folgender Methode: Anzahl der Zellen/ml Suspension = durchschnittliche Anzahl der Zellen in einer Reihe (10 Quadrate) x 10 die Gesamtzahl der Quadrate des Hämozytometers x Verdünnung mit Trypanblau x 1.000 (um das Ergebnis als Zellen/ml auszudrücken).
- Bringen Sie die Zellsuspension auf zwei Konzentrationen (5,6 x10 4 Zellen/ml und 1,12 x10 5 Zellen/ml), die den beiden Beschichtungsdichten (1 x 105 Zellen/cm2 und2 x 105 Zellen/cm2) entsprechen, mit CDCM.
- 50 μL der beiden Zellsuspensionen in die beiden vorbehandelten schwarzen 96-Well-Kulturplatten einsäen. Verteilen Sie die Zellen in den Platten mit einer Mehrkanalpipette von rechts von der Platte nach links und homogenisieren Sie zwischen jeder Säule, so dass die Verteilung der Zellen homogen ist.
- Fügen Sie 50 μL der Molekülbibliothek hinzu (z. B. die konditionierten Medien aus einer cDNA-Bibliothek, siehe unten), um mit einem vordefinierten Muster gescreent zu werden(Tabelle 1).
- Die Platten sieben Tage lang bei 37°C unter 5% CO2 ohne Medienwechsel inkubieren.
6. Zählen lebensfähiger Zellen
- Zu jeder Vertiefung der Platte werden 2,7 μM Calcein AM und 0,3 mM Ethidiumhomdimer hinzugefügt.
HINWEIS: Calcein dringt als Ethidiumhomdimer in die Zellen ein, die für große Moleküle undurchlässig sind. Im Zytoplasma lebender Zellen wird Calcein durch endogene Esterasen hydrolysiert, wird fluoreszierend, indem es bei 520 nm emittiert, wenn es bei 485 nm angeregt wird. Ethidium-Homodimer bindet an DNA auf toten Zellen. Die Ethidium-DNA-Homodimer-Bindung bewirkt, dass rote Fluoreszenz bei 635 nm nach Anregung bei 520 nm emittiert wird. - Inkubieren Sie die Platten für 1 Stunde bei Raumtemperatur in Abwesenheit von Licht.
- Lesen Sie die Fluoreszenz auf einem automatisierten Plattenleser, der aus einem invertierten Mikroskop besteht, das mit einer Quecksilberlampe mit zwei Anregungsfiltern bei 485 und 520 nm, zwei Emissionsfiltern bei 520 und 635 nm, einem Objektiv (x10), einer motorisierten Stufe, die von einem Prozessor gesteuert wird, und einer ladungsgekoppelten Gerätekamera (CCD) besteht. Diese Zählplattform wird von der Metamorph-Software19gesteuert. Es ermöglicht die gleichzeitige Erfassung von Fluoreszenzbildern lebender Zellen und toter Zellen in jeder Vertiefung der 96-Well-Platte, beginnend mit den Vertiefungen A1 in Richtung Bohrplatz A12, dann B12 in Richtung B1, C1 in Richtung C12 und so weiter (Tabelle I).
- Berechnen Sie die durchschnittliche Fläche A einer einzelnen Zelle anhand der 18 Vertiefungen der Negativkontrollen (Tabelle I).
- Zählen Sie die Zellen in jeder Vertiefung der Platte und wenden Sie die folgende Summenformel A x 29 / 20,7 an, um zu verhindern, dass Zelldoppel (Gruppierung von zwei Zellen) gezählt werden. Bewerten Sie die schützende Wirkung der Zapfen durch Moleküle als verhältnis zwischen der durchschnittlichen Zellzahl in den 4 Vertiefungen, in denen wir das Molekül getestet haben, und der durchschnittlichen Zellzahl in den 18 Vertiefungen der Negativkontrolle (siehe Tabelle 1 und ergänzende Abbildung 1).
- Kombinieren Sie die Ergebnisse der Platte, die mit 1 x 105 Zellen/cm2 ausgesät ist, mit der Platte, die mit 2 x 105 Zellen/cm2 gesät wurde, um den potenziellen Schutz (Lebensfähigkeitsverhältnis) durch jedes gescreente Molekül zu bewerten.
Ergebnisse
Wir beschreiben hier, wie das mit Zapfen angereicherte Kultursystem genutzt werden kann, um neuartige zapfenschützende Proteine zu identifizieren. Wir verwendeten dieses Protokoll, um eine normalisierte cDNA-Bibliothek aus Aderhaut- und Netzhautpigmentepithel aus 400 Augen von 8 Wochen alten Long-Evans-Ratten43zu screenen.
Diese Bibliothek enthält 6,0 x10 6 unabhängige Kolonien, die Einheiten bilden (CFUs) und hat eine durchschnittliche klonierte Einfügegröße von 2,1 Kilobase (kb), wobei mehr als 99% der rekombinanten Klone vorhanden sind. Pools von 100 Klonen aus dieser Bibliothek wurden vorübergehend in COS-1-Zellen transfiziert (0,1 μg Plasmid-DNA) und das konditionierte Medium (CM) von COS-1 wurde nach Inkubation für 48 Stunden in DMEM ohne Serum geerntet. Membranen oder Exosomen wurden nicht durch Ultrazentrifugation entfernt. Fünfzig Mikroliter von jedem CM wurden zu 4 Vertiefungen von zwei 96-Well-Platten hinzugefügt: eine mit 2 x 105 Zellen/cm2, die andere mit 4 x 105 Zellen/cm2 . CM aus COS-1-Zellen, die mit dem leeren Vektor pcDNA3.1 transfiziert wurden, der zum Aufbau der Bibliothek verwendet wurde, wurde als Negativkontrolle verwendet (Tabelle 1). Insgesamt wurden 2.112 Sätze von 100 Klonen, die 211.200 einzelnen Klonen entsprechen, in vier Kulturbrunnen und für zwei Aussaatbedingungen von kegelangereicherten Kulturen ausgewertet. Die beiden Bedingungen entsprechen zwei leicht unterschiedlichen Impfdichten, die es ermöglichen, die Schutzaktivität für insgesamt 1.689.600 Kulturbrunnen genauer zu beurteilen.
Unter den 42 Pools von Klonen mit einem Verhältnis größer als 2 haben die Pools 0080 und 0073 ein Lebensfähigkeitsverhältnis von 16 und 14 mal höher nach 7 Tagen Kultur als die Negativkontrolle, pcDNA3.1 (Ergänzende Abbildung 1). Diese Analyse ist unerlässlich, um die Pools von Interesse zu identifizieren. Jeder ausgewählte Pool von 100 Klonen wurde in 16 Sätze von 10 Klonen aus ihrem Glycerinbestand unterteilt. Diese Teilpools wurden nach der gleichen Methode in einer zweiten Screening-Runde (d.h. insgesamt 3.200 Kulturbrunnen) vorbereitet und getestet. Der Teilpool 0073-09 ergab die stärkste Lebensfähigkeitsquote (Ergänzende Abbildung 2A) und wurde unterteilt, um 16 einzelne Klone zu produzieren, die in einer dritten Runde des Screenings an kegelangereicherten Kulturen getestet wurden. Der Klon 0073-09-37 hebt sich mit einem Lebensfähigkeitsverhältnis von 2,5 deutlich von den anderen ab (Ergänzende Abbildung 2B). Die y-Achse hat eine andere Skala, auch wenn die Aussaatdichte die gleiche war wie in der ergänzenden Abbildung 2A. Wir haben dies häufig gesehen, wenn die Assays monatelang wöchentlich wiederholt werden. Nach der Analyse bestätigen diese Ergebnisse, dass der Klon 0073-09-37 eine robuste und reproduzierbare Wirkung auf das Überleben des Kegels hat. Der Test wurde unabhängig voneinander wiederholt (Abbildung 3A), und der Einsatz von 1,8 kb wurde sequenziert (Abbildung 3B).
Eine bioinformatische Analyse ergab, dass der Klon 0073-09-37, den wir Epithel-derived cone viability factor (EdCVF) nannten, drei offene Leserahmen (ORFs) enthält, von denen einer der am weitesten vorgelagerten (ORF1) für 84 Rückstände des C-terminalen Teils des Rattenproteins Zinkfingerprotein-180 (ZFP180, NP_653358) von 727 Aminosäurenkodiert 44. Die anderen beiden ORFs (ORF2 und ORF3) sind bei Mäusen viel weniger gut konserviert und fehlen bei anderen Säugetieren. Bei unabhängiger Prüfung übt nur ORF1 eine schützende Wirkung auf die Zapfen aus (Ergänzende Abbildung 3). ORF1 wurde als Glutathion-S-Transferase (GST)-Fusionsprotein hergestellt (Abbildung 4A). Das EdCVF-Protein wurde gereinigt und das GST-Tag entfernt (Abbildung 4B). EdCVF ist in der Lage, Diekendegeneration im kegelangereicherten Kultursystem zu verhindern (Abbildung 4C).
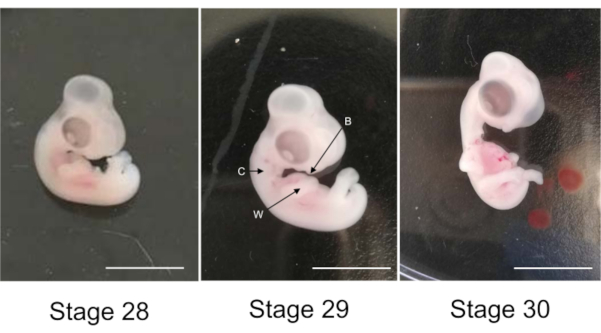
Abbildung 1: Hühnerembryonen in den Stadien28,29 und 30 der Entwicklung. Pfeile W: Flügel, C: Halsband und B: Schnabel. Maßstabsleiste 1 cm. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
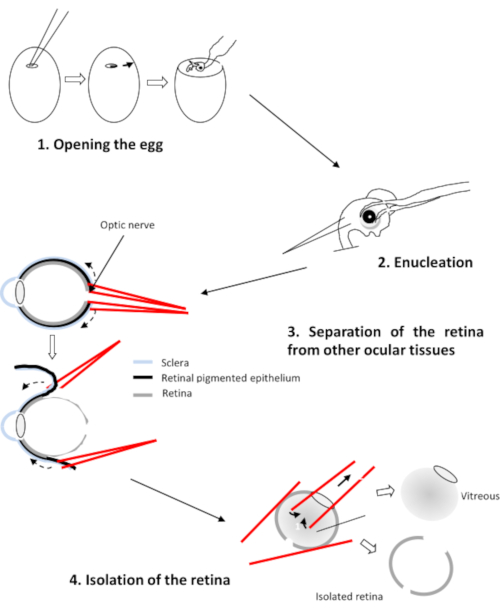
Abbildung 2: Dissektion der Netzhaut des Hühnerembros Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
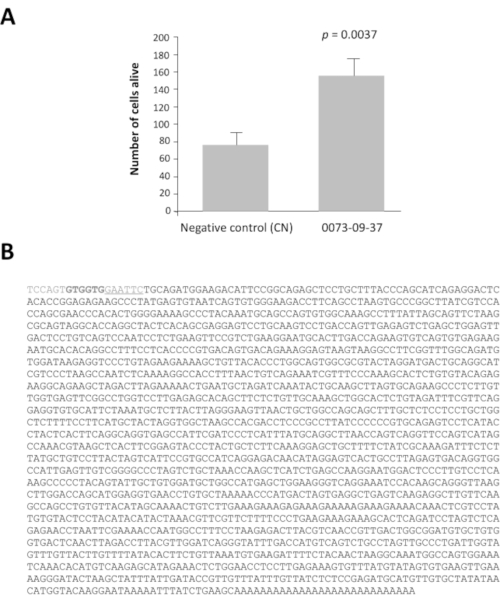
Abbildung 3: Epithel-abgeleiteter Zapfenlebensfähigkeitsfaktor (EdCVF), Klon 0073-09-37. A. (A). Erhöhte Lebensfähigkeit auf kegelangereicherter Kultur. B. Die Sequenz des cDNA-Klons 0073-09-37. Die unterstrichene Sequenz GAATTC ist die EcoRI-Einschränkungsseite, die zum Erstellen der Bibliothek verwendet wird. Die beiden Codons GTG in Fettdruck sind ungewöhnliche Übersetzungsinitiierungsseiten für EdCVF, die aus dem Vektor stammen. Statistische Analyse durch Schülertest. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
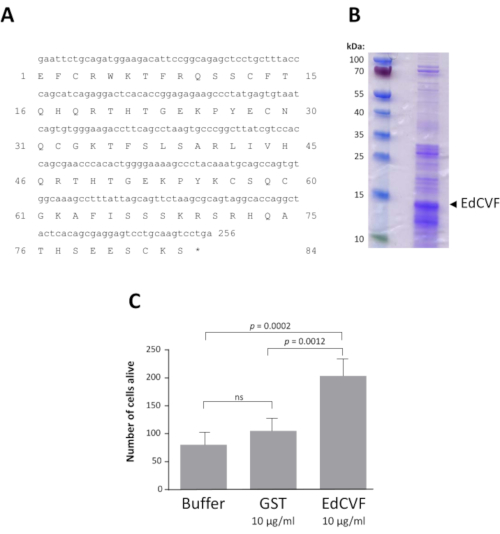
Abbildung 4: Rekombinante EdCVF-Aktivität. A. Sequenz von EdCVF in der Fusion mit Glutathion-S-Transferase (GST). B. Das gereinigte rekombinante EdCVF-Protein. C. (EN) Trophische Aktivität von GST-EdCVF auf Zapfen in Kultur. Statistische Analyse mit Tukeys Test. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung 1: Verhältnis der durchschnittlichen Zellzahl für einen cDNA-Pool und der Negativkontrolle, dem konditionierten Medium von COS-1-Zellen, die während der ersten Screening-Runde mit dem leeren Vektor pcDNA3.1 transfiziert wurden. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzend Abbildung 2: Anzahl der lebenden Zellen. A. (A). Die zweite Runde des Screenings mit Unterpools von 0073 Pool. B. Die dritte Runde des Screenings mit isolierten Klonen. CN: das konditionierte Medium von COS-1-Zellen, transfiziert mit dem leeren Vektor pcDNA3.1. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 3: Trophische Aktivität der drei offenen Leserahmen des isolierten Klons 0073-09-37. Statistische Analyse mit Dunnetts Test. Bitte klicken Sie hier, um diese Datei herunterzuladen.
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | |
| a | 1 | 1 | 1 | 1 | 2 | NC | 2 | 2 | 2 | 3 | 3 | NC |
| H | NC | 3 | 3 | 4 | 4 | 4 | NC | 4 | 5 | 5 | 5 | 5 |
| C | 6 | NC | 6 | 6 | 6 | 7 | 7 | NC | 7 | 7 | 8 | 8 |
| D | 8 | 8 | NC | 9 | 9 | 9 | 9 | 10 | NC | 10 | 10 | 10 |
| E | 11 | 11 | 11 | NC | 11 | 12 | 12 | 12 | 12 | NC | 13 | 13 |
| f | 13 | 13 | 14 | 14 | NC | 14 | 14 | 15 | 15 | 15 | NC | 15 |
| G | 16 | 16 | 16 | 16 | 17 | NC | 17 | 17 | 17 | 18 | 18 | NC |
| h | 18 | 18 | 19 | 19 | 19 | 19 | NC | 20 | 20 | 20 | NC | 20 |
| NC-Negativkontrollen | ||||||||||||
| 1-20 Positionen der getesteten Pools in vierfacher Ausführung | ||||||||||||
Tabelle 1: Plan der 96-Well-Platte für das Screening mit hohem Gehalt
Diskussion
Unter den vielen Parametern, die die Produktion einer mit Kegeln angereicherten Kultur aus Hühnerembryonen einschränken könnten, besteht der erste kritische Schritt darin, das Entwicklungsstadium der Embryonen in den geschlüpften Eiern genau zu identifizieren. Es wurde beobachtet, dass die Kultur von Zellen aus der Netzhaut von Embryonen bei ED8 (34. Stadium) nur 35% Photorezeptoren produziert, wobei die restlichen 65% aus anderen Neuronen bestehen4. Unabhängig von der Logistik, die angewendet wird, um die geschlüpften Eier zu erhalten, ist es notwendig, die Temperatur und die Inkubationszeit zu verfeinern und die Embryonen im Vergleich zu den Referenzbildern aller Entwicklungsstadien sorgfältig zu untersuchen42,45.
Ursprünglich wurde das kegelangereicherte Kultursystem mit der White Leghorn Sorte4entwickelt. Die weiße Farbe der Eier dieser Sorte wird in Frankreich nicht besonders geschätzt, daher haben wir eine Hühnersorte verwendet, die braune Eier produziert. Wir haben die I 657-Sorte verwendet, die durch Kreuzung von I 66 Hähnen mit JA57 Hühnern5hergestellt wird. Wir konnten die Eigenschaften der ursprünglichen Kulturen reproduzieren. Dies zeigt, dass der genetische Hintergrund des Huhns nicht entscheidend ist, um mit Zapfen angereicherte Kulturen zu erhalten.
Wir haben die Wirkung der individuellen Entfernung der Nahrungsergänzungsmittel im Kulturmedium nicht getestet, sondern beobachtet, dass Insulin eine entscheidende Rolle in Übereinstimmung mit der Wirkung von Insulin auf das Überleben von Zapfen in der rd1-Maus spielt, einem Modell der autosomal-rezessiven RP46. Trijodthyronin (T3) kann auch an der Differenzierung von netztinalen Vorläuferzellen des Hühnerembros in Zapfen entsprechend der Rolle des Schilddrüsenhormonrezeptors beim Schicksal von Netzhautzellen während der Entwicklung beteiligt sein40. Folglich kann das mit Zapfen angereicherte Kultursystem nicht verwendet werden, um Insulin durch Expressionsklonen zu identifizieren46.
Das mit Zapfen angereicherte Kultursystem beruht auf der Kultur primärer Neuronen und ist viel geeigneter als Methoden, die auf der Verwendung von immortalisierten Zellen wie der Zelllinie 661W24,25,26beruhen.
Das hier beschriebene Verfahren kann durch eine vorherige Elektroporation mit Plasmid DNA20modifiziert werden. Vor der Herstellung der Netzhautzellsuspension wird die gesamte Netzhaut in die Kammer eines maßgeschneiderten Elektroporators mit 120 μL 0,5 μg/μL Plasmid-DNA in 10 mM Tris-HCl pH 8,0, 1 mM EDTA gelegt. Fünf Impulse von 15 V für jeweils 50 ms werden durch 950 ms Intervall22getrennt angewendet. Versuche, störende RNA (RNAi) mit hilfe der Replikations-RCAS-Retroviren (Avian Splice) in zapfenangereicherte Kulturen zu liefern, waren erfolglos47. Dies ist sicherlich darauf zurückzuführen, dass in Abwesenheit von Serum und in Kulturen geringer Dichte retinale Vorläuferzellen nicht replizierend sind, eine Voraussetzung für die Vermehrung von Retroviren.
Wir haben das kegelangereicherte Kultursystem entwickelt, um trophische Faktoren zu identifizieren, die das Überleben von Kegeln mit Hilfe des Expressionsklonens fördern19. Um dies möglich zu machen, haben wir einen ersten Schritt des High-Content-Screenings mit konditioniertem Medium aus Pools von 100 Klonen durchgeführt. Selbst wenn die cDNAs aus der Bibliothek nach der Transfektion von COS-1-Zellen unter der Kontrolle eines starken CMV-Promotors exprimiert werden, bietet dies keine Garantie, dass alle von einzelnen cDNAs kodierten Proteine eine Konzentration erreichen, die ausreicht, um durch den Viability-Assay positiv bewertet zu werden. Dies ist eine große Einschränkung. In diesem Sinne ist jedes Screening nicht wirklich erschöpfend. Selbst wenn membranöse Proteine nicht aus dem konditionierten Medium entfernt wurden, ist die Konfiguration des Assays für die Identifizierung nicht diffusiver Faktoren ungünstig. Eine Alternative wäre, einzelne Klone nach Erhalt der Sequenz der cDNAs zu screenen, um Duplikationen bei der Analyse desselben Kandidatenproteins zu vermeiden. Dies wurde durch die Sequenzierung der retinalen cDNA-Bibliotheken initiiert, die wir verwendet haben43. Dieser Ansatz ist zwar rational, hat aber auch seine Grenzen. Die bioinformatische Analyse der cDNA-Sequenzen wird neben der Reduzierung der Redundanz unwiderstehlich die Priorisierung des Screenings bestimmter Klone auf der Grundlage von Wissen erzwingen. Dies wird nicht nachteilig sein, wenn schließlich die gesamte Bibliothek überprüft würde, auch wenn die dafür erforderliche Zeit erheblich verlängert wird. Aber ausnahmslos wird die Identität der Sequenz unsere Art und Weise beeinflussen, die Ergebnisse zu betrachten. Dies wird nicht neutral sein, da die Interpretation der Sequenz natürlich in Konkurrenz zu den experimentellen Daten stehen wird.
Die Identifizierung von EdCVF zeigt auch, dass High Content Screening technische Einschränkungen mit sich bringt. Aus der ersten Screening-Runde identifizierten wir zwei Pools mit hoher Aktivität (Ergänzende Abbildung 1). Der Pool 0073 führte zur erfolgreichen Identifizierung von EdCVF, während Pool 0080 nicht zu einem solchen Befund führte. Wir haben das Problem, das sich aus dem Verlust des aktiven Klons während der Vorbereitung von Sub-Pools ergeben könnte, nicht gelöst. Alternativ ist nicht ausgeschlossen, wenn auch nicht statistisch günstig, dass unter den cDNAs von Pool 0080 zwei Proteine synergistisch wirkten und ihre Aktivität nicht als einzelne Klone beobachtet werden konnte.
Die Identifizierung von Molekülen, die Zapfen schützen, durch Screening kleiner Moleküle ist eine zukünftige Anwendung des mit Zapfen angereicherten Kultursystems. Solche Moleküle werden für die Behandlung von Netzhautpathologien, für die die Gentherapie nicht der am besten geeignete Ansatz ist, von unschätzbarem Wert sein, da die altersbedingte Makuladegeneration nicht der Fall ist.
Offenlegungen
TL, J-AS und VF besitzen ein Patent auf die Verwendung von EdCVF zur Behandlung von Netzhautdegenerationen [WO2009071659 (A1). 12. Juni 2007].
Danksagungen
Die Autoren danken Jacques Bellalou, Lorelei Fournier, Emmanuelle Clérin, Frédéric Blond und dem Unternehmen agricole à responsabilité limitée (EARL Morizeau Dangers, Frankreich) für ihre unschätzbare Hilfe. Diese Arbeit wurde von Inserm, der Universität Sorbonne, der Agence Nationale pour la Recherche (ANR, Labex Lifesenses), der Stiftung Fighting Blindness (USA) und der IHU FOReSIGHT [ANR-18-IAHU-0001] unterstützt, unterstützt durch französische Staatsmittel, die von der ANR im Rahmen des Programms Investissements d'Avenir verwaltet werden.
Materialien
| Name | Company | Catalog Number | Comments |
| 96 black plates (Clear Button with lid Tissue culture treated) | Corning | 3603 | |
| Calcein AM | Thermo-Fisher scientific | C1430 | |
| CCD Camera | Photometrics | CoolSnap FX HQ | |
| CDPC (Cytidine 5′-diphosphocholine sodium salt dihydrate) | Sigma-Aldrich | C0256 | |
| CO2 Independant | Thermo-Fisher scientific | 18045-054 | |
| Curved forceps | Dutscher | 005093 | |
| DMEM Media | Thermo-Fisher scientific | 41966-029 | |
| DNAse | Sigma-Aldrich | D4263 | |
| Eggs incubator | FarmLine | M08 01 3100 | |
| Ethidium Homodimer | Thermo-Fisher scientific | E1169 | |
| Fœtal bovine serum | Thermo-Fisher scientific | 10270-098 | |
| Gentamycin | Thermo-Fisher scientific | 15710-049 | |
| Hydrocortisone | Sigma-Aldrich | H0880 | |
| ITS (insulin Transferine selenium) | Sigma-Aldrich | I1884 | |
| large straight pliers | Dutscher | 005074 | |
| Linoleic acid | Sigma-Aldrich | L8384 | |
| M199 medium | Thermo-Fisher scientific | 31150-022 | |
| Metamorph software | Metamorph | ||
| Microscope | NIKON | Eclipse TE2000 | |
| Motorized stage | Martzauzer | Mutlicontrol 2000 | |
| Optical filter switch | Shutter Instrument company | Lambda 10-2 | |
| PBS 1X | Thermo-Fisher scientific | 14190-086 | |
| Poly-L-lysine | Sigma-Aldrich | P6282 | |
| Progesterone | Sigma-Aldrich | P7556 | |
| Pursept A express | Fisher scientific | 11814110 | |
| Putriscine | Sigma-Aldrich | P5780 | |
| Sodium pyruvate | Sigma-Aldrich | S8636 | |
| straight forceps | Dutscher | 005092 | |
| Taurine | Sigma-Aldrich | T8691 | |
| Triiodothyronine | Sigma-Aldrich | T6397 | |
| Trypan blue | Thermo-Fisher scientific | 15250-061 | |
| Trypsine 0.25 % | Thermo-Fisher scientific | 25200-056 |
Referenzen
- Brownstein, C. D. . The Implicit Mind. , (2019).
- Heesy, C. P., Hall, M. I. The nocturnal bottleneck and the evolution of mammalian vision. Brain, Behavior and Evolution. 75 (3), 195-203 (2010).
- Borges, R., et al. Adaptive genomic evolution of opsins reveals that early mammals flourished in nocturnal environments. BMC Genomics. 19 (1), 121 (2018).
- Adler, R., Hatlee, M. Plasticity and differentiation of embryonic retinal cells after terminal mitosis. Science. 243 (4889), 391-393 (1989).
- Fintz, A. C., et al. Partial characterization of retina-derived cone neuroprotection in two culture models of photoreceptor degeneration. Investigative Ophthalmology & Visual Science. 44 (2), 818-825 (2003).
- Roska, B., Sahel, J. A. Restoring vision. Nature. 557 (7705), 359-367 (2018).
- Duncan, J. L., et al. Inherited Retinal Degenerations: Current Landscape and Knowledge Gaps. Translational Vision Science & Technology. 7 (4), 6 (2018).
- Russell, S., et al. Efficacy and safety of voretigene neparvovec (AAV2-hRPE65v2) in patients with RPE65-mediated inherited retinal dystrophy: a randomised, controlled, open-label, phase 3 trial. Lancet. 390 (10097), 849-860 (2017).
- RetNet. . Retinal Information Network. , (2020).
- Doonan, F., Donovan, M., Cotter, T. G. Activation of multiple pathways during photoreceptor apoptosis in the rd mouse. Investigative Ophthalmology & Visual Science. 46 (10), 3530-3538 (2005).
- Comitato, A., et al. Dominant and recessive mutations in rhodopsin activate different cell death pathways. Human Molecular Genetics. 25 (13), 2801-2812 (2016).
- Cronin, T., Leveillard, T., Sahel, J. A. Retinal degenerations: from cell signaling to cell therapy; pre-clinical and clinical issues. Current Gene Therapy. 7 (2), 121-129 (2007).
- Baumgartner, W. A., Baumgartner, A. M. Accounting for disagreements on average cone loss rates in retinitis pigmentosa with a new kinetic model: Its relevance for clinical trials. Medical Hypotheses. 89, 107-114 (2016).
- Leveillard, T., Sahel, J. A. Rod-derived cone viability factor for treating blinding diseases: from clinic to redox signaling. Science Translational Medicine. 2 (26), (2010).
- Leveillard, T., Sahel, J. A. Metabolic and redox signaling in the retina. Cellular and Molecular Life Sciences. 74 (20), 3649-3665 (2017).
- Clerin, E., Marussig, M., Sahel, J. A., Leveillard, T. Metabolic and Redox Signaling of the Nucleoredoxin-Like-1 Gene for the Treatment of Genetic Retinal Diseases. International Journal of Molecular Sciences. 21 (5), (2020).
- Fridlich, R., et al. The Thioredoxin-like Protein Rod-derived Cone Viability Factor (RdCVFL) Interacts with TAU and Inhibits Its Phosphorylation in the Retina. Molecular & Cellular Proteomics. 8 (6), 1206-1218 (2009).
- Mei, X., et al. The Thioredoxin Encoded by the Rod-Derived Cone Viability Factor Gene Protects Cone Photoreceptors Against Oxidative Stress. Antioxidants, Redox Signaling. 24 (16), 909-923 (2016).
- Leveillard, T., et al. Identification and characterization of rod-derived cone viability factor. Nature Genetics. 36 (7), 755-759 (2004).
- Vergara, M. N., Gutierrez, C., Canto-Soler, M. V. Efficient Gene Transfer in Chick Retinas for Primary Cell Culture Studies: An Ex-ovo Electroporation Approach. Journal of Visualized Experiments. (105), e52002 (2015).
- Fridlich, R., et al. The thioredoxin-like protein rod-derived cone viability factor (RdCVFL) interacts with TAU and inhibits its phosphorylation in the retina. Molecular & Cellular Proteomics. 8 (6), 1206-1218 (2009).
- Ait-Ali, N., et al. Rod-derived cone viability factor promotes cone survival by stimulating aerobic glycolysis. Cell. 161 (4), 817-832 (2015).
- Mei, X., et al. The Thioredoxin Encoded by the Rod-Derived Cone Viability Factor Gene Protects Cone Photoreceptors Against Oxidative Stress. Antioxidants & Redox Signaling. 24 (16), 909-923 (2016).
- Tan, E., et al. Expression of cone-photoreceptor-specific antigens in a cell line derived from retinal tumors in transgenic mice. Investigative Ophthalmology & Visual Science. 45 (3), 764-768 (2004).
- Wang, X. W., Tan, B. Z., Sun, M., Ho, B., Ding, J. L. Thioredoxin-like 6 protects retinal cell line from photooxidative damage by upregulating NF-kappaB activity. Free Radical Biology and Medicine. 45 (3), 336-344 (2008).
- Perron, N. R., Beeson, C., Rohrer, B. Early alterations in mitochondrial reserve capacity; a means to predict subsequent photoreceptor cell death. Journal of Bioenergetics and Biomembranes. 45 (1-2), 101-109 (2013).
- Krishnamoorthy, R. R., Clark, A. F., Daudt, D., Vishwanatha, J. K., Yorio, T. A forensic path to RGC-5 cell line identification: lessons learned. Investigative Ophthalmology & Visual Science. 54 (8), 5712-5719 (2013).
- Al-Ubaidi, M. R. RGC-5: are they really 661W? The saga continues. Experimental Eye Research. 119, 115 (2014).
- Clerin, E., et al. Vibratome sectioning mouse retina to prepare photoreceptor cultures. Journal of Visualized Experiments. (94), (2014).
- Hendrickson, A., Hicks, D. Distribution and density of medium- and short-wavelength selective cones in the domestic pig retina. Experimental Eye Research. 74 (4), 435-444 (2002).
- Bobu, C., Craft, C. M., Masson-Pevet, M., Hicks, D. Photoreceptor organization and rhythmic phagocytosis in the nile rat Arvicanthis ansorgei: a novel diurnal rodent model for the study of cone pathophysiology. Investigative Ophthalmology & Visual Science. 47 (7), 3109-3118 (2006).
- Saidi, T., Mbarek, S., Chaouacha-Chekir, R. B., Hicks, D. Diurnal rodents as animal models of human central vision: characterisation of the retina of the sand rat Psammomys obsesus. Graefes Arch Clin Exp Ophthalmol. 249 (7), 1029-1037 (2011).
- Traverso, V., Kinkl, N., Grimm, L., Sahel, J., Hicks, D. Basic fibroblast and epidermal growth factors stimulate survival in adult porcine photoreceptor cell cultures. Investigative Ophthalmology & Visual Science. 44 (10), 4550-4558 (2003).
- Balse, E., et al. Purification of mammalian cone photoreceptors by lectin panning and the enhancement of their survival in glia-conditioned medium. Investigative Ophthalmology & Visual Science. 46 (1), 367-374 (2005).
- Gagliardi, G., et al. Characterization and Transplantation of CD73-Positive Photoreceptors Isolated from Human iPSC-Derived Retinal Organoids. Stem Cell Reports. 11 (3), 665-680 (2018).
- Gagliardi, G., Ben M'Barek, K., Goureau, O. Photoreceptor cell replacement in macular degeneration and retinitis pigmentosa: A pluripotent stem cell-based approach. Progress in Retinal and Eye Research. 71, 1-25 (2019).
- Mears, A. J., et al. Nrl is required for rod photoreceptor development. Nature Genetics. 29 (4), 447-452 (2001).
- Swaroop, A., Kim, D., Forrest, D. Transcriptional regulation of photoreceptor development and homeostasis in the mammalian retina. Nature Reviews Neuroscience. 11 (8), 563-576 (2010).
- Kallman, A., et al. Investigating cone photoreceptor development using patient-derived NRL null retinal organoids. Nature Communications. 3 (1), 82 (2020).
- Eldred, K. C., et al. Thyroid hormone signaling specifies cone subtypes in human retinal organoids. Science. 362 (6411), (2018).
- Kole, C., et al. Otx2-Genetically Modified Retinal Pigment Epithelial Cells Rescue Photoreceptors after Transplantation. Molecular Therapy. 26 (1), 219-237 (2018).
- Hamburger, V., Hamilton, H. L. A series of normal stages in the development of the chick embryo. Journal of Morphology. 88 (1), 49-92 (1951).
- Kole, C., et al. Identification of an Alternative Splicing Product of the Otx2 Gene Expressed in the Neural Retina and Retinal Pigmented Epithelial Cells. PLoS One. 11 (3), 0150758 (2016).
- Shannon, M., Hamilton, A. T., Gordon, L., Branscomb, E., Stubbs, L. Differential expansion of zinc-finger transcription factor loci in homologous human and mouse gene clusters. Genome Research. 13 (6), 1097-1110 (2003).
- Stern, C. D., Holland, P. W. . Essential developmental biology: a practical approach. , (1993).
- Punzo, C., Kornacker, K., Cepko, C. L. Stimulation of the insulin/mTOR pathway delays cone death in a mouse model of retinitis pigmentosa. Nature Neuroscience. 12 (1), 44-52 (2009).
- Harpavat, S., Cepko, C. L. RCAS-RNAi: a loss-of-function method for the developing chick retina. BMC Developmental Biology. 6, 2 (2006).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten