Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Synthese von Masarimycin, einem niedermolekularen Inhibitor des grampositiven Bakterienwachstums
In diesem Artikel
Zusammenfassung
Es wird ein detailliertes Protokoll zur Herstellung des bakteriostatischen Diamid Masarimycin vorgestellt, einer niedermolekularen Sonde, die das Wachstum von Bacillus subtilis und Streptococcus pneumoniae hemmt, indem sie auf den Abbau der Zellwand abzielt. Seine Anwendung als chemische Sonde wird in Synergie-/Antagonismus-Assays und morphologischen Studien mit B. subtilis und S. pneumoniae nachgewiesen.
Zusammenfassung
Peptidoglycan (PG) in der Zellwand von Bakterien ist eine einzigartige makromolekulare Struktur, die Form und Schutz vor der Umgebung verleiht. Zentral für das Verständnis des Zellwachstums und der Zellteilung ist das Wissen, wie der PG-Abbau die Biosynthese und den Zellwandaufbau beeinflusst. Kürzlich wurde über die metabolische Markierung von PG durch die Einführung von modifizierten Zuckern oder Aminosäuren berichtet. Während eine chemische Abfrage von Biosyntheseschritten mit niedermolekularen Inhibitoren möglich ist, sind chemisch-biologische Werkzeuge zur Untersuchung des PG-Abbaus durch Autolysine unterentwickelt. Bakterielle Autolysine sind eine breite Klasse von Enzymen, die am eng koordinierten Abbau von PG beteiligt sind. Hier wird ein detailliertes Protokoll zur Herstellung einer niedermolekularen Sonde, Masarimycin, einem Inhibitor von N-Acetylglucosaminidase LytG in Bacillus subtilis, und des Zellwandstoffwechsels in Streptococcus pneumoniae vorgestellt. Die Herstellung des Inhibitors über mikrowellengestützte und klassische organische Synthese ist vorgesehen. Seine Anwendbarkeit als Werkzeug zur Untersuchung der grampositiven Physiologie in biologischen Assays wird vorgestellt.
Einleitung
Peptidoglycan (PG) ist ein netzartiges Polymer, das die Zellform und -struktur sowohl in grampositiven als auch in gramnegativen Bakterien 1,2 beschreibt. Dieses Heteropolymer ist eine Matrix von Aminozuckern, vernetzt durch kurze Peptide 3,4,5,6 mit einem Rückgrat, das aus β-(1,4)-verknüpften alternierenden N-Acetylglucosamin- (GlcNAc) und N-Acetylmuraminsäure (MurNAc)-Resten besteht (Abbildung 1)1. An den C-3-Lactylanteil von MurNAc ist das Stammpeptid gebunden. Der Metabolismus von PG beinhaltet ein eng koordiniertes System von biosynthetischen und abbaubaren Enzymen, um neues Material in die Zellwand einzubauen 7,8. Der Abbau von PG erfolgt durch Enzyme, die zusammen als Autolysine9 bezeichnet und basierend auf der Spezifität der gespaltenen Bindung weiter klassifiziert werden. Autolysine nehmen an vielen zellulären Prozessen teil, einschließlich Zellwachstum, Zellteilung, Motilität, PG-Reifung, Chemotaxis, Proteinsekretion, genetischer Kompetenz, Differenzierung und Pathogenität10,11. Die Entschlüsselung der spezifischen biologischen Funktionen einzelner Autolysine kann entmutigend sein, was zum Teil auf funktionelle Redundanz zurückzuführen ist. Jüngste biophysikalische 8,12,13 und Computerstudien 12 haben jedoch neue Einblicke in ihre Rolle im PG-Stoffwechsel geliefert. Darüber hinaus haben jüngste Berichte weitere Einblicke in die Synthese14 und membranvermittelten 15,16,17 Schritte im PG-Metabolismus gegeben. Ein gründliches Verständnis der Beziehung zwischen abbauenden und synthetischen Signalwegen des PG-Stoffwechsels könnte zu bisher unerschlossenen Antibiotikazielen führen.
Während es signifikante Fortschritte in der Methodik zur Untersuchung der Glykobiologie bei Eukaryoten gegeben hat, ist die bakterielle Glykobiologie und insbesondere der PG-Stoffwechsel nicht mit einer ähnlichen Geschwindigkeit fortgeschritten. Aktuelle chemische Ansätze zur Untersuchung des PG-Stoffwechsels umfassen fluoreszenzmarkierte Antibiotika18, fluoreszierende Sonden 19,20 und metabolische Markierung 21,22,23,24. Diese neuen Ansätze bieten neue Möglichkeiten, den bakteriellen Zellwandstoffwechsel zu hinterfragen. Während einige dieser Strategien in der Lage sind, PG in vivo zu markieren, können sie artspezifisch19 sein oder nur in Stämmen funktionieren, denen ein bestimmtes Autolysin25 fehlt. Viele PG-Markierungsstrategien sind für die Verwendung mit isoliertenZellwänden 26 oder mit in vitro rekonstituierten PG-Biosynthesewegen20,27,28 vorgesehen. Der Einsatz fluoreszierend markierter Antibiotika beschränkt sich derzeit auf biosynthetische Schritte und Transpeptidation18.
Das aktuelle Wissen über bakterielle Autolysine und ihre Rolle im Zellwandstoffwechsel stammt aus genetischen und in vitro biochemischen Analysen 11,29,30,31,32. Während diese Ansätze eine Fülle von Informationen über diese wichtige Klasse von Enzymen geliefert haben, kann die Entschlüsselung ihrer biologischen Rolle eine Herausforderung sein. Zum Beispiel führt die Deletion eines Autolysins aufgrund der funktionellen Redundanz33 in den meisten Fällen nicht dazu, dass das Bakterienwachstum gestoppt wird. Dies ist trotz ihrer impliziten Rolle beim Zellwachstum und der Zellteilung 7,12. Eine weitere Komplikation ist, dass die genetische Deletion von bakteriellen Autolysinen zu Meta-Phänotypen34 führen kann. Meta-Phänotypen entstehen aus dem komplexen Zusammenspiel zwischen dem von der genetischen Deletion betroffenen Signalweg und anderen miteinander verbundenen Wegen. Zum Beispiel kann ein Meta-Phänotyp über einen direkten Effekt wie das Fehlen eines Enzyms oder einen indirekten Effekt wie eine Störung der Regulatoren entstehen.
Derzeit gibt es nur wenige Inhibitoren von Glykosidasenautolysinen wie N-Acetylglucosaminidasen (GlcNAcase) und N-Acetylmuramidasen, die als chemische Sonden verwendet werden können, um den Abbau von PG zu untersuchen. Um dies zu beheben, wurde das Diamid-Masarimycin (früher als fgkc bezeichnet)identifiziert und 35 als bakteriostatischer Inhibitor des Bacillus subtilis-Wachstums charakterisiert, der auf das GlcNAcase LytG32 abzielt (Abbildung 1). LytG ist ein exo-wirkendes GlcNAcase36, ein Mitglied von Cluster 2 innerhalb der Glykosylhydrolasefamilie 73 (GH73). Es ist das wichtigste aktive GlcNAcase während des vegetativen Wachstums32. Unseres Wissens ist Masarimycin der erste Inhibitor eines PG-wirkenden GlcNAcase, der das Zellwachstum hemmt. Zusätzliche Studien von Masarimycin mit Streptococcus pneumoniae ergaben, dass Masarimycin wahrscheinlich den Zellwandstoffwechsel in diesem Organismushemmt 37. Hier wird die Herstellung von Masarimycin zur Verwendung als chemisch-biologische Sonde zur Untersuchung der Physiologie in den grampositiven Organismen B. subtilis und S. pneumoniae berichtet. Es werden Beispiele für die morphologische Analyse der Behandlung der inhibitorischen Konzentration unter dem Minimum mit Masarimycin sowie ein Synergie-/Antagonismus-Assay vorgestellt. Synergie- und Antagonismus-Assays mit Antibiotika mit klar definierten Wirkungsweisen können ein nützlicher Weg sein, um Verbindungen zwischen zellulären Prozessen zu erforschen38,39,40.
Protokoll
1. Allgemeine Methoden
HINWEIS: Alle Compounds wurden von Standardlieferanten bezogen und ohne weitere Reinigung verwendet.
- Führen Sie eine Dünnschichtchromatographie (TLC) auf einer mit Kieselgel XG F254 vorbeschichteten Aluminiumplatte durch. Erkennen Sie Flecken unter einer UV-Lampe, durch Eintauchen in p-Anisaldehydflecken oder durch Aussetzen von I2-Dampf.
- Zeichnen Sie alle Kernspinresonanzspektren (NMR) auf einem 400-MHz-Spektrometer auf.
ANMERKUNG: 1 H-NMR- und 13C-NMR-Spektren wurden auf Restlösungsmittelspitzen bezogen. Kopplungskonstanten sind in [Hz] und chemische Verschiebungen in [ppm] angegeben. - Record atmospheric pressure chemical ionization (APCI) Massenspektrometriespektren von Masarimycin auf einem kompakten Massenspektrometer, das mit einer atmosphärischen Feststoffanalysesonde ausgestattet ist.
2. Allgemeines Verfahren zur Herstellung von Masarimycin
HINWEIS: Führen Sie die folgenden Schritte in einem Abzug aus.
- Es wird eine 0,1 M Lösung in Methanol jedes Reaktanten hergestellt: Cyclohexylamin, Cyclohexylcarboxaldehyd, O-Iodbenzoesäure und Cyclohexylisocyanid35.
ACHTUNG: Cyclohexylamin, Cyclohexylisocyanid und Cyclohexylcarboxaldehyd sind brennbar. Sie können Hautkorrosion verursachen und orale, dermale, respiratorische oder reproduktive Toxizität induzieren. Halten Sie Verbindungen von offenen Flammen, heißen Oberflächen und Zündquellen fern. Tragen Sie einen geeigneten Haut- und Augenschutz, arbeiten Sie in einem gut belüfteten Bereich und vermeiden Sie das Einatmen von Dämpfen oder Nebel. Halten Sie die Flaschen zur Lagerung fest verschlossen und lagern Sie sie an einem kühlen, trockenen Ort. Lagern Sie Cyclohexylcarboxaldehyd in einem Exsikkator unter einerN2-Atmosphäre . - Mischen Sie 5 ml Cyclohexylamin (0,1 M Lösung in Methanol) und 5 ml Cyclohexylcarboxaldehyd (0,1 M in Methanol) in einem verschlossenen runden Bodenkolben und rühren Sie die Lösung mit einem magnetischen Rührstab auf einer Rühr-/Kochplatte für 30 min bei 40 °C in einem Sandbad. Überwachen Sie die Temperatur mit einem Thermometer, das etwa 1 cm unter der Sandoberfläche platziert ist.
- Nach 30 min werden 5 ml Cyclohexylisocyanid (0,1 m Lösung in Methanol) in die Lösung aus Schritt 2.2 gegeben und weitere 20 min bei 50 °C umgerührt. Zum Schluss werden 5 ml o-Jodbenzoesäure (0,1 m Lösung in Methanol) in das Reaktionsgemisch gegeben und bei 55 °C für 3-5 h weiter gerührt.
- Überwachen Sie den Fortschritt der Reaktion regelmäßig durch TLC etwa jede Stunde, nachdem das obige Reaktionsgemisch 3 h lang gerührt wurde.
- Schneiden Sie einen 3 cm x 6 cm großen Streifen aus TLC-Platte mit Aluminiumrücken. Zeichne mit einem Bleistift #2 eine Linie etwa 1 cm von unten. Platzieren Sie mit einer Glasmikrokapillare etwa 5 μL der Reaktionsmischung auf der TLC-Platte und lassen Sie sie trocknen.
- Zu einem 150-ml-Becherglas fügen Sie genügend mobile Phase (90: 10-Hexan: Isopropanol) hinzu, um den Boden des Becherglases zu bedecken. Legen Sie die obige TLC-Platte vorsichtig mit einer Pinzette in das Becherglas, um sicherzustellen, dass die TLC-Platte gleichmäßig in die mobile Phase eintritt. Decken Sie die Oberseite des Becherglases mit einem Stück Alufolie ab.
HINWEIS: Stellen Sie sicher, dass die mobile Phase die Linie und die gefleckte Probe nicht bedeckt. - Lassen Sie die mobile Phase die TLC-Platte hinauffahren, bis sie etwa 1 cm unter der Oberseite der Platte liegt. Entfernen Sie die TLC-Platte und zeichnen Sie mit einem Bleistift eine Linie, die die von der mobilen Phase zurückgelegte Strecke angibt. Lassen Sie die TLC-Platte in einem Abzug trocknen.
- Nach dem Trocknen legen Sie die TLC-Platte in ein Becherglas, das eine kleine Menge festes I2 enthält, und bedecken Sie das Becherglas mit einem Stück Zinnfolie. Überwachen Sie die TLC auf die Entwicklung von gelben/braunen Flecken. Entfernen Sie nach der Entwicklung die TLC-Platte und markieren Sie die Position der Spots mit einem Bleistift (Ergänzende Abbildung 1).
HINWEIS: Wenn I2 Flecken nicht markiert sind, löst sich der Fleck im Laufe der Zeit auf. Flecken können auch auf der TLC-Platte durch UV-Licht, p-Anisaldehyd-Färbung oder Kaliumpermanganatfärbung sichtbar gemacht werden (siehe Ergänzende Informationen). - Berechnen Sie dieRf-Werte für alle visualisierten Spots mit der folgenden Formel:
Rf =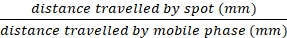
- Betrachten Sie die Reaktion als abgeschlossen, wenn nur eine Stelle mit Rf = 0,3 auf der TLC-Platte sichtbar ist. Entfernen Sie das Lösungsmittel in einem rotatorischen Verdampfer unter reduziertem Druck und trocknen Sie das Rohprodukt (erhalten als gelblich-braunes Öl) unter einem Hochvakuum, bis das gesamte Methanol verdampft ist.
- Lösen Sie das getrocknete Rohprodukt in 30 ml Ethylacetat auf und geben Sie es in einen separaten Trichter. Ethylacetat nacheinander mit 1 M HCl (2 x 30 ml), H2O (30 mL), gesättigterNaHCO3-Lösung (2 x 30 ml),H2O(30 ml) und gesättigter NaCl-Lösung (2 x 30 ml) extrahieren. Verwerfen Sie die wässrigen Schichten.
HINWEIS: Die Ethylacetatschicht ist die oberste Schicht in jeder der Extraktionen. Schütteln Sie bei jeder Extraktion den Separationstrichter, der das Ethylacetat und die wässrige Lösung (HCl, H2O,NaHCO3 oder NaCl) enthält, kräftig und lassen Sie die Schichten vollständig abscheiden. - Entfernen Sie die Ethylacetatschicht aus dem Separatorientrichter und sammeln Sie sie in einem Erlenmeyerkolben. Fügen Sie einen Spatel voll Na2SO 4 (wasserfrei) hinzu, um Restwasser aus Ethylacetat zu entfernen.
HINWEIS: Die Ethylacetatlösung gilt als trocken, wennNa2SO4 im Kolben frei läuft und nicht verklumpt. WennNa2 SO4 verklumpt, kann ein zusätzlicher Spatel Na2SO4 hinzugefügt werden. - Filtern Sie die getrocknete Ethylacetatlösung durch #1 Filterpapier, umNa2 SO4 zu entfernen. Waschen Sie das Filterpapier mit einer kleinen Menge Ethylacetat. Geben Sie die gefilterte Ethylacetatlösung in einen runden Bodenkolben und entfernen Sie das Lösungsmittel auf einem rotatorischen Verdampfer unter reduziertem Druck, um Masarimycin als Öl zu erhalten, sobald das gesamte Ethylacetat entfernt wurde.
- Das oben erhaltene Masarimycinöl wird in einer minimalen Menge (1-2 ml) von 9:1 Hexan: Isopropanol gelöst und auf einer magnetischen Rührplatte umgerührt, bis die gesamte Verbindung gelöst ist.
- Reinigen Sie das gelöste Masarimycin durch Flash-Chromatographie mit einer 12 g Normalphasen-Silica-Flash-Säule.
- Gleichen Sie die Blitzsäule mit 10 Säulenvolumina der mobilen Phase (99:1 Hexan: Isopropanol) aus, wobei das Gerät auf eine Durchflussrate von 15 ml/min eingestellt ist.
HINWEIS: Nachdem das Gleichgewicht abgeschlossen ist, stoppen Sie den Fluss und trennen Sie die Oberseite der Säule vom System. - Ziehen Sie gelöstes Masarimycin mit einer 5-ml-Spritze auf. Verbinden Sie die Spritze direkt mit der Oberseite der Gleichgewichtsblitzsäule und injizieren Sie die Lösung in die Säule. Schließen Sie die belastete Säule wieder an das Blitzchromatographiesystem an und initiieren Sie die Gradientenelution.
- Eludes Masarimycin aus der Säule mittels Gradientenelution zu einer Endkonzentration der mobilen Phase von 10:90 Hexan: Isopropanol über 12 Säulenvolumen. Überwachen Sie die Elution von Masarimycin durch Absorption bei 230 und 254 nm.
- Sammeln Sie die aus der Säule eluierten Verbindungen durch einen Fraktionssammler, der 20 ml Lösungsmittel pro Fraktion sammelt.
HINWEIS: Wenn kein Flash-Chromatographie-System verfügbar ist, kann die Reinigung von Masarimycin über eine Schwerkraftkieselsäuresäule mit einer mobilen 3:1 (Hexan: Ethylacetat) Phase durchgeführt werden. Fraktionen, die Masarimycin enthalten, können durch TLC unter Verwendung derselben mobilen Phase identifiziert werden. Die Visualisierung von TLC-Spots erfolgte entweder mit UV-Licht,I-2-Dampf oder Kaliumpermanganat-Färbung. - Identifizierung von Masarimycin-haltigen Fraktionen durch TLC (Schritte 2.5-2.9) oder Massenspektrometrie auf einem kompakten Massenspektrometer, das mit einer atmosphärischen Feststoffanalysesonde ausgestattet ist. Trocknen Sie das Endprodukt unter Vakuum (~0,3 mbar).
HINWEIS: Masarimycin wird routinemäßig als farbloses Öl oder Feststoff mit einer Ausbeute von 55% -70% in Bezug auf mmol von Cyclohexylcarboxaldehyd, das der Reaktion zugesetzt wird, erhalten. Berechnen Sie die Endausbeute von Masarimycin, indem Sie die Masse des gereinigten Masarimycins erhalten und die theoretische Ausbeute der Reaktion mit der folgenden Formel berechnen:
% Ausbeute = x 100%
x 100%
- Gleichen Sie die Blitzsäule mit 10 Säulenvolumina der mobilen Phase (99:1 Hexan: Isopropanol) aus, wobei das Gerät auf eine Durchflussrate von 15 ml/min eingestellt ist.
- Bestätigen Sie die Struktur von Masarimycin durch NMR.
- Lösen Sie ~ 10 mg Masarimycin-Probe in 0,5 mlCDCl 3 auf. Übertragen Sie die Lösung mit einer Pasteur-Pipette auf ein 5-mm-NMR-Rohr und verschließen Sie das Rohr. Legen Sie die NMR-Röhre in das Spektrometer.
- Erfassen Sie 1-H- und 13-C-NMR-Spektrenmit voreingestellten Experimenten des Herstellers. Chemische Schichtzuordnungen und repräsentative Spektren sind in den Ergänzungsabbildungen 3-4 enthalten.
- Masarimycin trocken oder in DMSO gelöst (25 mM Endkonzentration) bei -20 °C bis zur Anwendung lagern.
3. Mikrowellenverfahren zur Herstellung von Masarimycin
- Es werden 0,6 Mio. Lösungen von Cyclohexylamin, Cyclohexylcarboxaldehyd, Cyclohexylisocyanid und o-Iodbenzoesäure in Acetonitril hergestellt.
- Fügen Sie einen Rührstab und 10 ml Acetonitril zu einer Mikrowellenreaktionsdurchstechflasche aus Glas hinzu.
- 2 ml Cyclohexylamin (0,6 M in Acetonitril), 2 ml Cyclohexylcarboxaldehyd (0,6 M in Acetonitril) und 7 ml Acetonitril in die Durchstechflasche geben.
- Legen Sie die Mikrowellen-Reaktionsdurchstechflasche in das Mikrowellenkarussell. Rühren Sie die Mischung um, erhitzen Sie sie für 30 min bei 50 ° C bei einer Leistungseinstellung von 400 W und lassen Sie sie auf Raumtemperatur abkühlen.
- 2 ml o-Jodbenzoesäure (0,6 M in Methanol) und 2 ml Cyclohexylisocyanid (0,6 M in Acetonitril) in die Durchstechflasche geben. Rühren Sie die Mischung um, erhitzen Sie sie in der Mikrowelle für 40 min bei einer Leistungseinstellung von 400 W auf 100 °C und lassen Sie sie auf Raumtemperatur abkühlen.
- Überwachen Sie den Fortschritt der Reaktion mit TLC (90:10 Hexan: Isopropanol) unter Verwendung vonI2-Dampf nach Abschluss von Schritt 3.5.
HINWEIS: Wenn TLC zeigt, dass die Reaktion unvollständig ist (d. h. mehrere Flecken auf TLC), legen Sie die Reaktionsflasche wieder in die Mikrowelle und stellen Sie die in Schritt 3.5 beschriebenen Mikrowellenbedingungen ein. - Sobald die Reaktion abgeschlossen ist, gießen Sie die Lösung in einen 100 ml Rundbodenkolben und verdampfen Sie sie mit einem Rotationsverdampfer zum Trocknen.
- Befolgen Sie die Schritte 2.6-2.16 oben, um die wässrige Aufarbeitung, Reinigung und Charakterisierung von Masarimycin abzuschließen.
4. Synergie- und Antagonismus-Assay
- Züchten Sie Streptococcus pneumoniae R6 auf Mueller-Hinton (MH) Agarplatten mit 5% (v/v) Schafblut bei 37 °C unter anaeroben Bedingungen. Verwenden Sie in allen Experimenten Zellen der zweiten Passage, die in 5 ml MH-Brühe bei 37 ° C unter anaeroben Bedingungen gezüchtet wurden, bis OD600 ~ 0,4 beträgt.
- Setzen Sie die Inhibitoren Masarimycin und Optochin seriellen 1:2-Verdünnungen in entsprechenden Lösungsmitteln aus, wobei die resultierenden Konzentrationen die minimalen Inhibitorkonzentrationswerte (MIC) jedes Inhibitors flankieren.
- Machen Sie die anfängliche Verdünnung von Masarimycin in Dimethylsulfoxid (DMSO), bis eine Konzentration von 100 μM erreicht ist. Machen Sie ab diesem Punkt Masarimycin-Verdünnungen in MH-Brühe. Bereiten Sie die Optochin-Stammlösung (3,5 mM) vor, indem Sie handelsübliches Optochin (siehe Materialverzeichnis) in steriler MH-Brühe auflösen.
HINWEIS: Masarimycin-Stammlösungen wurden bei 25 mM in DMSO hergestellt.
- Machen Sie die anfängliche Verdünnung von Masarimycin in Dimethylsulfoxid (DMSO), bis eine Konzentration von 100 μM erreicht ist. Machen Sie ab diesem Punkt Masarimycin-Verdünnungen in MH-Brühe. Bereiten Sie die Optochin-Stammlösung (3,5 mM) vor, indem Sie handelsübliches Optochin (siehe Materialverzeichnis) in steriler MH-Brühe auflösen.
- Zu einer sterilen 96-Well-Mikrotiterplatte fügen Sie 2 μL Aliquots jeder Optochinverdünnung zu jeder Reihe der Platte hinzu. Zu derselben Platte fügen Sie 2 μL Aliquots jeder Masarimycin-Verdünnung zu jeder Spalte hinzu, um eine Reihe von Optochin- und Masarimycin-Konzentrationen auf der Platte zu erzeugen (Abbildung 2).
- Fügen Sie sterile MH-Brühe (93 μL) zu jeder Vertiefung hinzu, die die oben genannten Inhibitoren enthält. Die Mikrotiterplatten werden mit 5 μL Kultur (OD600 ~0,4) aus Schritt 4.1 beimpft.
HINWEIS: Die Impfung der 96-Well-Platte erfolgt typischerweise unter anaeroben Bedingungen an einem anaeroben Arbeitsplatz. Das endgültige Volumen im Bohrloch beträgt 100 μL. - Züchten Sie Kulturen für 18 h bei 37 °C unter anaeroben Bedingungen, gefolgt von der Zugabe von 30 μL 0,01% (m/v) Lösung von Resazurin-Natriumsalz. Inkubieren Sie die Platte bei Raumtemperatur für 15 Minuten, um die Bildung und Stabilisierung der Farbe zu ermöglichen.
HINWEIS: Resazurinlösung wird durch Auflösen der Verbindung in destilliertem Wasser hergestellt und kann bei 4 °C bis zu zwei Wochen gelagert werden. - Lesen Sie die Konzentrationswerte direkt von der Platte ab und weisen Sie die niedrigste Inhibitorkonzentration, für die kein Bakterienwachstum beobachtet wird (blaue Farbe), als [X] zu (siehe Schritt 4.7.1), d.h. die niedrigste inhibitorische Konzentration des Arzneimittels in Gegenwart des Co-Arzneimittels.
HINWEIS: Positives Bakterienwachstum wird in den Vertiefungen durch den rosa werdenden Resazurinfarbstoff identifiziert. MIC-Werte für jedes Arzneimittel allein (d. h. in Abwesenheit eines Co-Arzneimittels) werden auf ähnliche Weise unter Verwendung des Resazurin-MIC-Assays35 mit jedem Arzneimittel separat bestimmt (Ergänzende Abbildung 5). MICs in S. pneumoniae betragen 7,8 μM bzw. 15,85 μM für Masarimycin bzw. Optochin. - Bestimmen Sie die fraktionierte inhibitorische Konzentration (FIC) und den FIC-Index (FICI) mithilfe der folgenden Gleichungen.
- FIC= [X]/MIC x, wobei [X] (aus Schritt 4.6) die niedrigste inhibitorische Konzentration des Arzneimittels in Gegenwart des Co-Arzneimittels und MICx die niedrigste hemmende Konzentration des Arzneimittels in Abwesenheit des Co-Arzneimittels ist.
- FICI = FICMasarimycin + FICAntibiotikum
ANMERKUNG: FIC I < 0,5 = synergistisch, 0,5 <FIC I < 1 = additiv, 1 < FIC I < 4 = gleichgültig,FIC I > 4 = antagonistisch.
5. Morphologische Untersuchung
- Züchten Sie Bacillus subtilis 11774 auf Luria-Bertani (LB) Agarplatten (10 g/L Trypton, 5 g/L Hefeextrakt und 5 g/L NaCl) mit 1,5% Bacto-Agar bei 37 °C. Verwenden Sie in allen Experimenten Zellen der zweiten Passage, die in 5 ml LB-Brühe bei 37 ° C bis OD600 = 1 gezüchtet wurden. Züchten Sie S.pneumoniae auf die gleiche Weise wie in Schritt 4.1.
- Nach Erhalt einer Zellkulturdichte mit OD 600nm = 1 für B. subtilis oder OD600nm = 0,4 für S. pneumoniae wird Masarimycin mit einer Pipette zu dem als "behandelt" bezeichneten Kulturröhrchen bis zu einer Endkonzentration von 3,8 μM (0,75x MIC für B.subtilis) oder 5,85 μM (0,75x MIC für S.pneumoniae) zugegeben. Fügen Sie dem zweiten Kulturröhrchen mit der Bezeichnung "Steuerung" ein äquivalentes Volumen von DMSO hinzu.
- Für B.subtilis legen Sie die Proben 90 min lang in einen Inkubator bei 37 °C mit Schütteln bei 150 U/min. Bei S. pneumoniae die Zellen unter anaeroben Bedingungen ohne Schütteln inkubieren.
- Nach 90 min die Kulturen in einem 1:10-Gemisch (v/v) aus Kulturmedien und Fixierpuffer (20 mM HEPES, 1% Formaldehyd (pH 6,8)) bei 4 °C über Nacht chemisch fixieren. Nachdem die Fixierung abgeschlossen ist, tragen Sie 10-20 μL Proben mit einer Pipette auf Glasmikroskopobjektträger auf und lassen Sie sie an der Luft trocknen. Fixieren Sie die luftgetrockneten Proben, indem Sie die Glasobjektträger mit einem Bunsenbrenner erhitzen.
- Nach der Hitzefixierung die Proben unter Zugabe von 100 μL 0,1% (m/v) Methylenblau (Lösung in 20% (v/v) Ethanol) färben. Die gebeizten Objektträger für 10 min inkubieren und den überschüssigen Farbstoff mit dH2O wegwaschen. Dann erhitzen Sie die gefärbten Objektträger vorsichtig auf 60 ° C in einem Ofen für 15-20 Minuten, um die Zellen auf eine gemeinsame Fokusebene zu bringen.
- Verschließen Sie die gefärbten Proben, indem Sie ein Mikroskop-Deckglas über die gefärbten Zellen legen. Versiegeln Sie dann die Kanten mit Objektträgerzement. Legen Sie den versiegelten Objektträger auf den Mikroskoptisch und bringen Sie das Bild mit 100-facher Vergrößerung mittels Hellfeldmikroskopie in den Fokus.
- Legen Sie einen Tropfen Tauchöl auf den Objektträger und bringen Sie das Sichtfeld mit 1000-facher Vergrößerung in den Fokus. Erfassen Sie Mikroaufnahmen mit einer Kamera, die an das Mikroskop und die zugehörige Software angeschlossen ist. Erfassen Sie Bilder mit den Einstellungen für den automatischen Weißabgleich und die Blende in der Software.
HINWEIS: Alternativ können Bilder mit der Open-Source-Software ImageJ verarbeitet werden.
Ergebnisse
Masarimycin ist ein niedermolekularer bakteriostatischer Inhibitor von B. subtilis und S. pneumoniae und hemmt nachweislich das exo-wirkende GlcNAcase LytG in B. subtilis 35,37 und zielt in S. pneumoniae 37 auf die Zellwand ab. Masarimycin kann entweder durch die klassische oder mikrowellengestützte organische Synthese mit Ausbeuten im Bereich von 55% -70% effizient hergestellt werden. Die mikrowellenge...
Diskussion
Masarimycin ist ein einzelner mikromolarer bakteriostatischer Inhibitor des Wachstums von B. subtilis 35 und S. pneumoniae37. In B. subtilis wurde gezeigt, dass Masarimycin das GlcNAcase LytG35 hemmt, während das genaue molekulare Ziel in der Zellwand von S. pneumoniae nicht identifiziertwurde 37. Die Synthese von Masarimycin entweder mit dem klassischen organischen Synthese- oder Mikrowellenverfahr...
Offenlegungen
Reid, C. W. verfügt über geistiges Eigentum, das spezifische Anwendungen von Masarimycin betrifft.
Danksagungen
Die Forschung wurde von der National Science Foundation unter der Fördernummer 2009522 unterstützt. Die NMR-Analyse von Masarimycin wurde durch den National Science Foundation Major Research Instrumentation Program Award unter der Fördernummer 1919644 unterstützt. Alle Meinungen, Erkenntnisse, Schlussfolgerungen oder Empfehlungen, die in diesem Material zum Ausdruck gebracht werden, sind die der Autoren und spiegeln nicht unbedingt die Ansichten der National Science Foundation wider.
Materialien
| Name | Company | Catalog Number | Comments |
| 2-Iodobenzoic acid | SIGMA-ALDRICH | I7675-25G | corrosive, irritant, light yellow to orange-brown powder |
| 2-Propanol | SIGMA-ALDRICH | 109827-4L | flammable, irritant, colorless liquid |
| Acetonitrile | SIGMA-ALDRICH | 34851-4L | flammable, irritant, colorless liquid |
| Aluminum backed silica plates | Sorbtech | 4434126 | silica gel XG F254 on aluminum backed plates |
| chloroform-d | SIGMA-ALDRICH | 151823-50G | solvent for NMR |
| Compact Mass Spectrometer | Advion-Interchim | Advion CMS | compact mass spectrometer equiped with APCI source and atmospheric solids analysis probe |
| Corning Costar 96 well flat bottom plates-sterile | fisher chemical | 07-200-90 | for synergy/antagonism assays |
| cover slips | fisher chemical | 12-547 | for microscopy |
| Cyclohexanecarboxaldehyde | CHEM-IMPEX INT'L INC. | 24451 | flammable, irritant, colorless to pink liquid |
| Cyclohexyl isocyanide | SIGMA-ALDRICH | 133302-5G | irritant, colorless liquid, extremly unpleasant odor |
| Cyclohexylamine | SIGMA-ALDRICH | 240648-100ML | corrosive, flammable, irritant, colorless liquid unless contaminated |
| Ethyl acetate | SIGMA-ALDRICH | 537446-4L | flammable, irritant, colorless liquid |
| flash silica cartridge (12g) | Advion-Interchim | PF-50SIHP-F0012 | pack of flash silica columns (12g) for purification of masarimycin |
| formaldehyde | SIGMA-ALDRICH | F8775-25ML | fixing agent for microscopy |
| HEPES | SIGMA-ALDRICH | H8651-25G | buffer for microscopy fixing solution |
| Hexane, mixture of isomers | SIGMA-ALDRICH | 178918-4L | environmentally damaging, flammable, irritant, health hazard, colorless liquid |
| High performance compact mass spectrometer | Advion | expression | Atmospheric Solids Analysis Probe (ASAP), low resolution |
| High Vac | eppendorf | Vacufuge plus | vacuum aided by centrifugal force and temperature |
| Hydrochloric acid | SIGMA-ALDRICH | 258148-2.5L | corrosive, irritant, colorless liquid |
| hydrochloric acid | SIGMA-ALDRICH | 320331-2.5L | strong acid |
| immersion oil | fisher chemical | 12-365-19 | for microscopy |
| Iodine, resublimed crystals | Alfa Aesar | 41955 | environmentally damaging, irritant, health hazard, dark grey/purple crystals |
| Mestre Mnova | MestreLab Research | software for processing NMR spectra | |
| Methanol | SIGMA-ALDRICH | 439193-4L | flammable, toxic, health hazard, colorless liquid |
| methylene blue | SIGMA-ALDRICH | M9140-25G | microscopy stain for staining cell walls |
| meuller-hinton agar plates + 5% sheep blood | fisher chemical | B21176X | growth media for Streptococcus pneumoniae |
| meuller-hinton broth | fisher chemical | DF0757-17-6 | growth media for Streptococcus pneumoniae |
| microscope slides | fisher chemical | 22-310397 | for microscopy |
| Microwave Synthesis Labstation | MILESTONE | START SYNTH | device that requires the ventilation of a fume hood, equipped with synthesis carousel |
| NMR tubes | SIGMA-ALDRICH | Z562769-5EA | 5mm NMR tubes 600 MHz |
| Nuclear Magnetic Resonance (NMR) | Bruker | Ascend 400 | large superconducting magnet (400MHz) |
| optochin | fisher chemical | AAB21627MC | ethylhydrocupreine hydrochloride |
| petrie plates | Celltreat | 229695 | for preparing agar plates for bacterial growth |
| Primo Star Bright field/Phase contrast Microscope with ERc5s camera | Zeiss | for morphology studies | |
| puriFlash | interchim | XS520plus | flash chromatography purification system |
| resazurin | SIGMA-ALDRICH | R7017-1G | for synergy/antagonism assays |
| Rotary Evaporator | Heidolph | Hei-VAP Value "The Collegiate" | solvent evaporator |
| Sodium bicarbonate | SIGMA-ALDRICH | S6014-500G | irritant, white powder |
| Sodium chloride | fisher chemical | S271-1 | crystalline, colorless |
| Sodium chloride | SIGMA-ALDRICH | S5886-500G | for growth of B.subtilis and preparation of LB media |
| Sodium sulfate | SIGMA-ALDRICH | 7985592-500G | anhydrous, granular, white |
| tryptone | fisher chemical | BP1421-500 | for growth of B.subtilis and preparation of LB media |
| Whitney DG250 Workstation | Microbiology International | DG250 | anaerobic workstation. Anaerobic gas mixture used: 5% hydrogen, 10% carbon dioxide, balance nitrogen |
| yeast extract | fisher chemical | BP1422-500 | for growth of B.subtilis and preparation of LB media |
| Zen Lite (blue) software | Zeiss | for acquiring micrographs |
Referenzen
- Vollmer, W., Blanot, D., de Pedro, M. A. Peptidoglycan structure and architecture. FEMS Microbiology Review. 32 (2), 149-167 (2008).
- Munita, J. M., Bayer, A. S., Arias, C. A. Evolving resistance among Gram-positive pathogens. Clinical Infectious Diseases. 61, 48-57 (2015).
- Vollmer, W., Bertsche, U. Murein (peptidoglycan) structure, architecture and biosynthesis in Escherichia coli. Biochimica Biophysica Acta. 1778 (9), 1714-1734 (2008).
- Vollmer, W., Höltje, J. -. V. The architecture of the murein (peptidoglycan) in Gram-negative bacteria: vertical scaffold or horizontal layer(s). Journal of Bacteriology. 186 (18), 5978-5987 (2004).
- Clarke, A. J. Compositional analysis of peptidoglycan by high-performance anion-exchange chromatography. Analytical Biochemistry. 212 (2), 344-350 (1993).
- Kim, S. J., Chang, J., Singh, M. Peptidoglycan architecture of Gram-positive bacteria by solid-state NMR. Biochimica Biophysica Acta. 1848, 350-362 (2014).
- Koch, A. L., Doyle, R. J. Inside-to-outside growth and turnover of the wall of gram-positive rods. Journal of Theoretical Biology. 117 (1), 137-157 (1985).
- Beeby, M., Gumbart, J. C., Roux, B., Jensen, G. J. Architecture and assembly of the Gram-positive cell wall. Molecular Microbiology. 88 (4), 664-672 (2013).
- Shockman, G. D., Daneo-Moore, L., Kariyama, R., Massidda, O. Bacterial walls, peptidoglycan hydrolases, autolysins, and autolysis. Microbial Drug Resistance. 2 (1), 95-98 (1996).
- Dijkstra, A. J., Keck, W. Peptidoglycan as a barrier to transenvelope transport. Journal of Bacteriology. 178 (19), 5555-5562 (1996).
- Blackman, S. A., Smith, T. J., Foster, S. J. The role of autolysins during vegetative growth of Bacillus subtilis 168. Microbiology. 144, 73-82 (1998).
- Misra, G., Rojas, E. R., Gopinathan, A., Huang, K. C. Mechanical consequences of cell-wall turnover in the elongation of a Gram-positive bacterium. Biophysical Journal. 104 (11), 2342-2352 (2013).
- Wheeler, R., et al. Bacterial cell enlargement requires control of cell wall stiffness mediated by peptidoglycan hydrolases. mBio. 6 (4), 00660 (2015).
- Taguchi, A., Kahne, D., Walker, S. Chemical tools to characterize peptidoglycan synthases. Current Opinion in Chemical Biology. 53, 44-50 (2019).
- Welsh, M. A., Schaefer, K., Taguchi, A., Kahne, D., Walker, S. Direction of chain growth and substrate preferences of shape, elongation, division, and sporulation-family peptidoglycan glycosyltransferases. Journal of the American Chemial Society. 141 (33), 12994-12997 (2019).
- Rubino, F. A., et al. Detection of transport intermediates in the peptidoglycan flippase MurJ identifies residues essential for conformational cycling. Journal of the American Chemical Society. 142 (12), 5482-5486 (2020).
- Sjodt, M., et al. Structure of the peptidoglycan polymerase RodA resolved by evolutionary coupling analysis. Nature. 556 (7699), 118-121 (2018).
- Tiyanont, K., et al. Imaging peptidoglycan biosynthesis in Bacillus subtilis with fluorescent antibiotics. Proceedings of the National Academy of Science U S A. 103 (29), 11033-11038 (2006).
- Lebar, M. D., et al. Reconstitution of peptidoglycan cross-linking leads to improved fluorescent probes of cell wall synthesis. Journal of the American Chemical Society. 136 (31), 10874-10877 (2014).
- Do, T., Page, J. E., Walker, S. Uncovering the activities, biological roles, and regulation of bacterial cell wall hydrolases and tailoring enzymes. Journal of Biological Chemistry. 295 (10), 3347-3361 (2020).
- Liang, H., et al. Metabolic labelling of the carbohydrate core in bacterial peptidoglycan and its applications. Nature Communications. 8, 15015 (2017).
- DeMeester, K. E., et al. Metabolic incorporation of N-acetyl muramic acid probes into bacterial peptidoglycan. Current Protocol in Chemical Biology. 11 (4), 74 (2019).
- Lazor, K. M., et al. Use of Bioorthogonal N-acetylcysteamine (SNAc) analogues and peptidoglycan O-acetyltransferase B (PatB) to label peptidoglycan. The FASEB Journal. 32, 630 (2018).
- Wang, Y., Leimkuhler-Grimes, C. Fluorescent labeling of the carbohydrate backbone of peptidoglycan to track degradation in vivo. The FASEB Journal. 29, (2015).
- Kuru, E., et al. In probing of newly synthesized peptidoglycan in live bacteria with fluorescent D-amino acids. Angewandte Chemie International Edition. 51 (50), 12519-12523 (2012).
- Zhou, R., Chen, S., Recsei, P. A dye release assay for determination of lysostaphin activity. Analytical Biochemistry. 171 (1), 141-144 (1988).
- Qiao, Y., et al. Lipid II overproduction allows direct assay of transpeptidase inhibition by β-lactams. Nature Chemical Biology. 13 (7), 793-798 (2017).
- Lebar, M. D., et al. Forming cross-linked peptidoglycan from synthetic Gram-negative lipid II. Journal of the American Chemical Society. 135 (12), 4632-4635 (2013).
- Chen, R., Guttenplan, S. B., Blair, K. M., Kearns, D. B. Role of the D-dependent autolysins in Bacillus subtilis population heterogeneity. Journal of Bacteriology. 191 (18), 5775-5784 (2009).
- Yukie, S., Miki, K., Yoshio, N., Kuniaki, T., Yoshihisa, Y. Identification and characterization of an autolysin-encoding gene of Streptococcus mutans. Infection and Immunity. 73 (6), 3512-3520 (2005).
- Domenech, M., García, E., Moscoso, M. In vitro destruction of Streptococcus pneumoniae biofilms with bacterial and phage peptidoglycan hydrolases. Antimicrobial Agents and Chemotherapy. 55 (9), 4144-4148 (2011).
- Horsburgh, G. J., Atrih, A., Williamson, M. P., Foster, S. J. LytG of Bacillus subtilis is a novel peptidoglycan hydrolase: the major active glucosaminidase. Biochemistry. 42 (2), 257-264 (2003).
- Vermassen, A., et al. Cell wall hydrolases in bacteria: insight on the diversity of cell wall amidases, glycosidases and peptidases toward peptidoglycan. Frontiers in Microbiology. 10, 331 (2019).
- Martin-Galiano, A. J., Yuste, J., Cercenado, M. I., de la Campa, A. G. Inspecting the potential physiological and biomedical value of 44 conserved uncharacterised proteins of Streptococcus pneumoniae. BMC Genomics. 15, 652 (2014).
- Nayyab, S., et al. Diamide inhibitors of the Bacillus subtilis N-acetylglucosaminidase LytG that exhibit antibacterial activity. ACS Infectioius Diseases. 3 (6), 421-427 (2017).
- Lipski, A., et al. Structural and biochemical characterization of the β-N-acetylglucosaminidase from Thermotoga maritima: Toward rationalization of mechanistic knowledge in the GH73 family. Glycobiology. 25 (3), 319-330 (2014).
- Haubrich, B. A., et al. Inhibition of Streptococcus pneumoniae autolysins highlight distinct differences between chemical and genetic inactivation. bioRxiv. , 300541 (2020).
- Farha, M. A., et al. Inhibition of WTA synthesis blocks the cooperative action of PBPs and sensitizes MRSA to β-lactams. ACS Chemical Biology. 8 (1), 226-233 (2013).
- Lehár, J., et al. Chemical combination effects predict connectivity in biological systems. Molecular Systems Biology. 3 (1), 80 (2007).
- Farha, M. A., et al. Antagonism screen for inhibitors of bacterial cell wall biogenesis uncovers an inhibitor of undecaprenyl diphosphate synthase. Proceedings of the National Academy of Science U S A. 112 (35), 11048-11053 (2015).
- Palomino, J. C., et al. Resazurin microtiter assay plate: simple and inexpensive method for detection of drug resistance in Mycobacterium tuberculosis. Antimicrobial Agents and Chemotherapy. 46 (8), 2720-2722 (2002).
- Odds, F. C. Synergy, antagonism, and what the chequerboard puts between them. Journal of Antimicrobial Chemotherapy. 52 (1), 1 (2003).
- Arrigucci, R., Pozzi, G. Identification of the chain-dispersing peptidoglycan hydrolase LytB of Streptococcus gordonii. PLoS One. 12 (4), 0176117 (2017).
- Bai, X. -. H., et al. Structure of pneumococcal peptidoglycan hydrolase LytB reveals insights into the bacterial cell wall remodeling and pathogenesis. of Biological Chemistry. 289 (34), 23403-23416 (2014).
- Garcia, P., Gonzalez, M. P., Garcia, E., Lopez, R., Garcia, J. L. LytB, a novel pneumococcal murein hydrolase essential for cell separation. Molecular Microbiology. 31 (4), 1275-1281 (1999).
- Giladi, M., Altman-Price, N., Levin, I., Levy, L., Mevarech, M. FolM, a new chromosomally encoded dihydrofolate reductase in Escherichia coli. Journal of Bacteriology. 185 (23), 7015-7018 (2003).
- Chua, P. R., et al. Effective killing of the human pathogen Candida albicans by a specific inhibitor of non-essential mitotic kinesin Kip1p. Molecular Microbiology. 65 (2), 347-362 (2007).
- Rico-Lastres, P., et al. Substrate recognition and catalysis by LytB, a pneumococcal peptidoglycan hydrolase involved in virulence. Scientific Reports. 5, 16198 (2015).
- Vollmer, W., et al. The cell wall of Streptococcus pneumoniae. Microbiology Spectrum. 7 (3), (2019).
- Massidda, O., Nováková, L., Vollmer, W. From models to pathogens: how much have we learned about Streptococcus pneumoniae cell division. Environmental Microbiology. 15 (12), 3133-3157 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten