Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Synthèse de la masarimycine, une petite molécule inhibitrice de la croissance bactérienne à Gram positif
Dans cet article
Résumé
Un protocole détaillé est présenté pour la préparation du diamide bactériostatique masarimycine, une sonde à petites molécules qui inhibe la croissance de Bacillus subtilis et Streptococcus pneumoniae en ciblant la dégradation de la paroi cellulaire. Son application en tant que sonde chimique est démontrée dans des essais de synergie/antagonisme et des études morphologiques avec B. subtilis et S. pneumoniae.
Résumé
Le peptidoglycane (PG) dans la paroi cellulaire des bactéries est une structure macromoléculaire unique qui confère une forme et une protection contre l’environnement environnant. Au cœur de la compréhension de la croissance et de la division cellulaires se trouve la connaissance de la façon dont la dégradation du PG influence la biosynthèse et l’assemblage de la paroi cellulaire. Récemment, le marquage métabolique du PG par l’introduction de sucres ou d’acides aminés modifiés a été rapporté. Alors que l’interrogation chimique des étapes biosynthétiques avec de petites molécules inhibitrices est possible, les outils de biologie chimique pour étudier la dégradation du PG par les autolysines sont sous-développés. Les autolysines bactériennes sont une large classe d’enzymes qui sont impliquées dans la dégradation étroitement coordonnée du PG. Ici, un protocole détaillé est présenté pour la préparation d’une sonde à petite molécule, la masarimycine, qui est un inhibiteur de la N-acétylglucosaminidase LytG dans Bacillus subtilis, et le métabolisme de la paroi cellulaire chez Streptococcus pneumoniae. La préparation de l’inhibiteur par micro-ondes et la synthèse organique classique est fournie. Son applicabilité en tant qu’outil d’étude de la physiologie à Gram positif dans les essais biologiques est présentée.
Introduction
Le peptidoglycane (PG) est un polymère en forme de maille qui délimite la forme et la structure des cellules dans les bactéries Gram positif et Gram négatif 1,2. Cet hétéropolymère est une matrice de sucres aminés réticulés par de courts peptides 3,4,5,6 avec un squelette composé de résidus de N-acétylglucosamine (GlcNAc) et d’acide N-acétylmuramique (MurNAc) β(1,4)-liés (Figure 1)1. Attaché à la fraction lactyle C-3 de MurNAc est le peptide de la tige. Le métabolisme du PG implique un système étroitement coordonné d’enzymes biosynthétiques et dégradatives pour incorporer de nouveaux matériaux dans la paroi cellulaire 7,8. La dégradation du PG est effectuée par des enzymes collectivement appelées autolysines9 et classées en fonction de la spécificité de la liaison clivée. Les autolysines participent à de nombreux processus cellulaires, notamment la croissance cellulaire, la division cellulaire, la motilité, la maturation du PG, la chimiotaxie, la sécrétion de protéines, la compétence génétique, la différenciation et la pathogénicité10,11. Démêler les fonctions biologiques spécifiques des autolysines individuelles peut être intimidant, en partie à cause de la redondance fonctionnelle. Cependant, des études biophysiquesrécentes 8,12,13 et des études computationnelles12 ont fourni de nouvelles informations sur leurs rôles dans le métabolisme du PG. En outre, des rapports récents ont fourni des informations supplémentaires sur la synthèse14 et les 15,16,17 étapes médiées par membrane dans le métabolisme du PG. Une compréhension approfondie de la relation entre les voies dégradatives et synthétiques du métabolisme du PG pourrait donner lieu à des cibles antibiotiques inexploitées auparavant.
Bien qu’il y ait eu des progrès significatifs dans la méthodologie pour étudier la glycobiologie chez les eucaryotes, la glycobiologie bactérienne et, en particulier, le métabolisme pg n’a pas progressé à un rythme similaire. Les approches chimiques actuelles pour étudier le métabolisme du PG comprennent les antibiotiques marqués par fluorescence18, les sondes fluorescentes19,20 et le marquage métabolique 21,22,23,24. Ces nouvelles approches offrent de nouvelles façons d’interroger le métabolisme de la paroi cellulaire bactérienne. Bien que certaines de ces stratégies soient capables de marquer le PG in vivo, elles peuvent être spécifiques àl’espèce 19, ou ne fonctionner que dans des souches dépourvues d’une autolysine25 particulière. De nombreuses stratégies de marquage du PG sont destinées à être utilisées avec des parois cellulaires isolées26 ou avec des voies de biosynthèse de PG reconstituées in vitro 20,27,28. L’utilisation d’antibiotiques marqués par fluorescence est actuellement limitée aux étapes biosynthétiques et à la transpeptidation18.
Les connaissances actuelles sur les autolysines bactériennes et leur rôle dans le métabolisme de la paroi cellulaire proviennent de l’analyse génétique et biochimique in vitro 11,29,30,31,32. Bien que ces approches aient fourni une mine d’informations sur cette classe importante d’enzymes, il peut être difficile de déchiffrer leur rôle biologique. Par exemple, en raison de la redondance fonctionnelle33, la suppression d’une autolysine dans la plupart des cas n’entraîne pas l’arrêt de la croissance bactérienne. Ceci malgré leur rôle implicite dans la croissance et la divisioncellulaires 7,12. Une autre complication est que la délétion génétique des autolysines bactériennes peut donner naissance à des méta-phénotypes34. Les métaphénotypes proviennent de l’interaction complexe entre la voie affectée par la délétion génétique et d’autres voies interconnectées. Par exemple, un méta-phénotype peut survenir via un effet direct tel que l’absence d’une enzyme, ou un effet indirect tel qu’une perturbation des régulateurs.
Actuellement, il n’existe que quelques inhibiteurs des autolysines glycosidases telles que les N-acétylglucosaminidases (GlcNAcase) et les N-acétylmuramidases, qui peuvent être utilisées comme sondes chimiques pour étudier la dégradation du PG. Pour y remédier, le diamide masarimycine (précédemment appelé fgkc) a été identifié et caractérisé35 comme un inhibiteur bactériostatique de la croissance de Bacillus subtilis qui cible le GlcNAcase LytG32 (Figure 1). LytG est un GlcNAcase36 à action exo-agissante, membre du groupe 2 de la famille des glycosyl hydrolases 73 (GH73). C’est le principal GlcNAcase actif au cours de la croissance végétative32. À notre connaissance, la masarimycine est le premier inhibiteur d’un GlcNAcase à action PG qui inhibe la croissance cellulaire. Des études supplémentaires sur la masarimycine avec Streptococcus pneumoniae ont révélé que la masarimycine inhibe probablement le métabolisme de la paroi cellulaire dans cet organisme37. Ici, la préparation de masarimycine est rapportée pour une utilisation comme sonde de biologie chimique pour étudier la physiologie dans les organismes à Gram positif B. subtilis et S. pneumoniae. Des exemples d’analyse morphologique d’un traitement à concentration inhibitrice inférieure au minimum avec de la masarimycine, ainsi qu’un test de synergie/antagonisme sont présentés. Les tests de synergie et d’antagonisme utilisant des antibiotiques avec des modes d’action bien définis peuvent être un moyen utile d’explorer les connexions entre les processus cellulaires 38,39,40.
Protocole
1. Méthodes générales
REMARQUE: Tous les composés ont été achetés auprès de fournisseurs standard et utilisés sans autre purification.
- Effectuer une chromatographie sur couche mince (TLC) sur une plaque d’aluminium prélaquée de gel de silice XG F254. Détecter les taches sous une lampe UV, par immersion dans la tache de p-anisaldéhyde, ou en exposant à la vapeurI2 .
- Enregistrez tous les spectres de résonance magnétique nucléaire (RMN) sur un spectromètre de 400 MHz.
REMARQUE : 1spectre H-RMN et 13spectres C-RMN ont été référencés aux pics de solvant résiduels. Les constantes de couplage sont données en [Hz] et les décalages chimiques en [ppm]. - Enregistrer les spectres de spectrométrie de masse par ionisation chimique de pression atmosphérique (APCI) de la masarimycine sur un spectromètre de masse compact équipé d’une sonde d’analyse des solides atmosphériques.
2. Procédure générale de préparation de la masarimycine
REMARQUE: Effectuez les étapes ci-dessous dans une hotte aspirante.
- Préparer une solution de 0,1 M dans le méthanol de chaque réactif : cyclohexylamine, cyclohexyl carboxaldéhyde, acide o-iodobenzoïque et isocyanure de cyclohexyle35.
ATTENTION : La cyclohexylamine, l’isocyanure de cyclohexyle et le cyclohexylcarboxaldéhyde sont inflammables. Ils peuvent provoquer une corrosion cutanée et induire une toxicité buccale, cutanée, respiratoire ou reproductive. Gardez les composés à l’écart des flammes nues, des surfaces chaudes et des sources d’inflammation. Portez une protection appropriée de la peau et des yeux, travaillez dans un endroit bien ventilé et évitez l’inhalation de vapeurs ou de brouillard. Pour le stockage, gardez les bouteilles hermétiquement fermées et conservez-les dans un endroit frais et sec. Conserver le cyclohexyl carboxaldéhyde dans un dessiccateur sous atmosphère deN2 . - Mélanger 5 mL de cyclohexylamine (solution de 0,1 M dans le méthanol) et 5 mL de cyclohexyl carboxaldéhyde (0,1 M dans le méthanol) dans une fiole à fond rond coiffé et remuer la solution à l’aide d’une barre magnétique sur une plaque chauffante pendant 30 min à 40 °C dans un bain de sable. Surveillez la température à l’aide d’un thermomètre placé à environ 1 cm sous la surface du sable.
- Après 30 min, ajouter 5 mL d’isocyanure de cyclohexyle (solution de 0,1 M dans du méthanol) à la solution de l’étape 2.2 et remuer pendant 20 min supplémentaires à 50 °C. Enfin, ajouter 5 mL d’acide o-iodobenzoïque (solution 0,1 M dans du méthanol) au mélange réactionnel et continuer à agiter à 55 °C pendant 3-5 h.
- Surveiller périodiquement la progression de la réaction par TLC environ toutes les heures après que le mélange réactionnel ci-dessus ait été agité pendant 3 h.
- Coupez une bande de 3 cm x 6 cm de plaque TLC à dos d’aluminium. À l’aide d’un crayon #2, tracez une ligne à environ 1 cm du bas. À l’aide d’un microcapillaire en verre, repérez environ 5 μL du mélange réactionnel sur la plaque TLC et laissez-le sécher.
- À un bécher de 150 mL, ajouter suffisamment de phase mobile (hexane 90:10 : isopropanol) pour recouvrir le fond du bécher. À l’aide d’une pince à épiler, placez soigneusement la plaque TLC ci-dessus dans le bécher en veillant à ce que la plaque TLC entre uniformément dans la phase mobile. Couvrir le dessus du bécher avec un morceau de papier d’aluminium.
REMARQUE: Assurez-vous que la phase mobile ne couvre pas la ligne et l’échantillon tacheté. - Laissez la phase mobile remonter la plaque TLC jusqu’à ce qu’elle se trouve à environ 1 cm sous le haut de la plaque. Retirez la plaque TLC et, à l’aide d’un crayon, tracez une ligne indiquant la distance parcourue par la phase mobile. Laissez sécher la plaque TLC dans une hotte.
- Une fois séchée, placez la plaque TLC dans un bécher contenant une petite quantité de solide I2 et recouvrez le bécher d’un morceau de papier d’aluminium. Surveillez le TLC pour le développement de taches jaunes / brunes. Une fois développée, retirez la plaque TLC et marquez l’emplacement des taches à l’aide d’un crayon (Figure supplémentaire 1).
REMARQUE: Si I2 taches ne sont pas marquées, la tache se dissipera avec le temps. Les taches peuvent également être visualisées sur la plaque TLC par la lumière UV, la coloration au p-anisaldéhyde ou la coloration au permanganate de potassium (voir Informations supplémentaires). - Calculez les valeurs Rf pour tous les points visualisés à l’aide de la formule suivante :
Rf =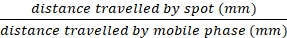
- Considérons la réaction comme terminée lorsqu’un seul point avec Rf = 0,3 est visible sur la plaque TLC. Retirer le solvant dans un évaporateur rotatif sous pression réduite et sécher le produit brut (obtenu sous forme d’huile brun jaunâtre) sous vide poussé jusqu’à ce que tout le méthanol soit évaporé.
- Dissoudre le produit brut séché dans 30 mL d’acétate d’éthyle et le transférer dans un entonnoir de séparation. Extraire séquentiellement l’acétate d’éthyle avec 1 M HCl (2 x 30 mL), H2O (30 mL), solution saturée de NaHCO 3 (2 x 30 mL),H2O (30 mL) et solution saturée de NaCl (2 x 30 mL). Jetez les couches aqueuses.
REMARQUE: La couche d’acétate d’éthyle est la couche supérieure dans chacune des extractions. Pour chaque extraction, agiter vigoureusement l’entonnoir séparateur contenant l’acétate d’éthyle et la solution aqueuse (HCl,H2O, NaHCO3 ou NaCl) et laisser les couches se séparer complètement. - Retirez la couche d’acétate d’éthyle de l’entonnoir de séparation et recueillez-la dans une fiole d’Erlenmeyer. Ajouter une spatule pleine de Na2SO4 (anhydre) pour éliminer l’eau résiduelle de l’acétate d’éthyle.
REMARQUE: La solution d’acétate d’éthyle est considérée comme sèche lorsque Na2SO4 dans la fiole coule librement et ne s’agglutine pas. Si Na2SO4 s’agglutine, une spatule supplémentaire de Na2SO4 peut être ajoutée. - Filtrer la solution d’acétate d’éthyle séchée à travers le papier filtre n ° 1 pour éliminer Na2SO4. Lavez le papier filtre avec une petite quantité d’acétate d’éthyle. Placer la solution d’acétate d’éthyle filtrée dans une fiole à fond rond et retirer le solvant sur un évaporateur rotatif sous pression réduite pour obtenir de la masarimycine sous forme d’huile une fois que tout l’acétate d’éthyle est retiré.
- Dissoudre l’huile de masarimycine obtenue ci-dessus en une quantité minimale (1-2 mL) d’hexane 9:1: isopropanol et remuer sur une plaque magnétique jusqu’à ce que tout le composé soit dissous.
- Purifier la masarimycine dissoute par chromatographie flash à l’aide d’une colonne flash de silice en phase normale de 12 g.
- Équilibrez la colonne flash avec 10 volumes de colonne de phase mobile (hexane 99:1 : isopropanol) avec l’instrument réglé à un débit de 15 mL/min.
REMARQUE: Une fois l’équilibrage terminé, arrêtez le flux et déconnectez le haut de la colonne du système. - Prélever la masarimycine dissoute à l’aide d’une seringue de 5 ml. Connectez la seringue directement au sommet de la colonne flash équilibrée et injectez la solution dans la colonne. Reconnectez la colonne chargée au système de chromatographie flash et lancez l’élution du gradient.
- Eluter masarimycine de la colonne en utilisant l’élution de gradient à une concentration finale de phase mobile de 10:90 hexane: isopropanol sur 12 volumes de colonne. Surveiller l’élution de la masarimycine par absorption à 230 et 254 nm.
- Recueillir les composés élués de la colonne par un collecteur de fraction qui recueille 20 mL de solvant par fraction.
REMARQUE: Si un système de chromatographie flash n’est pas disponible, la purification de la masarimycine peut être effectuée via une colonne de silice gravitaire avec une phase mobile 3: 1 (hexane: acétate d’éthyle). Les fractions contenant de la masarimycine peuvent être identifiées par TLC en utilisant la même phase mobile. La visualisation des taches TLC a été effectuée avec une lumière UV, de la vapeur I2 ou une coloration au permanganate de potassium. - Identifier les fractions contenant de la masarimycine par TLC (étapes 2.5-2.9) ou spectrométrie de masse sur un spectromètre de masse compact équipé d’une sonde d’analyse des solides atmosphériques. Sécher le produit final sous vide (~0,3 mbar).
REMARQUE: La masarimycine est systématiquement obtenue sous forme d’huile incolore ou solide avec un rendement de 55% à 70% par rapport au mmol de cyclohexyl carboxaldéhyde ajouté à la réaction. Calculer le rendement final de la masarimycine en obtenant la masse de la masarimycine purifiée et en calculant le rendement théorique de la réaction en utilisant la formule suivante:
% de rendement = x 100 %
x 100 %
- Équilibrez la colonne flash avec 10 volumes de colonne de phase mobile (hexane 99:1 : isopropanol) avec l’instrument réglé à un débit de 15 mL/min.
- Confirmer la structure de la masarimycine par RMN.
- Dissoudre ~10 mg d’échantillon de masarimycine dans 0,5 mL de CDCl3. À l’aide d’un pipet Pasteur, transférer la solution dans un tube RMN de 5 mm et boucher le tube. Placez le tube RMN dans le spectromètre.
- Acquérir des spectres RMN 1H et 13C à l’aide d’expériences prédéfinies par le fabricant. Les affectations de décalage chimique et les spectres représentatifs sont fournis dans les figures supplémentaires 3 à 4.
- Conserver la masarimycine sèche ou dissoute dans du DMSO (concentration finale de 25 mM) à -20 °C jusqu’à utilisation.
3. Procédure micro-ondes pour la préparation de la masarimycine
- Préparer des solutions 0,6 M de cyclohexylamine, de cyclohexylcarboxaldéhyde, d’isocyanure de cyclohexyle et d’acide o-iodobenzoïque dans l’acétonitrile.
- Ajouter une barre d’agitation et 10 mL d’acétonitrile dans un flacon de réaction au micro-ondes en verre.
- Ajouter 2 mL de cyclohexylamine (0,6 M dans l’acétonitrile), 2 mL de cyclohexyl carboxaldéhyde (0,6 M dans l’acétonitrile) et 7 mL d’acétonitrile dans le flacon.
- Placez le flacon de réaction au micro-ondes dans le carrousel à micro-ondes. Remuez le mélange, chauffez-le pendant 30 min à 50°C à une puissance de 400 W et laissez-le refroidir à température ambiante.
- Ajouter 2 mL d’acide o-iodobenzoïque (0,6 M dans le méthanol) et 2 mL d’isocyanure de cyclohexyle (0,6 M dans l’acétonitrile) au flacon. Remuez le mélange, chauffez-le à 100 °C au micro-ondes pendant 40 min à une puissance de 400 W et laissez-le refroidir à température ambiante.
- Surveiller la progression de la réaction par TLC (hexane 90:10 : isopropanol) à l’aide de vapeurI2 après la fin de l’étape 3.5.
REMARQUE : Si la TLC montre que la réaction est incomplète (c.-à-d. plusieurs taches sur la TLC), replacez le flacon de réaction dans le micro-ondes et réglez les conditions de micro-ondes décrites à l’étape 3.5. - Une fois la réaction terminée, versez la solution dans une fiole à fond rond de 100 mL et évaporez-la à sec à l’aide d’un évaporateur rotatif.
- Suivez les étapes 2.6 à 2.16 ci-dessus pour terminer le bilan aqueux, la purification et la caractérisation de la masarimycine.
4. Essai de synergie et d’antagonisme
- Cultiver Streptococcus pneumoniae R6 sur des plaques de gélose de Mueller-Hinton (MH) contenant 5 % (v/v) de sang de mouton à 37 °C dans des conditions anaérobies. Dans toutes les expériences, utiliser des cellules de deuxième passage cultivées dans 5 mL de bouillon MH à 37 °C dans des conditions anaérobies jusqu’à ce que l’OD600 soit d’environ 0,4.
- Soumettre les inhibiteurs masarimycine et optochine à des dilutions en série 1:2 dans des solvants respectifs, les concentrations résultantes flanquant les valeurs minimales de concentration inhibitrice (CMI) de chaque inhibiteur.
- Effectuer la dilution initiale de la masarimycine dans du diméthylsulfoxyde (DMSO) jusqu’à ce qu’une concentration de 100 μM soit atteinte. À partir de ce point, faites des dilutions de masarimycine dans le bouillon MH. Préparer la solution mère d’optochine (3,5 mM) en dissolvant l’optochine disponible dans le commerce (voir tableau des matériaux) dans un bouillon stérile de MH.
REMARQUE: Les solutions mères de masarimycine ont été fabriquées à 25 mM en DMSO.
- Effectuer la dilution initiale de la masarimycine dans du diméthylsulfoxyde (DMSO) jusqu’à ce qu’une concentration de 100 μM soit atteinte. À partir de ce point, faites des dilutions de masarimycine dans le bouillon MH. Préparer la solution mère d’optochine (3,5 mM) en dissolvant l’optochine disponible dans le commerce (voir tableau des matériaux) dans un bouillon stérile de MH.
- À une plaque stérile de microtitrage de 96 puits, ajouter 2 aliquotes μL de chaque dilution optochine à chaque rangée de la plaque. Sur la même plaque, ajouter 2 aliquotes μL de chaque dilution de masarimycine à chaque colonne pour créer un tableau de concentrations d’optochine et de masarimycine sur la plaque (Figure 2).
- Ajouter un bouillon stérile de MH (93 μL) à chaque puits contenant les inhibiteurs ci-dessus. Inoculer les plaques de microtitrage avec 5 μL de culture (OD600 ~ 0,4) à partir de l’étape 4.1.
REMARQUE: L’inoculation de la plaque de 96 puits se fait généralement dans des conditions anaérobies dans un poste de travail anaérobie. Le volume final dans le puits est de 100 μL. - Cultiver des cultures pendant 18 h à 37 °C dans des conditions anaérobies, suivies de l’ajout de 30 μL de solution à 0,01 % (m/v) de sel de résazurine sodique. Incuber la plaque à température ambiante pendant 15 min pour permettre la formation et la stabilisation de la couleur.
REMARQUE: La solution de résazurine est préparée en dissolvant le composé dans de l’eau distillée et peut être conservée à 4 ° C pendant deux semaines maximum. - Lisez directement les valeurs de concentration de la plaque et attribuez la concentration d’inhibiteur la plus faible pour laquelle aucune croissance bactérienne n’est observée (couleur bleue) comme [X] (voir étape 4.7.1), c’est-à-dire la concentration inhibitrice la plus faible du médicament en présence du co-médicament.
REMARQUE: La croissance bactérienne positive est identifiée dans les puits par le colorant de résazurine qui devient rose. Les valeurs de CMI pour chaque médicament seul (c.-à-d. en l’absence de co-médicament) sont déterminées de la même manière à l’aide du test de CMI de résazurine35 avec chaque médicament séparément (figure supplémentaire 5). Les CMI chez S. pneumoniae sont de 7,8 μM et 15,85 μM pour la masarimycine et l’optochine, respectivement. - Déterminer la concentration inhibitrice fractionnaire (FIC) et l’indice FIC (FICI) à l’aide des équations suivantes.
- FIC= [X]/MICx, où [X] (à partir de l’étape 4.6) est la concentration inhibitrice la plus faible du médicament en présence du co-médicament, et MICx est la concentration inhibitrice la plus faible du médicament en l’absence du co-médicament.
- FICI = FICmasarimycine +antibiotique FIC
NOTE: FICI < 0,5 = synergique, 0,5 < FICI < 1 = additif, 1 < FICI < 4 = indifférent, FICI > 4 = antagoniste.
5. Étude morphologique
- Cultivez Bacillus subtilis 11774 sur des plaques de gélose Luria-Bertani (LB) (10 g/L de tryptone, 5 g/L d’extrait de levure et 5 g/L de NaCl) contenant 1,5 % de gélose Bacto à 37 °C. Dans toutes les expériences, utiliser des cellules de deuxième passage cultivées dans 5 mL de bouillon LB à 37 °C jusqu’à OD600 = 1. Cultivez S.pneumoniae de la même manière qu’à l’étape 4.1.
- Après avoir obtenu une densité de culture cellulaire avec OD600nm = 1 pour B. subtilis, ou OD600nm = 0,4 pour S. pneumoniae, ajouter la masarimycine à l’aide d’une pipette au tube de culture étiqueté " traité » à une concentration finale de 3,8 μM (0,75x MIC pour B.subtilis), ou 5,85 μM (0,75x MIC pour S.pneumoniae). Au deuxième tube de culture étiqueté « témoin », ajoutez un volume équivalent de DMSO.
- Pour B.subtilis, placer les échantillons dans un incubateur à 37 °C pendant 90 min en agitant à 150 tr/min. Pour S. pneumoniae, incuber les cellules sans trembler dans des conditions anaérobies.
- Après 90 min, fixer chimiquement les cultures dans un mélange 1:10 (v/v) de milieux de culture et de tampon de fixation (20 mM HEPES, 1% de formaldéhyde (pH 6,8)) à 4 °C pendant la nuit. Une fois la fixation terminée, appliquez 10 à 20 μL d’échantillons sur des lames de microscope en verre à l’aide d’une pipette et laissez-les sécher à l’air. Fixez les échantillons séchés à l’air en chauffant les lames de verre à l’aide d’un brûleur Bunsen.
- Après fixation à la chaleur, tacher les échantillons avec l’ajout de 100 μL de bleu de méthylène à 0,1 % (m/v) (solution dans de l’éthanol à 20 % (v/v)). Incuber les lames tachées pendant 10 min et laver l’excès de colorant avec dH2O. Ensuite, chauffer doucement les lames colorées à 60 ° C dans un four pendant 15-20 min pour amener les cellules à un plan focal commun.
- Scellez les échantillons colorés en plaçant un couvercle de microscope sur les cellules colorées. Ensuite, scellez les bords à l’aide de ciment à lame de microscope. Placez la lame de microscope scellée sur la scène du microscope et mettez l’image au point à un grossissement de 100x à l’aide de la microscopie à champ lumineux.
- Placez une goutte d’huile d’immersion sur la lame du microscope et mettez le champ de vision au point en utilisant un grossissement de 1000x. Acquérir des micrographies à l’aide d’une caméra fixée au microscope et à son logiciel associé. Obtenez des images à l’aide des paramètres de balance des blancs et d’ouverture automatiques du logiciel.
REMARQUE: Alternativement, les images peuvent être traitées à l’aide du logiciel open source ImageJ.
Résultats
La masarimycine est une petite molécule inhibitrice bactériostatique de B. subtilis et S. pneumoniae et il a été démontré qu’elle inhibe l’exo-action GlcNAcase LytG chez B. subtilis35,37 et cible la paroi cellulaire chez S. pneumoniae37. La masarimycine peut être préparée efficacement par la synthèse organique classique ou assistée par micro-ondes avec des rendements compris entre 55% et 7...
Discussion
La masarimycine est un inhibiteur bactériostatique micromolaire unique de la croissance de B. subtilis35 et de S. pneumoniae37 . Chez B. subtilis, il a été démontré que la masarimycine inhibe le GlcNAcase LytG35, tandis que la cible moléculaire précise dans la paroi cellulaire de S. pneumoniae n’a pas été identifiée37. La synthèse de la masarimycine à l’aide de la synthèse organique ...
Déclarations de divulgation
Reid, C. W. a une propriété intellectuelle impliquant des applications spécifiques de la masarimycine.
Remerciements
La recherche a été soutenue par la National Science Foundation sous le numéro de subvention 2009522. L’analyse RMN de la masarimycine a été soutenue par l’attribution d’un important programme d’instrumentation de recherche de la National Science Foundation sous le numéro de subvention 1919644. Toutes les opinions, constatations et conclusions, ou recommandations exprimées dans ce document sont celles des auteurs et ne reflètent pas nécessairement les points de vue de la National Science Foundation.
matériels
| Name | Company | Catalog Number | Comments |
| 2-Iodobenzoic acid | SIGMA-ALDRICH | I7675-25G | corrosive, irritant, light yellow to orange-brown powder |
| 2-Propanol | SIGMA-ALDRICH | 109827-4L | flammable, irritant, colorless liquid |
| Acetonitrile | SIGMA-ALDRICH | 34851-4L | flammable, irritant, colorless liquid |
| Aluminum backed silica plates | Sorbtech | 4434126 | silica gel XG F254 on aluminum backed plates |
| chloroform-d | SIGMA-ALDRICH | 151823-50G | solvent for NMR |
| Compact Mass Spectrometer | Advion-Interchim | Advion CMS | compact mass spectrometer equiped with APCI source and atmospheric solids analysis probe |
| Corning Costar 96 well flat bottom plates-sterile | fisher chemical | 07-200-90 | for synergy/antagonism assays |
| cover slips | fisher chemical | 12-547 | for microscopy |
| Cyclohexanecarboxaldehyde | CHEM-IMPEX INT'L INC. | 24451 | flammable, irritant, colorless to pink liquid |
| Cyclohexyl isocyanide | SIGMA-ALDRICH | 133302-5G | irritant, colorless liquid, extremly unpleasant odor |
| Cyclohexylamine | SIGMA-ALDRICH | 240648-100ML | corrosive, flammable, irritant, colorless liquid unless contaminated |
| Ethyl acetate | SIGMA-ALDRICH | 537446-4L | flammable, irritant, colorless liquid |
| flash silica cartridge (12g) | Advion-Interchim | PF-50SIHP-F0012 | pack of flash silica columns (12g) for purification of masarimycin |
| formaldehyde | SIGMA-ALDRICH | F8775-25ML | fixing agent for microscopy |
| HEPES | SIGMA-ALDRICH | H8651-25G | buffer for microscopy fixing solution |
| Hexane, mixture of isomers | SIGMA-ALDRICH | 178918-4L | environmentally damaging, flammable, irritant, health hazard, colorless liquid |
| High performance compact mass spectrometer | Advion | expression | Atmospheric Solids Analysis Probe (ASAP), low resolution |
| High Vac | eppendorf | Vacufuge plus | vacuum aided by centrifugal force and temperature |
| Hydrochloric acid | SIGMA-ALDRICH | 258148-2.5L | corrosive, irritant, colorless liquid |
| hydrochloric acid | SIGMA-ALDRICH | 320331-2.5L | strong acid |
| immersion oil | fisher chemical | 12-365-19 | for microscopy |
| Iodine, resublimed crystals | Alfa Aesar | 41955 | environmentally damaging, irritant, health hazard, dark grey/purple crystals |
| Mestre Mnova | MestreLab Research | software for processing NMR spectra | |
| Methanol | SIGMA-ALDRICH | 439193-4L | flammable, toxic, health hazard, colorless liquid |
| methylene blue | SIGMA-ALDRICH | M9140-25G | microscopy stain for staining cell walls |
| meuller-hinton agar plates + 5% sheep blood | fisher chemical | B21176X | growth media for Streptococcus pneumoniae |
| meuller-hinton broth | fisher chemical | DF0757-17-6 | growth media for Streptococcus pneumoniae |
| microscope slides | fisher chemical | 22-310397 | for microscopy |
| Microwave Synthesis Labstation | MILESTONE | START SYNTH | device that requires the ventilation of a fume hood, equipped with synthesis carousel |
| NMR tubes | SIGMA-ALDRICH | Z562769-5EA | 5mm NMR tubes 600 MHz |
| Nuclear Magnetic Resonance (NMR) | Bruker | Ascend 400 | large superconducting magnet (400MHz) |
| optochin | fisher chemical | AAB21627MC | ethylhydrocupreine hydrochloride |
| petrie plates | Celltreat | 229695 | for preparing agar plates for bacterial growth |
| Primo Star Bright field/Phase contrast Microscope with ERc5s camera | Zeiss | for morphology studies | |
| puriFlash | interchim | XS520plus | flash chromatography purification system |
| resazurin | SIGMA-ALDRICH | R7017-1G | for synergy/antagonism assays |
| Rotary Evaporator | Heidolph | Hei-VAP Value "The Collegiate" | solvent evaporator |
| Sodium bicarbonate | SIGMA-ALDRICH | S6014-500G | irritant, white powder |
| Sodium chloride | fisher chemical | S271-1 | crystalline, colorless |
| Sodium chloride | SIGMA-ALDRICH | S5886-500G | for growth of B.subtilis and preparation of LB media |
| Sodium sulfate | SIGMA-ALDRICH | 7985592-500G | anhydrous, granular, white |
| tryptone | fisher chemical | BP1421-500 | for growth of B.subtilis and preparation of LB media |
| Whitney DG250 Workstation | Microbiology International | DG250 | anaerobic workstation. Anaerobic gas mixture used: 5% hydrogen, 10% carbon dioxide, balance nitrogen |
| yeast extract | fisher chemical | BP1422-500 | for growth of B.subtilis and preparation of LB media |
| Zen Lite (blue) software | Zeiss | for acquiring micrographs |
Références
- Vollmer, W., Blanot, D., de Pedro, M. A. Peptidoglycan structure and architecture. FEMS Microbiology Review. 32 (2), 149-167 (2008).
- Munita, J. M., Bayer, A. S., Arias, C. A. Evolving resistance among Gram-positive pathogens. Clinical Infectious Diseases. 61, 48-57 (2015).
- Vollmer, W., Bertsche, U. Murein (peptidoglycan) structure, architecture and biosynthesis in Escherichia coli. Biochimica Biophysica Acta. 1778 (9), 1714-1734 (2008).
- Vollmer, W., Höltje, J. -. V. The architecture of the murein (peptidoglycan) in Gram-negative bacteria: vertical scaffold or horizontal layer(s). Journal of Bacteriology. 186 (18), 5978-5987 (2004).
- Clarke, A. J. Compositional analysis of peptidoglycan by high-performance anion-exchange chromatography. Analytical Biochemistry. 212 (2), 344-350 (1993).
- Kim, S. J., Chang, J., Singh, M. Peptidoglycan architecture of Gram-positive bacteria by solid-state NMR. Biochimica Biophysica Acta. 1848, 350-362 (2014).
- Koch, A. L., Doyle, R. J. Inside-to-outside growth and turnover of the wall of gram-positive rods. Journal of Theoretical Biology. 117 (1), 137-157 (1985).
- Beeby, M., Gumbart, J. C., Roux, B., Jensen, G. J. Architecture and assembly of the Gram-positive cell wall. Molecular Microbiology. 88 (4), 664-672 (2013).
- Shockman, G. D., Daneo-Moore, L., Kariyama, R., Massidda, O. Bacterial walls, peptidoglycan hydrolases, autolysins, and autolysis. Microbial Drug Resistance. 2 (1), 95-98 (1996).
- Dijkstra, A. J., Keck, W. Peptidoglycan as a barrier to transenvelope transport. Journal of Bacteriology. 178 (19), 5555-5562 (1996).
- Blackman, S. A., Smith, T. J., Foster, S. J. The role of autolysins during vegetative growth of Bacillus subtilis 168. Microbiology. 144, 73-82 (1998).
- Misra, G., Rojas, E. R., Gopinathan, A., Huang, K. C. Mechanical consequences of cell-wall turnover in the elongation of a Gram-positive bacterium. Biophysical Journal. 104 (11), 2342-2352 (2013).
- Wheeler, R., et al. Bacterial cell enlargement requires control of cell wall stiffness mediated by peptidoglycan hydrolases. mBio. 6 (4), 00660 (2015).
- Taguchi, A., Kahne, D., Walker, S. Chemical tools to characterize peptidoglycan synthases. Current Opinion in Chemical Biology. 53, 44-50 (2019).
- Welsh, M. A., Schaefer, K., Taguchi, A., Kahne, D., Walker, S. Direction of chain growth and substrate preferences of shape, elongation, division, and sporulation-family peptidoglycan glycosyltransferases. Journal of the American Chemial Society. 141 (33), 12994-12997 (2019).
- Rubino, F. A., et al. Detection of transport intermediates in the peptidoglycan flippase MurJ identifies residues essential for conformational cycling. Journal of the American Chemical Society. 142 (12), 5482-5486 (2020).
- Sjodt, M., et al. Structure of the peptidoglycan polymerase RodA resolved by evolutionary coupling analysis. Nature. 556 (7699), 118-121 (2018).
- Tiyanont, K., et al. Imaging peptidoglycan biosynthesis in Bacillus subtilis with fluorescent antibiotics. Proceedings of the National Academy of Science U S A. 103 (29), 11033-11038 (2006).
- Lebar, M. D., et al. Reconstitution of peptidoglycan cross-linking leads to improved fluorescent probes of cell wall synthesis. Journal of the American Chemical Society. 136 (31), 10874-10877 (2014).
- Do, T., Page, J. E., Walker, S. Uncovering the activities, biological roles, and regulation of bacterial cell wall hydrolases and tailoring enzymes. Journal of Biological Chemistry. 295 (10), 3347-3361 (2020).
- Liang, H., et al. Metabolic labelling of the carbohydrate core in bacterial peptidoglycan and its applications. Nature Communications. 8, 15015 (2017).
- DeMeester, K. E., et al. Metabolic incorporation of N-acetyl muramic acid probes into bacterial peptidoglycan. Current Protocol in Chemical Biology. 11 (4), 74 (2019).
- Lazor, K. M., et al. Use of Bioorthogonal N-acetylcysteamine (SNAc) analogues and peptidoglycan O-acetyltransferase B (PatB) to label peptidoglycan. The FASEB Journal. 32, 630 (2018).
- Wang, Y., Leimkuhler-Grimes, C. Fluorescent labeling of the carbohydrate backbone of peptidoglycan to track degradation in vivo. The FASEB Journal. 29, (2015).
- Kuru, E., et al. In probing of newly synthesized peptidoglycan in live bacteria with fluorescent D-amino acids. Angewandte Chemie International Edition. 51 (50), 12519-12523 (2012).
- Zhou, R., Chen, S., Recsei, P. A dye release assay for determination of lysostaphin activity. Analytical Biochemistry. 171 (1), 141-144 (1988).
- Qiao, Y., et al. Lipid II overproduction allows direct assay of transpeptidase inhibition by β-lactams. Nature Chemical Biology. 13 (7), 793-798 (2017).
- Lebar, M. D., et al. Forming cross-linked peptidoglycan from synthetic Gram-negative lipid II. Journal of the American Chemical Society. 135 (12), 4632-4635 (2013).
- Chen, R., Guttenplan, S. B., Blair, K. M., Kearns, D. B. Role of the D-dependent autolysins in Bacillus subtilis population heterogeneity. Journal of Bacteriology. 191 (18), 5775-5784 (2009).
- Yukie, S., Miki, K., Yoshio, N., Kuniaki, T., Yoshihisa, Y. Identification and characterization of an autolysin-encoding gene of Streptococcus mutans. Infection and Immunity. 73 (6), 3512-3520 (2005).
- Domenech, M., García, E., Moscoso, M. In vitro destruction of Streptococcus pneumoniae biofilms with bacterial and phage peptidoglycan hydrolases. Antimicrobial Agents and Chemotherapy. 55 (9), 4144-4148 (2011).
- Horsburgh, G. J., Atrih, A., Williamson, M. P., Foster, S. J. LytG of Bacillus subtilis is a novel peptidoglycan hydrolase: the major active glucosaminidase. Biochemistry. 42 (2), 257-264 (2003).
- Vermassen, A., et al. Cell wall hydrolases in bacteria: insight on the diversity of cell wall amidases, glycosidases and peptidases toward peptidoglycan. Frontiers in Microbiology. 10, 331 (2019).
- Martin-Galiano, A. J., Yuste, J., Cercenado, M. I., de la Campa, A. G. Inspecting the potential physiological and biomedical value of 44 conserved uncharacterised proteins of Streptococcus pneumoniae. BMC Genomics. 15, 652 (2014).
- Nayyab, S., et al. Diamide inhibitors of the Bacillus subtilis N-acetylglucosaminidase LytG that exhibit antibacterial activity. ACS Infectioius Diseases. 3 (6), 421-427 (2017).
- Lipski, A., et al. Structural and biochemical characterization of the β-N-acetylglucosaminidase from Thermotoga maritima: Toward rationalization of mechanistic knowledge in the GH73 family. Glycobiology. 25 (3), 319-330 (2014).
- Haubrich, B. A., et al. Inhibition of Streptococcus pneumoniae autolysins highlight distinct differences between chemical and genetic inactivation. bioRxiv. , 300541 (2020).
- Farha, M. A., et al. Inhibition of WTA synthesis blocks the cooperative action of PBPs and sensitizes MRSA to β-lactams. ACS Chemical Biology. 8 (1), 226-233 (2013).
- Lehár, J., et al. Chemical combination effects predict connectivity in biological systems. Molecular Systems Biology. 3 (1), 80 (2007).
- Farha, M. A., et al. Antagonism screen for inhibitors of bacterial cell wall biogenesis uncovers an inhibitor of undecaprenyl diphosphate synthase. Proceedings of the National Academy of Science U S A. 112 (35), 11048-11053 (2015).
- Palomino, J. C., et al. Resazurin microtiter assay plate: simple and inexpensive method for detection of drug resistance in Mycobacterium tuberculosis. Antimicrobial Agents and Chemotherapy. 46 (8), 2720-2722 (2002).
- Odds, F. C. Synergy, antagonism, and what the chequerboard puts between them. Journal of Antimicrobial Chemotherapy. 52 (1), 1 (2003).
- Arrigucci, R., Pozzi, G. Identification of the chain-dispersing peptidoglycan hydrolase LytB of Streptococcus gordonii. PLoS One. 12 (4), 0176117 (2017).
- Bai, X. -. H., et al. Structure of pneumococcal peptidoglycan hydrolase LytB reveals insights into the bacterial cell wall remodeling and pathogenesis. of Biological Chemistry. 289 (34), 23403-23416 (2014).
- Garcia, P., Gonzalez, M. P., Garcia, E., Lopez, R., Garcia, J. L. LytB, a novel pneumococcal murein hydrolase essential for cell separation. Molecular Microbiology. 31 (4), 1275-1281 (1999).
- Giladi, M., Altman-Price, N., Levin, I., Levy, L., Mevarech, M. FolM, a new chromosomally encoded dihydrofolate reductase in Escherichia coli. Journal of Bacteriology. 185 (23), 7015-7018 (2003).
- Chua, P. R., et al. Effective killing of the human pathogen Candida albicans by a specific inhibitor of non-essential mitotic kinesin Kip1p. Molecular Microbiology. 65 (2), 347-362 (2007).
- Rico-Lastres, P., et al. Substrate recognition and catalysis by LytB, a pneumococcal peptidoglycan hydrolase involved in virulence. Scientific Reports. 5, 16198 (2015).
- Vollmer, W., et al. The cell wall of Streptococcus pneumoniae. Microbiology Spectrum. 7 (3), (2019).
- Massidda, O., Nováková, L., Vollmer, W. From models to pathogens: how much have we learned about Streptococcus pneumoniae cell division. Environmental Microbiology. 15 (12), 3133-3157 (2013).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon