È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Sintesi di Masarimycin, un inibitore di piccole molecole della crescita batterica Gram-positiva
In questo articolo
Riepilogo
Viene presentato un protocollo dettagliato per la preparazione della diamide batteriostatica masarimicina, una sonda a piccola molecola che inibisce la crescita di Bacillus subtilis e Streptococcus pneumoniae mirando alla degradazione della parete cellulare. La sua applicazione come sonda chimica è dimostrata in saggi di sinergia/antagonismo e studi morfologici con B. subtilis e S. pneumoniae.
Abstract
Il peptidoglicano (PG) nella parete cellulare dei batteri è una struttura macromolecolare unica che conferisce forma e protezione dall'ambiente circostante. Fondamentale per comprendere la crescita e la divisione cellulare è la conoscenza di come la degradazione del PG influenza la biosintesi e l'assemblaggio della parete cellulare. Recentemente, è stata riportata l'etichettatura metabolica di PG attraverso l'introduzione di zuccheri o amminoacidi modificati. Mentre l'interrogazione chimica dei passaggi biosintetici con inibitori di piccole molecole è possibile, gli strumenti di biologia chimica per studiare la degradazione del PG da parte delle autolisine sono sottosviluppati. Le autolisine batteriche sono un'ampia classe di enzimi coinvolti nella degradazione strettamente coordinata del PG. Qui, viene presentato un protocollo dettagliato per la preparazione di una sonda a piccola molecola, masarimycin, che è un inibitore della N-acetilglucosaminidasi LytG nel Bacillus subtilis e del metabolismo della parete cellulare nello Streptococcus pneumoniae. Viene fornita la preparazione dell'inibitore tramite sintesi organica classica e assistita da microonde. Viene presentata la sua applicabilità come strumento per studiare la fisiologia Gram-positiva nei saggi biologici.
Introduzione
Il peptidoglicano (PG) è un polimero a maglie che delinea la forma e la struttura cellulare nei batteri Gram-positivi e Gram-negativi 1,2. Questo eteropolimero è una matrice di amminozuccheri reticolati da peptidi corti 3,4,5,6 con una spina dorsale composta da residui alternati di N-acetilglucosamina (GlcNAc) e acido N-acetilmuramico (MurNAc) legati a β-(1,4) (Figura 1)1. Attaccato alla porzione di lattilo C-3 di MurNAc è il peptide dello stelo. Il metabolismo della PG coinvolge un sistema strettamente coordinato di enzimi biosintetici e degradativi per incorporare nuovo materiale nella parete cellulare 7,8. La degradazione del PG viene effettuata da enzimi collettivamente denominati autolisine9 e ulteriormente classificati in base alla specificità del legame scisso. Le autolisine partecipano a molti processi cellulari tra cui la crescita cellulare, la divisione cellulare, la motilità, la maturazione PG, la chemiotassi, la secrezione proteica, la competenza genetica, la differenziazione e la patogenicità10,11. Svelare le funzioni biologiche specifiche delle singole autolisine può essere scoraggiante, in parte a causa della ridondanza funzionale. Tuttavia, recenti studi biofisici 8,12,13 e computazionali12 hanno fornito nuove informazioni sui loro ruoli nel metabolismo pg. Inoltre, recenti rapporti hanno fornito ulteriori informazioni sulla sintesi14 e sui 15,16,17 passaggi mediati dalla membrana nel metabolismo pg. Una conoscenza approfondita della relazione tra vie degradative e sintetiche del metabolismo pg potrebbe dare origine a bersagli antibiotici precedentemente non sfruttati.
Mentre ci sono stati progressi significativi nella metodologia per studiare la glicobiologia negli eucarioti, la glicobiologia batterica e, in particolare, il metabolismo PG non è avanzato a un ritmo simile. Gli attuali approcci chimici per studiare il metabolismo pg includono antibiotici marcati fluorescentemente18, sonde fluorescenti 19,20 e etichettatura metabolica 21,22,23,24. Questi nuovi approcci stanno fornendo nuovi modi per interrogare il metabolismo della parete cellulare batterica. Mentre alcune di queste strategie sono in grado di etichettare PG in vivo, possono essere specie-specifiche19, o funzionare solo in ceppi privi di una particolare autolisina25. Molte strategie di marcatura PG sono destinate all'uso con pareti cellulari isolate26 o con vie di biosintesi PG ricostituite in vitro 20,27,28. L'uso di antibiotici marcati con fluorescenza è attualmente limitato alle fasi biosintetiche e alla transpeptidazione18.
Le attuali conoscenze sulle autolisine batteriche e sul loro ruolo nel metabolismo della parete cellulare provengono da analisi genetiche e biochimiche in vitro 11,29,30,31,32. Mentre questi approcci hanno fornito una ricchezza di informazioni su questa importante classe di enzimi, decifrare il loro ruolo biologico può essere difficile. Ad esempio, a causa della ridondanza funzionale33, la cancellazione di un'autolisina nella maggior parte dei casi non comporta l'arresto della crescita batterica. Questo nonostante il loro ruolo implicito nella crescita e divisione cellulare 7,12. Un'altra complicazione è che la delezione genetica delle autolisine batteriche può dare origine a meta-fenotipi34. I metafenotipi derivano dalla complessa interazione tra la via interessata dalla delezione genetica e altre vie interconnesse. Ad esempio, un meta-fenotipo può insorgere attraverso un effetto diretto come la mancanza di un enzima o un effetto indiretto come un'interruzione dei regolatori.
Attualmente, ci sono solo pochi inibitori delle autolisine della glicosidasi come N-acetilglucosaminidasi (GlcNAcase) e N-acetilmuramidasi, che possono essere utilizzati come sonde chimiche per studiare la degradazione del PG. Per risolvere questo problema, la diamide masarimicina (precedentemente definita come fgkc) è stata identificata e caratterizzatacon 35 come inibitore batteriostatico della crescita del Bacillus subtilis che prende di mira il GlcNAcase LytG32 (Figura 1). LytG è un GlcNAcase36 eso-agente, membro del cluster 2 all'interno della famiglia 73 della glicosil idrolasi (GH73). È il principale GlcNAcase attivo durante la crescita vegetativa32. Per quanto ne sappiamo, la masarimicina è il primo inibitore di un GlcNAcase ad azione PG che inibisce la crescita cellulare. Ulteriori studi sulla masarimicina con Streptococcus pneumoniae hanno scoperto che la masarimicina probabilmente inibisce il metabolismo della parete cellulare in questo organismo37. Qui, la preparazione di masarimicina è segnalata per l'uso come sonda di biologia chimica per studiare la fisiologia negli organismi Gram-positivi B. subtilis e S. pneumoniae. Vengono presentati esempi di analisi morfologica del trattamento con concentrazione inibitoria sub-minima con masarimicina, nonché un test di sinergia/antagonismo. I saggi di sinergia e antagonismo con antibiotici con modalità d'azione ben definite possono essere un modo utile per esplorare le connessioni tra i processi cellulari 38,39,40.
Protocollo
1. Metodi generali
NOTA: Tutti i composti sono stati acquistati da fornitori standard e utilizzati senza ulteriore purificazione.
- Eseguire la cromatografia a strato sottile (TLC) su una piastra di alluminio prefigurata con gel di silice XG F254. Rileva le macchie sotto una lampada UV, immergendoti nella macchia di p-anisaldeide o esponendo al vapore I2 .
- Registra tutti gli spettri di risonanza magnetica nucleare (NMR) su uno spettrometro a 400 MHz.
NOTA: 1H-NMR e 13spettri C-NMR sono stati riferiti ai picchi di solvente residuo. Le costanti di accoppiamento sono date in [Hz] e gli spostamenti chimici in [ppm]. - Registrare gli spettri di spettrometria di massa a ionizzazione chimica a pressione atmosferica (APCI) di masarimicina su uno spettrometro di massa compatto dotato di una sonda di analisi dei solidi atmosferici.
2. Procedura generale per la preparazione della masarimicina
NOTA: eseguire i passaggi seguenti in una cappa aspirante.
- Preparare una soluzione di 0,1 M in metanolo di ciascun reagente: cicloesilammina, cicloesil carbossildeide, acido o-iodobenzoico e cicloesil isocianuro35.
ATTENZIONE: La cicloesilammina, l'isocianuro di cicloesile e la carbossaldeide cicloesile sono infiammabili. Possono causare corrosione cutanea e indurre tossicità orale, cutanea, respiratoria o riproduttiva. Tenere i composti lontani da fiamme libere, superfici calde e fonti di accensione. Indossare un'adeguata protezione della pelle e degli occhi, lavorare in un'area ben ventilata ed evitare l'inalazione di vapori o nebbia. Per la conservazione, tenere le bottiglie ben chiuse e conservarle in un luogo fresco e asciutto. Conservare cicloesil carbossialdeide in un essiccatore in un'atmosfera N2 . - Mescolare 5 mL di cicloesilammina (0,1 M di soluzione in metanolo) e 5 mL di cicloesil carbossilaldeide (0,1 M in metanolo) in un matraccio a fondo tondo tappato e mescolare la soluzione utilizzando una barra magnetica su una piastra agitata/calda per 30 minuti a 40 °C in un bagno di sabbia. Monitorare la temperatura utilizzando un termometro posto a circa 1 cm sotto la superficie della sabbia.
- Dopo 30 minuti, aggiungere 5 mL di isocianuro di cicloesile (soluzione da 0,1 M in metanolo) alla soluzione dal punto 2.2 e mescolare per altri 20 minuti a 50 °C. Infine, aggiungere 5 mL di acido o-iodobenzoico (soluzione da 0,1 M in metanolo) alla miscela di reazione e continuare a mescolare a 55 °C per 3-5 ore.
- Monitorare periodicamente l'andamento della reazione da parte di TLC circa ogni ora dopo che la miscela di reazione di cui sopra è stata agitata per 3 ore.
- Tagliare una striscia di 3 cm x 6 cm di piastra TLC con supporto in alluminio. Usando una matita #2, disegna una linea a circa 1 cm dal basso. Utilizzando un microcapillare di vetro, individuare circa 5 μL della miscela di reazione sulla piastra TLC e lasciarla asciugare.
- A un becher da 150 ml, aggiungere abbastanza fase mobile (esano 90:10: isopropanolo) per coprire il fondo del becher. Utilizzando un paio di pinzette, posizionare con attenzione la piastra TLC sopra nel becher assicurandosi che la piastra TLC entri nella fase mobile in modo uniforme. Coprire la parte superiore del becher con un pezzo di carta stagnola.
NOTA: assicurarsi che la fase mobile non copra la linea e il campione individuato. - Lasciare che la fase mobile risalga la piastra TLC fino a quando non si trova a circa 1 cm sotto la parte superiore della piastra. Rimuovere la piastra TLC e, utilizzando una matita, disegnare una linea che indichi la distanza percorsa dalla fase mobile. Lasciare asciugare la piastra TLC in una cappa aspirante.
- Una volta essiccato, posizionare la piastra TLC in un becher contenente una piccola quantità di solido I2 e coprire il becher con un pezzo di stagnola. Monitorare la TLC per lo sviluppo di macchie gialle/marroni. Una volta sviluppato, rimuovere la piastra TLC e contrassegnare la posizione delle macchie con una matita (Figura supplementare 1).
NOTA: Se2 macchie non sono contrassegnate, la macchia si dissiperà nel tempo. Le macchie possono anche essere visualizzate sulla piastra TLC mediante luce UV, colorazione di p-anisaldeide o colorazione del permanganato di potassio (vedere Informazioni supplementari). - Calcolate i valori Rf per tutti i punti visualizzati utilizzando la seguente formula:
Rf =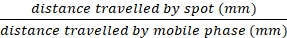
- Considerare la reazione completa quando solo un punto con Rf = 0,3 è visibile sulla piastra TLC. Rimuovere il solvente in un evaporatore rotativo a pressione ridotta e asciugare il prodotto grezzo (ottenuto come olio bruno-giallastro) sotto un alto vuoto fino a quando tutto il metanolo è evaporato.
- Sciogliere il prodotto greggio essiccato in 30 ml di acetato di etile e trasferirlo in un imbuto separatore. Estrarre l'acetato di etile in sequenza con 1 M HCl (2 x 30 mL), H2O (30 mL), soluzione satura di NaHCO3 (2 x 30 mL), H2O (30 mL) e soluzione di NaCl saturo (2 x 30 mL). Scartare gli strati acquosi.
NOTA: Lo strato di acetato di etile è lo strato superiore in ciascuna delle estrazioni. Per ogni estrazione, agitare vigorosamente l'imbuto separatore contenente l'acetato di etile e la soluzione acquosa (HCl, H2O, NaHCO3 o NaCl) e lasciare che gli strati si separino completamente. - Rimuovere lo strato di acetato di etile dall'imbuto separatore e raccoglierlo in un matraccio Erlenmeyer. Aggiungere una spatola piena di Na2SO4 (anidra) per rimuovere l'acqua residua dall'acetato di etile.
NOTA: La soluzione di acetato di etile è considerata secca quando Na2SO4 nel matraccio scorre liberamente e non si aggrega. Se Na2SO4 si aggrega, è possibile aggiungere una spatola aggiuntiva di Na2SO4 . - Filtrare la soluzione essiccata di acetato di etile attraverso la carta da filtro #1 per rimuovere Na2SO4. Lavare la carta da filtro con una piccola quantità di acetato di etile. Posizionare la soluzione filtrata di acetato di etile in un matraccio a fondo tondo e rimuovere il solvente su un evaporatore rotatorio a pressione ridotta per ottenere masarimicina come olio una volta rimosso tutto l'acetato di etile.
- Sciogliere l'olio di masarimicina ottenuto sopra in una quantità minima (1-2 ml) di esano 9:1: isopropanolo e mescolare su una piastra magnetica fino a quando tutto il composto è sciolto.
- Purificare la masarimicina disciolta mediante cromatografia flash utilizzando una colonna flash di silice in fase normale da 12 g.
- Equilibrare la colonna flash con 10 volumi di colonna di fase mobile (esano 99:1: isopropanolo) con lo strumento impostato ad una portata di 15 mL/min.
NOTA: al termine dell'equilibrio, arrestare il flusso e scollegare la parte superiore della colonna dal sistema. - Aspirare la masarimicina disciolta usando una siringa da 5 ml. Collegare la siringa direttamente alla parte superiore della colonna flash bilanciata e iniettare la soluzione nella colonna. Ricollegare la colonna caricata al sistema di cromatografia flash e avviare l'eluizione del gradiente.
- Elute masarimycin dalla colonna utilizzando l'eluizione gradiente ad una concentrazione di fase mobile finale di 10:90 esano: isopropanolo su 12 volumi di colonna. Monitorare l'eluizione di masarimicina attraverso l'assorbimento a 230 e 254 nm.
- Raccogliere i composti eluiti dalla colonna da un collettore di frazioni che raccoglie 20 ml di solvente per frazione.
NOTA: Se non è disponibile un sistema di cromatografia flash, la purificazione della masarimicina può essere eseguita tramite una colonna di silice gravitazionale con una fase mobile 3:1 (esano: acetato di etile). Le frazioni contenenti masarimicina possono essere identificate da TLC utilizzando la stessa fase mobile. La visualizzazione delle macchie TLC è stata effettuata con luce UV, vapore I2 o colorazione del permanganato di potassio. - Identificare le frazioni contenenti masarimicina mediante TLC (fasi 2.5-2.9) o spettrometria di massa su uno spettrometro di massa compatto dotato di una sonda di analisi dei solidi atmosferici. Asciugare il prodotto finale sotto vuoto (~0,3 mbar).
NOTA: La masarimicina è abitualmente ottenuta come olio incolore o solido con una resa del 55%-70% rispetto al mmol di cicloesil carbossialdeide aggiunto alla reazione. Calcola la resa finale della masarimicina ottenendo la massa della masarimicina purificata e calcolando la resa teorica della reazione usando la seguente formula:
% resa = x 100%
x 100%
- Equilibrare la colonna flash con 10 volumi di colonna di fase mobile (esano 99:1: isopropanolo) con lo strumento impostato ad una portata di 15 mL/min.
- Confermare la struttura della masarimicina mediante NMR.
- Sciogliere ~10 mg di campione di masarimicina in 0,5 ml di CDCl3. Utilizzando un pipetto Pasteur, trasferire la soluzione su un tubo NMR da 5 mm e tappare il tubo. Posizionare il tubo NMR nello spettrometro.
- Acquisisci spettri NMR 1H e 13C utilizzando esperimenti preimpostati dal produttore. Le assegnazioni dei turni chimici e gli spettri rappresentativi sono forniti nelle figure supplementari 3-4.
- Conservare la masarimicina asciutta o disciolta in DMSO (concentrazione finale di 25 mM) a -20 °C fino all'uso.
3. Procedura a microonde per la preparazione di masarimycin
- Preparare soluzioni 0,6 M di cicloesilammina, cicloesile carbossildeide, isocianuro di cicloesile e acido o-iodobenzoico in acetonitrile.
- Aggiungere una barra di agitazione e 10 ml di acetonitrile a un flaconcino di reazione a microonde di vetro.
- Aggiungere 2 mL di cicloesilammina (0,6 M in acetonitrile), 2 mL di cicloesil carbossildeide (0,6 M in acetonitrile) e 7 ml di acetonitrile al flaconcino.
- Posizionare il flaconcino di reazione a microonde nel carosello del microonde. Mescolare la miscela, riscaldarla per 30 minuti a 50°C con una potenza di 400 W e lasciarla raffreddare a temperatura ambiente.
- Aggiungere 2 mL di acido o-iodobenzoico (0,6 M in metanolo) e 2 mL di isocianuro di cicloesile (0,6 M in acetonitrile) al flaconcino. Mescolare la miscela, riscaldarla a 100 °C nel microonde per 40 minuti con una potenza di 400 W e lasciarla raffreddare a temperatura ambiente.
- Monitorare l'avanzamento della reazione mediante TLC (esano 90:10: isopropanolo) utilizzando il vapore I2 dopo il completamento del passaggio 3.5.
NOTA: se la TLC mostra che la reazione è incompleta (cioè più punti sulla TLC), riposizionare il flaconcino di reazione nel microonde e impostare le condizioni del microonde descritte al punto 3.5. - Una volta completata la reazione, versare la soluzione in un matraccio a fondo tondo da 100 ml ed evaporarla a secco utilizzando un evaporatore rotante.
- Seguire i passaggi 2.6-2.16 sopra per completare il workup acquoso, la purificazione e la caratterizzazione della masarimicina.
4. Analisi di sinergia e antagonismo
- Coltivare Streptococcus pneumoniae R6 su piastre di agar Mueller-Hinton (MH) contenenti il 5% (v/v) di sangue di pecora a 37 °C in condizioni anaerobiche. In tutti gli esperimenti, utilizzare cellule di secondo passaggio coltivate in 5 ml di brodo MH a 37 °C in condizioni anaerobiche fino a quando OD600 è ~ 0,4.
- Sottoporre gli inibitori masarimicina e optochina a diluizioni seriali 1:2 nei rispettivi solventi, con le concentrazioni risultanti che affiancano i valori minimi di concentrazione inibitoria (MIC) di ciascun inibitore.
- Effettuare la diluizione iniziale di masarimicina in dimetilsolfossido (DMSO) fino a raggiungere una concentrazione di 100 μM. Da questo punto, fare diluizioni di masarimicina in brodo MH. Preparare la soluzione madre di optochin (3,5 mM) sciogliendo l'optochina disponibile in commercio (vedere Tabella dei materiali) in brodo MH sterile.
NOTA: le soluzioni stock di Masarimycin sono state realizzate a 25 mM in DMSO.
- Effettuare la diluizione iniziale di masarimicina in dimetilsolfossido (DMSO) fino a raggiungere una concentrazione di 100 μM. Da questo punto, fare diluizioni di masarimicina in brodo MH. Preparare la soluzione madre di optochin (3,5 mM) sciogliendo l'optochina disponibile in commercio (vedere Tabella dei materiali) in brodo MH sterile.
- Su una piastra sterile di microtitolazione a 96 pozzetti, aggiungere aliquote da 2 μL di ciascuna diluizione optochina a ciascuna fila della piastra. Alla stessa piastra, aggiungere 2 aliquote μL di ogni diluizione di masarimicina a ciascuna colonna per creare una serie di concentrazioni di optochin e masarimycin sulla piastra (Figura 2).
- Aggiungere brodo MH sterile (93 μL) a ciascun pozzetto contenente gli inibitori di cui sopra. Inoculare le piastre di microtitolazione con 5 μL di coltura (OD600 ~ 0,4) dal punto 4.1.
NOTA: l'inoculazione della piastra a 96 pozzetti viene in genere eseguita in condizioni anaerobiche in una workstation anaerobica. Il volume finale nel pozzo è di 100 μL. - Coltivare colture per 18 ore a 37 °C in condizioni anaerobiche, seguite dall'aggiunta di 30 μL di soluzione allo 0,01% (m/v) di sale sodico di resazurina. Incubare la piastra a temperatura ambiente per 15 minuti per consentire la formazione e la stabilizzazione del colore.
NOTA: La soluzione di resazurina viene preparata sciogliendo il composto in acqua distillata e può essere conservata a 4 °C per un massimo di due settimane. - Leggere direttamente i valori di concentrazione dalla piastra e assegnare la più bassa concentrazione di inibitori per la quale non si osserva alcuna crescita batterica (colore blu) come [X] (vedere il punto 4.7.1), cioè la più bassa concentrazione inibitoria del farmaco in presenza del co-farmaco.
NOTA: La crescita batterica positiva è identificata nei pozzetti dal colorante resazurina che diventa rosa. I valori di MIC per ciascun farmaco da solo (cioè in assenza di co-farmaco) sono determinati in modo simile utilizzando il test resazurin MIC35 con ciascun farmaco separatamente (Figura supplementare 5). I MIC in S. pneumoniae sono 7,8 μM e 15,85 μM per masarimycin e optochin, rispettivamente. - Determinare la concentrazione inibitoria frazionaria (FIC) e l'indice FIC (FICI) utilizzando le seguenti equazioni.
- FIC= [X]/MICx, dove [X] (dal passo 4.6) è la più bassa concentrazione inibitoria del farmaco in presenza del co-farmaco, e MICx è la più bassa concentrazione inibitoria del farmaco in assenza del co-farmaco.
- FICI= FICmasarimicina +antibiotico FIC
NOTA: FICI < 0,5 = sinergico, 0,5 < FICI < 1 = additivo, 1 < FICI < 4 = indifferente, FICI > 4 = antagonista.
5. Studio morfologico
- Coltivare Bacillus subtilis 11774 su piastre di agar Luria-Bertani (LB) (10 g/L di triptone, 5 g/L di estratto di lievito e 5 g/L di NaCl) contenenti l'1,5% di Bacto agar a 37 °C. In tutti gli esperimenti, utilizzare cellule di secondo passaggio coltivate in 5 ml di brodo LB a 37 °C fino a OD600 = 1. Coltivare S.pneumoniae nello stesso modo del passaggio 4.1.
- Dopo aver ottenuto una densità di coltura cellulare con OD600nm = 1 per B. subtilis, o OD600nm = 0,4 per S. pneumoniae, aggiungere masarimycin usando una pipetta al tubo di coltura etichettato "trattato" ad una concentrazione finale di 3,8 μM (0,75x MIC per B.subtilis), o 5,85 μM (0,75x MIC per S.pneumoniae). Al secondo tubo di coltura etichettato "controllo", aggiungere un volume equivalente di DMSO.
- Per B.subtilis, posizionare i campioni in un incubatore a 37 °C per 90 minuti con agitazione a 150 giri/min. Per S. pneumoniae, incubare le cellule senza scuotere in condizioni anaerobiche.
- Dopo 90 minuti, fissare chimicamente le colture in una miscela 1:10 (v/v) di terreno di coltura e tampone di fissaggio (20 mM HEPES, 1% formaldeide (pH 6,8)) a 4 °C durante la notte. Al termine del fissaggio, applicare 10-20 μL di campioni su vetrini per microscopio di vetro utilizzando una pipetta e lasciarli asciugare all'aria. Fissare i campioni essiccati all'aria riscaldando i vetrini utilizzando un bruciatore Bunsen.
- Dopo il fissaggio a caldo, i campioni di colorazione con l'aggiunta di 100 μL di blu di metilene allo 0,1% (m/v) (soluzione in etanolo al 20% (v/v). Incubare i vetrini macchiati per 10 minuti e lavare via il colorante in eccesso con dH2O. Quindi, riscaldare delicatamente i vetrini macchiati a 60 ° C in un forno per 15-20 minuti per portare le celle su un piano focale comune.
- Sigillare i campioni macchiati posizionando un coprimicroscopio sulle cellule macchiate. Quindi, sigillare i bordi usando il cemento del vetrino del microscopio. Posizionare il vetrino del microscopio sigillato sul palco del microscopio e mettere a fuoco l'immagine con un ingrandimento di 100x utilizzando la microscopia a campo luminoso.
- Posizionare una goccia di olio per immersione sul vetrino del microscopio e mettere a fuoco il campo visivo utilizzando un ingrandimento di 1000x. Acquisire micrografie utilizzando una fotocamera collegata al microscopio e il relativo software associato. Acquisisci immagini utilizzando le impostazioni di bilanciamento automatico del bianco e apertura sul software.
NOTA: in alternativa, le immagini possono essere elaborate utilizzando il software open source ImageJ.
Risultati
Masarimycin è un inibitore batteriostatico a piccola molecola di B. subtilis e S. pneumoniae e ha dimostrato di inibire il GlcNAcase LytG eso-agente in B. subtilis35,37 e di colpire la parete cellulare in S. pneumoniae37. La masarimicina può essere preparata in modo efficiente sia dalla sintesi organica classica che da quella assistita da microonde con rese nell'intervallo 55%-70%. La sintesi assistita...
Discussione
Masarimycin è un singolo inibitore batteriostatico micromolare della crescita di B. subtilis35 e S. pneumoniae37 . In B. subtilis, masarimycin ha dimostrato di inibire il GlcNAcase LytG35, mentre il bersaglio molecolare preciso nella parete cellulare di S. pneumoniae non è stato identificato37. La sintesi di masarimicina utilizzando la classica sintesi organica o la procedura a microonde fornisce l...
Divulgazioni
Reid, C. W. ha proprietà intellettuale che coinvolge applicazioni specifiche di masarimycin.
Riconoscimenti
La ricerca è stata sostenuta dalla National Science Foundation con il numero di sovvenzioni 2009522. L'analisi NMR della masarimicina è stata supportata dal premio del programma di strumentazione di ricerca della National Science Foundation con il numero di sovvenzione 1919644. Eventuali opinioni, risultati e conclusioni o raccomandazioni espresse in questo materiale sono quelle degli autori e non riflettono necessariamente le opinioni della National Science Foundation.
Materiali
| Name | Company | Catalog Number | Comments |
| 2-Iodobenzoic acid | SIGMA-ALDRICH | I7675-25G | corrosive, irritant, light yellow to orange-brown powder |
| 2-Propanol | SIGMA-ALDRICH | 109827-4L | flammable, irritant, colorless liquid |
| Acetonitrile | SIGMA-ALDRICH | 34851-4L | flammable, irritant, colorless liquid |
| Aluminum backed silica plates | Sorbtech | 4434126 | silica gel XG F254 on aluminum backed plates |
| chloroform-d | SIGMA-ALDRICH | 151823-50G | solvent for NMR |
| Compact Mass Spectrometer | Advion-Interchim | Advion CMS | compact mass spectrometer equiped with APCI source and atmospheric solids analysis probe |
| Corning Costar 96 well flat bottom plates-sterile | fisher chemical | 07-200-90 | for synergy/antagonism assays |
| cover slips | fisher chemical | 12-547 | for microscopy |
| Cyclohexanecarboxaldehyde | CHEM-IMPEX INT'L INC. | 24451 | flammable, irritant, colorless to pink liquid |
| Cyclohexyl isocyanide | SIGMA-ALDRICH | 133302-5G | irritant, colorless liquid, extremly unpleasant odor |
| Cyclohexylamine | SIGMA-ALDRICH | 240648-100ML | corrosive, flammable, irritant, colorless liquid unless contaminated |
| Ethyl acetate | SIGMA-ALDRICH | 537446-4L | flammable, irritant, colorless liquid |
| flash silica cartridge (12g) | Advion-Interchim | PF-50SIHP-F0012 | pack of flash silica columns (12g) for purification of masarimycin |
| formaldehyde | SIGMA-ALDRICH | F8775-25ML | fixing agent for microscopy |
| HEPES | SIGMA-ALDRICH | H8651-25G | buffer for microscopy fixing solution |
| Hexane, mixture of isomers | SIGMA-ALDRICH | 178918-4L | environmentally damaging, flammable, irritant, health hazard, colorless liquid |
| High performance compact mass spectrometer | Advion | expression | Atmospheric Solids Analysis Probe (ASAP), low resolution |
| High Vac | eppendorf | Vacufuge plus | vacuum aided by centrifugal force and temperature |
| Hydrochloric acid | SIGMA-ALDRICH | 258148-2.5L | corrosive, irritant, colorless liquid |
| hydrochloric acid | SIGMA-ALDRICH | 320331-2.5L | strong acid |
| immersion oil | fisher chemical | 12-365-19 | for microscopy |
| Iodine, resublimed crystals | Alfa Aesar | 41955 | environmentally damaging, irritant, health hazard, dark grey/purple crystals |
| Mestre Mnova | MestreLab Research | software for processing NMR spectra | |
| Methanol | SIGMA-ALDRICH | 439193-4L | flammable, toxic, health hazard, colorless liquid |
| methylene blue | SIGMA-ALDRICH | M9140-25G | microscopy stain for staining cell walls |
| meuller-hinton agar plates + 5% sheep blood | fisher chemical | B21176X | growth media for Streptococcus pneumoniae |
| meuller-hinton broth | fisher chemical | DF0757-17-6 | growth media for Streptococcus pneumoniae |
| microscope slides | fisher chemical | 22-310397 | for microscopy |
| Microwave Synthesis Labstation | MILESTONE | START SYNTH | device that requires the ventilation of a fume hood, equipped with synthesis carousel |
| NMR tubes | SIGMA-ALDRICH | Z562769-5EA | 5mm NMR tubes 600 MHz |
| Nuclear Magnetic Resonance (NMR) | Bruker | Ascend 400 | large superconducting magnet (400MHz) |
| optochin | fisher chemical | AAB21627MC | ethylhydrocupreine hydrochloride |
| petrie plates | Celltreat | 229695 | for preparing agar plates for bacterial growth |
| Primo Star Bright field/Phase contrast Microscope with ERc5s camera | Zeiss | for morphology studies | |
| puriFlash | interchim | XS520plus | flash chromatography purification system |
| resazurin | SIGMA-ALDRICH | R7017-1G | for synergy/antagonism assays |
| Rotary Evaporator | Heidolph | Hei-VAP Value "The Collegiate" | solvent evaporator |
| Sodium bicarbonate | SIGMA-ALDRICH | S6014-500G | irritant, white powder |
| Sodium chloride | fisher chemical | S271-1 | crystalline, colorless |
| Sodium chloride | SIGMA-ALDRICH | S5886-500G | for growth of B.subtilis and preparation of LB media |
| Sodium sulfate | SIGMA-ALDRICH | 7985592-500G | anhydrous, granular, white |
| tryptone | fisher chemical | BP1421-500 | for growth of B.subtilis and preparation of LB media |
| Whitney DG250 Workstation | Microbiology International | DG250 | anaerobic workstation. Anaerobic gas mixture used: 5% hydrogen, 10% carbon dioxide, balance nitrogen |
| yeast extract | fisher chemical | BP1422-500 | for growth of B.subtilis and preparation of LB media |
| Zen Lite (blue) software | Zeiss | for acquiring micrographs |
Riferimenti
- Vollmer, W., Blanot, D., de Pedro, M. A. Peptidoglycan structure and architecture. FEMS Microbiology Review. 32 (2), 149-167 (2008).
- Munita, J. M., Bayer, A. S., Arias, C. A. Evolving resistance among Gram-positive pathogens. Clinical Infectious Diseases. 61, 48-57 (2015).
- Vollmer, W., Bertsche, U. Murein (peptidoglycan) structure, architecture and biosynthesis in Escherichia coli. Biochimica Biophysica Acta. 1778 (9), 1714-1734 (2008).
- Vollmer, W., Höltje, J. -. V. The architecture of the murein (peptidoglycan) in Gram-negative bacteria: vertical scaffold or horizontal layer(s). Journal of Bacteriology. 186 (18), 5978-5987 (2004).
- Clarke, A. J. Compositional analysis of peptidoglycan by high-performance anion-exchange chromatography. Analytical Biochemistry. 212 (2), 344-350 (1993).
- Kim, S. J., Chang, J., Singh, M. Peptidoglycan architecture of Gram-positive bacteria by solid-state NMR. Biochimica Biophysica Acta. 1848, 350-362 (2014).
- Koch, A. L., Doyle, R. J. Inside-to-outside growth and turnover of the wall of gram-positive rods. Journal of Theoretical Biology. 117 (1), 137-157 (1985).
- Beeby, M., Gumbart, J. C., Roux, B., Jensen, G. J. Architecture and assembly of the Gram-positive cell wall. Molecular Microbiology. 88 (4), 664-672 (2013).
- Shockman, G. D., Daneo-Moore, L., Kariyama, R., Massidda, O. Bacterial walls, peptidoglycan hydrolases, autolysins, and autolysis. Microbial Drug Resistance. 2 (1), 95-98 (1996).
- Dijkstra, A. J., Keck, W. Peptidoglycan as a barrier to transenvelope transport. Journal of Bacteriology. 178 (19), 5555-5562 (1996).
- Blackman, S. A., Smith, T. J., Foster, S. J. The role of autolysins during vegetative growth of Bacillus subtilis 168. Microbiology. 144, 73-82 (1998).
- Misra, G., Rojas, E. R., Gopinathan, A., Huang, K. C. Mechanical consequences of cell-wall turnover in the elongation of a Gram-positive bacterium. Biophysical Journal. 104 (11), 2342-2352 (2013).
- Wheeler, R., et al. Bacterial cell enlargement requires control of cell wall stiffness mediated by peptidoglycan hydrolases. mBio. 6 (4), 00660 (2015).
- Taguchi, A., Kahne, D., Walker, S. Chemical tools to characterize peptidoglycan synthases. Current Opinion in Chemical Biology. 53, 44-50 (2019).
- Welsh, M. A., Schaefer, K., Taguchi, A., Kahne, D., Walker, S. Direction of chain growth and substrate preferences of shape, elongation, division, and sporulation-family peptidoglycan glycosyltransferases. Journal of the American Chemial Society. 141 (33), 12994-12997 (2019).
- Rubino, F. A., et al. Detection of transport intermediates in the peptidoglycan flippase MurJ identifies residues essential for conformational cycling. Journal of the American Chemical Society. 142 (12), 5482-5486 (2020).
- Sjodt, M., et al. Structure of the peptidoglycan polymerase RodA resolved by evolutionary coupling analysis. Nature. 556 (7699), 118-121 (2018).
- Tiyanont, K., et al. Imaging peptidoglycan biosynthesis in Bacillus subtilis with fluorescent antibiotics. Proceedings of the National Academy of Science U S A. 103 (29), 11033-11038 (2006).
- Lebar, M. D., et al. Reconstitution of peptidoglycan cross-linking leads to improved fluorescent probes of cell wall synthesis. Journal of the American Chemical Society. 136 (31), 10874-10877 (2014).
- Do, T., Page, J. E., Walker, S. Uncovering the activities, biological roles, and regulation of bacterial cell wall hydrolases and tailoring enzymes. Journal of Biological Chemistry. 295 (10), 3347-3361 (2020).
- Liang, H., et al. Metabolic labelling of the carbohydrate core in bacterial peptidoglycan and its applications. Nature Communications. 8, 15015 (2017).
- DeMeester, K. E., et al. Metabolic incorporation of N-acetyl muramic acid probes into bacterial peptidoglycan. Current Protocol in Chemical Biology. 11 (4), 74 (2019).
- Lazor, K. M., et al. Use of Bioorthogonal N-acetylcysteamine (SNAc) analogues and peptidoglycan O-acetyltransferase B (PatB) to label peptidoglycan. The FASEB Journal. 32, 630 (2018).
- Wang, Y., Leimkuhler-Grimes, C. Fluorescent labeling of the carbohydrate backbone of peptidoglycan to track degradation in vivo. The FASEB Journal. 29, (2015).
- Kuru, E., et al. In probing of newly synthesized peptidoglycan in live bacteria with fluorescent D-amino acids. Angewandte Chemie International Edition. 51 (50), 12519-12523 (2012).
- Zhou, R., Chen, S., Recsei, P. A dye release assay for determination of lysostaphin activity. Analytical Biochemistry. 171 (1), 141-144 (1988).
- Qiao, Y., et al. Lipid II overproduction allows direct assay of transpeptidase inhibition by β-lactams. Nature Chemical Biology. 13 (7), 793-798 (2017).
- Lebar, M. D., et al. Forming cross-linked peptidoglycan from synthetic Gram-negative lipid II. Journal of the American Chemical Society. 135 (12), 4632-4635 (2013).
- Chen, R., Guttenplan, S. B., Blair, K. M., Kearns, D. B. Role of the D-dependent autolysins in Bacillus subtilis population heterogeneity. Journal of Bacteriology. 191 (18), 5775-5784 (2009).
- Yukie, S., Miki, K., Yoshio, N., Kuniaki, T., Yoshihisa, Y. Identification and characterization of an autolysin-encoding gene of Streptococcus mutans. Infection and Immunity. 73 (6), 3512-3520 (2005).
- Domenech, M., García, E., Moscoso, M. In vitro destruction of Streptococcus pneumoniae biofilms with bacterial and phage peptidoglycan hydrolases. Antimicrobial Agents and Chemotherapy. 55 (9), 4144-4148 (2011).
- Horsburgh, G. J., Atrih, A., Williamson, M. P., Foster, S. J. LytG of Bacillus subtilis is a novel peptidoglycan hydrolase: the major active glucosaminidase. Biochemistry. 42 (2), 257-264 (2003).
- Vermassen, A., et al. Cell wall hydrolases in bacteria: insight on the diversity of cell wall amidases, glycosidases and peptidases toward peptidoglycan. Frontiers in Microbiology. 10, 331 (2019).
- Martin-Galiano, A. J., Yuste, J., Cercenado, M. I., de la Campa, A. G. Inspecting the potential physiological and biomedical value of 44 conserved uncharacterised proteins of Streptococcus pneumoniae. BMC Genomics. 15, 652 (2014).
- Nayyab, S., et al. Diamide inhibitors of the Bacillus subtilis N-acetylglucosaminidase LytG that exhibit antibacterial activity. ACS Infectioius Diseases. 3 (6), 421-427 (2017).
- Lipski, A., et al. Structural and biochemical characterization of the β-N-acetylglucosaminidase from Thermotoga maritima: Toward rationalization of mechanistic knowledge in the GH73 family. Glycobiology. 25 (3), 319-330 (2014).
- Haubrich, B. A., et al. Inhibition of Streptococcus pneumoniae autolysins highlight distinct differences between chemical and genetic inactivation. bioRxiv. , 300541 (2020).
- Farha, M. A., et al. Inhibition of WTA synthesis blocks the cooperative action of PBPs and sensitizes MRSA to β-lactams. ACS Chemical Biology. 8 (1), 226-233 (2013).
- Lehár, J., et al. Chemical combination effects predict connectivity in biological systems. Molecular Systems Biology. 3 (1), 80 (2007).
- Farha, M. A., et al. Antagonism screen for inhibitors of bacterial cell wall biogenesis uncovers an inhibitor of undecaprenyl diphosphate synthase. Proceedings of the National Academy of Science U S A. 112 (35), 11048-11053 (2015).
- Palomino, J. C., et al. Resazurin microtiter assay plate: simple and inexpensive method for detection of drug resistance in Mycobacterium tuberculosis. Antimicrobial Agents and Chemotherapy. 46 (8), 2720-2722 (2002).
- Odds, F. C. Synergy, antagonism, and what the chequerboard puts between them. Journal of Antimicrobial Chemotherapy. 52 (1), 1 (2003).
- Arrigucci, R., Pozzi, G. Identification of the chain-dispersing peptidoglycan hydrolase LytB of Streptococcus gordonii. PLoS One. 12 (4), 0176117 (2017).
- Bai, X. -. H., et al. Structure of pneumococcal peptidoglycan hydrolase LytB reveals insights into the bacterial cell wall remodeling and pathogenesis. of Biological Chemistry. 289 (34), 23403-23416 (2014).
- Garcia, P., Gonzalez, M. P., Garcia, E., Lopez, R., Garcia, J. L. LytB, a novel pneumococcal murein hydrolase essential for cell separation. Molecular Microbiology. 31 (4), 1275-1281 (1999).
- Giladi, M., Altman-Price, N., Levin, I., Levy, L., Mevarech, M. FolM, a new chromosomally encoded dihydrofolate reductase in Escherichia coli. Journal of Bacteriology. 185 (23), 7015-7018 (2003).
- Chua, P. R., et al. Effective killing of the human pathogen Candida albicans by a specific inhibitor of non-essential mitotic kinesin Kip1p. Molecular Microbiology. 65 (2), 347-362 (2007).
- Rico-Lastres, P., et al. Substrate recognition and catalysis by LytB, a pneumococcal peptidoglycan hydrolase involved in virulence. Scientific Reports. 5, 16198 (2015).
- Vollmer, W., et al. The cell wall of Streptococcus pneumoniae. Microbiology Spectrum. 7 (3), (2019).
- Massidda, O., Nováková, L., Vollmer, W. From models to pathogens: how much have we learned about Streptococcus pneumoniae cell division. Environmental Microbiology. 15 (12), 3133-3157 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon