Method Article
Chemische Kartographie Ansätze zur Untersuchung der Trypanosomatideninfektion
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die Schritte zur Erstellung eines 3D-Modells der Metabolitenverteilung während der Trypanosomatideninfektion, einschließlich Probenentnahme, Metabolitenextraktion, überblick über die Datenerfassung von Flüssigkeitschromatographie-Tandem-Massenspektrometrie, 3D-Modellgenerierung und schließlich Datenvisualisierung.
Zusammenfassung
Pathogentropismus und Krankheitstropismus beziehen sich auf die Gewebestellen, die selektiv von Krankheitserregern besiedelt oder beschädigt werden, was zu lokalisierten Krankheitssymptomen führt. Zu den humaninfektiösen Trypanosomatidenparasiten gehören Trypanosoma cruzi, der Erreger der Chagas-Krankheit; Trypanosoma brucei, der Erreger der Schlafkrankheit; und Leishmania-Arten , Erreger der Leishmaniose. Zusammen betreffen sie 20 Millionen Menschen auf der ganzen Welt. Diese Parasiten zeigen einen spezifischen Tropismus: Herz, Speiseröhre, Dickdarm für T. cruzi, Fettgewebe, Bauchspeicheldrüse, Haut, Kreislaufsystem und Zentralnervensystem für T. brucei, Haut für dermotrope Leishmania-Stämme und Leber, Milz und Knochenmark für viscerotrope Leishmania-Stämme . Eine räumliche Perspektive ist daher unerlässlich, um die Pathogenese der Trypanosomatid-Krankheit zu verstehen. Die chemische Kartographie erzeugt 3D-Visualisierungen der Häufigkeit kleiner Moleküle, die durch Flüssigkeitschromatographie-Massenspektrometrie erzeugt werden, im Vergleich zu mikrobiologischen und immunologischen Parametern. Dieses Protokoll zeigt, wie die chemische Kartographie angewendet werden kann, um pathogene Prozesse während der Trypanosomatideninfektion zu untersuchen, beginnend mit der systematischen Gewebeentnahme und Metabolitenextraktion, gefolgt von der Erfassung von Daten zur Flüssigkeitschromatographie-Tandem-Massenspektrometrie und abschließend mit der Erstellung von 3D-Karten der Metabolitenverteilung. Diese Methode kann für mehrere Forschungsfragen verwendet werden, wie z.B. Nährstoffbedarf für die Gewebebesiedlung durch T. cruzi, T. brucei oder Leishmania, Immunmetabolismus an Infektionsorten und die Beziehung zwischen lokaler Gewebemetabolische Störung und klinischen Krankheitssymptomen, was zu einem umfassenden Einblick in die Pathogenese der Trypanosomatid-Krankheit führt.
Einleitung
Trypanosomatidenparasiten bestehen aus Leishmania-Arten, afrikanischen Trypanosomen (Trypanosoma brucei) und amerikanischen Trypanosomen (Trypanosoma cruzi). Leishmania protozoa verursachen Leishmaniose, die selbstheilende und selbstbegrenzte lokalisierte kutane Leishmaniose, mukokutane Leishmaniose, bei der das Schleimhautgewebe von Mund, Nase und Rachen geschädigt wird, und viszerale Leishmaniose mit Parasitentropismus zu den viszeralen Organen, die Fieber und Hepatosplenomegalie verursachen1,2 umfasst. T. brucei verursacht humane afrikanische Trypanosomiasis (HAT), auch bekannt als Schlafkrankheit, die hauptsächlich in afrikanischen Ländern berichtet wird3. Zu den klinischen Anzeichen und Symptomen gehören Hepatosplenomegalie, Fieber, Kopfschmerzen, Muskel-Skelett-Schmerzen, Lymphadenopathien und Anämie im hämo-lymphatischen Stadium, wenn Parasiten im Blutkreislauf und in der Lymphgefäße lokalisiert sind. Es folgt das meningo-enzephalitische Stadium, in dem Parasiten im zentralen Nervensystem lokalisiert sind und Schlafstörungen, Verhaltensänderungen und schließlich tödliches Komas verursachen4. T. cruzi verursacht die Chagas-Krankheit, die in Amerika endemisch ist. Infizierte Personen erleben ein anfängliches akutes Stadium, in der Regel asymptomatisch, mit breitem Parasitentropismus. Etwa 10%-30% der infizierten Personen erleben nach jahrzehntelanger Infektion chronische Symptome im Stadium, die durch Megaösophagus, Megakolon und kardiovaskuläre Komplikationen gekennzeichnet sind5,6.
Metabolomics untersucht kleine molekulare Spezies (50-1.500 Da), einschließlich biologischer Verbindungen aus dem primären oder sekundären Stoffwechsel und extern abgeleiteter Verbindungen wie Medikamente oder aus Lebensmitteln gewonnene Moleküle. Im Kontext von Wirt-Pathogen-Interaktionen kann die Metabolomik die Auswirkungen einer Infektion auf die Umgebungen der Wirtsmetaboliten untersuchen, was entscheidend für den Zugang zur Wirkung des Erregers auf den Wirt ist. Es kann auch die Anpassung von Krankheitserregern an die Ernährungs- und immunologische Umgebung des Wirts beurteilen7,8,9. Massenspektrometrie (MS) und Kernspinresonanzspektroskopie (NMR) sind gängige Metabolomik-Werkzeuge, die zur Identifizierung, Quantifizierung und Charakterisierung von Metaboliten verwendet werden. Dieser "Omics"-Ansatz kann auch auf die Entdeckung von Biomarkern und die Arzneimittelentwicklung angewendet werden10,11.
Angesichts des spezifischen Gewebetropismus von Trypanosomatiden-Parasiten können räumliche Metabolomik-Analysen einen signifikanten Einblick in die Pathogenese der von ihnen verursachten Krankheiten ermöglichen. Die Kartierung der räumlichen Verteilung der Metaboliten ergab Metaboliten, die lokal von einer chronischen Trypanosoma cruzi-Infektion im Herzgewebe der Maus und einer akuten und langfristigen Trypanosoma cruzi-Infektion im Magen-Darm-Trakt der Maus betroffen sind6,12,13. Insbesondere zeigte die chemische 3D-Kartographie eine Diskrepanz zwischen Parasitenpersistenz und metabolischen Veränderungen im Herzgewebe von chronisch Trypanosoma cruzi-infizierten Mäusen. Der Stoffwechsel war in den unteren und apikalen Segmenten des Herzens am stärksten gestört und stimmte mit den Stellen der Symptome der Chagas-Krankheit (herzapikale Aneurysmen) überein. Zu den Metabolitenfamilien, die durch eine Infektion an bestimmten kardialen Stellen gestört werden und mit dem Schweregrad der Erkrankung korrelieren, gehören Acylcarnitine und Glycerophosphocholine12,13,14. Im Magen-Darm-Trakt stimmten anhaltende Stoffwechselveränderungen mit den Symptomen der Chagas-Krankheit überein: Speiseröhre und Dickdarm. Im Gegensatz dazu ist der Stoffwechsel an Stellen, die nicht mit den Symptomen der Chagas-Krankheit assoziiert sind, wie dem Dünndarm, wieder normalisiert. Zu den Metaboliten, die lokal durch eine Infektion im Magen-Darm-Trakt gestört werden, gehören Acylcarnitine, Glycerophosphocholine, Kynurenin, Tryptophan und Cholsäure. Darüber hinaus ermöglichten diese Analysen die Identifizierung eines neuen metabolischen Mechanismus der Toleranz gegenüber der Chagas-Krankheit6. Die Anwendung dieser Methoden auf die Untersuchung der kutanen Leishmaniose zeigte signifikante metabolische Störungen an der Stelle der Läsion, aber auch spezifische metabolische Veränderungen im läsionsbenachbarten, makroskopisch gesunden Gewebe. Zum Beispiel wurde Glutamin an der Läsionsstelle erschöpft, während Glycerophosphocholine im m / z (Masse-Ladungs-Verhältnis) 200-299, 400-499, 500-599 und 600-699 an der Läsionsstelle signifikant erhöht waren. PC (O-34:1) war nur an läsionsbenachgebliebenen Stellen erhöht15.
Ziel dieses Manuskripts ist es, die Schritte zu demonstrieren, die notwendig sind, um 3D-Modelle der Metabolitenverteilung ("chemische Kartographie") zu generieren, wie sie auf Trypanosomatiden-Parasiteninfektionsmodelle angewendet werden (Abbildung 1). Dieser Ansatz baut auf mehreren kritischen Fortschritten im Zusammenhang mit der Metabolomik und der Metabolomik-Datenverarbeitung auf, insbesondere auf der Entwicklung einer Software zur Darstellung von Metabolomik-Daten auf 3D-Modellen16.
Protokoll
Alle beschriebenen Tierversuche wurden von der University of Oklahoma oder dem University of California San Diego Institutional Animal Care and Use Committee genehmigt. Alle Schritte im Umgang mit infektiösem Material wurden in einer Biosicherheitswerkbank (Klasse II, Typ A2) und gemäß den örtlichen Vorschriften durchgeführt.
1. Gewebeentnahme
- Infizieren Sie geeignete Trypanosomatiden-Infektionstiermodelle, im Allgemeinen Mäuse oder Hamster.
HINWEIS: Es gibt eine beträchtliche Vielfalt an Mausmodellen für trypanosomatiide Infektionen, abhängig von den gewünschten Symptomen, der Geschwindigkeit des Fortschreitens der Erkrankung, der Schwere der Erkrankung usw. Die Benutzer können nach Belieben wählen.- Für kutane Leishmaniose-Infektionsmodelle subkutan im Fußpolster oder intradermal im Ohr infizieren15.
- Bei viszeralen Leishmaniose-Infektionsmodellen intravenös infizieren1,17.
- Für Chagas-Krankheit und Schlafkrankheit Infektionsmodelle infizieren intraperitoneal6,12,18,19. Bestimmen Sie die zu verwendende Parasitendosis basierend auf dem Parasitenstamm und den geplanten Zeitpunkten.
- Planen Sie Schnittpositionen.
- Planen Sie, Schnitte mit mindestens 10 mg Gewebe pro Abschnitt zu erzeugen. Es ist am besten, 30-50 mg zu verwenden.
- Planen Sie für Infektionsmodelle der Chagas-Krankheit, Herz- und Magen-Darm-Segmente systematisch zu sammeln.
- Für kutane Leishmaniose-Infektionsmodelle sammeln Sie Läsionsgewebe und läsionsbenachbarte Proben.
- Für viszerale Leishmaniose-Infektionsmodelle planen Sie, Milz und mehrere Leberlappen zu sammeln. Zusätzliche Gewebestellen wie Fettgewebe können ebenfalls von Interesse sein.
VORSICHT: Proben von infizierten Tieren müssen im Rahmen des geeigneten, institutionell zugelassenen Biosicherheitsprotokolls behandelt werden. Dies beinhaltet in der Regel Anforderungen an die persönliche Schutzausrüstung (PSA) und nur das Öffnen von Röhrchen und das Sammeln von Proben in einer Biosicherheitswerkbank (Klasse II, Typ A2).
- Beschriften und wiegen Sie Tuben für die Homogenisierung.
- Verwenden Sie den für das verfügbare Homogenisierungssystem geeigneten Röhrchentyp. Verwenden Sie für einen TissueLyser 2 ml Mikrozentrifugenröhrchen.
- Einschläfern Sie Mäuse zu den gewünschten Infektionszeitpunkten unter Verwendung einer Isofluran-Überdosierung, wie von der institutionellen IACUC genehmigt oder gemäß dem von der IACUC genehmigten Protokoll.
- Schnittgewebe systematisch wie geplant, mit einem Schnitt pro Schlauch.
- Denken Sie daran, die Probenentnahmegeräte zwischen den Proben mit Extraktionslösungsmittel (50% Methanol in diesem Protokoll) zu waschen.
- Halten Sie den Röhrchendeckel offen, schnappen Sie sofort Gefrierproben in flüssigem Stickstoff ein.
VORSICHT: Verschließen Sie die Röhrchen erst, wenn flüssiger Stickstoff, der in die Röhre gelangt ist, vollständig verdampft ist, um zu verhindern, dass die Röhrchen explodieren, wenn sich Stickstoff ausdehnt. Ergreifen Sie angemessene Maßnahmen, um sicherzustellen, dass die Haut nicht mit flüssigem Stickstoff in Berührung kommt (verwenden Sie Kryohandschuhe und Pinzette, um die Schläuche zu halten). Tragen Sie einen Sicherheitsschutz. - Lagern Sie die Röhrchen auf Trockeneis, bis alle gewünschten Proben gesammelt wurden.
- Pausenpunkt: Lagern Sie die Proben bei -80 °C, bis sie für die Extraktion bereit sind.
- Wägen Sie Röhrchen, um das Gewicht der Gewebeprobe zu bestimmen. Datensatz in einer Tabelle.
- Bewahren Sie die Proben während des Wiegevorgangs gefroren auf: Röhrchen auf Trockeneis aufbewahren, schnell wiegen, sofort wieder auf Trockeneis legen. Lassen Sie die Proben während des Wiegens nicht auftauen.
2. Extraktion von Metaboliten
HINWEIS: Es dürfen nur Flüssigkeiten und Reagenzien in LC-MS-Qualität verwendet werden. Diese Methode wurde von Reference20 übernommen.
- Bereiten Sie alle Extraktionslösungsmittel (LC-MS-Klasse H2O, LC-MS-Methanol, gespickt mit 4 μM Sulfachlorpyridazin, LC-MS-Dichlormethan: Methanol mit 2 μM Sulfachlorpyridazin) in 1 L Glasflaschen unter Verwendung spezieller Glaswaren vor.
ANMERKUNG: Das aufzubereitende Volumen sollte auf der Grundlage von Probengewichten berechnet werden, wobei 500 μL Wasser pro 50 mg Probe, 500 μL Methanol mit 4 μM Sulfachlorpyridazin pro 50 mg Probe und 1.000 μL vorgefülltes Dichlormethan zu berücksichtigen sind: Methanol mit 2 μM Sulfachlorpyridazin pro 50 mg Probe, wodurch das berechnete Volumen um 10 % erhöht wird, um eine Ungenauigkeit des Pipettierens zu ermöglichen. Das Extraktionslösungsmittel mindestens über Nacht bei 4 °C lagern, um es vorzukühlen. - Führen Sie eine wasserbasierte Homogenisierung der Gewebeproben gemäß den unten genannten Schritten durch.
- Geben Sie eine 5-mm-Edelstahlperle (siehe Materialtabelle) mit einem Perlenspender zu jedem der 2 ml Mikrozentrifugenröhrchen, die Gewebeproben enthalten. Halten Sie die Röhren auf Eis.
- Stellen Sie ein leeres Röhrchen mit LC-MS-H2O her, das alle Schritte durchläuft und als Extraktionsrohling dient. Verwenden Sie das durchschnittliche H2O-Volumen aus Probenextraktionen. Geben Sie gekühlte LC-MS-Klasse H2O zu den gefrorenen Gewebeproben.
- Normalisieren Sie das Wasservolumen auf das Gewebegewicht durch Zugabe von 500 μL Wasser / 50 mg Probe unter Verwendung der in Schritt 1.7 berechneten Probengewichte.
VORSICHT: Wenn Sie mit biologisch gefährlichen Proben umgehen, befolgen Sie weiterhin das entsprechende, institutionell zugelassene Biosicherheitsprotokoll. Dies umfasst in der Regel Anforderungen an die persönliche Schutzausrüstung (PSA) und das Öffnen von Röhrchen nur innerhalb der Biosicherheitswerkbank (Klasse II, Typ A2).
- Normalisieren Sie das Wasservolumen auf das Gewebegewicht durch Zugabe von 500 μL Wasser / 50 mg Probe unter Verwendung der in Schritt 1.7 berechneten Probengewichte.
- Homogenisieren Sie Proben mit einer Geschwindigkeit von 25 Hz für 3 min mit einem Gewebehomogenisator (siehe Materialtabelle).
- Schließen Sie die Röhrchen fest, um ein Verschütten der Reagenzien während der Verarbeitung zu vermeiden.
- Sammeln Sie etwa 1/10 des Homogenisierungsvolumens für DNA-Extraktion, qPCR, proteinbasierte Analysen oder andere Analysen (falls gewünscht). In einem Mikrozentrifugenröhrchen oder einer 96-Well-Platte (je nach gesammeltem Volumen) bis zu 6 Monate bei -80 °C lagern, wenn DNA-Extraktionsexperimente an einem anderen Tag durchgeführt werden sollen. Längere Speicherdauern können möglich sein, wurden aber nicht getestet.
- Führen Sie DNA-Extraktionen mit einem handelsüblichen Kit für die DNA-Extraktion von Säugetieren (siehe Materialtabelle) aus Geweben durch, wie in Referenz13 beschrieben. Quantifizieren Sie die DNA-Ausbeute und lagern Sie die extrahierte DNA bei -20 °C. Eine akzeptable DNA-Quantität und -Qualität für qPCR wurde sogar bis zu 3 Jahre später beobachtet.
HINWEIS: Dieser Schritt kann an einem nachfolgenden Tag aus Metabolitenextraktion mit gefrorenem Homogenat durchgeführt werden. - Führen Sie qPCR wie in Referenz13 beschrieben durch, indem Sie 180 ng extrahierte DNA verwenden.
HINWEIS: Dies kann an DNA durchgeführt werden, die an einem früheren Tag gesammelt und bei -20 ° C eingefroren wurde.- Verwenden Sie im Falle von Studien zur T. cruzi-Infektion die folgenden Primer: ASTCGGCTGATCGTTTTCGA und AATTCCTCCAAGCAGCGGATA zur Quantifizierung der Parasitenspiegel21 und die folgenden Primer zur Normalisierung auf wirts-DNA-Spiegel: TCCCTCTCATCAGTTCTATTATCCCA und CAGCAAGCATCTATGCACTTAGACCCC22 (siehe Materialtabelle).
HINWEIS: Die empfohlenen qPCR-Zyklen sind wie folgt: Denatur bei 95 °C für 10 min; 40 Zyklen bei 95 °C für 30 s und dann 58 °C für 60 s und schließlich 72 °C für 60 s durchführen. Führen Sie eine Schmelzkurvenanalyse durch, die für den verfügbaren Thermocycler geeignet ist. Verarbeiten Sie Daten mit der ΔΔCt-Methode23, um die relative Parasitenlast zwischen den Probenahmestellen zu erhalten. Die absolute Quantifizierung kann erhalten werden, indem probenabgeleitete ΔΔCt-Werte mit einer Standardkurve verglichen werden, die aus bekannten Mengen von Parasiten erzeugt, in nicht infizierte Gewebeproben gespikt und wie in den Schritten 2.2 bis 2.2.4.1 extrahiert wird.
- Verwenden Sie im Falle von Studien zur T. cruzi-Infektion die folgenden Primer: ASTCGGCTGATCGTTTTCGA und AATTCCTCCAAGCAGCGGATA zur Quantifizierung der Parasitenspiegel21 und die folgenden Primer zur Normalisierung auf wirts-DNA-Spiegel: TCCCTCTCATCAGTTCTATTATCCCA und CAGCAAGCATCTATGCACTTAGACCCC22 (siehe Materialtabelle).
- Durchführung einer proteinbasierten Charakterisierung von Immunantworten unter Verwendung von multiplexierten Zytokin-Kits oder kommerziellen Standard-ELISA-Kits (siehe Materialtabelle), wie in Referenz13 auf dem gespeicherten Homogenat beschrieben.
- Führen Sie DNA-Extraktionen mit einem handelsüblichen Kit für die DNA-Extraktion von Säugetieren (siehe Materialtabelle) aus Geweben durch, wie in Referenz13 beschrieben. Quantifizieren Sie die DNA-Ausbeute und lagern Sie die extrahierte DNA bei -20 °C. Eine akzeptable DNA-Quantität und -Qualität für qPCR wurde sogar bis zu 3 Jahre später beobachtet.
- Sparen Sie mindestens die Hälfte der 500 μL des Homogenisierungsvolumens für die Metabolitenextraktion.
- Führen Sie eine wässrige Metabolitenextraktion durch.
HINWEIS: Die Lösungsmittelauswahl kann basierend auf den chemischen Eigenschaften der interessierenden Metaboliten angepasst werden.- Fügen Sie dem Homogenat eiskaltes Methanol in LC-MS-Qualität hinzu, das mit 4 μM Sulfachlorpyridazin versetzt ist, um eine Endkonzentration von 50% Methanol mit 2 μM Sulfachlorpyridazin in Wasser zu erreichen.
VORSICHT: Methanol ist brennbar und gefährlich. Verwenden Sie geeignete Sicherheitsverfahren, einschließlich der Handhabung in einem Abzug oder einer Biosicherheitswerkbank. - Homogenisieren Sie Proben in einem Gewebehomogenisator mit einer Geschwindigkeit von 25 Hz für 3 min. Zentrifugenproben bei 16.000 x g bei 4 °C für 10 min.
- Sammeln Sie ein gleiches Volumen an Überstand in eine 96-Well-Platte. Wählen Sie das Volumen, das dem kleinsten Volumen methanol + Wasser in allen Proben entspricht. Dies ist die wässrige Fraktion.
- Den verbleibenden wässrigen Homogenatüberstand bei -80 °C als Backup beiseite legen.
- Bewahren Sie die festen Rückstände auf Eis auf, während Sie die Überstände sammeln.
- Trockener wässriger Extraktionsüberstand bis zum Trocknen (~ 3 h oder über Nacht). Verwenden Sie maximale Geschwindigkeit und keine Heizung.
- Die getrocknete 96-Well-Platte bei -80 °C einfrieren.
- Fügen Sie dem Homogenat eiskaltes Methanol in LC-MS-Qualität hinzu, das mit 4 μM Sulfachlorpyridazin versetzt ist, um eine Endkonzentration von 50% Methanol mit 2 μM Sulfachlorpyridazin in Wasser zu erreichen.
- Führen Sie eine Extraktion organischer Metaboliten durch.
HINWEIS: Die Lösungsmittelauswahl kann basierend auf den chemischen Eigenschaften der interessierenden Metaboliten angepasst werden.- 1.000 μL je 50 mg der Probe des vorgekühlten Dichlormethans: Methanol, das mit 2 μM Sulfachlorpyridazin versetzt ist, in den festen Rückstand aus Schritt 2.3.4 gegeben.
VORSICHT: Verwenden Sie geeignete Sicherheitsverfahren beim Umgang mit Lösungsmitteln, einschließlich der Handhabung in einem Abzug mit einer guten Durchflussrate. - Homogenisieren Sie Proben in einem Gewebehomogenisator mit einer Geschwindigkeit von 25 Hz für 5 min. Zentrifugieren Sie die Proben bei 16.000 x g bei 4 °C für 10 min.
- Sammeln Sie ein gleiches Volumen an Überstand in eine 96-Well-Platte. Wählen Sie das Volumen, das dem kleinsten Volumen von Dichlormethan entspricht: Methanol, über alle Proben hinweg. Dies ist die organische Fraktion.
- Lagern Sie das Pellet bei -80 °C als Backup. Lagern Sie den restlichen organischen Extrakt bei -80 °C als Backup. Trocknen Sie den Bio-Extrakt über Nacht in einem Abzug an der Luft.
- Die getrocknete 96-Well-Platte bei -80 °C einfrieren.
- 1.000 μL je 50 mg der Probe des vorgekühlten Dichlormethans: Methanol, das mit 2 μM Sulfachlorpyridazin versetzt ist, in den festen Rückstand aus Schritt 2.3.4 gegeben.
3. LC-MS Datenerfassung
- Resuspend wässrige und organische Extrakte in jeweils 60 μL von 50% Methanol + 2 μM Sulfadimethoxin und kombinieren. Sonicate für 10 min; Dann zentrifugieren Sie für 10 min und übertragen Sie den Überstand auf eine saubere 96-Wellplate. Mit zonenfreier Plattendichtung versiegeln und die Platte in einen LC-Autosampler legen.
VORSICHT: Verwenden Sie geeignete Sicherheitsverfahren beim Umgang mit Lösungsmitteln, einschließlich der Handhabung in einem Abzug mit einer guten Durchflussrate. - Schließen Sie entsprechende mobile Phasen an das LC-System an (siehe Materialtabelle).
HINWEIS: Für positive Phase-Reversed-Phase-LC empfehlen die Autoren LC-MS-Grade H2O + 0,1% Ameisensäure als mobile Phase A und LC-MS-Grade Acetonitril + 0,1% Ameisensäure als mobile Phase B mit einer Durchflussrate von 0,5 ml / min und einem LC-Gradienten von 7,5 min. Empfohlene Gradientenschritte sind wie in Referenz6 veröffentlicht: 0-1 min, 2% B; 1-2,5 min, linearer Anstieg auf 98% B; 2,5-4,5 min, halten bei 98% B; 4,5-5,5 min, lineare Abnahme auf 2% B; 5,5-7,5 min, halten bei 2% B. - Stellen Sie sicher, dass das Instrument sauber ist. Kalibrieren Sie MS sowohl im positiven als auch im negativen Modus.
- Führen Sie eine MS-Leistungsbewertung durch, die für das Gerät geeignet ist.
- Erstellen Sie eine MS-Ausführungssequenz.
- Beginnen Sie mit 2 Leerproben, 2 Standards (6-Mischungen) und 5 gepoolten Qualitätskontrollen (QC) in einer Verdünnungsreihe, beginnend bei 2 μL Injektionsvolumen und schrittweise bis zu 30 μL Injektionsvolumen.
- Randomisieren Sie die Beispielreihenfolge.
- Führen Sie nach jeweils 12 Samples ein Leerzeichen und dann ein gepooltes QC aus.
- Schließen Sie die C8 LC-Säule (1,7 μm Partikelgröße, 100 Å Porengröße, 50 x 2,1 mm Länge x Innendurchmesser) an und überwachen Sie sie auf Leckagen und übermäßigen Gegendruck. Beheben Sie Probleme gemäß der Standardarbeitsanweisung des Instruments.
- Starten Sie die MS-Ausführungssequenz und sammeln Sie datenabhängige LC-MS/MS-Daten.
HINWEIS: Verwenden Sie für ein Q-Exactive Plus MS-Gerät beheizte Elektrospray-Ionisation und datenabhängige MS2-Erfassung (Top 5) im Positivenmodus, eine Auflösung von 70.000 für MS1 und 17.500 für MS2, AGC Target von 1E6 für MS1 und 2E5 für MS2, maximale IT von 100 ms für MS1 und MS2, Scanbereich bis 100-1500 m / z für MS1, und MS2-Isolationsfenster von 1 m/z. Stellen Sie das Mantelgas auf 35, das Auxgas auf 10 und das Kehrgas auf 0, die Sprühspannung auf 3,8 kV, die Kapillartemperatur auf 320 °C, den S-Linsen-HF-Pegel auf 50 und die Aux-Gastemperatur auf 350 °C.- Überprüfen Sie die Datenqualität: Überprüfen Sie die anfänglichen Leerzeichen (bestätigen Sie das Fehlen von Hauptspitzen), Standards (bestätigen Sie das Vorhandensein erwarteter Spitzen und der symmetrischen Peakform) und QCs (bestätigen Sie das Vorhandensein erwarteter Peaks, peak shape und erwarteter Peakintensität).
- Überwachen Sie regelmäßig die MS-Ausführung während der Ausführungssequenz.
- Sobald der Lauf beendet ist, überprüfen Sie die Daten auf verpasste Injektionen oder andere Fehler.
- Bewahren Sie die LC-Spalte wie vom Hersteller empfohlen auf. Proben entnehmen und bei -80 °C lagern.
- Laden Sie Rohdaten in das Datenrepository hoch.
ANMERKUNG: MassIVE (massive.ucsd.edu) wird empfohlen, um die nachgeschaltete Verbindung zur molekularen Vernetzung für Metabolitenannotationen zu ermöglichen24,25.
4. LC-MS Datenverarbeitung
- Konvertieren Sie RAW-Dateien mit MSConvert26 in ein offenes Format (.mzXML oder .mzML).
- Laden Sie Rohdaten und mzXML- oder mzML-Daten in das Datenrepository hoch.
- Feature-Tabelle generieren. Es gibt mehrere Tools, um dies zu tun (MZmine, MS-DIAL, openMS, XCMS, etc.27,28,29,30).
HINWEIS: MZmine wird empfohlen, da es kostenlos ist, Open-Source ist und mzXML-Dateien nach MSconvert direkt importieren kann, über grafische Benutzeroberflächenoptionen für die Verarbeitung von Daten und die Überwachung der Auswirkungen der Parameterauswahl verfügt und direkt in GNPS für molekulare Netzwerke exportieren kann24.
HINWEIS: Befolgen Sie die Werkzeugdokumentation und verwenden Sie die für das verfügbare Gerät geeigneten Parameter. Weitere Details finden Sie auch in Reference31.- Exportieren Sie die Feature-Tabelle in .csv Format.
5.3D Modellgenerierung
- Handzeichnen Sie ein 3D-Modell de novo, um es gemäß den unten genannten Schritten zu skalieren.
- Machen Sie ein Foto von dem Organ von Interesse.
- Führen Sie die folgenden Schritte in der SketchUp-Software (siehe Materialtabelle) wie unten beschrieben aus.
- Löschen Sie das Standardbild eines Mannes, das beim Öffnen der Software angezeigt wird.
- Klicken Sie auf Datei > Importieren , um ein Bild der interessierenden Organe zu importieren.
- Klicken Sie auf das Linien-Werkzeug und wählen Sie die Option Freihand . Verwenden Sie das Bleistiftwerkzeug, um die Umrisse der interessierenden Organe zu verfolgen und zu zeichnen. Stellen Sie sicher, dass Sie die Linie schließen, indem Sie den ganzen Weg zurück zum Ausgangspunkt zeichnen. Sobald die Linie erfolgreich geschlossen wurde, wird der gezeichnete Bereich automatisch schattiert angezeigt.
- Wählen Sie das Push/Pull-Werkzeug und ziehen Sie den schattierten Bereich nach oben, um die Zeichnung von 2D in 3D zu konvertieren.
- Organbild löschen: Wählen Sie die Schaltfläche Radiergummi , klicken Sie mit der rechten Maustaste auf das Bild und wählen Sie Löschen.
- Exportieren Sie die Datei im .dae-Format: Datei > exportieren Sie > 3D-Modell.
- Verbessern Sie den Realismus des Modells gemäß den unten genannten Schritten.
- Importieren Sie das Modell in die MeshLab-Software (siehe Materialtabelle): Öffnen Sie MeshLab und wählen Sie Datei > Netz importieren. Wählen Sie das im vorherigen Schritt generierte DAE-Modell aus. Ein Voröffner-Optionsmenü wird angezeigt. Wählen Sie OK aus.
- Wählen Sie im oberen Menü Wireframe aus. Wählen Sie dann: Filter > Remeshing, Vereinfachung und Rekonstruktion > Unterteilungsflächen: Mittelpunkt. Behalten Sie alle Werte als Standard bei, und wählen Sie Zweimal Anwenden aus. Überprüfen Sie die Drahtmodellansicht des Modells visuell, um sicherzustellen, dass es fein gerastert ist. Schließen Sie das Einblendmenü.
- Exportieren Sie das Modell im STL-Format: Datei > Netz exportieren als, und wählen Sie dann im Dropdown-Menü Dateityp die Option STL-Dateiformat (*.stl) aus. Klicken Sie auf Speichern. Wählen Sie im nächsten Einblendmenü die Option "OK".
- Öffnen Sie die Meshmixer-Software (siehe Materialtabelle). Klicken Sie auf die Schaltfläche Importieren (+). Wählen Sie die im vorherigen Schritt generierte STL-Datei aus. Verwenden Sie die Werkzeuge "Sculpt > Brushes > Drag und Sculpt > Brushes > "Inflate ", um die Oberflächen des Modells herauszuziehen, die abgerundet werden müssen.
- Sobald das Modell das gewünschte Aussehen hat, speichern Sie es im .stl-Format: Datei > Exportieren. Benennen Sie die Datei wie gewünscht und wählen Sie im Dropdown-Menü Dateityp die Option STL-Binärformat (*.stl) aus. Klicken Sie auf Speichern. Wenn ein Popup-Menü angezeigt wird, klicken Sie auf Weiter.
6. Generierung von Grundstücken
- Rufen Sie Koordinaten für die Positionen im 3D-Modell ab, die den Probenahmestellen entsprechen.
- Öffnen Sie das 3D-Modell aus Schritt 5.1.3.5 in der MeshLab-Software: Datei > Mesh importieren. Wählen Sie das in Schritt 5.1.3.5 generierte Modell aus. Klicken Sie im Popup-Fenster Post-Open-Verarbeitung auf OK.
- So rufen Sie x-, y- und z-Koordinaten für jeden Stichprobenpunkt ab: Wählen Sie das Auswahlpunkt-Werkzeug aus, und klicken Sie dann mit der rechten Maustaste in regelmäßigen Abständen über die 3D-Modelloberfläche. Sobald alle gewünschten Koordinaten ausgewählt wurden, klicken Sie im Popup-Fenster Formular auf die oberste Schaltfläche Speichern.
HINWEIS: Dadurch werden die Koordinaten im PP-Dateiformat exportiert. Diese Datei kann in einer Tabellenkalkulationssoftware geöffnet werden. - In Tabellenkalkulationssoftware: Öffnen Sie die in Schritt 6.1.2 generierte PP-Datei. Passen Sie die Datenanzeige mithilfe von > Text in Spalten > Trennzeichen an. Klicken Sie auf Weiter, wählen Sie dann Leertaste und dann Fertig stellen aus.
- Formatieren Sie es so, dass nur numerische Werte in den Tabellenkalkulationszellen verbleiben, indem Sie Home > Suchen & Auswählen > Ersetzen auswählen. Geben Sie im Feld Suchen nach Folgendes Folgendes ein: y=". Lassen Sie das Feld Ersetzen durch leer. Klicken Sie auf Alle ersetzen und dann auf OK. Wiederholen Sie dies für x=" und für z=" und für " />. Werte sind jetzt bereit für Schritt 6.2.
- Erstellen Sie die 'ili Feature-Tabelle. Diese Methode wurde von Reference16 übernommen.
- Erstellen Sie in einer Tabellenkalkulationssoftware die Featuretabelle. Zeilen entsprechen jeder Position und Spalten Daten.
HINWEIS: Die ersten Spalten müssen der Positionsname (Stichprobenname) sein, gefolgt von x-, y- und z-Koordinaten der Stichprobenpunkte, die in Schritt 6.1.2 (mit Spaltenüberschriften x, y, z) erhalten wurden. Die fünfte Spalte muss den Titel "radius" tragen. - Fügen Sie die entsprechenden Metadaten und die Häufigkeit der Metabolitenfunktionen in die nachfolgenden Tabellenkalkulationsspalten ein.
- Geben Sie in der Spalte "Radius" die gewünschte Größe der Probenahmepunkte ein, die im Modell visualisiert werden sollen. Ermitteln Sie die Werte für Radius empirisch: Geben Sie standardmäßig 1 ein und bewerten Sie dann, ob Radius oder Koordinaten in Schritt 6.3 angepasst werden müssen. Speichern Sie die Datei in .csv Format (Datei > Speichern unter) und wählen Sie dann im Dropdown-Menü CSV (durch Kommas getrennt) (*.csv) aus. Benennen Sie die Datei wie gewünscht. Klicken Sie auf Speichern.
- Erstellen Sie in einer Tabellenkalkulationssoftware die Featuretabelle. Zeilen entsprechen jeder Position und Spalten Daten.
- Öffnen Sie die Daten in 'ili (Software entwickelt von16).
- Öffnen Sie die 'ili Website (ili.embl.de). Wählen Sie Fläche aus. Ziehen Sie das erstellte 3D-Modell per Drag & Drop in das Browserfenster. Ziehen Sie die erstellte Feature-Tabelle per Drag & Drop in dasselbe Browserfenster.
- Verwenden Sie die Legende in der unteren rechten Ecke, um die gewünschte Datenspalte auf das 3D-Modell zu projizieren. Stellen Sie sicher, dass die in Schritt 6.2.1 ausgewählten Punkte und Radien mit den Probenahmestellen übereinstimmen. Passen Sie bei Bedarf die Werte in der Feature-Tabelle an oder wählen Sie zusätzliche Koordinaten im MeshLab aus (siehe Schritt 6.1.2).
HINWEIS: Die Visualisierung kann auch verbessert werden, indem Sie die > Rahmendeckkraft auswählen und den Schieberegler auf seinen Maximalwert einstellen. - Wählen Sie nacheinander jede Datenspalte aus, um die Verteilung dieses Metabolitenmerkmals im 3D-Modell zu bewerten.
HINWEIS: Dieser Ansatz kann auch verwendet werden, um nur bestimmte Metabolitenmerkmale von Interesse zu visualisieren, z. B. solche mit p < 0,05 oder einem bestimmten Faltenwechsel zwischen infizierten und nicht infizierten Proben, wie in externen statistischen Analysewerkzeugen bestimmt. Zu visualisierende Funktionen können auch außerhalb von 'ili durch maschinelle Lernansätze wie einen Random Forest ausgewählt werden.
- Führen Sie eine lineare / logarithmische Datenvisualisierung gemäß den unten genannten Schritten durch.
- Wählen Sie auf der Registerkarte Zuordnung oben rechts auf der Seite im Dropdown-Menü Skalieren die Option Linear oder Logarithmisch aus.
- Legen Sie den gleichen Maßstab für alle Plots fest, wenn Sie mehrere Features visualisieren.
HINWEIS: Die Website wählt automatisch das Minimum und Maximum für jede anzuzeigende Datenspalte aus. - Um die Skalierung manuell einzustellen, deaktivieren Sie die Option Auto Min/Max und geben Sie die gewünschte Skalierung ein. Alle Daten werden nun innerhalb derselben Skala angezeigt.
- Ändern Sie die Farbskala der Daten (Graustufen, Blau-Weiß-Rot usw.).
- Ändern Sie die Farbskala im Menü Zuordnung über das Dropdown-Menü Farbkarte in das gewünschte Farbschema. Stellen Sie sicher, dass das ausgewählte Farbschema farbenblind ist.
- Um die Farbe des 3D-Modells oder der Hintergrundfarbe zu ändern, verwenden Sie die Optionen Farbe und Hintergrund im Menü 3D.
- Blenden Sie 3D-Achsen aus, indem Sie im 3D-Menü das Feld Ursprung anzeigen deaktivieren.
- Speichern Sie das Bild durch Screenshots, mit dem Snipping-Tool oder der Tastenkombination Crtl + S für Windows oder Linux und
 + S unter OS X.
+ S unter OS X.
Ergebnisse
Die Anzahl der erhaltenen Metabolitenmerkmale hängt vom analysierten Gewebetyp und den Datenverarbeitungsparametern ab. Zum Beispiel wurde dieses Protokoll verwendet, um die räumlichen Auswirkungen der T. cruzi-Infektion auf das Metabolom des Magen-Darm-Trakts in einem Mausmodell der T. cruzi-Infektion zu analysieren. In früheren Arbeiten wurden männlichen C3H/HeJ intraperitoneal 1.000 CL + luc T. cruzi Parasiten injiziert32,6. Die Tiere wurden 12 oder 89 Tage nach der Infektion eingeschläfert, und eine chemische Kartographieanalyse von 13 zusammenhängenden Segmenten des Magen-Darm-Traktes wurde wie in diesem Protokoll beschrieben durchgeführt. Diese Analyse führte zu einer Feature-Tabelle mit 5.502 Features, die dann mit den in diesem Protokoll beschriebenen Schritten in 3D visualisiert wurden. Dieser Ansatz ermöglicht die Visualisierung von Metabolitenmerkmalen bei einzelnen Tieren, die an der Stelle hoher Parasitenlast hoch sind (Kynurenin, Abbildung 2B vs. Parasitenlast, Abbildung 2A), von Metaboliten mit differentieller Verteilung über Geweberegionen (Glutamin, Abbildung 2C) und Metabolitenmerkmalen, die in vergleichbaren Konzentrationen über Dünn- und Dickdarm gefunden werden (LPE 16:0 Abbildung 2D). ). Kynurenin wurde aufgrund seiner bekannten Beziehung zur Entzündung und früherer Veröffentlichungen über die Fähigkeit von Kynurenin-abgeleiteten Metaboliten, die T. cruzi-Last zu regulieren, für die Visualisierung ausgewählt33. Random-Forest-basierte Machine-Learning-Modelle hatten zuvor einen Zusammenhang zwischen Kynureninspiegeln und Infektionsstatus6 aufgedeckt. Glutamin wurde für eine Visualisierung ausgewählt, die auf früheren Publikationen basiert, die einen Zusammenhang zwischen der Verfügbarkeit von In-vitro-Glutamin und der Sensitivität des T. cruzi-Medikaments zeigen34. Die Differentialverteilung wurde mit Hilfe der logistischen Regression, p < 0,05, bestätigt. LPE 16:0 wurde nach visueller Inspektion der Daten ausgewählt, um Metabolitenmerkmale zu entdecken, die in vergleichbaren Konzentrationen an Gewebestellen gefunden wurden.
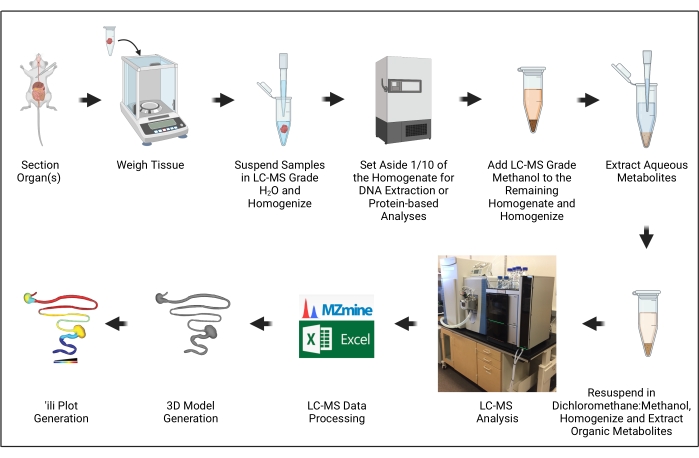
Abbildung 1: Protokollübersicht. Die Illustration wurde mit BioRender.com erstellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
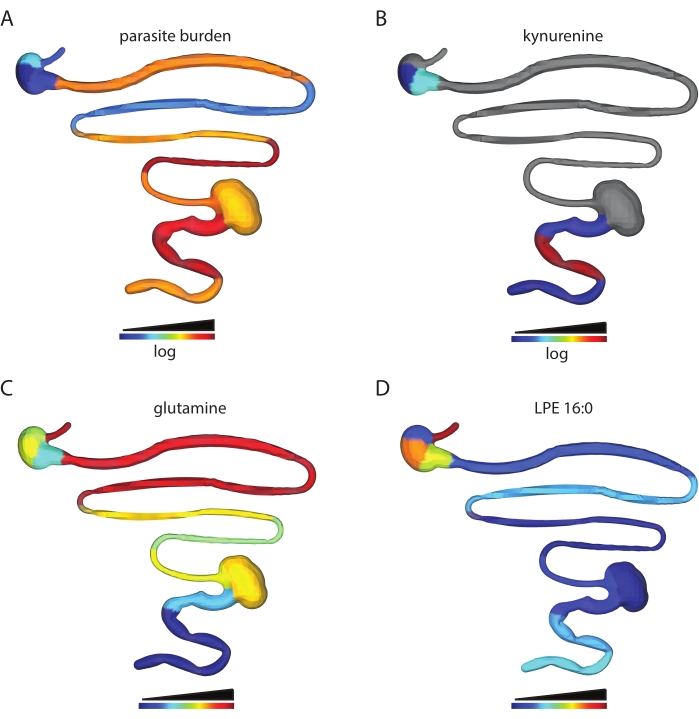
Abbildung 2: Chemische Kartographieanalyse. Männlichen C3H/HeJ-Mäusen wurden intraperitoneal 1.000 CL+luc T. cruzi Parasiten injiziert32. Die Tiere wurden 12 oder 89 Tage nach der Infektion eingeschläfert, und der Magen-Darm-Trakt wurde systematisch entnommen und abschnittiert (Schritt 1)6. Die Metaboliten wurden wie in Schritt 2 extrahiert und von LC-MS/MS analysiert.3D Modellgenerierung wurde mit der SketchUp-Software durchgeführt (Schritt 5), und die Daten wurden in 3D wie in Schritt 6 dargestellt. (A) Parasitenverteilung in einer bestimmten Maus, 12 Tage nach der Infektion. (B) Verteilung des Kynurenin-Metaboliten in derselben Maus, 12 Tage nach der Infektion. (C) Mittlere Glutaminverteilung über infizierte Mäuse, 89 Tage nach der Infektion. (D) Vergleichbare Mengen an m/z 454,292 Retentionszeit 2,929 min, annotiert als 2-Hexadecanoyl-sn-glycero-3-phosphoethanolamin (LPE 16:0), in derselben Maus wie in A und B im Dünndarm und Dickdarm. Stichproben und Daten wurden in6 generiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Das Verständnis von Trypanosomatid-Infektionen ist unerlässlich, um neuartige Arzneimittelentwicklungs- und Behandlungsansätze zu leiten. Diese chemische Kartographiemethode ist einzigartig aufgestellt, um umsetzbare Einblicke in die Beziehung zwischen Stoffwechsel und Trypanosomatid-Krankheitspathogenese zu liefern und so diesen translationalen Bedarf zu decken.
Bei der Metabolitenextraktion und ms-Analysen werden nur Lösungsmittel in LC-MS-Qualität empfohlen, um die Hintergrundkontamination zu verringern. Polymere Verunreinigungen35, die üblicherweise aus Paraffinfolien und/oder anderen Kunststoffen stammen36,37,38, sind nach Möglichkeit zu vermeiden. Insbesondere Parafilm darf niemals verwendet werden. Diese Aspekte sind von entscheidender Bedeutung, da die LC-MS-Datenqualität von den Materialien abhängt, die bei der Probenvorbereitung und Metabolitenextraktion verwendet werden. Die Datenqualität sollte vor der Erstellung von "ili-Plots" sichergestellt werden. Darüber hinaus erfordert die Erstellung dieser umfassenden räumlichen Metabolomik-Karten die Sammlung aller benachbarten Gewebeproben und die Extraktion von Metaboliten aus allen gesammelten Proben, um Lücken in diesen Karten zu vermeiden. Die Sammelverfahren, die Logistik der Metabolitenextraktion und die LC-MS-Analyse sowie die Kosten sollten daher entsprechend berücksichtigt und geplant werden.
Dieses Protokoll kann auf verschiedene Arten geändert werden, um die Benutzeranforderungen zu erfüllen. Beispielsweise beeinflussen die Polarität und Löslichkeit von Lösungsmitteln, die während der Metabolitenextraktion verwendet werden, welche Metaboliten nachgewiesen werden39. Um die Vielfalt der nachgewiesenen Metabolitenmerkmale für ungezielte chemische Kartographieanalysen zu maximieren, wird die Kombination mehrerer Extraktionsschritte und Lösungsmittel empfohlen. Zum Beispiel verwendet diese Methode Dichlormethan, Methanol und Wasser als Extraktionslösungsmittel, da sie einen genauen Nachweis von unpolaren und polaren Molekülen ermöglichen20,40. Diese Lösungsmittel sind jedoch nicht universell für jedes MS-Experiment geeignet, und forscher sollten Extraktionslösungsmittel basierend auf den Zielen ihres Projekts auswählen. Ebenso können verschiedene LC-MS/MS-Bedingungen verwendet werden, wie z.B. das Ersetzen der Umkehrphasenchromatographie durch die Normalphasenchromatographie. Alternative Säulen können auch für die Erfassung von Umkehrphasendaten anstelle der C8-Chromatographie verwendet werden, obwohl die C8-Chromatographie empirisch robuster gegenüber Gewebelipiden ist und eine geringere Verstopfungshäufigkeit aufweist. Konzeptionell können diese Protokolle auch auf andere massenspektrometrische Methoden wie Gaschromatographie-Massenspektrometrie usw. angewendet werden.
Ein alternativer Ansatz ist die massenspektrometrische Bildgebung. Im Gegensatz zu massenspektrometrischen Bildgebungsansätzen bewahrt die Flüssigkeitschromatographie-Massenspektrometrie die räumliche Information nicht von Natur aus10. Chemische Kartographieansätze schließen diese Lücke, indem sie den Probenstandort zum Zeitpunkt der Projektkonzeption, in die Probenmetadaten und in die Datenverarbeitungsschritte einbeziehen. Eine Stärke dieses chemischen Kartographieansatzes ist im Gegensatz zur massenspektrometrischen Bildgebung die Fähigkeit, zuverlässige Anmerkungen zu liefern (Metabolomics Standards Initiative Level 1 oder Level 2 Annotation Confidence41), im Gegensatz zur massenspektrometrischen Bildgebung, bei der der Großteil der Anwendungen nur für die Annotation auf genaue Masse angewiesen ist. Die massenspektrometrische Bildgebung ermöglicht eine feinkörnige räumliche Kartierung, manchmal bis auf die Einzelzellebene, z. B. 42,43. Im Gegensatz dazu ermöglichen chemische Kartographieansätze eine großräumige organübergreifende Kartierung der Metabolitenverteilung, ohne dass hochspezialisierte Kryosektionsfähigkeiten für ganze Tiere erforderlich sind. Die chemische Kartographie liefert ergänzende Beweise für die vielen räumlichen transkriptomischen Ansätze, die entwickelt werden, z.B. 44, mit dem Vorteil, dass sie sich auf die "Omics-Schicht" konzentriert, die dem Phänotyp am nächsten ist"45. Alternative Methoden zur Quantifizierung der Parasitenbelastung umfassen die Messung der Biolumineszenz zum Zeitpunkt der Probenentnahme6. Feine Segmente könnten auch gesammelt werden, um konfokale oder Elektronenmikroskopie zu ermöglichen, um die lokalisierte Parasitenbelastung und Gewebeschäden zu beurteilen. Das Wasserhomogenat, das in diesem Protokoll und früheren Publikationen13 für die Zytokinquantifizierung verwendet wird, könnte auch zur Quantifizierung proteinbasierter Marker für Gewebeschäden verwendet werden.
Es gibt auch mehrere Möglichkeiten, 3D-Modelle zu erhalten, die geeignet sind, die resultierenden LC-MS-Daten darzustellen. Zusätzlich zu der hier vorgeschlagenen Methode können Modelle vorgefertigt von verschiedenen Online-Anbietern erworben werden. Stellen Sie sicher, dass die Nutzungsbedingungen mit dem Verwendungszweck übereinstimmen, insbesondere in Bezug auf die Veröffentlichung. Modelle für große Organe können de novo mit 3D-Scannern nach Scanneranweisungen erstellt werden. Für die Generierung und Visualisierung von 3D-Modellen für die chemische Kartographie46 existieren Alternativen wie MATLAB, die aber vor allem vor der Entwicklung von 'ili16 implementiert wurden. MATLAB ist eine Datenanalyse- und Programmier-Tool-Suite, die eine Vielzahl von Anwendungen in vielen Bereichen bietet. MATLAB ist jedoch weder frei noch Open-Source und erfordert die Vertrautheit mit MATLAB-Schnittstellen, insbesondere wenn man bedenkt, dass MATLAB nicht für die Verarbeitung von Massenspektrometriedaten entwickelt wurde. Die Alternativen dieser vorgeschlagenen Methode, nämlich SketchUp, Meshlab und 'ili, sind frei zugänglich, benutzerfreundlich und bieten ähnliche Funktionen wie MATLAB für chemische Kartographiezwecke.
Diese Methode ist robust in Bezug auf probenpräparation und metabolitenextraktion. Die Fehlerbehebung ist meistens im LC-MS-Datenerfassungsschritt erforderlich. Dies würde den Rahmen dieses Artikels sprengen. Die Leser werden zu exzellenten Publikationen zur Fehlerbehebung bei der LC-MS-Datenerfassung weitergeleitet, darunter20,47. Ebenso geht die Komplexität der Metabolitenannotation über den Rahmen des Fokus dieser Methode auf die 3D-Modellgenerierung hinaus. Nützliche Referenzen zu diesem Thema sind24,25,48,49.
Während diese Methode die Pathogenese von Krankheiten effektiv erforscht, gibt es Einschränkungen für diesen Ansatz, von denen einige in jedem Metabolomics-Experiment üblich sind. Eine dieser Einschränkungen ist die geringe Annotationsrate der LC-MS-Features50, die von der Verfügbarkeit und Qualität der Referenzspektralbibliotheken abhängt. Eine weitere Einschränkung besteht darin, dass dieses Protokoll mRNA aufgrund der Inkompatibilität von RNA-Konservierungsreagenzien wie RNAlater mit der LC-MS/MS-Analyse nicht konserviert. Die Proteinqualität ist jedoch für nachgelagerte Analysen ausreichend und kann somit mRNA-basierte Analysen ersetzen.
Ein chemischer kartographischer Ansatz zur Infektionspathogenese spiegelt direkt wider, wie sich bakterielle, virale oder parasitäre Infektionen in Organsystemen entwickeln und lokalisierte Krankheiten verursachen. Die Analyse dieser regionalen Teilstichproben und die Generierung von 3D-Modellen vermittelt letztendlich, wie Metaboliten im dreidimensionalen Raum funktionieren, und beleuchtet diese bisher nicht erkannten räumlichen Dimensionen der Molekularbiologie. Unter Verwendung dieses Protokolls wurde beispielsweise die Metabolitenlokalisation mit der Parasitenbelastung von Trypanosoma cruzi verglichen. Die Ergebnisse klärten die Beziehung zwischen dem Erreger und dem Wirtsgewebe und zeigten auch die metabolische Dynamik der Symptomprogression der Chagas-Krankheit6. Chemische Kartographiemethoden wurden auch auf verschiedene Themen angewendet, wie z.B. die Interaktion zwischen Mensch und gebauter Umwelt51,52,53, die chemische Zusammensetzung von Organsystemen wie menschliche Haut46 und Lunge54 sowie pflanzenstoffwechsel und Umweltinteraktionen55. Zukünftige Anwendungen können die Bewertung der lokalen Krankheitstoleranz und -resilienz oder der Beziehung zwischen lokalen Metabolitenspiegeln, Pathogentropismus und Krankheitstropismus in Modellen über die Trypanosomatideninfektion hinaus umfassen. Dieser Ansatz sollte auch eine breite Anwendbarkeit haben, um die aktuellen pharmakokinetischen Protokolle zu erweitern, um die Beziehung zwischen lokalen Gewebearzneimittelspiegeln und Arzneimittelstoffwechsel vs. gesamtmetabolischem Kontext, Gewebeschäden und Pathogenclearance zu bewerten. Insgesamt ermöglicht die chemische Kartographie einzigartige Erkundungen der Metabolitenverteilung in verschiedenen Probentypen, mit Anwendungen wie Krankheitspathogenese, menschliche Gesundheit, Mensch-Umwelt-Interaktionen und mikrobielle Dynamik.
Offenlegungen
Keine zu meldenden Interessenkonflikte.
Danksagungen
Laura-Isobel McCall, PhD, ist Preisträgerin des Investigators in the Pathogenesis of Infectious Disease Award des Burroughs Wellcome Fund. Die Autoren möchten ferner die Unterstützung durch die NIH-Auszeichnungsnummer R21AI148886, ein Pilotstipendium des Oklahoma Center for Respiratory and Infectious Diseases (OCRID) unter der NIH-Preisnummer P20GM103648 und Startkapital der University of Oklahoma (an LIM) würdigen. Der Inhalt liegt ausschließlich in der Verantwortung der Autoren und stellt nicht unbedingt die offiziellen Ansichten der Geldgeber dar. Die Autoren möchten sich auch bei den Entwicklern der in diesem Protokoll verwendeten Tools bedanken. Gegebenenfalls wurden alle relevanten Publikationen zitiert.
Materialien
| Name | Company | Catalog Number | Comments |
| 1.5 mL Eppendorf tubes | NA | NA | any brand, as available |
| 1 L bottle, pyrex | NA | NA | any brand, as available |
| 1 L graduated cylinder, pyrex | NA | NA | any brand, as available |
| 2 mL SafeLock Eppendorf tubes | VWR | 20901-540 | use the appropriate tube model for the available tissue homogenizer |
| 3D model (de-novo generated according to protocol steps, or purchased) | NA | NA | as appropriate for system under investigation |
| 5 mm stainless steel bead | Qiagen | 69989 | |
| 96 well plate | Fisher | 3252449 | |
| AATTCCTCCAAGCAGCGGATA primer | NA | NA | any brand, as available; published in Piron, M. et al. Development of a real-time PCR assay for Trypanosoma cruzi detection in blood samples. Acta tropica. 103 (3), 195–200 (2007). |
| analytical balance | NA | NA | any brand, as available |
| ASTCGGCTGATCGTTTTCGA primer | NA | NA | any brand, as available; published in Piron, M. et al. Development of a real-time PCR assay for Trypanosoma cruzi detection in blood samples. Acta tropica. 103 (3), 195–200 (2007). |
| benchtop centrifuge with microcentrifuge, falcon tube and 96-well-plate capacity | NA | NA | any brand, as available |
| biosafety cabinet | NA | NA | class II, type A2; any brand, as available |
| CAGCAAGCATCTATGCACTTAG ACCCC primer | NA | NA | any brand, as available; published in Cummings, K.L., Tarleton, R.L. Rapid quantitation of Trypanosoma cruzi in host tissue by real-time PCR. Molecular and biochemical parasitology. 129 (1), 53–59 (2003). |
| camera | NA | NA | any brand, as available. A cellphone camera is adequate for this protocol |
| chemiluminescent-capable imaging system | NA | NA | any system, as available |
| cotton balls | NA | NA | any brand, as available |
| cryogloves | VWR | 97008-208 | replace with any brand, as available |
| dissection scissors | NA | NA | any brand, as available |
| dry ice | NA | NA | any brand, as available |
| extra-length forceps | NA | NA | any brand, as available |
| flammable-grade refrigerator | NA | NA | any brand, as available |
| freezer storage boxes for microcentrifuge tubes | NA | NA | any brand, as available |
| fume hood | NA | NA | any brand, as available |
| high-resolution mass spectrometer | NA | NA | any brand, as available, such as ThermoFisher Q-Exactive Plus (catalog number 0726030) |
| ice bucket | NA | NA | any brand, as available |
| ili software | ili.embl.de | NA | |
| isoflurane | Covetrus | 29405 | |
| large tupperware | NA | NA | any brand, as available; large enough to comfortably contain mouse, cotton ball |
| LC-MS grade acetonitrile | Fisher Optima | A955-4 | |
| LC-MS grade dicholoromethane | Fisher Optima | D151-4 | |
| LC-MS grade formic acid | Fisher Optima | A11750 | |
| LC-MS grade methanol | Fisher Optima | A456-4 | |
| LC-MS grade water | Fisher Optima | W64 | |
| liquid chromatography column | Phenomenex | 00B-4499-AN | may be changed to other brands and models as appropriate for the metabolites of interest |
| liquid chromatography column guard cartridge | Phenomenex | AJ0-8784 | may be changed to other brands and models as appropriate for the metabolites of interest |
| liquid chromatography column guard cartridge holder | Phenomenex | AJ0-9000 | may be changed to other brands and models as appropriate for the metabolites of interest |
| liquid nitrogen | NA | NA | any brand, as available |
| luciferin | Goldbio | LUCK-1G | |
| MeshLab software | https://www.meshlab.net/ | NA | |
| Meshmixer software | https://www.meshmixer.com/ | NA | |
| MS calibrant | NA | NA | appropriate one for available instrument |
| MS data processing software | NA | NA | multiple options available; authors recommend MZmine |
| MSConvert software | http://proteowizard.sourceforge.net/ | NA | |
| Nanodrop | ThermoFisher | ND-ONE-W | other nanodrop models are also suitable |
| p1000 pipet tips | NA | NA | use the appropriate brand to fit available pipettors |
| p1000 pipettor | NA | NA | any brand, as available |
| p20 pipette tips | NA | NA | use the appropriate brand to fit available pipettors |
| p20 pipettor | NA | NA | any brand, as available |
| p200 pipette tips | NA | NA | use the appropriate brand to fit available pipettors |
| p200 pipettor | NA | NA | any brand, as available |
| personal protective equipment (gloves, lab coat, safety glasses/goggles; faceshield) | NA | NA | any brand, as available |
| Q-Plex Mouse Cytokine - Screen (16-Plex) | Quansys biosciences | 110949MS | can replace with other protein-based cytokine assays such as other commercial cytokine ELISA kits |
| Quick-DNA Miniprep Plus Kit (200 preps) | Zymo | D4069 | replace with any brand of mammalian DNA extraction kit, as available |
| real-time thermocycler | NA | NA | any brand, as available |
| salt shaker or tea infuser | NA | NA | any brand; to contain isoflurane-soaked cotton ball and prevent contact with mouse skin |
| SketchUp software | https://www.sketchup.com/ | NA | |
| specimen forceps | NA | NA | any brand, as available |
| speedvac with microcentrifuge tube and 96-well-plate capacity | NA | NA | any brand, as available |
| spreadsheet software | https://www.microsoft.com/en-us/microsoft-365/excel | NA | can replace with other spreadsheet management software, as applicable |
| sulfachloropyridazine | Sigma | S9882-100G | |
| sulfadimethoxine | Sigma | S7007-10G | |
| Sybr green qPCR reaction mix | Fisher | A25780 | can replace with other Sybr green qPCR reaction mixes, as desired |
| TCCCTCTCATCAGTTCTAT GGCCCA primer | NA | NA | any brand, as available; published in Cummings, K.L., Tarleton, R.L. Rapid quantitation of Trypanosoma cruzi in host tissue by real-time PCR. Molecular and biochemical parasitology. 129 (1), 53–59 (2003). |
| tissue homogenizer | NA | NA | any brand, as available; for example, Qiagen TissueLyser II, catalog number 85300, with TissueLyser Adapter Set (2 x 24), catalog number 69982 |
| tissue samples | NA | NA | from appropriate infection model |
| TissueLyser single-bead dispenser | Qiagen | 69965 | |
| UHPLC | NA | NA | any brand, as available, such as ThermoFisher Vanquish (catalog number IQLAAAGABHFAPUMBHV) |
| ultra-low temperature freezer (-80) | NA | NA | any brand, as available |
| ultrasonic bath | NA | NA | any system, as available |
| wet ice | NA | NA | any brand, as available |
| zone-free sealing film | VWR | 490007-390 |
Referenzen
- McCall, L. -. I., et al. Adaptation of Leishmania donovani to and visceral environments: in vivo selection and proteomic analysis. Journal of Proteome Research. 14 (2), 1033-1059 (2015).
- McCall, L. -. I., Zhang, W. -. W., Matlashewski, G. Determinants for the development of visceral leishmaniasis disease. PLoS Pathogens. 9 (1), 1003053 (2013).
- de Castro Neto, A. L., da Silveira, J. F., Mortara, R. A. Comparative analysis of virulence mechanisms of trypanosomatids pathogenic to humans. Frontiers in Cellular and Infection Microbiology. 11, 669079 (2021).
- Franco, J. R., Simarro, P. P., Diarra, A., Jannin, J. G. Epidemiology of human African trypanosomiasis. Clinical Epidemiology. 6, 257-275 (2014).
- Ottilie, S., et al. Rapid chagas disease drug target discovery using directed evolution in drug-sensitive yeast. ACS Chemical Biology. 12 (2), 422-434 (2017).
- Hossain, E., et al. Mapping of host-parasite-microbiome interactions reveals metabolic determinants of tropism and tolerance in Chagas disease. Science Advances. 6 (30), (2020).
- Parab, A. R., McCall, L. -. I. Tryp-ing up metabolism: Role of metabolic adaptations in kinetoplastid disease pathogenesis. Infection and Immunity. 89 (4), (2021).
- Lewis, C. M., McCall, L. -. I., Sharp, R. R., Spicer, P. G. Ethical priority of the most actionable system of biomolecules: the metabolome. American Journal of Physical Anthropology. 171 (2), 177-181 (2020).
- Liu, Z., Ulrich vonBargen, R., McCall, L. I. Central role of metabolism in Trypanosoma cruzi tropism and Chagas disease pathogenesis. Current Opinion in Microbiology. 63, 204-209 (2021).
- Newsom, S. N., McCall, L. -. I. Metabolomics: Eavesdropping on silent conversations between hosts and their unwelcome guests. PLoS Pathogens. 14 (4), 1006926 (2018).
- Wishart, D. S., et al. HMDB: the Human Metabolome Database. Nucleic Acids Research. 35, 521-526 (2007).
- Dean, D. A., et al. Spatial metabolomics identifies localized chemical changes in heart tissue during chronic cardiac Chagas disease. PLoS Neglected Tropical Diseases. 15 (10), 0009819 (2021).
- McCall, L. -. I., et al. Mass spectrometry-based chemical cartography of a cardiac parasitic infection. Analytical Chemistry. 89 (19), 10414-10421 (2017).
- Hoffman, K., et al. Alterations to the cardiac metabolome induced by chronic infection relate to the degree of cardiac pathology. ACS Infectious Diseases. 7 (6), 1638-1649 (2021).
- Parab, A. R., et al. Dysregulation of glycerophosphocholines in the cutaneous lesion caused by Leishmania major in experimental murine models. Pathogens. 10 (5), 593 (2021).
- Protsyuk, I., et al. 3D molecular cartography using LC-MS facilitated by Optimus and 'ili software. Nature Protocols. 13 (1), 134-154 (2018).
- McCall, L. -. I., et al. Targeting ergosterol biosynthesis in Leishmania donovani: Essentiality of Sterol 14alpha-demethylase. PLOS Neglected Tropical Diseases. 9 (3), 0003588 (2015).
- Tear, W. F., et al. Selectivity and physicochemical optimization of repurposed Pyrazolo[1,5-]pyridazines for the treatment of human African trypanosomiasis. Journal of Medicinal Chemistry. 63 (2), 756-783 (2020).
- Klug, D. M., et al. Hit-to-lead optimization of benzoxazepinoindazoles as human African trypanosomiasis therapeutics. Journal of Medicinal Chemistry. 63 (5), 2527-2546 (2020).
- Want, E. J., et al. Global metabolic profiling of animal and human tissues via UPLC-MS. Nature Protocols. 8 (1), 17-32 (2013).
- Piron, M., et al. Development of a real-time PCR assay for Trypanosoma cruzi detection in blood samples. Acta Tropica. 103 (3), 195-200 (2007).
- Cummings, K. L., Tarleton, R. L. Rapid quantitation of Trypanosoma cruzi in host tissue by real-time PCR. Molecular and Biochemical Parasitology. 129 (1), 53-59 (2003).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2−ΔΔCT method. Methods. 25 (4), 402-408 (2001).
- Nothias, L. -. F., et al. Feature-based molecular networking in the GNPS analysis environment. Nature Methods. 17 (9), 905-908 (2020).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with global natural products social molecular networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Chambers, M. C., et al. A cross-platform toolkit for mass spectrometry and proteomics. Nature Biotechnology. 30 (10), 918-920 (2012).
- Tsugawa, H., et al. MS-DIAL: data-independent MS/MS deconvolution for comprehensive metabolome analysis. Nature Methods. 12 (6), 523-526 (2015).
- Sturm, M., et al. OpenMS - an open-source software framework for mass spectrometry. BMC Bioinformatics. 9, 163 (2008).
- Pluskal, T., Castillo, S., Villar-Briones, A., Oresic, M. MZmine 2: modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11, 395 (2010).
- Smith, C. A., Want, E. J., O'Maille, G., Abagyan, R., Siuzdak, G. XCMS: processing mass spectrometry data for metabolite profiling using nonlinear peak alignment, matching, and identification. Analytical Chemistry. 78 (3), 779-787 (2006).
- . FBMN with MZmine Available from: https://ccms-ucsd.github.io/GNPSDocumentation/featurebasedmolecularnetworking-with-mzmine2/ (2021)
- Lewis, M. D., et al. Bioluminescence imaging of chronic Trypanosoma cruzi infections reveals tissue-specific parasite dynamics and heart disease in the absence of locally persistent infection. Cellular Microbiology. 16 (9), 1285-1300 (2014).
- Knubel, C. P., et al. 3-Hydroxy kynurenine treatment controls T. cruzi replication and the inflammatory pathology preventing the clinical symptoms of chronic Chagas disease. PloS One. 6 (10), 26550 (2011).
- Dumoulin, P. C., Vollrath, J., Tomko, S. S., Wang, J. X., Burleigh, B. Glutamine metabolism modulates azole susceptibility in amastigotes. eLife. 9, (2020).
- Keller, B. O., Sui, J., Young, A. B., Whittal, R. M. Interferences and contaminants encountered in modern mass spectrometry. Analytica Chimica Acta. 627 (1), 71-81 (2008).
- Weaver, R., Riley, R. J. Identification and reduction of ion suppression effects on pharmacokinetic parameters by polyethylene glycol 400. Rapid Communications in Mass Spectrometry: RCM. 20 (17), 2559-2564 (2006).
- Larger, P. J., Breda, M., Fraier, D., Hughes, H., James, C. A. Ion-suppression effects in liquid chromatography-tandem mass spectrometry due to a formulation agent, a case study in drug discovery bioanalysis. Journal of Pharmaceutical and Biomedical Analysis. 39 (1-2), 206-216 (2005).
- Furey, A., Moriarty, M., Bane, V., Kinsella, B., Lehane, M. Ion suppression; a critical review on causes, evaluation, prevention and applications. Talanta. 115, 104-122 (2013).
- Lu, W., et al. Metabolite measurement: Pitfalls to avoid and practices to follow. Annual Review of Biochemistry. 86, 277-304 (2017).
- Masson, P., Alves, A. C., Ebbels, T. M. D., Nicholson, J. K., Want, E. J. Optimization and evaluation of metabolite extraction protocols for untargeted metabolic profiling of liver samples by UPLC-MS. Analytical Chemistry. 82 (18), 7779-7786 (2010).
- Sumner, L. W., et al. Proposed minimum reporting standards for chemical analysis. Metabolomics. 3 (3), 211-221 (2007).
- Rappez, L., et al. SpaceM reveals metabolic states of single cells. Nature Methods. 18 (7), 799-805 (2021).
- Yang, B., Tsui, T., Caprioli, R. M., Norris, J. L. Sample preparation and analysis of single cells using high performance MALDI FTICR mass spectrometry. Methods in Molecular Biology. 2064, 125-134 (2020).
- Longo, S. K., Guo, M. G., Ji, A. L., Khavari, P. A. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nature Reviews. Genetics. 22 (10), 627-644 (2021).
- Patti, G. J., Yanes, O., Siuzdak, G. Innovation: Metabolomics: the apogee of the omics trilogy. Nature Reviews. Molecular Cell Biology. 13 (4), 263-269 (2012).
- Bouslimani, A., et al. Molecular cartography of the human skin surface in 3D. Proceedings of the National Academy of Sciences of the United States of America. 112 (17), 2120-2129 (2015).
- Dudzik, D., Barbas-Bernardos, C., García, A., Barbas, C. Quality assurance procedures for mass spectrometry untargeted metabolomics. A review. Journal of Pharmaceutical and Biomedical Analysis. 147, 149-173 (2018).
- Alseekh, S., et al. Mass spectrometry-based metabolomics: a guide for annotation, quantification and best reporting practices. Nature Methods. 18 (7), 747-756 (2021).
- Chaleckis, R., Meister, I., Zhang, P., Wheelock, C. E. Challenges, progress and promises of metabolite annotation for LC-MS-based metabolomics. Current Opinion in Biotechnology. 55, 44-50 (2019).
- Viant, M. R., Kurland, I. J., Jones, M. R., Dunn, W. B. How close are we to complete annotation of metabolomes. Current Opinion in Chemical Biology. 36, 64-69 (2017).
- Kapono, C. A., et al. Creating a 3D microbial and chemical snapshot of a human habitat. Scientific Reports. 8 (1), 3669 (2018).
- Petras, D., et al. Mass spectrometry-based visualization of molecules associated with human habitats. Analytical Chemistry. 88 (22), 10775-10784 (2016).
- McCall, L. -. I., et al. Analysis of university workplace building surfaces reveals usage-specific chemical signatures. Building and Environment. 162, 106289 (2019).
- Garg, N., et al. Three-dimensional microbiome and metabolome cartography of a diseased human lung. Cell Host & Microbe. 22 (5), 705-716 (2017).
- Floros, D. J., et al. Mass spectrometry based molecular 3D-cartography of plant metabolites. Frontiers in Plant Science. 8, 429 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten