Method Article
Approcci cartografici chimici per studiare l'infezione da tripanosomatidi
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo protocollo descrive i passaggi per generare un modello 3D della distribuzione dei metaboliti durante l'infezione da tripanosomatidi, compresa la raccolta di campioni, l'estrazione di metaboliti, una panoramica della cromatografia liquida-acquisizione dei dati di spettrometria di massa tandem, generazione di modelli 3D e, infine, visualizzazione dei dati.
Abstract
Il tropismo patogeno e il tropismo della malattia si riferiscono alle posizioni dei tessuti colonizzate selettivamente o danneggiate dagli agenti patogeni, portando a sintomi localizzati della malattia. I parassiti tripanososamidi infettivi dell'uomo includono Trypanosoma cruzi, l'agente eziologico della malattia di Chagas; Trypanosoma brucei, l'agente eziologico della malattia del sonno; e le specie di Leishmania , agenti causali della leishmaniosi. Insieme, colpiscono 20 milioni di persone in tutto il mondo. Questi parassiti mostrano un tropismo specifico: cuore, esofago, colon per T. cruzi, tessuto adiposo, pancreas, pelle, sistema circolatorio e sistema nervoso centrale per T. brucei, pelle per ceppi di Leishmania dermotropica e fegato, milza e midollo osseo per ceppi viscerotropici di Leishmania . Una prospettiva spaziale è quindi essenziale per comprendere la patogenesi della malattia tripanosomatidica. La cartografia chimica genera visualizzazioni 3D dell'abbondanza di piccole molecole generate tramite cromatografia liquida-spettrometria di massa, rispetto ai parametri microbiologici e immunologici. Questo protocollo dimostra come la cartografia chimica possa essere applicata per studiare i processi patogeni durante l'infezione da tripanosomatidi, a partire dal campionamento sistematico dei tessuti e dall'estrazione dei metaboliti, seguita dall'acquisizione dei dati di cromatografia liquida-spettrometria di massa tandem e concludendo con la generazione di mappe 3D della distribuzione dei metaboliti. Questo metodo può essere utilizzato per molteplici domande di ricerca, come i requisiti nutrizionali per la colonizzazione dei tessuti da parte di T. cruzi, T. brucei o Leishmania, l'immunometabolismo nei siti di infezione e la relazione tra perturbazione metabolica tissutale locale e sintomi clinici della malattia, portando a una visione completa della patogenesi della malattia tripanosomatidica.
Introduzione
I parassiti tripanosomatidi sono costituiti da specie di Leishmania, tripanosomi africani (Trypanosoma brucei) e tripanosomi americani (Trypanosoma cruzi). I protozoi di leishmania causano leishmaniosi, che comprende l'auto-guarigione e l'auto-limitata leishmaniosi cutanea localizzata, la leishmaniosi mucocutanea in cui i tessuti mucosi della bocca, del naso e della gola si danneggiano, e la leishmaniosi viscerale con tropismo parassitario agli organi viscerali che causa febbre ed epatosplenomegalia1,2. T. brucei causa la tripanosomiasi africana umana (HAT), nota anche come malattia del sonno, segnalata principalmente nei paesi africani3. I segni e sintomi clinici includono epatosplenomegalia, febbre, mal di testa, dolori muscoloscheletrici, linfoadenopatie e anemia nella fase emo-linfatica quando i parassiti si localizzano nel flusso sanguigno e linfatici. Questo è seguito dallo stadio meningo-encefalitico, in cui i parassiti si localizzano nel sistema nervoso centrale e causano disturbi del sonno, alterazioni comportamentali e infine coma fatali4. T. cruzi causa la malattia di Chagas, endemica nelle Americhe. Gli individui infetti sperimentano uno stadio acuto iniziale, di solito asintomatico, con ampio tropismo parassitario. Circa il 10%-30% degli individui infetti sperimenta sintomi di stadio cronico dopo decenni di infezione, caratterizzati da megaesofago, megacolon e complicanze cardiovascolari5,6.
La metabolomica studia piccole specie molecolari (50-1.500 Da), compresi composti biologici del metabolismo primario o secondario e composti derivati esternamente come farmaci o molecole derivate dal cibo. Nel contesto delle interazioni ospite-patogeno, la metabolomica può esplorare l'impatto dell'infezione sugli ambienti del metabolita ospite, cruciale per accedere all'effetto del patogeno sull'ospite. Può anche valutare gli adattamenti patogeni all'ambiente nutrizionale e immunologico dell'ospite7,8,9. La spettroscopia di spettrometria di massa (MS) e la spettroscopia di risonanza magnetica nucleare (NMR) sono strumenti metabolomici comuni utilizzati per identificare, quantificare e caratterizzare i metaboliti. Questo approccio "omico" può essere applicato anche alla scoperta di biomarcatori e allo sviluppo di farmaci10,11.
Dato il tropismo tissutale specifico dei parassiti tripanosomatidi, le analisi metabolomiche spaziali possono consentire una visione significativa della patogenesi delle malattie che causano. La mappatura della distribuzione spaziale dei metaboliti ha rivelato metaboliti localmente colpiti dall'infezione cronica da Trypanosoma cruzi nel tessuto cardiaco del topo e dall'infezione acuta e a lungo termine da Trypanosoma cruzi nel tratto gastrointestinale del topo6,12,13. In particolare, la cartografia chimica 3D ha dimostrato una disconnessione tra la persistenza del parassita e le alterazioni metaboliche nel tessuto cardiaco di topi cronicamente infetti da Trypanosoma cruzi. Il metabolismo era più perturbato nei segmenti inferiori e apicali del cuore, in corrispondenza con i siti dei sintomi della malattia di Chagas (aneurismi apicali cardiaci). Le famiglie di metaboliti perturbate dall'infezione in specifici siti cardiaci e correlate alla gravità della malattia includono acilcarnitine e glicerofosfocoline12,13,14. Nel tratto gastrointestinale, le alterazioni metaboliche persistenti concordavano con i siti dei sintomi della malattia di Chagas: esofago e colon. Al contrario, il metabolismo viene ri-normalizzato in siti non associati ai sintomi della malattia di Chagas, come l'intestino tenue. I metaboliti localmente perturbati dall'infezione nel tratto gastrointestinale includono acilcarnitine, glicerofosfocoline, chinurenina, triptofano e acido colico. Inoltre, queste analisi hanno permesso l'identificazione di un nuovo meccanismo metabolico di tolleranza alla malattia di Chagas6. L'applicazione di questi metodi allo studio della leishmaniosi cutanea ha rivelato significative perturbazioni metaboliche nel sito della lesione, ma anche specifici cambiamenti metabolici nel tessuto macroscopicamente sano adiacente alla lesione. Ad esempio, la glutammina è stata impoverita nel sito della lesione, mentre le glicerofosfocoline nel m / z (rapporto massa / carica) 200-299, 400-499, 500-599 e 600-699 sono state significativamente aumentate nel sito della lesione. Il PC (O-34:1) è stato aumentato solo nei siti adiacenti alla lesione15.
L'obiettivo di questo manoscritto è quello di dimostrare i passaggi necessari per generare modelli 3D di distribuzione dei metaboliti ("cartografia chimica") applicati ai modelli di infezione da parassiti tripanosomatidi (Figura 1). Questo approccio si basa su diversi progressi critici nel contesto dell'elaborazione dei dati di metabolomica e metabolomica, in particolare lo sviluppo di un software per tracciare facilmente i dati metabolomici su modelli 3D16.
Protocollo
Tutti gli esperimenti sugli animali descritti sono stati approvati dall'Università dell'Oklahoma o dall'Università della California San Diego Institutional Animal Care and Use Committee. Tutte le fasi di manipolazione del materiale infettivo sono state eseguite all'interno di un armadio di biosicurezza (classe II, tipo A2) e secondo le normative locali.
1. Raccolta dei tessuti
- Infettare appropriati modelli animali di infezione da tripanosomatidi, generalmente topi o criceti.
NOTA: Esiste una notevole varietà di modelli murini per l'infezione da tripanosomatidi, a seconda dei sintomi desiderati da produrre, della velocità di progressione della malattia, della gravità della malattia, ecc. Gli utenti possono scegliere a loro piacimento.- Per i modelli di infezione da leishmaniosi cutanea, infettare per via sottocutanea nella pedana o per via intradermica nell'orecchio15.
- Per i modelli di infezione da leishmaniosi viscerale, infettare per via endovenosa1,17.
- Per i modelli di infezione da malattia di Chagas e malattia del sonno, infettare per via intraperitoneale6,12,18,19. Determinare la dose di parassita da utilizzare in base al ceppo parassitario e ai punti temporali pianificati.
- Pianificare le posizioni di sezionamento.
- Pianificare la generazione di sezioni con un minimo di 10 mg di tessuto per sezione. È meglio usare 30-50 mg.
- Per i modelli di infezione da malattia di Chagas, pianificare di raccogliere sistematicamente segmenti cardiaci e gastrointestinali.
- Per i modelli di infezione da leishmaniosi cutanea, raccogliere campioni di tessuto lesionale e lesioni adiacenti.
- Per i modelli di infezione da leishmaniosi viscerale, pianificare di raccogliere milza e lobi epatici multipli. Ulteriori siti tissutali come il tessuto adiposo possono anche essere di interesse da raccogliere.
ATTENZIONE: I campioni di animali infetti devono essere trattati secondo l'appropriato protocollo di biosicurezza approvato istituzionalmente. Ciò comporterà generalmente requisiti di dispositivi di protezione individuale (DPI) e solo l'apertura di tubi e la raccolta di campioni all'interno di un armadio di biosicurezza (classe II, tipo A2).
- Etichettare e pesare tubi per omogeneizzazione.
- Utilizzare il tipo di tubo appropriato per il sistema di omogeneizzazione disponibile. Per un TissueLyser, utilizzare 2 mL di tubi microcentrifuga.
- Eutanasia dei topi ai timepoint di infezione desiderati utilizzando il sovradosaggio di isoflurano come approvato dalla IACUC istituzionale o secondo il protocollo approvato dalla IACUC.
- Sezione tessuto sistematicamente come previsto, con una sezione per tubo.
- Ricordarsi di lavare l'attrezzatura per la raccolta dei campioni tra i campioni con solvente di estrazione (50% di metanolo in questo protocollo).
- Tenendo aperto il coperchio del tubo, far immediatamente scattare i campioni di congelamento in azoto liquido.
ATTENZIONE: non chiudere i tubi fino a quando l'azoto liquido entrato nel tubo non è completamente evaporato per evitare che i tubi esplodano mentre l'azoto si espande. Adottare misure adeguate per garantire che la pelle non venga a contatto con l'azoto liquido (utilizzare guanti criogenici e pinze per tenere i tubi). Indossare una visiera di sicurezza. - Conservare i tubi su ghiaccio secco fino a quando tutti i campioni desiderati sono stati raccolti.
- Punto di pausa: conservare i campioni a -80 °C fino al momento di effettuare estrazioni.
- Pesare i tubi per determinare il peso del campione di tessuto. Registra in un foglio di calcolo.
- Conservare i campioni congelati durante il processo di pesatura: tenere i tubi sul ghiaccio secco, pesare rapidamente, rimettere subito sul ghiaccio secco. Non lasciare che i campioni si scongelino durante la pesatura.
2. Estrazione del metabolita
NOTA: devono essere utilizzati solo liquidi e reagenti di grado LC-MS. Questo metodo è stato adattato da Reference20.
- Preparare tutti i solventi da estrazione (LC-MS grado H2O, LC-MS grado metanolo, chiodato con 4 μM di sulfaclorpiridazina, LC-MS grado diclorometano: metanolo con 2 μM di sulfaclorpiridazina) in flaconi di vetro da 1 L, utilizzando vetreria dedicata.
NOTA: il volume da preparare deve essere calcolato in base ai pesi del campione, considerando 500 μL di acqua per 50 mg di campione, 500 μL di metanolo con 4 μM di sulfaclorpiridazina per 50 mg di campione e 1.000 μL di diclorometano prechilled: metanolo con 2 μM di sulfaclorpiridazina per 50 mg di campione, aumentando il volume calcolato del 10% per consentire l'imprecisione del pipettaggio. Conservare il solvente di estrazione a 4 °C almeno durante la notte per pre-raffreddare. - Eseguire l'omogeneizzazione a base d'acqua dei campioni di tessuto secondo i passaggi indicati di seguito.
- Aggiungere una perla di acciaio inossidabile da 5 mm (vedi Tabella dei materiali) a ciascuna delle provette di microcentrifuga da 2 ml contenenti campioni di tessuto, utilizzando un distributore di perline. Tenere i tubi sul ghiaccio.
- Crea un tubo vuoto contenente H2O di grado LC-MS che passerà attraverso tutti i passaggi e fungerà da bianco di estrazione. Utilizzare il volume medio di H2O dalle estrazioni di campioni. Aggiungere il grado LC-MS H2O refrigerato ai campioni di tessuto congelato.
- Normalizzare il volume d'acqua al peso del tessuto aggiungendo 500 μL di acqua / 50 mg di campione, utilizzando i pesi del campione calcolati al punto 1.7.
ATTENZIONE: Se si maneggiano campioni biopericolosi, continuare a seguire il protocollo di biosicurezza appropriato e approvato istituzionalmente. Ciò comporterà generalmente requisiti per i dispositivi di protezione individuale (DPI) e l'apertura dei tubi solo all'interno dell'armadio di biosicurezza (classe II, tipo A2).
- Normalizzare il volume d'acqua al peso del tessuto aggiungendo 500 μL di acqua / 50 mg di campione, utilizzando i pesi del campione calcolati al punto 1.7.
- Omogeneizzare i campioni a una velocità di 25 Hz per 3 minuti utilizzando un omogeneizzatore tissutale (vedi Tabella dei materiali).
- Chiudere saldamente i tubi per evitare di versare i reagenti durante la lavorazione.
- Raccogliere circa 1/10 del volume di omogeneizzazione per l'estrazione del DNA, la qPCR, le analisi basate sulle proteine o altre analisi (se lo si desidera). Conservare in un tubo microcentrifuga o in una piastra a 96 pozzetti (a seconda del volume raccolto) per un massimo di 6 mesi a -80 °C, se gli esperimenti di estrazione del DNA devono essere eseguiti in un giorno diverso. Periodi di conservazione più lunghi possono essere possibili ma non sono stati testati.
- Eseguire estrazioni di DNA utilizzando qualsiasi kit commerciale standard per l'estrazione del DNA dei mammiferi (vedere Tabella dei materiali) dai tessuti come descritto nella referenza 13. Quantificare la resa del DNA e conservare il DNA estratto a -20 °C. Quantità e qualità del DNA accettabili per la qPCR sono state osservate anche fino a 3 anni dopo.
NOTA: Questo passaggio può essere eseguito su omogeneizzato congelato in un giorno successivo dall'estrazione del metabolita. - Eseguire qPCR come descritto in Reference13, utilizzando 180 ng di DNA estratto.
NOTA: Questo può essere eseguito su DNA raccolto in un giorno precedente e congelato a -20 °C.- Nel caso di studi sull'infezione da T. cruzi, utilizzare i seguenti primer: ASTCGGCTGATCGTTTTCGA e AATTCCTCCAAGCAGCGGATA per quantificare i livelli di parassiti21 e i seguenti primer per normalizzare ai livelli di DNA dell'ospite: TCCCTCTCATCAGTTCTATGGCCCA e CAGCAAGCATCTATGCACTTAGACCCC22 (vedere Tabella dei materiali).
NOTA: I cicli qPCR consigliati sono i seguenti: denatura a 95 °C per 10 min; eseguire 40 cicli a 95 °C per 30 s, e poi 58 °C per 60 s, e infine 72 °C per 60 s. Eseguire l'analisi della curva di fusione in modo appropriato per il termociclatore disponibile. Elaborare i dati utilizzando il metodo ΔΔCt23 per ottenere il carico parassitario relativo tra i siti di campionamento. La quantificazione assoluta può essere ottenuta confrontando i valori di ΔΔCt derivati dal campione con una curva standard generata da quantità note di parassiti, inseriti in campioni di tessuto non infetto ed estratti come nei passaggi da 2.2 a 2.2.4.1.
- Nel caso di studi sull'infezione da T. cruzi, utilizzare i seguenti primer: ASTCGGCTGATCGTTTTCGA e AATTCCTCCAAGCAGCGGATA per quantificare i livelli di parassiti21 e i seguenti primer per normalizzare ai livelli di DNA dell'ospite: TCCCTCTCATCAGTTCTATGGCCCA e CAGCAAGCATCTATGCACTTAGACCCC22 (vedere Tabella dei materiali).
- Eseguire la caratterizzazione basata sulle proteine delle risposte immunitarie utilizzando kit di citochine multiplexed o kit ELISA commerciali standard (vedere Tabella dei materiali) come descritto nella referenza 13 sull'omogeneizzato immagazzinato.
- Eseguire estrazioni di DNA utilizzando qualsiasi kit commerciale standard per l'estrazione del DNA dei mammiferi (vedere Tabella dei materiali) dai tessuti come descritto nella referenza 13. Quantificare la resa del DNA e conservare il DNA estratto a -20 °C. Quantità e qualità del DNA accettabili per la qPCR sono state osservate anche fino a 3 anni dopo.
- Risparmiare almeno la metà dei 500 μL del volume di omogeneizzazione per l'estrazione del metabolita.
- Eseguire l'estrazione del metabolita acquoso.
NOTA: La selezione del solvente può essere adattata in base alle proprietà chimiche dei metaboliti di interesse.- Aggiungere metanolo ghiacciato di grado LC-MS con 4 μM di sulfaclorpiridazina all'omogeneizzato per ottenere una concentrazione finale del 50% di metanolo con 2 μM di sulfaclorpiridazina in acqua.
ATTENZIONE: il metanolo è infiammabile e pericoloso. Utilizzare procedure di sicurezza appropriate, compresa la manipolazione all'interno di una cappa aspirante o di un armadio di biosicurezza. - Omogeneizzare i campioni in un omogeneizzatore tissutale a 25 Hz di velocità per 3 min. Centrifugare i campioni a 16.000 x g a 4 °C per 10 min.
- Raccogli un volume uguale di surnatante in una piastra a 96 pozzetti. Seleziona il volume in modo che corrisponda al più piccolo volume di metanolo + acqua combinato su tutti i campioni. Questa è la frazione acquosa.
- Mettere da parte l'eventuale surnatante omogeneizzato acquoso rimanente a -80 °C come backup.
- Mantenere il residuo solido sul ghiaccio mentre si raccolgono i supernatanti.
- Estrazione acquosa a secco surnatante fino ad asciugatura (~3 h o durante la notte). Utilizzare la massima velocità e nessun riscaldamento.
- Congelare la piastra essiccata a 96 pozzetti a -80 °C.
- Aggiungere metanolo ghiacciato di grado LC-MS con 4 μM di sulfaclorpiridazina all'omogeneizzato per ottenere una concentrazione finale del 50% di metanolo con 2 μM di sulfaclorpiridazina in acqua.
- Eseguire l'estrazione di metaboliti organici.
NOTA: La selezione del solvente può essere adattata in base alle proprietà chimiche dei metaboliti di interesse.- Aggiungere 1.000 μL per 50 mg del campione di diclorometano prechilled: metanolo con un picco di 2 μM di sulfaclorpiridazina al residuo solido dal punto 2.3.4.
ATTENZIONE: utilizzare procedure di sicurezza appropriate quando si maneggiano solventi, compresa la manipolazione all'interno di una cappa aspirante con una buona portata. - Omogeneizzare i campioni in un omogeneizzatore tissutale a 25 Hz di velocità per 5 min. Centrifugare i campioni a 16.000 x g a 4 °C per 10 min.
- Raccogli un volume uguale di surnatante in una piastra a 96 pozzetti. Selezionare il volume corrispondente al volume più piccolo di diclorometano: metanolo, su tutti i campioni. Questa è la frazione organica.
- Conservare il pellet a -80 °C come backup. Conservare l'estratto organico rimanente a -80 °C come backup. Asciugare all'aria l'estratto organico in una cappa aspirante durante la notte.
- Congelare la piastra essiccata a 96 pozzetti a -80 °C.
- Aggiungere 1.000 μL per 50 mg del campione di diclorometano prechilled: metanolo con un picco di 2 μM di sulfaclorpiridazina al residuo solido dal punto 2.3.4.
3. Acquisizione dati LC-MS
- Sospendere gli estratti acquosi e organici in 60 μL ciascuno del 50% di metanolo + 2 μM di sulfamimetossina e combinare. Sonicate per 10 min; quindi, centrifugare per 10 minuti e trasferire il surnatante in una piastra pulita a 96 pozzetti. Sigillare con guarnizione a piastre prive di zone e posizionare la piastra in un autocampionatore LC.
ATTENZIONE: utilizzare procedure di sicurezza appropriate quando si maneggiano solventi, compresa la manipolazione all'interno di una cappa aspirante con una buona portata. - Collegare le fasi mobili appropriate al sistema LC (vedere Tabella dei materiali).
NOTA: Per la LC inversa in modalità positiva, gli autori raccomandano LC-MS-grade H2O + 0,1% acido formico come fase mobile A e acetonitrile di grado LC-MS + acido formico allo 0,1% come fase B mobile con una portata di 0,5 ml / min e un gradiente LC di 7,5 minuti. I passi di pendenza consigliati sono quelli pubblicati in Reference6: 0-1 min, 2% B; 1-2,5 min, aumento lineare al 98% B; 2,5-4,5 min, tenuta al 98% B; 4,5-5,5 min, diminuzione lineare al 2% B; 5,5-7,5 min, tenere al 2% B. - Assicurarsi che lo strumento sia pulito. Calibrare la SM sia in modalità positiva che negativa.
- Eseguire la valutazione delle prestazioni della SM come appropriato per lo strumento.
- Creare una sequenza di esecuzione MS.
- Inizia con 2 spazi vuoti, 2 standard (6 miscele) e 5 controlli di qualità aggregati (QC) in una serie di diluizione, a partire da 2 μL di volume di iniezione e aumentando gradualmente fino a 30 μL di volume di iniezione.
- Randomizzare l'ordine del campione.
- Dopo ogni 12 campioni, eseguire uno spazio vuoto e quindi un QC in pool.
- Collegare la colonna C8 LC (dimensione delle particelle di 1,7 μm, dimensione dei pori di 100 Å, lunghezza 50 x 2,1 mm x diametro interno) e monitorare le perdite e l'eccessiva contropressione. Risolvere i problemi secondo la procedura operativa standard dello strumento.
- Avviare la sequenza di esecuzione MS e raccogliere dati LC-MS/MS dipendenti dai dati.
NOTA: per uno strumento Q-Exactive Plus MS, utilizzare la ionizzazione electrospray riscaldata e l'acquisizione MS2 dipendente dai dati (top 5) in modalità positiva, una risoluzione di 70.000 per MS1 e 17.500 per MS2, AGC Target di 1E6 per MS1 e 2E5 per MS2, IT massimo di 100 ms sia per MS1 che MS2, intervallo di scansione a 100-1500 m/z per MS1, e finestra di isolamento MS2 di 1 m/z. Impostare il gas della guaina a 35, il gas ausiliario a 10 e il gas di sweep a 0, la tensione di spruzzatura a 3,8 kV, la temperatura capillare a 320 °C, il livello RF della lente S a 50 e la temperatura del gas ausiliario a 350 °C.- Verificare la qualità dei dati: controllare gli spazi vuoti iniziali (confermare la mancanza di picchi principali), gli standard (confermare la presenza di picchi previsti e la forma simmetrica dei picchi) e i QC (confermare la presenza di picchi previsti, la forma del picco e l'intensità del picco prevista).
- Monitorare periodicamente l'esecuzione di MS durante la sequenza di esecuzione.
- Una volta terminata l'esecuzione, controllare i dati per eventuali iniezioni mancate o altri errori.
- Memorizzare la colonna LC come consigliato dal produttore. Rimuovere e conservare i campioni a -80 °C.
- Caricare i dati grezzi nel repository di dati.
NOTA: MassIVE (massive.ucsd.edu) è raccomandato per abilitare il collegamento a valle al collegamento molecolare per le annotazioni dei metaboliti24,25.
4. Trattamento dei dati LC-MS
- Convertire i file raw in formato aperto (.mzXML o .mzML) utilizzando MSConvert26.
- Caricare dati grezzi e dati mzXML o mzML nel repository di dati.
- Genera tabella delle feature. Ci sono più strumenti per farlo (MZmine, MS-DIAL, openMS, XCMS, ecc.27,28,29,30).
NOTA: MZmine è consigliato perché è gratuito, open source e può importare direttamente i file mzXML dopo MSconvert, ha opzioni di interfaccia utente grafica per l'elaborazione dei dati e il monitoraggio dell'impatto della selezione dei parametri e può esportare direttamente in GNPS per il networking molecolare24.
NOTA: seguire la documentazione dello strumento e utilizzare i parametri appropriati per lo strumento disponibile. Ulteriori dettagli sono disponibili anche in Reference31.- Esporta la tabella delle feature in formato .csv.
5.3D generazione del modello
- Disegna a mano il modello 3D de novo in scala secondo i passaggi indicati di seguito.
- Scatta una foto dell'organo di interesse.
- Esegui quanto segue nel software SketchUp (consulta Tabella dei materiali) come indicato di seguito.
- Eliminare l'immagine predefinita di un uomo che appare all'apertura del software.
- Fare clic su File > Importa per importare un'immagine degli organi di interesse.
- Fate clic sullo strumento Linee e selezionate l'opzione Mano libera . Usa lo strumento matita per tracciare e disegnare i contorni degli organi di interesse. Assicurarsi di chiudere la linea disegnando fino al punto di partenza. Una volta che la linea è stata chiusa con successo, l'area disegnata apparirà automaticamente ombreggiata.
- Selezionate lo strumento Push/Pull e visualizzate l'area ombreggiata per convertire il disegno da 2D a 3D.
- Eliminare l'immagine dell'organo: selezionare il pulsante Gomma , quindi fare clic con il pulsante destro del mouse sull'immagine e selezionare Cancella.
- Esporta il file in formato .dae: File > Esporta > modello 3D.
- Migliora il realismo del modello secondo i passaggi indicati di seguito.
- Importare il modello nel software MeshLab (vedere Tabella dei materiali): aprire MeshLab e selezionare File > Importa mesh. Selezionare il modello .dae generato nel passaggio precedente. Verrà visualizzato un menu Opzioni pre-apertura . Selezionare OK.
- Selezionate Wireframe nel menu in alto. Quindi, selezionate: Filtri > Rimescolamento, Semplificazione e ricostruzione > Superfici di suddivisione: Punto medio. Lasciare tutti i valori come predefiniti e selezionare Applica due volte. Ispezionate visivamente la vista wireframe del modello per assicurarvi che sia finemente grigliato. Chiudi il menu a comparsa.
- Esportare il modello in formato .stl: File > Esporta mesh come, quindi selezionare Formato file STL (*.stl) nel menu a discesa Tipo file . Clicca su Salva. Seleziona OK nel menu a comparsa successivo.
- Aprire il software Meshmixer (vedere Tabella dei materiali). Fare clic sul pulsante Importa (+ ). Selezionare il file .stl generato nel passaggio precedente. Utilizzate gli strumenti Scolpisci > pennelli > Trascina e scolpisci > Pennelli > Gonfia per estrarre le superfici del modello che devono essere arrotondate.
- Una volta che il modello ha l'aspetto desiderato, salvalo in formato .stl: File > Esporta. Assegnare al file il nome desiderato e selezionare Formato binario STL (*.stl) nel menu a discesa Salva come tipo. Clicca su Salva. Se viene visualizzato un menu a comparsa, fai clic su Continua.
6. 'ili generazione di appezzamenti
- Ottenere le coordinate per le posizioni nel modello 3D che corrispondono ai siti di campionamento.
- Aprire il modello 3D dal passaggio 5.1.3.5 nel software MeshLab : File > Import Mesh. Selezionate il modello generato nel passaggio 5.1.3.5. Fare clic su OK nella finestra pop-up Elaborazione post-apertura .
- Per ottenere le coordinate x, y e z per ogni punto di campionamento: selezionate lo strumento Selettori (PickPoints ), quindi fate clic con il pulsante destro del mouse a intervalli regolarmente distanziati sulla superficie del modello 3D. Una volta selezionate tutte le coordinate desiderate, fai clic sul pulsante Salva più in alto nella finestra pop-up Modulo .
NOTA: le coordinate verranno esportate in formato pp. Questo file può essere aperto in un software per fogli di calcolo. - Nel software del foglio di calcolo: aprire il file .pp generato nel passaggio 6.1.2. Regolare la visualizzazione dei dati utilizzando Dati > testo in colonne > delimitato. Fare clic su Avanti, quindi selezionare Spazio e fare clic su Fine.
- Riformattare in modo che solo i valori numerici rimangano nelle celle del foglio di calcolo selezionando Home > Trova e seleziona > Sostituisci. Nella casella Trova cosa, inserisci: y=". Lasciare vuota la casella Sostituisci con. Fare clic su Sostituisci tutto, quindi su OK. Ripetere per x=" e per z=" e per " />. I valori sono ora pronti per il passaggio 6.2.
- Crea la tabella delle feature 'ili. Questo metodo è stato adattato da Reference16.
- In un software per fogli di calcolo, creare la tabella delle funzionalità. Le righe corrispondono a ciascuna posizione e le colonne ai dati.
NOTA: le prime colonne devono essere il nome della posizione (nome del campione), seguito dalle coordinate x, y e z dei punti di campionamento ottenuti al passaggio 6.1.2 (con intestazioni di colonna x, y, z). La quinta colonna deve essere intitolata "raggio". - Incolla i metadati appropriati e l'abbondanza di funzionalità del metabolita nelle colonne successive del foglio di calcolo.
- Nella colonna "raggio", immettere la dimensione desiderata dei punti di campionamento da visualizzare sul modello. Determinare empiricamente i valori per il raggio: immettere 1 come valore predefinito, quindi valutare se il raggio o le coordinate devono essere regolati nel passaggio 6.3. Salvare il file in formato .csv (File > Salva con nome), quindi selezionare CSV (delimitato da virgole) (*.csv) nel menu a discesa. Assegnare al file il nome desiderato. Clicca su Salva.
- In un software per fogli di calcolo, creare la tabella delle funzionalità. Le righe corrispondono a ciascuna posizione e le colonne ai dati.
- Apri i dati in 'ili (software sviluppato da16).
- Apri il sito web 'ili (ili.embl.de). Selezionate Superficie (Surface). Trascina e rilascia il modello 3D creato nella finestra del browser. Trascinate e rilasciate la tabella delle feature creata nella stessa finestra del browser.
- Utilizzare la legenda nell'angolo in basso a destra per proiettare la colonna di dati desiderata sul modello 3D. Assicurarsi che le macchie e i raggi selezionati nel punto 6.2.1 corrispondano ai siti di campionamento. Se necessario, regolare i valori nella tabella delle feature o scegliere coordinate aggiuntive in MeshLab (vedere il passaggio 6.1.2).
NOTA: la visualizzazione può essere migliorata anche selezionando Spot > Border Opacity e impostando il dispositivo di scorrimento sul suo valore massimo. - Selezionare consecutivamente ogni colonna di dati per valutare la distribuzione di questa caratteristica metabolita sul modello 3D.
NOTA: Questo approccio può anche essere utilizzato per visualizzare solo specifiche caratteristiche metaboliche di interesse, ad esempio quelle con p < 0,05 o una certa variazione di piega tra campioni infetti e non infetti, come determinato in strumenti di analisi statistica esterni. Le funzionalità da visualizzare possono anche essere selezionate al di fuori di 'ili attraverso approcci di apprendimento automatico come una foresta casuale.
- Eseguire la visualizzazione dei dati lineari / di registro secondo i passaggi indicati di seguito.
- Utilizzando la scheda Mappatura in alto a destra della pagina, selezionare Lineare o Logaritmico nel menu a discesa Scala .
- Impostate la stessa scala per tutti i grafici se visualizzate più feature.
NOTA: il sito Web sceglie automaticamente il minimo e il massimo per ogni colonna di dati da visualizzare. - Per impostare manualmente la scala, deselezionate l'opzione Min/Max automatico e immettete il ridimensionamento desiderato. Tutti i dati verranno ora visualizzati all'interno della stessa scala.
- Modificare la scala dei colori dei dati (scala di grigi, blu-bianco-rosso, ecc.).
- Modificare la scala dei colori in base alla combinazione di colori desiderata nel menu Mappatura utilizzando il menu a discesa Mappa colori . Assicurarsi che la combinazione di colori selezionata sia compatibile con i daltonici.
- Per modificare il colore del modello 3D o il colore di sfondo, utilizzare le opzioni Colore e Sfondo nel menu 3D.
- Nascondete gli assi 3D deselezionando la casella Mostra origine (Show the Origin) nel menu 3D.3D.
- Salva l'immagine facendo screenshot, usando lo strumento di cattura o il tasto di scelta rapida Crtl + S per Windows o Linux e
 + S su OS X.
+ S su OS X.
Risultati
Il numero di caratteristiche metaboliche ottenute dipende dal tipo di tessuto analizzato e dai parametri di elaborazione dei dati. Ad esempio, questo protocollo è stato utilizzato per analizzare l'impatto spaziale dell'infezione da T. cruzi sul metaboloma del tratto gastrointestinale in un modello murino di infezione da T. cruzi. Nel lavoro precedente, il maschio C3H / HeJ è stato iniettato per via intraperitoneale con 1.000 cl + luc T. cruzi parassiti32,6. Gli animali sono stati sottoposti a eutanasia 12 o 89 giorni dopo l'infezione e un'analisi cartografica chimica di 13 segmenti contigui del tratto gastrointestinale è stata eseguita come descritto in questo protocollo. Questa analisi ha portato a una tabella di funzionalità di 5.502 feature, che sono state poi visualizzate in 3D utilizzando i passaggi descritti in questo protocollo. Questo approccio consente la visualizzazione delle caratteristiche dei metaboliti in singoli animali che sono elevati nel sito di elevato carico di parassiti (chinurenina, Figura 2B vs carico di parassiti, Figura 2A), di metaboliti con distribuzione differenziale tra le regioni tissutali (glutammina, Figura 2C) e caratteristiche dei metaboliti che si trovano a livelli comparabili nell'intestino tenue e crasso (LPE 16:0 Figura 2D ). La chinurenina è stata selezionata per la visualizzazione a causa della sua nota relazione con l'infiammazione e delle precedenti pubblicazioni sulla capacità dei metaboliti derivati dalla chinurenina di regolare il carico di T. cruzi33. Modelli casuali di apprendimento automatico basati su foreste avevano precedentemente rivelato un'associazione tra i livelli di chinurenina e lo stato di infezione6. La glutammina è stata selezionata per una visualizzazione basata su precedenti pubblicazioni che dimostrano una relazione tra disponibilità di glutammina in vitro e sensibilità al farmaco T. cruzi34. La distribuzione differenziale è stata confermata utilizzando la regressione logistica, p < 0,05. LPE 16:0 è stato selezionato dopo un'ispezione visiva dei dati per scoprire le caratteristiche dei metaboliti trovati a livelli comparabili tra i siti tissutali.
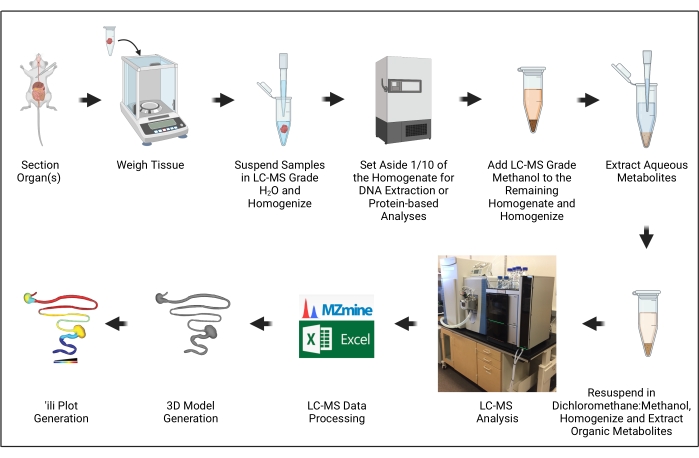
Figura 1: Panoramica del protocollo. L'illustrazione è stata creata con BioRender.com. Fare clic qui per visualizzare una versione più grande di questa figura.
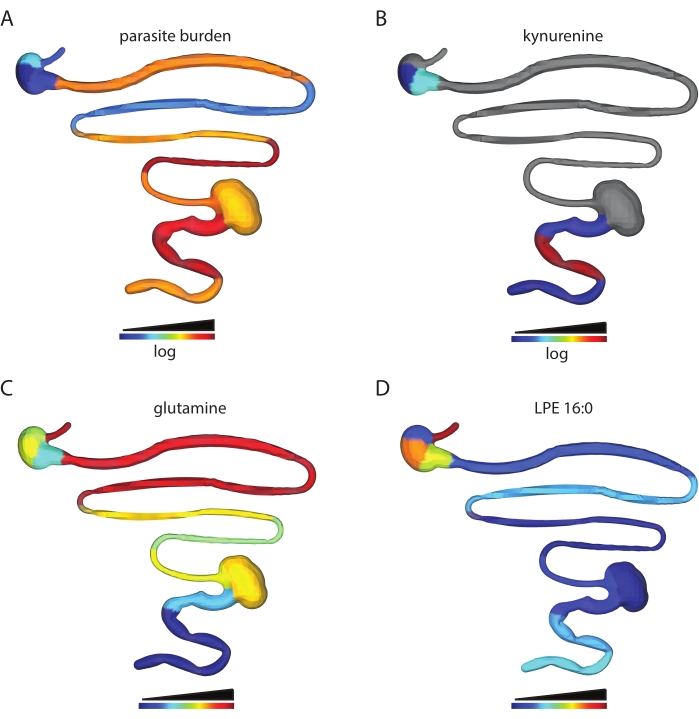
Figura 2: Analisi cartografica chimica. I topi maschi C3H/HeJ sono stati iniettati per via intraperitoneale con 1.000 parassiti CL+luc T. cruzi32. Gli animali sono stati sottoposti a eutanasia 12 o 89 giorni dopo l'infezione e il tratto gastrointestinale è stato raccolto e sezionato sistematicamente (fase 1)6. I metaboliti sono stati estratti come nella fase 2 e analizzati da LC-MS/MS.3D la generazione del modello è stata eseguita utilizzando il software SketchUp (passaggio 5) e i dati sono stati tracciati in 3D come nel passaggio 6. (A) Distribuzione del parassita in un topo specifico, 12 giorni dopo l'infezione. (B) Distribuzione del metabolita chinurenina nello stesso topo, 12 giorni dopo l'infezione. (C) Distribuzione media della glutammina tra topi infetti, 89 giorni dopo l'infezione. (D) Livelli comparabili di m/z 454.292 tempo di ritenzione 2.929 min, annotati come 2-esadecanoil-sn-glicero-3-fosfoetanolammina (LPE 16:0), nello stesso topo come in A e B nell'intestino tenue e nel colon. Campioni e dati sono stati generati in6. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Comprendere le infezioni da tripanosomatidi è essenziale per guidare lo sviluppo di nuovi farmaci e approcci terapeutici. Questo metodo di cartografia chimica è pronto a fornire informazioni utili sulla relazione tra metabolismo e patogenesi della malattia tripanosomatidica, rispondendo così a questa esigenza traslazionale.
Solo i solventi di grado LC-MS sono raccomandati durante l'estrazione dei metaboliti e le analisi ms, per ridurre la contaminazione di fondo. La contaminazione polimerica35, comunemente derivata da film di paraffina e/o altre materie plastiche36,37,38, deve essere evitata ove possibile. Parafilm, in particolare, non deve mai essere utilizzato. Questi aspetti sono cruciali poiché la qualità dei dati LC-MS dipende dai materiali utilizzati durante la preparazione del campione e l'estrazione dei metaboliti. La qualità dei dati dovrebbe essere garantita prima di generare grafici. Inoltre, la generazione di queste mappe metabolomiche spaziali complete richiede la raccolta di tutti i campioni di tessuto adiacenti e l'estrazione di metaboliti da tutti i campioni raccolti per evitare lacune in queste mappe. Le procedure di raccolta, la logistica dell'estrazione dei metaboliti e l'analisi LC-MS e i costi dovrebbero quindi essere considerati e pianificati di conseguenza.
Questo protocollo può essere modificato per soddisfare le esigenze degli utenti in diversi modi. Ad esempio, la polarità e la solubilità dei solventi utilizzati durante l'estrazione dei metaboliti influenzeranno i metaboliti rilevati39. Per massimizzare la diversità delle caratteristiche dei metaboliti rilevati per le analisi cartografiche chimiche non mirate, si raccomanda di combinare più fasi di estrazione e solventi. Ad esempio, questo metodo utilizza diclorometano, metanolo e acqua come solventi di estrazione perché consentono il rilevamento accurato di molecole non polari e polari20,40. Tuttavia, questi solventi non sono universalmente adatti per ogni esperimento di SM e i ricercatori dovrebbero selezionare i solventi da estrazione in base agli obiettivi del loro progetto. Allo stesso modo, possono essere utilizzate diverse condizioni LC-MS/ MS, come la sostituzione della cromatografia a fase inversa con la cromatografia a fase normale. Colonne alternative possono anche essere utilizzate per la raccolta di dati in fase inversa invece della cromatografia C8, anche se empiricamente, la cromatografia C8 è più robusta per i lipidi tissutali e ha una frequenza di intasamento inferiore. Concettualmente, questi protocolli possono essere applicati anche ad altri metodi di spettrometria di massa come la gascromatografia-spettrometria di massa, ecc.
Un approccio alternativo è l'imaging della spettrometria di massa. Infatti, a differenza degli approcci di imaging con spettrometria di massa, la cromatografia liquida-spettrometria di massa non conserva intrinsecamente le informazioni spaziali10. Gli approcci di cartografia chimica colmano questa lacuna includendo la posizione di campionamento al momento della concettualizzazione del progetto, nei metadati del campione e nelle fasi di elaborazione dei dati. Un punto di forza di questo approccio alla cartografia chimica, a differenza dell'imaging con spettrometria di massa, è la capacità di fornire annotazioni sicure (Metabolomics Standards Initiative livello 1 o livello 2 annotation confidence41), a differenza dell'imaging con spettrometria di massa in cui la maggior parte delle applicazioni si basa su una massa accurata solo per l'annotazione. L'imaging con spettrometria di massa consentirà una mappatura spaziale a grana fine, a volte fino al livello di singola cellula, ad esempio 42,43. Al contrario, gli approcci di cartografia chimica consentono la mappatura su larga scala tra organi della distribuzione dei metaboliti senza richiedere competenze di criosezione di animali interi altamente specializzate. La cartografia chimica fornisce prove complementari ai numerosi approcci trascrittomici spaziali in fase di sviluppo, ad esempio 44, con il vantaggio di concentrarsi sullo "strato omico più vicino al fenotipo"45. I metodi alternativi per la quantificazione del carico parassitario includono la misurazione della bioluminescenza al momento della raccolta del campione6. Segmenti fini potrebbero anche essere raccolti per consentire la microscopia confocale o elettronica per valutare il carico parassitario localizzato e il danno tissutale. L'omogeneizzato d'acqua, che viene utilizzato per la quantificazione delle citochine in questo protocollo e nelle pubblicazioni precedenti13, potrebbe anche essere utilizzato per quantificare i marcatori a base proteica del danno tissutale.
Esistono anche diversi modi per ottenere modelli 3D adatti a tracciare i dati LC-MS risultanti. Oltre al metodo suggerito qui, i modelli possono essere acquistati prefabbricati da vari fornitori online. Assicurarsi che i termini di utilizzo corrispondano all'uso previsto, in particolare per quanto riguarda la pubblicazione. I modelli per organi di grandi dimensioni possono essere generati de novo utilizzando scanner 3D secondo le istruzioni dello scanner. Esistono alternative come MATLAB per la generazione e la visualizzazione di modelli 3D per la cartografia chimica46, ma sono state implementate principalmente prima dello sviluppo di 'ili16. MATLAB è una suite di strumenti di analisi e programmazione dei dati che offre un'ampia varietà di applicazioni in molti campi. Tuttavia, MATLAB non è né gratuito né open-source e richiede familiarità con le interfacce MATLAB, soprattutto considerando che MATLAB non è stato sviluppato per l'elaborazione di dati di spettrometria di massa. Le alternative di questo metodo proposto, vale a dire SketchUp, Meshlab e 'ili, sono liberamente accessibili, facili da usare e offrono funzioni simili a MATLAB per scopi di cartografia chimica.
Questo metodo è robusto per quanto riguarda la preparazione del campione e l'estrazione del metabolita. La risoluzione dei problemi è spesso necessaria nella fase di acquisizione dati LC-MS. Questo esula dallo scopo di questo articolo. I lettori sono indirizzati a pubblicazioni eccellenti sulla risoluzione dei problemi di acquisizione dati LC-MS, tra cui 20,47. Allo stesso modo, le complessità dell'annotazione dei metaboliti esulano dallo scopo dell'attenzione di questo metodo sulla generazione di modelli 3D. Riferimenti utili su questo argomento includono24,25,48,49.
Mentre questo metodo esplora efficacemente la patogenesi della malattia, ci sono limitazioni a questo approccio, alcune delle quali sono comuni in qualsiasi esperimento di metabolomica. Una di queste limitazioni è il basso tasso di annotazione delle caratteristiche LC-MS50, che dipende dalla disponibilità e dalla qualità delle librerie spettrali di riferimento. Un'ulteriore limitazione è che questo protocollo non preserva l'mRNA a causa dell'incompatibilità dei reagenti di conservazione dell'RNA come RNAlater con l'analisi LC-MS/MS. Tuttavia, la qualità delle proteine è adeguata per le analisi a valle e quindi può sostituire le analisi basate sull'mRNA.
Un approccio cartografico chimico alla patogenesi delle infezioni riflette direttamente il modo in cui le infezioni batteriche, virali o parassitarie si sviluppano nei sistemi di organi e causano malattie localizzate. L'analisi di questi sottocampioni regionali e la generazione di modelli 3D alla fine trasmettono come i metaboliti funzionano nello spazio tridimensionale, facendo luce su queste dimensioni spaziali precedentemente non riconosciute della biologia molecolare. Utilizzando questo protocollo, ad esempio, la localizzazione dei metaboliti è stata confrontata con il carico di parassiti Trypanosoma cruzi. I risultati hanno chiarito la relazione tra l'agente patogeno e il tessuto ospite e hanno anche dimostrato le dinamiche metaboliche della progressione dei sintomi della malattia di Chagas6. I metodi di cartografia chimica sono stati applicati anche a vari argomenti, come l'interazione dell'ambiente costruito dall'uomo51,52,53, la composizione chimica di sistemi di organi come la pelle umana46 e i polmoni54 e il metabolismo vegetale e le interazioni ambientali55. Le applicazioni future possono comportare la valutazione della tolleranza e della resilienza localizzate alla malattia, o la relazione tra i livelli di metaboliti locali, il tropismo patogeno e il tropismo della malattia in modelli oltre l'infezione da tripanosomatidi. Questo approccio dovrebbe anche avere un'ampia applicabilità per espandere gli attuali protocolli di farmacocinetica, per valutare la relazione tra i livelli locali di farmaci tissutali e il metabolismo dei farmaci rispetto al contesto metabolico generale, al danno tissutale e alla clearance dei patogeni. Nel complesso, la cartografia chimica consente esplorazioni uniche delle distribuzioni dei metaboliti in vari tipi di campioni, con applicazioni che includono la patogenesi delle malattie, la salute umana, le interazioni uomo-ambiente e la dinamica microbica.
Divulgazioni
Nessun conflitto di interessi da segnalare.
Riconoscimenti
Laura-Isobel McCall, PhD, ha ricevuto un Investigators in the Pathogenesis of Infectious Disease Award dal Burroughs Wellcome Fund. Gli autori desiderano inoltre riconoscere il sostegno del numero di premio NIH R21AI148886, una sovvenzione pilota dell'Oklahoma Center for Respiratory and Infectious Diseases (OCRID) con il numero di premio NIH P20GM103648 e fondi di start-up dell'Università dell'Oklahoma (a LIM). Il contenuto è di esclusiva responsabilità degli autori e non rappresenta necessariamente le opinioni ufficiali dei finanziatori. Gli autori desiderano anche ringraziare gli sviluppatori degli strumenti utilizzati in questo protocollo. Sono state citate, ove applicabile, tutte le pubblicazioni pertinenti.
Materiali
| Name | Company | Catalog Number | Comments |
| 1.5 mL Eppendorf tubes | NA | NA | any brand, as available |
| 1 L bottle, pyrex | NA | NA | any brand, as available |
| 1 L graduated cylinder, pyrex | NA | NA | any brand, as available |
| 2 mL SafeLock Eppendorf tubes | VWR | 20901-540 | use the appropriate tube model for the available tissue homogenizer |
| 3D model (de-novo generated according to protocol steps, or purchased) | NA | NA | as appropriate for system under investigation |
| 5 mm stainless steel bead | Qiagen | 69989 | |
| 96 well plate | Fisher | 3252449 | |
| AATTCCTCCAAGCAGCGGATA primer | NA | NA | any brand, as available; published in Piron, M. et al. Development of a real-time PCR assay for Trypanosoma cruzi detection in blood samples. Acta tropica. 103 (3), 195–200 (2007). |
| analytical balance | NA | NA | any brand, as available |
| ASTCGGCTGATCGTTTTCGA primer | NA | NA | any brand, as available; published in Piron, M. et al. Development of a real-time PCR assay for Trypanosoma cruzi detection in blood samples. Acta tropica. 103 (3), 195–200 (2007). |
| benchtop centrifuge with microcentrifuge, falcon tube and 96-well-plate capacity | NA | NA | any brand, as available |
| biosafety cabinet | NA | NA | class II, type A2; any brand, as available |
| CAGCAAGCATCTATGCACTTAG ACCCC primer | NA | NA | any brand, as available; published in Cummings, K.L., Tarleton, R.L. Rapid quantitation of Trypanosoma cruzi in host tissue by real-time PCR. Molecular and biochemical parasitology. 129 (1), 53–59 (2003). |
| camera | NA | NA | any brand, as available. A cellphone camera is adequate for this protocol |
| chemiluminescent-capable imaging system | NA | NA | any system, as available |
| cotton balls | NA | NA | any brand, as available |
| cryogloves | VWR | 97008-208 | replace with any brand, as available |
| dissection scissors | NA | NA | any brand, as available |
| dry ice | NA | NA | any brand, as available |
| extra-length forceps | NA | NA | any brand, as available |
| flammable-grade refrigerator | NA | NA | any brand, as available |
| freezer storage boxes for microcentrifuge tubes | NA | NA | any brand, as available |
| fume hood | NA | NA | any brand, as available |
| high-resolution mass spectrometer | NA | NA | any brand, as available, such as ThermoFisher Q-Exactive Plus (catalog number 0726030) |
| ice bucket | NA | NA | any brand, as available |
| ili software | ili.embl.de | NA | |
| isoflurane | Covetrus | 29405 | |
| large tupperware | NA | NA | any brand, as available; large enough to comfortably contain mouse, cotton ball |
| LC-MS grade acetonitrile | Fisher Optima | A955-4 | |
| LC-MS grade dicholoromethane | Fisher Optima | D151-4 | |
| LC-MS grade formic acid | Fisher Optima | A11750 | |
| LC-MS grade methanol | Fisher Optima | A456-4 | |
| LC-MS grade water | Fisher Optima | W64 | |
| liquid chromatography column | Phenomenex | 00B-4499-AN | may be changed to other brands and models as appropriate for the metabolites of interest |
| liquid chromatography column guard cartridge | Phenomenex | AJ0-8784 | may be changed to other brands and models as appropriate for the metabolites of interest |
| liquid chromatography column guard cartridge holder | Phenomenex | AJ0-9000 | may be changed to other brands and models as appropriate for the metabolites of interest |
| liquid nitrogen | NA | NA | any brand, as available |
| luciferin | Goldbio | LUCK-1G | |
| MeshLab software | https://www.meshlab.net/ | NA | |
| Meshmixer software | https://www.meshmixer.com/ | NA | |
| MS calibrant | NA | NA | appropriate one for available instrument |
| MS data processing software | NA | NA | multiple options available; authors recommend MZmine |
| MSConvert software | http://proteowizard.sourceforge.net/ | NA | |
| Nanodrop | ThermoFisher | ND-ONE-W | other nanodrop models are also suitable |
| p1000 pipet tips | NA | NA | use the appropriate brand to fit available pipettors |
| p1000 pipettor | NA | NA | any brand, as available |
| p20 pipette tips | NA | NA | use the appropriate brand to fit available pipettors |
| p20 pipettor | NA | NA | any brand, as available |
| p200 pipette tips | NA | NA | use the appropriate brand to fit available pipettors |
| p200 pipettor | NA | NA | any brand, as available |
| personal protective equipment (gloves, lab coat, safety glasses/goggles; faceshield) | NA | NA | any brand, as available |
| Q-Plex Mouse Cytokine - Screen (16-Plex) | Quansys biosciences | 110949MS | can replace with other protein-based cytokine assays such as other commercial cytokine ELISA kits |
| Quick-DNA Miniprep Plus Kit (200 preps) | Zymo | D4069 | replace with any brand of mammalian DNA extraction kit, as available |
| real-time thermocycler | NA | NA | any brand, as available |
| salt shaker or tea infuser | NA | NA | any brand; to contain isoflurane-soaked cotton ball and prevent contact with mouse skin |
| SketchUp software | https://www.sketchup.com/ | NA | |
| specimen forceps | NA | NA | any brand, as available |
| speedvac with microcentrifuge tube and 96-well-plate capacity | NA | NA | any brand, as available |
| spreadsheet software | https://www.microsoft.com/en-us/microsoft-365/excel | NA | can replace with other spreadsheet management software, as applicable |
| sulfachloropyridazine | Sigma | S9882-100G | |
| sulfadimethoxine | Sigma | S7007-10G | |
| Sybr green qPCR reaction mix | Fisher | A25780 | can replace with other Sybr green qPCR reaction mixes, as desired |
| TCCCTCTCATCAGTTCTAT GGCCCA primer | NA | NA | any brand, as available; published in Cummings, K.L., Tarleton, R.L. Rapid quantitation of Trypanosoma cruzi in host tissue by real-time PCR. Molecular and biochemical parasitology. 129 (1), 53–59 (2003). |
| tissue homogenizer | NA | NA | any brand, as available; for example, Qiagen TissueLyser II, catalog number 85300, with TissueLyser Adapter Set (2 x 24), catalog number 69982 |
| tissue samples | NA | NA | from appropriate infection model |
| TissueLyser single-bead dispenser | Qiagen | 69965 | |
| UHPLC | NA | NA | any brand, as available, such as ThermoFisher Vanquish (catalog number IQLAAAGABHFAPUMBHV) |
| ultra-low temperature freezer (-80) | NA | NA | any brand, as available |
| ultrasonic bath | NA | NA | any system, as available |
| wet ice | NA | NA | any brand, as available |
| zone-free sealing film | VWR | 490007-390 |
Riferimenti
- McCall, L. -. I., et al. Adaptation of Leishmania donovani to and visceral environments: in vivo selection and proteomic analysis. Journal of Proteome Research. 14 (2), 1033-1059 (2015).
- McCall, L. -. I., Zhang, W. -. W., Matlashewski, G. Determinants for the development of visceral leishmaniasis disease. PLoS Pathogens. 9 (1), 1003053 (2013).
- de Castro Neto, A. L., da Silveira, J. F., Mortara, R. A. Comparative analysis of virulence mechanisms of trypanosomatids pathogenic to humans. Frontiers in Cellular and Infection Microbiology. 11, 669079 (2021).
- Franco, J. R., Simarro, P. P., Diarra, A., Jannin, J. G. Epidemiology of human African trypanosomiasis. Clinical Epidemiology. 6, 257-275 (2014).
- Ottilie, S., et al. Rapid chagas disease drug target discovery using directed evolution in drug-sensitive yeast. ACS Chemical Biology. 12 (2), 422-434 (2017).
- Hossain, E., et al. Mapping of host-parasite-microbiome interactions reveals metabolic determinants of tropism and tolerance in Chagas disease. Science Advances. 6 (30), (2020).
- Parab, A. R., McCall, L. -. I. Tryp-ing up metabolism: Role of metabolic adaptations in kinetoplastid disease pathogenesis. Infection and Immunity. 89 (4), (2021).
- Lewis, C. M., McCall, L. -. I., Sharp, R. R., Spicer, P. G. Ethical priority of the most actionable system of biomolecules: the metabolome. American Journal of Physical Anthropology. 171 (2), 177-181 (2020).
- Liu, Z., Ulrich vonBargen, R., McCall, L. I. Central role of metabolism in Trypanosoma cruzi tropism and Chagas disease pathogenesis. Current Opinion in Microbiology. 63, 204-209 (2021).
- Newsom, S. N., McCall, L. -. I. Metabolomics: Eavesdropping on silent conversations between hosts and their unwelcome guests. PLoS Pathogens. 14 (4), 1006926 (2018).
- Wishart, D. S., et al. HMDB: the Human Metabolome Database. Nucleic Acids Research. 35, 521-526 (2007).
- Dean, D. A., et al. Spatial metabolomics identifies localized chemical changes in heart tissue during chronic cardiac Chagas disease. PLoS Neglected Tropical Diseases. 15 (10), 0009819 (2021).
- McCall, L. -. I., et al. Mass spectrometry-based chemical cartography of a cardiac parasitic infection. Analytical Chemistry. 89 (19), 10414-10421 (2017).
- Hoffman, K., et al. Alterations to the cardiac metabolome induced by chronic infection relate to the degree of cardiac pathology. ACS Infectious Diseases. 7 (6), 1638-1649 (2021).
- Parab, A. R., et al. Dysregulation of glycerophosphocholines in the cutaneous lesion caused by Leishmania major in experimental murine models. Pathogens. 10 (5), 593 (2021).
- Protsyuk, I., et al. 3D molecular cartography using LC-MS facilitated by Optimus and 'ili software. Nature Protocols. 13 (1), 134-154 (2018).
- McCall, L. -. I., et al. Targeting ergosterol biosynthesis in Leishmania donovani: Essentiality of Sterol 14alpha-demethylase. PLOS Neglected Tropical Diseases. 9 (3), 0003588 (2015).
- Tear, W. F., et al. Selectivity and physicochemical optimization of repurposed Pyrazolo[1,5-]pyridazines for the treatment of human African trypanosomiasis. Journal of Medicinal Chemistry. 63 (2), 756-783 (2020).
- Klug, D. M., et al. Hit-to-lead optimization of benzoxazepinoindazoles as human African trypanosomiasis therapeutics. Journal of Medicinal Chemistry. 63 (5), 2527-2546 (2020).
- Want, E. J., et al. Global metabolic profiling of animal and human tissues via UPLC-MS. Nature Protocols. 8 (1), 17-32 (2013).
- Piron, M., et al. Development of a real-time PCR assay for Trypanosoma cruzi detection in blood samples. Acta Tropica. 103 (3), 195-200 (2007).
- Cummings, K. L., Tarleton, R. L. Rapid quantitation of Trypanosoma cruzi in host tissue by real-time PCR. Molecular and Biochemical Parasitology. 129 (1), 53-59 (2003).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2−ΔΔCT method. Methods. 25 (4), 402-408 (2001).
- Nothias, L. -. F., et al. Feature-based molecular networking in the GNPS analysis environment. Nature Methods. 17 (9), 905-908 (2020).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with global natural products social molecular networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Chambers, M. C., et al. A cross-platform toolkit for mass spectrometry and proteomics. Nature Biotechnology. 30 (10), 918-920 (2012).
- Tsugawa, H., et al. MS-DIAL: data-independent MS/MS deconvolution for comprehensive metabolome analysis. Nature Methods. 12 (6), 523-526 (2015).
- Sturm, M., et al. OpenMS - an open-source software framework for mass spectrometry. BMC Bioinformatics. 9, 163 (2008).
- Pluskal, T., Castillo, S., Villar-Briones, A., Oresic, M. MZmine 2: modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11, 395 (2010).
- Smith, C. A., Want, E. J., O'Maille, G., Abagyan, R., Siuzdak, G. XCMS: processing mass spectrometry data for metabolite profiling using nonlinear peak alignment, matching, and identification. Analytical Chemistry. 78 (3), 779-787 (2006).
- . FBMN with MZmine Available from: https://ccms-ucsd.github.io/GNPSDocumentation/featurebasedmolecularnetworking-with-mzmine2/ (2021)
- Lewis, M. D., et al. Bioluminescence imaging of chronic Trypanosoma cruzi infections reveals tissue-specific parasite dynamics and heart disease in the absence of locally persistent infection. Cellular Microbiology. 16 (9), 1285-1300 (2014).
- Knubel, C. P., et al. 3-Hydroxy kynurenine treatment controls T. cruzi replication and the inflammatory pathology preventing the clinical symptoms of chronic Chagas disease. PloS One. 6 (10), 26550 (2011).
- Dumoulin, P. C., Vollrath, J., Tomko, S. S., Wang, J. X., Burleigh, B. Glutamine metabolism modulates azole susceptibility in amastigotes. eLife. 9, (2020).
- Keller, B. O., Sui, J., Young, A. B., Whittal, R. M. Interferences and contaminants encountered in modern mass spectrometry. Analytica Chimica Acta. 627 (1), 71-81 (2008).
- Weaver, R., Riley, R. J. Identification and reduction of ion suppression effects on pharmacokinetic parameters by polyethylene glycol 400. Rapid Communications in Mass Spectrometry: RCM. 20 (17), 2559-2564 (2006).
- Larger, P. J., Breda, M., Fraier, D., Hughes, H., James, C. A. Ion-suppression effects in liquid chromatography-tandem mass spectrometry due to a formulation agent, a case study in drug discovery bioanalysis. Journal of Pharmaceutical and Biomedical Analysis. 39 (1-2), 206-216 (2005).
- Furey, A., Moriarty, M., Bane, V., Kinsella, B., Lehane, M. Ion suppression; a critical review on causes, evaluation, prevention and applications. Talanta. 115, 104-122 (2013).
- Lu, W., et al. Metabolite measurement: Pitfalls to avoid and practices to follow. Annual Review of Biochemistry. 86, 277-304 (2017).
- Masson, P., Alves, A. C., Ebbels, T. M. D., Nicholson, J. K., Want, E. J. Optimization and evaluation of metabolite extraction protocols for untargeted metabolic profiling of liver samples by UPLC-MS. Analytical Chemistry. 82 (18), 7779-7786 (2010).
- Sumner, L. W., et al. Proposed minimum reporting standards for chemical analysis. Metabolomics. 3 (3), 211-221 (2007).
- Rappez, L., et al. SpaceM reveals metabolic states of single cells. Nature Methods. 18 (7), 799-805 (2021).
- Yang, B., Tsui, T., Caprioli, R. M., Norris, J. L. Sample preparation and analysis of single cells using high performance MALDI FTICR mass spectrometry. Methods in Molecular Biology. 2064, 125-134 (2020).
- Longo, S. K., Guo, M. G., Ji, A. L., Khavari, P. A. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nature Reviews. Genetics. 22 (10), 627-644 (2021).
- Patti, G. J., Yanes, O., Siuzdak, G. Innovation: Metabolomics: the apogee of the omics trilogy. Nature Reviews. Molecular Cell Biology. 13 (4), 263-269 (2012).
- Bouslimani, A., et al. Molecular cartography of the human skin surface in 3D. Proceedings of the National Academy of Sciences of the United States of America. 112 (17), 2120-2129 (2015).
- Dudzik, D., Barbas-Bernardos, C., García, A., Barbas, C. Quality assurance procedures for mass spectrometry untargeted metabolomics. A review. Journal of Pharmaceutical and Biomedical Analysis. 147, 149-173 (2018).
- Alseekh, S., et al. Mass spectrometry-based metabolomics: a guide for annotation, quantification and best reporting practices. Nature Methods. 18 (7), 747-756 (2021).
- Chaleckis, R., Meister, I., Zhang, P., Wheelock, C. E. Challenges, progress and promises of metabolite annotation for LC-MS-based metabolomics. Current Opinion in Biotechnology. 55, 44-50 (2019).
- Viant, M. R., Kurland, I. J., Jones, M. R., Dunn, W. B. How close are we to complete annotation of metabolomes. Current Opinion in Chemical Biology. 36, 64-69 (2017).
- Kapono, C. A., et al. Creating a 3D microbial and chemical snapshot of a human habitat. Scientific Reports. 8 (1), 3669 (2018).
- Petras, D., et al. Mass spectrometry-based visualization of molecules associated with human habitats. Analytical Chemistry. 88 (22), 10775-10784 (2016).
- McCall, L. -. I., et al. Analysis of university workplace building surfaces reveals usage-specific chemical signatures. Building and Environment. 162, 106289 (2019).
- Garg, N., et al. Three-dimensional microbiome and metabolome cartography of a diseased human lung. Cell Host & Microbe. 22 (5), 705-716 (2017).
- Floros, D. J., et al. Mass spectrometry based molecular 3D-cartography of plant metabolites. Frontiers in Plant Science. 8, 429 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon