Method Article
Approches de cartographie chimique pour étudier l’infection à trypanosomatidé
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce protocole décrit les étapes pour générer un modèle 3D de la distribution des métabolites pendant l’infection par trypanosomatide, y compris la collecte d’échantillons, l’extraction de métabolites, un aperçu de l’acquisition de données de chromatographie de masse en tandem et de chromatographie liquide, la génération de modèles 3D et, enfin, la visualisation de données.
Résumé
Le tropisme des agents pathogènes et le tropisme des maladies désignent les emplacements tissulaires colonisés de manière sélective ou endommagés par des agents pathogènes, ce qui entraîne des symptômes localisés de la maladie. Les parasites trypanosomatidés infectieux par l’homme comprennent Trypanosoma cruzi, l’agent causal de la maladie de Chagas; Trypanosoma brucei, l’agent causal de la maladie du sommeil; et les espèces de Leishmania , agents responsables de la leishmaniose. Ensemble, ils touchent 20 millions de personnes à travers le monde. Ces parasites montrent un tropisme spécifique: cœur, œsophage, côlon pour T. cruzi, tissu adipeux, pancréas, peau, système circulatoire et système nerveux central pour T. brucei, peau pour les souches de Leishmania dermropiques et foie, rate et moelle osseuse pour les souches viscérotropes de Leishmania . Une perspective spatiale est donc essentielle pour comprendre la pathogenèse de la maladie trypanosomatide. La cartographie chimique génère des visualisations 3D de l’abondance de petites molécules générées par chromatographie liquide-spectrométrie de masse, par rapport aux paramètres microbiologiques et immunologiques. Ce protocole montre comment la cartographie chimique peut être appliquée pour étudier les processus pathogènes au cours de l’infection par trypanosomatide, en commençant par l’échantillonnage systématique des tissus et l’extraction des métabolites, suivis de l’acquisition de données par chromatographie liquide et spectrométrie de masse en tandem, et se terminant par la génération de cartes 3D de la distribution des métabolites. Cette méthode peut être utilisée pour de multiples questions de recherche, telles que les besoins en nutriments pour la colonisation tissulaire par T. cruzi, T. brucei ou Leishmania, l’immunométabolisme aux sites d’infection et la relation entre la perturbation métabolique tissulaire locale et les symptômes cliniques de la maladie, conduisant à un aperçu complet de la pathogenèse de la maladie trypanosomatide.
Introduction
Les parasites trypanosomatidés sont constitués d’espèces de Leishmania, de trypanosomes africains (Trypanosoma brucei) et de trypanosomes américains (Trypanosoma cruzi). Les protozoaires leishmaniaques provoquent la leishmaniose, qui comprend l’auto-guérison et la leishmaniose cutanée localisée auto-limitée, la leishmaniose muco-cutanée dans laquelle les tissus muqueux de la bouche, du nez et de la gorge sont endommagés, et la leishmaniose viscérale avec tropisme parasitaire aux organes viscéraux provoquant de la fièvre et de l’hépatosplénomégalie1,2. T. brucei est à l’origine de la trypanosomiase humaine africaine (THA), également connue sous le nom de maladie du sommeil, principalement signalée dans les pays africains3. Les signes et symptômes cliniques comprennent l’hépatosplénomégalie, la fièvre, les maux de tête, les douleurs musculo-squelettiques, les lymphadénopathies et l’anémie au stade hémo-lymphatique lorsque les parasites se localisent dans la circulation sanguine et lymphatique. Ceci est suivi par le stade méningo-encéphalitique, où les parasites se localisent dans le système nerveux central et provoquent des troubles du sommeil, une altération du comportement et éventuellement un comas mortel4. T. cruzi provoque la maladie de Chagas, endémique dans les Amériques. Les personnes infectées connaissent un stade aigu initial, généralement asymptomatique, avec un tropisme parasitaire large. Environ 10 % à 30 % des personnes infectées présentent des symptômes au stade chronique après des décennies d’infection, caractérisés par un mégaœsophage, un mégacôlon et des complications cardiovasculaires5,6.
La métabolomique étudie les petites espèces moléculaires (50-1 500 Da), y compris les composés biologiques issus du métabolisme primaire ou secondaire et les composés dérivés de l’extérieur tels que les médicaments ou les molécules dérivées des aliments. Dans le contexte des interactions hôte-pathogène, la métabolomique peut explorer l’impact de l’infection sur les environnements de métabolites de l’hôte, ce qui est crucial pour accéder à l’effet de l’agent pathogène sur l’hôte. Il peut également évaluer les adaptations des agents pathogènes à l’environnement nutritionnel et immunologique de l’hôte7,8,9. La spectrométrie de masse (SEP) et la spectroscopie par résonance magnétique nucléaire (RMN) sont des outils métabolomiques courants utilisés pour identifier, quantifier et caractériser les métabolites. Cette approche « omique » peut également être appliquée à la découverte de biomarqueurs et au développement de médicaments10,11.
Compte tenu du tropisme tissulaire spécifique des parasites trypanosomatidés, les analyses métabolomiques spatiales peuvent permettre de mieux comprendre la pathogenèse des maladies qu’ils provoquent. La cartographie de la distribution spatiale des métabolites a révélé des métabolites localement affectés par une infection chronique à Trypanosoma cruzi dans le tissu cardiaque de la souris et une infection aiguë et à long terme à Trypanosoma cruzi dans le tractus gastro-intestinal de la souris6,12,13. Plus précisément, la cartographie chimique 3D a démontré une déconnexion entre la persistance du parasite et les altérations métaboliques dans le tissu cardiaque de souris chroniquement infectées par Trypanosoma cruzi. Le métabolisme était le plus perturbé dans les segments inférieurs et apicals du cœur, correspondant aux sites de symptômes de la maladie de Chagas (anévrismes apicals cardiaques). Les familles de métabolites perturbées par l’infection à des sites cardiaques spécifiques et corrélées à la gravité de la maladie comprennent les acylcarnitines et la glycérophosphocholines12,13,14. Dans le tractus gastro-intestinal, des altérations métaboliques persistantes concordaient avec les sites de symptômes de la maladie de Chagas: œsophage et côlon. En revanche, le métabolisme est renormalisé à des sites non associés aux symptômes de la maladie de Chagas, tels que l’intestin grêle. Les métabolites localement perturbés par une infection dans le tractus gastro-intestinal comprennent les acylcarnitines, la glycérophosphocholines, la kynurénine, le tryptophane et l’acide cholique. De plus, ces analyses ont permis d’identifier un nouveau mécanisme métabolique de tolérance à la maladie de Chagas6. L’application de ces méthodes à l’étude de la leishmaniose cutanée a révélé des perturbations métaboliques importantes sur le site de la lésion, mais aussi des changements métaboliques spécifiques dans les tissus sains macroscopiquement adjacents à la lésion. Par exemple, la glutamine a été épuisée au site de la lésion, tandis que la glycérophosphocholines dans le m/z (rapport masse/charge) 200-299, 400-499, 500-599 et 600-699 a été significativement augmentée au site de la lésion. La PC (O-34:1) n’a été augmentée qu’aux sites adjacents aux lésions15.
L’objectif de ce manuscrit est de démontrer les étapes nécessaires pour générer des modèles 3D de distribution des métabolites (« cartographie chimique ») appliqués aux modèles d’infection parasitaire trypanosomatide (Figure 1). Cette approche s’appuie sur plusieurs avancées critiques dans le contexte du traitement des données métabolomiques et métabolomiques, en particulier le développement d’un logiciel 'ili pour tracer facilement les données métabolomiques sur des modèles 3D16.
Protocole
Toutes les expériences sur les animaux décrites ont été approuvées par l’Université de l’Oklahoma ou le Comité institutionnel de soins et d’utilisation des animaux de l’Université de Californie à San Diego. Toutes les étapes de manipulation des matières infectieuses ont été effectuées à l’intérieur d’une armoire de biosécurité (classe II, type A2) et conformément à la réglementation locale.
1. Prélèvement de tissus
- Infecter les modèles animaux d’infection trypanosomatidé appropriés, généralement des souris ou des hamsters.
REMARQUE: Il existe une variété considérable de modèles murins pour l’infection à la trypanosomatide, en fonction des symptômes souhaités à produire, de la vitesse de progression de la maladie, de la gravité de la maladie, etc. Les utilisateurs peuvent choisir à leur convenance.- Pour les modèles d’infection cutanée par leishmaniose, infecter par voie sous-cutanée dans le coussinet plantaire ou par voie intradermique dans l’oreille15.
- Pour les modèles d’infection par leishmaniose viscérale, infecter par voie intraveineuse1,17.
- Pour les modèles d’infection par la maladie de Chagas et la maladie du sommeil, infecter par voie intrapéritonéale6,12,18,19. Déterminer la dose de parasite à utiliser en fonction de la souche de parasite et des points de temps prévus.
- Planifier les positions de sectionnement.
- Prévoyez de générer des sections avec un minimum de 10 mg de tissu par section. Il est préférable d’utiliser 30-50 mg.
- Pour les modèles d’infection par la maladie de Chagas, prévoyez de collecter systématiquement les segments cardiaques et gastro-intestinaux.
- Pour les modèles d’infection par leishmaniose cutanée, prélever des échantillons de tissus lésionnels et adjacents aux lésions.
- Pour les modèles d’infection par la leishmaniose viscérale, prévoyez de recueillir la rate et plusieurs lobes du foie. D’autres sites tissulaires tels que le tissu adipeux peuvent également être intéressants à prélever.
MISE EN GARDE : Les échantillons provenant d’animaux infectés doivent être manipulés conformément au protocole de biosécurité approprié approuvé par l’établissement. Cela implique généralement des exigences en matière d’équipement de protection individuelle (EPI) et uniquement l’ouverture des tubes et le prélèvement d’échantillons à l’intérieur d’une armoire de biosécurité (classe II, type A2).
- Étiqueter et peser les tubes pour l’homogénéisation.
- Utilisez le type de tube approprié pour le système d’homogénéisation disponible. Pour un TissueLyser, utilisez des tubes de microcentrifugation de 2 mL.
- Euthanasier les souris aux moments d’infection souhaités en utilisant un surdosage d’isoflurane tel qu’approuvé par l’IACUC institutionnel ou selon le protocole approuvé par l’IACUC.
- Section systématique des tissus comme prévu, avec une section par tube.
- N’oubliez pas de laver l’équipement de prélèvement des échantillons entre les échantillons avec un solvant d’extraction (50% de méthanol dans ce protocole).
- En gardant le couvercle du tube ouvert, enclenchez immédiatement les échantillons de congélation dans de l’azote liquide.
ATTENTION : Ne fermez pas les tubes tant que l’azote liquide n’est pas entré dans le tube ne s’est pas complètement évaporé pour éviter que les tubes n’explosent à mesure que l’azote se dilate. Prenez les mesures adéquates pour vous assurer que la peau n’entre pas en contact avec de l’azote liquide (utilisez des gants cryogéniques et des pinces pour tenir les tubes). Portez un écran facial de sécurité. - Conservez les tubes sur de la glace carbonique jusqu’à ce que tous les échantillons souhaités aient été prélevés.
- Point de pause : conserver les échantillons à -80 °C jusqu’à ce qu’ils soient prêts à faire des extractions.
- Peser les tubes pour déterminer le poids de l’échantillon de tissu. Enregistrer dans une feuille de calcul.
- Gardez les échantillons congelés pendant le processus de pesée: gardez les tubes sur de la glace carbonique, pesez rapidement, remettez immédiatement sur de la glace sèche. Ne laissez pas les échantillons décongeler pendant la pesée.
2. Extraction des métabolites
REMARQUE: Seuls les liquides et réactifs de qualité LC-MS doivent être utilisés partout. Cette méthode a été adaptée de Reference20.
- Préparer tous les solvants d’extraction (qualité LC-MS H2O, méthanol de qualité LC-MS, enrichi de 4 μM de sulfachlorpyridazine, dichlorométhane de qualité LC-MS : méthanol enrichi de 2 μM de sulfachlorpyridazine) dans des bouteilles en verre de 1 L, à l’aide de verrerie dédiée.
REMARQUE : Le volume à préparer doit être calculé en fonction du poids de l’échantillon, en tenant compte de 500 μL d’eau par 50 mg d’échantillon, de 500 μL de méthanol enrichi de 4 μM de sulfachlorpyridazine par 50 mg d’échantillon et de 1 000 μL de dichlorométhane précraissé : méthanol enrichi de 2 μM de sulfachlorpyridazine par 50 mg d’échantillon, ce qui augmente le volume calculé de 10 % pour tenir compte de l’imprécision du pipetage. Conserver le solvant d’extraction à 4 °C au moins toute la nuit pour pré-refroidir. - Effectuer une homogénéisation à base d’eau des échantillons de tissus selon les étapes mentionnées ci-dessous.
- Ajouter une bille d’acier inoxydable de 5 mm (voir tableau des matériaux) à chacun des tubes de microcentrifugation de 2 mL contenant des échantillons de tissu, à l’aide d’un distributeur de billes. Gardez les tubes sur la glace.
- Fabriquez un tube vierge contenant du H2O de qualité LC-MS qui passera par toutes les étapes et servira d’ébauche d’extraction. Utilisez le volume moyen de H2O provenant des extractions d’échantillons. Ajouter le LC-MS réfrigéré de qualité H2O aux échantillons de tissus congelés.
- Normaliser le volume d’eau au poids des tissus en ajoutant 500 μL d’eau / 50 mg d’échantillon, en utilisant les poids d’échantillon calculés à l’étape 1.7.
MISE EN GARDE : Si vous manipulez des échantillons biodangereux, continuez de suivre le protocole de biosécurité approprié approuvé par l’établissement. Cela implique généralement des exigences pour l’équipement de protection individuelle (EPI) et les tubes d’ouverture uniquement à l’intérieur de l’armoire de biosécurité (classe II, type A2).
- Normaliser le volume d’eau au poids des tissus en ajoutant 500 μL d’eau / 50 mg d’échantillon, en utilisant les poids d’échantillon calculés à l’étape 1.7.
- Homogénéiser les échantillons à une vitesse de 25 Hz pendant 3 min à l’aide d’un homogénéisateur tissulaire (voir Tableau des matériaux).
- Fermez hermétiquement les tubes pour éviter de renverser les réactifs pendant le traitement.
- Collectez environ 1/10e du volume d’homogénéisation pour l’extraction de l’ADN, la qPCR, les analyses à base de protéines ou d’autres analyses (si vous le souhaitez). Conserver dans un tube de microcentrifugation ou une plaque de 96 puits (selon le volume collecté) jusqu’à 6 mois à -80 °C, si des expériences d’extraction d’ADN doivent être effectuées un jour différent. Des durées de stockage plus longues peuvent être possibles, mais n’ont pas été testées.
- Effectuer des extractions d’ADN à l’aide de n’importe quel kit commercial standard pour l’extraction d’ADN de mammifères (voir la Table des matériaux) à partir de tissus comme décrit dans la référence 13. Quantifier le rendement en ADN et stocker l’ADN extrait à -20 °C. Une quantité et une qualité d’ADN acceptables pour la qPCR ont été observées même jusqu’à 3 ans plus tard.
REMARQUE: Cette étape peut être effectuée sur l’homogénat congelé un jour suivant à partir de l’extraction des métabolites. - Effectuer la qPCR comme décrit dans la référence 13, en utilisant 180 ng d’ADN extrait.
REMARQUE: Cela peut être effectué sur l’ADN prélevé un jour précédent et congelé à -20 ° C.- Dans le cas d’études sur l’infection à T. cruzi, utilisez les amorces suivantes : ASTCGGCTGATCGTTTTCGA et AATTCCTCCAAGCAGCGGATA pour quantifier les niveaux de parasites21 et les amorces suivantes pour normaliser les niveaux d’ADN hôte : TCCCTCTCATCAGTTCTATGGCCCA et CAGCAAGCATCTATGCACTTAGACCCC22 (voir tableau des matériaux).
REMARQUE: Les cycles de qPCR recommandés sont les suivants: dénaturer à 95 ° C pendant 10 min; effectuer 40 cycles à 95 °C pendant 30 s, puis 58 °C pendant 60 s, et enfin 72 °C pendant 60 s. Effectuer une analyse de la courbe de fusion appropriée pour le thermocycleur disponible. Traiter les données à l’aide de la méthode ΔΔCt23 pour obtenir une charge parasitaire relative entre les sites d’échantillonnage. La quantification absolue peut être obtenue en comparant les valeurs ΔΔCt dérivées de l’échantillon à une courbe standard générée à partir de quantités connues de parasites, enrichie dans des échantillons de tissus non infectés et extraite comme aux étapes 2.2 à 2.2.4.1.
- Dans le cas d’études sur l’infection à T. cruzi, utilisez les amorces suivantes : ASTCGGCTGATCGTTTTCGA et AATTCCTCCAAGCAGCGGATA pour quantifier les niveaux de parasites21 et les amorces suivantes pour normaliser les niveaux d’ADN hôte : TCCCTCTCATCAGTTCTATGGCCCA et CAGCAAGCATCTATGCACTTAGACCCC22 (voir tableau des matériaux).
- Effectuer une caractérisation des réponses immunitaires à base de protéines à l’aide de kits de cytokines multiplexées ou de kits ELISA commerciaux standard (voir la table des matériaux) comme décrit dans la référence 13 sur l’homogénat stocké.
- Effectuer des extractions d’ADN à l’aide de n’importe quel kit commercial standard pour l’extraction d’ADN de mammifères (voir la Table des matériaux) à partir de tissus comme décrit dans la référence 13. Quantifier le rendement en ADN et stocker l’ADN extrait à -20 °C. Une quantité et une qualité d’ADN acceptables pour la qPCR ont été observées même jusqu’à 3 ans plus tard.
- Économisez au moins la moitié des 500 μL du volume d’homogénéisation pour l’extraction des métabolites.
- Effectuer une extraction aqueuse des métabolites.
REMARQUE: La sélection des solvants peut être adaptée en fonction des propriétés chimiques des métabolites d’intérêt.- Ajouter du méthanol glacé de qualité LC-MS enrichi de 4 μM de sulfachlorpyridazine à l’homogénat pour obtenir une concentration finale de 50 % de méthanol avec 2 μM de sulfachlorpyridazine dans l’eau.
ATTENTION : Le méthanol est inflammable et dangereux. Utilisez des procédures de sécurité appropriées, y compris la manipulation à l’intérieur d’une hotte ou d’une armoire de biosécurité. - Homogénéiser les échantillons dans un homogénéisateur tissulaire à une vitesse de 25 Hz pendant 3 min. Échantillons de centrifugeuse à 16 000 x g à 4 °C pendant 10 min.
- Collectez un volume égal de surnageant dans une plaque de 96 puits. Sélectionnez le volume qui correspond au plus petit volume de méthanol + eau combiné sur tous les échantillons. C’est la fraction aqueuse.
- Réserver tout surnageant homogène aqueux restant à -80 °C comme sauvegarde.
- Conservez les résidus solides sur la glace pendant la collecte des surnageants.
- Extraction aqueuse sèche surnageante jusqu’à ce qu’elle soit sèche (~3 h ou pendant la nuit). Utilisez la vitesse maximale et pas de chauffage.
- Lyophiliser la plaque séchée à 96 puits à -80 °C.
- Ajouter du méthanol glacé de qualité LC-MS enrichi de 4 μM de sulfachlorpyridazine à l’homogénat pour obtenir une concentration finale de 50 % de méthanol avec 2 μM de sulfachlorpyridazine dans l’eau.
- Effectuer l’extraction de métabolites organiques.
REMARQUE: La sélection des solvants peut être adaptée en fonction des propriétés chimiques des métabolites d’intérêt.- Ajouter 1 000 μL par 50 mg de l’échantillon de dichlorométhane précroché : méthanol enrichi de 2 μM de sulfachlorpyridazine au résidu solide de l’étape 2.3.4.
ATTENTION : Utilisez les procédures de sécurité appropriées lors de la manipulation de solvants, y compris la manipulation à l’intérieur d’une hotte avec un bon débit. - Homogénéiser les échantillons dans un homogénéisateur tissulaire à une vitesse de 25 Hz pendant 5 min. Centrifuger les échantillons à 16 000 x g à 4 °C pendant 10 min.
- Collectez un volume égal de surnageant dans une plaque de 96 puits. Sélectionnez le volume correspondant au plus petit volume de dichlorométhane : le méthanol, sur tous les échantillons. C’est la fraction organique.
- Conservez la pastille à -80 °C comme sauvegarde. Conservez le reste de l’extrait organique à -80 °C comme sauvegarde. Sécher à l’air libre l’extrait organique dans une hotte pendant la nuit.
- Lyophiliser la plaque séchée à 96 puits à -80 °C.
- Ajouter 1 000 μL par 50 mg de l’échantillon de dichlorométhane précroché : méthanol enrichi de 2 μM de sulfachlorpyridazine au résidu solide de l’étape 2.3.4.
3. Acquisition de données LC-MS
- Remettre en suspension des extraits aqueux et organiques en 60 μL chacun de 50% de méthanol + 2 μM de sulfadiméthoxine, et combiner. Sonicate pendant 10 min; ensuite, centrifugez pendant 10 min et transférez le surnageant dans une plaque propre de 96 puits. Scellez avec un joint de plaque sans zone et placez la plaque dans un échantillonneur automatique LC.
ATTENTION : Utilisez les procédures de sécurité appropriées lors de la manipulation de solvants, y compris la manipulation à l’intérieur d’une hotte avec un bon débit. - Connectez les phases mobiles appropriées au système LC (voir tableau des matériaux).
REMARQUE: Pour la LC en phase inversée en mode positif, les auteurs recommandent LC-MS-grade H2O + 0,1% d’acide formique comme phase mobile A et LC-MS-grade acétonitrile + 0,1% d’acide formique comme phase B mobile avec un débit de 0,5 mL / min et un gradient LC de 7,5 min. Les pas de gradient recommandés sont ceux publiés dans la Référence6 : 0-1 min, 2 % B ; 1-2,5 min, augmentation linéaire à 98% B; 2,5-4,5 min, tenir à 98% B; 4,5-5,5 min, diminution linéaire à 2% B; 5,5-7,5 min, tenir à 2% B. - Assurez-vous que l’instrument est propre. Calibrez la SEP en mode positif et négatif.
- Effectuer l’évaluation du rendement de la SP, le cas échéant pour l’instrument.
- Créez une séquence d’exécution MS.
- Commencez avec 2 ébauches, 2 étalons (6 mélanges) et 5 contrôles de qualité (CQ) regroupés dans une série de dilution, en commençant à 2 μL de volume d’injection et en augmentant progressivement jusqu’à 30 μL de volume d’injection.
- Randomisez l’ordre de l’échantillon.
- Après chaque 12 échantillons, exécutez un blanc, puis un QC groupé.
- Connectez la colonne C8 LC (taille des particules de 1,7 μm, taille des pores de 100 Å, longueur de 50 x 2,1 mm x diamètre interne) et surveillez les fuites et la contre-pression excessive. Résoudre les problèmes conformément à la procédure d’exploitation standard de l’instrument.
- Démarrez la séquence d’exécution MS et collectez les données LC-MS/MS dépendantes des données.
REMARQUE: Pour un instrument Q-Exactive Plus MS, utilisez l’ionisation par électropulvérisation chauffée et l’acquisition MS2 dépendante des données (top 5) en mode positif, une résolution de 70 000 pour MS1 et 17 500 pour MS2, une cible AGC de 1E6 pour MS1 et 2E5 pour MS2, une informatique maximale de 100 ms pour MS1 et MS2, une plage de balayage de 100-1500 m /z pour MS1, et fenêtre d’isolation MS2 de 1 m/z. Réglez le gaz de gaine à 35, le gaz auxiliaire à 10 et le gaz de balayage à 0, la tension de pulvérisation à 3,8 kV, la température capillaire à 320 °C, le niveau RF de la lentille S à 50 et la température du gaz auxiliaire à 350 °C.- Vérifier la qualité des données : vérifier les blancs initiaux (confirmer l’absence de pics majeurs), les normes (confirmer la présence de pics attendus et la forme symétrique des pics) et les QC (confirmer la présence des pics attendus, la forme des pics et l’intensité des pics attendus).
- Surveillez périodiquement l’exécution de MS pendant la séquence d’exécution.
- Une fois l’exécution terminée, vérifiez les données pour toute injection manquée ou d’autres erreurs.
- Conservez la colonne LC comme recommandé par le fabricant. Prélever et conserver les échantillons à -80 °C.
- Téléchargez les données brutes dans le référentiel de données.
REMARQUE : MassIVE (massive.ucsd.edu) est recommandé pour activer le lien en aval vers la mise en réseau moléculaire pour les annotations de métabolites24,25.
4. Traitement des données LC-MS
- Convertissez les fichiers bruts au format ouvert (.mzXML ou .mzML) à l’aide de MSConvert26.
- Téléchargez les données brutes et les données mzXML ou mzML dans le référentiel de données.
- Générez une table de fonctionnalités. Il existe plusieurs outils pour le faire (MZmine, MS-DIAL, openMS, XCMS, etc.27,28,29,30).
REMARQUE: MZmine est recommandé car il est gratuit, open-source et peut importer directement des fichiers mzXML après MSconvert, dispose d’options d’interface utilisateur graphique pour le traitement des données et la surveillance de l’impact de la sélection des paramètres, et peut exporter directement vers GNPS pour la mise en réseau moléculaire24.
REMARQUE : Suivez la documentation de l’outil et utilisez les paramètres appropriés à l’instrument disponible. Des détails supplémentaires peuvent également être trouvés dans Reference31.- Exportez la table des entités au format .csv.
5.3D génération de modèles
- Dessinez à la main un modèle 3D de novo à l’échelle selon les étapes mentionnées ci-dessous.
- Prenez une photo de l’organe d’intérêt.
- Effectuez les opérations suivantes dans le logiciel SketchUp (voir Tableau des matériaux) comme mentionné ci-dessous.
- Supprimez l’image par défaut d’un homme qui apparaît à l’ouverture du logiciel.
- Cliquez sur Fichier > Importer pour importer une image des organes d’intérêt.
- Cliquez sur l’outil Lignes et sélectionnez l’option Main levée . Utilisez l’outil crayon pour tracer et dessiner les contours des organes d’intérêt. Assurez-vous de fermer la ligne en remontant jusqu’au point de départ. Une fois la ligne fermée avec succès, la zone dessinée apparaîtra automatiquement ombrée.
- Sélectionnez l’outil Push/Pull et tirez vers le haut sur la zone ombrée pour convertir le dessin de 2D en 3D.
- Supprimer l’image de l’organe : sélectionnez le bouton Gomme , puis cliquez avec le bouton droit sur l’image et sélectionnez Effacer.
- Exporter le fichier au format .dae : Fichier > Exporter > modèle 3D.
- Améliorez le réalisme du modèle selon les étapes mentionnées ci-dessous.
- Importez le modèle dans le logiciel MeshLab (voir Table des matériaux) : ouvrez MeshLab et sélectionnez Fichier > Importer le maillage. Sélectionnez le modèle .dae généré à l’étape précédente. Un menu Options de pré-ouverture apparaîtra. Sélectionnez OK.
- Sélectionnez Wireframe dans le menu supérieur. Ensuite, sélectionnez : Filtres > Remeshing, Simplification et Reconstruction > Surfaces de subdivision : Point médian. Laissez toutes les valeurs par défaut et sélectionnez Appliquer deux fois. Inspectez visuellement la vue filaire du modèle pour vous assurer qu’elle est finement quadrillée. Fermez le menu contextuel.
- Exporter le modèle au format .stl : Fichier > Exporter le maillage sous, puis sélectionnez Format de fichier STL (*.stl) dans le menu déroulant Type de fichier . Cliquez sur Enregistrer. Sélectionnez OK dans le menu contextuel suivant.
- Ouvrez le logiciel Meshmixer (voir Tableau des matériaux). Cliquez sur le bouton Importer (+ ). Sélectionnez le fichier .stl généré à l’étape précédente. Utilisez les outils Sculpter > > Glisser et Sculpter > Pinceaux > Gonfler pour extraire les surfaces du modèle qui doivent être arrondies.
- Une fois que le modèle a l’apparence souhaitée, enregistrez-le au format .stl: Fichier > Exporter. Nommez le fichier comme vous le souhaitez et sélectionnez Format binaire STL (*.stl) dans le menu déroulant Type de fichier. Cliquez sur Enregistrer. Si un menu contextuel apparaît, cliquez sur Continuer.
6. Génération de parcelles
- Obtenez les coordonnées des positions dans le modèle 3D qui correspondent aux sites d’échantillonnage.
- Ouvrez le modèle 3D de l’étape 5.1.3.5 dans le logiciel MeshLab : Fichier > Importer un maillage. Sélectionnez le modèle généré à l’étape 5.1.3.5. Cliquez sur OK dans la fenêtre contextuelle Post-Open Processing .
- Pour obtenir les coordonnées x, y et z pour chaque point d’échantillonnage : sélectionnez l’outil Points de prélèvement , puis cliquez avec le bouton droit à intervalles réguliers sur la surface du modèle 3D. Une fois que toutes les coordonnées souhaitées ont été sélectionnées, cliquez sur le bouton Enregistrer le plus haut dans la fenêtre contextuelle Formulaire .
REMARQUE: Cela exportera les coordonnées au format de fichier .pp. Ce fichier peut être ouvert dans un tableur. - Dans un tableur : Ouvrez le fichier .pp généré à l’étape 6.1.2. Ajustez l’affichage des données à l’aide de Données > texte en colonnes > délimitées. Cliquez sur Suivant, puis sélectionnez Espace, puis cliquez sur Terminer.
- Reformatez pour que seules les valeurs numériques restent dans les cellules de la feuille de calcul en sélectionnant Accueil > Rechercher et sélectionner > Remplacer. Dans la zone Rechercher, entrez : y= ». Laissez la zone Remplacer par vide. Cliquez sur Remplacer tout, puis sur OK. Répétez pour x= » et pour z= » et pour " />. Les valeurs sont maintenant prêtes pour l’étape 6.2.
- Créez la table des fonctionnalités 'ili. Cette méthode a été adaptée de la référence 16.
- Dans un tableur, créez la table des fonctionnalités. Les lignes correspondent à chaque position et les colonnes aux données.
REMARQUE : Les premières colonnes doivent être le nom de la position (nom de l’échantillon), suivi des coordonnées x, y et z des points d’échantillonnage obtenus à l’étape 6.1.2 (avec les en-têtes de colonne x, y, z). La cinquième colonne doit s’intituler « rayon ». - Collez les métadonnées appropriées et l’abondance des caractéristiques métabolites dans les colonnes de feuille de calcul suivantes.
- Dans la colonne « rayon », entrez la taille souhaitée des points d’échantillonnage à visualiser sur le modèle. Déterminez empiriquement les valeurs du rayon : entrez 1 par défaut, puis évaluez si le rayon ou les coordonnées doivent être ajustés à l’étape 6.3. Enregistrez le fichier au format .csv (Fichier > Enregistrer sous), puis sélectionnez CSV (délimité par des virgules) (*.csv) dans le menu déroulant. Nommez le fichier comme vous le souhaitez. Cliquez sur Enregistrer.
- Dans un tableur, créez la table des fonctionnalités. Les lignes correspondent à chaque position et les colonnes aux données.
- Ouvrez les données dans 'ili (logiciel développé par16).
- Ouvrez le site Web 'ili (ili.embl.de). Sélectionnez Surface. Faites glisser et déposez le modèle 3D créé dans la fenêtre du navigateur. Faites glisser et déposez la table des fonctionnalités créée dans la même fenêtre de navigateur.
- Utilisez la légende dans le coin inférieur droit pour projeter la colonne de données souhaitée sur le modèle 3D. S’assurer que les taches et les rayons sélectionnés à l’étape 6.2.1 correspondent aux sites d’échantillonnage. Si nécessaire, ajustez les valeurs dans le tableau des fonctions ou choisissez des coordonnées supplémentaires dans le MeshLab (voir étape 6.1.2).
REMARQUE: La visualisation peut également être améliorée en sélectionnant les points > l’opacité de la bordure et en réglant le curseur sur sa valeur maximale. - Sélectionnez consécutivement chaque colonne de données pour évaluer la distribution de cette caractéristique métabolite sur le modèle 3D.
REMARQUE: Cette approche peut également être utilisée pour visualiser uniquement des caractéristiques de métabolites spécifiques d’intérêt, par exemple, ceux dont p < 0,05 ou un certain changement de pli entre les échantillons infectés et non infectés, tels que déterminés dans des outils d’analyse statistique externes. Les fonctionnalités à visualiser peuvent également être sélectionnées en dehors de 'ili grâce à des approches d’apprentissage automatique telles qu’une forêt aléatoire.
- Effectuez une visualisation linéaire/logarithmique des données selon les étapes mentionnées ci-dessous.
- À l’aide de l’onglet Mappage en haut à droite de la page, sélectionnez Linéaire ou Logarithmique dans le menu déroulant Echelle .
- Définissez la même échelle pour tous les tracés si vous visualisez plusieurs entités.
REMARQUE: Le site Web choisit automatiquement le minimum et le maximum pour chaque colonne de données à afficher. - Pour définir l’échelle manuellement, désélectionnez l’option Auto Min/Max et entrez la mise à l’échelle souhaitée. Toutes les données seront désormais affichées dans la même échelle.
- Modifiez l’échelle de couleur des données (niveaux de gris, bleu-blanc-rouge, etc.).
- Remplacez l’échelle de couleurs par le jeu de couleurs souhaité dans le menu Mappage à l’aide du menu déroulant Carte des couleurs. Assurez-vous que le jeu de couleurs sélectionné est compatible avec les daltoniens.
- Pour modifier la couleur du modèle 3D ou la couleur d’arrière-plan, utilisez les options Couleur et Arrière-plan dans le menu 3D.
- Masquez les axes 3D en désélectionnant la case Afficher l’origine sous le menu 3D.
- Enregistrez l’image en faisant une capture d’écran, à l’aide de l’outil de capture ou de la touche de raccourci Crtl + S pour Windows ou Linux et
 + S sous OS X.
+ S sous OS X.
Résultats
Le nombre de caractéristiques métabolites obtenues dépend du type de tissu analysé et des paramètres de traitement des données. Par exemple, ce protocole a été utilisé pour analyser l’impact spatial de l’infection à T. cruzi sur le métabolome du tractus gastro-intestinal dans un modèle murin d’infection à T. cruzi. Dans des travaux antérieurs, des C3H/HeJ mâles ont été injectés par voie intrapéritonéale avec 1 000 parasites CL + luc T. cruzi32,6. Les animaux ont été euthanasiés 12 ou 89 jours après l’infection, et une analyse cartographique chimique de 13 segments contigus du tractus gastro-intestinal a été effectuée comme décrit dans ce protocole. Cette analyse a conduit à un tableau de 5 502 entités, qui ont ensuite été visualisées en 3D à l’aide des étapes décrites dans ce protocole. Cette approche permet de visualiser les caractéristiques des métabolites chez les animaux individuels qui sont élevés sur le site d’une charge parasitaire élevée (kynurénine, Figure 2B vs charge parasitaire, Figure 2A), de métabolites ayant une distribution différentielle entre les régions tissulaires (glutamine, Figure 2C) et de caractéristiques métabolites qui se trouvent à des niveaux comparables dans le petit et le gros intestin (LPE 16:0 Figure 2D ). La kynurénine a été sélectionnée pour la visualisation en raison de sa relation connue avec l’inflammation et des publications antérieures sur la capacité des métabolites dérivés de la kynurénine à réguler la charge de T. cruzi33. Des modèles aléatoires d’apprentissage automatique basés sur la forêt avaient précédemment révélé une association entre les niveaux de kynurénine et le statut d’infection6. La glutamine a été sélectionnée pour une visualisation basée sur des publications antérieures démontrant une relation entre la disponibilité de la glutamine in vitro et la sensibilité au médicament T. cruzi34. La distribution différentielle a été confirmée par régression logistique, p < 0,05. LPE 16:0 a été sélectionné après inspection visuelle des données pour découvrir les caractéristiques des métabolites trouvés à des niveaux comparables sur les sites tissulaires.
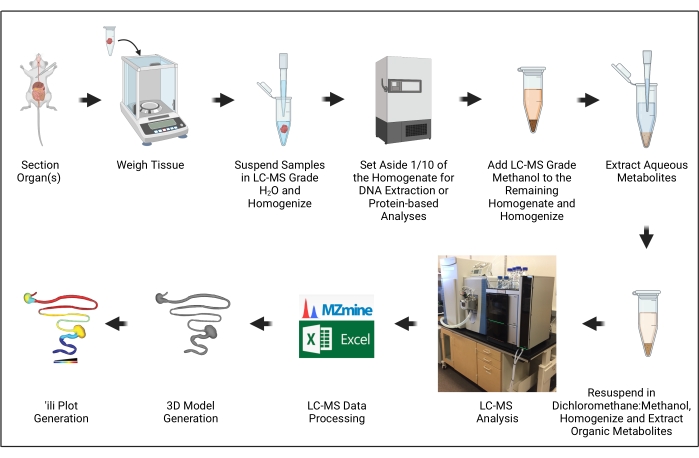
Figure 1 : Vue d’ensemble du protocole. L’illustration a été créée avec BioRender.com. Veuillez cliquer ici pour voir une version agrandie de cette figure.
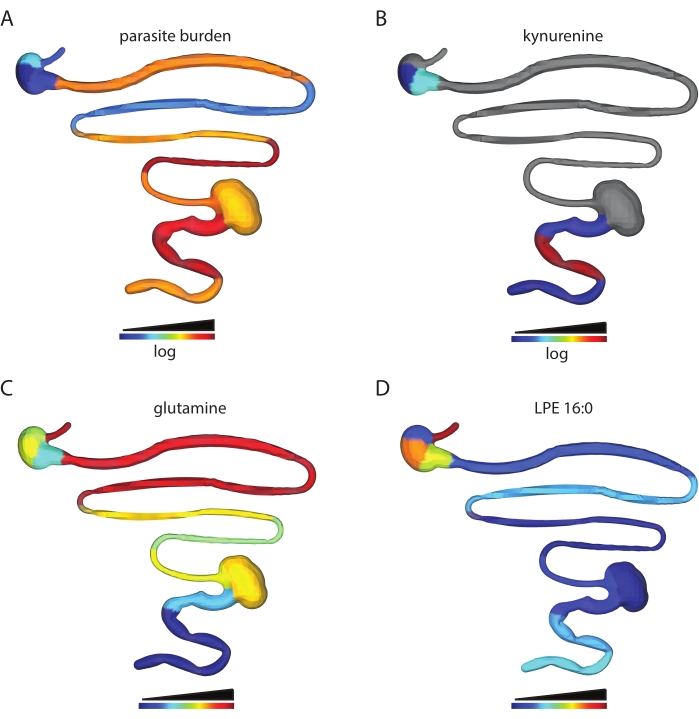
Figure 2 : Analyse cartographique chimique. Des souris mâles C3H/HeJ ont été injectées par voie intrapéritonéale avec 1 000 parasites CL+luc T. cruzi32. Les animaux ont été euthanasiés 12 ou 89 jours après l’infection, et le tractus gastro-intestinal a été prélevé et sectionné systématiquement (étape 1)6. Les métabolites ont été extraits comme à l’étape 2 et analysés par LC-MS/MS.3D génération du modèle a été effectuée à l’aide du logiciel SketchUp (étape 5), et les données ont été tracées en 3D comme à l’étape 6. (A) Distribution du parasite chez une souris spécifique, 12 jours après l’infection. (B) Distribution du métabolite de la kynurénine chez la même souris, 12 jours après l’infection. (C) Distribution moyenne de la glutamine chez les souris infectées, 89 jours après l’infection. (D) Niveaux comparables de temps de rétention m/z 454,292 2,929 min, annotés en 2-hexadécanoyl-sn-glycero-3-phosphoéthanolamine (LPE 16:0), chez la même souris que chez A et B dans l’intestin grêle et le côlon. Des échantillons et des données ont été générés en6. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Comprendre les infections à trypanosomatides est essentiel pour guider le développement de nouveaux médicaments et les approches thérapeutiques. Cette méthode de cartographie chimique est particulièrement bien placée pour fournir des informations exploitables sur la relation entre le métabolisme et la pathogenèse de la maladie trypanosomatide, répondant ainsi à ce besoin translationnel.
Seuls les solvants de qualité LC-MS sont recommandés lors de l’extraction des métabolites et des analyses MS, afin de réduire la contamination de fond. La contamination polymère35, généralement dérivée d’un film de paraffine et/ou d’autres plastiques36,37,38, doit être évitée dans la mesure du possible. Le parafilm, en particulier, ne doit jamais être utilisé. Ces aspects sont cruciaux car la qualité des données LC-MS dépend des matériaux utilisés lors de la préparation des échantillons et de l’extraction des métabolites. La qualité des données doit être assurée avant de générer des diagrammes. De plus, la génération de ces cartes métabolomiques spatiales complètes nécessite la collecte de tous les échantillons de tissus adjacents et l’extraction de métabolites à partir de tous les échantillons prélevés afin d’éviter les lacunes dans ces cartes. Les procédures de collecte, la logistique de l’extraction des métabolites et l’analyse LC-MS, ainsi que les coûts doivent donc être pris en compte et planifiés en conséquence.
Ce protocole peut être modifié pour répondre aux besoins des utilisateurs de plusieurs façons. Par exemple, la polarité et la solubilité des solvants utilisés lors de l’extraction des métabolites influenceront les métabolites détectés39. Pour maximiser la diversité des caractéristiques des métabolites détectés pour les analyses cartographiques chimiques non ciblées, il est recommandé de combiner plusieurs étapes d’extraction et solvants. Par exemple, cette méthode utilise le dichlorométhane, le méthanol et l’eau comme solvants d’extraction, car ils permettent une détection précise des molécules non polaires et polaires20,40. Cependant, ces solvants ne conviennent pas universellement à toutes les expériences sur la SEP, et les chercheurs devraient sélectionner les solvants d’extraction en fonction des objectifs de leur projet. De même, différentes conditions LC-MS/MS peuvent être utilisées, telles que le remplacement de la chromatographie en phase inversée par la chromatographie en phase normale. Des colonnes alternatives peuvent également être utilisées pour la collecte de données en phase inversée au lieu de la chromatographie en C8, bien que empiriquement, la chromatographie en C8 soit plus robuste pour les lipides tissulaires et ait une fréquence de colmatage plus faible. Conceptuellement, ces protocoles peuvent également être appliqués à d’autres méthodes de spectrométrie de masse telles que la chromatographie en phase gazeuse-spectrométrie de masse, etc.
Une autre approche est l’imagerie par spectrométrie de masse. En effet, contrairement aux approches d’imagerie par spectrométrie de masse, la chromatographie liquide-spectrométrie de masse ne préserve pas intrinsèquement l’information spatiale10. Les approches de cartographie chimique comblent cette lacune en incluant l’emplacement de l’échantillonnage au moment de la conceptualisation du projet, dans les métadonnées de l’échantillon et aux étapes de traitement des données. L’une des forces de cette approche de cartographie chimique, contrairement à l’imagerie par spectrométrie de masse, est la capacité de fournir des annotations fiables (confiance d’annotation de niveau 1 ou de niveau 2 de la Metabolomics Standards Initiative41), contrairement à l’imagerie par spectrométrie de masse où la majeure partie des applications reposent sur une masse précise uniquement pour l’annotation. L’imagerie par spectrométrie de masse permettra une cartographie spatiale à grain fin, parfois jusqu’au niveau d’une seule cellule, par exemple,42,43. En revanche, les approches de cartographie chimique permettent une cartographie inter-organes à grande échelle de la distribution des métabolites sans nécessiter de compétences hautement spécialisées en cryosection d’animaux entiers. La cartographie chimique fournit des preuves complémentaires aux nombreuses approches transcriptomiques spatiales en cours de développement, par exemple,44, avec l’avantage de se concentrer sur la « couche omique la plus proche du phénotype»45. D’autres méthodes de quantification de la charge parasitaire comprennent la mesure de la bioluminescence au moment du prélèvement de l’échantillon6. Des segments fins pourraient également être collectés pour permettre la microscopie confocale ou électronique afin d’évaluer la charge parasitaire localisée et les lésions tissulaires. L’homogénat d’eau, qui est utilisé pour la quantification des cytokines dans ce protocole et dans des publications antérieures13, pourrait également être utilisé pour quantifier les marqueurs à base de protéines des lésions tissulaires.
Il existe également plusieurs façons d’obtenir des modèles 3D adaptés pour tracer les données LC-MS résultantes. En plus de la méthode suggérée ici, les modèles peuvent être achetés pré-fabriqués auprès de divers fournisseurs en ligne. S’assurer que les conditions d’utilisation correspondent à l’utilisation prévue, en particulier en ce qui concerne la publication. Les modèles pour les grands organes peuvent être générés de novo à l’aide de scanners 3D selon les instructions du scanner. Des alternatives telles que MATLAB existent pour générer et visualiser des modèles 3D pour la cartographie chimique46, mais elles ont été principalement mises en œuvre avant le développement de 'ili16. MATLAB est une suite d’outils d’analyse et de programmation de données offrant une grande variété d’applications dans de nombreux domaines. Cependant, MATLAB n’est ni gratuit ni open-source, et il nécessite une familiarité avec les interfaces MATLAB, d’autant plus que MATLAB n’a pas été développé pour le traitement des données de spectrométrie de masse. Les alternatives de cette méthode proposée, à savoir SketchUp, Meshlab et 'ili, sont librement accessibles, conviviales et offrent des fonctions similaires à celles de MATLAB à des fins de cartographie chimique.
Cette méthode est robuste en ce qui concerne la préparation des échantillons et l’extraction des métabolites. Le dépannage est le plus souvent nécessaire à l’étape d’acquisition de données LC-MS. Cela dépasse le cadre de cet article. Les lecteurs sont dirigés vers d’excellentes publications sur le dépannage de l’acquisition de données LC-MS, y compris20,47. De même, les complexités de l’annotation des métabolites dépassent le cadre de l’accent mis par cette méthode sur la génération de modèles 3D. Les références utiles sur ce sujet incluent24,25,48,49.
Bien que cette méthode explore efficacement la pathogenèse de la maladie, il existe des limites à cette approche, dont certaines sont communes à toute expérience métabolomique. L’une de ces limitations est le faible taux d’annotation des fonctionnalités LC-MS50, qui dépend de la disponibilité et de la qualité des bibliothèques spectrales de référence. Une autre limitation est que ce protocole ne préserve pas l’ARNm en raison de l’incompatibilité des réactifs de préservation de l’ARN tels que l’ARNilatter avec l’analyse LC-MS/MS. Cependant, la qualité des protéines est adéquate pour les analyses en aval et peut donc remplacer les analyses à base d’ARNm.
Une approche cartographique chimique de la pathogenèse des infections reflète directement comment les infections bactériennes, virales ou parasitaires se développent dans les systèmes organiques et provoquent des maladies localisées. L’analyse de ces sous-échantillons régionaux et la génération de modèles 3D permettent en fin de compte de comprendre comment les métabolites fonctionnent dans l’espace tridimensionnel, mettant en lumière ces dimensions spatiales de la biologie moléculaire jusqu’alors méconnues. En utilisant ce protocole, par exemple, la localisation des métabolites a été comparée à la charge parasitaire de Trypanosoma cruzi. Les résultats ont clarifié la relation entre l’agent pathogène et le tissu hôte et ont également démontré la dynamique métabolique de la progression des symptômes de la maladie de Chagas6. Les méthodes de cartographie chimique ont également été appliquées à divers sujets, tels que l’interaction homme-environnement construit51,52,53, la composition chimique des systèmes organiques comme la peau humaine46 et les poumons54, et le métabolisme des plantes et les interactions environnementales55. Les applications futures peuvent impliquer l’évaluation de la tolérance et de la résilience localisées aux maladies, ou la relation entre les niveaux locaux de métabolites, le tropisme pathogène et le tropisme de la maladie dans des modèles au-delà de l’infection trypanosomatidée. Cette approche devrait également avoir une large applicabilité pour élargir les protocoles pharmacocinétiques actuels, pour évaluer la relation entre les niveaux locaux de médicaments tissulaires et le métabolisme des médicaments par rapport au contexte métabolique global, aux lésions tissulaires et à la clairance des agents pathogènes. Dans l’ensemble, la cartographie chimique permet d’explorer de manière unique les distributions de métabolites dans divers types d’échantillons, avec des applications telles que la pathogenèse des maladies, la santé humaine, les interactions homme-environnement et la dynamique microbienne.
Déclarations de divulgation
Aucun conflit d’intérêts à signaler.
Remerciements
Laura-Isobel McCall, Ph.D., est titulaire d’un prix Investigators in the Pathogenesis of Infectious Disease du Burroughs Wellcome Fund. Les auteurs souhaitent en outre reconnaître le soutien du numéro de prix R21AI148886 des NIH, d’une subvention pilote de l’Oklahoma Center for Respiratory and Infectious Diseases (OCRID) sous le numéro de prix niH P20GM103648 et des fonds de démarrage de l’Université de l’Oklahoma (à LIM). Le contenu relève de la seule responsabilité des auteurs et ne représente pas nécessairement les points de vue officiels des bailleurs de fonds. Les auteurs tiennent également à remercier les développeurs des outils utilisés dans ce protocole. Toutes les publications pertinentes ont été citées, le cas échéant.
matériels
| Name | Company | Catalog Number | Comments |
| 1.5 mL Eppendorf tubes | NA | NA | any brand, as available |
| 1 L bottle, pyrex | NA | NA | any brand, as available |
| 1 L graduated cylinder, pyrex | NA | NA | any brand, as available |
| 2 mL SafeLock Eppendorf tubes | VWR | 20901-540 | use the appropriate tube model for the available tissue homogenizer |
| 3D model (de-novo generated according to protocol steps, or purchased) | NA | NA | as appropriate for system under investigation |
| 5 mm stainless steel bead | Qiagen | 69989 | |
| 96 well plate | Fisher | 3252449 | |
| AATTCCTCCAAGCAGCGGATA primer | NA | NA | any brand, as available; published in Piron, M. et al. Development of a real-time PCR assay for Trypanosoma cruzi detection in blood samples. Acta tropica. 103 (3), 195–200 (2007). |
| analytical balance | NA | NA | any brand, as available |
| ASTCGGCTGATCGTTTTCGA primer | NA | NA | any brand, as available; published in Piron, M. et al. Development of a real-time PCR assay for Trypanosoma cruzi detection in blood samples. Acta tropica. 103 (3), 195–200 (2007). |
| benchtop centrifuge with microcentrifuge, falcon tube and 96-well-plate capacity | NA | NA | any brand, as available |
| biosafety cabinet | NA | NA | class II, type A2; any brand, as available |
| CAGCAAGCATCTATGCACTTAG ACCCC primer | NA | NA | any brand, as available; published in Cummings, K.L., Tarleton, R.L. Rapid quantitation of Trypanosoma cruzi in host tissue by real-time PCR. Molecular and biochemical parasitology. 129 (1), 53–59 (2003). |
| camera | NA | NA | any brand, as available. A cellphone camera is adequate for this protocol |
| chemiluminescent-capable imaging system | NA | NA | any system, as available |
| cotton balls | NA | NA | any brand, as available |
| cryogloves | VWR | 97008-208 | replace with any brand, as available |
| dissection scissors | NA | NA | any brand, as available |
| dry ice | NA | NA | any brand, as available |
| extra-length forceps | NA | NA | any brand, as available |
| flammable-grade refrigerator | NA | NA | any brand, as available |
| freezer storage boxes for microcentrifuge tubes | NA | NA | any brand, as available |
| fume hood | NA | NA | any brand, as available |
| high-resolution mass spectrometer | NA | NA | any brand, as available, such as ThermoFisher Q-Exactive Plus (catalog number 0726030) |
| ice bucket | NA | NA | any brand, as available |
| ili software | ili.embl.de | NA | |
| isoflurane | Covetrus | 29405 | |
| large tupperware | NA | NA | any brand, as available; large enough to comfortably contain mouse, cotton ball |
| LC-MS grade acetonitrile | Fisher Optima | A955-4 | |
| LC-MS grade dicholoromethane | Fisher Optima | D151-4 | |
| LC-MS grade formic acid | Fisher Optima | A11750 | |
| LC-MS grade methanol | Fisher Optima | A456-4 | |
| LC-MS grade water | Fisher Optima | W64 | |
| liquid chromatography column | Phenomenex | 00B-4499-AN | may be changed to other brands and models as appropriate for the metabolites of interest |
| liquid chromatography column guard cartridge | Phenomenex | AJ0-8784 | may be changed to other brands and models as appropriate for the metabolites of interest |
| liquid chromatography column guard cartridge holder | Phenomenex | AJ0-9000 | may be changed to other brands and models as appropriate for the metabolites of interest |
| liquid nitrogen | NA | NA | any brand, as available |
| luciferin | Goldbio | LUCK-1G | |
| MeshLab software | https://www.meshlab.net/ | NA | |
| Meshmixer software | https://www.meshmixer.com/ | NA | |
| MS calibrant | NA | NA | appropriate one for available instrument |
| MS data processing software | NA | NA | multiple options available; authors recommend MZmine |
| MSConvert software | http://proteowizard.sourceforge.net/ | NA | |
| Nanodrop | ThermoFisher | ND-ONE-W | other nanodrop models are also suitable |
| p1000 pipet tips | NA | NA | use the appropriate brand to fit available pipettors |
| p1000 pipettor | NA | NA | any brand, as available |
| p20 pipette tips | NA | NA | use the appropriate brand to fit available pipettors |
| p20 pipettor | NA | NA | any brand, as available |
| p200 pipette tips | NA | NA | use the appropriate brand to fit available pipettors |
| p200 pipettor | NA | NA | any brand, as available |
| personal protective equipment (gloves, lab coat, safety glasses/goggles; faceshield) | NA | NA | any brand, as available |
| Q-Plex Mouse Cytokine - Screen (16-Plex) | Quansys biosciences | 110949MS | can replace with other protein-based cytokine assays such as other commercial cytokine ELISA kits |
| Quick-DNA Miniprep Plus Kit (200 preps) | Zymo | D4069 | replace with any brand of mammalian DNA extraction kit, as available |
| real-time thermocycler | NA | NA | any brand, as available |
| salt shaker or tea infuser | NA | NA | any brand; to contain isoflurane-soaked cotton ball and prevent contact with mouse skin |
| SketchUp software | https://www.sketchup.com/ | NA | |
| specimen forceps | NA | NA | any brand, as available |
| speedvac with microcentrifuge tube and 96-well-plate capacity | NA | NA | any brand, as available |
| spreadsheet software | https://www.microsoft.com/en-us/microsoft-365/excel | NA | can replace with other spreadsheet management software, as applicable |
| sulfachloropyridazine | Sigma | S9882-100G | |
| sulfadimethoxine | Sigma | S7007-10G | |
| Sybr green qPCR reaction mix | Fisher | A25780 | can replace with other Sybr green qPCR reaction mixes, as desired |
| TCCCTCTCATCAGTTCTAT GGCCCA primer | NA | NA | any brand, as available; published in Cummings, K.L., Tarleton, R.L. Rapid quantitation of Trypanosoma cruzi in host tissue by real-time PCR. Molecular and biochemical parasitology. 129 (1), 53–59 (2003). |
| tissue homogenizer | NA | NA | any brand, as available; for example, Qiagen TissueLyser II, catalog number 85300, with TissueLyser Adapter Set (2 x 24), catalog number 69982 |
| tissue samples | NA | NA | from appropriate infection model |
| TissueLyser single-bead dispenser | Qiagen | 69965 | |
| UHPLC | NA | NA | any brand, as available, such as ThermoFisher Vanquish (catalog number IQLAAAGABHFAPUMBHV) |
| ultra-low temperature freezer (-80) | NA | NA | any brand, as available |
| ultrasonic bath | NA | NA | any system, as available |
| wet ice | NA | NA | any brand, as available |
| zone-free sealing film | VWR | 490007-390 |
Références
- McCall, L. -. I., et al. Adaptation of Leishmania donovani to and visceral environments: in vivo selection and proteomic analysis. Journal of Proteome Research. 14 (2), 1033-1059 (2015).
- McCall, L. -. I., Zhang, W. -. W., Matlashewski, G. Determinants for the development of visceral leishmaniasis disease. PLoS Pathogens. 9 (1), 1003053 (2013).
- de Castro Neto, A. L., da Silveira, J. F., Mortara, R. A. Comparative analysis of virulence mechanisms of trypanosomatids pathogenic to humans. Frontiers in Cellular and Infection Microbiology. 11, 669079 (2021).
- Franco, J. R., Simarro, P. P., Diarra, A., Jannin, J. G. Epidemiology of human African trypanosomiasis. Clinical Epidemiology. 6, 257-275 (2014).
- Ottilie, S., et al. Rapid chagas disease drug target discovery using directed evolution in drug-sensitive yeast. ACS Chemical Biology. 12 (2), 422-434 (2017).
- Hossain, E., et al. Mapping of host-parasite-microbiome interactions reveals metabolic determinants of tropism and tolerance in Chagas disease. Science Advances. 6 (30), (2020).
- Parab, A. R., McCall, L. -. I. Tryp-ing up metabolism: Role of metabolic adaptations in kinetoplastid disease pathogenesis. Infection and Immunity. 89 (4), (2021).
- Lewis, C. M., McCall, L. -. I., Sharp, R. R., Spicer, P. G. Ethical priority of the most actionable system of biomolecules: the metabolome. American Journal of Physical Anthropology. 171 (2), 177-181 (2020).
- Liu, Z., Ulrich vonBargen, R., McCall, L. I. Central role of metabolism in Trypanosoma cruzi tropism and Chagas disease pathogenesis. Current Opinion in Microbiology. 63, 204-209 (2021).
- Newsom, S. N., McCall, L. -. I. Metabolomics: Eavesdropping on silent conversations between hosts and their unwelcome guests. PLoS Pathogens. 14 (4), 1006926 (2018).
- Wishart, D. S., et al. HMDB: the Human Metabolome Database. Nucleic Acids Research. 35, 521-526 (2007).
- Dean, D. A., et al. Spatial metabolomics identifies localized chemical changes in heart tissue during chronic cardiac Chagas disease. PLoS Neglected Tropical Diseases. 15 (10), 0009819 (2021).
- McCall, L. -. I., et al. Mass spectrometry-based chemical cartography of a cardiac parasitic infection. Analytical Chemistry. 89 (19), 10414-10421 (2017).
- Hoffman, K., et al. Alterations to the cardiac metabolome induced by chronic infection relate to the degree of cardiac pathology. ACS Infectious Diseases. 7 (6), 1638-1649 (2021).
- Parab, A. R., et al. Dysregulation of glycerophosphocholines in the cutaneous lesion caused by Leishmania major in experimental murine models. Pathogens. 10 (5), 593 (2021).
- Protsyuk, I., et al. 3D molecular cartography using LC-MS facilitated by Optimus and 'ili software. Nature Protocols. 13 (1), 134-154 (2018).
- McCall, L. -. I., et al. Targeting ergosterol biosynthesis in Leishmania donovani: Essentiality of Sterol 14alpha-demethylase. PLOS Neglected Tropical Diseases. 9 (3), 0003588 (2015).
- Tear, W. F., et al. Selectivity and physicochemical optimization of repurposed Pyrazolo[1,5-]pyridazines for the treatment of human African trypanosomiasis. Journal of Medicinal Chemistry. 63 (2), 756-783 (2020).
- Klug, D. M., et al. Hit-to-lead optimization of benzoxazepinoindazoles as human African trypanosomiasis therapeutics. Journal of Medicinal Chemistry. 63 (5), 2527-2546 (2020).
- Want, E. J., et al. Global metabolic profiling of animal and human tissues via UPLC-MS. Nature Protocols. 8 (1), 17-32 (2013).
- Piron, M., et al. Development of a real-time PCR assay for Trypanosoma cruzi detection in blood samples. Acta Tropica. 103 (3), 195-200 (2007).
- Cummings, K. L., Tarleton, R. L. Rapid quantitation of Trypanosoma cruzi in host tissue by real-time PCR. Molecular and Biochemical Parasitology. 129 (1), 53-59 (2003).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2−ΔΔCT method. Methods. 25 (4), 402-408 (2001).
- Nothias, L. -. F., et al. Feature-based molecular networking in the GNPS analysis environment. Nature Methods. 17 (9), 905-908 (2020).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with global natural products social molecular networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Chambers, M. C., et al. A cross-platform toolkit for mass spectrometry and proteomics. Nature Biotechnology. 30 (10), 918-920 (2012).
- Tsugawa, H., et al. MS-DIAL: data-independent MS/MS deconvolution for comprehensive metabolome analysis. Nature Methods. 12 (6), 523-526 (2015).
- Sturm, M., et al. OpenMS - an open-source software framework for mass spectrometry. BMC Bioinformatics. 9, 163 (2008).
- Pluskal, T., Castillo, S., Villar-Briones, A., Oresic, M. MZmine 2: modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11, 395 (2010).
- Smith, C. A., Want, E. J., O'Maille, G., Abagyan, R., Siuzdak, G. XCMS: processing mass spectrometry data for metabolite profiling using nonlinear peak alignment, matching, and identification. Analytical Chemistry. 78 (3), 779-787 (2006).
- . FBMN with MZmine Available from: https://ccms-ucsd.github.io/GNPSDocumentation/featurebasedmolecularnetworking-with-mzmine2/ (2021)
- Lewis, M. D., et al. Bioluminescence imaging of chronic Trypanosoma cruzi infections reveals tissue-specific parasite dynamics and heart disease in the absence of locally persistent infection. Cellular Microbiology. 16 (9), 1285-1300 (2014).
- Knubel, C. P., et al. 3-Hydroxy kynurenine treatment controls T. cruzi replication and the inflammatory pathology preventing the clinical symptoms of chronic Chagas disease. PloS One. 6 (10), 26550 (2011).
- Dumoulin, P. C., Vollrath, J., Tomko, S. S., Wang, J. X., Burleigh, B. Glutamine metabolism modulates azole susceptibility in amastigotes. eLife. 9, (2020).
- Keller, B. O., Sui, J., Young, A. B., Whittal, R. M. Interferences and contaminants encountered in modern mass spectrometry. Analytica Chimica Acta. 627 (1), 71-81 (2008).
- Weaver, R., Riley, R. J. Identification and reduction of ion suppression effects on pharmacokinetic parameters by polyethylene glycol 400. Rapid Communications in Mass Spectrometry: RCM. 20 (17), 2559-2564 (2006).
- Larger, P. J., Breda, M., Fraier, D., Hughes, H., James, C. A. Ion-suppression effects in liquid chromatography-tandem mass spectrometry due to a formulation agent, a case study in drug discovery bioanalysis. Journal of Pharmaceutical and Biomedical Analysis. 39 (1-2), 206-216 (2005).
- Furey, A., Moriarty, M., Bane, V., Kinsella, B., Lehane, M. Ion suppression; a critical review on causes, evaluation, prevention and applications. Talanta. 115, 104-122 (2013).
- Lu, W., et al. Metabolite measurement: Pitfalls to avoid and practices to follow. Annual Review of Biochemistry. 86, 277-304 (2017).
- Masson, P., Alves, A. C., Ebbels, T. M. D., Nicholson, J. K., Want, E. J. Optimization and evaluation of metabolite extraction protocols for untargeted metabolic profiling of liver samples by UPLC-MS. Analytical Chemistry. 82 (18), 7779-7786 (2010).
- Sumner, L. W., et al. Proposed minimum reporting standards for chemical analysis. Metabolomics. 3 (3), 211-221 (2007).
- Rappez, L., et al. SpaceM reveals metabolic states of single cells. Nature Methods. 18 (7), 799-805 (2021).
- Yang, B., Tsui, T., Caprioli, R. M., Norris, J. L. Sample preparation and analysis of single cells using high performance MALDI FTICR mass spectrometry. Methods in Molecular Biology. 2064, 125-134 (2020).
- Longo, S. K., Guo, M. G., Ji, A. L., Khavari, P. A. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nature Reviews. Genetics. 22 (10), 627-644 (2021).
- Patti, G. J., Yanes, O., Siuzdak, G. Innovation: Metabolomics: the apogee of the omics trilogy. Nature Reviews. Molecular Cell Biology. 13 (4), 263-269 (2012).
- Bouslimani, A., et al. Molecular cartography of the human skin surface in 3D. Proceedings of the National Academy of Sciences of the United States of America. 112 (17), 2120-2129 (2015).
- Dudzik, D., Barbas-Bernardos, C., García, A., Barbas, C. Quality assurance procedures for mass spectrometry untargeted metabolomics. A review. Journal of Pharmaceutical and Biomedical Analysis. 147, 149-173 (2018).
- Alseekh, S., et al. Mass spectrometry-based metabolomics: a guide for annotation, quantification and best reporting practices. Nature Methods. 18 (7), 747-756 (2021).
- Chaleckis, R., Meister, I., Zhang, P., Wheelock, C. E. Challenges, progress and promises of metabolite annotation for LC-MS-based metabolomics. Current Opinion in Biotechnology. 55, 44-50 (2019).
- Viant, M. R., Kurland, I. J., Jones, M. R., Dunn, W. B. How close are we to complete annotation of metabolomes. Current Opinion in Chemical Biology. 36, 64-69 (2017).
- Kapono, C. A., et al. Creating a 3D microbial and chemical snapshot of a human habitat. Scientific Reports. 8 (1), 3669 (2018).
- Petras, D., et al. Mass spectrometry-based visualization of molecules associated with human habitats. Analytical Chemistry. 88 (22), 10775-10784 (2016).
- McCall, L. -. I., et al. Analysis of university workplace building surfaces reveals usage-specific chemical signatures. Building and Environment. 162, 106289 (2019).
- Garg, N., et al. Three-dimensional microbiome and metabolome cartography of a diseased human lung. Cell Host & Microbe. 22 (5), 705-716 (2017).
- Floros, D. J., et al. Mass spectrometry based molecular 3D-cartography of plant metabolites. Frontiers in Plant Science. 8, 429 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon