Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Rasterkraftmikroskopie zur Untersuchung der physikalischen Eigenschaften epidermaler Zellen lebender Arabidopsiswurzeln
In diesem Artikel
Zusammenfassung
Das Rasterkraftmikroskopie-Eindringprotokoll bietet die Möglichkeit, die Rolle der physikalischen Eigenschaften der Zellwand einer bestimmten Zelle eines Gewebes oder Organs während des normalen oder eingeschränkten Wachstums (d.h. unter Wasserdefizit) zu analysieren.
Zusammenfassung
Hier wird eine Methode beschrieben, um die physikalischen Eigenschaften der Zellwand epidermaler Zellen lebender Arabidopsiswurzeln durch Nanoindentationen mit einem Rasterkraftmikroskop (AFM) gekoppelt mit einem optischen inversen Fluoreszenzmikroskop zu charakterisieren. Die Methode besteht darin, kontrollierte Kräfte auf die Probe anzuwenden, während ihre Verformung gemessen wird, wodurch Parameter wie der scheinbare Elastizitätsmodul der Zellwände bei subzellulärer Auflösung quantifiziert werden können. Es erfordert eine sorgfältige mechanische Immobilisierung der Probe und die richtige Auswahl der Eindringkörper und Eindringtiefen. Obwohl es nur in äußeren Geweben verwendet werden kann, ermöglicht diese Methode die Charakterisierung mechanischer Veränderungen in pflanzlichen Zellwänden während der Entwicklung und ermöglicht die Korrelation dieser mikroskopischen Veränderungen mit dem Wachstum eines ganzen Organs.
Einleitung
Pflanzenzellen sind von einer Zellwand umgeben, die eine komplexe Struktur ist, die aus interagierenden Netzwerken von Polysacchariden, Proteinen, Metaboliten und Wasser besteht, deren Dicke je nach Zelltyp und Wachstumsphase von 0,1 bis zu mehreren μm variiert 1,2. Die mechanischen Eigenschaften der Zellwand spielen eine wesentliche Rolle beim Wachstum von Pflanzen. Niedrige Steifigkeitswerte der Zellwand wurden als Voraussetzung für Zellwachstum und Zellwanderweiterung vorgeschlagen, und es gibt zunehmend Hinweise darauf, dass alle Zellen mechanische Kräfte wahrnehmen, um ihre Funktionen zu erfüllen. Es wird jedoch noch diskutiert, ob Veränderungen in den physikalischen Eigenschaften der Zellwand das Zellschicksalbestimmen 2,3,4. Da sich Pflanzenzellen während der Entwicklung nicht bewegen, hängt die endgültige Form eines Organs davon ab, wie weit und in welche Richtung sich eine Zelle ausdehnt. Daher ist die Arabidopsis-Wurzel ein gutes Modell, um den Einfluss der physikalischen Eigenschaften der Zellwand auf die Zellexpansion zu untersuchen, da verschiedene Arten der Expansion in verschiedenen Regionen der Wurzel auftreten. So zeigt sich die anisotrope Expansion in der Dehnungszone und besonders deutlich in den Epidermiszellen5.
Die hier beschriebene Methode wurde verwendet, um die physikalischen Eigenschaften der Zellwand epidermaler Zellen auf der Nanoskala lebender Arabidopsis-Wurzeln mit einem Rasterkraftmikroskop (AFM) gekoppelt mit einem inversen Fluoreszenzphasenmikroskop6 zu charakterisieren. Für eine umfassende Überarbeitung der AFM-Technik lesen Sie 7,8,9.
Dieses Protokoll beschreibt eine grundlegende Probenvorbereitungsmethode und eine allgemeine Methode zur AFM-basierten Elastizitätsmessung pflanzlicher Zellwände.

Abbildung 1: Schematische Übersicht des Krafteindringexperiments in Arabidopsis-Wurzeln mittels Rasterkraftmikroskopie (AFM). Das Schema gibt einen Überblick über die Schritte eines Kraft-Eindring-Experiments von der Vorbereitung des Substrats zur festen Immobilisierung der Wurzelprobe (1-2), der Bestätigung der Wurzellebensfähigkeit durch Propidiumiodidfärbung (3), der Cantilever-Positionierung auf der Oberfläche einer länglichen Epidermiszelle der Primärwurzel (4-5), der Messung von Kraftkurven (6) und der Kraftkurvenverarbeitung zur Berechnung des scheinbaren Elastizitätsmoduls (7-8). EZ: Dehnungszone. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Protokoll
1. Vorbereitung des Pflanzenmaterials und Wachstumsbedingungen
- Um das benötigte Pflanzenmaterial zu erzeugen, sterilisieren Sie die Samen des Arabidopsis-Wildtyps und der mutierten Linien von Interesse.
HINWEIS: In diesem Protokoll haben wir Folgendes verwendet: ttl1: T-DNA-Insertionslinien Salk_063943 (für TTL1; AT1G53300) - Columbia-0 (Col-0) Wildtyp; die Procuste1 (prc1-1)-Mutante, die aus einer Knock-out-Mutation (Q720stop) im CESA6-Gen (AT5G64740) besteht, wie zuvor in10,11 beschrieben; und TTL1 x PRC1-1 Doppelmutante.- Geben Sie ein ungefähres Volumen von 50 μL Samen in ein Mikroröhrchen. Fügen Sie 500 μL 70% Ethanol + 0,01% Tween-Lösung bei Raumtemperatur hinzu. Mischen und 7 min inkubieren.
- Gießen Sie die Ethanollösung und fügen Sie 500 μL 20% Natriumhypochlorit hinzu. Mischen und 7 min inkubieren.
- Gießen Sie die Natriumhypochloritlösung und waschen Sie die Samen drei- bis viermal mit sterilem Wasser.
- Beschichtung
- Bereiten Sie im Voraus basal Murashige und Skoog (MS) medium12 + 1,5% Saccharose + 1% Agar (pH 5,7) vor (siehe Materialtabelle). Autoklavieren Sie das Medium. Bereiten Sie eine Laminar-Flow-Haube für die Arbeit in einer sterilen Umgebung vor und beschriften Sie die quadratischen Petrischale, die für das Experiment benötigt werden. Gießen Sie das Medium in die Petrischalen und lassen Sie das Medium erstarren.
- Platen Sie die sterilisierten Samen mit sterilen Pipettenspitzen auf quadratische Petrischale, die das Medium enthalten. Verteilen Sie die Samen gleichmäßig in zwei Reihen. Wenn die Samen trocken sind, schließen Sie die Deckel und verschließen Sie die Platten. Die Samen 4 Tage lang bei 4 °C im Dunkeln schichten.
HINWEIS: Eine Schichtung ist erforderlich, um die Keimung zu synchronisieren. - Platzieren Sie die Platten senkrecht in der Wachstumskammer in einem langen Tagesregime (16 h Licht/8 h dunkel) mit 25 μmol m2 s−1 Lichtstärke und 23 °C (Tag/Nacht). Wachsen Sie die Sämlinge für 7 Tage.
2. Osmotische Stressbehandlung (optional)
HINWEIS: Dieser Abschnitt enthält Details zum Wachstum von Arabidopsis-Wurzeln im osmotischen Potential von -1,2 MPa, geschätzt durch kryoskopisches Osmometer (Table of Materials). Dieser Teil kann je nach experimenteller Fragestellung weggelassen oder geändert werden.
- Basales MS-Medium + 1,5% Saccharose + 400 mM Mannit + 1% Agar (pH 5,7) zubereiten. Autoklavieren Sie das Medium. Gießen Sie das Medium in quadratische Petrischalen in die Laminar-Flow-Haube und lassen Sie das Medium erstarren.
- Mit einer Pinzette (Table of Materials) 5 Tage alte Sämlinge, die in basaler MS + 1,5% Saccharosemedium vorgezüchtet wurden, in quadratische Petrischalen legen, die das mit 400 mM Mannitol ergänzte Medium enthalten. Die Platten werden unter den gleichen Bedingungen wie in Schritt 1.2.3 senkrecht in die Wachstumskammer gestellt. Züchten Sie die Sämlinge unter dieser osmotischen Stressbedingung für 7 Tage.
HINWEIS: Um die Anpassung des Wurzelwachstums der Primärwurzel bei starkem osmotischen Stress zu bewerten, wird empfohlen, Sämlinge 5 Tage nach der Keimung zu verwenden, die unter kontrollierten Bedingungen gezüchtet werden, um sicherzustellen, dass die Wurzelmeristemgröße bereits festgelegt ist13. Alternativ kann die Analyse in jedem Entwicklungsstadium durchgeführt werden, um den Entwicklungseffekt der osmotischen Stresswirkung zu bestimmen.
3. Rasterkraftmikroskopie (AFM) Nanoindentationsexperimente
- Vorbereitung der Probe für Nanoindentationsexperimente
HINWEIS: Dies ist ein entscheidender Schritt für die Anwendung von AFM auf die Untersuchung lebender biologischer Proben. Die Probe muss fest mit dem Substrat verbunden sein, um während der Eindringmessungen mechanisch stabil zu sein. Gleichzeitig sollten die strukturellen Qualitäten der Probe erhalten bleiben. Eine effektive Strategie zur Stabilisierung einer großen Probe für AFM ist die Verwendung von Klebstoffen. Der Klebstoff muss schnell trocknen und darf nicht toxisch oder reaktiv mit dem umgebenden Medium sein. In diesem Protokoll wurden Styropor-Petrischalen als Substrat und nicht saurer Silikonkleber (Table of Materials) verwendet, um die Arabidopsis-Keimlinge zu binden.- Verteilen Sie eine dünne Schicht Silikonkleber mit einem Deckglas in die Petrischale. Lassen Sie den Kleber 45 s an der Luft.
- Legen Sie den Sämling mit einer Pinzette auf den Kleber und richten Sie ihn in eine Richtung aus, die den Kontakt zwischen den hervorstehenden Teilen des Sämlings und dem Ausleger vermeidet. Drücken Sie dann die Wurzel vorsichtig auf die Silikonkleberschicht, um sie fest zu binden. Lassen Sie den Sämling 45 s lang mit dem Klebstoff in Kontakt, bevor Sie mit den nächsten Schritten des Protokolls fortfahren.
- Inkubieren Sie die angeschlossenen Sämlinge mit 2 μg/ml Propidiumiodid (PI; Materialtabelle) 5 min im Dunkeln und dreimal vorsichtig mit 1x phosphatgepufferter Kochsalzlösung (PBS) waschen (Table of Materials).
- Legen Sie die PI-inkubierten Sämlinge in ein inverses Fluoreszenzmikroskop, das mit dem AFM gekoppelt ist. Um die Fluoreszenz zu beobachten, stellen Sie die Anregungs- und Emissionswellenlängen auf 572 nm bzw. 617 nm ein. Das Fehlen von Fluoreszenz in den epidermalen Zellen bestätigt die Lebensfähigkeit der Wurzeln.
- Bedecken Sie den Keimling mit 1x phosphatgepufferter Kochsalzlösung (PBS).
- Einrückungsprotokoll
HINWEIS: Führen Sie alle AFM-Messungen innerhalb von 1 h nach der Immobilisierung der Probe auf ihrem Träger bei Raumtemperatur durch. Der Raum, in dem sich das AFM befindet, sollte mit einer Klimaanlage auf 25 °C gehalten werden. Die PBS-Lösung sollte die gleiche Temperatur haben.- Montieren Sie einen Standard-Siliziumnitrid-Cantilever mit pyramidenförmiger Spitze (siehe Materialtabelle) in den AFM-Sondenhalter für Flüssigkeit (Table of Materials).
- Richten Sie den Laser auf dem Ausleger nahe an der Position der Spitze aus. Als nächstes bewegen Sie die Fotodiode, um den Laserspot in der Mitte des Detektors zu platzieren.
- Um die Durchbiegungsempfindlichkeit zu kalibrieren, positionieren Sie eine Styropor-Petrischale mit 1x PBS unter dem AFM.
HINWEIS: Dieser Schritt ist erforderlich, um den Einzug zu berechnen. Wenn eine Kraftmessung auf einer harten Oberfläche durchgeführt wird, gibt es keine Vertiefung in die Oberfläche, so dass jede Änderung der z-Scannerverschiebung der Cantilever-Durchbiegung entspricht. Daher ist es sehr wichtig, die Durchbiegungsempfindlichkeit auf einer harten Oberfläche zu kalibrieren. Die Verwendung einer weichen Oberfläche überschätzt die Durchbiegungsempfindlichkeit, was zu zu niedrigen Federkonstanten führt. - Stellen Sie den Photodetektor so ein, dass die berührungslose Auslenkung nahe 0 V liegt. Kalibrieren Sie die Durchbiegungsempfindlichkeit durch Ausführen einer Vertiefung (Kraftkurve) mit einer Rampengröße von 3 μm, einer Eindring- und Retraktionsrate von 0,6 μm/s und einer Triggerschwelle von 0,5 V.
- Um die Federkonstante des Auslegers zu kalibrieren, verwenden Sie das Dienstprogramm Thermal Tune der AFM-Software (Table of Materials), das für Sonden mit Federkonstanten kleiner als ca. 5 N/m empfohlen wird, oder verwenden Sie die in Referenz14 beschriebene Methode. Bevor Sie Thermal Tune in der Software aktivieren, stellen Sie sicher, dass die Sonde nicht mit der Probe interagiert.
- Geben Sie die Cantilever-Temperatur ein. Klicken Sie auf Calibrate > Thermal Tune oder auf das Thermal Tune-Symbol in der NanoScope-Symbolleiste . Wählen Sie einen Frequenzbereich (1-100 Hz).
- Klicken Sie im Bereich Thermal Tune auf Daten erfassen. Warten Sie, bis das AFM Daten erfasst hat, und klicken Sie dann auf die Schaltfläche Simple Harmonic Oscillator (Fluid).
- Stellen Sie die mittlere Filterbreite auf 3 ein. Passen Sie die PSD-Behälterbreite an, um das Rauschen in den erfassten Daten durch Mittelwertbildung zu reduzieren. Setzen Sie Anpassungsgrenzen um den ersten Resonanzgipfel.
- Klicken Sie auf Federkonstante berechnen k und dann im Popup-Fenster auf Ja und fragen Sie, ob der Benutzer diesen Wert verwenden möchte.
- Wiederholen Sie die oben beschriebenen Schritte dreimal und nehmen Sie manuell einen Durchschnitt der Federkonstantenwerte. Geben Sie diesen Durchschnittswert in das Feld Federkonstante in der Parameterliste Rampe in der PicoForce-Ansicht ein. Die Kalibrierung endet an dieser Stelle.
- Positionieren Sie die AFM-Sonde mit dem inversen Lichtmikroskop bei 10-facher, 20-facher und 40-facher Okularvergrößerung auf der Oberfläche der vierten länglichen epidermalen Zelle der Primärwurzel und stellen Sie sicher, dass sie in der Mitte der Zelle positioniert wird.
- Mit dem berechneten Federkonstantenwert (Schritt 3.2.5.5) erhalten Sie Kraftkurven mit einer Rampengröße von 3 μm, einer Triggerschwelle von 11 nN und einer Eindring- und Rückzugsrate von 0,6 μm/s an ausgewählten Punkten.
HINWEIS: Frühere Arbeiten15,16 fanden heraus, dass eine niedrige Frequenz von Eindringung und Rückzug dazu beiträgt, Hysterese und Widerstandskraft zu minimieren. - Erhalten Sie Kraftkurven von drei Zellen pro Wurzel für jede Behandlung (verwenden Sie drei biologische Replikate pro Behandlung). Erfassen Sie mindestens 150 Kraftkurven für jede Wurzel.
4. Messen Sie den scheinbaren Elastizitätsmodul
- Passen Sie jede Kraftkurve an das folgende Modell für pyramidale Eindringkörper17 an:
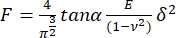 , wobei E der scheinbare Elastizitätsmodul der Probe ist,
, wobei E der scheinbare Elastizitätsmodul der Probe ist, 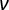 der Poisson-Modul der Probe und α der Halbwinkel zum Eckpunkt ist. Verwerfen Sie die Passungen mit einem Korrelationskoeffizienten (r2) < 0,99. Betrachten Sie das Poisson-Verhältnis für völlig inkompressibles Material:
der Poisson-Modul der Probe und α der Halbwinkel zum Eckpunkt ist. Verwerfen Sie die Passungen mit einem Korrelationskoeffizienten (r2) < 0,99. Betrachten Sie das Poisson-Verhältnis für völlig inkompressibles Material: 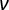 = 0,5 und die von den Herstellern angegebene Geometrie des Eindringkörpers.
= 0,5 und die von den Herstellern angegebene Geometrie des Eindringkörpers.
HINWEIS: Verwenden Sie die ersten 100 nm vom Kontaktpunkt der Belastungskurve, damit die Armatur so empfindlich wie möglich auf die Zellwandmechanik reagiert und der Einfluss des Turgordrucks auf die scheinbaren Elastizitätsmodulwerte8 reduziert wird. Die Anpassung erfolgte mit einem benutzerdefinierten MATLAB-Programm (erhältlich auf persönliche Anfrage durch Schreiben an J. C. B), das die Einstellung einer bestimmten Eindrucktiefe vom Kontaktpunkt der Belastungskurve aus ermöglicht. - Erstellen Sie ein normalisiertes Histogramm mit den scheinbaren Elastizitätsmoduldaten und passen Sie es an eine Gaußverteilung an. Verwerfen Sie Datenpunkte außerhalb des 95%-Konfidenzintervalls, und berechnen Sie sowohl das Histogramm als auch die Gaußsche Anpassung neu. Berechnen und melden Sie den Mittelwert und die Standardabweichung des scheinbaren Elastizitätsmoduls.
- Bestimmen Sie die Signifikanz der Vergleiche zwischen Gruppen durch mehrere Vergleichstests wie ANOVA.
Ergebnisse
Kraft-Eindring-Experimente
Der folgende Text stellt einige Ergebnisse vor, die erwartet werden, wenn ein Krafteinrückungsexperiment durchgeführt wird, um die typische Ausgabe zu zeigen, die zu erwarten ist, wenn das Protokoll gut ausgeführt wird.
Kraft-Weg-Kurven
Repräsentative Krafteindringdiagramme, die mit einrückenden lebenden Proben an einer Position in der Mitte der Zelle der Wurzeldehnungszone erhalten wurden, sind in Abb...
Diskussion
Die Zell- und Zellwandmechanik wird immer relevanter, um Erkenntnisse darüber zu gewinnen, wie die Mechanik Wachstumsprozesse beeinflusst. Da sich physikalische Kräfte über beträchtliche Entfernungen in festen Geweben ausbreiten, wird die Untersuchung von Veränderungen in den physikalischen Eigenschaften der Zellwand und wie sie wahrgenommen, kontrolliert, abgestimmt und auf das Wachstum der Pflanze beeinflusst werden, zu einem wichtigen Forschungsgebiet 2,3,8
Offenlegungen
Die Autoren haben keine Interessenkonflikte. Das MATLAB-Skript, das für die Anpassung der Daten verwendet wird, ist auf persönliche Anfrage erhältlich, indem Sie an J. C. B. schreiben.
Danksagungen
Diese Forschung wurde gefördert durch CSIC I+D 2018, Grant No. 95 (Mariana Sotelo Silveira).; CSIC Grupos (Omar Borsani) und PEDECIBA.
Materialien
| Name | Company | Catalog Number | Comments |
| 1 x Phosphate-Buffered Saline (PBS) | Include sodium chloride and phosphate buffer and is formulated to prevent osmotic shock and maintain water balance in living cells. | ||
| AFM software | Bruker, Billerica, MA, USA | ||
| Atomic force microscopy (AFM) | BioScope Catalyst, Bruker, Billerica, MA, USA | ||
| Catalyst Probe holder-fluid | Bruker, Billerica, MA, USA | CAT-FCH | A probe holder for the Bioscope Catalyst, designed for fluid operation in contact or Tapping Mode. Also compatible with air operation. |
| Cryoscopic osmometer; model OSMOMAT 030 | Gonotech, Berlin, Germany | ||
| Murashige & Skoog Medium | Duchess Biochemie | M0221 | Original concentration, (1962) |
| Optical inverted microscope coupled to the AFM | Olympus IX81, Miami, FL, USA | ||
| PEGAMIL | ANAEROBICOS S.R.L., Buenos Aires, Argentina | 100429 | Neutral, non acidic silicone glue |
| Petri dishes | Deltalab | 200201.B | Polystyrene, 55 x 14 mm, radiation sterile. |
| Propidium iodide | Sigma | P4170 | For root viability test. |
| Silicon nitride probe, DNP-10, cantilever A | Bruker, Billerica, MA, USA | DNP-10/A | For force modulation microscopy in liquid operation. Probe tip radius of 20-60 nm. 175-μm-long triangular cantilever, with a spring constant of 0.35 N/m. |
| Tweezers | Sigma | T4537 |
Referenzen
- Anderson, C. T., Kieber, J. J. Dynamic construction, perception, and remodeling of plant cell walls. Annual Review of Plant Biology. 71, 39-69 (2020).
- Roeder, A. H. K., et al. Fifteen compelling open questions in plant cell biology. The Plant Cell. 34 (1), 72-102 (2022).
- Zhang, B., Gao, Y., Zhang, L., Zhou, Y. The plant cell wall: Biosynthesis, construction, and functions. Journal of Integrative Plant Biology. 63 (1), 251-272 (2021).
- Hamant, O., Haswell, E. S. Life behind the wall: Sensing mechanical cues in plants. BMC Biology. 15 (59), 1-9 (2017).
- Scheres, B., Benfey, P., Dolan, L. Root development. The Arabidopsis Book. 1, 0101 (2002).
- Cuadrado-Pedetti, M. B., et al. The arabidopsis tetratricopeptide thioredoxin-like 1 gene is involved in anisotropic root growth during osmotic stress adaptation. Genes. 12 (2), 236 (2021).
- Milani, P., Braybrook, S. A., Boudaoud, A. Shrinking the hammer: micromechanical approaches to morphogenesis. Journal of Experimental Botany. 64 (15), 4651-4662 (2013).
- Braybrook, S. A. Measuring the elasticity of plant cells with atomic force microscopy. Methods in Cell Biology. 125, 237-254 (2015).
- Bidhendi, A. J., Geitmann, A. Methods to quantify primary plant cell wall mechanics. Journal of Experimental Botany. 70 (14), 3615-3648 (2019).
- Desnos, T., et al. Procuste1 mutants identify two distinct genetic pathways controlling hypocotyl cell elongation, respectively in dark- and light-grown Arabidopsis seedlings. Development. 122 (2), 683-693 (1996).
- Fagard, M., et al. Procuste1 encodes a cellulose synthase required for normal cell elongation specifically in roots and dark-grown hypocotyls of arabidopsis. The Plant Cell. 12 (12), 2409-2423 (2000).
- Murashige, T., Skoog, F. A revised medium for rapid growth and bio assays with tobacco tissue cultures. Physiologia Plantarum. 15 (3), 473-497 (1962).
- Perilli, S., Sabatini, S. Analysis of root meristem size development. Methods in Molecular Biology. 655, 177-187 (2010).
- Sader, J. E., et al. A virtual instrument to standardise the calibration of atomic force microscope cantilevers. Review of Scientific Instruments. 87 (9), 093711 (2016).
- Collinsworth, A. M., Zhang, S., Kraus, W. E., Truskey, G. A. Apparent elastic modulus and hysteresis of skeletal muscle cells throughout differentiation. American Journal of Physiology - Cell Physiology. 283 (4), 1219-1227 (2002).
- Mathur, A. B., Collinsworth, A. M., Reichert, W. M., Kraus, W. E., Truskey, G. A. Endothelial, cardiac muscle and skeletal muscle exhibit different viscous and elastic properties as determined by atomic force microscopy. Journal of Biomechanics. 34 (12), 1545-1553 (2001).
- Sirghi, L., Ponti, J., Broggi, F., Rossi, F. Probing elasticity and adhesion of live cells by atomic force microscopy indentation. European Biophysics Journal. 37 (6), 935-945 (2008).
- Peaucelle, A. AFM-based mapping of the elastic properties of cell walls: At tissue, cellular, and subcellular resolutions. Journal of Visualized Experiments: JoVE. (89), e51317 (2014).
- Peaucelle, A., et al. Pectin-induced changes in cell wall mechanics underlie organ initiation in Arabidopsis. Current Biology. 21 (20), 1720-1726 (2011).
- Fernandes, A. N., et al. Mechanical properties of epidermal cells of whole living roots of Arabidopsis thaliana: An atomic force microscopy study. Physical Review E. 85 (2), 21916 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten