Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Microscopie à force atomique pour étudier les propriétés physiques des cellules épidermiques des racines vivantes d’Arabidopsis
Dans cet article
Résumé
Le protocole d’indentation par microscopie à force atomique offre la possibilité de disséquer le rôle des propriétés physiques de la paroi cellulaire d’une cellule particulière d’un tissu ou d’un organe pendant une croissance normale ou contrainte (c’est-à-dire sous déficit hydrique).
Résumé
Une méthode est décrite ici pour caractériser les propriétés physiques de la paroi cellulaire des cellules épidermiques des racines vivantes d’Arabidopsis par nanoindentations avec un microscope à force atomique (AFM) couplé à un microscope optique à fluorescence inversée. La méthode consiste à appliquer des forces contrôlées à l’échantillon tout en mesurant sa déformation, ce qui permet de quantifier des paramètres tels que le module apparent de Young des parois cellulaires à des résolutions subcellulaires. Cela nécessite une immobilisation mécanique minutieuse de l’échantillon et une sélection correcte des pénétrateurs et des profondeurs d’indentation. Bien qu’elle ne puisse être utilisée que dans les tissus externes, cette méthode permet de caractériser les changements mécaniques dans les parois cellulaires végétales au cours du développement et permet la corrélation de ces changements microscopiques avec la croissance d’un organe entier.
Introduction
Les cellules végétales sont entourées d’une paroi cellulaire qui est une structure complexe composée de réseaux en interaction de polysaccharides, de protéines, de métabolites et d’eau dont l’épaisseur varie de 0,1 à plusieurs μm selon le type cellulaire et la phase de croissance 1,2. Les propriétés mécaniques de la paroi cellulaire jouent un rôle essentiel dans la croissance des plantes. De faibles valeurs de rigidité de la paroi cellulaire ont été proposées comme condition préalable à la croissance cellulaire et à l’expansion de la paroi cellulaire, et il est de plus en plus évident que toutes les cellules détectent les forces mécaniques pour remplir leurs fonctions. Cependant, il est encore débattu si les changements dans les propriétés physiques de la paroi cellulaire déterminent le destin cellulaire 2,3,4. Parce que les cellules végétales ne bougent pas pendant le développement, la forme finale d’un organe dépend de la distance et de la direction dans laquelle une cellule se développe. Ainsi, la racine d’Arabidopsis est un bon modèle pour étudier l’impact des propriétés physiques de la paroi cellulaire dans l’expansion cellulaire, car différents types d’expansion se produisent dans différentes régions de la racine. Par exemple, l’expansion anisotrope est évidente dans la zone d’élongation et particulièrement perceptible dans les cellules épidermiques5.
La méthode décrite ici a été utilisée pour caractériser les propriétés physiques de la paroi cellulaire des cellules épidermiques à l’échelle nanométrique des racines vivantes d’Arabidopsis à l’aide d’un microscope à force atomique (AFM) couplé à un microscope à phase de fluorescence inversée6. Pour une révision approfondie de la technique AFM, lire 7,8,9.
Ce protocole décrit une méthode de préparation d’échantillons de base et une méthode générale pour les mesures d’élasticité basées sur AFM des parois cellulaires végétales.

Figure 1 : Vue d’ensemble schématique de l’expérience d’indentation de force dans les racines d’Arabidopsis à l’aide de la microscopie à force atomique (AFM). Le schéma donne un aperçu des étapes d’une expérience de force-indentation depuis la préparation du substrat pour immobiliser fermement l’échantillon racinaire (1-2), la confirmation de la viabilité de la racine par coloration à l’iodure de propidium (3), le positionnement en porte-à-faux à la surface d’une cellule épidermique allongée de la racine primaire (4-5), la mesure des courbes de force (6) et le traitement de la courbe de force pour calculer le module de Young apparent (7-8). EZ : zone d’allongement. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
1. Préparation du matériel végétal et conditions de croissance
- Pour générer le matériel végétal nécessaire, stérilisez les graines de type sauvage d’Arabidopsis et les lignées mutantes d’intérêt.
REMARQUE: Dans ce protocole, nous avons utilisé ce qui suit: ttl1: Lignes d’insertion de l’ADN-T Salk_063943 (pour TTL1; AT1G53300) - Columbia-0 (Col-0) de type sauvage; le mutant Procuste1 (prc1-1), qui consiste en une mutation knock-out (Q720stop) dans le gène CESA6 (AT5G64740), comme décrit précédemment dans10,11; et TTL1 x PRC1-1 Double Mutant.- Mettez un volume approximatif de 50 μL de graines dans un microtube. Ajouter 500 μL d’éthanol à 70 % + solution de Tween à 0,01 % à température ambiante. Mélanger et incuber pendant 7 min.
- Verser la solution d’éthanol et ajouter 500 μL d’hypochlorite de sodium à 20%. Mélanger et incuber pendant 7 min.
- Versez la solution d’hypochlorite de sodium et lavez les graines trois à quatre fois avec de l’eau stérile.
- Placage
- Préparer à l’avance le milieu basal de Murashige et Skoog (MS)12 + 1,5 % de saccharose + 1 % de gélose (pH 5,7) (voir le tableau des matières). Autoclave le médium. Préparez une hotte à flux laminaire pour travailler dans un environnement stérile et étiquetez les boîtes de Petri carrées nécessaires à l’expérience. Versez le milieu dans les boîtes de Petri et laissez le milieu se solidifier.
- Plaquer les graines stérilisées à l’aide d’embouts de pipette stériles sur des boîtes de Petri carrées contenant le milieu. Répartir les graines uniformément sur deux rangées. Lorsque les graines sont sèches, fermez les couvercles et scellez les assiettes. Stratifier les graines à 4 °C dans l’obscurité pendant 4 jours.
REMARQUE : La stratification est nécessaire pour synchroniser la germination. - Placer les plaques verticalement dans la chambre de croissance dans un régime de jours longs (16 h de lumière / 8 h d’obscurité) avec une intensité lumineuse de 25 μmol m2 s−1 et 23 °C (jour/nuit). Cultivez les plants pendant 7 jours.
2. Traitement du stress osmotique (facultatif)
NOTE: Cette section fournit des détails sur la croissance des racines d’Arabidopsis dans le potentiel osmotique de -1,2 MPa tel qu’estimé par osmomètre cryoscopique (Tableau des matériaux). Cette partie peut être omise ou modifiée en fonction de la question expérimentale en question.
- Préparer le milieu MS basal + 1,5 % saccharose + 400 mM de mannitol + 1 % d’agar (pH 5,7). Autoclave le médium. Versez le milieu dans des boîtes de Petri carrées dans la hotte à flux laminaire et laissez le milieu se solidifier.
- À l’aide d’une pince à épiler (table des matières), placer les plants âgés de 5 jours pré-cultivés dans un milieu MS basal + 1,5 % saccharose dans des boîtes de Petri carrées contenant le milieu complété par 400 mM de mannitol. Placer les plaques verticalement dans la chambre de croissance dans les mêmes conditions qu’à l’étape 1.2.3. Cultivez les plantules dans cette condition de stress osmotique pendant 7 jours.
NOTE: Pour évaluer l’adaptation de la croissance racinaire de la racine primaire lors d’un stress osmotique sévère, il est suggéré d’utiliser des plantules 5 jours après la germination cultivées dans des conditions contrôlées pour s’assurer que la taille du méristème de la racine est déjà établie13. Alternativement, l’analyse peut être effectuée à chaque étape du développement pour déterminer l’effet sur le développement de l’action du stress osmotique.
3. Expériences de nanoindentation en microscopie à force atomique (AFM)
- Préparation de l’échantillon pour les expériences de nanoindentation
NOTE: Il s’agit d’une étape cruciale pour l’application de l’AFM à l’étude d’échantillons biologiques vivants. L’échantillon doit être solidement fixé au substrat pour être mécaniquement stable pendant les mesures d’indentation. Dans le même temps, les qualités structurelles des échantillons doivent être préservées. Une stratégie efficace pour stabiliser un gros échantillon pour AFM consiste à utiliser des adhésifs. L’adhésif doit sécher rapidement et ne doit pas être toxique ou réactif avec le milieu environnant. Dans ce protocole, des boîtes de Petri en polystyrène ont été utilisées comme substrat et de la colle de silicone non acide (Table des matériaux) a été utilisée pour lier les plants d’Arabidopsis.- Étaler une fine couche de colle silicone dans la boîte de Petri avec un couvercle. Laissez la colle en l’air pendant 45 s.
- Avec une pince à épiler, placez le plant sur la colle en l’orientant dans une direction qui évite le contact entre les parties saillantes du plant et le porte-à-faux. Ensuite, appuyez doucement la racine sur la couche de colle de silicone pour la lier fermement. Laissez le semis en contact avec la colle pendant 45 s avant de passer aux étapes suivantes du protocole.
- Incuber les plantules attachées avec 2 μg/mL d’iodure de propidium (PI; Tableau des matériaux) pendant 5 minutes à l’obscurité et les laver soigneusement trois fois avec 1x solution saline tamponnée au phosphate (PBS) (table des matières).
- Placez les plantules incubées par PI dans un microscope à fluorescence inversée couplé à l’AFM. Pour observer la fluorescence, réglez les longueurs d’onde d’excitation et d’émission à 572 nm et 617 nm, respectivement. L’absence de fluorescence à l’intérieur des cellules épidermiques confirme la viabilité des racines.
- Couvrir le semis avec 1x solution saline tamponnée au phosphate (PBS).
- Protocole d’indentation
NOTE: Effectuer toutes les mesures AFM dans les 1 h suivant l’immobilisation de l’échantillon sur son support à température ambiante. La pièce où se trouve l’AFM doit être maintenue à 25 °C avec un climatiseur. La solution de PBS doit être à la même température.- Montez un porte-à-faux standard en nitrure de silicium avec une pointe pyramidale (voir le tableau des matériaux) dans le support de sonde AFM pour fluide (tableau des matériaux).
- Alignez le laser sur le porte-à-faux près de la position de la pointe. Ensuite, déplacez la photodiode pour placer le spot laser au centre du détecteur.
- Pour calibrer la sensibilité de déflexion, placez une boîte de Petri en polystyrène avec 1x PBS sous l’AFM.
Remarque : Cette étape est nécessaire pour calculer l’indentation. Lorsqu’une mesure de force est effectuée sur une surface dure, il n’y a pas d’indentation dans la surface, de sorte que tout changement dans le déplacement du scanner z correspond à la déviation en porte-à-faux. Par conséquent, il est très important de calibrer la sensibilité à la déviation sur une surface dure. L’utilisation d’une surface molle surestimera la sensibilité à la déflexion, ce qui entraînera des constantes de ressort trop faibles. - Régler le photodétecteur de manière à ce que la flèche sans contact soit proche de 0 V. Calibrer la sensibilité à la déviation en effectuant une indentation (courbe de force), avec une taille de rampe de 3 μm, un taux d’indentation et de rétraction de 0,6 μm/s et un seuil de déclenchement de 0,5 V.
- Pour étalonner la constante de ressort du porte-à-faux, utilisez l’utilitaire Thermal Tune du logiciel AFM (Table of Materials) recommandé pour les sondes dont les constantes de ressort sont inférieures à environ 5 N/m ou utilisez la méthode décrite dans la référence14. Avant d’activer Thermal Tune dans le logiciel, assurez-vous que la sonde n’interagit pas avec l’échantillon.
- Entrez la température en porte-à-faux. Cliquez sur Calibrer > Réglage thermique ou sur l’icône Réglage thermique dans la barre d’outils NanoScope . Sélectionnez une gamme de fréquences (1-100 Hz).
- Cliquez sur Acquérir des données dans le panneau Réglage thermique. Attendez que l’AFM acquière des données, puis cliquez sur le bouton Simple Harmonic Oscillator (Fluid).
- Ajustez la largeur médiane du filtre à 3. Ajustez la largeur de la corbeille PSD pour réduire le bruit dans les données acquises en faisant la moyenne. Définissez des limites d’ajustement autour du premier pic de résonance.
- Cliquez sur Calculer la constante de ressort k, puis cliquez sur Oui dans la fenêtre contextuelle demandant si l’utilisateur souhaite utiliser cette valeur.
- Répétez les étapes décrites ci-dessus trois fois et prenez manuellement une moyenne des valeurs des constantes de ressort. Entrez cette valeur moyenne dans la zone Constante de ressort de la liste des paramètres Ramp de la vue PicoForce. L’étalonnage se termine à ce stade.
- À l’aide du microscope optique inversé à un grossissement de l’oculaire de 10x, 20x et 40x, positionnez la sonde AFM sur la surface de la quatrième cellule épidermique allongée de la racine primaire, en veillant à la positionner au centre de la cellule.
- Avec la valeur calculée de la constante de ressort (étape 3.2.5.5), obtenir des courbes de force avec une taille de rampe de 3 μm, un seuil de déclenchement de 11 nN et un taux d’indentation et de rétraction de 0,6 μm/s aux points sélectionnés.
REMARQUE: Des travaux antérieurs15,16 ont montré que la faible fréquence d’indentation et de rétraction aide à minimiser l’hystérésis et la force de traînée. - Obtenir des courbes de force à partir de trois cellules par racine pour chaque traitement (utiliser trois réplicas biologiques par traitement). Capturez au moins 150 courbes de force pour chaque racine.
4. Mesurer le module de Young apparent
- Ajustez chaque courbe de force au modèle suivant pour les pénétrateurs pyramidaux17:
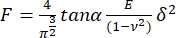 , où E est le module de Young apparent de l’échantillon,
, où E est le module de Young apparent de l’échantillon, 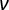 est le module de Poisson de l’échantillon et α est le demi-angle par rapport au sommet. Jetez les ajustements avec un coefficient de corrélation (r2) < 0,99. Considérons le rapport de Poisson pour un matériau totalement incompressible:
est le module de Poisson de l’échantillon et α est le demi-angle par rapport au sommet. Jetez les ajustements avec un coefficient de corrélation (r2) < 0,99. Considérons le rapport de Poisson pour un matériau totalement incompressible: 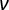 = 0,5 et la géométrie du pénétrateur donnée par les fabricants.
= 0,5 et la géométrie du pénétrateur donnée par les fabricants.
NOTA: Utiliser les 100 premiers nm du point de contact de la courbe de charge pour que le raccord soit aussi sensible que possible à la mécanique de la paroi cellulaire et pour réduire l’impact de la pression de turgescence sur les valeurs apparentes du modulede Young 8. Le montage a été réalisé à l’aide d’un programme MATLAB personnalisé (disponible sur demande personnelle en écrivant à J. C. B) qui permet de définir une profondeur d’indentation spécifique à partir du point de contact de la courbe de chargement. - Créez un histogramme normalisé avec les données de module de Young apparent et ajustez-le à une distribution gaussienne. Écartez les points de données en dehors de l’intervalle de confiance à 95 % et recalculez à la fois l’histogramme et l’ajustement gaussien. Calculer et rapporter la moyenne et l’écart-type du module de Young apparent.
- Déterminer l’importance des comparaisons entre les groupes par plusieurs tests de comparaison comme ANOVA.
Résultats
Expériences d’indentation de force
Le texte suivant présente certains résultats attendus lorsqu’une expérience d’indentation de force est menée pour montrer la sortie typique à attendre lorsque le protocole est bien exécuté.
Courbes force-déplacement
Les diagrammes d’indentation de force représentative qui ont été obtenus en indentant des échantillons vivants à une position placée au centre de la cellule de la zone d’élongation ...
Discussion
La mécanique des cellules et des parois cellulaires devient de plus en plus pertinente pour mieux comprendre comment la mécanique affecte les processus de croissance. Comme les forces physiques se propagent sur des distances considérables dans les tissus solides, l’étude des changements dans les propriétés physiques de la paroi cellulaire et comment ils sont détectés, contrôlés, réglés et impactent la croissance de la plante devient un domaine d’étude important <...
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts. Le script MATLAB utilisé pour ajuster les données est disponible sur demande personnelle en écrivant à J. C. B.
Remerciements
Cette recherche a été financée par CSIC I+D 2018, subvention n ° 95 (Mariana Sotelo Silveira). CSIC Grupos (Omar Borsani) et PEDECIBA.
matériels
| Name | Company | Catalog Number | Comments |
| 1 x Phosphate-Buffered Saline (PBS) | Include sodium chloride and phosphate buffer and is formulated to prevent osmotic shock and maintain water balance in living cells. | ||
| AFM software | Bruker, Billerica, MA, USA | ||
| Atomic force microscopy (AFM) | BioScope Catalyst, Bruker, Billerica, MA, USA | ||
| Catalyst Probe holder-fluid | Bruker, Billerica, MA, USA | CAT-FCH | A probe holder for the Bioscope Catalyst, designed for fluid operation in contact or Tapping Mode. Also compatible with air operation. |
| Cryoscopic osmometer; model OSMOMAT 030 | Gonotech, Berlin, Germany | ||
| Murashige & Skoog Medium | Duchess Biochemie | M0221 | Original concentration, (1962) |
| Optical inverted microscope coupled to the AFM | Olympus IX81, Miami, FL, USA | ||
| PEGAMIL | ANAEROBICOS S.R.L., Buenos Aires, Argentina | 100429 | Neutral, non acidic silicone glue |
| Petri dishes | Deltalab | 200201.B | Polystyrene, 55 x 14 mm, radiation sterile. |
| Propidium iodide | Sigma | P4170 | For root viability test. |
| Silicon nitride probe, DNP-10, cantilever A | Bruker, Billerica, MA, USA | DNP-10/A | For force modulation microscopy in liquid operation. Probe tip radius of 20-60 nm. 175-μm-long triangular cantilever, with a spring constant of 0.35 N/m. |
| Tweezers | Sigma | T4537 |
Références
- Anderson, C. T., Kieber, J. J. Dynamic construction, perception, and remodeling of plant cell walls. Annual Review of Plant Biology. 71, 39-69 (2020).
- Roeder, A. H. K., et al. Fifteen compelling open questions in plant cell biology. The Plant Cell. 34 (1), 72-102 (2022).
- Zhang, B., Gao, Y., Zhang, L., Zhou, Y. The plant cell wall: Biosynthesis, construction, and functions. Journal of Integrative Plant Biology. 63 (1), 251-272 (2021).
- Hamant, O., Haswell, E. S. Life behind the wall: Sensing mechanical cues in plants. BMC Biology. 15 (59), 1-9 (2017).
- Scheres, B., Benfey, P., Dolan, L. Root development. The Arabidopsis Book. 1, 0101 (2002).
- Cuadrado-Pedetti, M. B., et al. The arabidopsis tetratricopeptide thioredoxin-like 1 gene is involved in anisotropic root growth during osmotic stress adaptation. Genes. 12 (2), 236 (2021).
- Milani, P., Braybrook, S. A., Boudaoud, A. Shrinking the hammer: micromechanical approaches to morphogenesis. Journal of Experimental Botany. 64 (15), 4651-4662 (2013).
- Braybrook, S. A. Measuring the elasticity of plant cells with atomic force microscopy. Methods in Cell Biology. 125, 237-254 (2015).
- Bidhendi, A. J., Geitmann, A. Methods to quantify primary plant cell wall mechanics. Journal of Experimental Botany. 70 (14), 3615-3648 (2019).
- Desnos, T., et al. Procuste1 mutants identify two distinct genetic pathways controlling hypocotyl cell elongation, respectively in dark- and light-grown Arabidopsis seedlings. Development. 122 (2), 683-693 (1996).
- Fagard, M., et al. Procuste1 encodes a cellulose synthase required for normal cell elongation specifically in roots and dark-grown hypocotyls of arabidopsis. The Plant Cell. 12 (12), 2409-2423 (2000).
- Murashige, T., Skoog, F. A revised medium for rapid growth and bio assays with tobacco tissue cultures. Physiologia Plantarum. 15 (3), 473-497 (1962).
- Perilli, S., Sabatini, S. Analysis of root meristem size development. Methods in Molecular Biology. 655, 177-187 (2010).
- Sader, J. E., et al. A virtual instrument to standardise the calibration of atomic force microscope cantilevers. Review of Scientific Instruments. 87 (9), 093711 (2016).
- Collinsworth, A. M., Zhang, S., Kraus, W. E., Truskey, G. A. Apparent elastic modulus and hysteresis of skeletal muscle cells throughout differentiation. American Journal of Physiology - Cell Physiology. 283 (4), 1219-1227 (2002).
- Mathur, A. B., Collinsworth, A. M., Reichert, W. M., Kraus, W. E., Truskey, G. A. Endothelial, cardiac muscle and skeletal muscle exhibit different viscous and elastic properties as determined by atomic force microscopy. Journal of Biomechanics. 34 (12), 1545-1553 (2001).
- Sirghi, L., Ponti, J., Broggi, F., Rossi, F. Probing elasticity and adhesion of live cells by atomic force microscopy indentation. European Biophysics Journal. 37 (6), 935-945 (2008).
- Peaucelle, A. AFM-based mapping of the elastic properties of cell walls: At tissue, cellular, and subcellular resolutions. Journal of Visualized Experiments: JoVE. (89), e51317 (2014).
- Peaucelle, A., et al. Pectin-induced changes in cell wall mechanics underlie organ initiation in Arabidopsis. Current Biology. 21 (20), 1720-1726 (2011).
- Fernandes, A. N., et al. Mechanical properties of epidermal cells of whole living roots of Arabidopsis thaliana: An atomic force microscopy study. Physical Review E. 85 (2), 21916 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon