Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Phänotypisches Profiling von aus menschlichen Stammzellen gewonnenen dopaminergen Neuronen im Mittelhirn
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die Zellkultivierung menschlicher dopaminerger Neuronen im Mittelhirn, gefolgt von immunologischen Färbungen und der Generierung neuronaler phänotypischer Profile aus aufgenommenen mikroskopischen High-Content-Bildern, die die Identifizierung phänotypischer Variationen aufgrund genetischer oder chemischer Modulationen ermöglichen.
Zusammenfassung
Die Parkinson-Krankheit (PD) ist mit einer Reihe zellbiologischer Prozesse verbunden, die den Verlust von dopaminergen (mDA) Neuronen im Mittelhirn verursachen. Vielen aktuellen In-vitro-PD-Zellmodellen mangelt es an Komplexität und sie berücksichtigen nicht mehrere Phänotypen. Phänotypisches Profiling in humanen induzierten pluripotenten Stammzellen (iPSC)-abgeleiteten mDA-Neuronen kann diese Mängel beheben, indem gleichzeitig eine Reihe von neuronalen Phänotypen in einem Parkinson-relevanten Zelltyp parallel gemessen wird. In dieser Arbeit beschreiben wir ein Protokoll zur Gewinnung und Analyse phänotypischer Profile von kommerziell erhältlichen humanen mDA-Neuronen. Ein neuronenspezifisches Fluoreszenz-Färbepanel wird verwendet, um die mit Kern, α-Synuclein, Tyrosinhydroxylase (TH) und Mikrotubuli-assoziiertem Protein 2 (MAP2) verwandten Phänotypen zu visualisieren. Das beschriebene phänotypische Profilierungsprotokoll ist skalierbar, da es 384-Well-Platten, automatisches Liquid Handling und Hochdurchsatzmikroskopie verwendet. Der Nutzen des Protokolls wird anhand gesunder mDA-Neuronen von Spendern und mDA-Neuronen veranschaulicht, die die PD-verknüpfte G2019S-Mutation im Gen für die Leucin-reiche Repeat-Kinase 2 (LRRK2) tragen. Beide Zelllinien wurden mit dem LRRK2-Kinase-Inhibitor PFE-360 behandelt und phänotypische Veränderungen gemessen. Darüber hinaus zeigen wir, wie multidimensionale phänotypische Profile mit Hilfe von Clustering oder maschinellem Lernen gesteuerten überwachten Klassifikationsmethoden analysiert werden können. Das beschriebene Protokoll wird besonders für Forscher interessant sein, die an der Modellierung neuronaler Krankheiten arbeiten oder die Wirkung chemischer Verbindungen in menschlichen Neuronen untersuchen.
Einleitung
Bei der Parkinson-Krankheit (PD) sind verschiedene zellbiologische Prozesse gestört. Zum Beispiel wurden mitochondriale Dysfunktion, oxidativer Stress, Proteinabbaudefekte, Störung des vesikulären Transports und der endolysosomalen Funktion mit dem Verlust von dopaminergen (mDA) Neuronen im Mittelhirn in Verbindung gebracht, die häufig bei PD1 beobachtet werden. Daher scheint Parkinson mehrere Krankheitsmechanismen zu beinhalten, die miteinander interagieren und sich gegenseitig verschlimmern können. Eine nützliche Möglichkeit, dieses mechanistische Zusammenspiel zu untersuchen, ist die Erstellung eines umfassenden phänotypischen Fingerabdrucks oder Profils von dopaminergen (mDA) Neuronen im Mittelhirn.
Phänotypisches Profiling ist ein Ansatz, bei dem ein Profil einer Stichprobe auf der Grundlage einer Sammlung messbarer Merkmale erstellt wird, und zweitens werden auf der Grundlage dieses Profils Vorhersagen über eine Stichprobe getroffen 2,3. Das Ziel der Profilerstellung ist es, eine Vielzahl von Merkmalen zu erfassen, von denen einige zuvor möglicherweise nicht mit einer Krankheit oder Behandlung in Verbindung gebracht wurden3. Infolgedessen kann das Profiling unerwartete biologische Prozesse aufdecken. Die Erstellung von phänotypischen Profilen beruht in der Regel auf fluoreszenzgefärbten Zellen, und standardisierte Assays, wie z. B. Cell Painting, wurden entwickelt, um phänotypische Profile zu erstellen4. In jüngster Zeit wird phänotypisches Profiling beispielsweise für die Charakterisierung kleiner Moleküle oder die genaue Vorhersage von PD-Subtypen ausschließlich auf der Grundlage von patienteneigenen Fibroblasten eingesetzt 5,6. Trotz dieser Fortschritte wurde die phänotypische Profilerstellung bisher nur selten auf post-mitotisch differenzierte Zellen angewendet, wie z. B. von humanen induzierten pluripotenten Stammzellen (iPSC) abgeleitete mDA-Neuronen, die PD-assoziierte Mutationen wie LRRK2 G2019S exprimieren. Zu den wesentlichen Herausforderungen von iPSC-abgeleiteten Modellen gehören das Vorhandensein subtiler oder variabler pathologischer Merkmale über Differenzierungschargen oder Genotypen hinweg und die Tatsache, dass isolierte PD-Phänotypen nicht die volle Komplexität der Krankheit erfassen. Darüber hinaus sind neuronale iPSC-Modelle zwar physiologisch relevant, werden aber aufgrund von Bedenken hinsichtlich der technischen Komplexität nur selten in der PD-Wirkstoffforschung eingesetzt 7,8.
Wir haben zuvor eine robuste Methodik entwickelt, um mehrere Parkinson-bedingte pathophysiologische Phänotypen in menschlichen mDA-Neuronen zu messen, die sowohl empfindlich auf genetische als auch auf durch chemische Verbindungen induzierte phänotypische Veränderungen reagieren9. Dieser Artikel beschreibt detailliert eine weiter optimierte Version dieser Methodik, um phänotypische Profile aus mDA-Neuronen zu erstellen (Abbildung 1). Dieses Protokoll hat mehrere Vorteile gegenüber den zuvor beschriebenen phänotypischen Profiling-Ansätzen, wie z.B. die Verwendung hochwertiger mDA-Neuronen und die technische Reproduzierbarkeit. Dieses Protokoll ermöglicht zum ersten Mal ein phänotypisches Profiling in physiologisch relevanten post-mitotischen mDA-Neuronen nach chemischen Störungen in einer hochskalierbaren Weise. Vollständig differenzierte und kryokonservierte mDA-Neuronen sind kommerziell erhältlich, wodurch die Variabilität der Differenzierung von Charge zu Charge erheblich verringert wird. Zweitens kann die technische Variabilität durch ein klar definiertes Versuchsdesign (d. h. Kulturdauer oder Vermeidung von Randvertiefungen), automatisiertes Liquid Handling und automatisierte Mikroskopie weiter reduziert werden. Darüber hinaus werden hier die ersten Schritte der phänotypischen Profilanalyse unter Verwendung von unüberwachtem Clustering oder überwachten Klassifikationsansätzen skizziert, die zeigen, wie phänotypische Profiling-Daten analysiert werden können. Dieses Protokoll wird für Forscher von Nutzen sein, die sich für phänotypische Veränderungen von mDA-Neuronen interessieren, die durch genetische oder chemische Störungen induziert werden, insbesondere wenn ein hochgradig skalierbarer Studienaufbau erforderlich ist, z. B. bei Screening-Kampagnen oder wenn die Wirkung einer kleineren Anzahl von Verbindungen untersucht werden soll, z. B. um toxische Wirkungen zu bestimmen. Zusammenfassend wird erwartet, dass die Anwendung des phänotypischen Profilings menschlicher Neuronen eine wertvolle Technik ist, um komplexe krankheitsbedingte Phänotypen zu untersuchen und die zellulären Effekte von Wirkstoffkandidaten zu charakterisieren.
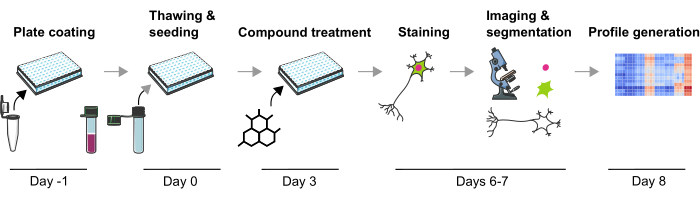
Abbildung 1: Schematische Darstellung des experimentellen Protokolls zur Generierung bildbasierter phänotypischer Profile aus humanen iPSC-abgeleiteten mDA-Neuronen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protokoll
1. Vorbereitung des Mediums und der Platten für das Neuronen-Seeding (Tag 1)
- Um die Platten für die Aussaat der Neuronen an Tag 1 vorzubereiten, erwärmen Sie Laminin kurz vor der Anwendung auf Raumtemperatur (RT). Bereiten Sie die Lamininlösung vor, indem Sie die Laminin-Stammlösung (0,1 mg/ml) 1/10 in kaltem PBS+/+ (mit Ca 2+ und Mg2+) verdünnen.
HINWEIS: Alle Reagenzien sind in der Materialtabelle aufgeführt. Die Zusammensetzung von Lösungen und Puffern ist in den Tabellen 1-4 beschrieben. - Dann werden 25 μl der Lamininlösung in jede Vertiefung einer Poly-D-Lysin (PDL) vorbeschichteten 384-Well-Platte gegeben und über Nacht bei 4 °C inkubiert. Die beschichteten Platten können bei 4 °C bis zu einer Woche gelagert werden.
HINWEIS: Das Protokoll kann hier bis zu einer Woche pausiert werden. Versiegeln Sie die Platten mit Kunststofffolie. - Bereiten Sie Complete Maintenance Media vor und lagern Sie es bis zu einem Monat bei 4 °C (Tabelle 1).
2. Auftauen von Neuronen (Tag 0)
- Um die Neuronen an Tag 0 aufzutauen, wird ein Wasserbad auf 37 °C vorgewärmt und das Complete Maintenance Media lichtgeschützt auf RT gestellt.
- Nehmen Sie das Fläschchen mit den im Handel erhältlichen gefrorenen Neuronen (siehe Materialtabelle) aus dem Flüssigstickstofftank und legen Sie es auf Trockeneis. Stellen Sie dann die Durchstechflasche für 2 Minuten in das Wasserbad. Sobald die Flüssigkeit vollständig aufgetaut ist, desinfizieren Sie die Durchstechflasche mit 70%igem Ethanol.
- Die aufgetauten Neuronen werden mit einer P1000-Pipette (ca. 370 μl) abgesaugt und in ein 50-ml-Zentrifugenröhrchen ohne Auf- und Abpipettieren überführt. Als nächstes wird die Durchstechflasche mit 630 μl Complete Maintenance Medium gespült und tropfenweise (45°-Winkel) in das 50-ml-Röhrchen gegeben. Während der Dosierung langsam rühren.
HINWEIS: Es ist wichtig, das Medium langsam Tropfen für Tropfen zu dosieren, um einen osmotischen Bruch der Zellen zu vermeiden. Ein Winkel von 45° und leichtes Rühren minimiert den hohen lokalen osmotischen Druck während des Pipettierens. - In ähnlicher Weise 1 ml vollständiges Erhaltungsmedium in das 50-ml-Zentrifugenröhrchen mit einer P1000-Pipette geben. Fügen Sie dann langsam 2 ml Complete Maintenance Medium hinzu. Während der Abgabe vorsichtig umrühren.
- Zähle die Zellen. Bereiten Sie ein Mikroröhrchen mit 10 μl Trypanblau und 10 μl Zellsuspension vor und geben Sie es in einen Objektträger der Zählkammer (10 μl). Führen Sie die Zählung mit einem automatischen Zellzähler (siehe Materialtabelle) oder manuell durch.
- Nach dem Zählen zentrifugieren Sie das 50-ml-Röhrchen mit den Neuronen bei 400 x g 5 Minuten lang bei RT und entfernen Sie den Überstand. Resuspendieren Sie das Pellet vorsichtig mit einer P1000-Pipette und 1 ml Complete Maintenance Medium. Geben Sie dann das erforderliche Volumen hinzu, um die gewünschte Konzentration zu erreichen (300.000 Zellen/ml, siehe auch nächster Schritt).
3. Aussaat von Neuronen auf präparierten Platten (Tag 0)
- Um Neuronen auf den vorbereiteten Platten an Tag 0 auszusäen, nehmen Sie die beschichteten Platten aus dem Kühlschrank, legen Sie sie unter die Zellkulturhaube und lassen Sie sie etwa 30 Minuten lang zu RT äquilibrieren.
- Saugen Sie kurz vor der Aussaat 15 μl Beschichtungslösung mit einem automatisierten Liquid Handler oder einer 16-Kanal-Pipette an. Lassen Sie ca. 10 μl pro Vertiefung stehen, um eine Beschädigung der Beschichtung zu vermeiden.
- Als nächstes werden 50 μl der Zelllösung mit 300.000 Zellen/ml (hergestellt in Schritt 2.6) pro Vertiefung mit einer 16-Kanal-Pipette abgegeben, was zu 15.000 ausgesäten Neuronen pro Vertiefung und einem Endvolumen pro Vertiefung von 60 μl führt.
- Vermeiden Sie bei einer 384-Well-Platte die Verwendung der Spalten 1, 2, 23, 24 und der Zeilen A, B, O und P, um mögliche Kanteneffekte zu minimieren, die sich auf die phänotypischen Profile auswirken können. Füllen Sie die unbenutzten leeren Vertiefungen mit 80 μl PBS. Die Platten werden bei 37 °C und 5 % CO2 inkubiert.
4. Mediumwechsel oder Compound-Behandlung (Tag 3)
- Abhängig von der Anzahl der Vertiefungen eine angemessene Menge des Complete Maintenance Mediums bei RT vorwärmen. Vor Licht schützen.
- Wenn ein Medienwechsel erforderlich ist, fahren Sie mit Schritt 4.4 fort. Wenn eine Behandlung der Verbindung gewünscht wird, überprüfen Sie die Konzentration der Verbindung und das verwendete Lösungsmittel (Wasser, DMSO, Methanol usw.).
- Bereiten Sie eine 1,5-fach konzentrierte Verbindungslösung für alle gewünschten zu testenden Konzentrationen vor. Bereiten Sie die Verdünnungen der Verbindung mit Complete Maintenance Medium vor.
HINWEIS: Verwenden Sie als neutrale Kontrolle das entsprechende Lösungsmittel in der gleichen Konzentration wie die getestete Verbindung. Wenn mehrere oder unbekannte Verbindungen in unterschiedlichen Konzentrationen verwendet werden, ist es ratsam, ein separates Dosis-Wirkungs-Experiment durchzuführen, um die Auswirkungen des Lösungsmittels auf das phänotypische Profil zu messen. - Wenn ein automatisches Pipettiersystem verwendet wird, geben Sie 60 μl der 1,5-fachen Verbindungslösung auf eine 384-Well-Aufbewahrungsplatte. Alternativ können Sie auch eine 16-Kanal-Pipette verwenden. Wenn ein Medienwechsel erforderlich ist, fügen Sie stattdessen 60 μl Complete Maintenance Medium hinzu.
- Mit dem automatischen Pipettiersystem werden 40 μl Medium pro Vertiefung von der neuronenhaltigen Platte abgesaugt und verworfen, um 20 μl pro Vertiefung zu erhalten. Geben Sie dann 40 μl/Well 1,5x Verbindungslösung von der 384-Well-Speicherplatte in jede Well, um die gewünschte Endkonzentration zu erhalten.
HINWEIS: Es ist wichtig, keinen vollständigen Medienwechsel durchzuführen, sondern immer Restmedium in der Vertiefung zu belassen, um eine Beschädigung des neuronalen Teppichs oder der Beschichtung zu vermeiden. - Falls die neuronale Kultur länger als die in diesem Protokoll beschriebenen 6 Tage dauert, wechseln Sie das Medium alle 2-3 Tage.
5. Neuronenfixierung und -färbung (Tage 6 bis 7)
- Um die Neuronen an den Tagen 6 und 7 zu fixieren und zu färben, verwenden Sie einen automatischen Liquid Handler für alle Abgabe- und Waschschritte. Alternativ können Sie auch eine 16-Kanal-Pipette verwenden.
- Bereiten Sie eine 10%ige Triton X-100-Lösung vor, indem Sie Triton X-100 in 1x PBS verdünnen. Vortex, bis die Lösung homogen ist. Bei 4 °C lagern.
- Um die Neuronen zu fixieren, geben Sie 20 μl/Well 16% PFA ab, was zu einer Endkonzentration von 4% führt. Inkubieren Sie die Platte für 30 Minuten bei RT und waschen Sie sie dreimal mit 1x PBS. Lassen Sie 20 μl/Well PBS nach der letzten Wäsche stehen.
VORSICHT: PFA ist als gefährliche Substanz anerkannt, von der bekannt ist, dass sie orale, dermale und respiratorische Toxizität verursacht. Es stellt auch eine Gefahr für die Augen dar und kann zu genetischen Mutationen und Krebs führen. Der richtige Umgang mit PFA erfordert die Verwendung geeigneter persönlicher Schutzausrüstung, wie z. B. Augen- und Gesichtsschutz, sowie die Sicherstellung einer ordnungsgemäßen Belüftung. Es ist wichtig, die Freisetzung von PFA in die Umwelt zu verhindern. - Für die Permeabilisierung und Blockierung wird eine 2-fache Blockierungslösung hergestellt (Tabelle 2).
- 20 μl/Well 2x Blockierungslösung (1x Endkonzentration) zugeben, 1 h bei RT inkubieren und einmal mit PBS waschen. Bewahren Sie 20 μl/Well PBS nach dem Waschen auf.
- Für die Färbung von Primärantikörpern (siehe Materialtabelle) ist ein 2-facher Primärfärbepuff herzustellen (Tabelle 3).
- 20 μl/Well 2x primären Färbepuffer (1x Endkonzentration) zugeben und über Nacht bei 4 °C inkubieren. Am nächsten Morgen an Tag 7 dreimal mit PBS waschen. Lassen Sie nach dem Waschen 20 μl/Wells PBS stehen.
- Für die Färbung von Sekundärantikörpern (siehe Materialtabelle) ist ein 2-facher sekundärer Färbepuff vorzubereiten (Tabelle 4).
- 20 μl/Well 2x sekundären Färbepuffer (1x Endkonzentration) hinzufügen, 2 h bei RT ohne Licht inkubieren und dreimal mit PBS waschen. Lassen Sie 100 μl PBS/Well nach der letzten Wäsche stehen.
- Fügen Sie der Platte eine Aluminiumversiegelung hinzu, um die Verdunstung zu minimieren. Alternativ können Sie die Platte auch mit Plastikfolie und Alufolie abdecken. Fahren Sie mit der Bildaufnahme fort oder lagern Sie die Platte bei 4 °C.
HINWEIS: Das Protokoll kann hier bis zu einer Woche pausiert werden. Wenn Hintergrundfluoreszenz beobachtet wird, sollten Sie die Blockierungszeit erhöhen. Wenn die Färbung nicht ausreicht, versuchen Sie, die Primärantikörperkonzentration oder die Inkubationszeit zu erhöhen.
- Fügen Sie der Platte eine Aluminiumversiegelung hinzu, um die Verdunstung zu minimieren. Alternativ können Sie die Platte auch mit Plastikfolie und Alufolie abdecken. Fahren Sie mit der Bildaufnahme fort oder lagern Sie die Platte bei 4 °C.
6. Bildgebung fluoreszenzgefärbter Neuronen (Tag 7)
- Nehmen Sie Bilder der plattierten, kultivierten und gefärbten Neuronen an Tag 7 auf. Idealerweise verwenden Sie ein automatisiertes konfokales Fluoreszenzmikroskop (siehe Materialtabelle). Alternativ können Sie Bilder auch manuell erfassen.
- Erfassen Sie die Hoechst-, TH-, α-Synuclein- und MAP2-Kanäle mit 405-nm-, 488-nm-, 561-nm- bzw. 647-nm-Lasern (Abbildung 2).
HINWEIS: Um eine ausreichende Menge an detaillierten Daten für die phänotypische Profilerstellung zu generieren, verwenden Sie ein 40-faches Objektiv und erfassen Sie 16 Felder/Wells mit Z-Stapeln, die aus 3 Z-Schichten bestehen, die durch 2 μm getrennt sind. Falls es pipettierbedingte Schäden im neuronalen Teppich gibt, versuchen Sie, eine Bildgebung dieser Bereiche zu vermeiden. - Stellen Sie je nach Mikroskop und Kamera die Belichtungszeiten und Anregungsintensitäten für jeden der vier Fluoreszenzkanäle separat ein, um einen optimalen Dynamikbereich der Fluoreszenzintensitäten zu erhalten.
HINWEIS: Bildgebungssoftware liefert oft ein Histogramm, um die ideale Belichtungszeit zu bestimmen. Ist das Histogramm im niedrigen Signalbereich zu stark nach links verschoben, dann ist die Belichtungszeit zu kurz oder die Anregungsintensität zu gering. Befindet sich rechts beim maximalen Signalpegel eine scharfe Klippe, so ist der Signalwert gesättigt. Reduzieren Sie in diesem Fall die Anregungsintensität oder verkürzen Sie die Belichtungszeit. - Speichern Sie Bilder in einem verlustfreien und offenen Format wie .tif.
7. Bildverarbeitung (Tag 8)
- Bildsegmentierung und phänotypische Merkmalsextraktion sind für die Erstellung quantitativer phänotypischer Profile erforderlich. Verwenden Sie die PhenoLink-Software für die Bildsegmentierung und Merkmalsextraktion (Materialtabelle). Eine Anleitung zur Installation von PhenoLink finden Sie im GitHub-Repository (https://github.com/Ksilink/PhenoLink).
HINWEIS: Die Bildsegmentierung wird verwendet, um verschiedene Objekte oder Regionen in einem Bild zu identifizieren und zu trennen, während die Extraktion phänotypischer Merkmale verwendet wird, um relevante Informationen aus diesen Regionen zu analysieren und zu extrahieren. Es stehen mehrere alternative Softwarelösungen zur Verfügung, wie z. B. CellProfiler10, ImageJ/FIJI 11, Napari 12 oder die Knime Analytics Platform13, um quantitative Informationen aus Mehrkanal-Fluoreszenzbildern zu extrahieren. - Führen Sie eine Bildsegmentierung für beleuchtungskorrigierte RAW-Bilder durch. Bestimmen Sie die jeweiligen Intensitätsschwellen des Fluoreszenzkanals empirisch pro Platte, so dass das Hintergrundsignal minimal ist und das gewünschte segmentierte Signal dem Signal im Rohbild entspricht. Platten, die am selben Tag verarbeitet und gefärbt werden, erfordern in der Regel vergleichbare Kanalintensitätsschwellen für die Segmentierung.
- Definieren Sie die Größe und Intensität des Zellkerns, um lebende von toten Zellen zu trennen. Wenn Sie 40x-Bilder verwenden, behalten Sie alle anderen Standardparameter bei und führen Sie die Software aus. Pro Well werden einhundertsechsundzwanzig quantitative Bildmerkmale berechnet (Ergänzende Tabelle 1).
- Verwenden Sie die resultierenden tabellarischen quantitativen Daten, um phänotypische Profile zu erstellen und phänotypische Profile aus verschiedenen Zelllinien oder Behandlungsbedingungen zu vergleichen. Jede Zeile entspricht einem biologischen Zustand (Well) und jede Spalte entspricht einem bestimmten phänotypischen Merkmal.
HINWEIS: Wir stellen eine Beispielausgabedatei zusammen mit der Datenanalyse-Pipeline zur Verfügung, um ihre Verwendung zu veranschaulichen (siehe Materialtabelle). Zusätzlich zeigt Abbildung 3 die Zusammensetzung eines phänotypischen Profils.
8. Erstellung und Visualisierung phänotypischer Profile (Tag 8)
- Wenn Sie Python und Jupyter nicht auf Ihrem Computer installiert haben, installieren Sie die Anaconda-Distribution, und öffnen Sie die Jupyter-Software. Laden Sie das bereitgestellte Jupyter-Notebook und alle anderen bereitgestellten Dateien herunter, und speichern Sie sie im selben Verzeichnis (siehe Materialtabelle). Öffnen Sie die Jupyter-Notebook-Datei mit der Jupyter-Software.
HINWEIS: Anaconda ist eine kostenlose Open-Source-Plattform für Programmiersprachen wie Python. Diese Plattform wird mit dem Python-Interpreter Jupyter geliefert, der das bereitgestellte Jupyter-Notebook ausführen kann, um phänotypische Profile (https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling) zu erstellen und zu analysieren. - Führen Sie das Jupyter-Notebook mithilfe der Jupyter-Software Zelle für Zelle aus. Die bereitgestellten Beispieldaten-, FTH- und .txt-Anforderungsdateien müssen sich im selben Verzeichnis wie das Jupyter-Notebook befinden. Jede Jupyter Notebook-Zelle ist mit Anmerkungen versehen, um ihre Funktionalität zu erläutern.
HINWEIS: Es ist wichtig, das Jupyter-Notebook in der richtigen Reihenfolge zu verwenden, beginnend mit dem Laden und Skalieren der Daten, und Zelle für Zelle bis zum Ende fortzufahren. Alle Daten- und Grafikausgaben werden in einem neu erstellten Ordner im Quellverzeichnis des Jupyter Notebooks gespeichert. Abbildung 4 zeigt den Workflow und die Workflow-Ausgabe.
Ergebnisse
Das phänotypische Profiling in mDA-Neuronen ist ein effizienter Weg, um mehrere Aspekte der Zellbiologie und ihre Veränderungen während der experimentellen Modulation zu quantifizieren. Um diese Methodik zu veranschaulichen, wurden in dieser Studie kryokonservierte LRRK2 G2019S und gesunde Spender-mDA-Neuronen verwendet. Diese Neuronen wurden seit ca. 37 Tagen differenziert, sind postmitotisch und exprimieren neuronale Marker (TUBB3 und MAP2) und dopaminerge Neuronenmarker, einschließlich Tyrosinhydroxylase (TH) in K...
Diskussion
Phänotypisches Profiling ist eine Technik zur Messung einer großen Anzahl von Phänotypen in Zellen durch Fluoreszenzfärbungen, Mikroskopie und Bildanalyse3. Phänotypische Profile können über Zelllinien oder andere experimentelle Bedingungen hinweg erstellt und verglichen werden, um komplexe Veränderungen in der Zellbiologie zu verstehen, die bei Verwendung einer einzigen Auslesung möglicherweise unbemerkt bleiben. Hier beschreiben wir die Anwendung von phänotypischem Profiling auf humane...
Offenlegungen
Alle Autoren sind bei Ksilink angestellt.
Danksagungen
Die Autoren bedanken sich bei allen Kolleginnen und Kollegen von Ksilink für ihre wertvolle Hilfe und die Diskussionen, die zur Gestaltung des vorgestellten Protokolls geführt haben.
Materialien
| Name | Company | Catalog Number | Comments |
| Anti- chicken – Alexa 647 | Jackson ImmunoRearch | 703-605-155 | Immunofluorescence |
| Anaconda | https://www.anaconda.com/download | ||
| Anti-Map2 | Novus | NB300-213 | Immunofluorescence |
| Anti-mouse - Alexa 488 | Thermo Fisher | A11001 | Immunofluorescence |
| Anti-rabbit - Alexa 555 | Thermo Fisher | A21429 | Immunofluorescence |
| Anti-Tyrosine Hydroxylase | Merck | T2928 | Immunofluorescence |
| Anti-α-synuclein | Abcam | 138501 | Immunofluorescence |
| Bravo Automated Liquid Handling Platform with 384ST head | Agilent | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Confocal microscope | Yokogawa | CV7000 | The use of an automated confocal fluorescence microscope is recommended to ensure image quality consistency. |
| Countess Automated cell counter | Invitrogen | Cell counting before seeding. Can also be done using a manual counting chamber. | |
| DPBS +/+ | Gibco | 14040-133 | Buffer for washing |
| EL406 Washer Dispenser | BioTek (Agilent) | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Formaldehyde Solution (PFA 16 %) | Euromedex | EM-15710-S | Fixation before staining |
| Hoechst 33342 | Invitrogen | H3570 | Nuclear staining |
| iCell Base Medium 1 | Fujifilm | M1010 | Base medium for neurons |
| iCell DPN, Donor#01279, Phenotype AHN, lot#106339, 1M | Fujifilm | C1087 | Apparently healthy donor |
| iCell DPN, Donor#11299, Phenotype LRRK2 G2019S, phenotype PD lot#106139 | Fujifilm | C1149 | Donor carrying LRRK2 G2019S mutation |
| iCell Nervous System Supplement | Fujifilm | M1031 | Supplement for base medium |
| iCell Neural Supplement B | Fujifilm | M1029 | Supplement for base medium |
| Jupyter Python Notebook | In-house development | https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling | Notebook to perform phenotypic profile visualization and classification from raw data. |
| Laminin | Biolamina | LN521 | Plate coating |
| PFE-360 | MedChemExpress | HY-120085 | LRRK2 kinase inhibitor |
| PhenoLink | In-house development | https://github.com/Ksilink/PhenoLink | Software for image analysis |
| PhenoPlate 384w, PDL coated | Perkin Elmer | 6057500 | Pre-coated plate for cell culture and imaging. This plate allows imaging of all wells using all objectives of the Yokogawa CV7000 microscope. |
| Storage plates Abgene 120 µL | Thermo Scientific | AB-0781 | Necessary for compound dispensing using the Vprep pipetting system. If not available, the use of an electronic multichannel pipette is recommended. |
| Triton | Sigma | T9284 | Permeabilization before lysis |
| Trypan Blue | Sigma | T8154-20ML | Determination of living cells |
| Vprep Pipetting System | Agilent | Medium change and compound dispensing. Alternatively, an electronic multichannel pipette can be used. |
Referenzen
- Panicker, N., Ge, P., Dawson, V. L., Dawson, T. M. The cell biology of Parkinson's disease. The Journal of Cell Biology. 220 (4), 202012095 (2021).
- Caicedo, J. C., et al. Data-analysis strategies for image-based cell profiling. Nature Methods. 14 (9), 849-863 (2017).
- Chandrasekaran, S. N., Ceulemans, H., Boyd, J. D., Carpenter, A. E. Image-based profiling for drug discovery: due for a machine-learning upgrade. Nature Reviews Drug Discovery. 20 (2), 145-159 (2021).
- Bray, M. -. A., et al. Cell Painting, a high-content image-based assay for morphological profiling using multiplexed fluorescent dyes. Nature Protocols. 11 (9), 1757-1774 (2016).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Ziegler, S., Sievers, S., Waldmann, H. Morphological profiling of small molecules. Cell Chemical Biology. 28 (3), 300-319 (2021).
- Cobb, M. M., Ravisankar, A., Skibinski, G., Finkbeiner, S. iPS cells in the study of PD molecular pathogenesis. Cell and Tissue Research. 373 (1), 61-77 (2018).
- Elitt, M. S., Barbar, L., Tesar, P. J. Drug screening for human genetic diseases using iPSC models. Human Molecular Genetics. 27 (R2), 89-98 (2018).
- Vuidel, A., et al. High-content phenotyping of Parkinson's disease patient stem cell-derived midbrain dopaminergic neurons using machine learning classification. Stem Cell Reports. 17 (10), 2349-2364 (2022).
- Stirling, D. R., Swain-Bowden, M. J., Lucas, A. M., Carpenter, A. E., Cimini, B. A., Goodman, A. CellProfiler 4: improvements in speed, utility and usability. BMC Bioinformatics. 22 (1), 433 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sofroniew, N., et al. . napari: a multi-dimensional image viewer for Python. , (2022).
- Berthold, M. R., et al. KNIME: The Konstanz Information Miner. Data Analysis, Machine Learning and Applications. , 319-326 (2008).
- Fathi, A., et al. Diverging Parkinson's Disease Pathology between patient-derived GBAN370S, LRRK2G2019S and engineered SNCAA53T iPSC-derived Dopaminergic Neurons. bioRxiv. , (2023).
- Wang, Y., Huang, H., Rudin, C., Shaposhnik, Y. Understanding How Dimension Reduction Tools Work: An Empirical Approach to Deciphering t-SNE, UMAP, TriMap, and PaCMAP for Data Visualization. Journal of Machine Learning Research. 22 (201), 1-73 (2021).
- Ke, G., et al. LightGBM: A highly efficient gradient boosting decision tree. Advances in Neural Information Processing Systems. 30, (2017).
- Avazzadeh, S., Baena, J. M., Keighron, C., Feller-Sanchez, Y., Quinlan, L. R. Modelling Parkinson's Disease: iPSCs towards Better Understanding of Human Pathology. Brain Sciences. 11 (3), 373 (2021).
- Sánchez-Danés, A., et al. Disease-specific phenotypes in dopamine neurons from human iPS-based models of genetic and sporadic Parkinson's disease. EMBO Molecular Medicine. 4 (5), 380-395 (2012).
- Oosterveen, T., et al. Pluripotent stem cell derived dopaminergic subpopulations model the selective neuron degeneration in Parkinson's disease. Stem Cell Reports. 16 (11), 2718-2735 (2021).
- Hughes, R. E., et al. Multiparametric high-content cell painting identifies copper ionophores as selective modulators of esophageal cancer phenotypes. ACS Chemical Biology. 17 (7), 1876-1889 (2022).
- Akbarzadeh, M., et al. Morphological profiling by means of the Cell Painting assay enables identification of tubulin-targeting compounds. Cell Chemical Biology. 29 (6), 1053-1064 (2022).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Way, G. P., et al. Morphology and gene expression profiling provide complementary information for mapping cell state. Cell Systems. 13 (11), 911-923 (2022).
- Feng, Y., Mitchison, T. J., Bender, A., Young, D. W., Tallarico, J. A. Multi-parameter phenotypic profiling: using cellular effects to characterize small-molecule compounds. Nature Reviews Drug Discovery. 8 (7), 567-578 (2009).
- Antonov, S. A., Novosadova, E. V. Current state-of-the-art and unresolved problems in using human induced pluripotent stem cell-derived dopamine neurons for parkinson's disease drug development. International Journal of Molecular Sciences. 22 (7), 3381 (2021).
- Miller, J. D., et al. Human iPSC-based modeling of late-onset disease via progerin-induced aging. Cell Stem Cell. 13 (6), 691-705 (2013).
- Bezard, E., Gross, C. E., Brotchie, J. M. Presymptomatic compensation in Parkinson's disease is not dopamine-mediated. Trends in Neurosciences. 26 (4), 215-221 (2003).
- Wu, Y., Le, W., Jankovic, J. Preclinical Biomarkers of parkinson disease. Archives of Neurology. 68 (1), 22-30 (2011).
- Verstraelen, P., et al. Systematic quantification of synapses in primary neuronal culture. iScience. 23 (9), 101542 (2020).
- Liu-Yesucevitz, L., et al. ALS-Linked mutations enlarge TDP-43-enriched neuronal RNA granules in the dendritic arbor. The Journal of Neuroscience. 34 (12), 4167-4174 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten