È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Profilo fenotipico dei neuroni dopaminergici del mesencefalo derivati da cellule staminali umane
In questo articolo
Riepilogo
Questo protocollo descrive la coltura cellulare di neuroni dopaminergici del mesencefalo umano, seguita da colorazione immunologica e generazione di profili fenotipici neuronali da immagini microscopiche acquisite ad alto contenuto che consentono l'identificazione di variazioni fenotipiche dovute a modulazioni genetiche o chimiche.
Abstract
Il morbo di Parkinson (PD) è legato a una serie di processi biologici cellulari che causano la perdita di neuroni dopaminergici del mesencefalo (mDA). Molti attuali modelli cellulari di PD in vitro mancano di complessità e non tengono conto di fenotipi multipli. Il profilo fenotipico nei neuroni mDA derivati da cellule staminali pluripotenti indotte umane (iPSC) può affrontare queste carenze misurando simultaneamente una serie di fenotipi neuronali in un tipo di cellula rilevante per la PD in parallelo. In questo articolo, descriviamo un protocollo per ottenere e analizzare i profili fenotipici da neuroni mDA umani disponibili in commercio. Un pannello di colorazione fluorescente specifico per i neuroni viene utilizzato per visualizzare i fenotipi correlati alla proteina nucleare, alla α-sinucleina, alla tirosina idrossilasi (TH) e alla proteina 2 associata ai microtubuli (MAP2). Il protocollo di profilazione fenotipica descritto è scalabile in quanto utilizza piastre a 384 pozzetti, manipolazione automatica dei liquidi e microscopia ad alto rendimento. L'utilità del protocollo è esemplificata utilizzando neuroni mDA donatori sani e neuroni mDA portatori della mutazione G2019S legata al PD nel gene della chinasi ripetuta 2 ricca di leucina (LRRK2). Entrambe le linee cellulari sono state trattate con l'inibitore della chinasi LRRK2 PFE-360 e sono stati misurati i cambiamenti fenotipici. Inoltre, dimostriamo come i profili fenotipici multidimensionali possano essere analizzati utilizzando metodi di classificazione supervisionati basati sul clustering o sull'apprendimento automatico. Il protocollo descritto interesserà in particolare i ricercatori che lavorano sulla modellazione delle malattie neuronali o che studiano gli effetti dei composti chimici nei neuroni umani.
Introduzione
Una varietà di processi biologici cellulari sono disturbati nella malattia di Parkinson (PD). Ad esempio, la disfunzione mitocondriale, lo stress ossidativo, i difetti di degradazione delle proteine, l'interruzione del traffico vescicolare e la funzione endolisosomiale sono stati associati alla perdita di neuroni dopaminergici del mesencefalo (mDA), sono comunemente osservati nella PD1. Pertanto, la malattia di Parkinson sembra coinvolgere molteplici meccanismi patologici che possono interagire e peggiorarsi a vicenda. Un modo utile per studiare questa interazione meccanicistica è la creazione di un'impronta digitale fenotipica completa o di un profilo dei neuroni dopaminergici del mesencefalo (mDA).
La profilazione fenotipica è un approccio che prevede la creazione di un profilo di un campione basato su una raccolta di caratteristiche misurabili e, in secondo luogo, comporta la formulazione di previsioni su un campione basato su questo profilo 2,3. L'obiettivo della profilazione è quello di acquisire una vasta gamma di caratteristiche, alcune delle quali potrebbero non essere state precedentemente associate a una malattia o a un trattamento3. Di conseguenza, la profilazione può rivelare processi biologici inaspettati. La profilazione fenotipica si basa tipicamente su cellule colorate con fluorescenza e saggi standardizzati, come il Cell Painting, sono stati sviluppati per creare profili fenotipici4. Recentemente, la profilazione fenotipica è stata, ad esempio, applicata per la caratterizzazione di piccole molecole o per la predizione accurata di sottotipi di PD basati esclusivamente su fibroblasti derivati da pazienti 5,6. Nonostante questi progressi, il profilo fenotipico è stato raramente applicato a cellule differenziate post-mitotiche, come i neuroni mDA derivati da cellule staminali pluripotenti indotte umane (iPSC) che esprimono mutazioni legate al PD come LRRK2 G2019S. Le sfide significative dei modelli derivati da iPSC includono la presenza di caratteristiche patologiche sottili o variabili tra i lotti di differenziazione o i genotipi e il fatto che i fenotipi isolati di PD non catturano l'intera complessità della malattia. Inoltre, mentre i modelli neuronali di iPSC sono fisiologicamente rilevanti, sono raramente utilizzati nei processi di scoperta di farmaci per la malattia di Parkinson a causa di preoccupazioni sulla complessità tecnica 7,8.
In precedenza abbiamo sviluppato una solida metodologia per misurare più fenotipi fisiopatologici correlati al PD nei neuroni mDA umani che sono sensibili ai cambiamenti fenotipici indotti da composti genetici e chimici9. Questo articolo descrive in dettaglio una versione ulteriormente ottimizzata di questa metodologia per creare profili fenotipici da neuroni mDA (Figura 1). Questo protocollo presenta diversi vantaggi rispetto agli approcci di profilazione fenotipica descritti in precedenza, come l'uso di neuroni mDA di alta qualità e la riproducibilità tecnica. Per la prima volta, questo protocollo consente la profilazione fenotipica nei neuroni mDA post-mitotici fisiologicamente rilevanti dopo perturbazioni chimiche in modo altamente scalabile. I neuroni mDA completamente differenziati e crioconservati sono disponibili in commercio, riducendo significativamente la variabilità della differenziazione da lotto a lotto. In secondo luogo, la variabilità tecnica può essere ulteriormente ridotta utilizzando un disegno sperimentale ben definito (ad esempio, la durata della coltura o evitando i pozzetti di bordo), la manipolazione automatizzata dei liquidi e la microscopia automatizzata. Inoltre, le fasi iniziali dell'analisi del profilo fenotipico utilizzando approcci di clustering non supervisionato o classificazione supervisionata sono descritte qui, indicando come possono essere analizzati i dati di profilazione fenotipica. Questo protocollo sarà utile per i ricercatori interessati ai cambiamenti fenotipici dei neuroni mDA indotti da perturbazioni genetiche o chimiche, in particolare quando è richiesta una configurazione di studio altamente scalabile, ad esempio durante le campagne di screening o quando gli effetti di un numero minore di composti devono essere studiati, ad esempio, per determinare gli effetti tossici. In sintesi, si prevede che l'applicazione del profilo fenotipico dei neuroni umani sia una tecnica preziosa per studiare fenotipi complessi correlati alla malattia e caratterizzare gli effetti cellulari dei farmaci candidati.
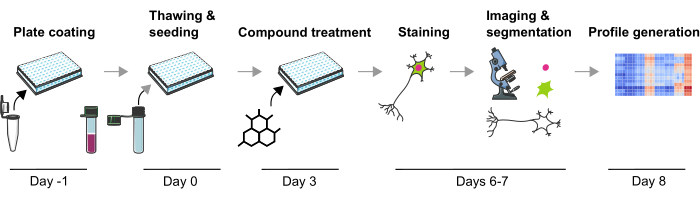
Figura 1: Rappresentazione schematica del protocollo sperimentale per generare profili fenotipici basati su immagini da neuroni mDA umani derivati da iPSC. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocollo
1. Preparazione del terreno e delle piastre per la semina dei neuroni (Giorno 1)
- Per preparare le piastre per la semina dei neuroni il giorno 1, riscaldare la laminina a temperatura ambiente (RT) appena prima dell'uso. Preparare la soluzione di Laminina diluendo la soluzione madre di Laminina (0,1 mg/mL) 1/10 in PBS+/+ freddo (con Ca 2+ e Mg2+).
NOTA: Tutti i reagenti sono elencati nell'Indice dei materiali. Le composizioni delle soluzioni e dei tamponi sono descritte nelle Tabelle 1-4. - Quindi, aggiungere 25 μL della soluzione di Laminina a ciascun pozzetto di una piastra a 384 pozzetti prerivestita di poli-D-lisina (PDL) e incubare per una notte a 4 °C. Le lastre rivestite possono essere conservate a 4 °C per un massimo di una settimana.
NOTA: Il protocollo può essere sospeso qui per un massimo di una settimana. Sigillare le piastre con pellicola di plastica. - Preparare i terreni di manutenzione completi e conservarli a 4 °C per un massimo di un mese (Tabella 1).
2. Scongelamento dei neuroni (Giorno 0)
- Per scongelare i neuroni il giorno 0, preriscaldare un bagno d'acqua a 37 °C e bilanciare il mezzo di mantenimento completo a RT, al riparo dalla luce.
- Estrarre la fiala contenente i neuroni congelati ottenuti in commercio (vedere Tabella dei materiali) dal serbatoio dell'azoto liquido e posizionarla su ghiaccio secco. Quindi posizionare il flaconcino a bagnomaria per 2 minuti. Una volta che il liquido è completamente scongelato, disinfettare il flaconcino con etanolo al 70%.
- Aspirare i neuroni scongelati con una pipetta P1000 (circa 370 μL) e trasferirli in una provetta da centrifuga da 50 mL senza pipettaggio su e giù. Successivamente, sciacquare il flaconcino con 630 μL di Medium di Mantenimento Completo, erogare goccia a goccia (angolo di 45°) nella provetta da 50 mL. Agitare lentamente durante l'erogazione.
NOTA: È fondamentale erogare il mezzo lentamente, goccia a goccia, per evitare la rottura osmotica delle cellule. L'angolo di 45° e la leggera agitazione riducono al minimo l'elevata pressione osmotica locale durante il pipettaggio. - Allo stesso modo, aggiungere 1 mL di terreno di mantenimento completo nella provetta da centrifuga da 50 mL con una pipetta P1000. Quindi, aggiungere lentamente 2 ml di terreno di mantenimento completo. Agitare con cura durante l'erogazione.
- Conta le celle. Preparare una microprovetta con 10 μL di Trypan Blue e 10 μL di sospensione cellulare e aggiungerla a un vetrino della camera di conteggio (10 μL). Eseguire il conteggio utilizzando un contatore di cellule automatizzato (vedere Tabella dei materiali) o manualmente.
- Dopo il conteggio, centrifugare la provetta da 50 mL contenente i neuroni a 400 x g per 5 minuti a RT e rimuovere il surnatante. Risospendere con cautela il pellet utilizzando una pipetta P1000 e 1 mL di Medium di Manutenzione Completa. Quindi aggiungere il volume necessario per raggiungere la concentrazione desiderata (300.000 cellule/mL, vedere anche il passaggio successivo).
3. Semina dei neuroni su piastre preparate (Giorno 0)
- Per seminare i neuroni sulle piastre preparate il giorno 0, estrarre le piastre rivestite dal frigorifero, posizionarle sotto la cappa di coltura cellulare e lasciarle equilibrare a RT per circa 30 minuti.
- Poco prima della semina, aspirare la soluzione di rivestimento da 15 μL con un manipolatore di liquidi automatizzato o una pipetta a 16 canali. Lasciare circa 10 μL per pozzetto per evitare danni al rivestimento.
- Successivamente, erogare 50 μL della soluzione cellulare contenente 300.000 cellule/mL (preparata nella fase 2.6) per pozzetto con una pipetta a 16 canali, ottenendo 15.000 neuroni seminati per pozzetto e un volume finale per pozzetto di 60 μL.
- In una piastra a 384 pozzetti, evitare di utilizzare le colonne 1, 2, 23, 24 e le righe A, B, O e P per ridurre al minimo i possibili effetti sui bordi che possono influire sui profili fenotipici. Riempire i pozzetti vuoti inutilizzati con 80 μL di PBS. Incubare le piastre a 37 °C e 5% di CO2 .
4. Cambio medio o trattamento composto (giorno 3)
- A seconda del numero di pozzetti, preriscaldare un volume appropriato di Complete Maintenance Medium a RT. Proteggere dalla luce.
- Se è necessario cambiare il fluido, procedere al punto 4.4. Se si desidera un trattamento del composto, verificare la concentrazione del composto e il solvente utilizzato (acqua, DMSO, metanolo, ecc.).
- Preparare una soluzione composta concentrata 1,5x per tutte le concentrazioni desiderate da testare. Preparare le diluizioni del composto utilizzando il mezzo di manutenzione completo.
NOTA: Come controllo neutro, utilizzare il rispettivo solvente alla stessa concentrazione del composto testato. Se si utilizzano composti multipli o sconosciuti a concentrazioni diverse, è consigliabile eseguire un esperimento dose-risposta separato per misurare gli effetti del solvente sul profilo fenotipico. - Se si utilizza un sistema di pipettaggio automatico, aggiungere 60 μL della soluzione composta 1,5x a una piastra di conservazione da 384 pozzetti. In alternativa, utilizzare una pipetta a 16 canali. Se è necessario sostituire il fluido, aggiungere invece 60 μL di mezzo di manutenzione completo.
- Utilizzando il sistema di pipettaggio automatico, aspirare ed eliminare 40 μL di terreno per pozzetto dalla piastra contenente neuroni per mantenere 20 μL/pozzetto. Quindi aggiungere 40 μL/pozzetto di soluzione composta 1,5x dalla piastra di conservazione a 384 pozzetti a ciascun pozzetto per ottenere la concentrazione finale desiderata.
NOTA: È fondamentale non eseguire cambi di terreno completi, ma lasciare sempre il terreno residuo nel pozzetto per evitare danni al tappeto neuronale o al rivestimento. - Nel caso in cui la coltura neuronale venga eseguita per più dei 6 giorni descritti in questo protocollo, cambiare il terreno ogni 2-3 giorni.
5. Fissazione e colorazione dei neuroni (giorni da 6 a 7)
- Per fissare e colorare i neuroni nei giorni 6 e 7, utilizzare un manipolatore di liquidi automatizzato per tutte le fasi di erogazione e lavaggio. In alternativa, utilizzare una pipetta a 16 canali.
- Preparare una soluzione di Triton X-100 al 10% diluendo Triton X-100 in 1x PBS. Vorticare fino a quando la soluzione non è omogenea. Conservare a 4 °C.
- Per fissare i neuroni, erogare 20 μL/pozzetto di PFA al 16% ottenendo una concentrazione finale del 4%. Incubare la piastra per 30 minuti a RT e lavarla tre volte con 1x PBS. Lasciare 20 μL/pozzetto di PBS dopo l'ultimo lavaggio.
ATTENZIONE: Il PFA è riconosciuto come una sostanza pericolosa nota per causare tossicità orale, cutanea e respiratoria. Rappresenta anche una minaccia per gli occhi e può portare a mutazioni genetiche e cancro. La corretta manipolazione del PFA richiede l'uso di adeguati dispositivi di protezione individuale, come la protezione degli occhi e del viso, oltre a garantire un'adeguata ventilazione. È importante prevenire il rilascio di PFA nell'ambiente. - Per la permeabilizzazione e il blocco, preparare una soluzione bloccante 2x (Tabella 2).
- Aggiungere 20 μL/pozzetto di soluzione bloccante 2x (concentrazione finale 1x), incubare per 1 ora a RT e lavare una volta con PBS. Conservare 20 μL/pozzetto di PBS dopo il lavaggio.
- Per la colorazione con anticorpi primari (vedere la tabella dei materiali), preparare un tampone di colorazione primario 2x (Tabella 3).
- Aggiungere 20 μL/pozzetto di 2 tamponi di colorazione primaria (1 concentrazione finale) e incubare per una notte a 4 °C. La mattina dopo, il giorno 7, lavare tre volte con PBS. Lasciare 20 μL/pozzetto di PBS dopo il lavaggio.
- Per la colorazione con anticorpi secondari (vedere la tabella dei materiali), preparare un tampone di colorazione secondario 2x (Tabella 4).
- Aggiungere 20 μL/pozzetto di tampone di colorazione secondario 2x (1x concentrazione finale), incubare per 2 ore a RT al riparo dalla luce e lavare tre volte con PBS. Lasciare 100 μL di PBS/pozzetto dopo l'ultimo lavaggio.
- Aggiungere una guarnizione in alluminio sulla piastra per ridurre al minimo l'evaporazione. In alternativa, coprire la piastra con pellicola di plastica e foglio di alluminio. Procedere all'acquisizione dell'immagine o conservare la lastra a 4 °C.
NOTA: Il protocollo può essere sospeso qui per un massimo di una settimana. Se si osserva una fluorescenza di fondo, prendere in considerazione l'aumento del tempo di blocco. Se la colorazione è insufficiente, provare ad aumentare la concentrazione di anticorpi primari o il tempo di incubazione.
- Aggiungere una guarnizione in alluminio sulla piastra per ridurre al minimo l'evaporazione. In alternativa, coprire la piastra con pellicola di plastica e foglio di alluminio. Procedere all'acquisizione dell'immagine o conservare la lastra a 4 °C.
6. Imaging di neuroni colorati con fluorescenza (Giorno 7)
- Acquisisci immagini dei neuroni piastre, coltivati e colorati il giorno 7. L'ideale sarebbe utilizzare un microscopio a fluorescenza confocale automatizzato (vedere la tabella dei materiali). In alternativa, è possibile acquisire le immagini manualmente.
- Acquisire i canali Hoechst, TH, α-sinucleina e MAP2 utilizzando rispettivamente laser da 405 nm, 488 nm, 561 nm e 647 nm (Figura 2).
NOTA: Per generare una quantità sufficiente di dati dettagliati per la profilazione fenotipica, utilizzare un obiettivo 40x e acquisire 16 campi/pozzetto utilizzando Z-stack costituiti da 3 fette Z separate da 2 μm. Nel caso in cui ci siano danni legati al pipettaggio nel tappeto neuronale, cercare di evitare l'imaging di queste aree. - A seconda del microscopio e della fotocamera, regolare separatamente i tempi di esposizione e le intensità di eccitazione per ciascuno dei quattro canali fluorescenti per ottenere una gamma dinamica ottimale delle intensità fluorescenti.
NOTA: Il software di imaging spesso fornisce un istogramma per determinare il tempo di esposizione ideale. Se l'istogramma è spostato troppo a sinistra nella gamma del segnale basso, il tempo di esposizione è troppo breve o l'intensità di eccitazione è troppo bassa. Se c'è un forte precipizio al livello massimo del segnale a destra, il valore del segnale è saturo. In questo caso, ridurre l'intensità dell'eccitazione o accorciare il tempo di esposizione. - Memorizza le immagini in un formato aperto e privo di perdite, ad esempio .tif.
7. Elaborazione delle immagini (giorno 8)
- La segmentazione dell'immagine e l'estrazione delle caratteristiche fenotipiche sono necessarie per la creazione di profili fenotipici quantitativi. Utilizzare il software PhenoLink per la segmentazione delle immagini e l'estrazione delle caratteristiche (Tabella dei materiali). Le istruzioni per l'installazione di PhenoLink sono disponibili nel repository GitHub (https://github.com/Ksilink/PhenoLink).
NOTA: La segmentazione delle immagini viene utilizzata per identificare e separare diversi oggetti o regioni in un'immagine, mentre l'estrazione di caratteristiche fenotipiche viene utilizzata per analizzare ed estrarre informazioni rilevanti da tali regioni. Sono disponibili diverse soluzioni software alternative, come CellProfiler 10, ImageJ/FIJI11, Napari 12 o Knime Analytics Platform13 per estrarre informazioni quantitative da immagini a fluorescenza multicanale. - Eseguite la segmentazione dell'immagine su immagini raw con correzione dell'illuminazione. Determinare empiricamente le rispettive soglie di intensità del canale fluorescente per lastra in modo che il segnale di fondo sia minimo e il segnale segmentato desiderato corrisponda al segnale nell'immagine grezza. Le lastre elaborate e colorate nello stesso giorno richiedono in genere soglie di intensità del canale comparabili per la segmentazione.
- Definisci la dimensione e l'intensità del nucleo per separare le cellule vive da quelle morte. Quando si utilizzano immagini 40x, mantenere tutti gli altri parametri predefiniti ed eseguire il software. Saranno calcolate centoventisei caratteristiche quantitative dell'immagine per pozzetto (Tabella supplementare 1).
- Utilizzare i dati quantitativi tabulari risultanti per costruire profili fenotipici e confrontare i profili fenotipici di diverse linee cellulari o condizioni di trattamento. Ogni riga corrisponde a una condizione biologica (pozzo) e ogni colonna corrisponde a una determinata caratteristica fenotipica.
NOTA: Forniamo un file di output di esempio insieme alla pipeline di analisi dei dati per illustrarne l'utilizzo (vedi Tabella dei materiali). Inoltre, la Figura 3 mostra la composizione di un profilo fenotipico.
8. Generazione e visualizzazione del profilo fenotipico (Giorno 8)
- Se non hai installato Python e Jupyter sul tuo computer, installa la distribuzione Anaconda e apri il software Jupyter. Scaricare il notebook Jupyter fornito e tutti gli altri file forniti e salvarli nella stessa directory (vedere Sommario dei materiali). Aprire il file del notebook Jupyter usando il software Jupyter.
NOTA: Anaconda è una piattaforma gratuita e open-source per linguaggi di programmazione come Python. Questa piattaforma viene fornita con l'interprete Python Jupyter che può eseguire il notebook Jupyter fornito per creare e analizzare profili fenotipici (https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling). - Eseguire il notebook Jupyter cella per cella usando il software Jupyter. I file .fth dei dati di esempio forniti e i file .txt dei requisiti devono trovarsi nella stessa directory del notebook Jupyter. Ogni cella del notebook Jupyter viene annotata per spiegarne la funzionalità.
NOTA: è fondamentale utilizzare il notebook Jupyter nell'ordine corretto, a partire dal caricamento e dal ridimensionamento dei dati e procedere cella per cella fino alla fine. Tutti i dati e l'output grafico verranno archiviati in una cartella appena creata nella directory di origine del notebook Jupyter. Nella Figura 4 vengono illustrati il flusso di lavoro e l'output del flusso di lavoro.
Risultati
La profilazione fenotipica nei neuroni mDA è un modo efficiente per quantificare molteplici aspetti della biologia cellulare e i loro cambiamenti durante la modulazione sperimentale. Per esemplificare questa metodologia, questo studio ha fatto uso di neuroni LRRK2 G2019S crioconservati e mDA di donatori sani. Questi neuroni sono stati differenziati per circa 37 giorni, sono marcatori neuronali post-mitotici ed espressi (TUBB3 e MAP2) e marcatori neuronali dopaminergici, tra cui la tirosina idrossilasi (TH) in combinazio...
Discussione
La profilazione fenotipica è una tecnica per misurare un gran numero di fenotipi nelle cellule applicando colorazioni fluorescenti, microscopia e analisi delle immagini3. I profili fenotipici possono essere ottenuti e confrontati tra linee cellulari o altre condizioni sperimentali per comprendere i cambiamenti complessi nella biologia cellulare che potrebbero passare inosservati quando si utilizza una singola lettura. Qui descriviamo l'applicazione del profilo fenotipico ai neuroni mDA umani deri...
Divulgazioni
Tutti gli autori sono dipendenti di Ksilink.
Riconoscimenti
Gli autori desiderano ringraziare tutti i colleghi di Ksilink per il loro prezioso aiuto e le discussioni che hanno portato alla progettazione del protocollo presentato.
Materiali
| Name | Company | Catalog Number | Comments |
| Anti- chicken – Alexa 647 | Jackson ImmunoRearch | 703-605-155 | Immunofluorescence |
| Anaconda | https://www.anaconda.com/download | ||
| Anti-Map2 | Novus | NB300-213 | Immunofluorescence |
| Anti-mouse - Alexa 488 | Thermo Fisher | A11001 | Immunofluorescence |
| Anti-rabbit - Alexa 555 | Thermo Fisher | A21429 | Immunofluorescence |
| Anti-Tyrosine Hydroxylase | Merck | T2928 | Immunofluorescence |
| Anti-α-synuclein | Abcam | 138501 | Immunofluorescence |
| Bravo Automated Liquid Handling Platform with 384ST head | Agilent | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Confocal microscope | Yokogawa | CV7000 | The use of an automated confocal fluorescence microscope is recommended to ensure image quality consistency. |
| Countess Automated cell counter | Invitrogen | Cell counting before seeding. Can also be done using a manual counting chamber. | |
| DPBS +/+ | Gibco | 14040-133 | Buffer for washing |
| EL406 Washer Dispenser | BioTek (Agilent) | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Formaldehyde Solution (PFA 16 %) | Euromedex | EM-15710-S | Fixation before staining |
| Hoechst 33342 | Invitrogen | H3570 | Nuclear staining |
| iCell Base Medium 1 | Fujifilm | M1010 | Base medium for neurons |
| iCell DPN, Donor#01279, Phenotype AHN, lot#106339, 1M | Fujifilm | C1087 | Apparently healthy donor |
| iCell DPN, Donor#11299, Phenotype LRRK2 G2019S, phenotype PD lot#106139 | Fujifilm | C1149 | Donor carrying LRRK2 G2019S mutation |
| iCell Nervous System Supplement | Fujifilm | M1031 | Supplement for base medium |
| iCell Neural Supplement B | Fujifilm | M1029 | Supplement for base medium |
| Jupyter Python Notebook | In-house development | https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling | Notebook to perform phenotypic profile visualization and classification from raw data. |
| Laminin | Biolamina | LN521 | Plate coating |
| PFE-360 | MedChemExpress | HY-120085 | LRRK2 kinase inhibitor |
| PhenoLink | In-house development | https://github.com/Ksilink/PhenoLink | Software for image analysis |
| PhenoPlate 384w, PDL coated | Perkin Elmer | 6057500 | Pre-coated plate for cell culture and imaging. This plate allows imaging of all wells using all objectives of the Yokogawa CV7000 microscope. |
| Storage plates Abgene 120 µL | Thermo Scientific | AB-0781 | Necessary for compound dispensing using the Vprep pipetting system. If not available, the use of an electronic multichannel pipette is recommended. |
| Triton | Sigma | T9284 | Permeabilization before lysis |
| Trypan Blue | Sigma | T8154-20ML | Determination of living cells |
| Vprep Pipetting System | Agilent | Medium change and compound dispensing. Alternatively, an electronic multichannel pipette can be used. |
Riferimenti
- Panicker, N., Ge, P., Dawson, V. L., Dawson, T. M. The cell biology of Parkinson's disease. The Journal of Cell Biology. 220 (4), 202012095 (2021).
- Caicedo, J. C., et al. Data-analysis strategies for image-based cell profiling. Nature Methods. 14 (9), 849-863 (2017).
- Chandrasekaran, S. N., Ceulemans, H., Boyd, J. D., Carpenter, A. E. Image-based profiling for drug discovery: due for a machine-learning upgrade. Nature Reviews Drug Discovery. 20 (2), 145-159 (2021).
- Bray, M. -. A., et al. Cell Painting, a high-content image-based assay for morphological profiling using multiplexed fluorescent dyes. Nature Protocols. 11 (9), 1757-1774 (2016).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Ziegler, S., Sievers, S., Waldmann, H. Morphological profiling of small molecules. Cell Chemical Biology. 28 (3), 300-319 (2021).
- Cobb, M. M., Ravisankar, A., Skibinski, G., Finkbeiner, S. iPS cells in the study of PD molecular pathogenesis. Cell and Tissue Research. 373 (1), 61-77 (2018).
- Elitt, M. S., Barbar, L., Tesar, P. J. Drug screening for human genetic diseases using iPSC models. Human Molecular Genetics. 27 (R2), 89-98 (2018).
- Vuidel, A., et al. High-content phenotyping of Parkinson's disease patient stem cell-derived midbrain dopaminergic neurons using machine learning classification. Stem Cell Reports. 17 (10), 2349-2364 (2022).
- Stirling, D. R., Swain-Bowden, M. J., Lucas, A. M., Carpenter, A. E., Cimini, B. A., Goodman, A. CellProfiler 4: improvements in speed, utility and usability. BMC Bioinformatics. 22 (1), 433 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sofroniew, N., et al. . napari: a multi-dimensional image viewer for Python. , (2022).
- Berthold, M. R., et al. KNIME: The Konstanz Information Miner. Data Analysis, Machine Learning and Applications. , 319-326 (2008).
- Fathi, A., et al. Diverging Parkinson's Disease Pathology between patient-derived GBAN370S, LRRK2G2019S and engineered SNCAA53T iPSC-derived Dopaminergic Neurons. bioRxiv. , (2023).
- Wang, Y., Huang, H., Rudin, C., Shaposhnik, Y. Understanding How Dimension Reduction Tools Work: An Empirical Approach to Deciphering t-SNE, UMAP, TriMap, and PaCMAP for Data Visualization. Journal of Machine Learning Research. 22 (201), 1-73 (2021).
- Ke, G., et al. LightGBM: A highly efficient gradient boosting decision tree. Advances in Neural Information Processing Systems. 30, (2017).
- Avazzadeh, S., Baena, J. M., Keighron, C., Feller-Sanchez, Y., Quinlan, L. R. Modelling Parkinson's Disease: iPSCs towards Better Understanding of Human Pathology. Brain Sciences. 11 (3), 373 (2021).
- Sánchez-Danés, A., et al. Disease-specific phenotypes in dopamine neurons from human iPS-based models of genetic and sporadic Parkinson's disease. EMBO Molecular Medicine. 4 (5), 380-395 (2012).
- Oosterveen, T., et al. Pluripotent stem cell derived dopaminergic subpopulations model the selective neuron degeneration in Parkinson's disease. Stem Cell Reports. 16 (11), 2718-2735 (2021).
- Hughes, R. E., et al. Multiparametric high-content cell painting identifies copper ionophores as selective modulators of esophageal cancer phenotypes. ACS Chemical Biology. 17 (7), 1876-1889 (2022).
- Akbarzadeh, M., et al. Morphological profiling by means of the Cell Painting assay enables identification of tubulin-targeting compounds. Cell Chemical Biology. 29 (6), 1053-1064 (2022).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Way, G. P., et al. Morphology and gene expression profiling provide complementary information for mapping cell state. Cell Systems. 13 (11), 911-923 (2022).
- Feng, Y., Mitchison, T. J., Bender, A., Young, D. W., Tallarico, J. A. Multi-parameter phenotypic profiling: using cellular effects to characterize small-molecule compounds. Nature Reviews Drug Discovery. 8 (7), 567-578 (2009).
- Antonov, S. A., Novosadova, E. V. Current state-of-the-art and unresolved problems in using human induced pluripotent stem cell-derived dopamine neurons for parkinson's disease drug development. International Journal of Molecular Sciences. 22 (7), 3381 (2021).
- Miller, J. D., et al. Human iPSC-based modeling of late-onset disease via progerin-induced aging. Cell Stem Cell. 13 (6), 691-705 (2013).
- Bezard, E., Gross, C. E., Brotchie, J. M. Presymptomatic compensation in Parkinson's disease is not dopamine-mediated. Trends in Neurosciences. 26 (4), 215-221 (2003).
- Wu, Y., Le, W., Jankovic, J. Preclinical Biomarkers of parkinson disease. Archives of Neurology. 68 (1), 22-30 (2011).
- Verstraelen, P., et al. Systematic quantification of synapses in primary neuronal culture. iScience. 23 (9), 101542 (2020).
- Liu-Yesucevitz, L., et al. ALS-Linked mutations enlarge TDP-43-enriched neuronal RNA granules in the dendritic arbor. The Journal of Neuroscience. 34 (12), 4167-4174 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon