É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Perfil Fenotípico de Neurônios Dopaminérgicos do Mesencéfalo Derivados de Células-Tronco Humanas
Neste Artigo
Resumo
Este protocolo descreve a cultura celular de neurônios dopaminérgicos do mesencéfalo humano, seguida de coloração imunológica e geração de perfis fenotípicos neuronais a partir de imagens microscópicas adquiridas de alto conteúdo, permitindo a identificação de variações fenotípicas devido a modulações genéticas ou químicas.
Resumo
A doença de Parkinson (DP) está ligada a uma série de processos biológicos celulares que causam perda de neurônios dopaminérgicos do mesencéfalo (mDA). Muitos modelos celulares atuais de DP in vitro carecem de complexidade e não levam em conta múltiplos fenótipos. O perfil fenotípico em neurônios mDA derivados de células-tronco pluripotentes induzidas humanas (iPSC) pode abordar essas deficiências medindo simultaneamente uma variedade de fenótipos neuronais em um tipo de célula relevante para DP em paralelo. Aqui, descrevemos um protocolo para obter e analisar perfis fenotípicos de neurônios mDA humanos comercialmente disponíveis. Um painel de coloração fluorescente neuron-específico é usado para visualizar os fenótipos nucleares, α-sinucleína, tirosina hidroxilase (TH) e proteína associada a microtúbulos 2 (MAP2) relacionados. O protocolo de perfil fenotípico descrito é escalável, pois utiliza placas de 384 poços, manuseio automático de líquidos e microscopia de alto rendimento. A utilidade do protocolo é exemplificada usando neurônios mDA de doadores saudáveis e neurônios mDA portadores da mutação G2019S ligada a PD no gene Leucine-rich repeat kinase 2 (LRRK2). Ambas as linhagens celulares foram tratadas com o inibidor de quinase LRRK2 PFE-360 e as alterações fenotípicas foram medidas. Além disso, demonstramos como perfis fenotípicos multidimensionais podem ser analisados usando métodos de classificação supervisionada orientados por clustering ou aprendizado de máquina. O protocolo descrito interessará particularmente aos pesquisadores que trabalham na modelagem de doenças neuronais ou no estudo dos efeitos de compostos químicos em neurônios humanos.
Introdução
Uma variedade de processos biológicos celulares são perturbados na doença de Parkinson (DP). Por exemplo, disfunção mitocondrial, estresse oxidativo, defeitos de degradação proteica, interrupção do tráfego vesicular e função endolisossômica têm sido associados à perda de neurônios dopaminérgicos mesencefálicos (mDA), são comumente observados na DP1. Portanto, a DP parece envolver múltiplos mecanismos patológicos que podem interagir e piorar uns com os outros. Uma maneira útil de investigar essa interação mecanicista é a criação de uma impressão digital fenotípica abrangente ou perfil de neurônios dopaminérgicos mesencefálicos (mDA).
O perfil fenotípico é uma abordagem que envolve a criação de um perfil de uma amostra com base em uma coleção de características mensuráveis e, em segundo lugar, envolve fazer previsões sobre uma amostra com base nesse perfil 2,3. O objetivo da criação de perfis é capturar uma gama diversificada de características, algumas das quais podem não ter sido previamente associadas a uma doença ou tratamento3. Como resultado, o perfilamento pode revelar processos biológicos inesperados. O perfil fenotípico tipicamente depende de células coradas fluorescentemente, e ensaios padronizados, como o Cell Painting, foram desenvolvidos para criar perfis fenotípicos4. Recentemente, o perfil fenotípico tem sido aplicado, por exemplo, para a caracterização de pequenas moléculas ou a predição precisa de subtipos de DP apenas com base em fibroblastos derivados do paciente 5,6. Apesar desses avanços, o perfil fenotípico raramente tem sido aplicado a células diferenciadas pós-mitóticas, como neurônios mDA derivados de células-tronco pluripotentes induzidas por humanos (iPSC) que expressam mutações ligadas à DP, como LRRK2 G2019S. Desafios significativos dos modelos derivados de iPSC incluem a presença de características patológicas sutis ou variáveis entre lotes de diferenciação ou genótipos, e o fato de que fenótipos isolados de DP não capturam toda a complexidade da doença. Além disso, embora os modelos neuronais de iPSC sejam fisiologicamente relevantes, eles raramente são usados em processos de descoberta de drogas para DP devido a preocupações com a complexidade técnica 7,8.
Desenvolvemos previamente uma metodologia robusta para medir múltiplos fenótipos fisiopatológicos relacionados à DP em neurônios humanos mDA que são sensíveis a alterações fenotípicas genéticas e químicas induzidas por compostosquímicos9. Este artigo descreve em detalhes uma versão otimizada dessa metodologia para criar perfis fenotípicos a partir de neurônios mDA (Figura 1). Este protocolo apresenta várias vantagens em relação às abordagens de perfil fenotípico descritas anteriormente, tais como o uso de neurônios mDA de alta qualidade e reprodutibilidade técnica. Pela primeira vez, este protocolo permite o perfil fenotípico em neurônios mDA pós-mitóticos fisiologicamente relevantes após perturbações químicas de forma altamente escalável. Neurônios mDA totalmente diferenciados e criopreservados estão comercialmente disponíveis, diminuindo significativamente a variabilidade da diferenciação lote a lote. Em segundo lugar, a variabilidade técnica pode ser ainda mais reduzida usando um desenho experimental bem definido (isto é, duração da cultura ou evitando poços de borda), manuseio automatizado de líquidos e microscopia automatizada. Adicionalmente, as etapas iniciais da análise do perfil fenotípico usando abordagens de agrupamento não supervisionado ou classificação supervisionada são descritas aqui, indicando como os dados de perfil fenotípico podem ser analisados. Este protocolo será útil para pesquisadores interessados em alterações fenotípicas de neurônios mDA induzidas por perturbações genéticas ou químicas, especificamente quando uma configuração de estudo altamente escalável é necessária, por exemplo, durante campanhas de triagem ou quando os efeitos de um número menor de compostos devem ser estudados, por exemplo, para determinar efeitos tóxicos. Em resumo, espera-se que a aplicação do perfil fenotípico de neurônios humanos seja uma técnica valiosa para estudar fenótipos complexos relacionados à doença e caracterizar os efeitos celulares de candidatos a fármacos.
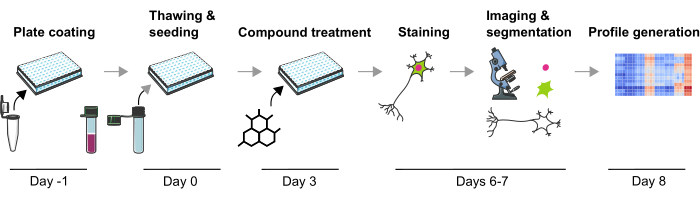
Figura 1: Representação esquemática do protocolo experimental para gerar perfis fenotípicos baseados em imagens a partir de neurônios mDA humanos derivados de iPSC. Clique aqui para ver uma versão maior desta figura.
Protocolo
1. Preparação do meio e placas para semeadura de neurônios (Dia 1)
- Para preparar as placas para semeadura de neurônios no Dia-1, aqueça a Laminina à temperatura ambiente (TR) imediatamente antes do uso. Preparar a solução de laminina diluindo a solução-mãe de laminina (0,1 mg/ml) 1/10 em PBS+/+ frio (com Ca 2+ e Mg2+).
NOTA: Todos os reagentes estão listados na Tabela de Materiais. As composições das soluções e dos tampões estão descritas nas Tabelas 1 a 4. - Em seguida, adicionar 25 μL da solução de laminina a cada poço de uma placa de 384 poços pré-revestida com poli-D-lisina (PDL) e incubar durante a noite a 4 °C. As placas revestidas podem ser armazenadas a 4 °C por até uma semana.
NOTA: O protocolo pode ser pausado aqui por até uma semana. Placas de vedação utilizando filme plástico. - Preparar Mídia de Manutenção Completa e armazenar a 4 °C por até um mês (Tabela 1).
2. Descongelamento de neurônios (Dia 0)
- Para descongelar os neurônios no Dia 0, pré-aqueça um banho-maria a 37 °C e equilibre o Meio de Manutenção Completo para RT, protegido da luz.
- Retire o frasco para injetáveis que contém os neurónios congelados obtidos comercialmente (ver Tabela de Materiais) do depósito de azoto líquido e coloque-o no gelo seco. Em seguida, coloque o frasco para injetáveis em banho-maria durante 2 minutos. Quando o líquido estiver completamente descongelado, desinfete o frasco para injetáveis com etanol a 70%.
- Aspirar os neurônios descongelados com uma pipeta P1000 (cerca de 370 μL) e transferi-los em um tubo centrífugo de 50 mL sem pipetagem ascendente e descendente. Em seguida, enxágue o frasco para injetáveis com 630 μL de Meio de Manutenção Completa, distribua gota a gota (ângulo de 45°) no tubo de 50 mL. Agitar lentamente durante a dispensação.
OBS: É fundamental dispensar o meio lentamente, gota a gota, para evitar ruptura osmótica das células. Um ângulo de 45° e agitação leve minimizam a alta pressão osmótica local durante a pipetagem. - Da mesma forma, adicione 1 mL de Meio de Manutenção Completa no tubo centrífugo de 50 mL com uma pipeta P1000. Em seguida, adicione lentamente 2 mL de Meio de Manutenção Completa. Agite cuidadosamente durante a dispensação.
- Conte as células. Preparar um microtubo com 10 μL de Azul de Tripano e 10 μL de suspensão celular e adicionar a uma lâmina de câmara de contagem (10 μL). Execute a contagem usando um contador de células automatizado (consulte Tabela de Materiais) ou manualmente.
- Após a contagem, centrifugar o tubo de 50 mL contendo os neurônios a 400 x g por 5 min no TR e remover o sobrenadante. Ressuspenda cuidadosamente o pellet usando uma pipeta P1000 e 1 mL de Meio de Manutenção Completa. Em seguida, adicione o volume necessário para atingir a concentração desejada (300.000 células/mL, veja também a próxima etapa).
3. Semeadura de neurônios em placas preparadas (Dia 0)
- Para semear neurônios nas placas preparadas no Dia 0, tire as placas revestidas da geladeira, coloque-as sob a tampa da cultura celular e deixe-as se equilibrar para RT por cerca de 30 min.
- Imediatamente antes da semeadura, aspirar a solução de revestimento de 15 μL com um manipulador de líquidos automatizado ou uma pipeta de 16 canais. Deixe aproximadamente 10 μL por poço para evitar danos ao revestimento.
- Em seguida, dispensar 50 μL da solução celular contendo 300.000 células/mL (preparada na etapa 2.6) por poço com uma pipeta de 16 canais, resultando em 15.000 neurônios semeados por poço e um volume final por poço de 60 μL.
- Em uma placa de 384 poços, evite usar as colunas 1, 2, 23, 24 e as linhas A, B, O e P para minimizar possíveis efeitos de borda que podem afetar os perfis fenotípicos. Encher os poços vazios não utilizados com 80 μL de PBS. Incubar as placas a 37 °C e 5% CO2.
4. Troca de meio ou tratamento composto (Dia 3)
- Dependendo do número de poços, pré-aqueça um volume apropriado de Meio de Manutenção Completa no RT.
- Se for necessária uma alteração média, avance para o passo 4.4. Se o tratamento composto for desejado, verifique a concentração de estoque do composto e o solvente usado (água, DMSO, metanol, etc.).
- Preparar uma solução de composto concentrado de 1,5x para todas as concentrações desejadas a serem testadas. Preparar as diluições do composto utilizando Meio de Manutenção Completa.
NOTA: Como controle neutro, utilizar o respectivo solvente na mesma concentração do composto testado. Se forem utilizados compostos múltiplos ou desconhecidos em concentrações diferentes, é aconselhável realizar um experimento dose-resposta separado para medir os efeitos do solvente no perfil fenotípico. - Se for utilizado um sistema de pipetagem automática, adicione 60 μL da solução composta de 1,5x a uma placa de armazenamento de 384 poços. Como alternativa, use uma pipeta de 16 canais. Se for necessária uma mudança de meio, adicione 60 μL de Meio de Manutenção Completa.
- Usando o sistema de pipetagem automática, aspirar e descartar 40 μL de meio por poço da placa contendo o neurônio para manter 20 μL/poço. Em seguida, adicione 40 μL/poço de solução composta de 1,5x da placa de armazenamento de 384 poços a cada poço para obter a concentração final desejada.
OBS: É fundamental não realizar trocas completas de meio, mas sempre deixar meio residual no poço para evitar danos ao tapete neuronal ou ao revestimento. - Caso a cultura neuronal seja realizada por mais de 6 dias descritos neste protocolo, troque o meio a cada 2-3 dias.
5. Fixação e coloração dos neurônios (Dias 6 a 7)
- Para fixar e manchar os neurônios nos dias 6 e 7, faça uso de um manipulador de líquidos automatizado para todas as etapas de dispensação e lavagem. Como alternativa, use uma pipeta de 16 canais.
- Prepare uma solução de Triton X-100 a 10% diluindo Triton X-100 em 1x PBS. Vórtice até que a solução fique homogênea. Conservar a 4 °C.
- Para fixar os neurônios, dispensar 20 μL/poço de PFA a 16%, resultando em uma concentração final de 4%. Incubar a placa por 30 min em TR e lavá-la três vezes com 1x PBS. Deixar 20 μL/poço de PBS após a última lavagem.
CUIDADO: O PFA é reconhecido como uma substância perigosa conhecida por causar toxicidade oral, dérmica e respiratória. Também representa uma ameaça para os olhos e pode levar a mutações genéticas e câncer. O manuseio adequado do PFA requer o uso de equipamentos de proteção individual adequados, como proteção ocular e facial, além de garantir ventilação adequada. É importante evitar a liberação de PFA no meio ambiente. - Para permeabilização e bloqueio, preparar uma solução de bloqueio 2x (Tabela 2).
- Adicionar 20 μL/poço de solução bloqueadora 2x (concentração final 1x), incubar por 1 h no TR e lavar uma vez com PBS. Manter 20 μL/poço de PBS após a lavagem.
- Para a coloração de anticorpos primários (ver Tabela de Materiais), prepare um tampão de coloração primário de 2x (Tabela 3).
- Adicionar 20 μL/poço de 2x tampão de coloração primário (concentração final de 1x) e incubar durante a noite a 4 °C. Na manhã seguinte, no dia 7, lave três vezes com PBS. Deixar 20 μL/poço de PBS após a lavagem.
- Para coloração de anticorpos secundários (ver Tabela de Materiais), prepare um tampão de coloração secundário de 2x (Tabela 4).
- Adicionar 20 μL/poço de 2x tampão de coloração secundário (1x concentração final), incubar por 2 h em TR longe da luz e lavar três vezes com PBS. Deixar 100 μL PBS/poço após a última lavagem.
- Adicione vedação de alumínio na placa para minimizar a evaporação. Alternativamente, cubra a placa usando filme plástico e papel alumínio. Proceder à aquisição da imagem ou conservar a placa a 4 °C.
NOTA: O protocolo pode ser pausado aqui por até uma semana. Se a fluorescência de fundo for observada, considere aumentar o tempo de bloqueio. Se a coloração for insuficiente, tente aumentar a concentração de anticorpos primários ou o tempo de incubação.
- Adicione vedação de alumínio na placa para minimizar a evaporação. Alternativamente, cubra a placa usando filme plástico e papel alumínio. Proceder à aquisição da imagem ou conservar a placa a 4 °C.
6. Imagem de neurônios corados fluorescentemente (Dia 7)
- Adquira imagens dos neurônios plaqueados, cultivados e corados no dia 7. O ideal é usar um microscópio de fluorescência confocal automatizado (ver Tabela de Materiais). Como alternativa, adquira imagens manualmente.
- Adquira os canais Hoechst, TH, α-sinucleína e MAP2 usando lasers de 405 nm, 488 nm, 561 nm e 647 nm, respectivamente (Figura 2).
NOTA: Para gerar uma quantidade suficiente de dados detalhados para o perfil fenotípico, use uma objetiva de 40x e adquira 16 campos/poço usando pilhas Z consistindo de 3 fatias Z separadas por 2 μm. Caso haja danos relacionados à pipetagem no tapete neuronal, tente evitar imagens dessas áreas. - Dependendo do microscópio e da câmera, ajuste os tempos de exposição e intensidades de excitação para cada um dos quatro canais fluorescentes separadamente para obter uma faixa dinâmica ideal das intensidades fluorescentes.
NOTA: O software de imagem geralmente fornece um histograma para determinar o tempo de exposição ideal. Se o histograma for deslocado muito para a esquerda na faixa de sinal baixo, o tempo de exposição é muito curto ou a intensidade de excitação é muito baixa. Se houver um penhasco acentuado no nível máximo do sinal à direita, o valor do sinal está saturado. Neste caso, reduza a intensidade da excitação ou diminua o tempo de exposição. - Armazene imagens em um formato aberto e sem perdas, como .tif.
7. Processamento de imagens (Dia 8)
- A segmentação das imagens e a extração das características fenotípicas são necessárias para a criação de perfis fenotípicos quantitativos. Utilize o software PhenoLink para segmentação de imagens e extração de recursos (Tabela de Materiais). As instruções para instalar o PhenoLink podem ser encontradas no repositório do GitHub (https://github.com/Ksilink/PhenoLink).
Observação : segmentação de imagem é usada para identificar e separar diferentes objetos ou regiões em uma imagem, enquanto extração de característica fenotípica é usada para analisar e extrair informações relevantes dessas regiões. Várias soluções de software alternativas, como CellProfiler10, ImageJ/FIJI 11, Napari 12 ou a Knime Analytics Platform13 estão disponíveis para extrair informações quantitativas de imagens de fluorescência multicanal. - Execute a segmentação de imagens em imagens brutas corrigidas por iluminação. Determine empiricamente os respectivos limiares de intensidade de canal fluorescente por placa, de modo que o sinal de fundo seja mínimo, e o sinal segmentado desejado corresponda ao sinal na imagem bruta. Placas processadas e coradas no mesmo dia normalmente requerem limiares de intensidade de canal comparáveis para segmentação.
- Defina o tamanho e a intensidade do núcleo para separar as células vivas das mortas. Ao usar imagens 40x, mantenha todos os outros parâmetros padrão e execute o software. Cento e vinte e seis características quantitativas da imagem serão calculadas por poço (Tabela Suplementar 1).
- Use os dados quantitativos tabulares resultantes para construir perfis fenotípicos e comparar perfis fenotípicos de diferentes linhagens celulares ou condições de tratamento. Cada linha corresponde a uma condição biológica (poço) e cada coluna corresponde a uma determinada característica fenotípica.
NOTA: Fornecemos um arquivo de saída de exemplo junto com o pipeline de análise de dados para ilustrar seu uso (consulte Tabela de Materiais). Além disso, a Figura 3 mostra a composição de um perfil fenotípico.
8. Geração e visualização do perfil fenotípico (Dia 8)
- Se você não tiver Python e Jupyter instalados em seu computador, instale o Anaconda Distribution e abra o software Jupyter. Baixe o notebook Jupyter fornecido e todos os outros arquivos fornecidos e salve-os no mesmo diretório (consulte a Tabela de Materiais). Abra o arquivo do bloco de anotações Jupyter usando o software Jupyter.
NOTA: Anaconda é uma plataforma gratuita e de código aberto para linguagens de programação como Python. Esta plataforma vem com o interpretador Python Jupyter que pode executar o notebook Jupyter fornecido para criar e analisar perfis fenotípicos (https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling). - Execute o notebook Jupyter célula por célula usando o software Jupyter. Os dados de exemplo fornecidos .fth e os arquivos .txt requisitos precisam estar localizados no mesmo diretório que o bloco de anotações Jupyter. Cada célula do bloco de anotações Jupyter é anotada para explicar sua funcionalidade.
NOTA: É crucial usar o bloco de anotações Jupyter na ordem correta, começando com o carregamento e dimensionamento de dados e prosseguir célula por célula até o final. Todos os dados e gráficos de saída serão armazenados em uma pasta recém-criada no diretório de origem do notebook Jupyter. A Figura 4 mostra o fluxo de trabalho e a saída do fluxo de trabalho.
Resultados
O perfil fenotípico em neurônios mDA é uma maneira eficiente de quantificar múltiplos aspectos da biologia celular e suas mudanças durante a modulação experimental. Para exemplificar essa metodologia, este estudo fez uso de neurônios mDA criopreservados LRRK2 G2019S e doadores saudáveis. Esses neurônios foram diferenciados por aproximadamente 37 dias, são pós-mitóticos e expressam marcadores neuronais (TUBB3 e MAP2) e marcadores de neurônios dopaminérgicos, incluindo tirosina hidroxilase (TH) em combinaç?...
Discussão
O perfil fenotípico é uma técnica para medir um grande número de fenótipos em células por meio da aplicação de colorações fluorescentes, microscopia e análise de imagens3. Perfis fenotípicos podem ser obtidos e comparados entre linhagens celulares ou outras condições experimentais para entender mudanças complexas na biologia celular que podem passar despercebidas ao usar uma única leitura. Descrevemos aqui a aplicação do perfil fenotípico em neurônios mDA humanos derivados de i...
Divulgações
Todos os autores são empregados por Ksilink.
Agradecimentos
Os autores gostariam de agradecer a todos os colegas da Ksilink por sua valiosa ajuda e discussões que levaram ao desenho do protocolo apresentado.
Materiais
| Name | Company | Catalog Number | Comments |
| Anti- chicken – Alexa 647 | Jackson ImmunoRearch | 703-605-155 | Immunofluorescence |
| Anaconda | https://www.anaconda.com/download | ||
| Anti-Map2 | Novus | NB300-213 | Immunofluorescence |
| Anti-mouse - Alexa 488 | Thermo Fisher | A11001 | Immunofluorescence |
| Anti-rabbit - Alexa 555 | Thermo Fisher | A21429 | Immunofluorescence |
| Anti-Tyrosine Hydroxylase | Merck | T2928 | Immunofluorescence |
| Anti-α-synuclein | Abcam | 138501 | Immunofluorescence |
| Bravo Automated Liquid Handling Platform with 384ST head | Agilent | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Confocal microscope | Yokogawa | CV7000 | The use of an automated confocal fluorescence microscope is recommended to ensure image quality consistency. |
| Countess Automated cell counter | Invitrogen | Cell counting before seeding. Can also be done using a manual counting chamber. | |
| DPBS +/+ | Gibco | 14040-133 | Buffer for washing |
| EL406 Washer Dispenser | BioTek (Agilent) | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Formaldehyde Solution (PFA 16 %) | Euromedex | EM-15710-S | Fixation before staining |
| Hoechst 33342 | Invitrogen | H3570 | Nuclear staining |
| iCell Base Medium 1 | Fujifilm | M1010 | Base medium for neurons |
| iCell DPN, Donor#01279, Phenotype AHN, lot#106339, 1M | Fujifilm | C1087 | Apparently healthy donor |
| iCell DPN, Donor#11299, Phenotype LRRK2 G2019S, phenotype PD lot#106139 | Fujifilm | C1149 | Donor carrying LRRK2 G2019S mutation |
| iCell Nervous System Supplement | Fujifilm | M1031 | Supplement for base medium |
| iCell Neural Supplement B | Fujifilm | M1029 | Supplement for base medium |
| Jupyter Python Notebook | In-house development | https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling | Notebook to perform phenotypic profile visualization and classification from raw data. |
| Laminin | Biolamina | LN521 | Plate coating |
| PFE-360 | MedChemExpress | HY-120085 | LRRK2 kinase inhibitor |
| PhenoLink | In-house development | https://github.com/Ksilink/PhenoLink | Software for image analysis |
| PhenoPlate 384w, PDL coated | Perkin Elmer | 6057500 | Pre-coated plate for cell culture and imaging. This plate allows imaging of all wells using all objectives of the Yokogawa CV7000 microscope. |
| Storage plates Abgene 120 µL | Thermo Scientific | AB-0781 | Necessary for compound dispensing using the Vprep pipetting system. If not available, the use of an electronic multichannel pipette is recommended. |
| Triton | Sigma | T9284 | Permeabilization before lysis |
| Trypan Blue | Sigma | T8154-20ML | Determination of living cells |
| Vprep Pipetting System | Agilent | Medium change and compound dispensing. Alternatively, an electronic multichannel pipette can be used. |
Referências
- Panicker, N., Ge, P., Dawson, V. L., Dawson, T. M. The cell biology of Parkinson's disease. The Journal of Cell Biology. 220 (4), 202012095 (2021).
- Caicedo, J. C., et al. Data-analysis strategies for image-based cell profiling. Nature Methods. 14 (9), 849-863 (2017).
- Chandrasekaran, S. N., Ceulemans, H., Boyd, J. D., Carpenter, A. E. Image-based profiling for drug discovery: due for a machine-learning upgrade. Nature Reviews Drug Discovery. 20 (2), 145-159 (2021).
- Bray, M. -. A., et al. Cell Painting, a high-content image-based assay for morphological profiling using multiplexed fluorescent dyes. Nature Protocols. 11 (9), 1757-1774 (2016).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Ziegler, S., Sievers, S., Waldmann, H. Morphological profiling of small molecules. Cell Chemical Biology. 28 (3), 300-319 (2021).
- Cobb, M. M., Ravisankar, A., Skibinski, G., Finkbeiner, S. iPS cells in the study of PD molecular pathogenesis. Cell and Tissue Research. 373 (1), 61-77 (2018).
- Elitt, M. S., Barbar, L., Tesar, P. J. Drug screening for human genetic diseases using iPSC models. Human Molecular Genetics. 27 (R2), 89-98 (2018).
- Vuidel, A., et al. High-content phenotyping of Parkinson's disease patient stem cell-derived midbrain dopaminergic neurons using machine learning classification. Stem Cell Reports. 17 (10), 2349-2364 (2022).
- Stirling, D. R., Swain-Bowden, M. J., Lucas, A. M., Carpenter, A. E., Cimini, B. A., Goodman, A. CellProfiler 4: improvements in speed, utility and usability. BMC Bioinformatics. 22 (1), 433 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sofroniew, N., et al. . napari: a multi-dimensional image viewer for Python. , (2022).
- Berthold, M. R., et al. KNIME: The Konstanz Information Miner. Data Analysis, Machine Learning and Applications. , 319-326 (2008).
- Fathi, A., et al. Diverging Parkinson's Disease Pathology between patient-derived GBAN370S, LRRK2G2019S and engineered SNCAA53T iPSC-derived Dopaminergic Neurons. bioRxiv. , (2023).
- Wang, Y., Huang, H., Rudin, C., Shaposhnik, Y. Understanding How Dimension Reduction Tools Work: An Empirical Approach to Deciphering t-SNE, UMAP, TriMap, and PaCMAP for Data Visualization. Journal of Machine Learning Research. 22 (201), 1-73 (2021).
- Ke, G., et al. LightGBM: A highly efficient gradient boosting decision tree. Advances in Neural Information Processing Systems. 30, (2017).
- Avazzadeh, S., Baena, J. M., Keighron, C., Feller-Sanchez, Y., Quinlan, L. R. Modelling Parkinson's Disease: iPSCs towards Better Understanding of Human Pathology. Brain Sciences. 11 (3), 373 (2021).
- Sánchez-Danés, A., et al. Disease-specific phenotypes in dopamine neurons from human iPS-based models of genetic and sporadic Parkinson's disease. EMBO Molecular Medicine. 4 (5), 380-395 (2012).
- Oosterveen, T., et al. Pluripotent stem cell derived dopaminergic subpopulations model the selective neuron degeneration in Parkinson's disease. Stem Cell Reports. 16 (11), 2718-2735 (2021).
- Hughes, R. E., et al. Multiparametric high-content cell painting identifies copper ionophores as selective modulators of esophageal cancer phenotypes. ACS Chemical Biology. 17 (7), 1876-1889 (2022).
- Akbarzadeh, M., et al. Morphological profiling by means of the Cell Painting assay enables identification of tubulin-targeting compounds. Cell Chemical Biology. 29 (6), 1053-1064 (2022).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Way, G. P., et al. Morphology and gene expression profiling provide complementary information for mapping cell state. Cell Systems. 13 (11), 911-923 (2022).
- Feng, Y., Mitchison, T. J., Bender, A., Young, D. W., Tallarico, J. A. Multi-parameter phenotypic profiling: using cellular effects to characterize small-molecule compounds. Nature Reviews Drug Discovery. 8 (7), 567-578 (2009).
- Antonov, S. A., Novosadova, E. V. Current state-of-the-art and unresolved problems in using human induced pluripotent stem cell-derived dopamine neurons for parkinson's disease drug development. International Journal of Molecular Sciences. 22 (7), 3381 (2021).
- Miller, J. D., et al. Human iPSC-based modeling of late-onset disease via progerin-induced aging. Cell Stem Cell. 13 (6), 691-705 (2013).
- Bezard, E., Gross, C. E., Brotchie, J. M. Presymptomatic compensation in Parkinson's disease is not dopamine-mediated. Trends in Neurosciences. 26 (4), 215-221 (2003).
- Wu, Y., Le, W., Jankovic, J. Preclinical Biomarkers of parkinson disease. Archives of Neurology. 68 (1), 22-30 (2011).
- Verstraelen, P., et al. Systematic quantification of synapses in primary neuronal culture. iScience. 23 (9), 101542 (2020).
- Liu-Yesucevitz, L., et al. ALS-Linked mutations enlarge TDP-43-enriched neuronal RNA granules in the dendritic arbor. The Journal of Neuroscience. 34 (12), 4167-4174 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados