Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Фенотипическое профилирование дофаминергических нейронов среднего мозга человека, полученных из стволовых клеток человека
В этой статье
Резюме
Этот протокол описывает культивирование клеток дофаминергических нейронов среднего мозга человека с последующим иммунологическим окрашиванием и генерацией нейрональных фенотипических профилей из полученных микроскопических изображений с высоким содержанием, позволяющих идентифицировать фенотипические вариации, обусловленные генетическими или химическими модуляциями.
Аннотация
Болезнь Паркинсона (БП) связана с рядом клеточных биологических процессов, которые вызывают потерю дофаминергических нейронов среднего мозга (мДА). Многие современные клеточные модели БП in vitro недостаточно сложны и не учитывают множественные фенотипы. Фенотипическое профилирование нейронов мДА, полученных из индуцированных плюрипотентных стволовых клеток человека (iPSC), может устранить эти недостатки путем одновременного измерения ряда фенотипов нейронов в PD-релевантном типе клеток. В данной статье мы опишем протокол получения и анализа фенотипических профилей коммерчески доступных нейронов мДА человека. Нейрон-специфическая флуоресцентная окрашивающая панель используется для визуализации фенотипов, связанных с ядерным α-синуклеином, тирозингидроксилазой (ТГ) и микротрубочками-ассоциированным белком 2 (MAP2). Описанный протокол фенотипического профилирования является масштабируемым, так как в нем используются 384-луночные планшеты, автоматическая работа с жидкостями и высокопроизводительная микроскопия. Полезность протокола проиллюстрирована на примере здоровых донорских нейронов mDA и нейронов mDA, несущих PD-сцепленную мутацию G2019S в гене лейцин-богатой повторной киназы 2 (LRRK2). Обе клеточные линии обрабатывали ингибитором киназы LRRK2 PFE-360 и измеряли фенотипические изменения. Кроме того, мы демонстрируем, как многомерные фенотипические профили могут быть проанализированы с помощью кластеризации или методов классификации с учителем на основе машинного обучения. Описанный протокол будет особенно интересен исследователям, работающим над моделированием заболеваний нейронов или изучающими эффекты химических соединений в нейронах человека.
Введение
При болезни Паркинсона (БП) нарушаются различные клеточные биологические процессы. Например, митохондриальная дисфункция, окислительный стресс, дефекты деградации белков, нарушение везикулярного транспорта и эндолизосомальной функции, связанные с потерей дофаминергических нейронов среднего мозга (мДА), обычно наблюдаются при БП1. Таким образом, болезнь Паркинсона, по-видимому, включает в себя несколько механизмов заболевания, которые могут взаимодействовать друг с другом и ухудшать друг друга. Одним из полезных способов исследования этого механистического взаимодействия является создание комплексного фенотипического отпечатка пальца или профиля дофаминергических нейронов среднего мозга (mDA).
Фенотипическое профилирование – это подход, который включает в себя создание профиля выборки на основе набора измеримых характеристик и, во-вторых, прогнозирование выборки на основе этого профиля 2,3. Цель профилирования состоит в том, чтобы зафиксировать широкий спектр признаков, некоторые из которых, возможно, ранее не были связаны с заболеванием или лечением3. В результате профилирование может выявить неожиданные биологические процессы. Фенотипическое профилирование, как правило, опирается на флуоресцентно окрашенные клетки, и для создания фенотипическихпрофилей были разработаны стандартизированные анализы, такие как Cell Painting. В последнее время фенотипическое профилирование, например, применяется для характеризации малых молекул или точного прогнозирования подтипов БП исключительно на основе фибробластов, полученных от пациентов 5,6. Несмотря на эти достижения, фенотипическое профилирование редко применялось к постмитотическим дифференцированным клеткам, таким как индуцированные плюрипотентные стволовые клетки человека (iPSC) нейроны mDA, которые экспрессируют PD-сцепленные мутации, такие как LRRK2 G2019S. К существенным проблемам моделей, полученных на основе ИПСК, относятся наличие малозаметных или вариабельных патологических признаков в группах дифференцировки или генотипах, а также тот факт, что изолированные фенотипы БП не охватывают всю сложность заболевания. Кроме того, несмотря на то, что нейронные модели ИПСК являются физиологически значимыми, они редко используются в процессах разработки лекарств для лечения БП из-за опасений по поводу технической сложности 7,8.
Ранее мы разработали надежную методологию для измерения нескольких патофизиологических фенотипов, связанных с болезнью Паркинсона, в нейронах мДА человека, которые чувствительны как к генетическим, так и к химическим соединениям фенотипическимизменениям. В данной статье подробно описывается дальнейшая оптимизированная версия этой методики для создания фенотипических профилей из нейронов mDA (рис. 1). Этот протокол имеет ряд преимуществ по сравнению с ранее описанными подходами к фенотипическому профилированию, такими как использование высококачественных нейронов mDA и техническая воспроизводимость. Впервые этот протокол позволяет осуществлять фенотипическое профилирование физиологически значимых постмитотических нейронов mDA после химических возмущений с высокой степенью масштабируемости. Полностью дифференцированные и криоконсервированные нейроны mDA коммерчески доступны, что значительно снижает вариабельность дифференцировки от партии к партии. Во-вторых, техническая вариабельность может быть дополнительно снижена за счет использования четко определенного плана эксперимента (т.е. продолжительности культивирования или отказа от краевых лунок), автоматизированной обработки жидкостей и автоматизированной микроскопии. Кроме того, здесь описаны начальные этапы анализа фенотипического профиля с использованием неконтролируемой кластеризации или контролируемой классификации, а также указано, как можно анализировать данные фенотипического профилирования. Этот протокол будет полезен исследователям, интересующимся фенотипическими изменениями нейронов mDA, индуцированными генетическими или химическими возмущениями, в частности, когда требуется высокомасштабируемая исследовательская установка, например, во время скрининговых кампаний или когда необходимо изучить влияние меньшего числа соединений, например, для определения токсических эффектов. Таким образом, предполагается, что применение фенотипического профилирования нейронов человека является ценным методом для изучения сложных фенотипов, связанных с заболеванием, и характеристики клеточных эффектов кандидатов в лекарственные препараты.
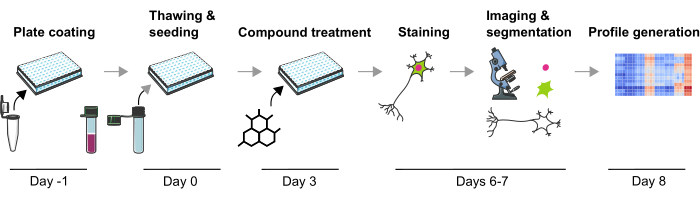
Рисунок 1: Схематическое изображение экспериментального протокола для создания фенотипических профилей на основе изображений из нейронов мДА человека, полученных из ИПСК. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
протокол
1. Подготовка среды и планшетов для посева нейронов (День 1)
- Чтобы подготовить планшеты к посеву нейронов в День-1, подогрейте ламинин до комнатной температуры (RT) непосредственно перед использованием. Приготовьте раствор ламинина, разбавив исходный раствор ламинина (0,1 мг/мл) 1/10 в холодном PBS+/+ (с Ca 2+ и Mg2+).
ПРИМЕЧАНИЕ: Все реагенты перечислены в таблице материалов. Составы растворов и буферов описаны в таблицах 1-4. - Затем добавьте 25 мкл раствора ламинина в каждую лунку 384-луночного планшета с полимер-D-лизином (PDL) и инкубируйте в течение ночи при 4 °C. Пластины с покрытием можно хранить при температуре 4 °C до одной недели.
ПРИМЕЧАНИЕ: Протокол может быть приостановлен на срок до одной недели. Запечатайте пластины с помощью полиэтиленовой пленки. - Подготовьте полный материал для технического обслуживания и храните при температуре 4 °C до одного месяца (Таблица 1).
2. Размораживание нейронов (День 0)
- Чтобы разморозить нейроны на 0-й день, разогрейте водяную баню до 37 °C и уравновешивайте Complete Maintenance Media до RT, защищенного от света.
- Выньте флакон с коммерчески полученными замороженными нейронами (см. Таблицу материалов) из резервуара с жидким азотом и поместите его на сухой лед. Затем поместите флакон на водяную баню на 2 минуты. Как только жидкость полностью оттает, продезинфицируйте флакон 70% этиловым спиртом.
- Аспирируйте размороженные нейроны с помощью пипетки P1000 (около 370 мкл) и перенесите их в центрифужную пробирку объемом 50 мл без пипетирования вверх-вниз. Затем промойте флакон 630 мкл Complete Maintenance Medium, капля за каплей (под углом 45°) в пробирку объемом 50 мл. Медленно перемешивайте во время дозирования.
ПРИМЕЧАНИЕ: Очень важно дозировать среду медленно, капля за каплей, чтобы избежать осмотического разрыва клеток. Угол наклона 45° и легкое перемешивание сводят к минимуму высокое локальное осмотическое давление во время пипетирования. - Аналогичным образом добавьте 1 мл питательной среды в центрифужную пробирку объемом 50 мл с помощью пипетки P1000. Затем медленно добавьте 2 мл Complete Maintenance Medium. Тщательно перемешайте во время дозирования.
- Подсчитайте ячейки. Приготовьте микропробирку с 10 мкл трипанового синего и 10 мкл клеточной суспензии и добавьте в предметное стекло счетной камеры (10 мкл). Выполняйте подсчет с помощью автоматического счетчика ячеек (см. Таблицу материалов) или вручную.
- После подсчета центрифугируют пробирку объемом 50 мл, содержащую нейроны, при 400 x g в течение 5 мин при RT и удаляют надосадочную жидкость. Осторожно ресуспендируйте гранулу с помощью пипетки P1000 и 1 мл средства для комплексного обслуживания. Затем добавьте необходимый объем, чтобы достичь желаемой концентрации (300 000 клеток/мл, см. также следующий шаг).
3. Посев нейронов на подготовленные планшеты (День 0)
- Чтобы посеять нейроны на подготовленные планшеты на день 0, достаньте планшеты с покрытием из холодильника, поместите их под колпак для клеточных культур и дайте им уравновеситься до RT примерно на 30 минут.
- Непосредственно перед посевом аспирируйте 15 мкл раствора для покрытия с помощью автоматического манипулятора жидкостями или 16-канальной пипетки. Оставьте примерно 10 мкл на лунку, чтобы предотвратить повреждение покрытия.
- Затем дозируют 50 мкл клеточного раствора, содержащего 300 000 клеток/мл (приготовленного на этапе 2.6), на лунку с помощью 16-канальной пипетки, в результате чего получается 15 000 засеянных нейронов на лунку и конечный объем на лунку 60 мкл.
- В 384-луночном планшете избегайте использования столбцов 1, 2, 23, 24 и строк A, B, O и P, чтобы свести к минимуму возможные краевые эффекты, которые могут повлиять на фенотипические профили. Заполните неиспользуемые пустые лунки 80 мкл PBS. Инкубируют планшеты при температуре 37 °C и 5% CO2.
4. Средняя смена или комбинированная обработка (день 3)
- В зависимости от количества лунок предварительно нагрейте соответствующий объем Complete Maintenance Medium at RT. Беречь от света.
- Если требуется изменение среды, перейдите к шагу 4.4. Если требуется обработка соединения, проверьте концентрацию исходного соединения и используемый растворитель (вода, ДМСО, метанол и т. д.).
- Приготовьте 1,5-кратный концентрированный раствор соединения для всех требуемых концентраций. Приготовьте компаундные разводы, используя Complete Maintenance Medium.
ПРИМЕЧАНИЕ: В качестве нейтрального контроля используйте соответствующий растворитель в той же концентрации, что и испытуемое соединение. Если используются несколько или неизвестных соединений в разных концентрациях, целесообразно провести отдельный эксперимент «доза-реакция» для измерения влияния растворителя на фенотипический профиль. - Если используется автоматическая система дозирования, добавьте 60 мкл 1,5-кратного раствора компаунда в 384-луночный накопительный планшет. В качестве альтернативы можно использовать 16-канальную пипетку. Если требуется смена среды, добавьте вместо этого 60 мкл Complete Maintenance Medium.
- Используя автоматическую систему дозирования, отсасывайте и выбрасывайте 40 мкл среды на лунку из нейронсодержащей пластины, чтобы сохранить 20 мкл/лунку. Затем добавьте 40 мкл/лунку 1,5-кратного раствора соединения из 384-луночного накопителя в каждую лунку для получения желаемой конечной концентрации.
ПРИМЕЧАНИЕ: Очень важно не выполнять полную смену среды, а всегда оставлять остаточную среду в лунке, чтобы предотвратить повреждение нейронального ковра или покрытия. - В случае, если культивирование нейронов проводится более 6 дней, описанных в данном протоколе, следует менять среду каждые 2-3 дня.
5. Фиксация и окрашивание нейронов (6-7 дни)
- Чтобы зафиксировать и окрасить нейроны на 6-й и 7-й день, используйте автоматизированный манипулятор жидкости для всех этапов дозирования и промывки. В качестве альтернативы можно использовать 16-канальную пипетку.
- Приготовьте 10% раствор Triton X-100, разбавив Triton X-100 в 1x PBS. Встряхивайте до тех пор, пока раствор не станет однородным. Хранить при температуре 4 °C.
- Чтобы зафиксировать нейроны, дозируйте 20 мкл/лунку 16% PFA, в результате чего конечная концентрация составляет 4%. Инкубируйте планшет в течение 30 минут при RT и промойте его три раза с помощью 1x PBS. Оставьте 20 мкл/лунку PBS после последней стирки.
ВНИМАНИЕ: PFA признан опасным веществом, которое, как известно, вызывает пероральную, кожную и респираторную токсичность. Он также представляет угрозу для глаз и может привести к генетическим мутациям и раку. Правильное обращение с PFA требует использования соответствующих средств индивидуальной защиты, таких как защита глаз и лица, а также обеспечения надлежащей вентиляции. Важно не допустить попадания PFA в окружающую среду. - Для пермеабилизации и блокировки приготовьте 2-кратный раствор для блокировки (табл. 2).
- Добавьте 20 мкл/лунку 2-кратного блокирующего раствора (1-кратная конечная концентрация), инкубируйте в течение 1 ч при RT и промойте один раз PBS. Храните 20 мкл/лунку PBS после стирки.
- Для окрашивания первичными антителами (см. Таблицу материалов) приготовьте 2-кратный первичный окрашивающий буфер (Таблица 3).
- Добавьте 20 мкл/лунку 2-кратного первичного окрашивающего буфера (1-кратная конечная концентрация) и инкубируйте в течение ночи при 4 °C. На следующее утро на 7-й день трижды умойтесь PBS. После промывки оставьте 20 мкл/лунку PBS.
- Для окрашивания вторичными антителами (см. Таблицу материалов) приготовьте 2-кратный буфер для вторичного окрашивания (Таблица 4).
- Добавьте 20 мкл/лунку 2-кратного вторичного окрашивающего буфера (1-кратная конечная концентрация), инкубируйте в течение 2 ч при RT вдали от света и трижды промойте PBS. Оставьте 100 мкл PBS/хорошо после последней стирки.
- Добавьте алюминиевое уплотнение на пластину, чтобы свести к минимуму испарение. Как вариант, накройте тарелку полиэтиленовой пленкой и алюминиевой фольгой. Перейдите к получению изображения или храните пластину при температуре 4 °C.
ПРИМЕЧАНИЕ: Протокол может быть приостановлен на срок до одной недели. Если наблюдается фоновая флуоресценция, рассмотрите возможность увеличения времени блокировки. Если окрашивания недостаточно, попробуйте увеличить концентрацию первичных антител или время инкубации.
- Добавьте алюминиевое уплотнение на пластину, чтобы свести к минимуму испарение. Как вариант, накройте тарелку полиэтиленовой пленкой и алюминиевой фольгой. Перейдите к получению изображения или храните пластину при температуре 4 °C.
6. Визуализация флуоресцентно окрашенных нейронов (7-й день)
- Получите изображения покрытых, культивируемых и окрашенных нейронов на 7-й день. В идеале следует использовать автоматизированный конфокальный флуоресцентный микроскоп (см. Таблицу материалов). Кроме того, можно получить изображения вручную.
- Для получения каналов Хёхста, TH, α-синуклеина и MAP2 используются лазеры с длиной волны 405 нм, 488 нм, 561 нм и 647 нм соответственно (рис. 2).
ПРИМЕЧАНИЕ: Чтобы получить достаточное количество подробных данных для фенотипического профилирования, используйте 40-кратный объектив и получите 16 полей/лунку с помощью Z-стеков, состоящих из 3 Z-срезов, разделенных 2 мкм. В случае повреждений нейронного ковра, связанных с пипетированием, старайтесь избегать визуализации этих областей. - В зависимости от микроскопа и камеры отрегулируйте время экспозиции и интенсивность возбуждения для каждого из четырех флуоресцентных каналов отдельно, чтобы получить оптимальный динамический диапазон интенсивностей флуоресценции.
ПРИМЕЧАНИЕ: Программное обеспечение для обработки изображений часто предоставляет гистограмму для определения идеального времени экспозиции. Если гистограмма слишком сильно смещена влево в низком диапазоне сигнала, значит, время экспозиции слишком короткое или интенсивность возбуждения слишком мала. Если на максимальном уровне сигнала справа есть резкий обрыв, то значение сигнала насыщено. В этом случае уменьшите интенсивность возбуждения или сократите время экспозиции. - Храните изображения в открытом формате без потерь, таком как .tif.
7. Обработка изображений (День 8)
- Сегментация изображений и выделение фенотипических признаков необходимы для создания количественных фенотипических профилей. Используйте программное обеспечение PhenoLink для сегментации изображений и извлечения признаков (Таблица материалов). Инструкцию по установке PhenoLink можно найти в репозитории GitHub (https://github.com/Ksilink/PhenoLink).
ПРИМЕЧАНИЕ: Сегментация изображения используется для идентификации и разделения различных объектов или областей на изображении, в то время как извлечение фенотипических признаков используется для анализа и извлечения релевантной информации из этих областей. Существует несколько альтернативных программных решений, таких как CellProfiler10, ImageJ/FIJI 11, Napari 12 или Knime Analytics Platform13, для извлечения количественной информации из многоканальных флуоресцентных изображений. - Выполняйте сегментацию изображений на необработанных изображениях с коррекцией освещенности. Определите соответствующие пороги интенсивности флуоресцентного канала опытным путем для каждой пластины, чтобы фоновый сигнал был минимальным, а требуемый сегментированный сигнал соответствовал сигналу в необработанном изображении. Пластины, обработанные и окрашенные в один и тот же день, обычно требуют сопоставимых пороговых значений интенсивности канала для сегментации.
- Определите размер и интенсивность ядра, чтобы отделить живые клетки от мертвых. При использовании 40-кратных изображений сохраните все остальные параметры по умолчанию и запустите программное обеспечение. На одну скважину будет рассчитано сто двадцать шесть количественных характеристик изображения (Дополнительная таблица 1).
- Используйте полученные табличные количественные данные для построения фенотипических профилей и сравнения фенотипических профилей из различных клеточных линий или условий лечения. Каждая строка соответствует биологическому состоянию (колодцу), а каждый столбец соответствует определенному фенотипическому признаку.
ПРИМЕЧАНИЕ: Мы приводим пример выходного файла вместе с конвейером анализа данных, чтобы проиллюстрировать его использование (см. Таблицу материалов). Кроме того, на рисунке 3 показан состав фенотипического профиля.
8. Генерация и визуализация фенотипического профиля (День 8)
- Если на вашем компьютере не установлены Python и Jupyter, установите дистрибутив Anaconda и откройте программное обеспечение Jupyter. Загрузите прилагаемую записную книжку Jupyter и все остальные предоставленные файлы и сохраните их в том же каталоге (см. Таблицу материалов). Откройте файл записной книжки Jupyter с помощью программного обеспечения Jupyter.
ПРИМЕЧАНИЕ: Anaconda — это бесплатная платформа с открытым исходным кодом для языков программирования, таких как Python. Эта платформа поставляется с интерпретатором Python Jupyter, который может выполнять предоставленную записную книжку Jupyter для создания и анализа фенотипических профилей (https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling). - Выполните ячейку за ячейкой записной книжки Jupyter с помощью программного обеспечения Jupyter. Предоставленные примеры данных в формате FTH и файлы .txt требований должны находиться в том же каталоге, что и записная книжка Jupyter. Каждая ячейка записной книжки Jupyter снабжена аннотациями, поясняющими ее функциональные возможности.
ПРИМЕЧАНИЕ: Крайне важно использовать записную книжку Jupyter в правильном порядке, начиная с загрузки и масштабирования данных, и продолжая ячейку за ячейкой до конца. Все выходные данные и графика будут храниться во вновь созданной папке в исходном каталоге записной книжки Jupyter. На рисунке 4 показан рабочий процесс и выходные данные рабочего процесса.
Результаты
Фенотипическое профилирование нейронов мДА является эффективным способом количественной оценки различных аспектов клеточной биологии и их изменений во время экспериментальной модуляции. Чтобы проиллюстрировать эту методологию, в этом исследовании использовались криоконсервирова...
Обсуждение
Фенотипическое профилирование — это метод измерения большого числа фенотипов в клетках с помощью флуоресцентного окрашивания, микроскопии и анализа изображений3. Фенотипические профили могут быть получены и сравнены между клеточными линиями или другими эксперименталь?...
Раскрытие информации
Все авторы являются сотрудниками компании «Ксилинк».
Благодарности
Авторы хотели бы поблагодарить всех коллег из «Ксилинк» за их ценную помощь и обсуждения, которые привели к разработке представленного протокола.
Материалы
| Name | Company | Catalog Number | Comments |
| Anti- chicken – Alexa 647 | Jackson ImmunoRearch | 703-605-155 | Immunofluorescence |
| Anaconda | https://www.anaconda.com/download | ||
| Anti-Map2 | Novus | NB300-213 | Immunofluorescence |
| Anti-mouse - Alexa 488 | Thermo Fisher | A11001 | Immunofluorescence |
| Anti-rabbit - Alexa 555 | Thermo Fisher | A21429 | Immunofluorescence |
| Anti-Tyrosine Hydroxylase | Merck | T2928 | Immunofluorescence |
| Anti-α-synuclein | Abcam | 138501 | Immunofluorescence |
| Bravo Automated Liquid Handling Platform with 384ST head | Agilent | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Confocal microscope | Yokogawa | CV7000 | The use of an automated confocal fluorescence microscope is recommended to ensure image quality consistency. |
| Countess Automated cell counter | Invitrogen | Cell counting before seeding. Can also be done using a manual counting chamber. | |
| DPBS +/+ | Gibco | 14040-133 | Buffer for washing |
| EL406 Washer Dispenser | BioTek (Agilent) | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Formaldehyde Solution (PFA 16 %) | Euromedex | EM-15710-S | Fixation before staining |
| Hoechst 33342 | Invitrogen | H3570 | Nuclear staining |
| iCell Base Medium 1 | Fujifilm | M1010 | Base medium for neurons |
| iCell DPN, Donor#01279, Phenotype AHN, lot#106339, 1M | Fujifilm | C1087 | Apparently healthy donor |
| iCell DPN, Donor#11299, Phenotype LRRK2 G2019S, phenotype PD lot#106139 | Fujifilm | C1149 | Donor carrying LRRK2 G2019S mutation |
| iCell Nervous System Supplement | Fujifilm | M1031 | Supplement for base medium |
| iCell Neural Supplement B | Fujifilm | M1029 | Supplement for base medium |
| Jupyter Python Notebook | In-house development | https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling | Notebook to perform phenotypic profile visualization and classification from raw data. |
| Laminin | Biolamina | LN521 | Plate coating |
| PFE-360 | MedChemExpress | HY-120085 | LRRK2 kinase inhibitor |
| PhenoLink | In-house development | https://github.com/Ksilink/PhenoLink | Software for image analysis |
| PhenoPlate 384w, PDL coated | Perkin Elmer | 6057500 | Pre-coated plate for cell culture and imaging. This plate allows imaging of all wells using all objectives of the Yokogawa CV7000 microscope. |
| Storage plates Abgene 120 µL | Thermo Scientific | AB-0781 | Necessary for compound dispensing using the Vprep pipetting system. If not available, the use of an electronic multichannel pipette is recommended. |
| Triton | Sigma | T9284 | Permeabilization before lysis |
| Trypan Blue | Sigma | T8154-20ML | Determination of living cells |
| Vprep Pipetting System | Agilent | Medium change and compound dispensing. Alternatively, an electronic multichannel pipette can be used. |
Ссылки
- Panicker, N., Ge, P., Dawson, V. L., Dawson, T. M. The cell biology of Parkinson's disease. The Journal of Cell Biology. 220 (4), 202012095 (2021).
- Caicedo, J. C., et al. Data-analysis strategies for image-based cell profiling. Nature Methods. 14 (9), 849-863 (2017).
- Chandrasekaran, S. N., Ceulemans, H., Boyd, J. D., Carpenter, A. E. Image-based profiling for drug discovery: due for a machine-learning upgrade. Nature Reviews Drug Discovery. 20 (2), 145-159 (2021).
- Bray, M. -. A., et al. Cell Painting, a high-content image-based assay for morphological profiling using multiplexed fluorescent dyes. Nature Protocols. 11 (9), 1757-1774 (2016).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Ziegler, S., Sievers, S., Waldmann, H. Morphological profiling of small molecules. Cell Chemical Biology. 28 (3), 300-319 (2021).
- Cobb, M. M., Ravisankar, A., Skibinski, G., Finkbeiner, S. iPS cells in the study of PD molecular pathogenesis. Cell and Tissue Research. 373 (1), 61-77 (2018).
- Elitt, M. S., Barbar, L., Tesar, P. J. Drug screening for human genetic diseases using iPSC models. Human Molecular Genetics. 27 (R2), 89-98 (2018).
- Vuidel, A., et al. High-content phenotyping of Parkinson's disease patient stem cell-derived midbrain dopaminergic neurons using machine learning classification. Stem Cell Reports. 17 (10), 2349-2364 (2022).
- Stirling, D. R., Swain-Bowden, M. J., Lucas, A. M., Carpenter, A. E., Cimini, B. A., Goodman, A. CellProfiler 4: improvements in speed, utility and usability. BMC Bioinformatics. 22 (1), 433 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sofroniew, N., et al. . napari: a multi-dimensional image viewer for Python. , (2022).
- Berthold, M. R., et al. KNIME: The Konstanz Information Miner. Data Analysis, Machine Learning and Applications. , 319-326 (2008).
- Fathi, A., et al. Diverging Parkinson's Disease Pathology between patient-derived GBAN370S, LRRK2G2019S and engineered SNCAA53T iPSC-derived Dopaminergic Neurons. bioRxiv. , (2023).
- Wang, Y., Huang, H., Rudin, C., Shaposhnik, Y. Understanding How Dimension Reduction Tools Work: An Empirical Approach to Deciphering t-SNE, UMAP, TriMap, and PaCMAP for Data Visualization. Journal of Machine Learning Research. 22 (201), 1-73 (2021).
- Ke, G., et al. LightGBM: A highly efficient gradient boosting decision tree. Advances in Neural Information Processing Systems. 30, (2017).
- Avazzadeh, S., Baena, J. M., Keighron, C., Feller-Sanchez, Y., Quinlan, L. R. Modelling Parkinson's Disease: iPSCs towards Better Understanding of Human Pathology. Brain Sciences. 11 (3), 373 (2021).
- Sánchez-Danés, A., et al. Disease-specific phenotypes in dopamine neurons from human iPS-based models of genetic and sporadic Parkinson's disease. EMBO Molecular Medicine. 4 (5), 380-395 (2012).
- Oosterveen, T., et al. Pluripotent stem cell derived dopaminergic subpopulations model the selective neuron degeneration in Parkinson's disease. Stem Cell Reports. 16 (11), 2718-2735 (2021).
- Hughes, R. E., et al. Multiparametric high-content cell painting identifies copper ionophores as selective modulators of esophageal cancer phenotypes. ACS Chemical Biology. 17 (7), 1876-1889 (2022).
- Akbarzadeh, M., et al. Morphological profiling by means of the Cell Painting assay enables identification of tubulin-targeting compounds. Cell Chemical Biology. 29 (6), 1053-1064 (2022).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Way, G. P., et al. Morphology and gene expression profiling provide complementary information for mapping cell state. Cell Systems. 13 (11), 911-923 (2022).
- Feng, Y., Mitchison, T. J., Bender, A., Young, D. W., Tallarico, J. A. Multi-parameter phenotypic profiling: using cellular effects to characterize small-molecule compounds. Nature Reviews Drug Discovery. 8 (7), 567-578 (2009).
- Antonov, S. A., Novosadova, E. V. Current state-of-the-art and unresolved problems in using human induced pluripotent stem cell-derived dopamine neurons for parkinson's disease drug development. International Journal of Molecular Sciences. 22 (7), 3381 (2021).
- Miller, J. D., et al. Human iPSC-based modeling of late-onset disease via progerin-induced aging. Cell Stem Cell. 13 (6), 691-705 (2013).
- Bezard, E., Gross, C. E., Brotchie, J. M. Presymptomatic compensation in Parkinson's disease is not dopamine-mediated. Trends in Neurosciences. 26 (4), 215-221 (2003).
- Wu, Y., Le, W., Jankovic, J. Preclinical Biomarkers of parkinson disease. Archives of Neurology. 68 (1), 22-30 (2011).
- Verstraelen, P., et al. Systematic quantification of synapses in primary neuronal culture. iScience. 23 (9), 101542 (2020).
- Liu-Yesucevitz, L., et al. ALS-Linked mutations enlarge TDP-43-enriched neuronal RNA granules in the dendritic arbor. The Journal of Neuroscience. 34 (12), 4167-4174 (2014).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены