Method Article
La importancia de la concentración de la proteína correcta para la cinética y la determinación de afinidad en el análisis de la estructura-función
En este artículo
Resumen
Nosotros aplicamos la etiqueta sin análisis de la interacción de proteínas utilizando Biacore X100 para el análisis de estructura-función de la unión de varios mutantes cistatina B a la papaína través de la caracterización cinética. Libre de calibración de análisis de concentración (ACCP) mide la concentración de proteína con actividad de unión retenido sin la necesidad de una curva patrón. Nos muestran que la confirmación de las concentraciones de uso de la ACCP aumenta la fiabilidad de los análisis cinético y que las constantes cinéticas se puede determinar de forma fiable, incluso si la actividad de una proteína recombinante se reduce.
Resumen
En este estudio, se explora la interacción entre el B cisteína proteasa bovina cistatina inhibidor y una forma catalíticamente inactiva de la papaína (Fig. 1), una proteasa cisteína planta, por análisis en tiempo real sin etiquetas usando X100 Biacore. Varias variantes B cistatina con mutaciones puntuales en las áreas de interacción con la papaína, que se producen. Para cada variante B cistatina que determinar su concentración de unión específica con la calibración sin el análisis de concentración (ACCP) y comparar los valores obtenidos con la concentración de proteínas totales, determinada por un
Protocolo

Figura 1. La estructura tridimensional de la cistatina del complejo B (azul) con papaína (amarillo). Mutado residuos de la cistatina B se muestran en rojo.
1. Principios de la Marca de análisis sin la interacción con los sistemas de Biacore
En un experimento de unión típica etiqueta sin utilizar un sistema de Biacore, una biomolécula llamado el "ligando" se une a la superficie de un chip sensor. Un sistema de flujo de canales trae su pareja de unión, denominada "analito", en contacto con la superficie del chip, donde la detección se lleva a cabo. Cuando el analito se une al ligando, el cambio resultante en la acumulación de masa en la superficie se detecta por la resonancia de plasmones superficiales (SPR). La respuesta de SPR es proporcional a la cantidad de analito vinculante.
Dado que la unión se mide en tiempo real, la cinética de asociación y de las constantes de disociación de velocidad para una interacción específica puede ser determinada. A partir de estas constantes, es posible calcular la afinidad como la constante de disociación de equilibrio. También es posible calcular la afinidad de los datos del estado de unión estable. Una metodología similar se puede también utilizar para determinar la concentración de una proteína que se une específicamente a un ligando en la superficie.
2. Analizar las propiedades cinéticas de la interacción proteína-proteína
En X100 Biacore, análisis cinético se puede realizar utilizando un solo ciclo de la cinética. En un experimento de un solo ciclo de la cinética, una serie de concentraciones del analito se inyecta en un ciclo único análisis sin regeneración de la superficie en medio de inyecciones. Por lo tanto, de un solo ciclo de la cinética permite el análisis de cinética cuando es difícil encontrar las condiciones adecuadas de regeneración.
Una vez que los datos de un experimento cinético se ha recogido, X100 Biacore software de evaluación genera los valores de k a, d k, y D K ajustando los datos a un modelo de interacción.
3. Enfoques para determinar la concentración de proteína
Analizar las interacciones entre biomoléculas es importante para entender su función. La caracterización de la unión de las proteínas con otras proteínas, de ácido nucleico, o las pequeñas moléculas es fundamental para la investigación bioquímica, y se emplea en muchas otras áreas, incluyendo el descubrimiento de fármacos.
Para una medición exacta de la cinética de la interacción entre dos proteínas que interactúan, es esencial conocer la concentración de la proteína se une específicamente en la muestra experimental que se utiliza como sustancia analizada. Una lectura de un espectrofotómetro de 280 o ensayos colorimétricos, tales como el empleo de un reactivo de Bradford se utiliza comúnmente para determinar la concentración de proteína total. Sin embargo, las impurezas de proteínas puede afectar el resultado. Más importante aún, las dos formas activa e inactiva de la proteína se incluyen en la concentración de proteína total. Particularmente en el caso de las proteínas recombinantes, que puede estar inactiva debido al doblez incorrecta, es importante para determinar el porcentaje de proteína que se une específicamente en la muestra.
En X100 Biacore, la concentración en relación con la actividad de unión específica puede ser determinada por la comparación de los niveles de respuesta obligatoria a una curva de calibración derivada de un estándar conocido, o mediante el uso de la Libre de Calibración metodología más recientemente introducido el análisis de concentración (ACCP). ACCP no se basa en un estándar. Por lo tanto, CFCA es particularmente útil en el caso del estudio de las formas mutantes de las proteínas, donde las normas no suelen estar disponibles.
En un experimento de la ACCP, el tipo de unión inicial se mide a diferentes caudales en condiciones cuando la difusión de la muestra a la superficie del chip es la velocidad. El coeficiente de difusión de la sustancia analizada, las dimensiones de la celda de flujo y las velocidades de flujo se tienen en cuenta en el cálculo de la concentración de la unión específica de la tasa de 1,2 consolidación inicial.
En un experimento de cinética, la concentración de la sustancia analizada se utiliza en el cálculo de la cinética de velocidad de asociación constante y la afinidad de los datos experimentales. ACCP y la medición cinética en los sistemas de Biacore ambos se basan en las propiedades de la interacción misma. Por lo tanto, con la concentración de unión específica determinada por el análisis de Biacore, en lugar de la concentración de proteína total, aumenta la fiabilidad de los resultados.
4. Utilizando el análisis de Biacore para caracterizar la interacción entre la cistatina B y la papaína
B cistatina mamíferos es un inhibidor reversible, competitiva a proteínas y fuerte unión de cisteína proteinasas papaína-como, sobre todo la catepsina B, H, K, L y S. Estas proteínas están implicadas principalmente en nonsla degradación intracelular de proteínas electiva. Cistatinas se supone que protegen a las células y los tejidos de la proteólisis inadecuado por estas enzimas. También inactivar proteasas cisteína a partir de parásitos y virus y puede participar en la defensa contra la invasión de estos agentes infecciosos. Además, cistatinas inhibir varias cisteína proteinasas de plantas, tales como la papaína, que se utiliza con frecuencia como una enzima modelo en los estudios de estructura-función. La estructura tridimensional del complejo B cistatina con papaína 3 muestra que la interacción entre las dos proteínas está dominado por los contactos hidrofóbicos, lo que, desde el lado de inhibidor, se proporcionan por la N-y C-terminal y dos lazos de la horquilla ( Figura 1). En este estudio, se analiza la importancia del extremo C-terminal y el segundo bucle de unión de la cistatina B para unirse a la papaína mediante el uso de la cistatina B bovina variantes que contienen mutaciones puntuales en las áreas de interacción con papaína. En primer lugar, determinar la concentración de unión específica de las cuatro variantes de la cistatina B utilizados, así como la de la proteína de tipo salvaje. Una vez que las concentraciones de unión específica de la cistatina B activa se determinan las constantes de velocidad y la afinidad de tipo salvaje y las variantes mutantes de la cistatina B vinculante a la papaína se miden utilizando X100 Biacore.
5. Instrumentos y reactivos
- Las variantes B cistatina Cys3Ser/His75Gly, Cys3Ser/Leu73Gly, Cys3Ser/Tyr97Ala y Cys3Ser se producen a lo descrito previamente 4. Todos los mutantes contienen una sustitución adicional de cisteína a serina en la posición 3, para evitar la formación de dímeros vinculados disulfuro inactivos de la cistatina B.
- El objetivo de la papaína también se prepara como se describió anteriormente 5. Es S-(metiltio) papaína (MMTS-la papaína) que tiene un grupo metiltio unido a Cys25 en el activo fisura, lo que hace la proteasa catalíticamente inactiva.
- X100 X100 Biacore con Biacore Package Plus se utiliza para medir y analizar las concentraciones de uniones específicas y cinética de unión.
- MMTS-la papaína se inmoviliza en CM5 chip sensor con Biacore Kit de acoplamiento Amine.
- Las carreras se llevan a cabo a 25 ° C, y el buffer es de 0,01 M Hepes a pH 7,4, NaCl 0,15 M, EDTA 0,0034 M, y 0,05% de polisorbato 20.
- Entre cada variante de la cistatina B, la superficie del sensor se regenera con NaOH 20 mM durante 30 segundos a un caudal de 10 l / min.
6.Immobilization del ligando MMTS-la papaína
La unión covalente de MMTS-la papaína para el análisis de la ACCP
CFCA se basa en la medición de la tasa de unión en las condiciones en que se limita la velocidad de difusión de moléculas de analito en la superficie (limitación de transporte masivo). Esto se ve favorecido por los niveles altos de inmovilización del ligando. Inmovilización de la papaína se MMTS-configurar y ejecutar utilizando el asistente de la inmovilización de software de control de Biacore X100.
- Activar la superficie de la celda de flujo 2 de la inyección de una mezcla de succinimida (NHS) y carbodiimida (EDC) por 7 minutos, con un caudal de 10 l / min. Una celda de flujo se deja sin cambios para ser utilizados como superficie de referencia.
- Inyectar MMTS-la papaína en 50μg/ml en tampón de acetato sódico a pH 4,5 durante 15 minutos a un caudal de 5 l / min.
- Etanolamina se inyecta durante 7 minutos con un caudal de 10 l / min para desactivar el resto de ésteres activos.
El procedimiento de acoplamiento todo debe resultar en ~ 3000 RU-MMTS papaína inmovilizada en la celda de flujo 2.
La unión covalente de MMTS-la papaína para el análisis cinético
El mismo procedimiento se utiliza para inmovilizar MMTS-la papaína para el análisis cinético, con la única diferencia es que en el análisis cinético, el nivel de inmovilización debe ser baja para evitar que la tasa de unión cada vez limitada por la difusión. Una celda de flujo se deja sin modificar en este paso y con el fin de ser utilizado como superficie de referencia.
- Después de la activación de la superficie con la mezcla de NHS y EDC como en 7.1, MMTS-la papaína se inyecta en 1μg/ml durante 50 segundos. Después de que la superficie se desactiva con una inyección de etanolamina como en el paso 7.3.
El nivel de acoplamiento debe ser de aproximadamente 50 RU después de este procedimiento.
7. La cistatina determinar la concentración de B utilizando el ensayo de CFCA
- El experimento de la ACCP está configurado de acuerdo con el asistente de la ACCP en X100 Biacore Package Plus. Aproximadamente el 10 nM proteína que se inyecta en los caudales de 10 y 100 l / min por duplicado en 48 segundos. La superficie se regenera con 20 mM NaOH entre cada ciclo.
- Incluyen las inyecciones en blanco para cada caudal.
- La concentración de proteína se determina a partir del enlace de datos (Figura 3) utilizando ªe CFCA función de la evaluación de X100 Biacore Además el software de evaluación de paquetes.
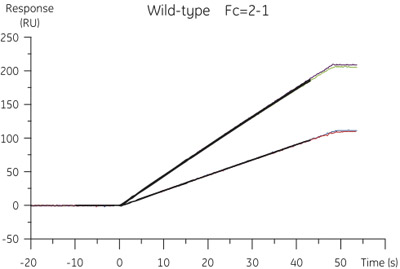
Figura 2. Los resultados del análisis de la ACCP B. cistatina de tipo salvaje Los datos específicos de unión de concentración fue extraído de la sensorgrams obtenidos en los caudales de 10 y 100 l / min.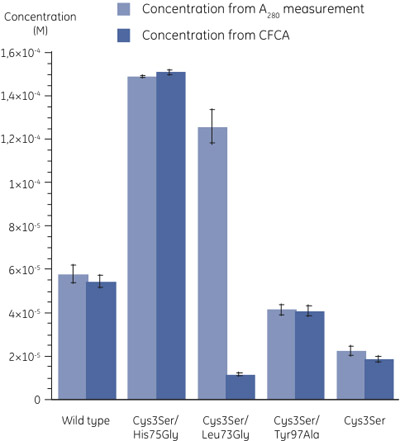
Concentraciones de la figura 3. De los mutantes determinado utilizando una medida 280 (n = 3) y la ACCP (n = 2). Los errores estándar se indican mediante barras de error. Los coeficientes de absorción molar se calcula como se describe en (6). Una gran diferencia en los valores de concentración de Cys3Ser/Leu73Gly mutante determinado por los dos métodos se pueden observar. - Al comparar la concentración determinada por una medición de 280 con que por CFCA, es claro que, mientras que en la mayoría de los casos la proteína se encuentra en plena actividad, para la variante Cys3Ser/Leu73Gly, la fracción de la proteína activa es muy baja (Figura 3). La siguiente tabla muestra las actividades de las variantes B cistatina relacionados con la concentración total de un 280.
Muestra CFCA en relación con un 280 (%) De tipo salvaje 94 Cys3Ser/His75Gly 101 Cys3Ser/Leu73Gly 9 Cys3Ser/Tyr97Ala 99 Cys3Ser 83
8. La medición de la cinética de la cistatina B unión a la papaína
- Basándose en las mediciones CFCA, preparar una serie de concentraciones de dos veces que van desde 2,5 a 40 nm para cada una de las variantes de la cistatina B.
- El experimento de cinética se ha configurado mediante el asistente de la cinética en X100 Biacore con el enfoque de ciclo único. La superficie no se regenera entre las inyecciones, pero después del final de cada ciclo de análisis utilizando 20 mM NaOH como solución de la regeneración.
- Configurar el experimento de manera que cada ciclo con la muestra está flanqueada por un ciclo en blanco, donde se inyecta buffer en lugar de muestra.
- Utilice la función de evaluación de la cinética en X100 Biacore para realizar automáticamente las sustracciones en blanco de referencia resta de datos antes de instalar un modelo de interacción de 1:1.
- Los datos experimentales obtenidos para la unión de las variantes B cistatina a la papaína se muestra en la Figura 4. La siguiente tabla resume la asociación y las constantes de disociación y la tasa de las constantes de disociación de equilibrio obtenidos en el análisis cinético.
Muestra k a (M -1 s -1) k d (s -1) K D (M) De tipo salvaje 1,8 x 10 6 0.41 x 10 -3 2.3 x 10 -10 Cys3Ser/His75Gly 1,1 x 10 6 1.7 x 10 -3 1,5 x 10 -9 Cys3Ser/Leu73Gly 1,1 x 10 6 23 x 10 -3 2,2 x 10 -8 Cys3Ser/Tyr97Ala 1.7 x 10 6 12 x 10 -3 7,1 x 10 -9 Cys3Ser 0,9 x 10 6 0.53 x 10 -3 5.8 x 10 -10 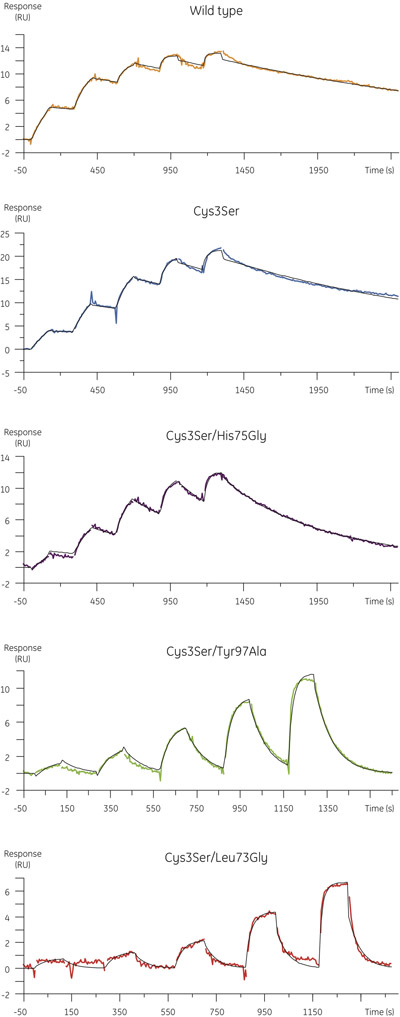
Figura 4. Perfiles cinéticos para las variantes B cistatina unión a MMTS-la papaína determina usando la cinética de un solo ciclo. Sensorgrams muestran en blanco y resta de datos de referencia con el ajuste cinético de un modelo de interacción 1:01 superpuestas en negro. - Mientras que las tasas de asociación de los mutantes son comparables a la de la proteína de tipo salvaje (Figura 5, panel superior), las constantes de velocidad de disociación de los mutantes pueden ser más altos en cerca de dos órdenes de magnitud, con un aumento correspondiente en el equilibrio de disociación constante (Figura 5, panel inferior).

Figura 5. Los cálculos de las constantes de velocidad y la afinidad se basaron en las concentraciones obtenidas a partir de la ACCP. Los cambios de k a, d k y K D, asociada con las mutaciones se representan en los gráficos. - Dado que la concentración de la muestra es un parámetro en el cálculo de la tasa constante de asociación de los datos experimentales, esimportante que es correcto. Utilizando la concentración basada en la medición de un 280 en lugar de la ACCP llevaría a la conclusión de que Leu73 es importante para la velocidad de asociación, que la sustitución parece ser el resultado de cerca de 10 veces más lenta que la asociación de las variantes B cistatina otros. Sin embargo, la concentración de unión específica medida por la ACCP es sólo el 10% del total de proteínas en este caso. Cuando esto se toma en cuenta este aspecto se pone de manifiesto que la tasa de asociación para Leu73 es similar a la de las otras variantes. Por lo tanto, CFCA permite la evaluación correcta de un k K y D lo que hace posible la interpretación adecuada del mecanismo de interacción.
Discusión
En este trabajo, cuatro mutantes y B de tipo salvaje cistatina fueron producidos con el fin de evaluar la importancia del lazo de unión segundo y C-terminal para la interacción entre la cistatina B y papaína. Este estudio demostró la ventaja y facilidad de uso de X100 Biacore para determinar la concentración y la unión específica para analizar la cinética de las interacciones proteína-proteína para entender la relación estructura-función. Hemos demostrado que la medición de la concentración de proteínas totales no revela las variantes de proteínas que han reducido la actividad de unión, que en este caso presenta un error de medición importante en la determinación de la afinidad de unión y la cinética. Las concentraciones de unión específica de las variantes de la cistatina B se determinaron utilizando CFCA con X100 Biacore. Usando las mediciones de concentración de CFCA como insumo para el análisis cinético resultó en una tasa fiable y constantes de afinidad, lo que permite la correcta interpretación del mecanismo de interacción.
Disminución de las afinidades de los mutantes se debe casi exclusivamente a un aumento de k d-valor, mientras que k se vio afectado un poco. Este comportamiento indica que tanto la segunda región lazo de unión y el extremo C-terminal no son importantes para el tipo de unión del inhibidor a la papaína. Por el contrario, contribuir a la afinidad de unión principalmente por mantener el inhibidor unido a la enzima una vez que el complejo se ha formado.
Divulgaciones
Materiales
| Name | Company | Catalog Number | Comments |
| Biacore™ X100 System | GE Healthcare | BR-1100-73 | http://www.biacore.com/lifesciences/products/systems_overview/x100/system_information/index.html |
| Biacore™ X100 Plus Package | GE Healthcare | BR-1007-98 | http://www.biacore.com/lifesciences/products/systems_overview/x100/system_information/index.html |
| Sensor Chip CM5 | GE Healthcare | BR-1000-14 | http://www.biacore.com/lifesciences/products/systems_overview/x100/system_information/index.html |
| Amine Coupling Kit | GE Healthcare | BR-1000-50 | |
| Acetate buffer pH 4.5, 50 ml | GE Healthcare | BR-1003-50 | |
| HBS-EP+ buffer 10X, 4 x 50 ml | GE Healthcare | BR-1008-26 | |
| Plastic Vials 11 mm | GE Healthcare | BR-1002-87 | |
| Rubber caps, type 2 | GE Healthcare | BR-1004-11 |
Referencias
- Christensen, L. L. H. Theoretical analysis of protein concentration determination using biosensor technology under conditions of partial mass transport limitation. Anal. Biochem. 249, 153-164 (1997).
- Sigmundsson, K., Mâsson, G., Rice, R., Beauchemin, N., Öbrink, B. Determination of active concentrations and association and dissociation rate constants of interacting biomolecules: an analytical solution to the theory for kinetic and mass transport limitations in biosensor technology and its experimental verification. Biochemistry. 41 (26), 8263-8276 (2002).
- Stubbs, M. T., Laber, B., Bode, W., Huber, R., Jerala, R., Lenarcic, B., Turk, V. The refined 2.4 A X-ray crystal structure of recombinant human stefin B in complex with the cysteine proteinase papain: a novel type of proteinase inhibitor interaction. EMBO J. 9 (6), 1939-1947 (1990).
- Pol, E., Björk, I. Importance of the second binding loop and the C-terminal end of cystatin B (Stefin B) for inhibition of cysteine proteinases. Biochemistry. 38 (32), 10519-10526 (1999).
- Björk, I., Pol, E., Raub-Segall, E., Abrahamson, M., Rowan, A. D., Mort, J. S. Differential changes in the association and dissociation rate constants for binding of cystatins to target proteinases occurring on N-terminal truncation of the inhibitors indicate that the interaction mechanism varies with different enzymes. Biochem. J. 299, 219-225 (1994).
- Pace, C. N., Vajdos, F., Fee, L., Grimsley, G., Gray, T. How to measure and predict the molar absorption coefficient of a protein. ProteinScience. 4 (11), 2411-2423 (1995).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados