Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Selección de los aptámeros de amiloide β-proteínas, el agente causante de la enfermedad de Alzheimer
En este artículo
Resumen
Los aptámeros son ribo-/deoxyribo-oligonucleotides corto seleccionado por In-vitro Métodos de la evolución basada en la afinidad de un objetivo específico. Los aptámeros son herramientas de reconocimiento molecular con la versatilidad de las aplicaciones terapéuticas, diagnósticas y de investigación. Demostramos métodos para la selección de aptámeros de amiloide β-proteínas, el agente causal de la enfermedad de Alzheimer.
Resumen
La enfermedad de Alzheimer (EA) es una enfermedad progresiva, dependiente de la edad, enfermedades neurodegenerativas con un curso insidioso que hace que su diagnóstico presintomático difícil 1. Diagnóstico del TDA definitiva se logra sólo postmortem, estableciendo así el diagnóstico presintomático, precoz de la EA es crucial para desarrollar y administrar tratamientos eficaces 2,3.
Β-amiloide, proteína (Aß) es fundamental para la patogénesis de la EA. Asambleas soluble, Aß oligoméricas se cree que afecta a la neurotoxicidad de la disfunción sináptica y la pérdida de neuronas en el año 4,5. Diversas formas de Aß soluble asambleas se han descrito, sin embargo, sus interrelaciones y la importancia de la etiología y la patogénesis de AD son complejos y no se entiende bien 6. Herramientas específicas para el reconocimiento molecular pueden desentrañar las relaciones entre los conjuntos de Aß y facilitar la detección y caracterización de estos conjuntos de principios en el curso de la enfermedad antes de aparecer los síntomas. Reconocimiento molecular comúnmente se basa en anticuerpos. Sin embargo, una clase alternativa de herramientas de reconocimiento molecular, aptámeros, ofrece importantes ventajas con respecto a los anticuerpos 7,8. Los aptámeros son oligonucleótidos generados por in-vitro de selección: la evolución sistemática de ligandos por enriquecimiento exponencial (SELEX) 9,10. SELEX es un proceso iterativo que, al igual que la evolución darwiniana, permite la selección, amplificación, el enriquecimiento y la perpetuación de una propiedad, por ejemplo, ávida, específica, de unión al ligando (aptámeros) o la actividad catalítica (ribozimas y ADNzimas).
Pesar de la aparición de aptámeros como herramientas de la biotecnología moderna y la medicina 11, han sido poco utilizados en el campo de amiloide. Pocos o ARN aptámeros ssDNA han sido seleccionados contra las distintas formas de proteínas priónicas (PrP) 12-16. Un aptámero ARN generados contra PrP bovina recombinante ha demostrado que reconocer bovina PrP-β de 17 años, soluble, oligómero, β-hoja-rica variante de conformación de larga duración PrP que forma fibrillas de amiloide 18. Aptámeros generado con las formas monoméricas y varios de β fibrilar se encontraron 2-microglobulina (β 2 m) para enlazar las fibrillas de ciertas proteínas amiloidogénica otros, además de las fibrillas de β 2 m 19. Ylera et al. describe aptámeros de ARN seleccionados en contra inmovilizado monoméricas Aβ40 20. Inesperadamente, estos aptámeros obligado fibrilar Aβ40. En conjunto, estos datos plantean varias cuestiones importantes. ¿Por qué aptámeros seleccionado frente a las proteínas monoméricas reconocer sus formas poliméricas? Podría aptámeros contra las formas monoméricas y / o de las proteínas oligoméricas amiloidogénica se obtiene? Para abordar estas cuestiones, hemos intentado seleccionar aptámeros para covalentemente estabilizado oligoméricas Aβ40 21 generados usando foto-inducido de entrecruzamiento de las proteínas sin modificar (picup) 22,23. Similar a los hallazgos previos 17,19,20, estos aptámeros reaccionaron con fibrillas de Aß y otras proteínas amiloidogénica probable que el reconocimiento de un amiloide potencialmente estructural común aptatope 21. A continuación, presentamos la metodología SELEX utilizados en la producción de estos aptámeros 21.
Protocolo
Parte 1: preparación de la proteína y el cross-linking
Inicialmente, la proteína utilizada para SELEX se trata previamente con 1,1,1,3,3,3-hexafluoro-2-propanol (HFIP) para obtener homogénea, global sin preparación, como se describió previamente 23. Este paso es necesario porque preformados agregados inducir la agregación de las proteínas de rápida amiloidogénica, resultando en una mala reproducibilidad experimental 24, y no son deseables para la selección de aptámeros de forma desagregada, no fibrilar formas de la proteína.
- Pesar 800 mg ~ (~ 180 nmol) puro Aβ40 usando una microbalanza. Transferir el péptido liofilizado en seco en un recipiente etiquetado, revestido con silicio, bajo adsorbente de 1,6 ml tubos de microcentrífuga.
- Disolver el péptido en el 100% 1,1,1,3,3,3-hexafluoro-2-propanol (HFIP, 400 l) para obtener 0,5 mM solución de péptidos como se describió previamente 23.
- Divida esta solución en cuatro alícuotas de 100 l de tal manera que cada tubo contiene aproximadamente 45 nmol Aβ40 nominalmente. Proceder a la eliminación de HFIP como hemos descrito anteriormente 23.
- Antes de solubilización de los péptidos HFIP tratados en el buffer de reacciones de reticulación, preparar el cross-linking y reactivos de enfriamiento. Pesar persulfato de amonio (APS, 228,2 MW g / mol) y preparar una solución de 40 mM en fosfato sódico 10 mM, pH 7,4. Mezclar con un agitar hasta que la solución es clara.
- Prepare una solución 2 mM de Tris (2,2-bipiridilo) dichlororuthenium (II) hexahidratado (RuBpy, MW 748,63 g / mol) en fosfato sódico 10 mM, pH 7,4. Mezclar con un vórtice y verificar su completa disolución. Proteger el tubo de la luz con papel de aluminio.
- Prepare el reactivo de enfriamiento. Pesar ditiotreitol (DTT, 154,5 MW g / mol) y se disuelven en agua destilada o en 10 mM fosfato de sodio, pH 7,4, a 1 M.
- Disolver el péptido HFIP tratados exactamente como se describe anteriormente 23,25 pero el objetivo de obtener ~ 60 mM solución de péptidos.
- Realizar picup para generar una mezcla de oligómeros Aβ40 como se describió anteriormente 23. Una reacción típica picup se lleva a cabo en un volumen de 20 l en la concentración final de proteínas, RuBpy, y APS de 30 M, de 0,05 mm y 1 mm, respectivamente 23. En este caso, la mezcla utilizada para picup contiene 108 proteínas l, 6 RuBpy l, 6 APS l, y 1 l de TDT y la concentración de proteínas, RuBpy, y APS son duplicó en relación con la reacción típica. Esto reduce el número de reacciones picup a realizar y aumenta la concentración de proteínas de la desalación.
Parte 2: Desalinización de la preparación de proteína
Antes de utilizar la proteína para SELEX, la desalinización se realiza para eliminar los reactivos de entrecruzamiento utilizado para picup. Esta mezcla de reacción picup contiene los reactivos de entrecruzamiento y ~ 55 proteínas M (concentración nominal).
- Retire la tapa superior de una columna de la desalación de 5 ml, el apoyo de la columna por un pie, coloque un vaso de precipitados debajo de la salida de la columna, y quitar el tapón de salida. Deja que el flujo de buffer de almacenamiento a través y recoger en el vaso.
- Equilibrar la columna en 10 mM de acetato de amonio, pH 8,3. Añadir 5 cama de resina volúmenes (25 ml) de este buffer en 3 ml de alícuotas a la columna y permitir que fluya a través.
- Mientras tanto, la etiqueta 8 bajo adsorbente, de silicio recubierto de 1,6 ml tubos y colocarlos en un bastidor de tubo.
- Después de la columna se equilibra, aplicar una alícuota de 0,5 hasta 0,7 ml de la mezcla de reacción picup por columna por uso individual de desalación (columnas se pueden lavar, almacenar y equilibrada para su posterior uso). Dejar que la mezcla de proteínas para empapar en la columna de resina y deseche el flujo continuo en el vaso.
- Coloque el tubo de la primera colección por debajo de la salida de la columna. Añadir 0,5 ml de solución tampón de acetato a la columna y recoger los primeros 0,5 ml de la fracción que fluye a través.
- Repita los pasos 2.4 y 2.5, y obtener hasta ocho fracciones de 0,5 ml en los tubos correspondientes.
- Número de otra serie de 8 bajo adsorbente, revestido con silicio, pequeños tubos de 0,6 ml y colocarlos en una rejilla. Etiqueta de otro tubo como "blanco". Saque 150 l de cada fracción y la transferencia en el etiquetado de 0,6 ml tubos. Utilice 20 l de SDS-PAGE análisis (Figura 1) como se muestra anteriormente 23.
- Transferencia de 150 l de acetato de amonio 10 mM, pH 8,3, en el tubo en blanco. Utilice este tubo para fijar el blanco en un espectrofotómetro.
- Medir la absorbancia en 130 l de cada fracción en λ = 280 nm en una cubeta de cuarzo.
- Después de la absorbancia se mide, se combinan las fracciones con mayor contenido de proteínas (por lo general las fracciones 3-5), mezclar suavemente con una pipeta, y mantener una alícuota de 10 l para el análisis de aminoácidos para determinar la concentración de proteína real (no mostrado).
- Dividir la muestra en varias alícuotas de proteínas nmol ~ 2 por tubo y liofilizar las muestras en un liofilizador.
- Después de la terminación de la liofilización, el tratamiento de las muestras con el 100% HFIP como antes.
- Guarde los tubos a -20 ° C. Use un tubo para cada ciclo de SELEX (abajo).
Parte 3: Ampliación de la síntesis al azar de la biblioteca ssDNA por PCR
La síntesis de la biblioteca ssDNA se utiliza aquí para SELEX incluyó a 49 nucleótidos al azar (A: T: G: C = 25:25:25:25%) flanqueado por las regiones constantes que comprende sitios de clonación (Bam HI, Eco RI) y un promotor T7, como descrito previamente 26.
- Para ampliar la biblioteca, crear un estándar de la reacción de PCR de la siguiente manera: 202 l de agua desionizada, 40 ml de ADN Taq 10x tampón de la polimerasa, 2 l plantilla de ADN de cadena simple (0,5 nmol), 120 l 25 mM MgCl 2, 28 l 10 mM desoxinucleósidos mezcla trifosfato (dNTPs), una imprimación l delantero (300 pmol), un cebador inverso l (300 pmol), y 4 l de Taq polimerasa.
- Realizar la reacción de PCR utilizando un termociclador con los siguientes valores: 5 min a 94 ° C para la desnaturalización inicial de 20 ciclos cada uno de los 94 ° C durante 30 segundos, 52 ° C durante 30 segundos para el recocido, 72 ° C durante 30 segundos para extensión, y la extensión final a 72 ° C durante 7 minutos.
- Tras la finalización de la reacción de PCR, purificar el producto de ADN amplificado utilizando el kit de purificación de Qiagen QiaQuick PCR de acuerdo con las instrucciones del fabricante. Por lo general, en estos experimentos la concentración y el rendimiento de ADN de 160 ± 10 ng / l en 50 microlitros.
- Compruebe que la cantidad de ADN después de la PCR por electroforesis en gel de agarosa al 2%.
Parte 4: Generación de los 32 P-RNA marcado por la transcripción in vitro
- Establecer la reacción de transcripción en un anillo con tapa, de 1,6 ml del tubo de acuerdo con las instrucciones del fabricante con algunas modificaciones de la siguiente manera: 20 l buffer 5x transcripción T7, 7,5 l cada uno de mM RATP 100, rGTP, rUTP, 1 l 100 mM RCTP, 2 l α 32 P-CTP (3000 Ci / mmol), 5.10 mg purificada plantilla de ADN (~ l 30-40 de producto purificado de ADN), 10 l mezcla de enzimas, y completar el volumen final de 100 l mediante la adición de agua libre de nucleasa.
- Mezclar suavemente la solución con una pipeta, se centrifuga la mezcla y se incuba a 37 ° C durante la noche.
- Al final de la reacción, la plantilla de ADN tiene que ser eliminado. Añadir RQ1 RNasa libre de DNasa a una concentración de 1 U / mg de ADN y se incuba durante 4 horas a 37 ° C para digerir la plantilla de ADN.
- Después de 4 horas, extraer el ARN mediante la adición de 1 volumen de citrato saturada de fenol: cloroformo: alcohol isoamílico (125:24:1, pH 4,7). Mezcle por un vórtice de ~ 1 min y centrifugar a 16.000 g durante 2 min.
- Transferir la fase superior acuosa a un tubo nuevo o descartar la fase final por aspiración con una micropipeta. Añadir 1 volumen de cloroformo: alcohol isoamílico (24:1), mezclar por un vórtice de 1 min y centrifugar como se describe en 4.4.
- Transferir la fase superior acuosa a un tubo nuevo o descartar la fase final por aspiración con una micropipeta. Cloroformo residual puede ser removido por realizar una vuelta rápida (10 segundos) en una microcentrífuga y la eliminación de la fase final con una micropipeta. En este paso, es más fácil para eliminar la fase final en lugar del sobrenadante.
- Para precipitar el ARN, añadir 0,1 volúmenes equivalentes de acetato de sodio 3M, pH 5,2, y un volumen equivalente de 2-propanol. Mezclar y colocar en un congelador a -20 ° C durante 15 min.
- Después de 15 minutos, girar a toda velocidad, preferiblemente en una microcentrífuga refrigerada a 4 ° C, durante 20-30 minutos para precipitar el producto de ARN.
- Aspirar el sobrenadante con cuidado, lavar el precipitado de ARN con 0,5 ml de etanol al 70%, se centrifuga a 4 ° C y descartar el etanol por aspiración.
- Transferir el tubo que contiene el precipitado de ARN a un bloque de calor seco y el pellet a 37 ° C durante 5 min.
- Disolver la muestra de ARN en tampón STE 150 mM, pH 8,0 (siempre con la ilustración ProbeQuant G-50 microcolumnas) o el agua libre de nucleasa a un volumen idéntico al de la reacción es decir, in-vitro de transcripción, 100 l (paso 4.1).
- Calentar el tubo que contiene el producto de RNA a 70 ° C durante 10 minutos en un bloque de calor y mezclar por un vórtice para facilitar la disolución del ARN.
- Centrifugar a velocidad máxima durante 1 minuto a temperatura ambiente.
- Mantener una alícuota de 1 l de ARN en una etiqueta de 0,6 ml de tubo para el recuento de centelleo y TBE-urea electroforesis en gel de poliacrilamida (Parte 6).
Parte 5: Eliminación de los nucleótidos no incorporados, la desalinización de ARN, y recuento de centelleo
Para eliminar los nucleótidos no incorporados, se utilizan dos columnas de G-50 de acuerdo a las instrucciones del fabricante.
- Invertir columnas y mezclar por un vórtice para volver a suspender la resina.
- Desprenda el cierre inferior de las columnas en la perforación con la herramienta de plástico en el kit y asegúrese de dejar la salida sin tocar. Afloje el tapón de un cuarto de vuelta y colocar las columnas en los tubos de recolección de limpia que en el G-50 Kit.
- Girar las columnas de los tubos de recolección a 730 g durante 1 minuto para extraer el tampón de almacenamiento.
- La transferencia de columnas a dos nuevos O-ring con tapa, de 1,6 ml y tubos de carga de 50 l de 32 P-etiquetados muestra de ARN por columna directamente sobre la resina sin perturbar la resina.
- Giran a 730 g durante 2 minutos para recoger el ARN purificado etiquetados. Después de este paso, descartar las columnas.
- La transferencia de un l de G-50-ARN purificado a un tubo de 0.6 ml para el recuento de centelleo y la electroforesis (Parte 6). Tienda de valores de ARN a -20 ° C hasta su uso para SELEX. Es deseable que el ARN se utiliza dentro de 2 días después de la etiqueta para evitar su degradación y pérdida de actividad.
- Utilice los dos 1-l alícuotas de ARN de 4,14 y 5,6 pasos para medir las cuentas por minuto (cpm / l) utilizando un contador de centelleo. Aquí, nosotros usamos la Triathler sobremesa contador de centelleo.
- La transferencia de los tubos que contienen la muestra de ARN en el interior del adaptador de plástico utilizado para contar P 32, siempre con Triathler. Coloque el tubo y el adaptador dentro de la cámara de recuento, cierre la tapa de la cámara, elegir 32 contando P, y pulse Iniciar para comenzar a contar.
- Calcular la etiqueta%, es decir la incorporación, la incorporación% de α-32 P CTP = (cpm / l para "G-50" muestra ÷ cpm / l de "TOTAL" de la muestra) x 100. Muestra total es la muestra antes del G-50 la purificación.
- Después de recuento de centelleo y el cálculo de la incorporación por ciento, se calcula el rendimiento de ARN y la actividad específica del ARN. Por ejemplo, si el recuento de un grupo de ARN antes (237.370 cpm / l) y después de la purificación del G-50 (191 330 cpm / l) la incorporación de rendimiento del 81%, entonces el siguiente se calculan antes de obtener el rendimiento de ARN:
Desde el paso 4.1, la cantidad de α-32 P CTP en 3000 Ci / mmol y 10 Ci / l es: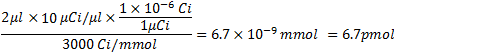 .
.
El monto de la etiqueta de CTP se utiliza: .
.
Monto total de la CTP se utiliza 100006.7pmol. El peso molecular de ARN se calcula de la siguiente manera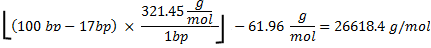
en 321,45 g / mol 27 es la masa media de rNTPs, 100 pb es el número de bases en la secuencia, y 17 pares de bases es el número de bases en el promotor T7 que no se transcriben. Sustracción de 61,96 g / mol de peso molecular de oligonucleótidos tiene en cuenta la eliminación de HPO 2 (63.98) y la adición de dos átomos de hidrógeno (2,02) 27. Porque, de los 4 rNTPs, CTP es el reactivo limitante, si todos los CTP se incorporara la masa teórica de ARN producido habría sido: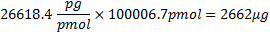 .
.
Sin embargo, debido a la incorporación del 81%, ahora esta masa es 2.156 mg. Cuentas por g de ARN sería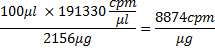
donde 100 l es el volumen de reacción de transcripción, y en términos de moles: 26618.4 mg / mmol x 8874 cpm / mg = 2.4x10 8 cpm / mol. A partir de aquí, la cantidad de ARN en nmol y su volumen adecuada que se utiliza para la incubación con la proteína se puede calcular, por ejemplo, 5 l de ARN que contienen ~ 4 nmol ARN, es decir,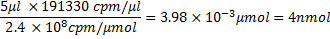
En estos experimentos, a 300 pmol de la proteína nmol nmol 1 y 4 a 100 nmol de ARN se utiliza 21.
Parte 6: Caracterización de los productos de ARN mediante electroforesis y autorradiografía
- Para preparar las muestras para la electroforesis, el uso de las alícuotas de 1 l de ARN antes y después del G-50 la purificación de los pasos de 4,14 y 5,6, respectivamente. Agregar 4 tampón STE y 5 l l 2x Novex TBE-Urea tampón de la muestra.
- El calor de las muestras a 70 º C durante 5 minutos como se recomienda para la electroforesis de ARN (calefacción se considera que son innecesarias porque la resolución de gel de las muestras de RNA con y sin calefacción es el mismo en esta configuración experimental).
- Reunir a un 6% de TBE-urea-gel de poliacrilamida en el aparato del gel de ejecución, llenar el interior y exterior con cámaras de 1x TBE Novex-tampón de urea. Lave los pocillos del gel prefabricados utilizando el buffer de una pipeta de 1 ml.
- Centrifugar los tubos de ARN y la carga de las muestras (10 en total l) con carga de gel-tips. Correr el gel a 180 V durante 50 min.
- Después de 50 min, desmontar el gel, rompiendo además el molde de plástico, retirar y desechar sólo el papel más corto del molde de gel, pero deja el respaldo de más de soporte para el gel.
- Limpie la superficie del área de trabajo con Decon (o descontaminante otros radiactividad apropiado) asegurarse de que todos los puntos de contaminación radiactiva se eliminan.
- Coloque dos capas de plástico Saran Wrap, envuelva el gel y el soporte de plástico en la envoltura de plástico de dos IMPLÍCITAS.
- Exponer el gel envueltos en plástico a una hoja de unutoradiography película de rayos X dentro de un cassette de exposición en el cuarto oscuro, bajo la luz de seguridad. Deja el cassette a -20 ° C durante 60-90 min.
- Desarrollar la película en el cuarto oscuro bajo la luz a salvo después de 60-90 minutos con un revelador automático de películas (Figura 2).
Parte 7: incubación de la proteína ARN y vinculante filtro
En primer lugar, el ARN y las proteínas se incuban en una solución y las secuencias de ARN que se unen a las proteínas se separan de las carpetas no. Como SELEX progreso ciclos, la unión de filtro dará una indicación de la proteína de unión a ARN de enriquecimiento.
- Incubar el ARN a 90 ° C durante 10 minutos para la desnaturalización y luego a temperatura ambiente durante 10 minutos para la renaturalización lento.
- Disolver la proteína del paso 2.12 en 8 l de NaOH 60 mM y añadir 36 l de nucleasa libre de agua destilada. Sonicar la mezcla durante 1 minuto y añadir 36 l 2x tampón de unión a ARN (20 mM Tris, 300 mM NaCl, 10 mM MgCl 2, pH 7,5).
- Mezcle la cantidad apropiada de ARN con 20 l de 10x tampón de unión a ARN y se alcance un volumen de 200 l mediante la adición de agua libre de nucleasa. Marque el tubo "control negativo".
- Mix 20 l de proteína y la cantidad deseada de ARN con 20 l de 10x tampón de unión a ARN y completar el volumen a 200 ml con agua libre de nucleasa. La etiqueta del tubo de "reacción".
- Mezclar e incubar los tubos durante 30 minutos a temperatura ambiente. Mientras tanto, preparar a los filtros y la configuración del filtro de unión para el siguiente paso.
- Adjunte una de 125 ml de brazo lateral frasco a una toma de aspiración. Coloque una pre-limpieza, soporte de vidrio poroso para el filtro en el matraz de brazo lateral.
- Equilibrar tres membranas de filtro en 3.2 ml de buffer 1x unión al ARN en una placa de 35x10 mm de Petri durante 10-15 min. El primer filtro se utiliza para ajustar la succión al vacío, la segunda se utilizará para el ARN-alone, control negativo vinculante de filtro, y el tercero se utiliza para la unión del filtro de la mezcla de ARN-proteína.
- Después de 30 minutos, se centrifuga la reacción de unión y los tubos de control a toda velocidad por 5 min a temperatura ambiente.
- A su vez en el vacío y el lugar de la primera membrana porosa en el cristal. Mediante una micropipeta, goteo 0,5 ml de tampón de unión al ARN en la membrana y ajustar el vacío para permitir el flujo lento de cada gota de búfer a través de la membrana.
- Lugar de la segunda membrana sobre el vidrio poroso y con la misma velocidad de flujo, aplicar el control negativo-en la membrana.
- Aplicar 4x0.5 ml alícuotas de 1x tampón de unión a ARN para lavar la membrana y descartar el filtrado. Tenga en cuenta que si antes de la limpieza que se desea, el flujo continuo se mantiene para la extracción de RNA. Pre-limpieza elimina las secuencias de ARN que se unen al filtro.
- Quitar la membrana de control negativo y el lugar en un correspondiente marcado con 1,6 ml de tubos para recuento de centelleo.
- Vuelva a colocar el vidrio poroso con una pre-limpieza segundo soporte de vidrio poroso.
- Lugar el tercer disco en el vidrio poroso y aplicar la mezcla de reacción. Lave el filtro como en el paso 7.11 y deseche el flujo continuo. El número de lavados se puede aumentar en los ciclos posteriores SELEX para aumentar el rigor de las condiciones de SELEX.
- Retire el disco de filtro y coloque en un tubo de 1.6 ml con la etiqueta "mezcla de reacción" y mantener para recuento de centelleo.
- Realizar recuento de centelleo como en el paso 5.8 y tomar nota de la cuenta para los filtros respectivos.
- Calcular el nivel de la radioactividad del filtro con destino en comparación con la cantidad total de radiactividad aplicada a las membranas (vinculante%). Esto le dará una indicación de enriquecimiento de unión a medida que avanza SELEX.
Parte 8: extracción de ARN de los filtros
ARN se extrae de los filtros para obtener las secuencias que se unen a la proteína. Estas secuencias se amplifican para el ciclo de SELEX siguiente.
- Después de recuento de centelleo, quitar el enlace de reacción filtro del tubo (desde el paso 7.15) y ponerlo en un lugar limpio, seco, 35x10 mm, una placa de Petri.
- Utilice un paño limpio de bisturí y unas pinzas para cortar la membrana en trozos pequeños.
- Usando las pinzas, sustituir las piezas cortadas de la membrana en el mismo tubo marcado en el paso 8.1.
- Añadir 400 l tampón de elución (7 M urea, 3 mM EDTA, 100 mM de citrato sódico, pH 5,0) e incubar el tubo a 95 ° C durante 10 min.
- Centrifugar el tubo a una velocidad superior a la temperatura ambiente, aspirar y recoger la mantequilla de extracción en un tubo nuevo etiquetado.
- Medir el recuento de la radiactividad que queda en el tubo que contiene las piezas de la membrana por recuento de centelleo para evaluar la eficacia de la extracción.
- Repita el proceso de extracción (8.4 a 8.6 pasos) tres veces. La eficacia después de 3 extracciones suele ser ~ 95-96%.
- En los tubos que contienen los extractos de ARN, añadir un volumen (400 l) de citrato-saturada (pH 4,7) fenol: cloroformo: alcohol isoamílico (125:24:1). Mezcle por un vórtice de ~ 1 min y centrifugadoge a 16.000 g durante 2 min.
- Transferir la fase superior acuosa a un tubo nuevo o descartar la fase final por aspiración con una micropipeta.
- Añadir 1 volumen de cloroformo: alcohol isoamílico (24:1), mezclar por un vórtice de 1 min y centrifugar a 16.000 g durante 2 min.
- Transferir la fase superior acuosa a un tubo nuevo o descartar la fase final por aspiración con una micropipeta.
- Para precipitar el ARN, añadir 0,1 volúmenes de acetato de sodio 3 M, pH 5,2, el glucógeno 3-4 l (10 mg / l) como el ARN coprecipitant, y un volumen equivalente de 2-propanol. Mezclar y colocar en un congelador a -20 ° C durante la noche.
- Girar a toda velocidad, preferiblemente en una microcentrífuga a 4 ° C, durante 20-30 minutos para precipitar el ARN del paso 8.12.
- Después de la centrifugación, el ARN se separa en una fase líquida que es apenas visible. Aspirar el sobrenadante con cuidado, sin desalojar a esta fase de precipitación apenas visible en la parte inferior del tubo.
- Lave el ARN "pellet" con 0,5 ml de etanol al 70%, se centrifuga durante 5 minutos a velocidad máxima a 4 ° C y descartar el etanol por aspiración sin desplazar la fase de precipitación apenas visible.
- Disolver el precipitado de ARN en 50 l tampón STE y proceder a la purificación de G-50 como en el paso 5.
Parte 9: La transcripción inversa y PCR para la continuación de los ciclos de SELEX
Para continuar con el siguiente ciclo de SELEX, el ARN tiene que ser la transcripción inversa a ADN y amplificación por PCR.
- En 5 etiquetados 0,6 ml tubos, mezcle 3 l de la purifica, G-50-desalado ARN con 2 l de ocho veces diluido en primer inversa. "Control negativo." Etiqueta de un tubo
- Incubar la mezcla a 70 ° C durante 5 minutos, y luego en hielo durante otros 5 minutos para permitir la hibridación de los cebadores para el ARN.
- A esta mezcla sobre el hielo, añadir 6,4 l agua libre de nucleasa, 4 l ImProm-II tampón de reacción 5x, 1.6 l 25 mM MgCl 2, 1 l 10 mM de mezcla dNTP, un inhibidor de la ribonucleasa RNasein l, 1 l ImProm-II de la transcriptasa inversa sumando un total de 15 microlitros.
- Para el control negativo, añadir 7,4 l de nucleasa libre de agua y dejar de lado la transcriptasa inversa. Marque los tubos en consecuencia. El control negativo se incluye para verificar que la contaminación de ADN de un ciclo anterior no se amplía para el ciclo de SELEX siguiente. Este paso pone a prueba la eficacia de la digestión de la plantilla de ADN por RQ1 RNasa libre de DNasa en el paso 4.3.
- Use un termociclador para la incubación de la mezcla de reacción a 25 ° C durante 5 min, 42 ° C durante 1 hora para el recocido y la extensión del ADN primer capítulo, respectivamente, seguido de 70 ° C durante 15 min para inactivar la transcriptasa inversa.
- Después de la reacción de transcripción inversa, establecer la mezcla de PCR. Añadir 30 l de nucleasa libre de agua, 10 l de 10x tampón Taq, 30 l 25 mM MgCl 2, 7 l 10 mM de mezcla dNTP, 1 l de cada cebador, y una polimerasa Taq l en todos los tubos.
- Ejecute el programa de PCR como en la parte 3 de 9-14 ciclos.
- El producto se purifica el ADN como en el paso 3.3.
- Mix 8 l de producto de ADN con 2 l colorante de carga de ADN y electroforesis en agarosa al 2% a 100 V durante 15-20 minutos (Figura 3).
- Producto de ADN está listo para ser transcrito a ARN marcado como en la parte 4 y se utiliza para el siguiente ciclo de SELEX.
Parte 10: Resultados de Representante
En los experimentos SELEX, la naturaleza de Aβ40 oligómeros utilizarse como el destino, la calidad de ARN para cada ciclo, y con éxito la extracción de RNA y la amplificación después de cada ciclo son importantes. Hemos utilizado picup para generar una mezcla de oligómeros Aβ40 SELEX después de la depuración y eliminación de la reticulación reactivos. Los experimentos de la desalación se describe en la Parte 2 por lo general conducen a la pérdida de proteínas 50-55%. La cantidad y calidad de proteínas se pueden evaluar mediante medidas de absorbancia (λ = 280) y SDS-PAGE (Figura 1). El perfil de absorbancia media de Aβ40 eluidos de cinco experimentos individuales superponer un típico perfil de SDS-PAGE de Aβ40 eluye en uno de esos experimentos se muestran en la Figura 1. Los datos muestran que la proteína consistente eluye de la columna de las fracciones 3-5 y los reactivos de entrecruzamiento eluir después de la fracción 6 (absorbancia mayor en la fracción 7, Figura 1). SDS-PAGE se muestra la típica distribución de oligómeros Aβ40 22. Esta distribución es reproducible después de las fracciones proteicas fueron liofilizadas (2,11), tratados con HFIP (2,11), resolubilizado (7,1), y re-analizados por SDS-PAGE.
Integridad del ARN para cada ciclo de SELEX también es importante para la progresión de SELEX iterativo, especialmente cuando nucleasa susceptibles ribo-oligonucleótidos se utilizan. Después de la amplificación del ARN y el etiquetado (Parte 4), la calidad de ARN marcado puede ser evaluada por electroforesis en gel de TBE-urea-poliacrilamida. Un perfil típico de un intacta la etiqueta del producto antes y después de ARN G-50 de purificación (PArt. 5) se muestra en la Figura 2.
Después de cada ciclo de SELEX, el ARN se extrae de la membrana después de filtrar vinculante (Parte 8) y la transcripción inversa (Parte 9) en el ADN para la amplificación por PCR (Paso 9.5). La plantilla de ADN de un ciclo anterior se utiliza para generar 32 P-RNA marcado (Parte 4) para el próximo ciclo. Si alguna plantilla de la contaminación de ADN de un ciclo anterior persiste en el etiquetado de productos del ARN después de las reacciones de transcripción in vitro (Parte 4), la eficiencia de los ciclos de SELEX se reducirá, exigiendo más ciclos. Para controlar esto, después de cada ciclo de SELEX y la correspondiente transcripción inversa-reacción de PCR, electroforesis de agarosa se lleva a cabo (9.12). Ausencia de productos de ADN en los tubos de reacción de control negativo (9,4) indica eliminación con éxito de la plantilla de ADN procedentes de un ciclo de SELEX anterior (Figura 3). Si la amplificación del ADN por PCR se observa en el tubo de control negativo, se recomienda que la duración de la incubación con RQ1 DNasa (4.3) se prolonga. El fabricante recomienda la duración de la incubación con RQ1 DNasa es de 15 minutos, sin embargo, encontramos que ya incubaciones (4-5 h) se requiere para eliminar totalmente la plantilla de ADN (Figura 3).
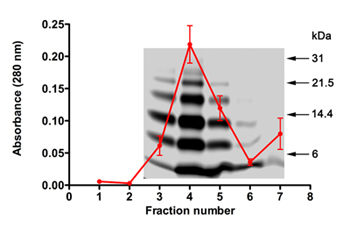
Figura 1. SDS-PAGE y el perfil de absorción de picup generado, desaladas Aβ40 oligómeros. El perfil de absorción de 5 columnas individuales de desalación se promedian y se superpone en un gel representativo. Marcadores de peso molecular se muestran a la derecha.

Figura 2. TBE-urea-poliacrilamida electroforesis en gel de ARN producto antes y después del G-50 la purificación. La dirección de migración del producto ARN del cátodo al ánodo, como se indica.
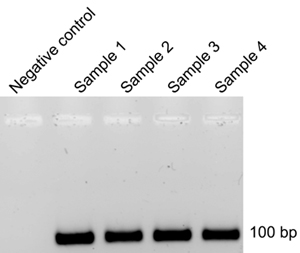
Figura 3. Electroforesis en gel de agarosa de los productos de ADN después de la transcripción inversa y amplificación por PCR.
Discusión
El punto de partida del proceso SELEX es la síntesis de oligonucleótidos de una biblioteca al azar por lo general con 10 12 -10 15 secuencias. En el ADN SELEX, esta biblioteca se utiliza inmediatamente después de una piscina ssDNA se genera, mientras que en el ARN SELEX, ha demostrado aquí, la biblioteca de ADN de cadena simple se convierte primero en un grupo de ARN enzimáticamente por la transcripción in vitro. Entonces, SELEX se realiza iterativamente mediante el cual cada ciclo c...
Divulgaciones
Agradecimientos
Este trabajo fue apoyado por becas AG030709 de NIH / NIA y 07-65798 del Departamento de Salud de California. Reconocemos Margaret M. Condron para la síntesis de péptidos y análisis de aminoácidos, la Dra. Elizabeth F. Neufeld para ayudar y apoyar a los pasos iniciales del proyecto, el Dr. Chi-Hong Chen B. de proporcionar apoyo y reactivos, y el Dr. Andrew D. . Ellington útil para los debates.
Materiales
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| Aβ40 | UCLA Biopolymers Laboratory | Lyophilized powder | ||
| MX5 Automated-S Microbalance | Mettler Toledo | |||
| Silicon-coated, 1.6-ml tubes | Denville Scientific | C19033 or C19035 | ||
| 1,1,1,3,3,3-hexafluoro-2-propanol (HFIP) | TCI America | H0424 | Use in a fume hood. | |
| Ammonium persulfate | Sigma | A-7460 | Vortex until the solution is clear. APS is prepared freshly each time and should be used within 48 h. | |
| Tris(2,2-bipridyl)dichlororuthenium(II) hexahydrate | Sigma | 224758-1G | Vortex until the solution is clear. Cover the RuBpy tube with foil to protect the reagent from ambient light. RuBpy is prepared freshly each time and should be used within 48 h. | |
| Dithiothreitol (DTT) | Sigma | 43815 | ||
| D-Salt™ Excellulose™ desalting columns | Thermo Scientific | 20449 | ||
| Ammonium acetate | Fisher Scientific | A637-500 | ||
| Silicon-coated, 0.6-ml tubes | Denville Scientific | C19063 | ||
| Novex Tricine Gels (10–20%) | Invitrogen | EC6625B0X | 10-well; mini size (8 cm X 8 cm); 25 μl loading volume per well; separation range 5 kDa to 40 kDa | |
| Quartz cuvette | Hellma | 105.250-QS | ||
| Beckman DU 640 spectrophotometer | Beckman | |||
| ssDNA library | Integrated DNA Technologies | Custom-ordered | The library was designed to contain 49 random nucleotides flanked by two constant regions containing primer-binding and cloning sites: 5'-TAA TAC GAC TCA CTA TAG GGA ATT CCG CGT GTG C (N:25:25:25:25%) (N)49 G TCC GTT CGG GAT CCT C-3' | |
| Taq DNA polymerase | USB Corporation | 71160 | Recombinant Thermus aquaticus DNA Polymerase supplied with 10× PCR Buffer and a separate tube of 25 mM MgCl2 for routine PCR. | |
| PCR Nucleotide Mix, 10 mM solution | USB Corporation | 77212 | (10 mM each dATP, dCTP, dGTP, dTTP) | |
| Forward primer | Integrated DNA Technologies | Custom-ordered | 5'-TAA TAC GAC TCA CTA TAG GGA ATT CCG CGT GTG C-3' | |
| Reverse primer | Integrated DNA Technologies | Custom-ordered | 5'-GAG GAT CCC GAA CGG AC-3' | |
| Thermal cycler | Denville Scientific | Techne TC-312 | ||
| QIAquick PCR Purification Kit (50) | QIAGEN | 28104 | ||
| Agarose | Denville Scientific | CA3510-8 | ||
| Conical, sterile 1.6-ml tubes with caps attached with O-rings | Denville Scientific | C19040-S | ||
| RiboMAX™ Large Scale RNA Production System–T7 | Promega | P1300 | The kit contains: 120 μl Enzyme Mix (RNA polymerase, recombinant RNasin® ribonuclease inhibitor and recombinant inorganic pyrophosphatase); 240 μl transcription 5 buffer; 100 μl each of 4 rNTPs, 100 mM; 110 U RQ1 RNase-free DNase, 1 U/μl; 10 μl linear control DNA, 1 mg/ml; 1 ml 3M sodium acetate (pH 5.2); 1.25 ml nuclease-Free water | |
| α-32P-cytidine 5'-triphosphate, 250 μCi (9.25 MBq), | Perkin Elmer | BLU008H250UC | Specific Activity: 3000 Ci (111 TBq)/mmol, 50 mM Tricine (pH 7.6) | |
| Citrate-saturated phenol:chloroform:isoamyl alcohol (125:24:1, pH 4.7) | Sigma (Fluka) | 77619 | ||
| Chloroform:Isoamyl alcohol (24:1) | Sigma | C0549 | ||
| Absolute ethanol for molecular biology | Sigma | E7023 | ||
| Z216-MK refrigerated microcentrifuge | Denville Scientific | C0216-MK | ||
| illustra ProbeQuant™ G-50 Micro Columns | GE Healthcare | Obtained from Fisher Scientific (45-001-487) | Prepacked with Sephadex™ G-50 DNA Grade and pre-equilibrated in STE buffer containing 0.15% Kathon as Biocide | |
| Triathler Bench-top Scintillation counter | Hidex Oy, Turku, Finland | Triathler LSC Model: 425-034 | ||
| Novex® TBE-Urea Sample Buffer (2×) | Invitrogen | LC6876 | ||
| 6% TBE-Urea Gels 1.0 mm, 10 wells | Invitrogen | EC6865BOX | ||
| Novex® TBE Running Buffer (5×) | Invitrogen | LC6675 | ||
| Radioactivity decontaminant | Fisher Scientific | 04-355-67 | ||
| Gel-loading tips | Denville Scientific | P3080 | ||
| XCell SureLock Mini-Cell | Invitrogen | EI0001 | XCell SureLock Mini-Cell | |
| Autoradiography film | Denville Scientific | E3018 | Use in complete darkness | |
| Autoradiography film, Hyperfilm™ ECL | Amersham Biosciences | RPN3114K | Can be used under red safe light. | |
| Membrane discs | Millipore | GSWP02500 | Mixed cellulose ester, hydrophilic, 0.22-μm disc membranes | |
| Fritted glass support base for 125-ml flask | VWR | 26316-696 | ||
| Petri dishes | Fisher Scientific | 08-757-11YZ | ||
| Urea | Fisher Scientific | AC32738-0050 | ||
| EDTA | Fisher Scientific | 118430010 | ||
| Glycogen | Sigma | G1767 | ||
| 2-Propanol for molecular biology | Sigma | I9516 | ||
| Recombinant RNase inhibitor | USB Corporation | 71571 | ||
| ImProm-II™Reverse Transcription System | Promega | A3802 | ||
| Recombinant RNase inhibitor | USB Corporation | 71571 | ||
| RapidRun™ Loading Dye | USB Corporation | 77524 |
Referencias
- Monien, B. H., Apostolova, L. G., Bitan, G. Early diagnostics and therapeutics for Alzheimer's disease-how early can we get there. Expert. Rev. Neurother. 6, 1293-1306 (2006).
- Nestor, P. J., Scheltens, P., Hodges, J. R. Advances in the early detection of Alzheimer's disease. Nat. Med. 10, S34-S41 (2004).
- Kawas, C. H. Clinical practice. Early Alzheimer's disease. N. Engl. J. Med. 349, 1056-1063 (2003).
- Haass, C., Selkoe, D. J. Soluble protein oligomers in neurodegeneration: lessons from the Alzheimer's amyloid β-peptide. Nat. Rev. Mol. Cell. Biol. 8, 101-112 (2007).
- Kirkitadze, M. D., Bitan, G., Teplow, D. B. Paradigm shifts in Alzheimer's disease and other neurodegenerative disorders: the emerging role of oligomeric assemblies. J. Neurosci. Res. 69, 567-577 (2002).
- Rahimi, F., Shanmugam, A., Bitan, G. Structure-function relationships of pre-fibrillar protein assemblies in Alzheimer's disease and related disorders. Curr. Alzheimer Res. 5, 319-341 (2008).
- Jayasena, S. D. Aptamers: an emerging class of molecules that rival antibodies in diagnostics. Clin. Chem. 45, 1628-1650 (1999).
- Bunka, D. H., Stockley, P. G. Aptamers come of age - at last. Nat. Rev. Microbiol. 4, 588-596 (2006).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Lee, J. F., Stovall, G. M., Ellington, A. D. Aptamer therapeutics advance. Curr. Opin. Chem. Biol. 10, 282-289 (2006).
- Weiss, S. RNA aptamers specifically interact with the prion protein PrP. J. Virol. 71, 8790-8797 (1997).
- Bibby, D. F. Application of a novel in vitro selection technique to isolate and characterise high affinity DNA aptamers binding mammalian prion proteins. J. Virol. Methods. 151, 107-115 (2008).
- Rhie, A. Characterization of 2'-fluoro-RNA aptamers that bind preferentially to disease-associated conformations of prion protein and inhibit conversion. J. Biol. Chem. 278, 39697-39705 (2003).
- King, D. J., Safar, J. G., Legname, G., Prusiner, S. B. Thioaptamer interactions with prion proteins: sequence-specific and non-specific binding sites. J. Mol. Biol. 369, 1001-1014 (2007).
- Proske, D. Prion-protein-specific aptamer reduces PrPSc formation. ChemBioChem. 3, 717-725 (2002).
- Murakami, K., Nishikawa, F., Noda, K., Yokoyama, T., Nishikawa, S. Anti-bovine prion protein RNA aptamer containing tandem GGA repeat interacts both with recombinant bovine prion protein and its β isoform with high affinity. Prion. 2, 73-80 (2008).
- Luhrs, T., Zahn, R., Wuthrich, K. Amyloid formation by recombinant full-length prion proteins in phospholipid bicelle solutions. J. Mol. Biol. 357, 833-841 (2006).
- Bunka, D. H. Production and characterization of RNA aptamers specific for amyloid fibril epitopes. J. Biol. Chem. 282, 34500-34509 (2007).
- Ylera, F., Lurz, R., Erdmann, V. A., Furste, J. P. Selection of RNA aptamers to the Alzheimer's disease amyloid peptide. Biochem. Biophys. Res. Commun. 290, 1583-1588 (2002).
- Rahimi, F., Murakami, K., Summers, J. L., Chen, C. H., Bitan, G. RNA aptamers generated against oligomeric Aβ40 recognize common amyloid aptatopes with low specificity but high sensitivity. PLoS ONE. 4, e7694-e7694 (2009).
- Bitan, G., Lomakin, A., Teplow, D. B. Amyloid β-protein oligomerization: prenucleation interactions revealed by photo-induced cross-linking of unmodified proteins. J. Biol. Chem. 276, 35176-35184 (2001).
- Rahimi, F., Maiti, P., Bitan, G. Photo-induced cross-linking of unmodified proteins (PICUP) applied to amyloidogenic peptides. J. Vis. Exp. , (2009).
- Bitan, G., Fradinger, E. A., Spring, S. M., Teplow, D. B. Neurotoxic protein oligomers-what you see is not always what you get. Amyloid. 12, 88-95 (2005).
- Bitan, G. Structural study of metastable amyloidogenic protein oligomers by photo-induced cross-linking of unmodified proteins. Methods Enzymol. 413, 217-236 (2006).
- Chen, C. H., Chernis, G. A., Hoang, V. Q., Landgraf, R. Inhibition of heregulin signaling by an aptamer that preferentially binds to the oligomeric form of human epidermal growth factor receptor-3. Proc. Natl. Acad. Sci. USA. 100, 9226-9231 (2003).
- Adams, D. S. . Lab math: a handbook of measurements, calculations, and other quantitative skills for use at the bench. , (2003).
- Gopinath, S. C. Methods developed for SELEX. Anal. Bioanal. Chem. 387, 171-182 (2007).
- Takahashi, T., Tada, K., Mihara, H. RNA aptamers selected against amyloid β-peptide (Aβ) inhibit the aggregation of Aβ. Mol. Biosyst. 5, 986-991 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados