É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Seleção de Aptamers para proteína β-amilóide, o agente causador da doença de Alzheimer
Neste Artigo
Resumo
Aptâmeros são ribo-/deoxyribo-oligonucleotides curtas selecionados por In-vitro Métodos de evolução com base na afinidade por um alvo específico. Aptâmeros são ferramentas de reconhecimento molecular com o versátil aplicações terapêuticas, de diagnóstico e pesquisa. Demonstramos métodos de selecção dos aptâmeros para a proteína β-amilóide, o agente causador da doença de Alzheimer.
Resumo
Doença de Alzheimer (DA) é uma progressiva, dependente da idade desordem, neurodegenerativas com um curso insidioso que torna seu diagnóstico pré-sintomático difícil 1. AD diagnóstico definitivo é obtido post-mortem só, estabelecendo assim o diagnóstico, pré-sintomático precoce da AD é crucial para o desenvolvimento e administração de terapias eficazes 2,3.
Β-amilóide-proteína (Aâ) é central para a patogênese AD. Solúvel, oligomérica assembléias Aâ Acredita-se que afetam a disfunção subjacente neurotoxicidade sináptica e perda de neurônios na AD 4,5. Várias formas de assembléias Aâ solúveis têm sido descritos, no entanto, suas inter-relações e relevância para a etiologia e patogenia AD são complexas e não bem compreendido 6. Ferramentas específicas de reconhecimento molecular pode desvendar as relações entre os conjuntos Aâ e facilitar a detecção e caracterização destes conjuntos no início do curso da doença antes que os sintomas surjam. Reconhecimento molecular comumente se baseia em anticorpos. No entanto, uma classe alternativa de ferramentas de reconhecimento molecular, aptâmeros, oferece vantagens importantes em relação aos anticorpos 7,8. Aptâmeros são oligonucleotides gerados por in-vitro de seleção: a evolução sistemática de ligantes por enriquecimento exponencial (SELEX) 9,10. SELEX é um processo iterativo que, semelhante à evolução darwinista, permite a amplificação de seleção, o enriquecimento, ea perpetuação de uma propriedade, por exemplo, ávido, específico, ligante de ligação (aptâmeros) ou atividade catalítica (ribozimas e DNAzymes).
Apesar da emergência de aptâmeros como ferramentas em biotecnologia moderna e da medicina 11, eles têm sido subutilizados no campo amilóide. RNA poucos ou aptâmeros ssDNA foram selecionados contra diversas formas da proteína príon (PrP) 12-16. Um aptamer RNA recombinante contra gerada PrP bovina foi mostrado para reconhecer bovina PrP-β 17, um solúvel, oligomérica, β-folhas ricas variante conformacional de full-length PrP que forma fibrilas amilóides 18. Aptâmeros gerados usando formas monomérica e vários de β fibrilar 2-microglobulina (β 2 m) foram encontradas para ligar fibrilas de algumas outras proteínas amiloidogênicas além β 2 m fibrilas 19. Ylera et al. descrito aptâmeros RNA selecionada imobilizada contra monomérica Aβ40 20. Inesperadamente, esses aptâmeros ligado fibrilar Aβ40. Em conjunto, esses dados levantam várias questões importantes. Por que aptâmeros selecionados contra proteínas monoméricas reconhecer suas formas poliméricas? Poderia aptâmeros contra as formas monoméricas e / ou de proteínas oligoméricas amiloidogênicas ser obtido? Para responder a essas questões, buscou-se selecionar aptâmeros para covalentemente estabilizado oligomérica Aβ40 21 gerados usando foto-induzida cross-linking de proteínas não modificadas (picup) 22,23. Similar aos resultados anteriores 17,19,20, esses aptâmeros reagiu com fibrilas de Aâ e várias outras proteínas amiloidogênicas provável o reconhecimento de um amilóide potencialmente estrutural comum aptatope 21. Aqui, apresentamos a metodologia SELEX utilizados na produção destes aptâmeros 21.
Protocolo
Parte 1: preparação de proteína e cross-linking
Inicialmente, a proteína usada para SELEX é pré-tratados com 1,1,1,3,3,3-hexafluoro-2-propanol (HFIP) para obter homogênea, agregar-free preparações, como descrito anteriormente 23. Esta etapa é necessária porque pré-formados agregados induzir a agregação rápida de proteínas amiloidogênicas, resultando em reprodutibilidade experimental pobres 24, e são indesejáveis para a seleção de aptâmeros para desagregados, não-fibrilar formas da proteína.
- Pesar ~ mcg 800 (~ 180 nmol) pura Aβ40 usando uma microbalança. Transferência do peptídeo seco liofilizado em rotulados, revestido a silicone, de baixo adsorvente tubos de 1,6 ml de microcentrífuga.
- Dissolver o peptídeo em 100% 1,1,1,3,3,3-hexafluoro-2-propanol (HFIP, 400 mL) para obter solução 0,5 mM de peptídeo como descrito anteriormente 23.
- Divida essa solução em quatro 100-mL alíquotas de tal forma que cada tubo contém ~ 45 nmol Aβ40 nominalmente. Proceder à remoção de HFIP como descrito anteriormente 23.
- Antes de solubilizar os peptídeos HFIP tratados em tampão de cross-linking reações, preparar o cross-linking e reagentes de extinção. Pesar persulfato de amônio (APS, 228,2 MW g / mol) e preparar uma solução de 40 mM em fosfato de sódio 10 mM, pH 7.4. Misture usando um vortex até que a solução é clara.
- Prepare dois solução mM de Tris (2,2-bipyridyl) dichlororuthenium hexa (II) (RuBpy, MW 748,63 g / mol) em 10 mM fosfato de sódio, pH 7.4. Misture usando um vortex e verificar a completa dissolução. Proteger o tubo de luz usando uma folha de alumínio.
- Prepare o reagente de têmpera. Pesar ditiotreitol (DTT, 154,5 MW g / mol) e dissolver em água deionizada ou em 10 mM fosfato de sódio, pH 7,4, a 1 M.
- Dissolver o peptídeo HFIP-tratados exatamente como descrito anteriormente 23,25, mas visam obter ~ 60 mM solução peptídeo.
- Executar picup para gerar uma mistura de Aβ40 oligoméricos como descrito anteriormente 23. Uma reação típica picup é realizada em um volume de 20 l, onde a concentração final de proteína, RuBpy e APS são 30 mM, 0,05 mM e 1 mM, respectivamente 23. Aqui, a mistura usada para picup contém 108 mL de proteína, 6 RuBpy mL, 6 APS mL e 1 mL TDT ea concentração de proteína, RuBpy e APS são todos dobrou em relação à reação típica. Isto reduz o número de reações picup a ser realizado e aumenta a concentração de proteína para a dessalga.
Parte 2: Dessalinização a preparação de proteínas
Antes de utilizar a proteína para SELEX, dessalinização é realizada para remover os reagentes de ligação cruzada utilizado para picup. Esta mistura de reação picup contém os reagentes cross-linking e proteína M ~ 55 (concentração nominal).
- Remova a tampa superior de uma coluna de dessalinização de 5 ml, suporte da coluna por uma posição, coloque um copo abaixo da saída da coluna, e retirar a ficha de saída. Permitir o fluxo de buffer de armazenamento através de e recolher no copo.
- Equilibrar a coluna em 10 mM acetato de amónio, pH 8.3. Adicionar 5 resina cama-volumes (25 ml) deste buffer em 3-ml alíquotas para a coluna e permitir que ele flua.
- Enquanto isso, etiqueta 8 low-adsorvente, silício revestido de 1,6 ml tubos e colocá-los em um rack de tubos.
- Depois que a coluna é equilibrada, aplicar uma alíquota de 0,5-0,7 ml da mistura de reação picup por coluna por uso único dessalinização (colunas podem ser lavados, armazenados e equilibrada para mais tarde usa). Permitir a mistura de proteína de molho na resina coluna e descartar o fluxo através do copo.
- Coloque o tubo de coleta abaixo da primeira saída da coluna. Adicionar 0,5 ml de tampão de acetato para a coluna e recolher a fração de 0,5 ml de primeira fluindo.
- Repita os passos 2.4 e 2.5, e recolher até oito de 0,5 ml frações nos tubos correspondentes.
- Definir um outro número, de 8 de baixa adsorvente, revestido a silicone, pequenos tubos de 0,6 ml e colocá-los em um rack. Rótulo outro tubo como "em branco". Retire 150 mL de cada fração e transferir para a rotulados 0,6 ml tubos. Use 20 l de SDS-PAGE análise (Figura 1) como mostrado anteriormente 23.
- Transferência de 150 mL de acetato de amônio 10 mM, pH 8,3, para dentro do tubo branco. Use este tubo para definir o espaço em branco em espectrofotômetro.
- Medir a absorvância em 130 mL de cada fração de λ = 280 nm em uma cubeta de quartzo.
- Após a absorbância é medido, combine as frações com o maior teor de proteína (tipicamente frações 3-5), misture delicadamente com uma pipeta, e manter uma alíquota de 10 mL para análise de aminoácidos para determinar a concentração de proteína real (não mostrados).
- Dividir a amostra em alíquotas múltiplas de proteínas ~ nmol 2 por tubo e lyophilize as amostras em um liofilizador.
- Após a conclusão da liofilização, tratar as amostras com HFIP 100% como antes.
- Armazenar os tubos a -20 ° C. Use um tubo para cada ciclo SELEX (abaixo).
Parte 3: Ampliação da sintéticos aleatórios ssDNA biblioteca por PCR
A biblioteca ssDNA sintética usada aqui para SELEX incluídos 49 nucleotídeos randomizados (A: T: G: C = 25:25:25:25%) ladeado por regiões constantes que compreende locais de clonagem (Bam HI, Eco RI) e um promotor T7, como descrito anteriormente 26.
- Para ampliar a biblioteca, criar uma reação de PCR padrão da seguinte maneira: 202 mL de água deionizada, 40 mL tampão 10x Taq DNA polimerase, 2 modelo ssDNA mL (0,5 nmol), 120 mL 25 mM MgCl 2, 28 mL 10 mM mix trifosfato desoxinucleótidos (dNTPs), 1 mL de primer para a frente (300 pmol), um primer reverso mL (300 pmol), e 4 mL de Taq polimerase.
- Realizar a reação de PCR utilizando um termociclador com as seguintes configurações: 5 min a 94 ° C para desnaturação inicial, 20 ciclos de cada um dos 94 ° C por 30 segundos, 52 ° C por 30 segundos para anelamento, 72 ° C por 30 segundos para extensão, extensão e final a 72 ° C por 7 min.
- Após a conclusão da reação de PCR, purificar o produto de DNA amplificado usando o kit Qiagen QIAquick purificação PCR, conforme instruções do fabricante. Geralmente, nesses experimentos a concentração e rendimento de DNA são 160 ± 10 ng / mL em 50 ul.
- Verificar a quantidade de DNA após a PCR por eletroforese em gel de agarose 2%.
Parte 4: Geração de 32 P-rotulados RNA por transcrição in vitro
- Configurar a reação de transcrição em um O-ring-capped, tubo de 1,6 ml de acordo com as instruções do fabricante, com algumas modificações da seguinte forma: 20 tampão de transcrição mL 5x T7, 7,5 mL cada um dos 100 mM RATP, rGTP, rUTP, 1 ml 100 mM RCTP, 2 l α 32 P-CTP (3000 Ci / mmol), 50-10 mcg purificada modelo de DNA (~ 30-40 mL de DNA purificado do produto), 10 mL mistura de enzimas, e compõem o volume final de 100 l pela adição de água livre de nuclease.
- Misturar a solução suavemente por uma pipeta, centrifugar a mistura e incubar a 37 ° C durante a noite.
- No final da reação, o modelo de DNA tem de ser removido. Adicionar RQ1 RNase DNase a uma concentração de 1 U / mg de DNA modelo e incubar por 4 h, a 37 ° C para digerir o modelo de DNA.
- Após 4 h, extrair o RNA, adicionando um volume de citrato-saturada de fenol: clorofórmio: álcool isoamílico (125:24:1, pH 4,7). Misture por um vórtice de ~ 1 min e centrifugar a 16.000 g por 2 min.
- Transferir a fase superior, aquosa para um novo tubo ou descartar a fase final por aspiração utilizando uma micropipeta. Adicionar 1 volume de clorofórmio: álcool isoamílico (24:1), misture por um vortex por 1 min e centrifugar conforme descrito em 4.4.
- Transferir a fase superior, aquosa para um novo tubo ou descartar a fase final por aspiração utilizando uma micropipeta. Clorofórmio residual pode ser removido através da realização de um giro rápido (10 segundos) em uma microcentrífuga e remoção da fase de fundo com uma micropipeta. Nesta etapa, é mais fácil de remover a fase de fundo ao invés do sobrenadante.
- Para precipitar o RNA, adicionar 0,1 volume equivalente de acetato de sódio 3M, pH 5,2, e 1 volume equivalente de 2-propanol. Misture e coloque em um freezer -20 ° C por 15 min.
- Após 15 min, giram em alta velocidade, de preferência em uma microcentrífuga refrigerada a 4 ° C, por 20-30 min para precipitar o produto RNA.
- Aspirar o sobrenadante com cuidado, lavar o pellet de RNA com 0,5 ml de etanol 70%, centrifugar a 4 ° C e descartar o etanol por aspiração.
- Transferir o tubo contendo o pellet de RNA a um bloco de calor e secar o pellet a 37 ° C por 5 min.
- Dissolver a amostra de RNA em 150 mM tampão STE, pH 8.0 (fornecido com o ProbeQuant illustra G-50 microcolunas) ou água livre de nuclease para um volume idêntico ao do in-vitro a transcrição de reação, ou seja, 100 l (passo 4.1).
- Aqueça o tubo contendo o produto RNA a 70 ° C por 10 min em um bloco de calor e misture por um vórtice para facilitar a dissolução do RNA.
- Centrífuga em alta velocidade por 1 min em temperatura ambiente.
- Manter uma alíquota de 1 mL-de RNA em um tubo de 0,6 ml rotulados para contagem de cintilação e TBE-uréia eletroforese em gel de poliacrilamida (Parte 6).
Parte 5: Remoção de nucleotídeos unincorporated, dessalinização RNA, e de cintilação contagem
Para remover os nucleotídeos unincorporated, use dois G-50 colunas de acordo com as instruções do fabricante.
- Inverter colunas e misture por um vortex para voltar a suspender a resina.
- Pressão fora do fechamento inferior das colunas de perfuração utilizando a ferramenta de plástico fornecido com o kit e certifique-se de deixar a saída intocada. Solte a tampa um quarto de volta e coloque as colunas em tubos de coleta limpa fornecidas no G-50 Kit.
- Girar as colunas nos tubos de coleta em 730 g por 1 min para remover o tampão de armazenamento.
- Transferência de colunas para dois novos O-ring-capped, 1,6 ml tubos e carregar 50 ul de 32 P-rotulados amostra de RNA por coluna diretamente sobre a resina, sem perturbar a resina.
- Giram em 730 g por 2 min para coletar o RNA purified rotulados. Após esta etapa, descartar as colunas.
- Transferência de 1 ml de G-50-RNA purificado a um tubo de 0,6 ml para contagem de cintilação e eletroforese (Parte 6). Loja de ações RNA a -20 ° C até o uso de SELEX. É desejável que o RNA é usado dentro de dois dias após a rotulagem, para evitar sua degradação e perda de atividade.
- Use as duas alíquotas de 1 mL de RNA passos 4,14 e 5,6 para medir as contagens por minuto (cpm / mL), utilizando um contador de cintilação. Aqui, usamos o Triathler contador de cintilação bancada.
- Transferir os tubos contendo a amostra de RNA no interior do adaptador plástico usado para contagem de 32 P, desde que com Triathler. Coloque o tubo ea placa dentro da câmara de contagem, feche a tampa da câmara, escolha de contagem P 32, e pressione start para iniciar a contagem.
- Calcule%, isto é a incorporação de etiquetas, a incorporação de α% 32 P-CTP = (cpm / mL para "G-50" amostra ÷ cpm / mL para a amostra "TOTAL") x 100. TOTAL da amostra é a amostra antes de G-50 de purificação.
- Após a contagem de cintilação e cálculo de incorporação por cento, calcular o rendimento de RNA e atividade específica de RNA. Por exemplo, se a conta para um pool de RNA antes (237.370 cpm / mL) e depois G-50 de purificação (191.330 cpm / mL) incorporação de rendimento de 81%, então o seguinte são calculados antes de obter o rendimento de RNA:
A partir do passo 4.1, a quantidade de α 32 P-CTP em 3000 Ci mmol / e 10 μCi / mL é: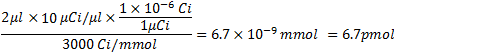 .
.
A quantidade de unlabeled CTP utilizada é: .
.
Quantidade total de CTP é utilizada 100006.7pmol. O peso molecular do RNA é calculada da seguinte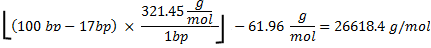
onde 321,45 g / mol 27 é a massa média de rNTPs, 100 bp é o número de bases na seqüência, e 17 bp é o número de bases no promotor T7, que não são transcritos. Subtração de 61,96 g / mol a partir do peso molecular oligonucleotídeo leva em conta a remoção de HPO 2 (63,98) e da adição de dois átomos de hidrogênio (2,02) 27. Porque, dos quatro rNTPs, CTP é o reagente limitante, se todos os CTP deveriam ser incorporados a massa teórica de RNA produzida teria sido: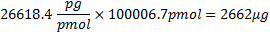 .
.
No entanto, por causa da incorporação de 81%, essa massa é agora 2156 mg. Contagens por mg de RNA seria, então,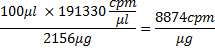
onde 100 l é o volume de reação de transcrição, e em termos de moles: 26.618,4 g / mol x 8,874 cpm / mg = 2.4x10 8 cpm / mol. A partir daqui, a quantidade de RNA em nmol e seu volume adequada utilizada para incubação com proteína pode ser calculado por exemplo, 5 mL de RNA conterá ~ 4 nmol RNA, ou seja,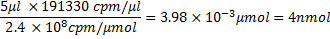
Nestes experimentos, a 300 pmol a 1 nmol de proteína e 4 nmol a 100 nmol RNA foram usados 21.
Parte 6: Caracterização do produto RNA por eletroforese e autoradiografia
- Para preparar as amostras para eletroforese, use as alíquotas de 1 mL de RNA antes e depois G-50 a partir das etapas de purificação de 4,14 e 5,6, respectivamente. Adicione 4 mL tampão STE, 5 l 2x Novex tampão de amostra TBE-uréia.
- Aqueça as amostras a 70 ° C por 5 min como normalmente recomendado para RNA eletroforese (aquecimento foi encontrado para ser desnecessária, pois a resolução gel das amostras de RNA com e sem aquecimento é o mesmo nesta configuração experimental).
- Montar um 6% gel de poliacrilamida TBE-uréia-gel no aparelho de execução, preencher o interior e exterior com câmaras 1x tampão TBE Novex-uréia Running. Lave os poços do gel pré-moldado usando o buffer de uma pipeta de 1 ml.
- Centrifugue os tubos RNA e carregar as amostras (10 total mL), utilizando gel de carregamento de dicas. Executar o gel a 180 V por 50 min.
- Após 50 min, desmontar o gel por quebrando o molde de plástico, remover e descartar apenas o forro mais curto do molde gel, mas deixar o mais apoio como suporte para o gel.
- Limpe a superfície da área de trabalho com a Decon (ou descontaminante radioatividade outro apropriado) certificando-se de todos os pontos contaminantes radioativos são removidos.
- Lay out de duas camadas de plástico Saran wrap; embrulhar o gel eo suporte plástico no envoltório de plástico de dois dobraram.
- Expõe o gel envolto em plástico para uma folha de umutoradiography filme x-ray dentro de um cassete de exposição no quarto escuro, sob luz de segurança. Deixe a cassete a -20 ° C por 60-90 min.
- Desenvolver o filme na sala escura sob luz de segurança depois de 60-90 min usando um desenvolvedor de filme automático (Figura 2).
Parte 7: RNA de incubação da proteína e obrigatória de filtro
Primeiro, o RNA e proteínas são incubados em solução e, em seguida, as seqüências de RNA que se ligam à proteína são separados dos não-ligantes. Como SELEX progresso ciclos, encadernação filtro dará uma indicação de enriquecimento protéico RNA vinculativo.
- Incubar a RNA a 90 ° C por 10 min para desnaturação e, em seguida, em temperatura ambiente por 10 min para renaturalização lento.
- Dissolver a proteína a partir do passo 2,12 em 8 mL 60 mM NaOH e adicionar 36 água livre de nuclease mL deionizada. Sonicate a mistura durante 1 min e adicione 36 mL de buffer 2x RNA binding (20 mM Tris, 300 mM NaCl, 10 mM MgCl 2, pH 7,5).
- Misture a quantidade adequada de RNA com 20 mL de buffer RNA 10x vinculativo e completar o volume para 200 mL pela adição de água livre de nuclease. Rotular o tubo "controle negativo."
- Misture 20 mL de proteína ea quantidade desejada de RNA com 20 mL de buffer RNA 10x vinculativo e perfazer o volume a 200 mL com água livre de nuclease. Rótulo do tubo de "reação".
- Misturar e incubar os tubos durante 30 min à temperatura ambiente. Enquanto isso prepare os filtros e da instalação de filtro de ligação para a próxima etapa.
- Anexar um frasco de 125 ml-lado-braço para uma entrada de vácuo. Coloque uma pré-limpeza, suporte de vidro poroso para o filtro no frasco lado-braço.
- Equilibrar três membranas de filtro em 2-3 ml de 1X tampão RNA obrigatório em uma placa de Petri 35x10 mm por 10-15 min. O primeiro filtro será usado para ajustar a sucção a vácuo, o segundo será utilizado para RNA-alone, negativa de controle de ligação de filtro, eo terceiro será utilizado para a ligação do filtro da mistura de RNA-proteína.
- Após 30 min, centrifugar a reação de ligação e os tubos controle em alta velocidade por 5 min à temperatura ambiente.
- Ligue o vácuo e colocar a membrana primeiro no vidro poroso. Usando uma micropipeta, gotejamento 0,5 ml de RNA do buffer de ligação para a membrana e ajustar o vácuo para permitir o fluxo lento de cada gota de buffer através da membrana.
- Coloque a segunda membrana sobre o vidro poroso e usando o mesmo caudal, aplicar o controle negativo sobre a membrana.
- Aplicar-4x0.5 ml alíquotas de 1x tampão RNA vinculativo para lavar a membrana e descartar o escoamento. Note que se pre-clearing é desejado, o fluxo é mantido por meio de extração de RNA. Pré-limpeza remove seqüências de RNA que se ligam ao filtro.
- Remover a membrana negativo de controle e coloque em uma correspondentemente marcado com tubo 1,6 ml para contagem de cintilação.
- Substituir o vidro poroso com um suporte de vidro pré-limpos segundo poroso.
- Coloque o terceiro disco no vidro poroso e aplicar a mistura de reação. Lavar o filtro como no passo 7,11 e descartar o escoamento. O número de lavagens pode ser aumentada em ciclos mais tarde SELEX para aumentar o rigor das condições SELEX.
- Remova o disco de filtro e coloque dentro de um tubo de 1,6 ml rotulados como "mistura de reação" e manter a contagem de cintilação.
- Realizar a contagem de cintilação como no passo 5,8 e anotar as contagens para os filtros respectivos.
- Calcular o nível da radioactividade filtro ligado em relação ao montante total da radioatividade aplicada às membranas (ligação%). Isto lhe dará uma indicação de enriquecimento de ligação com o progresso da SELEX.
Parte 8: extração de RNA a partir dos filtros
RNA é extraído dos filtros para obter as seqüências que se ligam à proteína. Essas seqüências são amplificados para o ciclo SELEX seguinte.
- Após a contagem de cintilação, remova a reação de ligação do filtro do tubo (a partir do passo 7.15) e coloque em um pano limpo, seco, placa de Petri 35x10 mm.
- Use um bisturi limpo e um par de pinças para cortar a membrana em pedaços pequenos.
- Usando a pinça, substituir as peças de corte da membrana no mesmo tubo rotulado a partir do passo 8.1.
- Adicionar 400 mL tampão de eluição (7 M uréia, 3 mM EDTA, citrato de sódio 100 mM, pH 5,0) e incubar os tubos a 95 ° C por 10 min.
- Centrifugar o tubo em velocidade superior à temperatura ambiente, aspirado, e recolher a manteiga de extração em um novo tubo rotulado.
- Medir a contagem de radioatividade restante no tubo que contém as peças membrana por cintilação contagem para avaliar a eficiência de extração.
- Repita o processo de extração (passos 8,4-8,6) três vezes. A eficiência após três extrações normalmente é ~ 95-96%.
- Nos tubos contendo os extratos de RNA, adicionar 1 volume (400 L) de citrato-saturada (pH 4,7) fenol: clorofórmio: álcool isoamílico (125:24:1). Misture por um vórtice de ~ 1 min e centrifugaçãoge a 16.000 g por 2 min.
- Transferir a fase superior, aquosa para um novo tubo ou descartar a fase final por aspiração utilizando uma micropipeta.
- Adicionar 1 volume de clorofórmio: álcool isoamílico (24:1), misture por um vortex por 1 min e centrifugar a 16.000 g por 2 min.
- Transferir a fase superior, aquosa para um novo tubo ou descartar a fase final por aspiração utilizando uma micropipeta.
- Para precipitar o RNA, adicionar 0,1 volume de acetato de sódio 3 M, pH 5,2, o glicogênio mL 3-4 (10 mcg / mL) como RNA coprecipitant, e um volume equivalente de 2-propanol. Misture e coloque em um freezer -20 ° C durante a noite.
- Giram em velocidade máxima, de preferência em uma microcentrífuga a 4 ° C, por 20-30 min para precipitar o RNA a partir do passo 8.12.
- Após a centrifugação, o RNA separa em uma fase líquida que é pouco visível. Aspirar o sobrenadante com cuidado, sem desalojar esta fase pouco visível precipitante na parte inferior do tubo.
- Lave o RNA "pellet" com 0,5 ml de etanol 70%, centrifugar por 5 min em velocidade máxima a 4 ° C e desprezar o etanol por aspiração sem desalojar a fase pouco visível precipitante.
- Dissolver o sedimento RNA em 50 mL tampão STE e siga para G-50 de purificação como no passo 5.
Parte 9: A transcrição reversa e PCR para os ciclos de continuar SELEX
Para avançar para o próximo ciclo de SELEX, RNA tem de ser reversamente transcrito para DNA e amplificado pela PCR.
- Em 5 rotulados 0,6 ml tubos, misture 3 mL da purificado, G-50-dessalinizada RNA com 2 l de 8 vezes diluída de primer reverso. "Controle negativo." Tubo de um rótulo
- Incubar a mistura a 70 ° C por 5 min, e depois no gelo por mais 5 minutos para permitir a hibridização do primer para o RNA.
- Para esta mistura sobre o gelo, adicione 6,4 água livre de nuclease mL, 4 mL ImProm-II tampão de reação 5x, 1,6 mL 25 mM MgCl 2, 1 ml 10 mM dNTP mix, um inibidor da ribonuclease mL RNasein, 1 ml ImProm-II Transcriptase Reversa perfazendo um total de 15 mL.
- Para o controle negativo, adicionar 7,4 de água livre de nuclease mL e deixar de fora da transcriptase reversa. Identifique os tubos de acordo. O controle negativo é incluído para verificar se a contaminação de DNA de um ciclo anterior não é amplificado para o ciclo SELEX seguinte. Esta etapa testa a eficácia da digestão de DNA por modelo RQ1 RNase DNase no passo 4.3.
- Use um termociclador para incubar a mistura de reação a 25 ° C por 5 min, 42 ° C durante 1 h para o recozimento e extensão da vertente do primeiro DNA, respectivamente, seguido de 70 ° C por 15 min para inativar a transcriptase reversa.
- Após a reação de transcrição reversa, configurar o mix de PCR. Adicione 30 de água livre de nuclease mL, 10 ml tampão Taq 10x, 30 mL 25 mM MgCl 2, 7 mL 10 mM dNTP mix, 1 ml de cada primer, e 1 mL de polimerase Taq em todos os tubos.
- Execute o programa de PCR como na Parte 3 de 9-14 ciclos.
- Purificar o DNA do produto como no passo 3.3.
- Mix 8 mL do produto DNA com 2 mL de corante DNA carga e electroforese em agarose 2% em 100 V para min 15-20 (Figura 3).
- Produto DNA está pronto para ser transcrito para RNA rotulados como na Parte 4 e utilizado para o próximo ciclo de SELEX.
Parte 10: Resultados Representante
Em experimentos SELEX, a natureza da Aβ40 oligômeros usado como o destino, a qualidade de RNA usada para cada ciclo, e extração de RNA de sucesso e de amplificação após cada ciclo são importantes. Usamos picup para gerar uma mistura oligomérica Aβ40 para SELEX após purificação e remoção do cross-linking reagentes. Os experimentos dessalinização descrito na Parte 2 geralmente levam a perda de proteína 50-55%. A quantidade de proteína e de qualidade pode ser avaliada através de medições de absorbância (λ = 280) e SDS-PAGE (Figura 1). O perfil de absorbância média de Aβ40 eluatos de 5 experiências individuais sobrepondo um perfil de SDS-PAGE típico de Aβ40 eluída em um desses experimentos são mostrados na Figura 1. Os dados mostram que a proteína consistentemente elui fora da coluna, em frações 3-5 e os reagentes cross-linking eluir após a fração 6 (absorbância aumento na fração de 7, Figura 1). SDS-PAGE mostra a distribuição de oligômero típico Aβ40 22. Esta distribuição foi reprodutível após a frações protéicas foram liofilizadas (2,11), tratados com HFIP (2.11), ressolubilizam (7.1), e re-analisados por SDS-PAGE.
Integridade do RNA para cada ciclo SELEX também é importante para a progressão da SELEX iterativo, especialmente quando nuclease sensíveis ribo-oligonucleotides são usados. Depois de RNA a amplificação e rotulagem (Parte 4), a qualidade do RNA rotulado pode ser avaliada por TBE-uréia-eletroforese em gel de poliacrilamida. Um perfil típico de um produto intacto RNA rotulados antes e depois do G-50 de purificação (Part 5) é mostrado na Figura 2.
Após cada ciclo SELEX, RNA é extraído da membrana após filtro de ligação (Parte 8) e reverse-transcrito (Parte 9) para a DNA para amplificação por PCR (Passo 9.5). O modelo de DNA a partir de um ciclo anterior é então usado para gerar 32 P-rotulados RNA (Parte 4) para o próximo ciclo. Se algum modelo de DNA contaminante de um ciclo anterior persistir no produto RNA marcado após reações de transcrição in vitro (Parte 4), a eficiência dos ciclos SELEX será reduzido, exigindo mais ciclos. Para controlar isso, após cada ciclo SELEX ea correspondente transcrição reversa-reação de PCR, eletroforese em agarose é realizada (9.12). Ausência de DNA do produto nos tubos de controle de reação negativa (9,4) indica a remoção bem sucedida do modelo de DNA proveniente de um ciclo de SELEX anterior (Figura 3). Se a amplificação do DNA por PCR é observado no tubo controle negativo, é aconselhável que a duração da incubação com RQ1 DNase (4.3) ser prolongada. O fabricante recomenda-duração da incubação com RQ1 DNase é de 15 min, no entanto, descobrimos que as incubações mais tempo (4-5 h) foram obrigados a retirar o DNA template completamente (Figura 3).
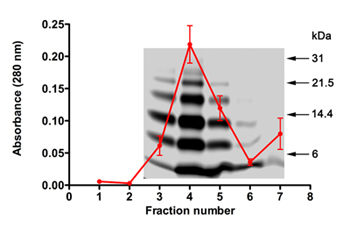
Figura 1. SDS-PAGE e perfil de absorção de picup gerado, dessalinizada Aβ40 oligômeros. O perfil de absorção de 5 colunas individuais dessalinização foi em média e sobreposto a um gel representativo. Marcadores de peso molecular são mostrados à direita.

Figura 2. TBE-uréia-eletroforese em gel de poliacrilamida do RNA do produto antes e depois do G-50 de purificação. A direção da migração do produto RNA é de cátodo para ânodo, como indicado.
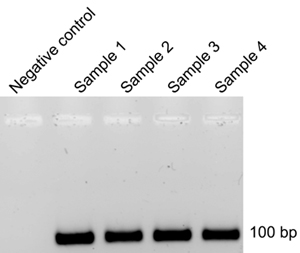
Figura 3. Eletroforese em gel de Agarose do produto DNA após transcrição reversa e amplificação por PCR.
Discussão
O ponto de partida do processo SELEX é a síntese de uma biblioteca de oligonucleotídeos aleatórios tipicamente contendo 10 12 -10 15 seqüências. No DNA SELEX, esta biblioteca é usada diretamente depois de um pool de ssDNA é gerada, enquanto que no RNA SELEX, demonstrado aqui, a biblioteca ssDNA é convertido primeiro a um pool de RNA enzimaticamente por in-vitro de transcrição. Então, é realizada iterativamente SELEX que cada ciclo compõe a exposição e ligação de oligonucl...
Divulgações
Agradecimentos
Este trabalho foi suportado por concessões de NIH AG030709 / NIA e 07-65798 do Departamento de Saúde Pública da Califórnia. Reconhecemos Margaret M. Condron para peptídeo síntese e análise de aminoácidos, Dr. Elizabeth Neufeld F. para ajudar e apoiar os passos iniciais do projeto, Dr. Chi-Hong B. Chen para dar suporte e reagentes, e André, Dr. D . Ellington para discussões úteis.
Materiais
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| Aβ40 | UCLA Biopolymers Laboratory | Lyophilized powder | ||
| MX5 Automated-S Microbalance | Mettler Toledo | |||
| Silicon-coated, 1.6-ml tubes | Denville Scientific | C19033 or C19035 | ||
| 1,1,1,3,3,3-hexafluoro-2-propanol (HFIP) | TCI America | H0424 | Use in a fume hood. | |
| Ammonium persulfate | Sigma | A-7460 | Vortex until the solution is clear. APS is prepared freshly each time and should be used within 48 h. | |
| Tris(2,2-bipridyl)dichlororuthenium(II) hexahydrate | Sigma | 224758-1G | Vortex until the solution is clear. Cover the RuBpy tube with foil to protect the reagent from ambient light. RuBpy is prepared freshly each time and should be used within 48 h. | |
| Dithiothreitol (DTT) | Sigma | 43815 | ||
| D-Salt™ Excellulose™ desalting columns | Thermo Scientific | 20449 | ||
| Ammonium acetate | Fisher Scientific | A637-500 | ||
| Silicon-coated, 0.6-ml tubes | Denville Scientific | C19063 | ||
| Novex Tricine Gels (10–20%) | Invitrogen | EC6625B0X | 10-well; mini size (8 cm X 8 cm); 25 μl loading volume per well; separation range 5 kDa to 40 kDa | |
| Quartz cuvette | Hellma | 105.250-QS | ||
| Beckman DU 640 spectrophotometer | Beckman | |||
| ssDNA library | Integrated DNA Technologies | Custom-ordered | The library was designed to contain 49 random nucleotides flanked by two constant regions containing primer-binding and cloning sites: 5'-TAA TAC GAC TCA CTA TAG GGA ATT CCG CGT GTG C (N:25:25:25:25%) (N)49 G TCC GTT CGG GAT CCT C-3' | |
| Taq DNA polymerase | USB Corporation | 71160 | Recombinant Thermus aquaticus DNA Polymerase supplied with 10× PCR Buffer and a separate tube of 25 mM MgCl2 for routine PCR. | |
| PCR Nucleotide Mix, 10 mM solution | USB Corporation | 77212 | (10 mM each dATP, dCTP, dGTP, dTTP) | |
| Forward primer | Integrated DNA Technologies | Custom-ordered | 5'-TAA TAC GAC TCA CTA TAG GGA ATT CCG CGT GTG C-3' | |
| Reverse primer | Integrated DNA Technologies | Custom-ordered | 5'-GAG GAT CCC GAA CGG AC-3' | |
| Thermal cycler | Denville Scientific | Techne TC-312 | ||
| QIAquick PCR Purification Kit (50) | QIAGEN | 28104 | ||
| Agarose | Denville Scientific | CA3510-8 | ||
| Conical, sterile 1.6-ml tubes with caps attached with O-rings | Denville Scientific | C19040-S | ||
| RiboMAX™ Large Scale RNA Production System–T7 | Promega | P1300 | The kit contains: 120 μl Enzyme Mix (RNA polymerase, recombinant RNasin® ribonuclease inhibitor and recombinant inorganic pyrophosphatase); 240 μl transcription 5 buffer; 100 μl each of 4 rNTPs, 100 mM; 110 U RQ1 RNase-free DNase, 1 U/μl; 10 μl linear control DNA, 1 mg/ml; 1 ml 3M sodium acetate (pH 5.2); 1.25 ml nuclease-Free water | |
| α-32P-cytidine 5'-triphosphate, 250 μCi (9.25 MBq), | Perkin Elmer | BLU008H250UC | Specific Activity: 3000 Ci (111 TBq)/mmol, 50 mM Tricine (pH 7.6) | |
| Citrate-saturated phenol:chloroform:isoamyl alcohol (125:24:1, pH 4.7) | Sigma (Fluka) | 77619 | ||
| Chloroform:Isoamyl alcohol (24:1) | Sigma | C0549 | ||
| Absolute ethanol for molecular biology | Sigma | E7023 | ||
| Z216-MK refrigerated microcentrifuge | Denville Scientific | C0216-MK | ||
| illustra ProbeQuant™ G-50 Micro Columns | GE Healthcare | Obtained from Fisher Scientific (45-001-487) | Prepacked with Sephadex™ G-50 DNA Grade and pre-equilibrated in STE buffer containing 0.15% Kathon as Biocide | |
| Triathler Bench-top Scintillation counter | Hidex Oy, Turku, Finland | Triathler LSC Model: 425-034 | ||
| Novex® TBE-Urea Sample Buffer (2×) | Invitrogen | LC6876 | ||
| 6% TBE-Urea Gels 1.0 mm, 10 wells | Invitrogen | EC6865BOX | ||
| Novex® TBE Running Buffer (5×) | Invitrogen | LC6675 | ||
| Radioactivity decontaminant | Fisher Scientific | 04-355-67 | ||
| Gel-loading tips | Denville Scientific | P3080 | ||
| XCell SureLock Mini-Cell | Invitrogen | EI0001 | XCell SureLock Mini-Cell | |
| Autoradiography film | Denville Scientific | E3018 | Use in complete darkness | |
| Autoradiography film, Hyperfilm™ ECL | Amersham Biosciences | RPN3114K | Can be used under red safe light. | |
| Membrane discs | Millipore | GSWP02500 | Mixed cellulose ester, hydrophilic, 0.22-μm disc membranes | |
| Fritted glass support base for 125-ml flask | VWR | 26316-696 | ||
| Petri dishes | Fisher Scientific | 08-757-11YZ | ||
| Urea | Fisher Scientific | AC32738-0050 | ||
| EDTA | Fisher Scientific | 118430010 | ||
| Glycogen | Sigma | G1767 | ||
| 2-Propanol for molecular biology | Sigma | I9516 | ||
| Recombinant RNase inhibitor | USB Corporation | 71571 | ||
| ImProm-II™Reverse Transcription System | Promega | A3802 | ||
| Recombinant RNase inhibitor | USB Corporation | 71571 | ||
| RapidRun™ Loading Dye | USB Corporation | 77524 |
Referências
- Monien, B. H., Apostolova, L. G., Bitan, G. Early diagnostics and therapeutics for Alzheimer's disease-how early can we get there. Expert. Rev. Neurother. 6, 1293-1306 (2006).
- Nestor, P. J., Scheltens, P., Hodges, J. R. Advances in the early detection of Alzheimer's disease. Nat. Med. 10, S34-S41 (2004).
- Kawas, C. H. Clinical practice. Early Alzheimer's disease. N. Engl. J. Med. 349, 1056-1063 (2003).
- Haass, C., Selkoe, D. J. Soluble protein oligomers in neurodegeneration: lessons from the Alzheimer's amyloid β-peptide. Nat. Rev. Mol. Cell. Biol. 8, 101-112 (2007).
- Kirkitadze, M. D., Bitan, G., Teplow, D. B. Paradigm shifts in Alzheimer's disease and other neurodegenerative disorders: the emerging role of oligomeric assemblies. J. Neurosci. Res. 69, 567-577 (2002).
- Rahimi, F., Shanmugam, A., Bitan, G. Structure-function relationships of pre-fibrillar protein assemblies in Alzheimer's disease and related disorders. Curr. Alzheimer Res. 5, 319-341 (2008).
- Jayasena, S. D. Aptamers: an emerging class of molecules that rival antibodies in diagnostics. Clin. Chem. 45, 1628-1650 (1999).
- Bunka, D. H., Stockley, P. G. Aptamers come of age - at last. Nat. Rev. Microbiol. 4, 588-596 (2006).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Lee, J. F., Stovall, G. M., Ellington, A. D. Aptamer therapeutics advance. Curr. Opin. Chem. Biol. 10, 282-289 (2006).
- Weiss, S. RNA aptamers specifically interact with the prion protein PrP. J. Virol. 71, 8790-8797 (1997).
- Bibby, D. F. Application of a novel in vitro selection technique to isolate and characterise high affinity DNA aptamers binding mammalian prion proteins. J. Virol. Methods. 151, 107-115 (2008).
- Rhie, A. Characterization of 2'-fluoro-RNA aptamers that bind preferentially to disease-associated conformations of prion protein and inhibit conversion. J. Biol. Chem. 278, 39697-39705 (2003).
- King, D. J., Safar, J. G., Legname, G., Prusiner, S. B. Thioaptamer interactions with prion proteins: sequence-specific and non-specific binding sites. J. Mol. Biol. 369, 1001-1014 (2007).
- Proske, D. Prion-protein-specific aptamer reduces PrPSc formation. ChemBioChem. 3, 717-725 (2002).
- Murakami, K., Nishikawa, F., Noda, K., Yokoyama, T., Nishikawa, S. Anti-bovine prion protein RNA aptamer containing tandem GGA repeat interacts both with recombinant bovine prion protein and its β isoform with high affinity. Prion. 2, 73-80 (2008).
- Luhrs, T., Zahn, R., Wuthrich, K. Amyloid formation by recombinant full-length prion proteins in phospholipid bicelle solutions. J. Mol. Biol. 357, 833-841 (2006).
- Bunka, D. H. Production and characterization of RNA aptamers specific for amyloid fibril epitopes. J. Biol. Chem. 282, 34500-34509 (2007).
- Ylera, F., Lurz, R., Erdmann, V. A., Furste, J. P. Selection of RNA aptamers to the Alzheimer's disease amyloid peptide. Biochem. Biophys. Res. Commun. 290, 1583-1588 (2002).
- Rahimi, F., Murakami, K., Summers, J. L., Chen, C. H., Bitan, G. RNA aptamers generated against oligomeric Aβ40 recognize common amyloid aptatopes with low specificity but high sensitivity. PLoS ONE. 4, e7694-e7694 (2009).
- Bitan, G., Lomakin, A., Teplow, D. B. Amyloid β-protein oligomerization: prenucleation interactions revealed by photo-induced cross-linking of unmodified proteins. J. Biol. Chem. 276, 35176-35184 (2001).
- Rahimi, F., Maiti, P., Bitan, G. Photo-induced cross-linking of unmodified proteins (PICUP) applied to amyloidogenic peptides. J. Vis. Exp. , (2009).
- Bitan, G., Fradinger, E. A., Spring, S. M., Teplow, D. B. Neurotoxic protein oligomers-what you see is not always what you get. Amyloid. 12, 88-95 (2005).
- Bitan, G. Structural study of metastable amyloidogenic protein oligomers by photo-induced cross-linking of unmodified proteins. Methods Enzymol. 413, 217-236 (2006).
- Chen, C. H., Chernis, G. A., Hoang, V. Q., Landgraf, R. Inhibition of heregulin signaling by an aptamer that preferentially binds to the oligomeric form of human epidermal growth factor receptor-3. Proc. Natl. Acad. Sci. USA. 100, 9226-9231 (2003).
- Adams, D. S. . Lab math: a handbook of measurements, calculations, and other quantitative skills for use at the bench. , (2003).
- Gopinath, S. C. Methods developed for SELEX. Anal. Bioanal. Chem. 387, 171-182 (2007).
- Takahashi, T., Tada, K., Mihara, H. RNA aptamers selected against amyloid β-peptide (Aβ) inhibit the aggregation of Aβ. Mol. Biosyst. 5, 986-991 (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados