A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
מבחר Aptamers עבור חלבון β עמילואיד, הסוכן סיבתי של מחלת אלצהיימר
In This Article
Summary
Aptamers הם ribo-/deoxyribo-oligonucleotides קצר נבחר על ידי חוץ גופית שיטות האבולוציה מבוסס על זיקה יעד ספציפי. Aptamers הם כלים לזיהוי מולקולרי עם יישומים טיפולית, אבחון, מחקר מגוונות. אנו מדגימים שיטות הבחירה של aptamers לחלבון β עמילואיד, הסוכן סיבתי של מחלת אלצהיימר.
Abstract
מחלת אלצהיימר (AD) הוא פרוגרסיבי, תלויי גיל, הפרעת ניווניות עם קורס חתרני שהופכת presymptomatic האבחנה שלה קשה 1. אבחנה ברורה לספירה מושגת רק נתיחה שלאחר המוות, ובכך לבסס אבחנה presymptomatic, מוקדם של אלצהיימר הוא חיוני לפיתוח טיפולים יעילים לניהול 2,3.
עמילואיד β-חלבון (Aβ) הוא מרכזי בהיווצרות לספירה. מסיסים, מכלולים Aβ oligomeric הם האמינו הבסיסית neurotoxicity להשפיע על תפקוד סינפטי ואיבוד נוירון לספירה 4,5. צורות שונות של מכלולים Aβ מסיסים תוארו, לעומת זאת, יחסי הגומלין שלהם רלוונטיות האטיולוגיה לספירה בפתוגנזה הם מורכבים ולא הבין היטב 6. כלים ספציפיים ההכרה המולקולרית עשויה לפתור את היחסים בין מכלולים Aβ ולאפשר זיהוי ואפיון של מכלולים אלה מוקדם במהלך המחלה לפני שהתסמינים מופיעים. הכרה מולקולרית בדרך כלל מסתמכת על נוגדנים. עם זאת, מעמד חלופי של כלי הכרה, aptamers מולקולרית, מציעה יתרונות חשובים ביחס נוגדנים 7,8. Aptamers הם oligonucleotides שנוצר על ידי בחירה ב-vitro: התפתחות שיטתית של ligands על ידי העשרה מעריכי (Selex) 9,10. Selex היא תהליך חוזר ונשנה זה, בדומה לאבולוציה הדרווינית, מאפשר בחירה, הגברה, העשרה, והנצחה של הנכס, למשל, נלהב, ספציפית, מחייב ליגנד (aptamers) או פעילות קטליטית (ribozymes ו DNAzymes).
למרות הופעתה של aptamers ככלים בתחום הביוטכנולוגיה והרפואה המודרנית 11, הם כבר לא מנוצלים בתחום עמילואיד. RNA מעטים או aptamers ssDNA נבחרו נגד צורות שונות של חלבונים Prion (PRP) 12-16. Aptamer RNA שנוצר נגד PRP רקומביננטי שור הוצגה להכיר שור PRP-β 17, מסיסים, oligomeric, גיליון β-עשירים גרסה קונפורמציה של PRP באורך מלא שנוצר סיבי עמילואיד 18. Aptamers שנוצר באמצעות טפסים monomeric וכמה סיבי β 2-microglobulin (β 2 מ ') נמצאו לאגד הסיבים של חלבונים מסוימים amyloidogenic אחרים מלבד β 2 מ' הסיבים 19. Ylera et al. תיאר aptamers RNA שנבחר נגד משותקת monomeric Aβ40 20. באופן בלתי צפוי, aptamers אלה קשורים סיבי Aβ40. בסך הכל, הנתונים הללו מעלים שאלות חשובות. מדוע נבחר aptamers נגד חלבונים monomeric לזהות צורות פולימריות שלהם? יכול aptamers נגד צורות monomeric ו / או oligomeric של חלבונים amyloidogenic לקבל? כדי לענות על השאלות האלה, ניסינו לבחור aptamers עבור קוולנטית מיוצב oligomeric Aβ40 21 שנוצר באמצעות תמונה-Induced cross-linking של חלבונים ללא שינוי (PICUP) 22,23. בדומה לממצאים קודמים 17,19,20, aptamers אלה הגיבו הסיבים של Aβ וכמה חלבונים אחרים amyloidogenic סביר הכרה עמילואיד משותף פוטנציאלי מבניים aptatope 21. כאן, אנו מציגים את המתודולוגיה Selex המשמשים לייצור של אלה aptamers 21.
Protocol
חלק 1: הכנת חלבון cross-linking
בתחילה, חלבון המשמש Selex הוא pretreated עם 1,1,1,3,3,3-hexafluoro-2-propanol (HFIP) כדי להשיג הומוגניות, במצטבר ללא הכנות, כפי שתוארו קודם לכן 23. צעד זה הוא הכרחי משום מראש יצרו אגרגטים לגרום להצטברות מהירה של חלבונים amyloidogenic, וכתוצאה מכך שחזור הניסוי מסכן 24, והם רצויים לבחירת aptamers עבור unaggregated, לא סיבי צורות של החלבון.
- לשקול את ~ 800 מיקרוגרם (~ 180 nmol) טהור Aβ40 באמצעות microbalance. העברת הפפטיד lyophilized יבש לתוך שכותרתו, סיליקון מצופה, בעלי כושר ספיגה נמוך 1.6-מ"ל צינורות microfuge.
- ממיסים את הפפטיד ב 100% 1,1,1,3,3,3-hexafluoro-2-propanol (HFIP, 400 μl) להשיג 0.5 פפטיד פתרון mM כפי שתואר לעיל 23.
- מחלקים את הפתרון הזה לארבעה 100-μl aliquots כזה שפופרת מכילה ~ 45 nmol Aβ40 להלכה. המשך הסרת HFIP כפי שתואר לעיל 23.
- לפני solubilizing HFIP שטופלו פפטידים במאגר עבור cross-linking תגובות, להכין את cross-linking ו ריאגנטים מרווה. לשקול את persulfate אמוניום (APS, MW 228.2 g / mol) ולהכין פתרון של 40 מ"מ ב 10 mM נתרן פוספט, pH 7.4. מערבבים בעזרת מערבולת עד הפתרון הוא ברור.
- הכן פתרון 2 מ"מ טריס (2,2-bipyridyl) dichlororuthenium (II) hexahydrate (RuBpy, MW 748.63 g / mol) ב 10 mM נתרן פוספט, pH 7.4. מערבבים בעזרת מערבולת לאמת התפרקות מוחלטת. הגן על הצינור מן האור באמצעות רדיד אלומיניום.
- הכן מגיב מרווה. לשקול את dithiothreitol (DTT, MW 154.5 g / mol) ו להתמוסס במים deionized או פוספט-10 mM נתרן, pH 7.4, עד 1 מ '
- ממיסים את הפפטיד HFIP שטופלו בדיוק כפי שתואר קודם לכן 23,25 אלא שואפים להשיג ~ 60 פפטיד פתרון מיקרומטר.
- בצע PICUP ליצור תערובת של Aβ40 oligomeric כפי שתואר לעיל 23. תגובה PICUP טיפוסי מבוצע בהיקף של 20 μl שבו ריכוזי הסופי של החלבון, RuBpy ו APS הם 30 מיקרומטר, 0.05 מ"מ, 1 מ"מ, 23 בהתאמה. הנה, את התערובת המשמשים PICUP מכיל חלבון 108 μl, 6 RuBpy μl, 6 APS μl, ו 1 DTT μl ואת הריכוז של חלבון, RuBpy ו APS הם הכפילו את כל יחסית התגובה הטיפוסית. פעולה זו מפחיתה את מספר התגובות PICUP להתבצע ומגדיל את ריכוז החלבון עבור desalting.
חלק 2: Desalting הכנת חלבון
לפני השימוש חלבון עבור Selex, desalting מבוצע כדי להסיר את ריאגנטים cross-linking המשמש PICUP. תערובת זו מכילה את התגובה PICUP ריאגנטים cross-linking ו ~ 55 חלבון מיקרומטר (ריכוז נומינלי).
- הסר את המכסה העליון את עמודה 5 מ"ל desalting, תמיכה העמודה על ידי דוכן, במקום כוס מתחת לשקע את הטור, ולהסיר את התקע לשקע. תן את זרימת למאגר אחסון דרך לאסוף לתוך כוס.
- לאזן את העמודה אצטט 10 mM אמוניום, pH 8.3. הוסף 5 שרף המיטה כרכים (25 מ"ל) של חיץ זה 3-ml aliquots לעמודה ולאפשר לו לזרום.
- בינתיים, תווית 8 נמוך בעלי כושר ספיגה, סיליקון מצופה 1.6-מ"ל צינורות למקם אותם מתלה צינור.
- אחרי הטור הוא equilibrated, להחיל aliquot 0.5-0.7-מ"ל של תערובת התגובה PICUP לכל עמודה לכל אחד להשתמש desalting (עמודות ניתן לכבס, לאחסן, ו equilibrated עבור משתמש אחר). אפשר תערובת חלבון לספוג לתוך שרף את העמודה וזורקים את הזרימה דרך בספל.
- מניחים את הצינור הקולקציה הראשונה מתחת לשקע העמודה. הוסף 0.5 מ"ל אצטט חיץ לעמודה ולאסוף את החלק הראשון 0.5-מ"ל זורם דרך.
- חזור על שלבים 2.4 ו -2.5, ולאסוף עד שמונה 0.5-מ"ל שברים של צינורות המקביל.
- קבוצה נוספת מספר 0.6-ml 8 צינורות בעלי כושר ספיגה נמוך, מצופה סיליקון, קטנים ומניחים אותם במעמד. לייבל אחר צינור כמו "ריק". קח את 150 μl של כל חלק ולהעביר לתוך 0.6-מ"ל צינורות שכותרתו. השתמש 20 μl לניתוח SDS-PAGE (איור 1) כפי שהוצג קודם 23.
- העברת 150 μl 10 mM אמוניום אצטט, pH 8.3, לתוך הצינור ריק. השתמש באפשרות זו כדי להגדיר את צינור ריק ספקטרופוטומטר.
- מדוד את ספיגת ב 130 μl של כל חלק ב λ = 280 ננומטר קובט קוורץ.
- לאחר ספיגת נמדדת, לשלב את השברים עם תוכן החלבון הגבוהה ביותר (בדרך כלל שברים 3-5), לערבב בעדינות בעזרת פיפטה, ולשמור aliquot 10-μl לניתוח חומצת אמינו על מנת לקבוע את ריכוז החלבון בפועל (לא מוצג).
- מחלקים את המדגם לתוך aliquots מרובות של חלבון 2 ~ nmol לכל צינור lyophilize דגימות lyophilizer.
- לאחר השלמת lyophilization, לטפל בדגימות עם HFIP 100% כמו קודם.
- אחסן את צינורות ב -20 ° C. השתמש באחת צינור לכל מחזור Selex (להלן).
חלק 3: הגברה של הסינתטי אקראי ssDNA ספריה על ידי ה-PCR
הספרייה ssDNA סינתטי המשמש כאן Selex כללה 49 נוקליאוטידים אקראיים (A: T: G: C = 25:25:25:25%) מוקף האזורים קבוע הכולל אתרים שיבוט (Bam HI, Eco RI) לבין האמרגן T7, כמו שתואר קודם לכן 26.
- כדי להגביר את הספרייה, הקים תגובה רגילה PCR כדלקמן: 202 μl מים deionized, 40 פולימראז μl 10x תקי למאגר הדנ"א, 2 μl תבנית ssDNA (0.5 nmol), 120 μl 25 מ"מ MgCl 2, 28 deoxynucleoside μl 10 mM לערבב טריפוספט (dNTPs), 1 פריימר קדימה μl (300 pmol), 1 פריימר הפוך μl (300 pmol), ו 4 μl פולימראז תקי.
- בצע את תגובת ה-PCR באמצעות Cycler תרמי עם ההגדרות הבאות: 5 דקות ב 94 מעלות denaturation ההתחלתי, 20 מחזורים כל אחד 94 מעלות צלזיוס למשך 30 שניות, 52 ° C למשך 30 שניות חישול, 72 ° C למשך 30 שניות הרחבה, והרחבה הסופי 72 מעלות צלזיוס למשך 7 דקות.
- לאחר השלמת התגובה PCR, לטהר את מוצר ה-DNA הגברה באמצעות PCR QiaQuick Qiagen ערכת טיהור לפי הוראות היצרן. באופן כללי, בניסויים אלה ריכוז ואת התשואה של ה-DNA הם 160 ± 10 ng / μl ב μl 50.
- בדוק את כמות ה-DNA לאחר PCR על ידי אלקטרופורזה agarose 2% ג'ל.
חלק 4: דור P-32 שכותרתו RNA על ידי שעתוק חוץ גופית
- הגדר את התגובה שעתוק בתוך צינור O-Ring הכתיר 1.6-מ"ל, על פי הוראות היצרן עם כמה שינויים כדלקמן: 20 חיץ μl 5x T7 שעתוק, 7.5 μl כל אחד 100 מ"מ rATP, rGTP, rUTP, 1 μl 100 mM rCTP, 2 μl α 32 P-CTP (3000 Ci / mmol), 50-10 מיקרוגרם מטוהרים תבנית ה-DNA (~ μl 30-40 מטוהרים של מוצר ה-DNA), אנזים לערבב 10 μl, ולעשות את הווליום הסופי μl 100 על ידי הוספת nuclease ללא מים.
- מערבבים בעדינות את הפתרון על ידי טפטפת, צנטריפוגה את התערובת, ואת לדגור על 37 מעלות צלזיוס למשך הלילה.
- בסוף התגובה, את תבנית ה-DNA יש להסיר. הוסף RQ1 RNase ללא DNase ריכוז של U 1 / מיקרוגרם של ה-DNA התבנית דגירה של 4 שעות ב 37 ° C לעכל את תבנית ה-DNA.
- לאחר 4 שעות, לחלץ את RNA ע"י הוספת 1 נפח של ציטרט רווי פנול: כלורופורם: אלכוהול isoamyl (125:24:1, pH 4.7). מערבבים על ידי מערבולת דקות ~ 1 ו צנטריפוגות ב g 16,000 דקות 2.
- מעבירים את השלב העליון, מימית לצינור טריים או להשליך את השלב התחתון על ידי שאיפה באמצעות micropipette. הוסף 1 נפח של כלורופורם: isoamyl אלכוהול (24:1), תערובת של מערבולת דקות 1 ו צנטריפוגות כמתואר 4.4.
- מעבירים את השלב העליון, מימית לצינור טריים או להשליך את השלב התחתון על ידי שאיפה באמצעות micropipette. כלורופורם שיורית ניתן להסיר על ידי ביצוע סיבוב מהיר (10 שניות) ב microcentrifuge והסרה של השלב התחתון עם micropipette. בשלב זה, קל יותר להסיר את השלב התחתון ולא supernate.
- כדי לזרז את הרנ"א, להוסיף 0.1 בנפח המקבילה של נתרן אצטט 3M, pH 5.2, ו 1-נפח המקבילה של 2-propanol. מערבבים ומניחים במקפיא -20 מעלות צלזיוס במשך 15 דקות.
- לאחר 15 דקות, הספין במלוא המהירות, רצוי microcentrifuge בקירור ב 4 ° C, במשך 20-30 דקות כדי לזרז את המוצר RNA.
- לשאוב supernate בזהירות, לשטוף את גלולה RNA עם 0.5 מ"ל של אתנול 70%, צנטריפוגות ב 4 ° C ו להתעלמות אתנול על ידי שאיפה.
- העברת הצינור המכיל את גלולה RNA לחסום חום יבש גלולה בשעה 37 ° C ל 5 דקות.
- ממיסים את דגימת רנ"א חיץ 150 מ"מ STE, pH 8.0 (בתנאי עם ProbeQuant illustra G-50 microcolumns) או nuclease ללא מים בנפח זהה לזה של חוץ גופית התגובה שעתוק, כלומר 100 μl (שלב 4.1).
- מחממים את שפופרת המכילה את המוצר RNA על 70 מעלות צלזיוס במשך 10 דקות בתוך גוש חום ומערבבים ידי מערבולת כדי להקל על פירוק RNA.
- צנטריפוגה במהירות שיא 1 דקות בטמפרטורת החדר.
- שמור aliquot 1-μl של רנ"א בצינור 0.6-מ"ל שכותרתו עבור ספירת scintillation ו-TBE אוריאה ג'ל אלקטרופורזה polyacrylamide (חלק 6).
חלק 5: הסרה של נוקלאוטידים מאוגדים, desalting RNA, הנצנץ לספור
כדי להסיר את נוקלאוטידים מאוגדים, להשתמש בשני G-50 עמודות על פי הוראות היצרן.
- היפוך העמודות ומערבבים ידי מערבולת כדי resuspend שרף.
- הצמד סגר את החלק התחתון של העמודים על ניקוב שימוש בכלי פלסטיק המסופק בערכה ולוודא לעזוב לשקע ללא שינוי. שחרר את מכסה רבע סיבוב והמקום עמודות לתוך צינורות אוסף נקי הניתנים Gערכת -50.
- ספין העמודות צינורות לאיסוף גרם 730 דקות 1 להסיר את החיץ אחסון.
- העברת עמודות לשני חדש O-Ring הכתיר, 1.6-מ"ל צינורות לטעון 50 μl של 32 P-שכותרתו מדגם RNA לכל עמודה ישירות על גבי שרף ללא מביך שרף.
- ספין ב 730 גרם 2 דקות כדי לאסוף את RNA שכותרתו מטוהרים. לאחר שלב זה, למחוק את העמודות.
- העברת 1 μl של G-50-RNA מטוהרים אל צינור 0.6-ml עבור ספירת הנצנץ ואת אלקטרופורזה (חלק 6). חנות מניות RNA ב -20 ° C עד לשימוש Selex. רצוי כי RNA משמש בתוך 2 ימים לאחר תיוג, כדי למנוע השפלה שלה והפסד של פעילות.
- שימוש שני 1-μl aliquots RNA מן הצעדים 4.14 ו - 5.6 כדי למדוד את ספירות לדקה (CPM / μl) בעזרת מונה נצנץ. כאן, אנו משתמשים הספסל העליון Triathler מונה נצנץ.
- העברת צינורות המכילים את דגימת רנ"א מתאם בתוך הפלסטיק המשמש לספור P 32, מסופק עם Triathler. שים את הצינור ואת מתאם בתוך החדר הספירה, לסגור את המכסה של החדר, לבחור 32 לספור P ולחץ להתחיל להתחיל לספור.
- חישוב תווית% ההתאגדות כלומר, שילוב של α 32% P-CTP = (CPM / μl עבור "G-50" מדגם ÷ לדקה / μl למדגם "סך הכל") x 100. המדגם הוא מדגם לפני טיהור G-50.
- לאחר ספירת scintillation וחישוב ההתאגדות אחוזים, לחשב את התשואה של רנ"א פעילות ספציפית של רנ"א. לדוגמה, אם נחשב בבריכה של רנ"א לפני (237,370 עותקים לדקה / μl) ואחרי ה-G-50 טיהור (191,330 עותקים לדקה / μl) ההתאגדות תשואה 81%, אזי הבא מחושבים לפני השגת תשואה של רנ"א:
החל שלב 4.1, כמות α 32 P-CTP ב 3000 Ci / mmol ו 10 μCi / μl היא: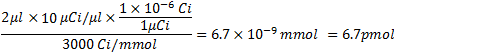 .
.
כמות ללא תווית CTP בשימוש הוא: .
.
סך CTP בשימוש הוא 100006.7pmol. משקל מולקולרי של רנ"א מחושב כדלקמן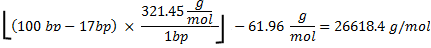
איפה 321.45 g / mol 27 הוא מסה הממוצעת של rNTPs, 100 נ"ב הוא מספר בסיסים ברצף, ו -17 נ"ב הוא מספר בסיסים האמרגן T7 שאינם עיבד. חיסור של 61.96 גר '/ שומה מ משקל מולקולרי oligonucleotide הלוקח בחשבון את הסרת HPO 2 (63.98) ואת התוספת של שני אטומי מימן (2.02) 27. מכיוון, של 4 rNTPs, CTP הוא מגיב מגביל, אם כל CTP היו להיות משולב המסה התיאורטית של RNA המיוצר היה: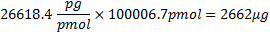 .
.
עם זאת, בגלל שילוב 81%, מסה זו היא כעת 2,156 מיקרוגרם. ספירת לכל מיקרוגרם של רנ"א יהיה אז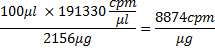
היכן 100 μl הוא נפח תגובה שעתוק, והן מבחינת שומות: 26618.4 מיקרוגרם / μmol x 8874 עותקים לדקה / מיקרוגרם = 2.4x10 8 עותקים לדקה / μmol. מכאן, כמות רנ"א nmol נפח הראוי לשמש הדגירה עם חלבון ניתן לחשב למשל, 5 RNA μl יכיל ~ 4 nmol RNA, כלומר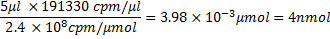
בניסויים אלו, 300 pmol לחלבון nmol 1 ו -4 nmol עד 100 nmol RNA שימשו 21.
חלק 6: אפיון המוצר RNA על ידי אלקטרופורזה ו autoradiography
- כדי להכין את דגימות אלקטרופורזה, השתמש 1-μl aliquots של רנ"א לפני ואחרי טיהור G-50 מן הצעדים 4.14 ו - 5.6, בהתאמה. הוסף חוצץ 4 STE μl ו 5 μl 2x Novex TBE-דשן מאגר דוגמאות.
- מחממים את דגימות ב 70 מעלות צלזיוס למשך 5 דקות כפי המומלץ בדרך כלל אלקטרופורזה RNA (חימום נמצאה מיותרת כי ההחלטה ג'ל של דגימות רנ"א עם ובלי חימום היא אותה התקנה זו ניסיוני).
- להרכיב 6% TBE-urea-polyacrylamide ג'ל במנגנון ג'ל פועל, למלא את החדרים מבפנים החוצה עם הצפת 1x TBE-דשן Novex ריצה. יש לשטוף את הבארות של הג'ל precast באמצעות חיץ על ידי 1-מ"ל פיפטה.
- צנטריפוגה צינורות RNA ו לטעון את דגימות (10 בסך הכל μl) באמצעות ג'ל טעינת עצות. הרץ את הג'ל על V 180 דקות 50.
- לאחר 50 דקות, לפרק את הג'ל על ידי מתפרקת תבנית פלסטיק, להסיר ולסלק רק גיבוי קצרים יותר של עובש ג'ל, אבל להשאיר את הגיבוי יותר כתמיכה עבור הג'ל.
- נקו את פני השטח של אזור העבודה עם Decon (או רדיואקטיביות אחרים decontaminant המתאימה) כדי לוודא את כל הנקודות זיהום רדיואקטיבי יוסרו.
- פריסת שתי שכבות של פלסטיק ניילון נצמד, לעטוף את הג'ל ואת גיבוי פלסטיק לעטוף שני העמיס הפלסטיק.
- לחשוף את הג'ל עטופה בפלסטיק לגיליון שלutoradiography רנטגן הסרט בתוך קלטת חשיפה בחדר חשוך תחת אור בטוח. השאירו את הקלטת ב -20 מעלות צלזיוס למשך 60-90 דקות.
- לפתח את הסרט בחדר חשוך תחת אור בטוח לאחר 60-90 דקות בעזרת מפתח הסרט אוטומטי (איור 2).
חלק 7: הדגירה RNA חלבון מחייב מסנן
ראשית, רנ"א ועל חלבונים מודגרת בתמיסה ולאחר מכן את רצפי רנ"א להיקשר לחלבון מופרדים הלא קלסרים. כפי התקדמות מחזורים Selex, מחייב לסנן ייתן אינדיקציה העשרה חלבון-RNA מחייב.
- דגירה RNA על 90 מעלות צלזיוס למשך 10 דקות ולאחר מכן עבור denaturation בטמפרטורת החדר למשך 10 דקות עבור renaturation איטי.
- ממיסים את חלבון מן הצעד 2.12 ב 8 μl 60 mM NaOH ומוסיפים מים 36 μl nuclease ללא deionized. Sonicate את התערובת במשך 1 דקות ולהוסיף 36 חיץ μl 2x RNA מחייב (20 mM טריס, 300 mM NaCl, 10 mM MgCl 2, pH 7.5).
- מערבבים את הכמות המתאימה של RNA עם 20 חיץ μl RNA 10x מחייב לפצות את נפח μl 200 על ידי הוספת nuclease ללא מים. לייבל הצינור "שליטה שלילית".
- לערבב חלבון 20 μl ואת הסכום הרצוי של רנ"א עם 20 חיץ μl RNA 10x מחייב לפצות את נפח μl 200 עם מים nuclease חינם. לייבל הצינור "תגובה".
- מערבבים דגירה צינורות למשך 30 דקות בטמפרטורת החדר. בינתיים מכינים את המסננים ההתקנה לסנן מחייבת עבור לשלב הבא.
- צרף 125-ml לצד הזרוע הבקבוק אל כניסת אבק. מניחים מראש לנקות, תמיכה זכוכית נקבובי עבור המסנן על הבקבוק בצד הזרוע.
- לאזן 3 ממברנות מסנן 2-3 מ"ל של חיץ RNA 1x מחייב בצלחת פטרי 35x10 מ"מ 10-15 דקות. המסנן הראשון ישמש להתאמת יניקה ואקום, השני ישמש RNA-לבד, שלילי שליטה מחייב לסנן, והשלישי ישמש מחייב סינון של התערובת RNA-חלבון.
- לאחר 30 דקות, צנטריפוגה התגובה מחייב צינורות לשלוט במהירות שיא עבור 5 דקות בטמפרטורת החדר.
- הפעל את הריק ואת המקום הראשון על קרום הזכוכית נקבובי. שימוש micropipette, לטפטף 0.5 מ"ל של חיץ RNA מחייב על הממברנה ולהתאים את הוואקום כדי לאפשר זרימה איטית של ירידה כל חיץ דרך הממברנה.
- מניחים את קרום second אל הכוס נקבוביים באמצעות קצב הזרימה אותו, להחיל את השליטה שלילי על הממברנה.
- החל 4x0.5-ml aliquots של חיץ RNA 1x מחייב לשטוף את הממברנה וזורקים את הזרימה דרך. שים לב שאם מראש ניקוי הוא הרצוי, את הזרימה דרך נשמרת להפקת RNA. טרום ניקוי מסיר רצפי RNA אשר נקלטות על ידי המסנן.
- הסר את הממברנה שלילי מלאה מקום לתוך צינור 1.6-מ"ל בהתאמה שכותרתו עבור ספירת הנצנץ.
- החלפת זכוכית נקבובי עם תמיכה כוס שנייה טרום לנקות נקבובי.
- הנח את הדיסק third על הזכוכית נקבוביים להחיל את תערובת התגובה. שטפו את המסנן כמו בשלב 7.11 וזורקים את הזרימה דרך. מספר שוטף ניתן להגדיל במחזורים Selex מאוחר יותר כדי להגדיל את החמרת התנאים Selex.
- הסר את הדיסק במקום לסנן לתוך צינור 1.6-מ"ל שכותרתה "תערובת התגובה" ולשמור על הנצנץ הספירה.
- בצע scintillation לספור כמו בשלב 5.8 ורושם את ספירת עבור מסננים, בהתאמה.
- חשב את רמת הרדיואקטיביות לסנן הנכנס לעומת הסכום הכולל של רדיואקטיביות להחיל את הקרומים (מחייב%). זה ייתן אינדיקציה העשרה מחייב עם התקדמות Selex.
חלק 8: מיצוי RNA מן המסננים
RNA מופק מסננים כדי לקבל את רצפי אשר נקלטות על ידי החלבון. רצפים אלה מוגבר במחזור Selex הבא.
- לאחר ספירת הנצנץ, להסיר את התגובה מחייב-מסנן מהצינור (משלב 7.15) ואת המקום לתוך נקייה ויבשה צלחת, פטרי 35x10 מ"מ.
- השתמש אזמל נקי פינצטה לחתוך את קרום לחתיכות קטנות.
- בעזרת פינצטה, להחליף את הקצוות החתוכים של הממברנה בצינור שכותרתו אותו משלב 8.1.
- הוסף חוצץ elution 400 μl (7 מ אוריאה, 3 mM EDTA, סודיום ציטראט 100 מ"מ, pH 5.0) ו דגירה הצינור ב 95 מעלות 10 דקות.
- צנטריפוגה הצינור במהירות שיא בטמפרטורת החדר, לשאוב, לאסוף את החמאה לתוך צינור השאיבה תווית חדשה.
- מדוד את הרדיואקטיביות ספירת הנותרים בצינור המכיל את פיסות קרום ידי scintillation לספור על מנת להעריך את יעילות השאיבה.
- חזור על תהליך החילוץ (צעדים 8.4-8.6) שלוש פעמים. יעילות לאחר 3 עקירות בדרך כלל ~ 95-96%.
- בתוך הצינורות המכיל את תמציות RNA, להוסיף 1 נפח (400 μl) של ציטרט רווי (pH 4.7) פנול: כלורופורם: אלכוהול isoamyl (125:24:1). מערבבים על ידי מערבולת של ~ 1 דקות ו centrifuge ב גר 16,000 דקות 2.
- מעבירים את השלב העליון, מימית לצינור טריים או להשליך את השלב התחתון על ידי שאיפה באמצעות micropipette.
- הוסף 1 נפח של כלורופורם: isoamyl אלכוהול (24:1), תערובת של מערבולת דקות 1 ו צנטריפוגות ב g 16,000 דקות 2.
- מעבירים את השלב העליון, מימית לצינור טריים או להשליך את השלב התחתון על ידי שאיפה באמצעות micropipette.
- כדי לזרז את הרנ"א, להוסיף נפח של 0.1 מ 3 נתרן אצטט, pH 5.2, גליקוגן μl 3-4 (10 מיקרוגרם / μl) כמו coprecipitant RNA, ו 1 מקביל בנפח של 2-propanol. מערבבים ומניחים במקפיא ° C -20 לילה.
- ספין בשיא המהירות, רצוי microcentrifuge בבית 4 ° C, במשך 20-30 דקות כדי לזרז את הרנ"א משלב 8.12.
- לאחר צנטריפוגה, RNA מפריד בשלב נוזל כי הוא בקושי נראה. לשאוב supernate בקפידה ללא פירוק השלב הזה נמהר שבקושי ניתן היה להבחין בחלק התחתון של הצינור.
- שטפו את הרנ"א "גלולה" עם 0.5 מ"ל של אתנול 70%, צנטריפוגה 5 דקות במהירות שיא ב -4 ° C ו להתעלמות אתנול על ידי שאיפה ללא פירוק שלב נמהר בקושי נראית לעין.
- ממיסים את גלולה רנ"א חיץ 50 STE μl והמשך טיהור G-50 כמו בשלב 5.
חלק 9: שעתוק הפוך ו PCR להמשך מחזורים Selex
כדי להמשיך למחזור הבא של Selex, RNA צריך להיות הפוך, עיבד את דנ"א מוגבר על ידי PCR.
- ב 5 שכותרתו 0.6-מ"ל צינורות, לערבב 3 μl של מטוהרים, G-50-RNA desalted עם μl 2 של פריימר 8 מתקפלת בדילול מלא, הפוך. "שליטה שלילית". תווית אחת צינור
- דגירה את התערובת על 70 מעלות צלזיוס למשך 5 דקות, ולאחר מכן על קרח למשך 5 דקות אחר כדי לאפשר הכלאה של פריימר אל RNA.
- לתערובת זו על הקרח, להוסיף 6.4 מים μl nuclease חינם, חוצץ 4 μl ImProm-II תגובה 5x, 1.6 μl 25 מ"מ MgCl 2, 1 לערבב μl dNTP 10 מ"מ, 1 μl אינהיביטור RNasein Ribonuclease, 1 μl ImProm-II Reverse transcriptase ממציא סך של μl 15.
- לשליטה השלילי, להוסיף 7.4 מים μl nuclease ללא ולהשאיר את reverse transcriptase. לייבל הצינורות בהתאם. הבקרה השלילית נכללת כדי לוודא זיהום דנ"א במחזור הקודם לא מוגבר במחזור Selex הבא. צעד זה בדיקות האפקטיביות של מערכת העיכול של תבנית ה-DNA על ידי DNase RQ1 RNase ללא בשלב 4.3.
- השתמש Cycler תרמי כדי לדגור את תערובת התגובה של 25 מעלות צלזיוס למשך 5 דקות, 42 ° C ל h 1 עבור חישול הרחבה של גדיל הדנ"א הראשונה, בהתאמה, ואחריהם 70 מעלות צלזיוס למשך 15 דקות כדי להשבית reverse transcriptase.
- לאחר תגובה הפוכה שעתוק, להגדיר את התמהיל PCR. הוסף 30 μl מים nuclease חינם, 10 חיץ μl 10x תקי, 30 μl 25 MgCl 2, mM 7 mM μl 10 לערבב dNTP, 1 μl של כל צבע יסוד, ו 1 פולימראז תקי μl בכל הצינורות.
- הפעל את תוכנית ה-PCR כמו חלק 3 עבור 9-14 מחזורים.
- לטהר את מוצר ה-DNA כמו בשלב 3.3.
- מיקס 8 μl של מוצר ה-DNA עם 2 צבע טעינת μl דנ"א electrophorese agarose על 2% ב V 100 דקות 15-20 (איור 3).
- מוצר ה-DNA הוא מוכן להיות עיבד ל RNA שכותרתו כמו חלק 4 ומשמש למחזור הבא של Selex.
פרק 10: תוצאות נציג
בניסויים Selex, אופי Aβ40 oligomers לשמש יעד, איכות RNA המשמש לכל מחזור, ומצליח מיצוי רנ"א הגברה לאחר כל מחזור חשובים. השתמשנו PICUP ליצור תערובת oligomeric Aβ40 עבור Selex לאחר טיהור וסילוק של cross-linking ריאגנטים. הניסויים שתוארו desalting חלק 2 בדרך כלל להוביל לאובדן חלבון 50-55%. כמות חלבון איכותי ניתן להעריך באמצעות מדידות ספיגת (λ = 280) ו SDS-PAGE (איור 1). הפרופיל הממוצע של ספיגת Aβ40 eluates מתוך 5 ניסויים בודדים שכיסה פרופיל טיפוסי SDS-PAGE של Aβ40 eluted באחד הניסויים האלה הם שמוצג באיור 1. הנתונים מראים כי החלבון בעקביות elutes את העמודה שברים 3-5 ואת ריאגנטים cross-linking elute לאחר שבריר 6 (ספיגת גדל ב -7 שבר, איור 1). SDS-PAGE מציג את התפלגות טיפוסית Aβ40 oligomer 22. הפצה זו היתה לשחזור לאחר שברים חלבון היו lyophilized (2.11), שטופלו HFIP (2.11), resolubilized (7.1), ו מחדש ונותחו על ידי SDS-PAGE.
Integrity של רנ"א לכל מחזור Selex חשוב גם עבור איטרטיבי התקדמות Selex, במיוחד כאשר nuclease-רגישים ribo-oligonucleotides משמשים. אחרי הגברה RNA וסימון (חלק 4), איכות RNA שכותרתו ניתן להעריך על ידי אלקטרופורזה TBE-urea-polyacrylamide ג'ל. פרופיל טיפוסי של מוצר שלם RNA שכותרתו לפני ואחרי ה-G-50 טיהור (Pאמנות 5) מוצג באיור 2.
לאחר כל מחזור Selex, RNA מופק הממברנה לאחר סינון מחייב (חלק 8) ו - reverse-עיבד (חלק 9) ל-DNA של הגברה PCR (שלב 9.5). תבנית ה-DNA ממעגל קודמת לאחר מכן נעשה שימוש כדי לייצר 32 P-שכותרתו RNA (חלק 4) למחזור הבא. אם חלק תבנית ה-DNA לזהם ממעגל קודמת נמשכת במוצר RNA שכותרתו לאחר חוץ גופית תגובות שעתוק (חלק 4), היעילות של מחזורי Selex יופחת, תובעני יותר מחזורים. כדי לשלוט על זה, לאחר כל מחזור Selex והתגובה המקביל הפוכה שעתוק PCR, אלקטרופורזה agarose מבוצע (9.12). היעדרות של מוצר ה-DNA בשלילה שליטה צינורות תגובה (9.4) מצביעה על הסרה מוצלחת של תבנית ה-DNA שמקורם במחזור Selex הקודמים (תרשים 3). אם הגברה DNA על ידי ה-PCR הוא ציין בצינור שלילי לשלוט, מומלץ כי משך הדגירה עם RQ1 DNase (4.3) להיות ממושך. יצרן המומלץ משך הדגירה עם RQ1 DNase הוא 15 דקות, לעומת זאת, מצאנו כי incubations יותר (4-5 שעות) נדרשו להסיר את תבנית ה-DNA לחלוטין (איור 3).
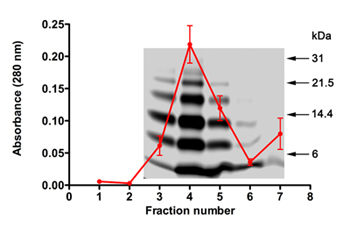
באיור 1. SDS-PAGE ופרופיל ספיגת PICUP שנוצר, Aβ40 oligomers desalted. הפרופיל ספיגת מתוך 5 עמודות desalting הפרט היה בממוצע ו overlain על ג'ל נציג. סמנים משקל מולקולרי מוצגים בצד ימין.

איור 2. TBE-urea-polyacrylamide ג'ל אלקטרופורזה של המוצר RNA לפני ואחרי טיהור G-50. כיוון הנדידה של המוצר RNA הוא מן הקתודה לאנודה כמצוין.
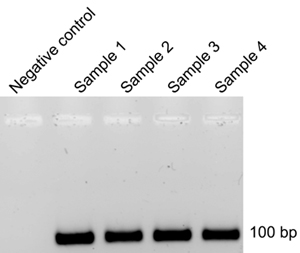
3. איור ג'ל אלקטרופורזה agarose של מוצר ה-DNA לאחר תעתוק הפוך הגברה-PCR.
Discussion
נקודת המוצא של התהליך Selex הוא סינתזה של ספריה oligonucleotide אקראי המכיל בדרך כלל 10 -10 12 15 רצפים. ב-DNA Selex, ספריה זו משמשת מיד לאחר בריכה ssDNA שנוצר, ואילו RNA Selex, הפגינו כאן, הספרייה ssDNA מומר הראשון בריכת RNA enzymatically ידי שעתוק חוץ גופית. ואז, Selex מבוצע iteratively לפיה כל מחזור כו...
Disclosures
Acknowledgements
עבודה זו נתמכה על ידי מענקים AG030709 מ-NIH / NIA ו 07-65798 מן המחלקה לבריאות הציבור בקליפורניה. אנו מכירים מרגרט מ Condron לסינתזה פפטיד וניתוח חומצת אמינו, ד"ר אליזבת פ 'נויפלד לסיוע ותמיכה בשלבים הראשוניים של הפרויקט, ד"ר צ'י קונג ב חן למתן תמיכה ריאגנטים, וד"ר אנדרו D . אלינגטון לדיונים מועילים.
Materials
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| Aβ40 | UCLA Biopolymers Laboratory | Lyophilized powder | ||
| MX5 Automated-S Microbalance | Mettler Toledo | |||
| Silicon-coated, 1.6-ml tubes | Denville Scientific | C19033 or C19035 | ||
| 1,1,1,3,3,3-hexafluoro-2-propanol (HFIP) | TCI America | H0424 | Use in a fume hood. | |
| Ammonium persulfate | Sigma | A-7460 | Vortex until the solution is clear. APS is prepared freshly each time and should be used within 48 h. | |
| Tris(2,2-bipridyl)dichlororuthenium(II) hexahydrate | Sigma | 224758-1G | Vortex until the solution is clear. Cover the RuBpy tube with foil to protect the reagent from ambient light. RuBpy is prepared freshly each time and should be used within 48 h. | |
| Dithiothreitol (DTT) | Sigma | 43815 | ||
| D-Salt™ Excellulose™ desalting columns | Thermo Scientific | 20449 | ||
| Ammonium acetate | Fisher Scientific | A637-500 | ||
| Silicon-coated, 0.6-ml tubes | Denville Scientific | C19063 | ||
| Novex Tricine Gels (10–20%) | Invitrogen | EC6625B0X | 10-well; mini size (8 cm X 8 cm); 25 μl loading volume per well; separation range 5 kDa to 40 kDa | |
| Quartz cuvette | Hellma | 105.250-QS | ||
| Beckman DU 640 spectrophotometer | Beckman | |||
| ssDNA library | Integrated DNA Technologies | Custom-ordered | The library was designed to contain 49 random nucleotides flanked by two constant regions containing primer-binding and cloning sites: 5'-TAA TAC GAC TCA CTA TAG GGA ATT CCG CGT GTG C (N:25:25:25:25%) (N)49 G TCC GTT CGG GAT CCT C-3' | |
| Taq DNA polymerase | USB Corporation | 71160 | Recombinant Thermus aquaticus DNA Polymerase supplied with 10× PCR Buffer and a separate tube of 25 mM MgCl2 for routine PCR. | |
| PCR Nucleotide Mix, 10 mM solution | USB Corporation | 77212 | (10 mM each dATP, dCTP, dGTP, dTTP) | |
| Forward primer | Integrated DNA Technologies | Custom-ordered | 5'-TAA TAC GAC TCA CTA TAG GGA ATT CCG CGT GTG C-3' | |
| Reverse primer | Integrated DNA Technologies | Custom-ordered | 5'-GAG GAT CCC GAA CGG AC-3' | |
| Thermal cycler | Denville Scientific | Techne TC-312 | ||
| QIAquick PCR Purification Kit (50) | QIAGEN | 28104 | ||
| Agarose | Denville Scientific | CA3510-8 | ||
| Conical, sterile 1.6-ml tubes with caps attached with O-rings | Denville Scientific | C19040-S | ||
| RiboMAX™ Large Scale RNA Production System–T7 | Promega | P1300 | The kit contains: 120 μl Enzyme Mix (RNA polymerase, recombinant RNasin® ribonuclease inhibitor and recombinant inorganic pyrophosphatase); 240 μl transcription 5 buffer; 100 μl each of 4 rNTPs, 100 mM; 110 U RQ1 RNase-free DNase, 1 U/μl; 10 μl linear control DNA, 1 mg/ml; 1 ml 3M sodium acetate (pH 5.2); 1.25 ml nuclease-Free water | |
| α-32P-cytidine 5'-triphosphate, 250 μCi (9.25 MBq), | Perkin Elmer | BLU008H250UC | Specific Activity: 3000 Ci (111 TBq)/mmol, 50 mM Tricine (pH 7.6) | |
| Citrate-saturated phenol:chloroform:isoamyl alcohol (125:24:1, pH 4.7) | Sigma (Fluka) | 77619 | ||
| Chloroform:Isoamyl alcohol (24:1) | Sigma | C0549 | ||
| Absolute ethanol for molecular biology | Sigma | E7023 | ||
| Z216-MK refrigerated microcentrifuge | Denville Scientific | C0216-MK | ||
| illustra ProbeQuant™ G-50 Micro Columns | GE Healthcare | Obtained from Fisher Scientific (45-001-487) | Prepacked with Sephadex™ G-50 DNA Grade and pre-equilibrated in STE buffer containing 0.15% Kathon as Biocide | |
| Triathler Bench-top Scintillation counter | Hidex Oy, Turku, Finland | Triathler LSC Model: 425-034 | ||
| Novex® TBE-Urea Sample Buffer (2×) | Invitrogen | LC6876 | ||
| 6% TBE-Urea Gels 1.0 mm, 10 wells | Invitrogen | EC6865BOX | ||
| Novex® TBE Running Buffer (5×) | Invitrogen | LC6675 | ||
| Radioactivity decontaminant | Fisher Scientific | 04-355-67 | ||
| Gel-loading tips | Denville Scientific | P3080 | ||
| XCell SureLock Mini-Cell | Invitrogen | EI0001 | XCell SureLock Mini-Cell | |
| Autoradiography film | Denville Scientific | E3018 | Use in complete darkness | |
| Autoradiography film, Hyperfilm™ ECL | Amersham Biosciences | RPN3114K | Can be used under red safe light. | |
| Membrane discs | Millipore | GSWP02500 | Mixed cellulose ester, hydrophilic, 0.22-μm disc membranes | |
| Fritted glass support base for 125-ml flask | VWR | 26316-696 | ||
| Petri dishes | Fisher Scientific | 08-757-11YZ | ||
| Urea | Fisher Scientific | AC32738-0050 | ||
| EDTA | Fisher Scientific | 118430010 | ||
| Glycogen | Sigma | G1767 | ||
| 2-Propanol for molecular biology | Sigma | I9516 | ||
| Recombinant RNase inhibitor | USB Corporation | 71571 | ||
| ImProm-II™Reverse Transcription System | Promega | A3802 | ||
| Recombinant RNase inhibitor | USB Corporation | 71571 | ||
| RapidRun™ Loading Dye | USB Corporation | 77524 |
References
- Monien, B. H., Apostolova, L. G., Bitan, G. Early diagnostics and therapeutics for Alzheimer's disease-how early can we get there. Expert. Rev. Neurother. 6, 1293-1306 (2006).
- Nestor, P. J., Scheltens, P., Hodges, J. R. Advances in the early detection of Alzheimer's disease. Nat. Med. 10, S34-S41 (2004).
- Kawas, C. H. Clinical practice. Early Alzheimer's disease. N. Engl. J. Med. 349, 1056-1063 (2003).
- Haass, C., Selkoe, D. J. Soluble protein oligomers in neurodegeneration: lessons from the Alzheimer's amyloid β-peptide. Nat. Rev. Mol. Cell. Biol. 8, 101-112 (2007).
- Kirkitadze, M. D., Bitan, G., Teplow, D. B. Paradigm shifts in Alzheimer's disease and other neurodegenerative disorders: the emerging role of oligomeric assemblies. J. Neurosci. Res. 69, 567-577 (2002).
- Rahimi, F., Shanmugam, A., Bitan, G. Structure-function relationships of pre-fibrillar protein assemblies in Alzheimer's disease and related disorders. Curr. Alzheimer Res. 5, 319-341 (2008).
- Jayasena, S. D. Aptamers: an emerging class of molecules that rival antibodies in diagnostics. Clin. Chem. 45, 1628-1650 (1999).
- Bunka, D. H., Stockley, P. G. Aptamers come of age - at last. Nat. Rev. Microbiol. 4, 588-596 (2006).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Lee, J. F., Stovall, G. M., Ellington, A. D. Aptamer therapeutics advance. Curr. Opin. Chem. Biol. 10, 282-289 (2006).
- Weiss, S. RNA aptamers specifically interact with the prion protein PrP. J. Virol. 71, 8790-8797 (1997).
- Bibby, D. F. Application of a novel in vitro selection technique to isolate and characterise high affinity DNA aptamers binding mammalian prion proteins. J. Virol. Methods. 151, 107-115 (2008).
- Rhie, A. Characterization of 2'-fluoro-RNA aptamers that bind preferentially to disease-associated conformations of prion protein and inhibit conversion. J. Biol. Chem. 278, 39697-39705 (2003).
- King, D. J., Safar, J. G., Legname, G., Prusiner, S. B. Thioaptamer interactions with prion proteins: sequence-specific and non-specific binding sites. J. Mol. Biol. 369, 1001-1014 (2007).
- Proske, D. Prion-protein-specific aptamer reduces PrPSc formation. ChemBioChem. 3, 717-725 (2002).
- Murakami, K., Nishikawa, F., Noda, K., Yokoyama, T., Nishikawa, S. Anti-bovine prion protein RNA aptamer containing tandem GGA repeat interacts both with recombinant bovine prion protein and its β isoform with high affinity. Prion. 2, 73-80 (2008).
- Luhrs, T., Zahn, R., Wuthrich, K. Amyloid formation by recombinant full-length prion proteins in phospholipid bicelle solutions. J. Mol. Biol. 357, 833-841 (2006).
- Bunka, D. H. Production and characterization of RNA aptamers specific for amyloid fibril epitopes. J. Biol. Chem. 282, 34500-34509 (2007).
- Ylera, F., Lurz, R., Erdmann, V. A., Furste, J. P. Selection of RNA aptamers to the Alzheimer's disease amyloid peptide. Biochem. Biophys. Res. Commun. 290, 1583-1588 (2002).
- Rahimi, F., Murakami, K., Summers, J. L., Chen, C. H., Bitan, G. RNA aptamers generated against oligomeric Aβ40 recognize common amyloid aptatopes with low specificity but high sensitivity. PLoS ONE. 4, e7694-e7694 (2009).
- Bitan, G., Lomakin, A., Teplow, D. B. Amyloid β-protein oligomerization: prenucleation interactions revealed by photo-induced cross-linking of unmodified proteins. J. Biol. Chem. 276, 35176-35184 (2001).
- Rahimi, F., Maiti, P., Bitan, G. Photo-induced cross-linking of unmodified proteins (PICUP) applied to amyloidogenic peptides. J. Vis. Exp. , (2009).
- Bitan, G., Fradinger, E. A., Spring, S. M., Teplow, D. B. Neurotoxic protein oligomers-what you see is not always what you get. Amyloid. 12, 88-95 (2005).
- Bitan, G. Structural study of metastable amyloidogenic protein oligomers by photo-induced cross-linking of unmodified proteins. Methods Enzymol. 413, 217-236 (2006).
- Chen, C. H., Chernis, G. A., Hoang, V. Q., Landgraf, R. Inhibition of heregulin signaling by an aptamer that preferentially binds to the oligomeric form of human epidermal growth factor receptor-3. Proc. Natl. Acad. Sci. USA. 100, 9226-9231 (2003).
- Adams, D. S. . Lab math: a handbook of measurements, calculations, and other quantitative skills for use at the bench. , (2003).
- Gopinath, S. C. Methods developed for SELEX. Anal. Bioanal. Chem. 387, 171-182 (2007).
- Takahashi, T., Tada, K., Mihara, H. RNA aptamers selected against amyloid β-peptide (Aβ) inhibit the aggregation of Aβ. Mol. Biosyst. 5, 986-991 (2009).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved