È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Selezione di Aptamers per β-amiloide proteina, l'agente eziologico della malattia di Alzheimer
In questo articolo
Riepilogo
Aptameri sono ribo-/deoxyribo-oligonucleotides corti selezionati In vitro Metodi evoluzione sulla base di affinità per un target specifico. Aptameri sono strumenti di riconoscimento molecolare con versatili applicazioni terapeutiche, diagnostiche e di ricerca. Dimostriamo metodi per la selezione di aptameri per la β-amiloide, proteina, l'agente eziologico della malattia di Alzheimer.
Abstract
Malattia di Alzheimer (AD) è un progressista, età-dipendente, malattia neurodegenerativa con un corso insidioso che rende difficile la diagnosi presintomatica 1. DC diagnosi definitiva si ottiene solo post-mortem, stabilendo così presintomatica, diagnosi precoce di Alzheimer è cruciale per lo sviluppo e l'amministrazione terapie efficaci 2,3.
Proteina β-amiloide (Aβ) è fondamentale per la patogenesi dell'Alzheimer. Solubile, le assemblee oligomeriche Aβ si ritiene di influenzare neurotossicità sottostante disfunzione sinaptica e perdita di neuroni in AD 4,5. Varie forme di Aβ solubile assemblee sono stati descritti, tuttavia, le loro interrelazioni e la pertinenza ad eziologia e la patogenesi dC sono complessi e non ben compreso 6. Specifici strumenti di riconoscimento molecolare può svelare i rapporti tra assemblee Aβ e facilitare l'individuazione e caratterizzazione di queste assemblee nelle prime fasi del decorso della malattia prima che i sintomi emergere. Il riconoscimento molecolare si basa generalmente su anticorpi. Tuttavia, una classe alternativa di strumenti di riconoscimento molecolare, aptameri, offre importanti vantaggi rispetto agli anticorpi 7,8. Aptameri oligonucleotidi sono generate da in-vitro selezione: evoluzione sistematica dei ligandi di arricchimento esponenziale (SELEX) 9,10. SELEX è un processo iterativo che, simile a quello dell'evoluzione darwiniana, permette la selezione, l'amplificazione, l'arricchimento, e la perpetuazione di una proprietà, ad esempio, avido, specifico, di legame (aptameri) o l'attività catalitica (ribozimi e DNAzymes).
Nonostante la comparsa di aptameri come strumenti nel campo della biotecnologia moderna e della medicina 11, sono stati sottoutilizzati nel campo amiloide. RNA pochi o aptameri ssDNA sono stati selezionati contro varie forme di proteine prioniche (PrP) 12-16. Un aptamero RNA generati contro ricombinante PrP bovina ha dimostrato di riconoscere bovina PrP-β 17, una solubile, oligomerici, β-sheet ricco di variante conformazionale di full-length PrP che si forma fibrille amiloidi 18. Aptameri generati utilizzando forme monomeriche e molti dei β fibrillare sono stati trovati 2-microglobulina (β 2 m) per legare fibrille di alcune altre proteine amiloidogenica β oltre 2 m fibrille 19. Ylera et al. descritto aptameri RNA selezionato contro immobilizzato monomerico Aβ40 20. Inaspettatamente, queste aptameri legato fibrillare Aβ40. Complessivamente, questi dati sollevano diverse questioni importanti. Perché aptameri selezionati contro proteine monomeriche riconoscere loro forme polimeriche? Potrebbe aptameri contro le forme monomerica e / o oligomerici di proteine amiloidogenica ottenere? Per rispondere a queste domande, abbiamo cercato di selezionare aptameri per covalentemente stabilizzato oligomerici Aβ40 21 generati utilizzando foto-indotta cross-linking delle proteine non modificato (PICUP) 22,23. Simile a risultati precedenti 17,19,20, questi aptameri reagito con fibrille di altre proteine Aβ e diversi amiloidogenico probabilmente riconoscere un amiloide potenzialmente comuni strutturali aptatope 21. Qui, vi presentiamo la metodologia SELEX utilizzati nella produzione di questi aptameri 21.
Protocollo
Parte 1: preparazione di proteine e di cross-linking
Inizialmente, la proteina utilizzata per SELEX è pretrattati con 1,1,1,3,3,3-esafluoro-2-propanolo (HFIP) per ottenere omogeneo, aggregato senza preparazioni, come descritto in precedenza 23. Questo passaggio è necessario perché preformato aggregati indurre una rapida aggregazione di proteine amiloidogenica, con conseguente scarsa riproducibilità sperimentale 24, e sono indesiderabili per la selezione di aptameri per non aggregati, non fibrillare forme della proteina.
- Pesare ~ 800 mcg (~ 180 nmol) puro Aβ40 utilizzando una microbilancia. Trasferire il peptide secco liofilizzato in etichetta, siliconato, a basso adsorbenti 1,6 ml tubi microcentrifuga.
- Sciogliere il peptide in 100% 1,1,1,3,3,3-esafluoro-2-propanolo (HFIP, 400 microlitri) per ottenere una soluzione 0,5 mM peptide come descritto in precedenza 23.
- Dividere la soluzione in quattro aliquote tale che ogni tubo contiene ~ 45 nmol Aβ40 nominalmente 100 microlitri. Procedere alla rimozione del HFIP come descritto in precedenza 23.
- Prima solubilizzante il HFIP trattati con peptidi in tampone per il cross-linking reazioni, preparare il cross-linking e reagenti di raffreddamento. Pesare persolfato di ammonio (APS, MW 228,2 g / mol) e preparare una soluzione di 40 mm in 10 mM di fosfato di sodio, pH 7.4. Mescolare con un vortice fino a quando la soluzione è limpida.
- Preparare soluzione 2 mM di Tris (2,2-bipyridyl) dichlororuthenium (II) esaidrato (RuBpy MO 748,63 g / mol) in 10 mM fosfato di sodio, pH 7.4. Mescolare con un vortice e verificare la completa dissoluzione. Proteggere il tubo dalla luce con un foglio di alluminio.
- Preparare il reagente tempra. Pesare ditiotreitolo (DTT, MW 154,5 g / mol) e si sciolgono in acqua deionizzata o in 10-mM fosfato di sodio, pH 7.4, a 1 M.
- Sciogliere il HFIP trattati con peptide esattamente come descritto in precedenza 23,25, ma lo scopo di ottenere ~ 60 soluzione peptide micron.
- Eseguire PICUP per generare una miscela di oligomerici Aβ40 come descritto in precedenza 23. Una reazione tipica PICUP viene eseguita in un volume di 20 l in cui le concentrazioni finali di proteine, RuBpy, e APS sono 30 micron, 0,05 mm e 1 mm, rispettivamente 23. Qui, la miscela utilizzata per PICUP contiene 108 microlitri di proteine, 6 RuBpy microlitri, 6 APS microlitri, e 1 DTT microlitri e la concentrazione di proteine, RuBpy, e APS sono raddoppiate rispetto alla reazione tipica. Questo riduce il numero di reazioni PICUP da eseguire e aumenta la concentrazione di proteine per la dissalazione.
Parte 2: Dissalazione la preparazione di proteine
Prima di utilizzare la proteina per SELEX, dissalazione viene effettuata per rimuovere i reagenti reticolazione utilizzato per PICUP. Questa miscela di reazione PICUP contiene i reagenti cross-linking e mM ~ 55 proteine (concentrazione nominale).
- Togliere il tappo superiore off a 5 ml colonna dissalazione, sostenere la colonna da uno stand, posizionare un bicchiere sotto l'uscita della colonna, e rimuovere il tappo di scarico. Lasciate che il flusso del buffer di archiviazione tramite e raccogliere nel becher.
- Equilibrare la colonna in 10 mM ammonio acetato, pH 8.3. Aggiungere 5 volumi di resina letto (25 ml) di questo buffer in 3-ml aliquote alla colonna e permettergli di fluire attraverso.
- Nel frattempo, etichetta 8 basso adsorbenti, siliconato da 1,6 ml, tubi e metterli in un rack tubo.
- Dopo che la colonna è equilibrata, applicare un 0,5-0,7 ml aliquota della miscela di reazione PICUP per colonna dissalazione per uso singolo (colonne possono essere lavati, memorizzati, ed equilibrato per un successivo uso). Lasciare che la miscela proteica di penetrare nel resina colonna e scartare il deflusso nel bicchiere.
- Posizionare il tubo prima collezione sotto l'uscita della colonna. Aggiungere 0,5 ml di tampone acetato alla colonna e raccogliere i primi 0,5 ml frazione scorre.
- Ripetere i passi 2.4 e 2.5, e raccogliere fino a otto 0,5 ml frazioni nei tubi corrispondente.
- Numero di un altro set di 8 a bassa adsorbenti, siliconato, piccoli tubi di 0,6 ml e metterli in un rack. Etichetta un altro tubo come "vuoto". Togliere 150 ml di ogni frazione e il trasferimento in etichetta 0,6 ml tubi. Usare 20 l per SDS-PAGE analisi (Figura 1) come mostrato in precedenza 23.
- Trasferire 150 microlitri di acetato di ammonio 10 mM, pH 8.3, nel tubo vuoto. Utilizzare questo tubo per impostare il vuoto in uno spettrofotometro.
- Misurare l'assorbanza in 130 ml di ogni frazione a λ = 280 nm in una cuvetta al quarzo.
- Dopo l'assorbanza è misurata, unire le frazioni con il più alto contenuto proteico (tipicamente frazioni 3-5), mescolare delicatamente con una pipetta, e mantenere un 10-microlitri aliquota per l'analisi di aminoacidi per determinare l'effettiva concentrazione di proteine (non mostrato).
- Dividere il campione in diverse aliquote di circa 2 proteine nmol per tubo e liofilizzare i campioni in un liofilizzatore.
- Dopo il completamento della liofilizzazione, trattare i campioni con il 100% HFIP come prima.
- Conservare le provette a -20 ° C. Utilizzare un tubo per ogni ciclo di SELEX (sotto).
Parte 3: Amplificazione della sintesi casuale ssDNA biblioteca mediante PCR
La biblioteca sintetico ssDNA usato qui per SELEX incluso 49 nucleotidi randomizzati (A: T: G: C = 25:25:25:25%) affiancata da regioni costanti che comprende siti di clonazione (Bam HI, Eco RI) e un promotore T7, come descritto in precedenza 26.
- Per amplificare la biblioteca, impostare una reazione standard di PCR come segue: 202 microlitri di acqua deionizzata, 40 microlitri di buffer 10x Taq DNA polimerasi, 2 modello microlitri ssDNA (0,5 nmol), 120 microlitri 25 mM MgCl 2, 28 microlitri 10 deossinucleosidici mix mM trifosfato (dNTPs), 1 fondo microlitri in avanti (300 pmol), 1 fondo retromarcia microlitri (300 pmol), e 4 microlitri Taq polimerasi.
- Eseguire la reazione di PCR utilizzando un termociclatore con le seguenti impostazioni: 5 minuti a 94 ° C per la denaturazione iniziale, ogni 20 cicli di 94 ° C per 30 sec, 52 ° C per 30 secondi per la ricottura, 72 ° C per 30 secondi per estensione, e l'estensione finale a 72 ° C per 7 min.
- Dopo il completamento della reazione PCR, purificare il prodotto DNA amplificato utilizzando il kit Qiagen QIAquick purificazione PCR secondo le istruzioni del produttore. Generalmente, in questi esperimenti la concentrazione e la resa del DNA sono 160 ± 10 ng / mL in 50 microlitri.
- Verificare l'importo del DNA dopo PCR del 2%, elettroforesi su gel di agarosio.
Parte 4: Generazione di 32 P-etichettati RNA in vitro di trascrizione
- Impostare la reazione di trascrizione in un O-ring-capped, 1,6 ml tubo secondo le istruzioni del produttore con alcune modifiche come segue: 20 l 5x tampone trascrizione T7, 7,5 microlitri ciascuno dei mM RATP 100, rGTP, rUTP, 1 ml 100 mM rCTP, 2 microlitri α 32 P-CTP (3000 Ci / mmol), 5-10 mg purificato modello di DNA (~ 30-40 microlitri di DNA purificato prodotto), 10 microlitri miscela di enzimi, e portare il volume finale di 100 ul con l'aggiunta di acqua priva di nucleasi.
- Mescolare delicatamente la soluzione con una pipetta, centrifuga la miscela, e incubare a 37 ° C durante la notte.
- Alla fine della reazione, il modello del DNA deve essere rimosso. Aggiungi RQ1 DNasi RNasi-free per una concentrazione di 1 U / mg di DNA stampo e incubare per 4 ore a 37 ° C a digerire il modello del DNA.
- Dopo 4 ore, estrarre l'RNA con l'aggiunta di 1 volume di citrato saturi fenolo: cloroformio: alcool isoamilico (125:24:1, pH 4,7). Mescolare con un vortice di ~ 1 min e centrifugare a 16.000 g per 2 min.
- Trasferimento in alto, fase acquosa in una nuova provetta o eliminare la fase inferiore di aspirazione mediante una micropipetta. Aggiungere 1 volume di cloroformio: alcool isoamilico (24:1), mescolare con un vortex per 1 min e centrifugare, come descritto in 4.4.
- Trasferimento in alto, fase acquosa in una nuova provetta o eliminare la fase inferiore di aspirazione mediante una micropipetta. Cloroformio residuo può essere rimosso effettuando un giro veloce (10 secondi) in una microcentrifuga e la rimozione della fase di fondo con una micropipetta. In questa fase, è più facile rimuovere la fase di fondo piuttosto che il supernate.
- Per precipitare l'RNA, aggiungere 0,1 volume equivalente di acetato di sodio 3M, pH 5,2, e 1-volume equivalente di 2-propanolo. Mescolare e mettere in un congelatore di -20 ° C per 15 min.
- Dopo 15 minuti, girare alla massima velocità, preferibilmente in una microcentrifuga refrigerato a 4 ° C, per 20-30 minuti per far precipitare il prodotto RNA.
- Aspirare il supernate attenzione, lavare il pellet di RNA con 0,5 ml di etanolo al 70%, centrifugare a 4 ° C e scartare l'etanolo mediante aspirazione.
- Trasferire la provetta contenente il pellet di RNA a un blocco di calore e asciugare il pellet a 37 ° C per 5 min.
- Sciogliere il campione di RNA in 150 mM tampone STE, pH 8.0 (fornito con il ProbeQuant illustrazioni G-50 microcolonne) o acqua priva di nucleasi a un volume identico a quello della reazione in vitro la trascrizione ad esempio, 100 l (punto 4.1).
- Riscaldare il tubo contenente il prodotto RNA a 70 ° C per 10 minuti in un blocco di calore e mescolare con un vortice per facilitare la dissoluzione RNA.
- Centrifugare alla massima velocità per 1 minuto a temperatura ambiente.
- Tenere un 1-microlitri aliquota di RNA in una etichettato 0,6 ml di tubo per il conteggio a scintillazione e TBE-urea elettroforesi su gel di poliacrilammide (parte 6).
Parte 5: rimozione di nucleotidi prive di personalità giuridica, la desalinizzazione RNA, e il conteggio a scintillazione
Per rimuovere i nucleotidi prive di personalità giuridica, utilizzare due G-50 colonne secondo le istruzioni del produttore.
- Invertire le colonne e mescolare con un vortice di risospendere la resina.
- Snap off la chiusura inferiore della colonna a perforazione utilizzando lo strumento di plastica fornita nel kit e assicurarsi di lasciare intatta la presa. Allentare il tappo di un quarto di giro e posto le colonne in provette di raccolta pulita fornita nel G-50 Kit.
- Spin le colonne nei tubi di raccolta a 730 g per 1 min per rimuovere il buffer di memoria.
- Trasferimento colonne di due nuovi O-ring-capped, 1,6 ml tubi e carico di 50 l di 32 P-marcato campione di RNA per colonna direttamente sulla resina senza perturbare la resina.
- Spin a 730 g per 2 minuti per raccogliere le RNA purificato etichettati. Dopo questo passo, scartare le colonne.
- Trasferire 1 ml di G-50-RNA purificato a 0,6 ml di tubo di scintillazione per il conteggio e l'elettroforesi (Parte 6). Conservare magazzino RNA a -20 ° C fino al momento dell'uso per SELEX. È auspicabile che l'RNA viene utilizzato entro 2 giorni dopo l'etichettatura al fine di evitare il degrado e la perdita di attività.
- Utilizzare le due 1-microlitri aliquote RNA passi da 4,14 e 5,6 per misurare i conteggi al minuto (cpm / mL) con un contatore a scintillazione. Qui, si usa il banco contatore a scintillazione Triathler.
- Trasferire i tubi contenenti i campioni di RNA all'interno l'adattatore in plastica utilizzati per 32 il conteggio P, dotato di Triathler. Mettere il tubo e l'adattatore all'interno della camera di conteggio, chiudere il coperchio della camera, scegliere 32 contando P e premere start per iniziare il conteggio.
- Calcolare etichetta%, cioè l'incorporazione, l'incorporazione% di α-32 P = CTP (cpm / mL per "G-50" campione ÷ cpm / mL per campione "TOTALE") x 100. Campione totale è il campione prima G-50 di purificazione.
- Dopo la conta delle scintillazioni e calcolo di incorporazione per cento, calcolare il rendimento di RNA e l'attività specifica di RNA. Per esempio, se i conteggi per un pool di RNA prima (237.370 cpm / mL) e dopo il G-50 di purificazione (191.330 cpm / mL) l'incorporazione di rendimento 81%, quindi la seguente sono calcolati prima di ottenere la resa di RNA:
Dal punto 4.1, la quantità di α-32 P CTP a 3000 Ci / mmol e 10 μCi / mL è: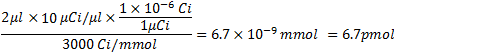 .
.
La quantità di CTP senza etichetta utilizzata è: .
.
Importo totale dei CTP utilizzato è 100006.7pmol. Il peso molecolare di RNA è calcolato come segue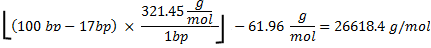
cui 321,45 g / mol 27 è la massa media dei rNTPs, 100 bp è il numero di basi della sequenza, e 17 bp è il numero di basi nel promotore T7 che non sono trascritti. Sottrazione di 61,96 g / mole dal peso molecolare oligonucleotide prende in considerazione la rimozione di HPO 2 (63,98) e l'aggiunta di due atomi di idrogeno (2.02) 27. Perché, dei 4 rNTPs, CTP è il reagente limitante, se tutti i CTP sono stati da incorporare massa teorica di RNA prodotto sarebbe stato: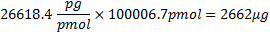 .
.
Tuttavia, a causa del 81% incorporazione, questa massa è 2156 mg. Conteggi per mg di RNA sarebbe poi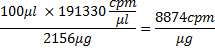
dove 100 l è il volume di reazione di trascrizione, sia in termini di moli: 26618,4 mg / mmol x 8874 cpm / mg = 2.4x10 8 cpm / mmol. Da qui, la quantità di RNA in nmol e il suo volume impiega nel caso delle proteine con l'incubazione può essere calcolato ad esempio, 5 microlitri di RNA conterrà ~ 4 nmol RNA, cioè,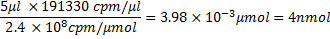
In questi esperimenti, 300 pmol a 1 nmol di proteine e 4 nmol a 100 nmol RNA sono stati utilizzati 21.
Caratterizzazione del prodotto RNA mediante elettroforesi e autoradiografia: parte 6
- Per preparare i campioni per elettroforesi, utilizzare l'1-microlitri aliquote di RNA prima e dopo il G-50 di purificazione dai passi 4,14 e 5,6, rispettivamente. Aggiungere 4 tampone microlitri STE e 5 microlitri 2x Novex TBE-Urea Sample Buffer.
- Riscaldare i campioni a 70 ° C per 5 minuti come di solito raccomandato per l'elettroforesi di RNA (riscaldamento è risultato inutile perché la risoluzione gel dei campioni di RNA con e senza riscaldamento è lo stesso in questa configurazione sperimentale).
- Assemblare un 6% TBE-urea-poliacrilammide gel in gel-running apparecchio, riempire l'interno e le Camere di fuori con 1x Novex TBE-Urea tampone di corsa. Sciacquare i pozzetti del gel prefabbricati utilizzando il buffer da una pipetta da 1 ml.
- Centrifugare le provette RNA e caricare i campioni (10 in totale microlitri) con gel-loading punte. Attivare il gel a 180 V per 50 min.
- Dopo 50 minuti, smontare il gel rompendo a parte lo stampo di plastica, rimuovere ed eliminare solo l'appoggio più corto dello stampo gel, ma lasciare l'appoggio più come un supporto per il gel.
- Pulire la superficie dell'area di lavoro con Decon (o altro decontaminante radioattività appropriato) assicurandosi che tutti i punti contaminanti radioattivi vengono rimossi.
- Stendere due strati di pellicola trasparente di plastica, avvolgere il gel e il supporto in plastica a due moltiplicato involucro di plastica.
- Esporre il gel avvolto in un foglio di plastica di unutoradiography pellicola di raggi X all'interno di una cassetta di esposizione nella camera oscura, sotto la luce di sicurezza. Lasciare la cassetta a -20 ° C per 60-90 minuti.
- Sviluppare il film in camera oscura a luce sicuro dopo 60-90 minuti con uno sviluppatore automatico film (Figura 2).
Parte 7: incubazione delle proteine RNA e vincolante filtro
In primo luogo, l'RNA e proteine sono incubati in soluzione e poi le sequenze di RNA che si legano alla proteina sono separate dal non-raccoglitori. Con il progredire SELEX cicli, vincolante filtro darà un'indicazione di arricchimento proteico RNA vincolanti.
- Incubare l'RNA a 90 ° C per 10 min per denaturazione e poi a temperatura ambiente per 10 minuti per rinaturazione lento.
- Sciogliere la proteina da 2,12 passo a 8 microlitri 60 mm NaOH e aggiungere 36 microlitri di nucleasi senza acqua deionizzata. Sonicare l'impasto per 1 minuto e aggiungere 36 microlitri di buffer 2x legame RNA (20 mM Tris, 300 mM NaCl, 10 mM MgCl 2, pH 7,5).
- Mescolare la giusta quantità di RNA con 20 l di buffer 10x RNA vincolante e portare al volume di 200 l con l'aggiunta di acqua priva di nucleasi. L'etichetta del "controllo negativo." Tubo
- Mix 20 microlitri di proteine e la quantità desiderata di RNA con 20 microlitri di buffer 10x RNA vincolante e portare al volume di 200 l con acqua priva di nucleasi. Etichetta la "reazione" del tubo.
- Miscelare e incubare le provette per 30 minuti a temperatura ambiente. Nel frattempo preparare i filtri e la configurazione del filtro di legame per il prossimo passo.
- Collegare un 125 ml braccio laterale pallone in una bocca d'aspirazione. Posizionare un pre-lavaggio, supporto poroso di vetro per il filtro sul braccio laterale pallone.
- Equilibrare 3 membrane filtranti in 2-3 ml di 1x tampone RNA vincolante in un 35x10 mm piastra di Petri per 10-15 min. Il primo filtro verrà utilizzato per regolare l'aspirazione a vuoto, il secondo sarà utilizzato per l'RNA-alone, controllo negativo vincolante filtro, e il terzo sarà utilizzato per il legame del filtro di RNA-proteina miscela.
- Dopo 30 minuti, centrifugare la reazione di legame e le provette di controllo alla massima velocità per 5 minuti a temperatura ambiente.
- Accendere il vuoto e il luogo della membrana prima sul vetro poroso. Usando una micropipetta, goccia a goccia 0,5 ml di tampone RNA vincolante sulla membrana e regolare il vuoto per permettere il flusso lento di ogni goccia di buffer attraverso la membrana.
- Posizionare la seconda membrana sul vetro poroso e utilizzando lo stesso tasso di flusso, applicare il controllo negativo sulla membrana.
- Applicare 4x0.5 ml aliquote di 1x tampone RNA vincolante per lavare la membrana e scartare il flow-through. Si noti che se pre-clearing è desiderato, il flow-through viene mantenuto per l'estrazione di RNA. Pre-clearing rimuove sequenze di RNA che si legano al filtro.
- Rimuovere il controllo negativo membrana e posto in una altrettanto marcata con 1,6 ml di tubo per il conteggio di scintillazione.
- Sostituire il vetro poroso con un pre-lavaggio secondo supporto di vetro poroso.
- Mettere il terzo disco sul vetro poroso e applicare la miscela di reazione. Lavare il filtro come al punto 7.11 e scartare il flow-through. Il numero di lavaggi può essere aumentato nei cicli più tardi SELEX per aumentare la severità delle condizioni di SELEX.
- Rimuovere il disco di filtro e posto in un tubo da 1,6 ml con l'etichetta "miscela di reazione" e conservare per il conteggio di scintillazione.
- Eseguire il conteggio a scintillazione come al punto 5.8 e annotare i conteggi per i filtri dei rispettivi.
- Calcolare il livello del filtro legato radioattività rispetto alla quantità totale di radioattività applicata alle membrane (vincolante%). Questo darà un'indicazione di arricchimento legame con il progredire della SELEX.
Parte 8: estrazione di RNA dai filtri
RNA viene estratto dai filtri per ottenere le sequenze che si legano alla proteina. Queste sequenze sono amplificati per il ciclo SELEX successivo.
- Dopo la conta delle scintillazioni, rimuovere l'associazione-reazione filtro dal tubo (passo da 7,15) e posto in un ambiente pulito, asciutto, 35x10 mm piastra di Petri.
- Utilizzare un panno pulito bisturi e un paio di pinzette per tagliare la membrana in piccoli pezzi.
- Con le pinze, sostituire i pezzi tagliati della membrana nella stessa provetta etichettata dal punto 8.1.
- Aggiungere 400 microlitri di buffer di eluizione (7 M urea, 3 mM EDTA, 100 mM citrato di sodio, pH 5,0) e incubare il tubo a 95 ° C per 10 min.
- Centrifugare la provetta alla massima velocità a temperatura ambiente, aspirare, e raccogliere il burro in una provetta di estrazione nuova etichetta.
- Misurare i conteggi radioattività residua nel tubo contenente i pezzi membrana da scintillazione contando per valutare l'efficienza di estrazione.
- Ripetere il processo di estrazione (passi 8,4-8,6) tre volte. L'efficienza dopo 3 estrazioni di solito è ~ 95-96%.
- Nei tubi contenenti gli estratti di RNA, aggiungere 1 volume (400 mL) di citrato-saturo (pH 4,7) fenolo: cloroformio: alcool isoamilico (125:24:1). Mescolare con un vortice di ~ 1 min e centrifugage a 16.000 g per 2 min.
- Trasferimento in alto, fase acquosa in una nuova provetta o eliminare la fase inferiore di aspirazione mediante una micropipetta.
- Aggiungere 1 volume di cloroformio: alcool isoamilico (24:1), mescolare con un vortex per 1 min e centrifugare a 16000 g per 2 min.
- Trasferimento in alto, fase acquosa in una nuova provetta o eliminare la fase inferiore di aspirazione mediante una micropipetta.
- Per precipitare l'RNA, aggiungere 0,1 volume di 3 acetato di sodio M, pH 5,2, 3-4 glicogeno microlitri (10 mg / mL) come RNA coprecipitant, e 1 equivalente volume di 2-propanolo. Mescolare e mettere in un congelatore di -20 ° C durante la notte.
- Girare alla massima velocità, preferibilmente in una microcentrifuga a 4 ° C, per 20-30 minuti per precipitare l'RNA dal punto 8.12.
- Dopo la centrifugazione, l'RNA si separa in una fase liquida, che è appena visibile. Aspirare il supernate attenzione a non staccare questa fase appena visibile precipitato sul fondo della provetta.
- Lavare l'RNA "pellet", con 0,5 ml di etanolo al 70%, centrifugare per 5 minuti a velocità massima a 4 ° C ed eliminare l'etanolo mediante aspirazione a non staccare la fase appena visibile precipitante.
- Sciogliere il pellet di RNA in 50 microlitri di buffer STE e procedere al G-50 di purificazione come al punto 5.
Parte 9: trascrizione inversa e PCR per continuare i cicli di SELEX
Per procedere, per il prossimo ciclo di SELEX, l'RNA deve essere retrotrascritto al DNA e amplificato mediante PCR.
- In 5 etichettato 0,6 ml tubi, mescolare 3 ml della purificato, G-50-dissalate RNA con 2 ml di 8-volte-Primer diluito inverso. 'Controllo negativo.' Etichetta di un tubo
- Incubare la miscela a 70 ° C per 5 minuti, e poi in ghiaccio per un altro min 5 per consentire ibridazione del primer a RNA.
- A questa miscela sul ghiaccio, aggiungere 6,4 microlitri di nucleasi senza acqua, 4 microlitri ImProm-II tampone di reazione 5x, 1,6 microlitri 25 mM MgCl 2, 1 ml 10 mM dNTP mix, 1 ml inibitore della ribonucleasi RNasein, 1 ml ImProm-II della trascrittasi inversa per un totale di 15 microlitri.
- Per il controllo negativo, aggiungere 7,4 microlitri acqua priva di nucleasi e lasciare fuori la trascrittasi inversa. Etichetta i tubi di conseguenza. Il controllo negativo è incluso per verificare che la contaminazione del DNA da un ciclo precedente non è amplificato per il ciclo SELEX successivo. Questa fase test l'efficacia della digestione di DNA da RQ1 RNasi-free DNasi al punto 4.3.
- Utilizzare un termociclatore per incubare la miscela di reazione a 25 ° C per 5 min, 42 ° C per 1 ora per la ricottura e l'estensione del primo filamento di DNA, rispettivamente, seguiti da 70 ° C per 15 minuti per inattivare la trascrittasi inversa.
- Dopo la reazione di trascrizione inversa, impostare il mix PCR. Aggiungere 30 microlitri acqua priva di nucleasi, 10 microlitri di buffer 10x Taq, 30 microlitri 25 mM MgCl 2, 7 microlitri 10 mM dNTP mix, 1 ml di ogni primer, e 1 microlitri Taq polimerasi in tutti i tubi.
- Eseguire il programma PCR come nella parte 3 per 9-14 cicli.
- Purificare il prodotto del DNA come al punto 3.3.
- Mix 8 ml di prodotto DNA con 2 colorante microlitri carico del DNA e elettroforesi su agarosio al 2% a 100 V per 15-20 minuti (Figura 3).
- DNA prodotto è pronto per essere trascritto a RNA etichettati come nella parte 4 e utilizzato per il prossimo ciclo di SELEX.
Parte 10: Risultati Rappresentante
Negli esperimenti SELEX, la natura del Aβ40 oligomeri usati come destinazione, la qualità di RNA utilizzati per ogni ciclo, e di successo estrazione di RNA e dopo ogni ciclo di amplificazione sono importanti. Abbiamo usato per generare un PICUP oligomerica Aβ40 miscela per SELEX dopo la purificazione e la rimozione del cross-linking reagenti. Gli esperimenti dissalazione di cui alla parte 2 di solito portare alla perdita di proteine 50-55%. La quantità di proteine e la qualità può essere valutata mediante misure di assorbanza (λ = 280) e SDS-PAGE (Figura 1). Il profilo medio di assorbanza Aβ40 eluati da 5 esperimenti individuali sovrapponendo una tipica SDS-PAGE profilo di Aβ40 eluita in uno di questi esperimenti sono mostrati in Figura 1. I dati mostrano che la proteina eluisce costantemente al largo della colonna in frazioni 3-5 e reagenti reticolazione eluire frazione dopo 6 (assorbanza maggiore nella frazione 7, Figura 1). SDS-PAGE mostra il tipico Aβ40 distribuzione oligomeri 22. Questa distribuzione è riproducibile dopo le frazioni proteiche sono state liofilizzate (2.11), trattati con HFIP (2.11), resolubilized (7,1), e ri-analizzati mediante SDS-PAGE.
L'integrità di RNA per ogni ciclo di SELEX è importante anche per la progressione iterativo di SELEX, specialmente quando nucleasi suscettibile ribo-oligonucleotidi sono utilizzati. Dopo l'amplificazione di RNA e di etichettatura (parte 4), la qualità del RNA marcata può essere valutata da TBE-urea-poliacrilammide gel elettroforesi. Un profilo tipico di un prodotto etichettato RNA intatto prima e dopo il G-50 di purificazione (PArt. 5) è illustrato nella figura 2.
Dopo ogni ciclo di SELEX, l'RNA viene estratto dalla membrana filtrante dopo vincolante (parte 8) e retrotrascritto (parte 9) per l'amplificazione del DNA per PCR (Step 9.5). Il modello di DNA da un ciclo precedente viene quindi utilizzato per generare 32 P-marcato RNA (parte 4) per il ciclo successivo. Se qualche modello di DNA contaminante da un ciclo precedente persiste nel prodotto etichettato RNA dopo in vitro le reazioni di trascrizione (parte 4), l'efficienza dei cicli di SELEX sarà ridotto, chiedendo più cicli. Per controllare per questo, dopo ogni ciclo di SELEX e la corrispondente trascrizione inversa reazione di PCR, elettroforesi su agarosio viene eseguita (9.12). Assenza di prodotto DNA in senso negativo di controllo provette (9,4) indica la rimozione di successo del modello di DNA proveniente da un ciclo di SELEX precedente (Figura 3). Se amplificazione del DNA mediante PCR è osservata nel controllo negativo tubo, è consigliabile che la durata della incubazione con RQ1 DNasi (4,3) essere prolungata. Il produttore raccomanda durata incubazione con RQ1 DNasi è di 15 minuti, però, abbiamo scoperto che l'incubazione più lungo (4-5 h) è stato richiesto di rimuovere il DNA stampo completamente (Figura 3).
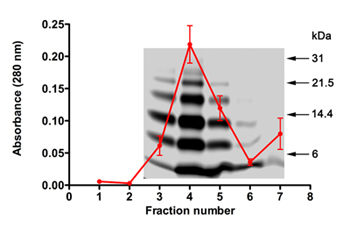
Figura 1. SDS-PAGE e il profilo di assorbimento PICUP generati, dissalato Aβ40 oligomeri. Il profilo di assorbimento da 5 colonne singole dissalazione era media e ricoperte su un gel di rappresentanza. Marcatori peso molecolare vengono visualizzati sulla destra.

Figura 2. TBE-urea-poliacrilammide gel elettroforesi di RNA prodotto prima e dopo il G-50 di purificazione. La direzione di migrazione del prodotto RNA è dal catodo all'anodo, come indicato.
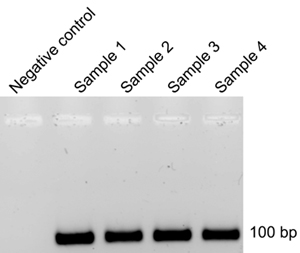
Figura 3. Agarosio elettroforesi su gel di prodotto del DNA dopo trascrizione inversa e PCR.
Discussione
Il punto di partenza del processo di SELEX è sintesi di una libreria oligonucleotide casuale tipicamente contiene 10 12 -10 15 sequenze. Nel DNA SELEX, questa libreria è utilizzata direttamente dopo un pool di ssDNA viene generato, mentre in RNA SELEX, dimostrato qui, la biblioteca ssDNA viene convertito prima a un pool di RNA enzimaticamente da trascrizione in vitro. Poi, SELEX viene eseguito iterativamente per cui ogni ciclo comprende l'esposizione e vincolante di oligonucleotidi p...
Divulgazioni
Riconoscimenti
Questo lavoro è stato sostenuto dalle concessioni AG030709 dal NIH / NIA e 07-65798 del California Department of Public Health. Riconosciamo Margaret M. Condron per la sintesi di peptidi e analisi degli aminoacidi, la dottoressa Elizabeth F. Neufeld per aiutare e sostenere i passi iniziali del progetto, il Dr. B. Chi-Hong Chen di sostegno e di reagenti e Dr. Andrew D . Ellington per le discussioni utili.
Materiali
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| Aβ40 | UCLA Biopolymers Laboratory | Lyophilized powder | ||
| MX5 Automated-S Microbalance | Mettler Toledo | |||
| Silicon-coated, 1.6-ml tubes | Denville Scientific | C19033 or C19035 | ||
| 1,1,1,3,3,3-hexafluoro-2-propanol (HFIP) | TCI America | H0424 | Use in a fume hood. | |
| Ammonium persulfate | Sigma | A-7460 | Vortex until the solution is clear. APS is prepared freshly each time and should be used within 48 h. | |
| Tris(2,2-bipridyl)dichlororuthenium(II) hexahydrate | Sigma | 224758-1G | Vortex until the solution is clear. Cover the RuBpy tube with foil to protect the reagent from ambient light. RuBpy is prepared freshly each time and should be used within 48 h. | |
| Dithiothreitol (DTT) | Sigma | 43815 | ||
| D-Salt™ Excellulose™ desalting columns | Thermo Scientific | 20449 | ||
| Ammonium acetate | Fisher Scientific | A637-500 | ||
| Silicon-coated, 0.6-ml tubes | Denville Scientific | C19063 | ||
| Novex Tricine Gels (10–20%) | Invitrogen | EC6625B0X | 10-well; mini size (8 cm X 8 cm); 25 μl loading volume per well; separation range 5 kDa to 40 kDa | |
| Quartz cuvette | Hellma | 105.250-QS | ||
| Beckman DU 640 spectrophotometer | Beckman | |||
| ssDNA library | Integrated DNA Technologies | Custom-ordered | The library was designed to contain 49 random nucleotides flanked by two constant regions containing primer-binding and cloning sites: 5'-TAA TAC GAC TCA CTA TAG GGA ATT CCG CGT GTG C (N:25:25:25:25%) (N)49 G TCC GTT CGG GAT CCT C-3' | |
| Taq DNA polymerase | USB Corporation | 71160 | Recombinant Thermus aquaticus DNA Polymerase supplied with 10× PCR Buffer and a separate tube of 25 mM MgCl2 for routine PCR. | |
| PCR Nucleotide Mix, 10 mM solution | USB Corporation | 77212 | (10 mM each dATP, dCTP, dGTP, dTTP) | |
| Forward primer | Integrated DNA Technologies | Custom-ordered | 5'-TAA TAC GAC TCA CTA TAG GGA ATT CCG CGT GTG C-3' | |
| Reverse primer | Integrated DNA Technologies | Custom-ordered | 5'-GAG GAT CCC GAA CGG AC-3' | |
| Thermal cycler | Denville Scientific | Techne TC-312 | ||
| QIAquick PCR Purification Kit (50) | QIAGEN | 28104 | ||
| Agarose | Denville Scientific | CA3510-8 | ||
| Conical, sterile 1.6-ml tubes with caps attached with O-rings | Denville Scientific | C19040-S | ||
| RiboMAX™ Large Scale RNA Production System–T7 | Promega | P1300 | The kit contains: 120 μl Enzyme Mix (RNA polymerase, recombinant RNasin® ribonuclease inhibitor and recombinant inorganic pyrophosphatase); 240 μl transcription 5 buffer; 100 μl each of 4 rNTPs, 100 mM; 110 U RQ1 RNase-free DNase, 1 U/μl; 10 μl linear control DNA, 1 mg/ml; 1 ml 3M sodium acetate (pH 5.2); 1.25 ml nuclease-Free water | |
| α-32P-cytidine 5'-triphosphate, 250 μCi (9.25 MBq), | Perkin Elmer | BLU008H250UC | Specific Activity: 3000 Ci (111 TBq)/mmol, 50 mM Tricine (pH 7.6) | |
| Citrate-saturated phenol:chloroform:isoamyl alcohol (125:24:1, pH 4.7) | Sigma (Fluka) | 77619 | ||
| Chloroform:Isoamyl alcohol (24:1) | Sigma | C0549 | ||
| Absolute ethanol for molecular biology | Sigma | E7023 | ||
| Z216-MK refrigerated microcentrifuge | Denville Scientific | C0216-MK | ||
| illustra ProbeQuant™ G-50 Micro Columns | GE Healthcare | Obtained from Fisher Scientific (45-001-487) | Prepacked with Sephadex™ G-50 DNA Grade and pre-equilibrated in STE buffer containing 0.15% Kathon as Biocide | |
| Triathler Bench-top Scintillation counter | Hidex Oy, Turku, Finland | Triathler LSC Model: 425-034 | ||
| Novex® TBE-Urea Sample Buffer (2×) | Invitrogen | LC6876 | ||
| 6% TBE-Urea Gels 1.0 mm, 10 wells | Invitrogen | EC6865BOX | ||
| Novex® TBE Running Buffer (5×) | Invitrogen | LC6675 | ||
| Radioactivity decontaminant | Fisher Scientific | 04-355-67 | ||
| Gel-loading tips | Denville Scientific | P3080 | ||
| XCell SureLock Mini-Cell | Invitrogen | EI0001 | XCell SureLock Mini-Cell | |
| Autoradiography film | Denville Scientific | E3018 | Use in complete darkness | |
| Autoradiography film, Hyperfilm™ ECL | Amersham Biosciences | RPN3114K | Can be used under red safe light. | |
| Membrane discs | Millipore | GSWP02500 | Mixed cellulose ester, hydrophilic, 0.22-μm disc membranes | |
| Fritted glass support base for 125-ml flask | VWR | 26316-696 | ||
| Petri dishes | Fisher Scientific | 08-757-11YZ | ||
| Urea | Fisher Scientific | AC32738-0050 | ||
| EDTA | Fisher Scientific | 118430010 | ||
| Glycogen | Sigma | G1767 | ||
| 2-Propanol for molecular biology | Sigma | I9516 | ||
| Recombinant RNase inhibitor | USB Corporation | 71571 | ||
| ImProm-II™Reverse Transcription System | Promega | A3802 | ||
| Recombinant RNase inhibitor | USB Corporation | 71571 | ||
| RapidRun™ Loading Dye | USB Corporation | 77524 |
Riferimenti
- Monien, B. H., Apostolova, L. G., Bitan, G. Early diagnostics and therapeutics for Alzheimer's disease-how early can we get there. Expert. Rev. Neurother. 6, 1293-1306 (2006).
- Nestor, P. J., Scheltens, P., Hodges, J. R. Advances in the early detection of Alzheimer's disease. Nat. Med. 10, S34-S41 (2004).
- Kawas, C. H. Clinical practice. Early Alzheimer's disease. N. Engl. J. Med. 349, 1056-1063 (2003).
- Haass, C., Selkoe, D. J. Soluble protein oligomers in neurodegeneration: lessons from the Alzheimer's amyloid β-peptide. Nat. Rev. Mol. Cell. Biol. 8, 101-112 (2007).
- Kirkitadze, M. D., Bitan, G., Teplow, D. B. Paradigm shifts in Alzheimer's disease and other neurodegenerative disorders: the emerging role of oligomeric assemblies. J. Neurosci. Res. 69, 567-577 (2002).
- Rahimi, F., Shanmugam, A., Bitan, G. Structure-function relationships of pre-fibrillar protein assemblies in Alzheimer's disease and related disorders. Curr. Alzheimer Res. 5, 319-341 (2008).
- Jayasena, S. D. Aptamers: an emerging class of molecules that rival antibodies in diagnostics. Clin. Chem. 45, 1628-1650 (1999).
- Bunka, D. H., Stockley, P. G. Aptamers come of age - at last. Nat. Rev. Microbiol. 4, 588-596 (2006).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Lee, J. F., Stovall, G. M., Ellington, A. D. Aptamer therapeutics advance. Curr. Opin. Chem. Biol. 10, 282-289 (2006).
- Weiss, S. RNA aptamers specifically interact with the prion protein PrP. J. Virol. 71, 8790-8797 (1997).
- Bibby, D. F. Application of a novel in vitro selection technique to isolate and characterise high affinity DNA aptamers binding mammalian prion proteins. J. Virol. Methods. 151, 107-115 (2008).
- Rhie, A. Characterization of 2'-fluoro-RNA aptamers that bind preferentially to disease-associated conformations of prion protein and inhibit conversion. J. Biol. Chem. 278, 39697-39705 (2003).
- King, D. J., Safar, J. G., Legname, G., Prusiner, S. B. Thioaptamer interactions with prion proteins: sequence-specific and non-specific binding sites. J. Mol. Biol. 369, 1001-1014 (2007).
- Proske, D. Prion-protein-specific aptamer reduces PrPSc formation. ChemBioChem. 3, 717-725 (2002).
- Murakami, K., Nishikawa, F., Noda, K., Yokoyama, T., Nishikawa, S. Anti-bovine prion protein RNA aptamer containing tandem GGA repeat interacts both with recombinant bovine prion protein and its β isoform with high affinity. Prion. 2, 73-80 (2008).
- Luhrs, T., Zahn, R., Wuthrich, K. Amyloid formation by recombinant full-length prion proteins in phospholipid bicelle solutions. J. Mol. Biol. 357, 833-841 (2006).
- Bunka, D. H. Production and characterization of RNA aptamers specific for amyloid fibril epitopes. J. Biol. Chem. 282, 34500-34509 (2007).
- Ylera, F., Lurz, R., Erdmann, V. A., Furste, J. P. Selection of RNA aptamers to the Alzheimer's disease amyloid peptide. Biochem. Biophys. Res. Commun. 290, 1583-1588 (2002).
- Rahimi, F., Murakami, K., Summers, J. L., Chen, C. H., Bitan, G. RNA aptamers generated against oligomeric Aβ40 recognize common amyloid aptatopes with low specificity but high sensitivity. PLoS ONE. 4, e7694-e7694 (2009).
- Bitan, G., Lomakin, A., Teplow, D. B. Amyloid β-protein oligomerization: prenucleation interactions revealed by photo-induced cross-linking of unmodified proteins. J. Biol. Chem. 276, 35176-35184 (2001).
- Rahimi, F., Maiti, P., Bitan, G. Photo-induced cross-linking of unmodified proteins (PICUP) applied to amyloidogenic peptides. J. Vis. Exp. , (2009).
- Bitan, G., Fradinger, E. A., Spring, S. M., Teplow, D. B. Neurotoxic protein oligomers-what you see is not always what you get. Amyloid. 12, 88-95 (2005).
- Bitan, G. Structural study of metastable amyloidogenic protein oligomers by photo-induced cross-linking of unmodified proteins. Methods Enzymol. 413, 217-236 (2006).
- Chen, C. H., Chernis, G. A., Hoang, V. Q., Landgraf, R. Inhibition of heregulin signaling by an aptamer that preferentially binds to the oligomeric form of human epidermal growth factor receptor-3. Proc. Natl. Acad. Sci. USA. 100, 9226-9231 (2003).
- Adams, D. S. . Lab math: a handbook of measurements, calculations, and other quantitative skills for use at the bench. , (2003).
- Gopinath, S. C. Methods developed for SELEX. Anal. Bioanal. Chem. 387, 171-182 (2007).
- Takahashi, T., Tada, K., Mihara, H. RNA aptamers selected against amyloid β-peptide (Aβ) inhibit the aggregation of Aβ. Mol. Biosyst. 5, 986-991 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon