Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Выбор Аптамеры для β-амилоида белка, возбудитель болезни Альцгеймера
В этой статье
Резюме
Аптамеры короткие ribo-/deoxyribo-oligonucleotides выбран В пробирке Эволюции методов, основанных на сродством к конкретной цели. Аптамеры молекулярные инструменты признания с универсальным терапевтических, диагностических и исследовательских целях. Мы демонстрируем методы отбора аптамеров для амилоидных β-белок, возбудитель болезни Альцгеймера.
Аннотация
Alzheimer's disease (AD) is a progressive, age-dependent, neurodegenerative disorder with an insidious course that renders its presymptomatic diagnosis difficult1. Definite AD diagnosis is achieved only postmortem, thus establishing presymptomatic, early diagnosis of AD is crucial for developing and administering effective therapies2,3.
Amyloid β-protein (Aβ) is central to AD pathogenesis. Soluble, oligomeric Aβ assemblies are believed to affect neurotoxicity underlying synaptic dysfunction and neuron loss in AD4,5. Various forms of soluble Aβ assemblies have been described, however, their interrelationships and relevance to AD etiology and pathogenesis are complex and not well understood6. Specific molecular recognition tools may unravel the relationships amongst Aβ assemblies and facilitate detection and characterization of these assemblies early in the disease course before symptoms emerge. Molecular recognition commonly relies on antibodies. However, an alternative class of molecular recognition tools, aptamers, offers important advantages relative to antibodies7,8. Aptamers are oligonucleotides generated by in-vitro selection: systematic evolution of ligands by exponential enrichment (SELEX)9,10. SELEX is an iterative process that, similar to Darwinian evolution, allows selection, amplification, enrichment, and perpetuation of a property, e.g., avid, specific, ligand binding (aptamers) or catalytic activity (ribozymes and DNAzymes).
Despite emergence of aptamers as tools in modern biotechnology and medicine11, they have been underutilized in the amyloid field. Few RNA or ssDNA aptamers have been selected against various forms of prion proteins (PrP)12-16. An RNA aptamer generated against recombinant bovine PrP was shown to recognize bovine PrP-β17, a soluble, oligomeric, β-sheet-rich conformational variant of full-length PrP that forms amyloid fibrils18. Aptamers generated using monomeric and several forms of fibrillar β2-microglobulin (β2m) were found to bind fibrils of certain other amyloidogenic proteins besides β2m fibrils19. Ylera et al. described RNA aptamers selected against immobilized monomeric Aβ4020. Unexpectedly, these aptamers bound fibrillar Aβ40. Altogether, these data raise several important questions. Why did aptamers selected against monomeric proteins recognize their polymeric forms? Could aptamers against monomeric and/or oligomeric forms of amyloidogenic proteins be obtained? To address these questions, we attempted to select aptamers for covalently-stabilized oligomeric Aβ4021 generated using photo-induced cross-linking of unmodified proteins (PICUP)22,23. Similar to previous findings17,19,20, these aptamers reacted with fibrils of Aβ and several other amyloidogenic proteins likely recognizing a potentially common amyloid structural aptatope21. Here, we present the SELEX methodology used in production of these aptamers21.
протокол
Часть 1: подготовка Белок и сшивания
Первоначально, белка, используемого для SELEX предварительно обрабатывается с 1,1,1,3,3,3-гексафтор-2-пропанол (HFIP), чтобы получить однородную, совокупный без подготовки, как описано выше 23. Этот шаг необходим, потому что предварительно сформированных агрегатов вызывают быстрое агрегации амилоидогенный белков, что приводит к плохой воспроизводимости экспериментальных 24, и нежелательны для выбора аптамеры для unaggregated, не фибриллярные формы белка.
- Взвесьте из ~ 800 мкг (~ 180 нмоль) чистой Aβ40 использованием микровесов. Передача сухого лиофилизированного пептида в подписанные, кремния, покрытые оболочкой, низким адсорбента 1,6-мл пробирки и отцентрифугировать.
- Растворите пептида в 100% 1,1,1,3,3,3-гексафтор-2-пропанол (HFIP, 400 мкл), чтобы получить 0,5 мМ пептида раствора, как описано ранее 23.
- Разделите это решение в четыре 100-мкл аликвоты так, что каждая трубка содержит ~ 45 нмоль Aβ40 номинально. Приступить к удалению HFIP, как описано выше 23.
- Перед солюбилизирующие HFIP обработанных пептидов в буфер для сшивания реакций, подготовить сшивания и тушения реагентов. Отвешивать персульфата аммония (APS, МВт 228,2 г / моль) и приготовить раствор из 40 мМ в 10 мМ фосфат натрия, рН 7,4. Смешайте использованием вихревой пока раствор не станет прозрачным.
- Подготовка 2 мМ раствора Трис (2,2-бипиридил) dichlororuthenium (II) гексагидрат (RuBpy, МВт 748,63 г / моль) в 10 мМ фосфат натрия, рН 7,4. Смешайте использованием вихря и проверить полного растворения. Защита труб от света с помощью алюминиевой фольги.
- Подготовка тушение реагента. Отвешивать дитиотреитол (DTT, МВт 154,5 г / моль) и растворить в деионизованной воде или в 10-мМ фосфата натрия, рН 7,4, до 1 м.
- Растворите HFIP обработанных пептидных именно так, как описано выше 23,25, но целью получения ~ 60 мкМ пептида решение.
- Выполните PICUP генерировать смеси олигомерных Aβ40, как описано выше 23. Типичная реакция PICUP выполняется в объеме 20 мкл, где конечные концентрации белка, RuBpy и APS являются 30 мкм, 0,05 мм, и 1 мМ, соответственно, 23. Здесь смесь, используемая для PICUP содержит 108 мкл белка, 6 мкл RuBpy, 6 мкл APS, и 1 мкл DTT и концентрацию белка, RuBpy и APS все удвоилось по сравнению с типичной реакцией. Это снижает число реакций PICUP должны быть выполнены и повышает концентрацию белка для обессоливания.
Часть 2: Обессоливание белковый препарат
Перед использованием белка для SELEX, обессоливания осуществляется для удаления сшивающих реагентов, используемых для PICUP. Эта смесь содержит PICUP реакции сшивания реагентов и ~ 55 мкМ белка (номинальная концентрация).
- Снимите верхнюю крышку с 5-мл обессоливания колонки, поддержка колонки стоят, место стакан ниже выходе из колонки, и удалить розетки вилку. Пусть поток хранения буфера через и собирают в стакан.
- Равновесия колонки в 10 мМ ацетата аммония, рН 8,3. Добавьте 5 смолой объемов слоя (25 мл) этого буфера в 3-мл аликвоты в колонну и позволить ей течь через.
- Тем временем, этикетка минимум 8-адсорбент, кремний покрытием 1,6-мл пробирки и поместите их в трубки стойку.
- После колонна находится в равновесии, применять 0,5-0.7-мл аликвоты смеси PICUP реакции на столбец на одного человека обессоливания (столбцы можно мыть, хранятся и уравновешенной для последующего использования). Разрешить белковая смесь, чтобы впитать в колонку смолы и отбросить проточные в стакан.
- Место первой трубки коллекция ниже выходе из колонки. Добавить 0,5 мл ацетатного буфера в колонну и собирать первые 0,5-мл фракция, протекающий через.
- Повторите шаги 2.4 и 2.5, и собрать до восьми 0,5-мл фракций в соответствующих труб.
- Количество другой набор минимум 8-адсорбент, кремния, покрытые оболочкой, небольшой 0,6-мл пробирки и поместите их в стойку. Этикетка другую трубку, как "пустой". Возьмите 150 мкл каждой фракции и передачи в меченых 0,6-мл пробирках. Используйте 20 мкл для SDS-PAGE анализа (рис. 1), как показано ранее 23.
- Передача 150 мкл 10 мМ ацетата аммония, рН 8,3, в пустую трубу. Используйте эту трубку, чтобы установить пробел в спектрофотометр.
- Измерить абсорбцию в 130 мкл каждой фракции при λ = 280 нм в кварцевой кювете.
- После поглощения измеряется, объединить фракции с высоким содержанием белка (как правило, фракции 3-5), осторожно перемешать с помощью пипетки, и держать 10-мкл для аминокислотного анализа, чтобы определить реальную концентрацию белка (не показаны).
- Разделите образца в нескольких аликвоты ~ 2 нмоль белка на трубе и lyophilize образцов в лиофилизатор.
- После завершения лиофилизации, лечить образцов с 100% HFIP по-прежнему.
- Магазин трубы при температуре -20 ° C. Используйте одну трубку для каждого цикла SELEX (см. ниже).
Часть 3: Усиление синтетических случайных оцДНК библиотеки с помощью ПЦР
Синтетической библиотеки оцДНК используется здесь для SELEX включены 49 рандомизированных нуклеотидов (Т: G: C = 25:25:25:25%) окружении постоянной регионах включающий клонирования объектов (Bam HI, Eco RI) и промоутером T7, как описано выше 26.
- Для усиления библиотека, созданы стандартные реакции ПЦР следующим образом: 202 мкл деионизированной воды, 40 мкл 10х Taq ДНК-полимераза буфер, 2 мкл шаблон оцДНК (0,5 нмоль), 120 мкл 25 мМ MgCl 2, 28 мкл 10 мМ дезоксинуклеозид трифосфат смеси (дНТФ), 1 мкл прямого праймера (300 пмоль), 1 мкл праймера обратном (300 пмоль) и 4 мкл Taq-полимеразы.
- Выполните ПЦР-реакции с помощью термоциклер со следующими настройками: 5 мин при 94 ° C для начальной денатурации, 20 циклов каждого из 94 ° С в течение 30 сек, 52 ° С в течение 30 сек для отжига, 72 ° С в течение 30 сек расширение, а окончательное расширение при 72 ° С в течение 7 мин.
- После завершения реакции ПЦР, очищают амплифицированной ДНК продукта с использованием ПЦР Qiagen QiaQuick очистки комплект в соответствии с инструкциями производителя. Как правило, в этих экспериментах концентрация и выход ДНК 160 ± 10 нг / мкл в 50 мкл.
- Проверьте количество ДНК после ПЦР на 2% гель-электрофореза в агарозном.
Часть 4: Поколение 32 Р-меченых РНК в пробирке транскрипции
- Настройка транскрипции реакцию в уплотнительное кольцо крышки, 1,6 мл трубки в соответствии с инструкциями изготовителя с некоторыми изменениями следующим образом: 20 мкл 5x транскрипции Т7 буфера, 7,5 мкл 100 мМ RATP, rGTP, rUTP, 1 мкл 100 мМ rCTP, 2 мкл α 32 P-CTP (3000 Ки / ммоль), 5-10 мкг очищенной ДНК-матрицы (~ 30-40 мкл очищенной ДНК продукта), 10 мкл смеси ферментов, и составляют окончательный объем до 100 мкл , добавив нуклеазы без воды.
- Смешайте раствор осторожно пипеткой, центрифуги смеси и инкубировать при температуре 37 ° С в течение ночи.
- В конце реакции, ДНК-матрицы должна быть удалена. Добавить RQ1 РНКазы без ДНКазы в концентрации 1 ЕД / мкг ДНК шаблон и инкубировать в течение 4 ч при 37 ° С, чтобы переварить ДНК.
- Через 4 часа, извлечение РНК, добавив 1 объем цитрата насыщенных фенол: хлороформ: изоамиловый спирт (125:24:1, рН 4,7). Смешайте по вихрь в течение ~ 1 мин и центрифуге при 16000 г в течение 2 мин.
- Передача верхний, водной фазы в новую пробирку или отказаться от нижней фазе стремлением использовании микропипетки. Добавить 1 объем хлороформ: изоамиловый спирт (24:1), смешать вихрем в течение 1 мин и центрифуги, как описано в п. 4.4.
- Передача верхний, водной фазы в новую пробирку или отказаться от нижней фазе стремлением использовании микропипетки. Остаточная хлороформ может быть удалена, выполняя быстрое вращение (10 секунд) в микроцентрифужных и удаления нижней фазе с микропипетки. На этом этапе, это легче удалить нижнюю фазу, а не supernate.
- Чтобы осадок РНК, добавить 0,1-объем эквивалент 3М ацетата натрия, рН 5,2, и 1-объеме, эквивалентном 2-пропанола. Смешайте и поставьте в -20 ° C морозилку на 15 мин.
- Через 15 минут вращаются на максимальной скорости, предпочтительно в охлажденном микроцентрифужных при 4 ° С, в течение 20-30 мин для осаждения продукта РНК.
- Аспирируйте supernate тщательно, вымыть РНК гранул с 0,5 мл 70% этанола, центрифуги при 4 ° С и отбросить этанола аспирации.
- Передача трубки, содержащей РНК гранулы для тепла блоков и сухих гранул при 37 ° С в течение 5 мин.
- Растворите образца РНК в 150 мМ STE буфере, рН 8,0 (при условии, с иллюстрации ProbeQuant G-50 микроколонок) или нуклеазы без воды до объема идентична в пробирке транскрипции реакции т.е. 100 мкл (шаг 4.1).
- Тепло трубки, содержащей продукт РНК при 70 ° С в течение 10 мин в блоке тепла и микс от вихрей для облегчения РНК роспуска.
- Центрифуга на максимальной скорости в течение 1 мин при комнатной температуре.
- Держите 1-мкл РНК в меченых 0,6-мл трубку для сцинтилляционного счета и КЭ-мочевины полиакриламидном геле (Часть 6).
Часть 5: Удаление неинкорпорированных нуклеотидов РНК обессоливания, и сцинтилляционных счет
Для удаления неинкорпорированных нуклеотидов, использование двух G-50 столбцов в соответствии с инструкциями производителя.
- Обратить колонн и микс от вихря ресуспендирования смолы.
- Оснастку со дна закрытие перфорации колонны на использование пластиковых инструмент, предоставляемый в комплекте и не забудьте оставить выходе нетронутыми. Ослабьте крышку на четверть оборота и место столбцов в чистые трубы коллекции представлены в G-50 Комплект.
- Спиновые столбцов в коллекции труб на 730 г в течение 1 мин, чтобы удалить хранения буфера.
- Передача колонны к двум новым уплотнительным кольцом крышками, 1,6-мл пробирки и нагрузки 50 мкл 32 P-меченой РНК пробы в колонку непосредственно на смолу без возмущающих смолы.
- Спиновые на 730 г в течение 2 мин для сбора очищенной меченой РНК. После этого шага, отказаться от колонны.
- Передача 1 мкл G-50-очищенной РНК 0,6-мл трубку для сцинтилляционного счета и электрофорез (Часть 6). Магазин фондовом РНК при температуре -20 ° С до использования для SELEX. Желательно, чтобы РНК используется в течение 2 дней после маркировки, чтобы избежать ее деградации и потере активности.
- Использование двух 1-мкл РНК аликвот от шагов, 4,14 и 5,6 для измерения импульсов в минуту (стр / мкл) с помощью сцинтилляционного счетчика. Здесь мы используем Triathler настольный сцинтилляционных счетчиков.
- Передача пробирки, содержащие образцы РНК внутри пластиковый адаптер используется для подсчета P 32, при условии, с Triathler. Положите трубку и адаптер в счетной камере, закройте крышку камеры, выбрать 32 P счета, и нажать старт, чтобы начать подсчет.
- Рассчитать% этикетке включения т.е.% включения α-32 P = CTP (CPM / мкл для "G-50" образца ÷ копий в минуту / мкл для "TOTAL" образец) х 100. ИТОГО образца образца до G-50 очистки.
- После подсчета сцинтилляционных и расчет процентов включения, рассчитывать доходность РНК и удельная активность РНК. Например, если рассчитывает на бассейн РНК до (237 370 копий в минуту / мкл) и после того, G-50 очистки (191 330 копий в минуту / мкл) дают 81% включения, то следующие рассчитываются до получения выхода РНК:
Начиная с этапа 4.1, количество α-32 P CTP в 3000 Ки / ммоль и 10 мкКи / мкл: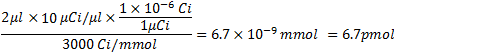 .
.
Количество немеченого CTP используется: .
.
Общая сумма CTP используется 100006.7pmol. Молекулярная масса РНК вычисляется следующим образом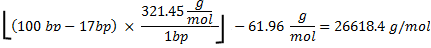
где 321,45 г / моль 27, средняя масса rNTPs, 100 б.п. является число баз в последовательности, и 17 б.п. является число баз в промоутер T7, не транскрибируется. Вычитание 61,96 г / моль от олигонуклеотида молекулярной массы учитывает удаление HPO 2 (63,98) и добавлением двух атомов водорода (2.02) 27. Потому что, 4 rNTPs, CTP является лимитирующим реагентом, если все CTP должны были быть включены теоретические масса РНК производства было бы: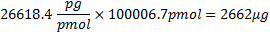 .
.
Однако из-за 81% инкорпорации, эта масса в настоящее время 2156 мкг. Графы на мкг РНК затем будет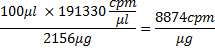
где 100 мкл объема транскрипции реакции, так и в плане родинок: 26618,4 мкг / мкмоль х 8874 копий в минуту / мкг = 2.4x10 8 копий в минуту / мкмоль. Отсюда, количество РНК в нмоль и соответствующий объем используемых для инкубации с белком может быть рассчитана, например, 5 мкл РНК будет содержать ~ 4 нмоль РНК, т. е.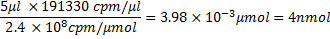
В этих экспериментах, 300 пмоль на 1 белок нмоль и 4 нмоль до 100 нмоль РНК были использованы 21.
Часть 6: Характеристика продукта РНК с помощью электрофореза и авторадиографии
- Для подготовки образцов для электрофореза, использования 1-мкл аликвот РНК до и после G-50 очистки от шагов, 4,14 и 5,6, соответственно. Добавить 4 мкл буфера STE и 5 мкл 2x Novex КЭ-мочевины Пример буфера.
- Тепло образцов при 70 ° C в течение 5 минут, как обычно рекомендуется для электрофореза РНК (отопление оказалось ненужным, поскольку гель разрешением образцов РНК и без отопления в этом же экспериментальной установки).
- Соберите 6% КЭ-карбамидо-полиакриламидном геле в гель-работает аппарат, заполните внутри и снаружи камеры с 1x Novex КЭ-мочевины Запуск буфера. Промыть лунки геля сборных использованием буфера 1-мл пипетки.
- Центрифуга РНК труб и загрузить образцы (10 мкл общего) с помощью гель-погрузка советы. Выполнить геля при 180 В в течение 50 мин.
- Через 50 мин, разобрать геля разваливается пластиковых форм, удалить и уничтожить только короче поддержке гель плесень, но оставить больше поддержки в качестве поддержки для геля.
- Очистите поверхность рабочей зоны с Decon (или другого соответствующего деконтаминант радиоактивности) убедившись, что все загрязнения радиоактивными пятна удаляются.
- Выложить два слоя пластиковых Саран обернуть; обернуть гель и пластиковые подложки в двух курсировали полиэтиленовой пленкой.
- Expose гель, завернутые в пластик, чтобы листutoradiography рентгеновской пленки внутри экспозиции кассету в темной комнате, в безопасных света. Оставьте кассеты при температуре -20 ° С в течение 60-90 мин.
- Разработка пленку в темной комнате в безопасных свет после 60-90 мин с использованием автоматического разработчика пленки (рис. 2).
Часть 7: РНК инкубации белка и фильтр обязательным
Во-первых, РНК и белка инкубируют в растворе, а затем РНК последовательности, которые связываются с белком отделяются от не-связующих. Поскольку прогресс SELEX циклов, фильтр обязательным даст указание белка связывание РНК обогащения.
- Инкубируйте РНК при 90 ° С в течение 10 мин для денатурации и затем при комнатной температуре в течение 10 мин для медленного ренатурации.
- Растворите белка из шаг 2,12 в 8 мкл 60 мМ NaOH и добавляют 36 мкл нуклеазы без деионизированной водой. Разрушать ультразвуком смесь в течение 1 мин и добавить 36 мкл 2x связывание РНК буфера (20 мМ Tris, 300 мМ NaCl, 10 мМ MgCl 2, рН 7,5).
- Смешайте необходимое количество РНК с 20 мкл 10х буфера для связывания РНК и составляют объема до 200 мкл, добавив нуклеазы без воды. Этикетка трубки "негативного контроля".
- Смешать 20 мкл белка и необходимое количество РНК с 20 мкл 10х буфера для связывания РНК и составляют объема до 200 мкл с нуклеазы без воды. Этикетка трубки «реакции».
- Смешать и инкубировать пробирки в течение 30 мин при комнатной температуре. Тем временем подготовить фильтры и фильтры-связывающий установки для следующего шага.
- Прикрепить 125-мл побочных руку к колбе вакуум на входе. Место предварительно очищенное, пористые стекла для поддержки фильтра на стороне руке колбу.
- Равновесия 3 фильтра мембран в 2-3 мл 1x связывание РНК буфера в 35x10 мм чашки Петри в течение 10-15 мин. Первый фильтр будет использоваться для регулировки вакуумного всасывания, вторая будет использоваться для РНК-одиночку, негативного контроля фильтра обязательными, а третий будет использоваться для фильтра связывание РНК-белок смеси.
- Через 30 мин центрифуга обязательной реакции и контроль труб при максимальной скорости в течение 5 мин при комнатной температуре.
- Включите вакуумный и место первой мембраной на пористое стекло. Использование микропипетки, капельно 0,5 мл буфера для связывания РНК на мембрану и регулировать вакуум, чтобы медленный поток каждого буфера падение через мембрану.
- Место вторую мембрану на пористое стекло и, используя тот же расход, применять негативного контроля на мембране.
- Применить 4x0.5-мл аликвоты 1x связывание РНК буфера для промывки мембраны и отбросить проточные. Заметим, что если предварительно очистив желательно, проточные сохраняется для экстракции РНК. Предварительная очистка удаляет последовательности РНК, которые связываются с фильтром.
- Удалить негативного контроля мембраны и поместить в соответственно меченных 1,6-мл трубку для сцинтилляционного счета.
- Замените пористого стекла с предварительно очищенное второй пористый носитель стекла.
- Место третий круг на пористое стекло и применить реакционной смеси. Мыть фильтры, как в пункте 7.11 и отбросить проточные. Число моющие средства могут быть увеличены в более поздних SELEX циклы увеличения жесткости SELEX условиях.
- Снимите фильтр диск и поместить в 1,6-мл трубки с надписью "реакционной смеси" и сохранить для сцинтилляционных счет.
- Выполните сцинтилляционного счета, как в пункте 5.8 и запишите рассчитывает на соответствующие фильтры.
- Рассчитать уровень фильтра связана радиоактивность по сравнению с общим количеством радиоактивности применяется для мембран (% обязательные). Это даст указание обязательным обогащением, как прогрессирует SELEX.
Часть 8: РНК извлечения из фильтров
РНК извлекается из фильтров для получения последовательности, которые связываются с белком. Эти последовательности усиливаются к следующему циклу SELEX.
- После сцинтилляционного счета, удалить привязку реакции фильтр из трубки (с шагом 7,15) и поместить в чистый, сухой, 35x10 мм чашки Петри.
- Используйте чистую скальпеля и пинцета, чтобы сократить мембраны на мелкие кусочки.
- Использование пинцета, замените нарезанные кусочки мембраны в том же помечены трубки с шага 8.1.
- Добавить 400 мкл буфера элюирования (7 М мочевины, 3 мМ ЭДТА, 100 мМ цитрат натрия, рН 5,0) и инкубировать трубке при 95 ° С в течение 10 мин.
- Центрифуга трубки на максимальной скорости при комнатной температуре, аспирация, и собирать добычу масло в новых меченых трубки.
- Измерьте оставшееся рассчитывает радиоактивности в пробирку, содержащую мембраны произведения сцинтилляционных счет оценить эффективность экстракции.
- Повторите процесс экстракции (шаги 8.4-8.6) трижды. Эффективность через 3 экстракции, как правило, ~ 95-96%.
- В пробирки, содержащие экстракты РНК, добавить 1 объем (400 мкл) цитрат-насыщенных (рН 4,7) фенол: хлороформ: изоамиловый спирт (125:24:1). Смешайте по вихрь в течение ~ 1 мин и центрифугированияGE в 16000 г в течение 2 мин.
- Передача верхний, водной фазы в новую пробирку или отказаться от нижней фазе стремлением использовании микропипетки.
- Добавить 1 объем хлороформ: изоамиловый спирт (24:1), смешать вихрем в течение 1 мин и центрифуге при 16000 г в течение 2 мин.
- Передача верхний, водной фазы в новую пробирку или отказаться от нижней фазе стремлением использовании микропипетки.
- Чтобы осадок РНК, добавить 0,1 объема 3 М ацетата натрия, рН 5,2, 3-4 мкл гликогена (10 мкг / мкл), а РНК соосадителя и 1 объеме, эквивалентном 2-пропанола. Смешайте и поставьте в морозильник -20 ° С в течение ночи.
- Спиновые на максимальной скорости, желательно в микроцентрифужных при 4 ° С, в течение 20-30 мин для осаждения РНК из шаг 8.12.
- После центрифугирования, РНК отделяется в жидкой фазе, которая едва видна. Аспирируйте supernate тщательно, не выбивать это едва видимого осадка фазы в нижней части трубы.
- Вымойте РНК "шарик" с 0,5 мл 70% этанола, центрифуги в течение 5 минут на максимальной скорости при 4 ° С и отбросить этанола стремлением без выбивании едва видимого осадка фазу.
- Растворите гранулы РНК в 50 мкл буфера STE и приступить к G-50 очистки, как в шаге 5.
Часть 9: обратной транскрипции и ПЦР для продолжения цикла SELEX
Для перехода к следующему циклу SELEX, РНК должна быть обратной транскрипции с ДНК и усиливаются с помощью ПЦР.
- В 5 помечены 0,6-мл пробирки, смешать 3 мкл очищенной, G-50-обессоленной РНК с 2 мкл 8-кратный разведенного обратного праймера. Этикетка одна трубка "негативного контроля".
- Выдержите смесь при температуре 70 ° С в течение 5 мин, а затем на льду в течение еще 5 минут, чтобы гибридизация праймера для РНК.
- К этой смеси на лед, добавьте 6,4 мкл нуклеазы без воды, 4 мкл ImProm-II 5x буфера реакции, 1,6 мкл 25 мМ MgCl 2, 1 мкл 10 мМ дНТФ смеси, 1 мкл рибонуклеазы RNasein ингибитора, 1 мкл ImProm-II обратной транскриптазы , составляющих в общей сложности 15 мкл.
- Для отрицательного контроля, добавить 7,4 мкл нуклеазы без воды и оставить в обратной транскриптазы. Этикетка трубы соответственно. Отрицательный контроль включен, чтобы убедиться, что загрязняющие ДНК из предыдущего цикла не усиливается на следующий цикл SELEX. Этот шаг тесты эффективности переваривания ДНК шаблон RQ1 РНКазы без ДНКазы в шаге 4.3.
- Используйте термоциклер для инкубации реакционной смеси при 25 ° С в течение 5 мин, 42 ° С в течение 1 ч для отжига и расширение первой цепи ДНК, соответственно, затем 70 ° С в течение 15 мин для инактивации обратной транскриптазы.
- После обратной транскрипции реакции, создать ПЦР смеси. Добавить 30 мкл нуклеазы без воды, 10 мкл 10х буфера Taq, 30 мкл 25 мМ MgCl 2, 7 мкл 10 мМ дНТФ смеси, 1 мкл каждого праймера, и 1 мкл Taq-полимеразы во всех трубах.
- Запустите программу ПЦР в части 3 для 9-14 циклов.
- Purify ДНК продукт, как в пункте 3.3.
- Смешать 8 мкл ДНК-продукта с 2 мкл красителя загрузки ДНК и electrophorese на 2% агарозном при 100 В в течение 15-20 мин (рис. 3).
- ДНК продукт готов к транскрибируется в РНК, как меченый в части 4 и используется для следующего цикла SELEX.
Часть 10: Представитель Результаты
В SELEX экспериментах, характер Aβ40 олигомеров используются в качестве цели, качество РНК используется для каждого цикла, и успешное извлечение РНК и амплификации после каждого цикла имеют важное значение. Мы использовали PICUP генерировать олигомерных Aβ40 смеси для SELEX после очистки и удаления сшивающих реагентов. Обессоливания экспериментах, описанных в Части 2, обычно приводят к 50-55% потере белка. Количество белка и качества можно оценить с помощью поглощения измерений (λ = 280) и SDS-PAGE (рис. 1). Средний профиль поглощения Aβ40 элюатов от 5 отдельных экспериментов наложения типичных SDS-PAGE профилем Aβ40 элюировали в одном из этих опытов показаны на рисунке 1. Данные показывают, что белок последовательно элюируется из колонки во фракциях 3-5 и сшивающих реагентов элюируются после фракция 6 (увеличилась доля поглощения в 7, Рисунок 1). SDS-PAGE показана типичная Aβ40 олигомер распределения 22. Это распределение было воспроизводимых после белковые фракции лиофилизировали (2,11), получавших HFIP (2,11), resolubilized (7,1), и повторно проанализированы SDS-PAGE.
Целостность РНК для каждого цикла SELEX также имеет важное значение для итерационных прогрессирование SELEX, особенно когда нуклеазы восприимчивых к рибо-олигонуклеотиды используются. После усиления РНК и маркировки (часть 4), качество меченой РНК можно оценить с помощью КЭ-карбамидо-электрофореза в полиакриламидном геле. Типичный профиль нетронутыми отметку РНК до и после G-50 очистки (Pстатья 5) показан на рисунке 2.
После каждого цикла SELEX, РНК извлекается из мембранных фильтров после связывания (часть 8) и обратной транскрипции (часть 9) с ДНК для ПЦР-амплификации (шаг 9.5). ДНК-матрицы от предыдущего цикла, затем используется для генерации 32 P-меченой РНК (Часть 4) для следующего цикла. Если некоторые загрязняющие ДНК шаблон из предыдущего цикла сохраняется в соответствующую отметку РНК после в пробирке транскрипции реакций (Часть 4), эффективность циклов SELEX будет снижена, требуя более циклов. Для контроля за этого после каждого цикла SELEX и соответствующие обратной транскрипции ПЦР-реакции, электрофорез в агарозном выполняется (9.12). Отсутствие ДНК продукта в отрицательной контрольной реакции труб (9,4) указывает на успешное удаление шаблона ДНК, происходящих из предыдущего цикла SELEX (рис. 3). Если амплификации ДНК методом ПЦР наблюдается негативного контроля трубы, он сообщил, что продолжительность инкубации с RQ1 ДНКазы (4,3) быть продлен. Производителя рекомендуемая продолжительность инкубации с RQ1 ДНКазы составляет 15 мин, однако, мы обнаружили, что больше инкубации (4-5 ч), необходимой для удаления шаблона ДНК полностью (рис. 3).
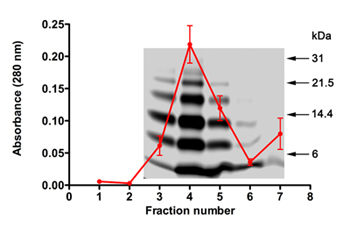
Рисунок 1. SDS-PAGE и поглощение профиля PICUP генерируемые, обессоленной Aβ40 олигомеров. Абсорбцию профиль из 5 индивидуальных обессоливания колонн усредняются и перекрыты на репрезентативной геля. Молекулярные маркеры вес показано на рисунке справа.

Рисунок 2. КЭ-карбамидо-электрофореза в полиакриламидном геле продуктов РНК до и после G-50 очистки. Миграции направление продукт РНК от катода к аноду, как указано.
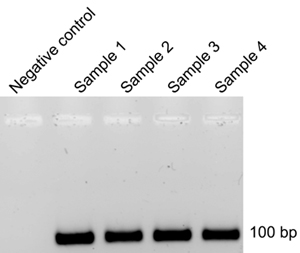
Рисунок 3. Агарозном гель-электрофореза ДНК продукт после обратной транскрипции и ПЦР-амплификации.
Обсуждение
Отправной точкой процесса SELEX является синтез случайных библиотеки олигонуклеотидных обычно содержащих 10 12 -10 15 последовательностей. В ДНК SELEX, эта библиотека используется непосредственно после бассейна оцДНК создается, тогда как в РНК SELEX, продемонстрировали здесь, в библ?...
Раскрытие информации
Благодарности
Эта работа была поддержана грантами AG030709 из NIH / НИА и 07-65798 от Калифорнийского департамента здравоохранения. Мы признаем, Маргарет М. Кондрон для синтеза пептидов и аминокислотного анализа, доктор Элизабет Ф. Нейфельд для оказания помощи и поддержки начальных этапов проекта, доктор Чи-Хун Б. Чена за оказание поддержки и реагентов, а также д-р Эндрю D . Эллингтона за полезные обсуждения.
Материалы
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| Aβ40 | UCLA Biopolymers Laboratory | Lyophilized powder | ||
| MX5 Automated-S Microbalance | Mettler Toledo | |||
| Silicon-coated, 1.6-ml tubes | Denville Scientific | C19033 or C19035 | ||
| 1,1,1,3,3,3-hexafluoro-2-propanol (HFIP) | TCI America | H0424 | Use in a fume hood. | |
| Ammonium persulfate | Sigma | A-7460 | Vortex until the solution is clear. APS is prepared freshly each time and should be used within 48 h. | |
| Tris(2,2-bipridyl)dichlororuthenium(II) hexahydrate | Sigma | 224758-1G | Vortex until the solution is clear. Cover the RuBpy tube with foil to protect the reagent from ambient light. RuBpy is prepared freshly each time and should be used within 48 h. | |
| Dithiothreitol (DTT) | Sigma | 43815 | ||
| D-Salt™ Excellulose™ desalting columns | Thermo Scientific | 20449 | ||
| Ammonium acetate | Fisher Scientific | A637-500 | ||
| Silicon-coated, 0.6-ml tubes | Denville Scientific | C19063 | ||
| Novex Tricine Gels (10–20%) | Invitrogen | EC6625B0X | 10-well; mini size (8 cm X 8 cm); 25 μl loading volume per well; separation range 5 kDa to 40 kDa | |
| Quartz cuvette | Hellma | 105.250-QS | ||
| Beckman DU 640 spectrophotometer | Beckman | |||
| ssDNA library | Integrated DNA Technologies | Custom-ordered | The library was designed to contain 49 random nucleotides flanked by two constant regions containing primer-binding and cloning sites: 5'-TAA TAC GAC TCA CTA TAG GGA ATT CCG CGT GTG C (N:25:25:25:25%) (N)49 G TCC GTT CGG GAT CCT C-3' | |
| Taq DNA polymerase | USB Corporation | 71160 | Recombinant Thermus aquaticus DNA Polymerase supplied with 10× PCR Buffer and a separate tube of 25 mM MgCl2 for routine PCR. | |
| PCR Nucleotide Mix, 10 mM solution | USB Corporation | 77212 | (10 mM each dATP, dCTP, dGTP, dTTP) | |
| Forward primer | Integrated DNA Technologies | Custom-ordered | 5'-TAA TAC GAC TCA CTA TAG GGA ATT CCG CGT GTG C-3' | |
| Reverse primer | Integrated DNA Technologies | Custom-ordered | 5'-GAG GAT CCC GAA CGG AC-3' | |
| Thermal cycler | Denville Scientific | Techne TC-312 | ||
| QIAquick PCR Purification Kit (50) | QIAGEN | 28104 | ||
| Agarose | Denville Scientific | CA3510-8 | ||
| Conical, sterile 1.6-ml tubes with caps attached with O-rings | Denville Scientific | C19040-S | ||
| RiboMAX™ Large Scale RNA Production System–T7 | Promega | P1300 | The kit contains: 120 μl Enzyme Mix (RNA polymerase, recombinant RNasin® ribonuclease inhibitor and recombinant inorganic pyrophosphatase); 240 μl transcription 5 buffer; 100 μl each of 4 rNTPs, 100 mM; 110 U RQ1 RNase-free DNase, 1 U/μl; 10 μl linear control DNA, 1 mg/ml; 1 ml 3M sodium acetate (pH 5.2); 1.25 ml nuclease-Free water | |
| α-32P-cytidine 5'-triphosphate, 250 μCi (9.25 MBq), | Perkin Elmer | BLU008H250UC | Specific Activity: 3000 Ci (111 TBq)/mmol, 50 mM Tricine (pH 7.6) | |
| Citrate-saturated phenol:chloroform:isoamyl alcohol (125:24:1, pH 4.7) | Sigma (Fluka) | 77619 | ||
| Chloroform:Isoamyl alcohol (24:1) | Sigma | C0549 | ||
| Absolute ethanol for molecular biology | Sigma | E7023 | ||
| Z216-MK refrigerated microcentrifuge | Denville Scientific | C0216-MK | ||
| illustra ProbeQuant™ G-50 Micro Columns | GE Healthcare | Obtained from Fisher Scientific (45-001-487) | Prepacked with Sephadex™ G-50 DNA Grade and pre-equilibrated in STE buffer containing 0.15% Kathon as Biocide | |
| Triathler Bench-top Scintillation counter | Hidex Oy, Turku, Finland | Triathler LSC Model: 425-034 | ||
| Novex® TBE-Urea Sample Buffer (2×) | Invitrogen | LC6876 | ||
| 6% TBE-Urea Gels 1.0 mm, 10 wells | Invitrogen | EC6865BOX | ||
| Novex® TBE Running Buffer (5×) | Invitrogen | LC6675 | ||
| Radioactivity decontaminant | Fisher Scientific | 04-355-67 | ||
| Gel-loading tips | Denville Scientific | P3080 | ||
| XCell SureLock Mini-Cell | Invitrogen | EI0001 | XCell SureLock Mini-Cell | |
| Autoradiography film | Denville Scientific | E3018 | Use in complete darkness | |
| Autoradiography film, Hyperfilm™ ECL | Amersham Biosciences | RPN3114K | Can be used under red safe light. | |
| Membrane discs | Millipore | GSWP02500 | Mixed cellulose ester, hydrophilic, 0.22-μm disc membranes | |
| Fritted glass support base for 125-ml flask | VWR | 26316-696 | ||
| Petri dishes | Fisher Scientific | 08-757-11YZ | ||
| Urea | Fisher Scientific | AC32738-0050 | ||
| EDTA | Fisher Scientific | 118430010 | ||
| Glycogen | Sigma | G1767 | ||
| 2-Propanol for molecular biology | Sigma | I9516 | ||
| Recombinant RNase inhibitor | USB Corporation | 71571 | ||
| ImProm-II™Reverse Transcription System | Promega | A3802 | ||
| Recombinant RNase inhibitor | USB Corporation | 71571 | ||
| RapidRun™ Loading Dye | USB Corporation | 77524 |
Ссылки
- Monien, B. H., Apostolova, L. G., Bitan, G. Early diagnostics and therapeutics for Alzheimer's disease-how early can we get there. Expert. Rev. Neurother. 6, 1293-1306 (2006).
- Nestor, P. J., Scheltens, P., Hodges, J. R. Advances in the early detection of Alzheimer's disease. Nat. Med. 10, S34-S41 (2004).
- Kawas, C. H. Clinical practice. Early Alzheimer's disease. N. Engl. J. Med. 349, 1056-1063 (2003).
- Haass, C., Selkoe, D. J. Soluble protein oligomers in neurodegeneration: lessons from the Alzheimer's amyloid β-peptide. Nat. Rev. Mol. Cell. Biol. 8, 101-112 (2007).
- Kirkitadze, M. D., Bitan, G., Teplow, D. B. Paradigm shifts in Alzheimer's disease and other neurodegenerative disorders: the emerging role of oligomeric assemblies. J. Neurosci. Res. 69, 567-577 (2002).
- Rahimi, F., Shanmugam, A., Bitan, G. Structure-function relationships of pre-fibrillar protein assemblies in Alzheimer's disease and related disorders. Curr. Alzheimer Res. 5, 319-341 (2008).
- Jayasena, S. D. Aptamers: an emerging class of molecules that rival antibodies in diagnostics. Clin. Chem. 45, 1628-1650 (1999).
- Bunka, D. H., Stockley, P. G. Aptamers come of age - at last. Nat. Rev. Microbiol. 4, 588-596 (2006).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Lee, J. F., Stovall, G. M., Ellington, A. D. Aptamer therapeutics advance. Curr. Opin. Chem. Biol. 10, 282-289 (2006).
- Weiss, S. RNA aptamers specifically interact with the prion protein PrP. J. Virol. 71, 8790-8797 (1997).
- Bibby, D. F. Application of a novel in vitro selection technique to isolate and characterise high affinity DNA aptamers binding mammalian prion proteins. J. Virol. Methods. 151, 107-115 (2008).
- Rhie, A. Characterization of 2'-fluoro-RNA aptamers that bind preferentially to disease-associated conformations of prion protein and inhibit conversion. J. Biol. Chem. 278, 39697-39705 (2003).
- King, D. J., Safar, J. G., Legname, G., Prusiner, S. B. Thioaptamer interactions with prion proteins: sequence-specific and non-specific binding sites. J. Mol. Biol. 369, 1001-1014 (2007).
- Proske, D. Prion-protein-specific aptamer reduces PrPSc formation. ChemBioChem. 3, 717-725 (2002).
- Murakami, K., Nishikawa, F., Noda, K., Yokoyama, T., Nishikawa, S. Anti-bovine prion protein RNA aptamer containing tandem GGA repeat interacts both with recombinant bovine prion protein and its β isoform with high affinity. Prion. 2, 73-80 (2008).
- Luhrs, T., Zahn, R., Wuthrich, K. Amyloid formation by recombinant full-length prion proteins in phospholipid bicelle solutions. J. Mol. Biol. 357, 833-841 (2006).
- Bunka, D. H. Production and characterization of RNA aptamers specific for amyloid fibril epitopes. J. Biol. Chem. 282, 34500-34509 (2007).
- Ylera, F., Lurz, R., Erdmann, V. A., Furste, J. P. Selection of RNA aptamers to the Alzheimer's disease amyloid peptide. Biochem. Biophys. Res. Commun. 290, 1583-1588 (2002).
- Rahimi, F., Murakami, K., Summers, J. L., Chen, C. H., Bitan, G. RNA aptamers generated against oligomeric Aβ40 recognize common amyloid aptatopes with low specificity but high sensitivity. PLoS ONE. 4, e7694-e7694 (2009).
- Bitan, G., Lomakin, A., Teplow, D. B. Amyloid β-protein oligomerization: prenucleation interactions revealed by photo-induced cross-linking of unmodified proteins. J. Biol. Chem. 276, 35176-35184 (2001).
- Rahimi, F., Maiti, P., Bitan, G. Photo-induced cross-linking of unmodified proteins (PICUP) applied to amyloidogenic peptides. J. Vis. Exp. , (2009).
- Bitan, G., Fradinger, E. A., Spring, S. M., Teplow, D. B. Neurotoxic protein oligomers-what you see is not always what you get. Amyloid. 12, 88-95 (2005).
- Bitan, G. Structural study of metastable amyloidogenic protein oligomers by photo-induced cross-linking of unmodified proteins. Methods Enzymol. 413, 217-236 (2006).
- Chen, C. H., Chernis, G. A., Hoang, V. Q., Landgraf, R. Inhibition of heregulin signaling by an aptamer that preferentially binds to the oligomeric form of human epidermal growth factor receptor-3. Proc. Natl. Acad. Sci. USA. 100, 9226-9231 (2003).
- Adams, D. S. . Lab math: a handbook of measurements, calculations, and other quantitative skills for use at the bench. , (2003).
- Gopinath, S. C. Methods developed for SELEX. Anal. Bioanal. Chem. 387, 171-182 (2007).
- Takahashi, T., Tada, K., Mihara, H. RNA aptamers selected against amyloid β-peptide (Aβ) inhibit the aggregation of Aβ. Mol. Biosyst. 5, 986-991 (2009).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены