このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
アミロイドβ-蛋白質、アルツハイマー病の原因物質のためのアプタマーの選択
要約
アプタマーは、で選択された短いribo-/deoxyribo-oligonucleotidesです体外特定のターゲットの親和性に基づく進化の方法。アプタマーは、多目的な治療、診断、および研究アプリケーションとの分子認識ツールです。我々は、アミロイドβ-蛋白質、アルツハイマー病の原因物質のためのアプタマーの選択のための方法を示しています。
要約
アルツハイマー病(AD)は1、その発症前診断が困難なレンダリング狡猾なコースを持つプログレッシブ、年齢依存性、神経変性疾患である。明確なADの診断は、このように、ADの発症前、早期診断が効果的な治療法2,3の開発と管理のための非常に重要である確立する、唯一の事後分析を実現しています。
アミロイドβ-蛋白質は(Aβ)ADの病因の中心です。水溶性、オリゴマーAβアセンブリはAD 4,5におけるシナプス機能障害とニューロンの損失の基礎となる神経毒性に影響を及ぼすと考えられている。可溶性Aβアセンブリの様々な形態が記載されている、しかし、それらの相互関係と、ADの病因および病因に関連性は複雑で、ウェル6理解されていない。症状が現れる前に、特定の分子認識ツールでは、Aβアセンブリ間の関係を解明し、疾患の過程で早期にこれらのアセンブリの検出と特徴付けを促進することができる。分子認識は、一般的に抗体に依存しています。しかし、分子認識ツールの代替クラス、アプタマーは、抗体7,8の相対重要な利点を提供しています。指数関数的濃縮(SELEX)9,10によるリガンドの系統進化:アプタマーは、 試験管内の選択によって生成されたオリゴヌクレオチドである。 SELEXは、ダーウィンの進化論に似て、可能にする、という反復的なプロセスであり、選択、増幅、濃縮、およびプロパティの永続化、など、熱心な、特定の、リガンド結合(アプタマー)または触媒活性(リボザイムとDNAzymes)。
現代のバイオテクノロジーと医学11のツールとしてのアプタマーの出現にもかかわらず、彼らはアミロイドの分野で十分に活用されています。いくつかのRNAまたはssDNAのアプタマーは、プリオン蛋白質(プリオン)12月16日の様々な形態に対して選択されています。組換えウシのPrPに対して生成されたRNAアプタマーは、アミロイド線維の18を形成する完全長のPrPの水溶性、オリゴマー、β-シートが豊富な立体配座の変異型、ウシのPrP -β17を認識することが示された。アプタマーは、線維のβ2 -ミクログロブリン(β2 m)の単量体といくつかの形式を使用して生成されたβ2メートルフィブリル19に加えて、特定の他のアミロイドタンパク質の線維を結合することが見出された。 Ylera ら 。固定化単量体のAβ4020に対して選択されたRNAアプタマーを説明した。不意に、これらのアプタマーは、線維Aβ40をバインド。完全に、これらのデータはいくつかの重要な問題を提起する。なぜ単量体のタンパク質に対して選択されたアプタマーは、それらのポリマー形態を認識するのですか?アミロイド生成性タンパク質の単量体及び/またはオリゴマーの形態に対するアプタマーが得られるか?これらの質問に対処するために、我々は、光誘起架橋無修正のタンパク質(PICUP)22,23を使用して生成された共有結合で安定化オリゴマーAβ4021のアプタマーを選択することを試みた。前回の調査結果17,19,20と同様に、これらのアプタマーは、おそらく構造aptatope 21潜在的に共通のアミロイドを認識Aβおよび他のいくつかのアミロイド蛋白質の線維と反応した。ここで、我々は、これらのアプタマー21の製造に使用されるSELEX方法論を提示する。
プロトコル
パート1:蛋白質の調製と架橋
当初は、SELEXに使用される蛋白質は、以前は23を説明するように、均質な、集約のない準備を取得するために1,1,1,3,3,3 -ヘキサフルオロ-2 -プロパノール(HFIP)で前処理されています。このステップは、事前に形成された凝集体は、貧しい人々の実験の再現性24で、その結果、アミロイド蛋白質の急速な凝集を誘導するために必要であるとタンパク質の非集約、非繊維状の形態のためのアプタマーの選択のために望ましくないです。
- 天秤を用いて純粋〜800μgの(〜180 nmol)をAβ40を秤量。 、シリコンコーティングされた、低吸着剤1.6 mlのマイクロチューブラベル付けに凍結乾燥ペプチドを転送します。
- 以前に23を説明するように0.5mMのペプチド溶液を得ることを100%1,1,1,3,3,3 -ヘキサフルオロ-2 -プロパノール(HFIP、400μlの)のペプチドを溶かす。
- 各チューブは、名目上〜45 nmolのAβ40を含むように4つの100μlのアリコートにこのソリューションを割ります。以前に23を説明するようにHFIPを除去するために進んでください。
- 架橋反応のためのバッファにHFIP処理ペプチドを可溶化する前に、架橋と消光試薬を準備する。過硫酸アンモニウム(APS、MW 228.2グラム/モル)を秤量し、10mMリン酸ナトリウムの40mMのpH7.4の溶液を調製。溶液が透明になるまでボルテックスを用いて混ぜる。
- 10 mMリン酸ナトリウム中のトリス(2,2 - ビピリジル)ジクロロ(II)六水和物(RuBpy、MW 748.63グラム/モル)、pH7.4の2mMの溶液を調製します。ボルテックスを用いて混合し、完全な溶解を確認してください。アルミ箔を使用して光からチューブを保護します。
- 消光試薬を準備します。ジチオスレイトール(DTT、MW 154.5グラム/モル)を秤量し、1 Mに、脱イオン水で、または10 mMのリン酸ナトリウム、pH7.4中に溶解
- HFIP処理ペプチドを溶解正確に、以前に23,25説明するが、〜60μMペプチド溶液を得ることを目指しています。
- 以前に23を説明するようにオリゴマーAβ40の混合物を生成するPICUPを実行します。典型的なPICUPの反応は、タンパク質、RuBpy、およびAPSの最終濃度が30μM、0.05 mMの、それぞれ23 1 mMの、ある20μlの体積で行われます。ここで、PICUPのために使用される混合物は、108μlのタンパク質、6μlのRuBpy、6μlのAPS、および1μlのDTTとタンパク質の濃度、RuBpy、とAPSが含まれていますすべての典型的な反応への相対倍増されています。これは、実行するPICUP反応の数が減少し、脱塩のためのタンパク質濃度を増加させる。
パート2:タンパク質調製物を脱塩
SELEXのためにタンパク質を使用する前に、脱塩がPICUPのために使用される架橋試薬を除去するために実行されます。このPICUPの反応混合物は、架橋試薬と〜55μMのタンパク質(名目濃度)が含まれています。
- 5 mlの脱塩カラムをオフにトップキャップを外し、スタンドで列をサポートして、カラムの出口下にビーカーを置き、コンセントプラグを取り外します。を介してストレージバッファの流れを聞かせてビーカーに集める。
- の10 mM酢酸アンモニウム、pHは8.3でカラムを平衡化する。列に3 mlのアリコートで、このバッファーの5樹脂床のボリューム(25 ml)を入れて、そのまま流れるようにできます。
- 一方、ラベル下位8吸着剤、シリコンでコーティングされた1.6 - mlチューブとチューブのラックに置いてください。
- カラムが平衡になった後、(列は後で使用するため保存され、洗浄し、そして平衡化することができます)、単一の脱塩の使用ごとにカラムごとPICUPの反応混合物の0.5〜0.7 mlのアリコートを適用します。タンパク質の混合物がカラムの樹脂に浸透し、ビーカーにフロースルーを破棄することができます。
- カラムの出口の下の最初のコレクションチューブを置きます。カラムに0.5mlの酢酸緩衝液を追加し、流れる最初の0.5 mlの画分を集める。
- ステップ2.4および2.5を繰り返して、そして対応するチューブの8つの0.5 mlのフラクションまで収集。
- 数別の下位8吸着剤、シリコンでコーティングされた、小型の0.6 mlチューブのセットとラックに配置します。として別のチューブにラベルを付ける"空白。"標識した0.6 mlチューブに各画分と、転送の150μlを取り出してください。前述の23に示す(図1)SDS - PAGE解析のための20μlを使用する。
- ブランクチューブに150μlの10 mM酢酸アンモニウム、pHは8.3、転送する。分光光度計で空白を設定するには、このチューブを使用してください。
- 石英キュベットにλ= 280nmにおける各画分130μlの吸光度を測定します。
- 吸光度を測定した後、最高のタンパク質含有量(通常は分画3-5)で分画を組み合わせて、ピペットを用いて混合し、実際のタンパク質濃度(図示せず)を決定するアミノ酸分析のために、10μlのアリコートを保つ。
- チューブあたり〜2ナノモルタンパク質の複数のアリコートにサンプルを分割し、凍結乾燥機でサンプルを凍結乾燥。
- 凍結乾燥終了後、以前のように100%のHFIPのサンプルを扱う。
- -20℃でチューブを保存するそれぞれのSELEXサイクル(以下)に対応するチューブを使用してください。
パート3:PCRによる合成ランダムなssDNAライブラリの増幅
クローニング部位(Bam HIで、 エコ RI)およびT7プロモーターを含む定常領域に挟まれた、としてSELEXのためにここで使用される合成のssDNAライブラリは49の無作為化ヌクレオチドを(C = 25:25:25:25%:T::G A)が含ま以前に26を説明した。
- 202μlの脱イオン水、40μlの10 × Taq DNAポリメラーゼバッファー、2μlのssDNAのテンプレート(0.5 nmol)を、120μlの25 mMのMgCl 2、28μLの10mMのデオキシヌクレオシド三リン酸の混合:ライブラリを増幅するために、次のように標準的なPCR反応をセットアップする(dNTPsを)、1μlのフォワードプライマー(300 pmol)を、1μlの逆方向のプライマー(300 pmol)を、4μlのTaqポリメラーゼ。
- 次の設定でサーマルサイクラーを用いたPCR反応行います:94℃で5分·初期変性C、アニーリング30秒、30秒、52 ° Cで20サイクル94℃のそれぞれ、72℃で30秒Cの拡張子、および7分間72℃での最終拡張子。
- PCR反応終了後、メーカーの指示に従ってキアゲンQIAクイックPCR精製キットを用いて増幅DNA産物を精製する。一般に、これらの実験で濃度とDNAの収率は160 ±50μlのでは10 ng /μLです。
- 2%アガロースゲル電気泳動によってPCR後のDNAの量を確認してください。
パート4: インビトロ転写により32 P -ラベルされたRNAの生成
- 20μlの5倍のT7転写バッファー、7.5μlの100mMのRATP、rGTP、rUTP、1μlの100の各々次のように多少の変更をメーカーの指示にしたがってO -リングに覆われた、1.6 - mlチューブで転写反応を設定するmMのrCTP、2μlのα32 P - CTP(3000 CI / mmol)を、5〜10μgのは、DNAテンプレート(DNA産物を精製したの〜30から40μl)を、10μlの酵素ミックス、そして100μlに、最終的なボリュームを構成するの精製ヌクレアーゼフリー水を添加することにより。
- ピペットで静かに溶液を混合、混合物を遠心分離し、37℃で一晩インキュベートする。
- 反応の終了時に、DNAテンプレートは削除する必要があります。 37℃4時間の鋳型DNAとインキュベートの1 U /μgの濃度にRQ1 RNaseフリーのDNaseを追加° CのDNAテンプレートを消化する。
- クロロホルム:イソアミルアルコール(125:24:1、pHは4.7)4時間後、クエン酸飽和フェノールの1ボリュームを追加することにより、RNAを抽出する。 2分間16,000 gで約1分間、遠心分離のためのボルテックスで混ぜる。
- 新しいチューブに上層、水相を転送したり、マイクロピペットを用いて吸引して底相を捨てる。イソアミルアルコール(24:1)、として4.4で説明される1分間、遠心分離のためのボルテックスして混合:クロロホルムの1ボリュームを追加します。
- 新しいチューブに上層、水相を転送したり、マイクロピペットを用いて吸引して底相を捨てる。残留クロロホルムは、マイクロピペットで底相のマイクロ遠心と除去の迅速なスピン(10秒)を行うことにより削除することができます。このステップでは、それは下の段階ではなく、上清を除去する方が簡単です。
- RNAを沈殿させる場合、3Mの酢酸ナトリウムの0.1体積当量、pHは5.2、および2 - プロパノール、1 - ボリュームの同等のものを追加。 15分間、-20℃の冷凍庫でのミックスと場所。
- 後15分、トップスピードでスピン、好ましくは4で冷蔵微量の° Cは、20〜30分のためにRNAの生成物を沈殿する。
- 注意深く上清を吸引、70%エタノールを0.5ml、4℃で遠心してRNAペレットを洗浄し、吸引によってエタノールを捨てる。
- ヒートブロックにRNAペレットを含むチューブに移し、5分間37℃でペレット℃で乾燥させる。
- インビトロ転写反応すなわち、100μlの(ステップ4.1)と同じボリュームに150mMのSTEバッファー、pH 8.0(illustra ProbeQuant G - 50ミクロに付属)またはヌクレアーゼフリー水でRNAサンプルを溶解する。
- ° C渦による熱ブロックとミックスで10分間RNAの溶解を容易にするために70でRNAの生成物を含むチューブを加熱する。
- 室温で1分間、最高速度で遠心する。
- シンチレーション計数とTBE -尿素ポリアクリルアミドゲル電気泳動(その6)のための標識0.6 mlチューブにRNAの1μlを分注しておいてください。
パート5:非法人のヌクレオチドの除去、RNA脱塩、およびシンチレーション計数
取り込まれなかったヌクレオチドを削除するには、製造元の指示に従って、2つのG - 50カラムを使用してください。
- 樹脂を再懸濁するためにボルテックスして、列とミックスを反転。
- キットに付属のプラスチック製のツールを使用して、ミシン目のカラムの底部閉鎖を折りますとコンセントはそのままにしておいてください。キャップの4分の1回転緩め、Gで提供されるクリーンなコレクションチューブにカラムを配置-50キット。
- ストレージのバッファを削除するには、1分間730 gでコレクションチューブにカラムをスピン。
- 二つの新しいO -リングに覆われた、1.6 - mlチューブにカラムを移し、樹脂を乱すことなく、直接、樹脂上にカラムごとに32 P -標識したRNAサンプル50μlをロードする。
- 精製した標識RNAを収集するために2分間730 gでスピン。このステップの後、カラムを破棄する。
- シンチレーション計測と電気泳動(第6報)0.6 mlのチューブにG - 50で精製したRNAの1μlを移す。 SELEXのため使用するまで-20℃で保存する株式RNA。それは、RNAはその分解や活性の損失を避けるために、標識後2日以内に使用されていることが望ましい。
- シンチレーションカウンターを用いて分あたりのカウント数(cpm /μl)を測定するステップ4.14および5.6〜2個の1μLRNAのアリコートを使用してください。ここで、我々はTriathlerのベンチトップ型シンチレーションカウンターを使用してください。
- Triathlerに付属の32 Pのカウントに使用されるプラスチック製のアダプターの内部にRNAサンプルの入ったチューブを、転送する。 、カウントチャンバー内チューブとアダプターを入れるチャンバーの蓋を閉じ、32 Pのカウントを選択し、カウントを開始する開始キーを押します。
- %ラベルの取り込みすなわち、α32 P - CTP =("G - 50"サンプル"TOTAL"サンプル用÷ CPM /μL用CPM /μL)× 100%の取り込みを計算する。全サンプルでは、G - 50精製前のサンプルです。
- パーセント取り込みのシンチレーション計数と計算した後、RNAの収量とRNAの比活性を計算する。前(237370 CPM /μL)とG - 50精製(191330 CPM /μL)後のRNAのプールの数が81%混入をもたらすたとえば、次は、RNAの収量を得る前に計算されます。
ステップ4.1から、3000 CI /モルおよび/ 10μCiの添加でα32 P - CTPの量は、次のとおりです。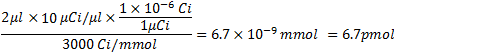 。
。
使用されるラベルのないCTPの量は、次のとおりです。 。
。
CTP使用済みの合計量は100006.7pmolです。 RNAの分子量は、次のように計算されます。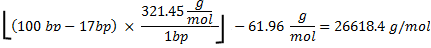
321.45グラム/モル27 rNTPsの平均質量である、100 bpのは、シーケンス内の塩基の数であり、17 bpは転写されていないT7プロモーターの塩基数です。オリゴヌクレオチド分子量から61.96グラム/モルの減算は、アカウントのHPO 2の除去(63.98)と2つの水素原子(2.02)27の加算になります。これは、すべてのCTPが生成されるRNAの理論的質量を組み込むことにした場合、4 rNTPsから、CTPは、制限試薬であり、されていました: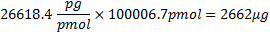 。
。
しかし、81%の取り込みの、この質量は2156μgです。 RNAのμgのあたりのカウント数は、次のようになります。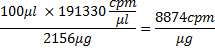
ここで、100μlのは、転写反応の量であり、モルの観点から:26618.4μgの/マイクロモルX 8874 CPM /μgの= 2.4x10 8 CPM /マイクロモル。例えば、計算することができますここから、nmolのとその適切な量のRNAの量は、タンパク質とのインキュベーションのために使用される、5μlのRNAは、〜4 nmolのRNA、すなわち、含まれています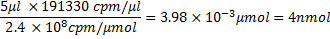
これらの実験では、100 nmolのRNAから1 nmolのタンパク質と4 nmolの〜300 pmolの21を使用した。
第6部:電気泳動とオートラジオグラフィーによるRNAの製品の特性評価
- 電気泳動用サンプルを準備するには、それぞれ、ステップ4.14および5.6〜G - 50精製の前と後のRNAの1μlを使用してください。 4μlのSTEバッファーと5μlの2倍NOVEX TBE -尿素サンプルバッファーを追加します。
- 70でサンプルを加熱°通常RNAの電気泳動(とと加熱することなくRNAサンプルのゲルの分解能は、この実験では同じであるため、加熱が不要であることが判明した)のために推奨されているように5分間。
- ゲル - 実行中の装置で6%TBE -尿素 - ポリアクリルアミドゲルを組み立てる、1X NOVEX TBE -尿素ランニングバッファーで内側と外側のチャンバーを埋める。 1 mlのピペットでバッファを使用して、プレキャストゲルのウェルをすすぎます。
- RNAのチューブを遠心し、ゲルローディングチップを使用してサンプル(10μlの合計)を読み込みます。 50分間180 Vで泳動する。
- 50分後、、プラスチック金型を分解してゲルを分解除去し、ゲル型の唯一の短いバッキングを破棄するが、ゲルのためのサポートとして長く支持しておきます。
- Decon(または他の適切な放射能の除染装置)は、すべての汚染放射性スポットが削除されていることを確認して作業領域の表面を清掃してください。
- プラスチック製のサランラップの二つの層のレイアウトを、二pliedラップでゲルとプラスチック製のバッキングを包む。
- のシートにビニールに包んでゲルを公開安全光の下で暗い部屋での露光カセット内部のutoradiography X線フィルム、。 60〜90分間、-20℃でカセットにしておきます。
- 自動フィルム現像機(図2)を使用して60から90分後に安全な光の下で暗い部屋で映画を開発する。
パート7:RNAのタンパク質のインキュベーションおよびフィルター結合
最初に、RNAとタンパク質が溶液中でインキュベートされ、その後タンパク質に結合するRNA配列は、非バインダーから分離されています。 SELEXサイクルの進捗状況として、フィルター結合はタンパク質のRNA結合濃縮の指標を与える。
- 変性のための10分間、その後ゆっくりと再生のための10分間、室温で90℃でRNAをインキュベートする。
- 8μlの60 mM NaOHにステップ2.12から蛋白質を溶かし、36μlヌクレアーゼフリー脱イオン水を加える。 1分間混合物を超音波洗浄し、36μlの2倍のRNA結合バッファー(20mMトリス、300mMのNaCl、10mMのMgCl 2、pH 7.5)を追加。
- 20μlの10xのRNA結合バッファーでRNAの適切な量を混合し、ヌクレアーゼフリー水を添加することにより200μlにボリュームを構成しています。チューブラベル"負の制御。"
- 20μlの10xのRNA結合バッファーで20μlのタンパク質やRNAの所望の量を混合し、ヌクレアーゼフリー水200μlにボリュームを構成しています。チューブ"反応"というラベルを付けます。
- 混合し、室温で30分間チューブをインキュベートする。一方、フィルタと次のステップのためのフィルタ結合セットアップを準備。
- 真空入口に125 mlの枝付きフラスコを取り付けます。枝付きフラスコでのフィルタのプレ洗浄、多孔質ガラスの支持を置きます。
- 10〜15分間35x10 mmのペトリ皿に1倍RNA結合バッファーを2-3 mlの3フィルターメンブレンを平衡化させます。最初のフィルタは、真空吸引を調整するために使用される、秒は負の制御フィルタの結合、RNA単独で使用され、3番目はRNA -タンパク質混合物のフィルター結合のために使用されます。
- 30分後、室温で5分間、トップスピードでの結合反応と制御のチューブを遠心してください。
- 真空をオンにして、多孔質ガラス上に第1膜を置きます。マイクロピペットを用いて、メンブレンにRNAを結合緩衝液0.5mlを滴下し、膜を介して各バッファのドロップの遅い流れを可能にするために真空を調整する。
- 多孔質ガラス上に第2の膜を配置し、同じ流速を用いて、メンブレンに負の制御を適用する。
- メンブレンを洗浄し、フロースルーを捨てるために1 × RNA結合バッファーの4x0.5 - mlのアリコートを適用します。前洗浄が必要な場合は、フロースルーは、RNA抽出のために保持されていることに注意してください。前洗浄は、フィルターに結合するRNA配列を除去。
- シンチレーションカウントのための相応標識1.6 mlチューブに陰性コントロール細胞膜と場所を削除します。
- プレ洗浄2の多孔質ガラスをサポートするように多孔質ガラスを交換してください。
- 多孔質ガラスの上に3台目のディスクを配置し、反応混合物を適用する。ステップ7.11のようにフィルターを洗浄し、フロースルーを捨てる。洗浄回数は、SELEX条件のストリンジェンシーを高めるために、後でSELEXサイクルで増加することができます。
- "反応混合物"というラベルの付いた1.6 mlチューブにフィルターディスクと場所を削除し、シンチレーション計数のためにしてください。
- ステップ5.8としてカウントシンチレーションを実行し、各フィルタのカウントをメモしておきましょう。
- 膜(%結合)に適用される放射能の総量に比べてフィルターに結合した放射能のレベルを計算する。これは、SELEXの進行に結合濃縮の指標を与える。
パート8:フィルタからのRNA抽出
RNAがタンパク質に結合する配列を取得するためのフィルタから抽出されます。これらのシーケンスは、次のSELEXサイクルのために増幅される。
- シンチレーション計数した後、きれいな、乾燥した、35x10 mmのペトリ皿にチューブからフィルター結合反応(ステップ7.15から)と場所を削除する。
- 小さな断片に膜をカットするクリーンなメスとピンセットを使用してください。
- ピンセットを使用して、ステップ8.1から同じラベルの付いたチューブの膜のカット部分を交換してください。
- 400μlの溶出バッファーを(7 M尿素、3mMのEDTA、100mMのクエン酸ナトリウム、pH5.0)を加え、10分間95℃でチューブをインキュベートする。
- 、室温で最高速度でチューブを遠心吸引し、新しいラベルが付いたチューブに抽出バターを収集する。
- 抽出効率を評価するためにシンチレーション計数により、膜の部分を含むチューブに残っている放射能のカウントを測定します。
- 三度抽出プロセス(ステップ8.4から8.6)を繰り返します。 3回抽出後の効率は通常95から96パーセントを〜です。
- クロロホルム:イソアミルアルコール(125:24:1)RNA抽出物を含むチューブに、クエン酸飽和の1ボリューム(400μl)を液(pH 4.7)フェノールを追加します。 〜1分と遠心のためのボルテックスして混合2分間16,000 gでGE。
- 新しいチューブに上層、水相を転送したり、マイクロピペットを用いて吸引して底相を捨てる。
- イソアミルアルコール(24:1)、2分間16,000 gで1分間、遠心するためのボルテックスして混合:クロロホルムの1ボリュームを追加します。
- 新しいチューブに上層、水相を転送したり、マイクロピペットを用いて吸引して底相を捨てる。
- RNAを沈殿させるために、3 M酢酸ナトリウム0.1容積、pHは5.2、RNA coprecipitant図3-4μlをグリコーゲン(10μg/μLの)、及び2 - プロパノール、1体積当量を追加します。一晩-20℃の冷凍庫でのミックスと場所。
- ステップ8.12からRNAを沈殿させるために4℃遠心° C、20〜30分のために好ましくは、最高速度で回転する。
- 遠心分離後、RNAはかろうじて見える程度である液相に分離します。チューブの底にこのかろうじて目に見える沈殿相を外れなく慎重に上清を吸引除去する。
- 70%エタノール0.5mlをRNA"ペレット"を洗う、4の最高速度で5分間遠心分離℃、かろうじて目に見える沈殿相を外れなしで吸引してエタノールを捨てる。
- 50μlのSTEバッファーにRNAペレットを溶解し、手順5のようにG - 50精製に進みます。
パート9:継続SELEXサイクルのための逆転写とPCR
SELEXの次のサイクルに進むには、RNAはDNAへ逆転写とPCRによって増幅にする必要があります。
- 5で0.6 mlチューブには、ラベルが付いて精製を3μl、8倍希釈したリバースプライマー2μlのとG - 50 -脱塩RNAを混ぜる。ラベルつのチューブ"ネガティブコントロール。"
- 70℃で5分間、そしてその後RNAに対するプライマーのハイブリダイゼーションを可能にするために別の氷上で5分間混合物をインキュベートする。
- 氷の上にこの混合物に、6.4μlヌクレアーゼフリー水、4μlのImProm - II 5倍反応緩衝液、1.6μlの25 mMのMgCl 2、1μlの10mMのdNTPミックス、1μlのRNaseinのリボヌクレアーゼインヒビター、1μlのImProm - II逆転写酵素を追加する15μlの合計を構成する。
- ネガティブコントロールのために、7.4μlヌクレアーゼフリー水を追加し、逆転写酵素を残す。それに応じてチューブにラベルを付けます。ネガティブコントロールは、前のサイクルからのDNAのコンタミが次のSELEXサイクルで増幅されていないことを確認するために含まれています。この手順は、手順4.3でのRQ1 RNaseフリーのDNaseによるDNAテンプレートの消化の有効性をテストします。
- アニーリング、70に続いて、それぞれ一本鎖DNA、、の延長のための1時間5分、42℃25℃で反応混合物をインキュベートするサーマルサイクラーを使用して℃で15分間の逆転写酵素を失活させる。
- 逆転写反応後、PCRミックスを設定します。 30μlのヌクレアーゼフリー水、10μlの10 × Taq緩衝、30μlの25 mMのMgCl 2、7μlの10mMのdNTPミックス、各プライマーを1μl、およびすべてのチューブに1μlのTaqポリメラーゼを追加。
- 9月14日サイクルの第3部のようにPCRプログラムを実行します。
- ステップ3.3のようなDNA産物を精製する。
- 15〜20分間(図3)の100 Vで2%アガロースに2μlのDNAローディング色素と電気泳動させるとDNA産物の8μlを混合します。
- DNA産物は、パート4のように標識されたRNAに転写し、SELEX法の次のサイクルのために使用できるようになります。
パート10:代表的な結果
SELEX実験では、各サイクル後のAβ40ターゲットとして使用するオリゴマー、各サイクルで使用されるRNAの品質、そして成功したRNAの抽出と増幅の性質は重要である。我々は、精製および架橋試薬を除去した後、SELEX用オリゴマーAβ40混合物を生成するためにPICUPを使用していました。第2部で説明されている脱塩実験は、通常50〜55%蛋白質の損失につながる。タンパク質の量と質は、吸光度測定(λ= 280)とSDS - PAGE(図1)を用いて評価することができる。これらの実験の一つで溶出したAβ40の標準的なSDS - PAGEのプロファイルを重ねて5個々の実験からAβ40溶出液の平均吸光度プロファイルを図1に示されています。データは、タンパク質が一貫してフラクション3-5および分数6以降溶出架橋試薬(フラクション7、図1の増加吸光度)でカラムをオフに溶出することを示している。 SDS - PAGEは、典型的なAβ40オリゴマーの分布22を示しています。タンパク質画分は、(2.11)凍結乾燥HFIP(2.11)、resolubilized(7.1)で処理し、SDS - PAGEによって再分析された後に、このディストリビューションに再現可能です。
それぞれのSELEXサイクルのためのRNAの完全性は、ヌクレアーゼ感受性リボ - ヌクレオチドが使用されている場合は特に、SELEXの進行を反復するためにも重要です。 RNA増幅とラベリング(パート4)の後、ラベルされたRNAの品質は、TBE -尿素 - ポリアクリルアミドゲル電気泳動によって評価することができる。 G - 50精製(P前と後そのまま標識RNA製品の典型的なプロフィールアート5)図2に示します。
それぞれのSELEXサイクルの後、RNAをPCR増幅(ステップ9.5)のためのDNAへのフィルタの結合(その8)と(その9)逆転写後の膜から抽出されます。前のサイクルからのDNAテンプレートは、次のサイクルのための32 P標識RNAを(パート4)を生成するために使用されます。前のサイクルからいくつかの汚染DNAテンプレートは、 インビトロ転写反応(パート4)の後に標識されたRNAの製品で解決しない場合は、SELEXサイクルの効率は、より多くのサイクルを要求し、削減されます。これを制御するために、それぞれのSELEXサイクルと対応する逆転写PCR反応後、アガロース電気泳動では、(9.12)が実行されます。陰性コントロール反応チューブ(9.4)でDNA産物の不在は、以前のSELEXサイクル(図3)から発信されたDNAテンプレートが正常に削除を示します。 PCRによるDNAの増幅は陰性コントロールチューブで観察されている場合、それはRQ1 DNase処理(4.3)とのインキュベーションの持続時間が延長されることをお勧めします。 RQ1 DNaseで、製造元が推奨するインキュベーション時間は15分である、しかし、我々は長いインキュベーション(4-5時間)は(図3)完全にテンプレートDNAを除去するのに必要とされたことがわかった。
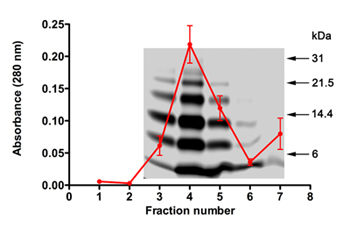
図1 SDS - PAGEとPICUPが生成した、脱塩Aβ40オリゴマーの吸光度プロファイル。 5個々の脱塩カラムから吸光度プロファイルを平均し、代表的なゲルで覆われました。分子量マーカーを右側に表示されます。

G - 50精製前後のRNA製品2。TBE -尿素-ポリアクリルアミドゲル電気泳動図 。示されるようにRNAの製品の移行の方向は、カソードからアノードになります。
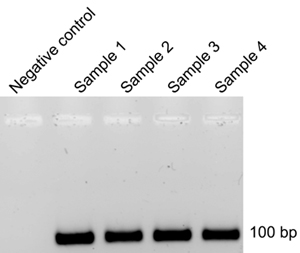
逆転写とPCR増幅後のDNA産物の3。アガロースゲル電気泳動図 。
ディスカッション
SELEXプロセスの出発点は、通常、10 12 -10 15配列を含む、ランダムオリゴヌクレオチドライブラリーの合成です。 RNA SELEXで、ここに示したのに対し、DNA SELEXでは、このライブラリは、ssDNAのプールが生成された後に直接使用され、ssDNAのライブラリは、 インビトロ転写によって酵素的にRNAのプールに最初に変換されます。各サイクルが曝露と目的のターゲットへのオリゴ...
開示事項
謝辞
この作品は、公衆衛生のカリフォルニア部からNIH / NIAと07から65798からの補助金AG030709によってサポートされていました。我々は、サポートおよび試薬を提供するためのペプチドの合成とアミノ酸分析のためのマーガレットM.コンドロン、プロジェクトの最初のステップを支援し、サポートするための博士エリザベスF.ニューフェルド、博士カイ香港B.チェンを認識し、博士とアンドリューD有用な議論のため。エリントン。
資料
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| Aβ40 | UCLA Biopolymers Laboratory | Lyophilized powder | ||
| MX5 Automated-S Microbalance | Mettler Toledo | |||
| Silicon-coated, 1.6-ml tubes | Denville Scientific | C19033 or C19035 | ||
| 1,1,1,3,3,3-hexafluoro-2-propanol (HFIP) | TCI America | H0424 | Use in a fume hood. | |
| Ammonium persulfate | Sigma | A-7460 | Vortex until the solution is clear. APS is prepared freshly each time and should be used within 48 h. | |
| Tris(2,2-bipridyl)dichlororuthenium(II) hexahydrate | Sigma | 224758-1G | Vortex until the solution is clear. Cover the RuBpy tube with foil to protect the reagent from ambient light. RuBpy is prepared freshly each time and should be used within 48 h. | |
| Dithiothreitol (DTT) | Sigma | 43815 | ||
| D-Salt™ Excellulose™ desalting columns | Thermo Scientific | 20449 | ||
| Ammonium acetate | Fisher Scientific | A637-500 | ||
| Silicon-coated, 0.6-ml tubes | Denville Scientific | C19063 | ||
| Novex Tricine Gels (10–20%) | Invitrogen | EC6625B0X | 10-well; mini size (8 cm X 8 cm); 25 μl loading volume per well; separation range 5 kDa to 40 kDa | |
| Quartz cuvette | Hellma | 105.250-QS | ||
| Beckman DU 640 spectrophotometer | Beckman | |||
| ssDNA library | Integrated DNA Technologies | Custom-ordered | The library was designed to contain 49 random nucleotides flanked by two constant regions containing primer-binding and cloning sites: 5'-TAA TAC GAC TCA CTA TAG GGA ATT CCG CGT GTG C (N:25:25:25:25%) (N)49 G TCC GTT CGG GAT CCT C-3' | |
| Taq DNA polymerase | USB Corporation | 71160 | Recombinant Thermus aquaticus DNA Polymerase supplied with 10× PCR Buffer and a separate tube of 25 mM MgCl2 for routine PCR. | |
| PCR Nucleotide Mix, 10 mM solution | USB Corporation | 77212 | (10 mM each dATP, dCTP, dGTP, dTTP) | |
| Forward primer | Integrated DNA Technologies | Custom-ordered | 5'-TAA TAC GAC TCA CTA TAG GGA ATT CCG CGT GTG C-3' | |
| Reverse primer | Integrated DNA Technologies | Custom-ordered | 5'-GAG GAT CCC GAA CGG AC-3' | |
| Thermal cycler | Denville Scientific | Techne TC-312 | ||
| QIAquick PCR Purification Kit (50) | QIAGEN | 28104 | ||
| Agarose | Denville Scientific | CA3510-8 | ||
| Conical, sterile 1.6-ml tubes with caps attached with O-rings | Denville Scientific | C19040-S | ||
| RiboMAX™ Large Scale RNA Production System–T7 | Promega | P1300 | The kit contains: 120 μl Enzyme Mix (RNA polymerase, recombinant RNasin® ribonuclease inhibitor and recombinant inorganic pyrophosphatase); 240 μl transcription 5 buffer; 100 μl each of 4 rNTPs, 100 mM; 110 U RQ1 RNase-free DNase, 1 U/μl; 10 μl linear control DNA, 1 mg/ml; 1 ml 3M sodium acetate (pH 5.2); 1.25 ml nuclease-Free water | |
| α-32P-cytidine 5'-triphosphate, 250 μCi (9.25 MBq), | Perkin Elmer | BLU008H250UC | Specific Activity: 3000 Ci (111 TBq)/mmol, 50 mM Tricine (pH 7.6) | |
| Citrate-saturated phenol:chloroform:isoamyl alcohol (125:24:1, pH 4.7) | Sigma (Fluka) | 77619 | ||
| Chloroform:Isoamyl alcohol (24:1) | Sigma | C0549 | ||
| Absolute ethanol for molecular biology | Sigma | E7023 | ||
| Z216-MK refrigerated microcentrifuge | Denville Scientific | C0216-MK | ||
| illustra ProbeQuant™ G-50 Micro Columns | GE Healthcare | Obtained from Fisher Scientific (45-001-487) | Prepacked with Sephadex™ G-50 DNA Grade and pre-equilibrated in STE buffer containing 0.15% Kathon as Biocide | |
| Triathler Bench-top Scintillation counter | Hidex Oy, Turku, Finland | Triathler LSC Model: 425-034 | ||
| Novex® TBE-Urea Sample Buffer (2×) | Invitrogen | LC6876 | ||
| 6% TBE-Urea Gels 1.0 mm, 10 wells | Invitrogen | EC6865BOX | ||
| Novex® TBE Running Buffer (5×) | Invitrogen | LC6675 | ||
| Radioactivity decontaminant | Fisher Scientific | 04-355-67 | ||
| Gel-loading tips | Denville Scientific | P3080 | ||
| XCell SureLock Mini-Cell | Invitrogen | EI0001 | XCell SureLock Mini-Cell | |
| Autoradiography film | Denville Scientific | E3018 | Use in complete darkness | |
| Autoradiography film, Hyperfilm™ ECL | Amersham Biosciences | RPN3114K | Can be used under red safe light. | |
| Membrane discs | Millipore | GSWP02500 | Mixed cellulose ester, hydrophilic, 0.22-μm disc membranes | |
| Fritted glass support base for 125-ml flask | VWR | 26316-696 | ||
| Petri dishes | Fisher Scientific | 08-757-11YZ | ||
| Urea | Fisher Scientific | AC32738-0050 | ||
| EDTA | Fisher Scientific | 118430010 | ||
| Glycogen | Sigma | G1767 | ||
| 2-Propanol for molecular biology | Sigma | I9516 | ||
| Recombinant RNase inhibitor | USB Corporation | 71571 | ||
| ImProm-II™Reverse Transcription System | Promega | A3802 | ||
| Recombinant RNase inhibitor | USB Corporation | 71571 | ||
| RapidRun™ Loading Dye | USB Corporation | 77524 |
参考文献
- Monien, B. H., Apostolova, L. G., Bitan, G. Early diagnostics and therapeutics for Alzheimer's disease-how early can we get there. Expert. Rev. Neurother. 6, 1293-1306 (2006).
- Nestor, P. J., Scheltens, P., Hodges, J. R. Advances in the early detection of Alzheimer's disease. Nat. Med. 10, S34-S41 (2004).
- Kawas, C. H. Clinical practice. Early Alzheimer's disease. N. Engl. J. Med. 349, 1056-1063 (2003).
- Haass, C., Selkoe, D. J. Soluble protein oligomers in neurodegeneration: lessons from the Alzheimer's amyloid β-peptide. Nat. Rev. Mol. Cell. Biol. 8, 101-112 (2007).
- Kirkitadze, M. D., Bitan, G., Teplow, D. B. Paradigm shifts in Alzheimer's disease and other neurodegenerative disorders: the emerging role of oligomeric assemblies. J. Neurosci. Res. 69, 567-577 (2002).
- Rahimi, F., Shanmugam, A., Bitan, G. Structure-function relationships of pre-fibrillar protein assemblies in Alzheimer's disease and related disorders. Curr. Alzheimer Res. 5, 319-341 (2008).
- Jayasena, S. D. Aptamers: an emerging class of molecules that rival antibodies in diagnostics. Clin. Chem. 45, 1628-1650 (1999).
- Bunka, D. H., Stockley, P. G. Aptamers come of age - at last. Nat. Rev. Microbiol. 4, 588-596 (2006).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Lee, J. F., Stovall, G. M., Ellington, A. D. Aptamer therapeutics advance. Curr. Opin. Chem. Biol. 10, 282-289 (2006).
- Weiss, S. RNA aptamers specifically interact with the prion protein PrP. J. Virol. 71, 8790-8797 (1997).
- Bibby, D. F. Application of a novel in vitro selection technique to isolate and characterise high affinity DNA aptamers binding mammalian prion proteins. J. Virol. Methods. 151, 107-115 (2008).
- Rhie, A. Characterization of 2'-fluoro-RNA aptamers that bind preferentially to disease-associated conformations of prion protein and inhibit conversion. J. Biol. Chem. 278, 39697-39705 (2003).
- King, D. J., Safar, J. G., Legname, G., Prusiner, S. B. Thioaptamer interactions with prion proteins: sequence-specific and non-specific binding sites. J. Mol. Biol. 369, 1001-1014 (2007).
- Proske, D. Prion-protein-specific aptamer reduces PrPSc formation. ChemBioChem. 3, 717-725 (2002).
- Murakami, K., Nishikawa, F., Noda, K., Yokoyama, T., Nishikawa, S. Anti-bovine prion protein RNA aptamer containing tandem GGA repeat interacts both with recombinant bovine prion protein and its β isoform with high affinity. Prion. 2, 73-80 (2008).
- Luhrs, T., Zahn, R., Wuthrich, K. Amyloid formation by recombinant full-length prion proteins in phospholipid bicelle solutions. J. Mol. Biol. 357, 833-841 (2006).
- Bunka, D. H. Production and characterization of RNA aptamers specific for amyloid fibril epitopes. J. Biol. Chem. 282, 34500-34509 (2007).
- Ylera, F., Lurz, R., Erdmann, V. A., Furste, J. P. Selection of RNA aptamers to the Alzheimer's disease amyloid peptide. Biochem. Biophys. Res. Commun. 290, 1583-1588 (2002).
- Rahimi, F., Murakami, K., Summers, J. L., Chen, C. H., Bitan, G. RNA aptamers generated against oligomeric Aβ40 recognize common amyloid aptatopes with low specificity but high sensitivity. PLoS ONE. 4, e7694-e7694 (2009).
- Bitan, G., Lomakin, A., Teplow, D. B. Amyloid β-protein oligomerization: prenucleation interactions revealed by photo-induced cross-linking of unmodified proteins. J. Biol. Chem. 276, 35176-35184 (2001).
- Rahimi, F., Maiti, P., Bitan, G. Photo-induced cross-linking of unmodified proteins (PICUP) applied to amyloidogenic peptides. J. Vis. Exp. , (2009).
- Bitan, G., Fradinger, E. A., Spring, S. M., Teplow, D. B. Neurotoxic protein oligomers-what you see is not always what you get. Amyloid. 12, 88-95 (2005).
- Bitan, G. Structural study of metastable amyloidogenic protein oligomers by photo-induced cross-linking of unmodified proteins. Methods Enzymol. 413, 217-236 (2006).
- Chen, C. H., Chernis, G. A., Hoang, V. Q., Landgraf, R. Inhibition of heregulin signaling by an aptamer that preferentially binds to the oligomeric form of human epidermal growth factor receptor-3. Proc. Natl. Acad. Sci. USA. 100, 9226-9231 (2003).
- Adams, D. S. . Lab math: a handbook of measurements, calculations, and other quantitative skills for use at the bench. , (2003).
- Gopinath, S. C. Methods developed for SELEX. Anal. Bioanal. Chem. 387, 171-182 (2007).
- Takahashi, T., Tada, K., Mihara, H. RNA aptamers selected against amyloid β-peptide (Aβ) inhibit the aggregation of Aβ. Mol. Biosyst. 5, 986-991 (2009).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved