Method Article
Microarrays de ADN: Control de calidad de la muestra, la hibridación de la matriz y de escaneo
En este artículo
Resumen
Se demuestra el uso de microarrays de ADN para la expresión de perfiles del sistema nervioso. Se describe el control de ARN de calidad, etiquetado de la muestra, y la hibridación de la matriz y de exploración.
Resumen
Microarrays de perfiles de expresión del sistema nervioso proporciona un enfoque de gran alcance para la identificación de las actividades de genes en diferentes etapas de desarrollo, diferentes estados fisiológicos o patológicos, la respuesta al tratamiento, y, en general, cualquier condición que está siendo probado experimentalmente 1. Perfiles de expresión de los tejidos neurales requiere el aislamiento de ARN de alta calidad, la amplificación del ARN aislado y la hibridación de microarrays de ADN. En este artículo se describen los protocolos de los experimentos de microarrays reproducible a partir de tejido del tumor cerebral 2. Vamos a empezar realizando un análisis de control de calidad de aislados de muestras de ARN con 2100 de Agilent Bioanalyzer "lab-on-a-chip" de tecnología. De alta calidad las muestras de ARN son esenciales para el éxito de cualquier experimento de microarrays, y el Bioanalyzer 2100 proporciona una medición rápida y cuantitativa de la calidad de la muestra. Muestras de ARN se amplifican y etiquetados mediante la realización de la transcripción reversa para obtener ADNc, seguido por la transcripción in vitro en presencia de nucleótidos marcados para producir la etiqueta cRNA. Mediante el uso de un kit de marcaje de dos colores, que se etiqueta de la muestra experimental con Cy3 y una muestra de referencia con Cy5. Ambas muestras serán combinadas y se hibridan a las matrices K 4x44 de Agilent. Dos colores arrays ofrecen la ventaja de una comparación directa entre dos muestras de ARN, lo que aumenta la precisión de las mediciones, en particular para los pequeños cambios en los niveles de expresión, ya que las dos muestras de ARN se hibridan de manera competitiva a una sola micromatriz. Las matrices se analizarán en las dos longitudes de onda correspondientes, y la proporción de Cy3 a Cy5 señal para cada función se utiliza como una medida directa de la abundancia relativa del ARNm correspondiente. Este análisis identifica los genes que se expresan diferencialmente en respuesta a las condiciones experimentales se está probando.
Protocolo
1. Análisis de calidad de control de la muestra de ARN con 2100 Bioanalyzer
Antes de empezar,
- Desnaturalizar la escalera y las muestras a 70 ° C durante 10 min. Enfriar inmediatamente en hielo.
- Limpie los electrodos con el instrumento RNaseZAP durante 1 minuto, seguido por RNasa libre de agua durante 10 segundos. Permita que los electrodos se seque al aire.
- Preparar la matriz de gel de pipeta 550 l de la matriz de gel en un filtro de giro incluye en el kit. Centrifugar a 1500 rcf durante 10 min a temperatura ambiente. Alícuota del filtrado de gel en 65 alícuotas y se almacenan a 4 ° C durante un máximo de un mes.
- Para preparar la estación de cebado de chips,
- Inserte una jeringa de 1 ml a través del clip y en el adaptador luer lock.
- Ajuste de la placa base a la posición C aflojando el tornillo en la base, el levantamiento de la placa y volver a apretar el tornillo.
- Ajuste el clip de la jeringa hasta la primera posición.
- Equilibre el concentrado de tinte a la temperatura ambiente. Vortex durante 10 segundos. Añadir 1 l de medio de contraste para una alícuota de 65 l de gel filtrados. Vortex bien y se centrifuga a 13.000 rcf durante 10 min a temperatura ambiente. Utilizar en un día.
Cuando esté listo para ejecutar sus muestras, - Posición de un chip en la estación de cebado. En este protocolo se está utilizando ARN de 6.000 fichas nano. Para cargar el gel,
- Pipetear 9 l de la mezcla de gel colorante en la bien marcada con un fondo de "G" sobre un fondo negro. Asegúrese de que su punta se coloca en el fondo del pozo.
- La posición del émbolo de la jeringa de 1 ml. Cerca de la estación de cebado. Asegúrese de que un clic.
- Presione el émbolo lentamente pero sin pausa hasta que se lleva a cabo por el clip.
- Espere 30 segundos. Suelte el clip.
- Deje que el émbolo sube por sí mismo. Después de que deje de moverse, esperar unos segundos y tire del émbolo hacia atrás a la posición de 1 ml. Abra la estación de cebado.
- Pipetear 9 l de la mezcla de gel colorante en cada uno de los dos pozos de la marca "G".
- Pipeta de 5 l de la marca en cada uno de los 12 pozos de la muestra y en la escalera también.
- Pipeta de 1 l de la escalera en escalera y preparada.
- Pipeta de 1 l de cada muestra en los pocillos de la muestra.
- Pipeta de 1 l de la marca en cada muestra pozo abandonado.
- Vortex el chip durante 1 minuto a 2.400 rpm.
- Para ejecutar el chip,
- Inicie el software de expertos para el 2100.
- Coloque el chip y cierre la tapa. Si el instrumento está en línea, aparecerá un icono si la tapa está abierta o cerrada y qué tipo de chip se inserta. Asegúrese de que el puerto correcto está seleccionado.
- Realizar el ensayo dentro de los 5 min de la carga de las muestras para evitar la evaporación.
2. Amplificación y etiquetado
Para prepararse para el análisis de microarrays, el RNA de muestras se amplifican y etiquetado, por lo general en una reacción basada en la RNA polimerasa T7 3. CDNA de doble cadena se genera por la transcripción reversa. cDNA se utiliza en una reacción de transcripción in vitro para generar lo que se conoce como cRNA. Esta reacción se lleva a cabo en presencia de ribonucleótidos etiquetado, la producción de cantidades de microgramos de ARN marcado para la hibridación de la matriz. La elección de los métodos de amplificación / etiquetado depende de la plataforma de microarrays después de ser utilizado. En esta sección, se describe la generación de la etiqueta fluorescente del ARN utilizando rápida Agilent kit etiquetado Amp.
- Pipeta de 50 ng a 5 g de ARN en un tubo de 1,5 ml. El volumen no debe superar los 8,3 l. Si es necesario, el concentrado de la muestra de RNA por centrifugación al vacío o precipitación con alcohol.
- Añadir 1,2 l de imprimación T7.
- Llevar a un volumen final de 11,5 ml con agua libre de RNasa.
- Se incuba a 65 ° C durante 10 minutos para desnaturalizar el ARN. Recomendamos el uso de un termociclador con una tapa caliente para evitar la condensación en la parte superior del tubo.
- Durante los 10 minutos de incubación, cálido Buffer 5X primer capítulo a 80 ° C durante 5 min. Vortex y centrifugar. Mantener a temperatura ambiente hasta que esté listo para su uso.
- Después de la incubación de 10 minutos, enfriar rápidamente por la desnaturalización de muestras de ARN en el hielo.
- Prepare la mezcla maestra cDNA. Por reacción, añadir lo siguiente:
- 4 l Buffer 5X primer capítulo
- 2 l 0,1 M TDT
- 1 l 10 mM dNTPs
- 1 l MMLV-RT enzima
- 0,5 l de salida RNasa.
- Mezcle los componentes invirtiendo el tubo 4 veces. No vórtice. Girar hacia abajo para recoger el contenido en la parte inferior del tubo.
- Añadir 8,5 l de mezcla maestra por muestra. Mix de pipeta hacia arriba y abajo.
- Incubar durante 2 horas a 40 ° C, seguido por 10 min a 65 ° C. Recomendamos el uso de un termociclador con una tapa caliente.
- La transferencia de los tubos en hielo durante 5 min.
- Prepare la mezcla maestra de transcripción. Por reacción, añadir lo siguiente:
- 15,3 l agua libre de nucleasa
- 20 lTampón de transcripción 4X
- 6 l 0,1 M TDT
- 8 PNT l
- 6.4 l 50% de PEG (PEG debe precalentado a 40 ° C y vortex para asegurar la resuspensión)
- 0,5 l de RNasa de salida
- 0.6 pirofosfatasa inorgánica l
- 0,8 l T7 RNA polimerasa
- 2,4 l CTP Cy3 o Cy5
- En pocas palabras girar las muestras a recoger el contenido en la parte inferior del tubo.
- Añadir 60 l de mezcla de transcripción maestro por cada muestra.
- Se incuba a 40 ° C durante 2 horas.
- Añadir 20 l de agua libre de RNasa.
- Purificar el cRNA marcado para eliminar los nucleótidos no incorporados. Recomendamos el uso de columnas RNeasy Mini Qiagen.
- Cuantificar la etiqueta cRNA. Recomendamos el uso de un espectrofotómetro NanoDrop2000 en el modo de medición de microarrays. Asegúrese de seleccionar el ARN-40 como el tipo de muestra.
- Registro de concentración de la muestra, el rendimiento (calculado como la concentración multiplicada por el volumen) y la actividad específica (calculada en 1000, multiplicado por la ración de la concentración de colorante sobre la concentración de cRNA). Para la hibridación de microarrays, es necesario para lograr un rendimiento de al menos 825 ng y una actividad específica de al menos 8 pmol / mg.
3. Microarrays de hibridación
- Encienda la estación de hibridación y regulado a 65 ° C por lo menos 2 horas antes de la hibridación se inicia.
- Para preparar la muestra de la hibridación, mezcla lo siguiente en un tubo de 1,5 ml:
- 825 ng Cy3-cRNA (normalmente es el "tratamiento" de la muestra)
- 825 ng Cy5-cRNA (normalmente es el de "referencia" de la muestra)
- 11 l agente de bloqueo de 10 veces.
- Añadir RNasa libre de agua hasta un volumen final de 52,8 l
- Añadir 2,2 l de amortiguación fragmentación.
- Vortex a baja velocidad para mezclar.
- Se incuba a 60 ° C durante exactamente 30 minutos. Este es el paso cRNA fragmentación, lo que genera fragmentos etiquetados para la hibridación de la matriz. Es muy importante para la incubación de exactamente como se describe. Se recomienda el uso de un termociclador con una tapa caliente. Es muy importante para la incubación de exactamente como se describe para generar sondas de la longitud media correcta. La fragmentación excesiva o insuficiente, dará lugar a falsos negativos o falsos positivos.
- Añadir 55 l de tampón de hibridación 2X para detener la fragmentación. Mezclar con la pipeta, teniendo especial cuidado de no introducir burbujas.
- Centrifugar durante 1 minuto a velocidad máxima para recoger el contenido en la parte inferior del tubo. Si se observan burbujas, ya centrífuga para eliminarlos.
- Coloque el tubo en hielo, protegido de la luz. Cargue la muestra en la matriz tan pronto como sea posible.
- Para cargar la matriz, primero preparar la cámara de hibridación. Este protocolo describe el uso de arrays 4x44K Agilent con el sistema de hibridación Roche-Nimblegen y cámaras A4 mezclador.
- Coloque una diapositiva de microarrays en la parte de código de barras de montaje / desmontaje de herramientas, en primer lugar.
- Abrir una mesa de mezclas A4 y exponer el adhesivo. La celebración de la matriz y el montaje / desmontaje de herramientas para impedir cualquier movimiento, coloque la mezcla A4 en la diapositiva, comenzando en el extremo. Asegúrese de que esté correctamente alineado con la herramienta. Pegue la mesa de mezclas a lo largo de la diapositiva matriz.
- Tire del extremo de la mesa de mezclas para eliminar la diapositiva / dispositivo de mezclas.
- Con la herramienta rebuzno, la prensa a lo largo de la junta adhesiva para asegurarse de que está bien pegado a theslide. Las pequeñas burbujas de aire atrapadas entre el adhesivo y deslice la puede ver sobre un fondo oscuro. Tómese un tiempo extra para eliminar estas burbujas con la herramienta de rebuznar. Su matriz está ahora listo para ser cargado.
- Utilice una pipeta de desplazamiento positivo para carga de 100 l de la muestra de la hibridación. Presione la punta firmemente en el interior del orificio del puerto y luego prescindir lenta y suavemente para que el aire no quede atrapado en la zona de la matriz. Nunca trate de succionar la muestra una copia de seguridad. Una vez que toda la muestra se ha dispensado, mantener la punta en el orificio del puerto, sin soltar el émbolo. Cuando la muestra abarca toda la gama y el líquido comienza a salir del orificio de ventilación, eliminar rápidamente la pipeta. Sólo suelte el émbolo una vez que la punta está lejos de la diapositiva.
- Frote suavemente el exceso de líquido en ambos puertos. Asegúrese de que usted no extraer la muestra de la propia cámara.
- Cubrir los orificios con pegatinas con pinzas.
- Poner la diapositiva en la estación de hibridación, y compruebe que los orificios de la vejiga están colocados correctamente en las juntas tóricas. Esto asegurará una mezcla adecuada durante la hibridación.
- Cierre la tapa deslizante y la tapa de la estación. Conjunto de la estación de mezclar el modo B.
- Hibridar la matriz a 65 ° C durante 17 horas.
- Prepare el tampón de lavado 2 mediante la adición de Triton X-102 a una concentración final de 0,005%. Mantener a 37 ° C durante la noche.
4. Microarrays de lavado
- Añadir Triton X-102 a un tampón de lavado a una concentración final de 0,005%. Mantener a temperatur ambientee.
- Cuando la hibridación es terminado, llenar un vaso plato "A" con el tampón de lavado 1. El plato debe ser suficientemente grande para contener la herramienta de montaje / desmontaje y permitir que algunas maniobras también. Todos los lavados se debe hacer en recipientes de vidrio. Evite los platos de plástico, ya que tienden a lixiviar los compuestos resultantes de fondo en alta en la matriz.
- Ponga una rejilla de diapositivas y una barra agitadora en una placa de tinción "B". Rellenar con un tampón de lavado, asegurándose de que cubre el bastidor. Colocar en una placa de agitación a temperatura ambiente.
- Ponga una barra de agitación en un plato de tinción vacío "C" y colocar en una placa de agitación.
- Eliminar la matriz y lo puso en la herramienta de montaje / desmontaje. Sumergir a toda la asamblea en el plato "A".
- Mientras sostiene la herramienta de montaje / desmontaje y deslice la de los lados opuestos con una mano, con cuidado retire el mezclador A4. Asegúrese de que no se rasque el área de la matriz.
- Rápidamente el lugar de la diapositiva en el soporte en la tinción de plato "B". A partir de ahora, la exposición al aire se debe minimizarse, ya que el tinte Cy5 es sensible a la capa de ozono. No lavar más de 8 conjuntos a la vez.
- Lávese las manos por un minuto de agitación a velocidad media.
- Rellene tinción plato "C" con tampón de lavado precalentado 2. Transferencia de las diapositivas y de lavado de 1 minuto.
- Muy lentamente quitar la rejilla de diapositivas de las manchas plato "C" para reducir al mínimo las gotas dejaron atrás en la diapositiva.
- Seco de la diapositiva al hacer girar durante 2 minutos. Si una centrífuga compatible no está disponible, se centrifuga la diapositiva en un tubo cónico de 50 ml o un golpe de gas argón en él.
- Colocar los portaobjetos en un tubo cónico de 50 ml y llenarlo con gas argón. Exploración de inmediato para evitar la pérdida de señal. Se recomienda el uso del escáner GenePix 4000B de Molecular Devices.
5. Los resultados representativos:
Buenas muestras de la calidad del ARN total debe producir sólo dos picos más importantes se ejecutan en un Bioanalyzer de control de calidad, correspondientes a las dos principales especies de ARN ribosomal. Algunos la degradación del ARN se verá como una prueba antes de que el primer pico de ARN ribosomal. Severamente degradados, el ARN de baja calidad se muestran un pico ancho y una serie de picos en los tiempos de retención baja, mientras que los dos picos de ARN ribosomal será de muy baja intensidad o no ser identificada en absoluto. Para ver ejemplos de la salida de 2100 Bioanalyzer, véase la figura 1.
El experto 2100 software calculará un número de la integridad del ARN, o RIN, como una medida cuantitativa de la calidad del ARN. Muestras de alta calidad, con valores de RIN superior a 9, son, evidentemente, mejor para las aplicaciones de microarrays. Sin embargo, se han utilizado muestras con valores de RIN tan bajo como 5,2 en la generación de datos de microarrays de calidad razonable.
Matrices de buena calidad debe producir una señal de alto en el PMT valores relativamente bajos. En nuestro experimento, la mayoría de las transcripciones se espera que estén presentes en niveles similares en la muestra experimental y de referencia; cambios masivos, muy extendida en la expresión de genes probablemente carecen de importancia biológica. Por lo tanto, la mayor parte de la matriz debe tener un color amarillento en vez de verde o rojo. Señales de buena calidad también debe estar en un rango dinámico de tal manera que los histogramas de señal totalmente superponen. Para ejemplos, véase la figura 2.
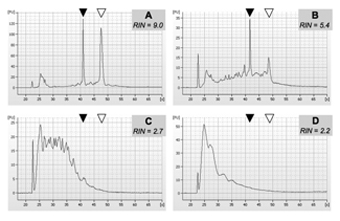
Figura 1. Ejemplo de cuatro muestras de ARN aislado de los tejidos humanos tumor cerebral. RNA fue aislado utilizando el protocolo presentado en el punto 3.1.1. Calidad de la muestra se evaluó utilizando el Bioanalyzer 2100 en un chip 6000 de ARN como se describe en el punto 3.1.3. Muestras sean representativas de cuatro distintas cualidades de ARN: una, muy buena la calidad del ARN (RIN> 9), B, parcialmente degradados ARN (RIN 5-6, tenga en cuenta el pico significativamente menor de 28S ARNr), C, muy degradados ARN (RIN < 3), D, casi totalmente degradadas ARN (RIN = 2). Hemos utilizado con éxito muestras con características similares o superiores a B (RIN> 5.2) para aplicaciones de microarrays de aguas abajo. Puntas de flecha sólida y abierta indicar la posición del 18S y 28S rRNA especies, respectivamente.
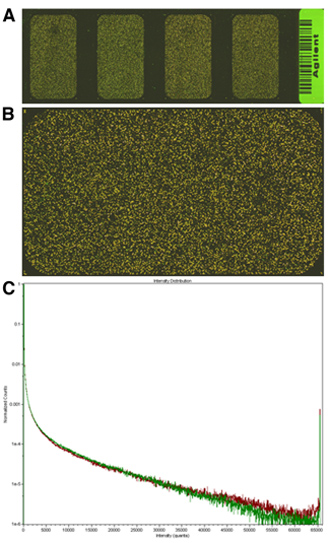
Figura 2. Ejemplos de imágenes de la matriz y los diagramas de dispersión. A ver, conjunto de diapositivas, que muestra las cuatro matrices en el formato 4X44K Agilent, B, escaneo de alta resolución de una de las áreas de la matriz, tenga en cuenta que ninguna de las señales (rojo o verde) es predominante; C, gráfico de dispersión de la imagen en B, tenga en cuenta que las señales se solapan por completo y no más de 1x10 -6 características se encuentran en la intensidad de saturación.
Discusión
Perfiles de expresión con microarrays de ADN proporciona un método sencillo para identificar genes expresados diferencialmente entre las dos muestras biológicas. El éxito de los experimentos de perfiles de expresión requieren alta calidad de las muestras de ARN, el etiquetado y la hibridación robusta. En nuestra experiencia, las matrices comerciales y equipos de etiquetado de Agilent proporcionan datos de alta calidad a un costo razonable. Mientras Agilent también ofrece su hibridación y maquinaria de exploración, estamos a favor del sistema de hibridación de Roche-Nimblegen y el GenePix 4000B escáner de microarrays de Molecular Devices. Tenga en cuenta que la unidad de hibridación Roche-Nimblegen era conocido como el sistema de hibridación MAUI. Después de la reciente adquisición corporativa de Roche, los mezcladores A4 han sido descontinuados. A partir del momento de escribir esto, todavía se pueden encontrar a través de otros vendedores como los diagnósticos Kreatech ( www.kreatech.com ; este sitio web también ofrece una herramienta de amplia compatibilidad conveniente), pero a largo plazo la disponibilidad de stock es incierta. Otros sistemas de hibridación están disponibles (por ejemplo, de Agilent), sin embargo, si mezcladores compatibles se pueden encontrar por la matriz utilizada, se recomienda el sistema de la Roche-NimbleGen por su calidad y reproducibilidad.
Divulgaciones
Agradecimientos
Este trabajo fue apoyado por becas de ED de la Fundación James S. McDonnell 21 Programa de Premios del siglo XXI, y el Premio a la Innovación Nuevo Director del NIH. GAB fue apoyado en parte por una beca postdoctoral del Instituto de Medicina Regenerativa de California.
Materiales
| Name | Company | Catalog Number | Comments |
| 2100 Bioanalyzer | Agilent Technologies | G2943CA | Includes chip priming station, vortexer, software and laptop computer |
| 2100 Bioanalyzer electrophoresis set | Agilent Technologies | G2947CA | |
| RNA 6000 Nano kit | Agilent Technologies | 5067-1511 | includes size ladder, marker, gel matrix, dye, electrode cleaners, spin filters and nanochips |
| RNase Zap | Applied Biosystems | AM9780M | |
| Quick Amp labeling kit, two-color | Agilent Technologies | 5190-0444 | |
| RNeasy mini kit | Qiagen | 74104 | |
| Gene Expression Hybridization Kit | Agilent Technologies | 5188-542 | Includes Blocking agent, Fragmentation buffer and Hybridization buffer |
| Gene Expression Wash Buffer Kit | Agilent Technologies | 5188-5327 | Includes Wash Buffers 1 and 2 and Triton X-102 |
| Hybridization Station | Roche Group | 05223652001 | 4-slide model |
| Hybridization System Accesory Kit | Roche Group | 05327695001 | Contains verification assembly for testing mixing, replacement O-rings, disassembly tool, forceps and brayer |
| A4 mixer hybridization chambers | |||
| Whole Human Genome (4x44) Oligo Microarray | Agilent Technologies | G4112F | |
| Positive displacement pipette | Gilson, Inc. | F148504 | |
| Capillary pistons for positive displacement pipette | Gilson, Inc. | F148415 | |
| Microarray dryer | Nimblegen | 05223636001 | |
| Microarray fluorescent scanner, Axon GenePix 4000B | Molecular Devices | GENEPIX4000B | |
| GenePix Pro 6.0 software | Molecular Devices |
Referencias
- Diaz, E. One Decade Later: What has Gene Expression Profiling Told us About Neuronal Cell Types, Brain Function and Disease. Curr Genomics. 10, 318-318 (2009).
- Barisone, G. A. Expression profiling in Neuroscience, edited by Ioannis Karamanos. , SPRINGER SCIENCE+BUSINESS MEDIA, LLC. New York. (2010).
- Gelder, R. N. V. an Amplified RNA synthesized from limited quantities of heterogeneous cDNA. Proc Natl Acad Sci U S A. 87, 1663-1663 (1990).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados